É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Citometria de fluxo e assassinas naturais celular Lytic Actividade em sangue total humano
Neste Artigo
Resumo
This work describes an advanced workflow for the accurate and fast determination of NK (Natural Killer) cell count and NK cell cytotoxicity in human blood samples.
Resumo
a citotoxicidade das células NK é uma medida utilizada para determinar o efeito de uma intervenção externa sobre a função da célula NK. No entanto, a precisão e reprodutibilidade deste ensaio pode ser considerado instável, quer devido a erros do utilizador ou por causa da sensibilidade das células NK para manipulação experimental. Para eliminar estes problemas, um fluxo de trabalho que eles se reduz a um mínimo foi estabelecida e é apresentada aqui. Para ilustrar, obtivemos amostras de sangue, em vários momentos, desde corredores (n = 4), que foram submetidos a uma intensa sessão de exercício. Primeiro, as células NK são simultaneamente identificados e isolados através de marcação de CD56 e de triagem magnética baseada, directamente a partir de sangue inteiro e de tão pouco como um mililitro. As células NK são ordenados removido de quaisquer anticorpos de reagentes ou de nivelamento. Eles podem ser contados, a fim de estabelecer uma contagem de células NK precisa por mililitro de sangue. Em segundo lugar, as células NK (células efectoras ordenados ou E) pode ser misturado com 3,3'-Diotadecyloxacarbocyanine perclorato (DIO) marcaram células K562 (células alvo ou T) a um ensaio-óptima de 1: 5 razão T: E, e analisadas usando um imaging fluxo-citómetro que permite a visualização de cada evento e a eliminação de qualquer falsa positiva ou falsos negativos (como doublets ou células efetoras). Este fluxo de trabalho pode ser completada em cerca de 4 h, e permite a resultados muito estáveis mesmo quando se trabalha com amostras humanas. Quando disponíveis, as equipes de pesquisa pode testar várias intervenções experimentais em seres humanos, e comparar as medidas em toda a vários pontos de tempo, sem comprometer a integridade dos dados.
Introdução
células assassinas naturais são um elemento essencial do sistema imune inato. Embora eles são muito regulado, eles têm a capacidade de reconhecer e eliminar células anormais através de contacto célula-a-célula e sem activação prévia 1. Como tal, eles formam uma linha rápida de defesa contra as infecções. O exercício, especialmente intensa, tem sido demonstrado para deprimir o sistema imunitário de forma transiente 2, 3, 4, 5. As células NK são particularmente propensos para este efeito 4, 6, 7, de forma eficaz criar uma janela de aumento da sensibilidade à doença. Assim, o estudo de intervenções antes, durante ou após o exercício intenso com o objetivo de reduzir o seu impacto sobre a função das células NK é de particular interesse para o bem-estar dos atletas pós-competições.
No entanto, o estudo de tais intervenções é complicada por vários factores: 1) As células NK são escassas 8, a cerca de 1% do compartimento de células brancas do sangue; 2) As células NK são muito sensíveis ao stress e dependem de exposição constante a condições fisiológicas para permanecer viável e estável no decurso da experimentação; e 3) ensaios padrão para determinar a citotoxicidade das células NK, como Ficoll gradientes 9 e solte ensaios de 10, não são confiáveis e inconsistente. A variabilidade inerente de amostras humanas único composto estas questões. amostras humanas frescas colhidas durante as intervenções são bastante regulamentado e difícil de obter, pelo menos, quando comparado com as amostras de animais ou linhas celulares imortalizadas. Isso reduz as oportunidades para repetir experiências ou adicionar participantes para o estudo de coorte para alcançar limiares estatísticos significativos. Coletivamente, estas questões apoiar a necessidade de um protocolo simplificado que permite foR tanto de alto rendimento e uma análise de alta fiabilidade da actividade lítica de células NK em amostras humanas.
Nós estabelecemos um fluxo de trabalho que encurta o tempo necessário para identificar, isolar e células NK teste de sangue total humano, minimizando a exposição a factores externos. O método de optimizar o uso de dois instrumentos, um separador magnético à base de células e um fluxo de imagens citómetro, e, um T específica optimizado-ensaio: razão E para permitir a detecção das diminuições ou aumentos de citotoxicidade de células NK.
Protocolo
NOTA: Todos os procedimentos de recolha de sangue foram conduzidos de acordo com as diretrizes estabelecidas pela Appalachian State University (ASU) Institutional Review Board (IRB).
1. Recolha de Sangue Total
- Tenha um phlebotomist certificada tirar sangue de acordo com as diretrizes da Organização Mundial de Saúde.
- Tirar sangue em um 4 ml tubo de colheita de sangue contendo ácido Di-Potássio etilenodiaminotetracético (EDTA K 2). Inverta tubo de recolha de sangue de acordo com as instruções do fabricante. Mantenha tubo de coleta de sangue à temperatura ambiente num agitador de bancada até à separação.
2. Preparação de células alvo marcadas com DiO
- Crescer as células K562 em completa de Iscove Modified Meios de Dulbecco (IMDM) suplementado com 10% de soro fetal de bovino (FBS) e 1% de penicilina estreptomicina durante várias semanas, antes do ensaio para assegurar a saúde das células. Ajuste o concentratde iões das células a 3 x 10 5 células / mL diariamente através da realização de uma contagem de células e subsequente passagem de células. Executar uma mudança de meio completo a cada 2 a 3 dias.
- No dia do ensaio, executar uma contagem de células utilizando uma mistura 1: 1 hemocitómetro.
- Retirar 10 mL do frasco K562 e coloque em 1,5 mL tubo.
- Adicionar 10 ul de corante azul de tripano para o mesmo tubo de 1,5 ml para um factor de diluição de 1: 1.
- Agite suavemente o tubo para misturar K562 células e tripan corante azul.
- Permitir K562 corante azul de tripano de células-se a incubar durante 1 minuto à temperatura ambiente.
- Remover 15 mL de células K562 coradas de tubo.
- Pipeta hemocitómetro para contagem de células.
- Contagem de células nas quatro esquinas, bem como o quadrado do meio para um total de cinco quadrados.
- Calcular a contagem de células utilizando a equação:
Total de células / ml = Total de células contadas X (fator de diluição / Número de quadrados) X 10.000 / mL
- Ressuspender as células alvo K562 a uma densidade final de 1 x 10 6 culas / ml em meio de cultura IMDM isento de soro.
- Para células alvo K562 não coradas, adicionar 10 ml de células alvo K562 para um tubo de 15 ml a uma densidade de 1 x 10 6 células / ml para um total de 10 x 10 6 células K562. Colocar em uma incubadora a 37 ° C com 5% de CO 2 até que esteja pronto para uso.
- Para células alvo K562 DiO manchado, adicionam-se 10 ml de células K562 alvo para um tubo de 15 ml a uma densidade de 1 x 10 6 células / ml para um total de 10 x 10 6 células K562.
- Adicionar 1 mL de solução-DiO rotulagem de células por ml de suspensão de células em 15 ml de tubo designada por coloração DiO e suavemente vórtice. Por exemplo, adicionar 10 ul de solução de marcação celular DiO a 10 x 10 6 K562 células / ml num volume de 1 x 10 6 células / ml.
- Incubar solução K562-DiO durante 20 min a 37 ° C com 5% de CO 2 em um tubo de 15 ml.
- Segueing de incubação, adicionar 3 ml de solução salina à temperatura ambiente fosfato tamponada (PBS) a uma solução K562-DiO contendo 15 ml de tubo.
- Centrifugar durante 10 minutos a 135 xg à temperatura ambiente.
- Cuidadosamente remover o sobrenadante sem perturbar o pelete de células com uma pipeta de volume ajustável 1000 ul.
- Adicionar 10 ml de IMDM fresco para pellet contendo células 15 mL tubo.
- Suavemente o tubo de vórtice para ressuspender as células.
- Repita os passos 2.7 a 2.10 por mais duas vezes.
- Células de loja em um 37 ° C incubadora com 5% de CO 2 até que esteja pronto para uso.
NOTA: As células podem ser armazenadas no incubador durante até 24 horas, mas é preferível usá-los no mesmo dia.
3. Preparação de Controles
- Transferir o seguinte em separados e devidamente rotulados tubos de 1,5 ml:
- Adicionar 500 mL de IMDM fresco contendo ressuspenso DIO-rotulados K562 células para o "Double positiva" rotuladas 1,5 tubo mL.
- Adicionar 500 mL de IMDM fresco contendo novamente suspensas células K562 DIO-etiquetados para a "única dio" rotulados 1,5 tubo mL.
- Adicionar 500 mL de IMDM fresco contendo células K562 não marcadas ressuspendidas em a "iodeto de propídio (PI), apenas" marcado tubo 1,5 mL.
- Coloque a ponta positiva Duplo e PI apenas tubos em um 55 ° C banho de água durante 5 minutos.
- Após a 5 min tiver decorrido, remover os tubos e limpe com 70% de etanol.
- Adicionar 10 L de PI ao Duplo positiva e PI apenas 1,5 tubos mL.
- Coloque todos os três controlos de células alvo K562 na incubadora a 37 ° C durante 30 min.
- Após os 30 min de incubação tiver decorrido, centrifugar os três controlos de células alvo K562 durante 2 minutos a 163 x g.
- Cuidadosamente remover o sobrenadante sem perturbar o sedimento celular.
- Ressuspender cada controlo com 20 mL de meio fresco cultura de células IMDM, e deixar na incubadora a 37 ° C com 5% de CO 2 durante pelo menos30 min para o sinal DiO óptima.
NOTA: Os controles são agora pronto para ser executado através do fluxo de imagem citômetro.
4. NK celular separação automatizada
- Ligue separador de células e permitir que o ciclo de start-up para terminar.
- Certifique-se de que todas as garrafas iluminações de fluidos são verdes e que o frasco de resíduos está vazio.
- Obter uma temperatura de 15 mL suporte de tubos quarto.
NOTA: A seleção é baseado em volumes de amostra. Por exemplo, para um volume de menos do que 3 ml de utilizar um suporte de tubos de 15 ml e para um volume de mais de 3 mL usar um suporte para tubos de 50 mL. - tubos de amostra de etiqueta em conformidade (Repita por amostra / participante): Participante 1 Whole Amostra de Sangue; Participante 1 fracção negativa; Participante 1 fracção positiva.
- Gentilmente pipeta 1,5 ml de sangue total a partir do Passo 1.2 em "Whole Amostra de Sangue" tubo de 15 mL.
- Coloque 15 ml tubo adequadamente rotulado como "Whole Amostra de Sangue" do Passo 4.5 e rotulado 15 mL "fracção negativa" e"fracção positiva" tubos de Passo 4.4 no suporte de tubos. Utilize suporte de amostras thefollowing set-up: Fileira (R1) A: Whole Amostra de Sangue, Row (R2) B: fração magneticamente marcados positiva,: Negativo, fração não marcado, Row (R4) C.
- Insira o rack separador para o MiniSampler para autolabeling.
- Seleccionar "Reagente" no menu e realçar a posição em que o frasco vai ser colocado na prateleira de separação.
- Selecione "Ler Reagente" para activar o leitor de código 2D.
- Colocar o frasco do reagente apropriado em frente do leitor de código de 2D entre 0,5-2,5 cm da tampa do leitor de código.
NOTA: Por exemplo, o reagente necessário para a separação de células NK é CD56. - Segure frasco de reagente em um ângulo na frente do leitor de código 2D para leitura ideal.
- Colocar o frasco de reagente para a posição adequada cremalheira separador.
- Destacar as posições desejadas na aba Separação na tela.
- No submenu Labeling, atribuir um auprograma tolabeling.
- Atribuir a célula de separação MicroBeads Reagente CD56 para acumular posições 1, 2, 3 e 4.
- Selecione o programa de separação "posselwb".
- Selecione a opção "lavar" programa de lavagem.
- Inserir um volume de amostra de 1.500 mL no submenu "Volume" usando o teclado numérico.
- Selecione a opção "Enter" no teclado.
- Selecione "Executar" para iniciar a separação de células.
- Selecione "OK" para confirmar que tampão suficiente disponível para o processamento de todas as amostras.
5. Contagem de Células NK Separação Após celular
- Imediatamente após a separação de células com o separador de células, recuperar a fração positiva. Deixe em temperatura ambiente. Esta fracção contém a população de células NK puro desejado.
- Para cada amostra individual, executar uma contagem de células utilizando um hemocitómetro, como por Etapa 2.2.
- Após os cálculos, registre a contagem de células.
6. Ensaio de Citotoxicidade Preparação da Amostra
- Preparar e rotular tubos de 1,5 ml para cada amostra / participante em conformidade.
- Pipeta desejada proporção de células NK e células K562 DIO-rotulados para cada tubo.
NOTA: Por exemplo, a proporção desejada de células alvo K562 e células efectoras NK é de 1: 5. - Centrifugar durante 5 minutos a 135 x g.
- Cuidadosamente remover o sobrenadante sem perturbar o sedimento celular.
- Ressuspender NK-DiO marcado mistura de células K562 em 500? L de meio celular NK sem interleucina-2 (IL-2) e 2-mercaptoetanol (2-ME) (incompleta meios de cultura de células NK).
NOTA: O meio de cultura de células NK é incompleta Meio Essencial Mínimo de Eagle com bicarbonato de sódio, sem L-glutamina, ribonucleósidos e desoxiribonucleósidos. - Adicionar 5 mL de PI a cada tubo.
- Centrifugar durante 2 minutos a 163 x g.
- Incubar as células a 37 ° C durante 2 h.
- Após 2 h de incubação, centrifuGE durante 2 minutos a 163 x g.
- Cuidadosamente remover o sobrenadante sem perturbar o sedimento celular.
- Ressuspender as células em 25 ul incompleta meios de cultura celular NK.
7. Preparação de espontânea ( "S") Amostra
- Pipetar 500 ul de células K562 DIO-marcados (concentração de 1 x 10 6 células / ml) no tubo de 1,5 mL.
- Adicionar 10 ul de PI de cada um dos tubos.
- tubo de centrifugação durante 2 minutos a 163 x g.
- Incubar as células a 37 ° C durante 2 h.
- Após 2 h de incubação, centrifuga-se durante 2 min a 163 x g.
- Cuidadosamente remover o sobrenadante sem perturbar o sedimento celular.
- Ressuspender as células em 25 ul incompleta Meio Essencial (MEM-α) Meios de cultura de células alfa-mínimos.
8. Aquisição de Dados com imagens de fluxo Citómetro
- Pressione o botão verde no interior da porta da frente da imagem de citometria de fluxo para ligar o instrumento.
- Ligue all computadores associados com a imagiologia de fluxo FACSCalibur.
- Inicie o fluxo de imagem citômetro software.
- Clique no botão "Iniciar" para limpar o sistema e preparar a linha de amostra.
- Uma vez que o "Startup" estiver concluída, feche a janela "calibrações".
- Atribuir canais: no lado superior esquerdo, clique em cada canal, a fim de atribuir-lhes.
- No lado direito, clique no botão gráfico de dispersão para criar 4 scatterplots: Raw Max Pixel _MC_6 vs Area_M06, Raw Max Pixel _MC_2 vs Area_M02, Raw Max Pixel _MC_5 vs Area_M05 e FieldArea_M01 vs AspectRatio_M01.
- Comece analisando amostras usando primeiro o controle "Double positiva".
- Clique em "Load".
- Coloque o tubo de 1,5 ml com a amostra "Double positiva" de Passos 3,4 para 3,9 no carregador de amostra.
- Selecione o objetivo 40X sob a guia "Ampliação".
- Ligue a 405 mW e642 lasers MW.
- Ligue o canal "Brightfield".
- Clique em "Selecionar intensidade."
- Com base na amostra de controlo "Duplo positiva", determinar a intensidade desejada para o laser de 405 mW de modo que o detector não está sobrecarregado.
Nota: Por exemplo, a intensidade ideal para este experimento foi fixado em 11 mW.
- Após a desejada set-up é conseguido, adquirir dados.
- Sob a guia "Aquisição Arquivo", digite em um texto nome personalizado. Selecione uma pasta para salvar o arquivo (s) de dados.
- Digite o número de células de adquirir ao lado de "Collect". Tipicamente, este número varia entre 1.000 a 10.000.
- Clique em "Adquirir".
NOTA: Uma vez que o número desejado de células é adquirido, o arquivo de dados é salvo automaticamente na pasta selecionada anteriormente.
- Após a aquisição for concluída, carregar a amostra de controlo seguinte - DiO único controle.
- Clique em "Load".
- Coloque o tubo de 1,5 mL com o "único dio" amostra para o carregador de amostra.
- Deixar o objetivo 40X sob a guia "Ampliação" selecionado.
- Deixar o laser 405 mW ligado.
- Desligue a mW laser de 642 e o canal "Brightfield".
NOTA: Agora que a desejada configuração foi alcançado, os dados podem ser adquiridos. - Sob a guia "Arquivo Aquisição", digite em um texto nome personalizado e escolha uma pasta para salvar o arquivo (s) de dados. Digite o número de células de adquirir ao lado de "Collect.". Normalmente este número é de 1.000.
- Clique em "Adquirir".
NOTA: Uma vez que o número desejado de células é adquirido o arquivo de dados é salvo automaticamente na pasta selecionada anteriormente.
- Repita o passo 8,10 para o "único PI" amostra de controlo. As amostras experimentais estão agora prontos para serem recolhidos.
- Lidar com as remaining amostras experimentais, incluindo as "amostras" S "espontânea" da seguinte forma:
- Deixar o objetivo 40X sob a guia "Ampliação" selecionado.
- Ligue a 405 mW e 642 lasers MW.
- Ligue o canal "Brightfield".
- Clique em "Intensidade Set."
- Sob a guia "Arquivo Aquisição", digite em um texto nome personalizado e escolha uma pasta para salvar o arquivo (s) de dados. Digite em um texto nome personalizado.
- Digite o número de células de adquirir ao lado de "Recolha".
- Clique no botão "Acquire".
NOTA: Uma vez que o número desejado de células é adquirido o arquivo de dados é salvo automaticamente na pasta selecionada anteriormente.
- Repita o passo 8,12 para todas as amostras experimentais.
- Depois de todos os dados experimentais e arquivos foram recolhidos, clique no botão "Shutdown" para esterilizar o sistema.
9. ImaginAnálise de amostras g Citómetro de fluxo
- Abra o fluxo de imagiologia citómetro de aplicação de software de análise.
- Em "Arquivo", abra um arquivo .RIF experimental.
- Construir uma nova matriz usando os arquivos .RIF única cor (OID somente controlar e controle somente-PI, criado durante as etapas de 8.10 e 8.11), selecionando "Criar um novo Matrix" sob a guia de Compensação no fluxo de imagens citômetro software.
NOTA: O software irá pedir para a seleção dos arquivos de uma só cor e fundi-los para criar um arquivo de matriz (extensão de arquivo .ctm), que está a ser selecionado para aplicar a compensação de canal. - Criar gráficos de pontos utilizando a função de "blocos de construção" do software.
- Criar um ponto-plot (BrightFieldGradient_RMS vs frequência) para a porta do células focalizados. Ligue para o portão "Focus" (Figura 1A).
- Usando o portão "Focus", criar um gráfico de pontos de Bright Área Campo vs Campo brilhante proporção para g comeu as células singlet. Ligue para o portão "Single" (Figura 1B).
- Usando o portão "Single", criar um gráfico de pontos de Intensity_MC_Ch02 vs Intensity_MC_Ch5. Utilize este plano para identificar e células dupla positivos (metas, mortos) gate DIO-positiva apenas células (alvos, vivo) e PI-DiO (Figura 1C).
NOTA: Todas as parcelas descrito nos passos 9.4.1, 9.4.2 e 9.4.3 podem ser criados usando a função de "blocos de construção" do software.
- Clique na função de estatísticas do gráfico de pontos para acessar os números de células de cada portão.
- Calcula-se a percentagem de alvos na amostra mortas espontânea e amostras experimentais utilizando a seguinte fórmula:
% alvos mortos na amostra = (alvos #dead x 100) / (# viver alvos + #dead alvos) - Calcular a citotoxicidade utilizando a seguinte fórmula:
% Citotoxicidade = [(Experimental mortos mortos-espontânea) / (morto 100 espontânea)] x100
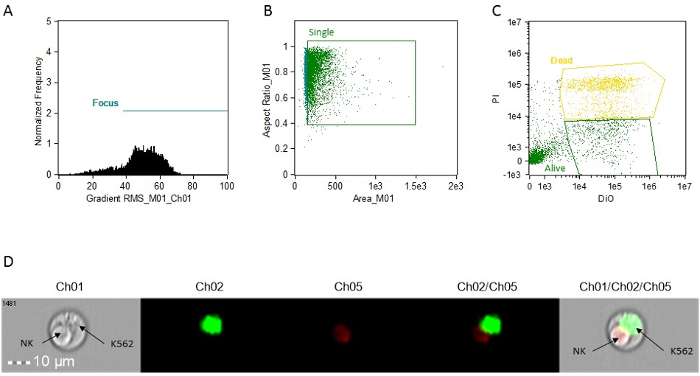
Figura 1: histogramas representativos, gráficos de dispersão e imagens para análise da atividade citotóxica. Análise (A) células foco. (B) Análise única célula. Análise (C) coloração das células alvo. Todas as determinações são feitas usando a imagem anexada para cada evento. Isso pode ser acessado no software de análise, basta clicar sobre o evento nos gráficos. (D) imagem representativa de um evento de gibão, mostrando uma célula NK apoptótica e um alvo K562 ao vivo. Ch01, Brightfield. CH02, a DIO. CH05, PI. Por favor clique aqui para ver uma versão maior desta figura.
Resultados
Determinação do número de células NK
O efeito de funcionamento pesado na contagem de células NK no sangue total foi medida, utilizando o protocolo de exercício descrito na Figura 2. Amostras de sangue foram colhidas antes do exercício, imediatamente após o exercício, 1,5 h após o exercício, e, finalmente, 24 e 48 h após a coleta de sangue inicial. A concentração de células NK por mililitro de sangue total foi medido para cada corredor (Figur...
Discussão
O método descrito neste estudo mede directamente a actividade específica de células NK de um indivíduo em resposta a estímulos (neste caso particular, o exercício prolongado). Tipicamente, as células NK são isolados a partir de um do sangue utilizando um gradiente de densidade ou de triagem de células, usando uma combinação de marcadores. Embora estes métodos sejam largamente usadas, elas têm muitas desvantagens: eles são morosos, envolvem manipulações múltiplas, e dependem fortemente do usuário. Como ...
Divulgações
The authors have nothing to disclose.
Agradecimentos
This project was supported by Agriculture and Food Research Initiative Competitive Grant no. 2100-68003-30395 from the USDA National Institute of Food and Agriculture.
Materiais
| Name | Company | Catalog Number | Comments |
| K-562 lymphoblasts | ATCC | CCL-243 | |
| Iscove's Modified Dulbecco's Media | ATCC | 30-2005 | High glucose, with L-Glutamine, with HEPES, Sterile-filtered |
| Alpha Minimum Essential medium | ATCC | CRL-2407 | Without ribonucleosides and deoxyribonucleosides but with 2 mM L-glutamine and 1.5 g/L sodium bicarbonate |
| Trypan Blue Solution 0.4% | Amresco | K940-100ML | Tissue culture grade |
| Propridium Iodide Staining Solution | BD Pharmingen | 51-66211E | |
| Vybranto DiO cell-labeling solution | Vybranto | V-22886 | |
| autoMACS Pro Separator | Miltenyi Biotec | 130-092-545 | |
| autoMACS Running Buffer | Miltenyi Biotec | 130-091-221 | |
| autoMACS Washing Buffer | Miltenyi Biotec | 130-092-987 | |
| autoMACS Columns | Miltenyi Biotec | 130-021-101 | |
| Whole Blood CD56 MicroBeads, human | Miltenyi Biotec | 130-090-875 | |
| ImageStream X Mark II Imaging Flow Cytometer | EMD Millipore | ||
| Speedbeads | Amnis Corporation | 400030 | |
| 0.4-0.7% Hypochlorite (Sterilizer) | VWR | JT9416-1 | |
| Coulter Clenz | Beckman Coulter | 8546929 | |
| 70% Isopropanol (Debubbler) | EMD Millipore | 1.3704 | |
| D-PBS (Sheath fluid) | EMD Millipore | BSS-1006-B (1X) | No calcium or magnesium |
| INSPIRE Software | EMD Millipore | Version Mark II, September 2013 | |
| Ideas Application Software | EMD Millipore | Version 6.1, July 2014 |
Referências
- Cerwenka, A., Lanier, L. L. Natural killer cells, viruses and cancer. Nat Rev Immunol. 1 (1), 41-49 (2001).
- Nieman, D. C. Exercise, infection, and immunity. Int J Sports Med. 15, 131-141 (1994).
- Romeo, J., Warnberg, J., Pozo, T., Marcos, A. Physical activity, immunity and infection. Proc Nutr Soc. 69 (3), 390-399 (2010).
- Nieman, D. C. Marathon training and immune function. Sports Med. 37 (4-5), 412-415 (2007).
- Simpson, R. J., Kunz, H., Agha, N., Graff, R. Exercise and the Regulation of Immune Functions. Prog Mol Biol Transl Sci. 135, 355-380 (2015).
- Walsh, N. P., et al. Position statement. Part one: Immune function and exercise. Exerc Immunol Rev. 17, 6-63 (2011).
- Timmons, B. W., Cieslak, T. Human natural killer cell subsets and acute exercise: a brief review. Exerc Immunol Rev. 14, 8-23 (2008).
- Westermann, J., Pabst, R. Distribution of lymphocyte subsets and natural killer cells in the human body. Clin Investig. 70 (7), 539-544 (1992).
- Boyum, A. Isolation of leucocytes from human blood. Further observations. Methylcellulose, dextran, and ficoll as erythrocyteaggregating agents. Scand J Clin Lab Invest Suppl. 97, 31-50 (1968).
- McMillan, R., Scott, J. L. Leukocyte labeling with 51-Chromium. I. Technic and results in normal subjects. Blood. 32 (5), 738-754 (1968).
- Berk, L. S., et al. The effect of long endurance running on natural killer cells in marathoners. Med Sci Sports Exerc. 22 (2), 207-212 (1990).
- McAnulty, L. S., et al. Effect of blueberry ingestion on natural killer cell counts, oxidative stress, and inflammation prior to and after 2.5 h of running. Appl Physiol Nutr Metab. 36 (6), 976-984 (2011).
- Millard, A. L., et al. Brief Exercise Increases Peripheral Blood NK Cell Counts without Immediate Functional Changes, but Impairs their Responses to ex vivo Stimulation. Front Immunol. 4, 125 (2013).
- Janeway, C. A., Travers, P., Walport, M., Shlomchik, M. J. . Immunobiology: The Immune System in Health and Disease. 5th edn. , (2001).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados