É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Projeto Experimental de Laser Microdissecção RNA-Seq: Lições de uma análise do desenvolvimento foliar do milho
Neste Artigo
Resumo
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Resumo
Genes com funções importantes no desenvolvimento têm frequentemente padrões de expressão espacial e / ou temporalmente limitados. Muitas vezes, estes transcritos do gene não são detectados ou não são identificados como diferencialmente expressos (DE) em análises transcriptomic de órgãos vegetais inteiros. Laser Microdissecção RNA-Seq (LM RNA-Seq) é uma ferramenta poderosa para identificar genes que são DE em domínios específicos de desenvolvimento. No entanto, a escolha de domínios celulares para microdissect e comparar, e a precisão dos microdissecações são cruciais para o sucesso dos experimentos. Aqui, dois exemplos ilustram as considerações de design para experiências transcriptômica; LM uma análise de RNA-Seq para identificar genes que são dé ao longo do eixo proximal-distal folha de milho, e uma segunda experiência para identificar genes que estão em liguleless1 DE-R (LG1-R) mutantes em comparação com o tipo selvagem. Os principais elementos que contribuíram para o sucesso desses experimentos foram detalhados histológica e em siTU hibridação análises da região a ser analisada, selecção de primórdios foliares nas fases de desenvolvimento equivalentes, a utilização de pontos de referência morfológicas para seleccionar regiões de microdissecação, e microdissecação de domínios precisamente medidos. Este documento proporciona um protocolo detalhado para a análise de domínios de desenvolvimento por LM RNA-Seq. Os dados aqui apresentados ilustram como a região selecionada para microdissection irá afetar os resultados obtidos.
Introdução
A folha de milho é um modelo ideal para estudar a formação de campos de desenvolvimento durante a morfogénese, uma vez que tem uma fronteira distinta entre a lâmina e a bainha que é passível de dissecção genética (Figura 1A). Durante as fases iniciais do desenvolvimento foliar, uma faixa linear de células menores, a banda preligule (PLB), subdivide o primórdio foliar em domínios pré-lâmina e pré-bainha. A ligule franja-like e aurículas triangulares desenvolver a partir da PLB (Figura 1A, C, D). telas genéticas identificaram mutações que perturbam o limite lâmina de bainha. Por exemplo, liguleless1 recessivo (LG1) mutações excluir o ligule e aurículas 1, 2, 3, 4 (Figura 1B). A hibridação in situ revelou que LG1 transcrição acumula no PLB e ligule emergentes, tornando-se um excelente marcador para o desenvolvimento ligule 5, 6 (Figura 1E).
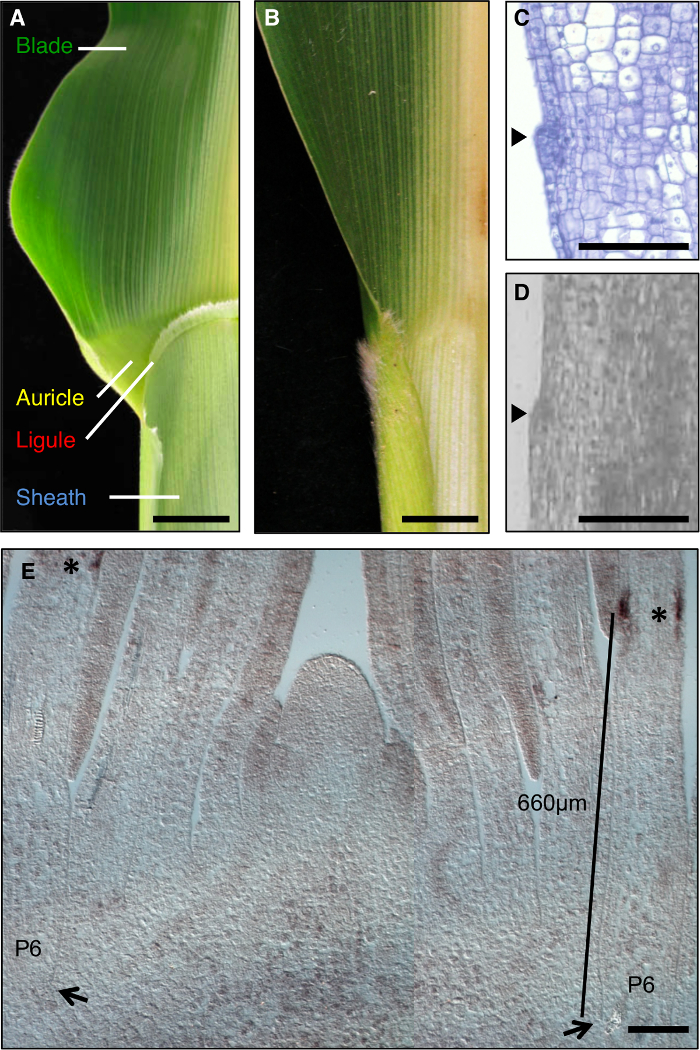
Figura 1: Tipo selvagem e folhas de milho liguleless1-R. (A) a região Blade-bainha limite da folha de tipo selvagem maduro mostrando estruturas ligule e aurícula. (B) a região Blade-bainha limite da maturidade liguleless1-R folha que mostra ausência de estruturas ligule e aurícula. Folhas em A e B foram cortadas pela metade ao longo da nervura central. (C) longitudinal secção através primórdio foliar de tipo selvagem. Amostra foi processada e corados para análise histológica. A lígula iniciadora é evidente como uma colisão que sobressai a partir do plano da folha (ponta de seta). (D) seita Longitudinalíon através primórdio folha de tipo selvagem. Amostra foi processada por LM como descrito no texto. Arrowhead indica iniciar ligule. (E) lg1 hibridização in situ de corte longitudinal lateral do ápice filmagem. Os asteriscos indicam acumulação transcrição lg1 na PLB do primórdio foliar P6. As setas indicam a base do P6 primórdio. A barra indica a medição a partir da base de o primórdio da PLB. As barras de escala em A e B = 20 mm. As barras de escala em Ce = 100 uM. Esta figura foi modificado a partir da referência 6 (Copyright: Sociedade Americana de biólogos da planta). Por favor clique aqui para ver uma versão maior desta figura.
Neste estudo, LM RNA-Seq foi empregue para identificar um conjunto de genes que são expressos diferencialmente (DE) para a lâmina de bainha limite em relação a outras partes do primórdio foliar e de IDE genes ntify que estão DE em mutantes lg1-R em relação aos irmãos de tipo selvagem. LM RNA-Seq é um método de quantificação de acumulação de transcrição em células específicas ou domínios celulares 7. sistemas lineares combinar um laser e um microscópio com uma câmera digital. tecido é seccionado montadas em lâminas e visualizaram pelo microscópio. O software LM normalmente inclui ferramentas de desenho que permitem ao usuário para delinear qualquer região selecionada para microdissection. Os cortes a laser ao longo da linha, e o tecido selecionado é catapultado para fora da lâmina e em um tubo suspenso acima do slide. LM permite que o usuário microdissect domínios precisos, incluindo camadas de células específicas e até mesmo células individuais 8, 9. ARN pode então ser extraído a partir do tecido microdissecados. Subsequentemente, o componente de RNA-Seq utiliza a próxima geração de sequenciação para sequenciar bibliotecas de cDNA geradas a partir do RNA extraído 10,= "xref"> 11.
Principais vantagens do LM RNA-seq são a capacidade de quantificar a acumulação de transcrição em domínios bem definidos ea capacidade ao perfil todo o transcriptoma simultaneamente 7. A técnica é particularmente adequada para sondagem eventos precoces de desenvolvimento, onde a região de interesse é muitas vezes microscópica. Estudos anteriores têm utilizado LM combinado com a tecnologia de microarrays para estudar processos de desenvolvimento em plantas 9, 12, 13. RNA-Seq tem a vantagem de quantificar transcritos de uma vasta gama dinâmica, incluindo os genes de baixo-expresso, e informação da sequência anterior não é necessária 10, 11. Além disso, LM RNA-Seq tem o potencial para destacar genes desenvolvente importantes que podem ser perdidas em telas de mutagénese devido à redundância genética ou a letalidade da perda de de-função mutante.
Genes Developmentally importantes, como sheath1 estreita (NS1) e em forma de taça cotyledon2 (CUC2), muitas vezes têm padrões de expressão específicas de apenas uma ou algumas células 17, 18, 19, 20. Muitos são expressos apenas durante as fases iniciais de desenvolvimento e não no órgão maduro. Quando órgãos inteiros ou grandes domínios são analisados, estes transcritos específicos de células são diluídas e não pode ser detectada em análises mais convencionais. Ao permitir análises de domínios bem definidos, LM RNA-Seq permite que estes genes específicos de tecido a ser identificados e quantificados.
factores cruciais para o sucesso das experiências descritas aqui foram uma análise histológica minuciosa que orientou a seleção do estágio de desenvolvimento apropriado e de domínio para análise e MeasureMe precisant de domínios de tecido celular para LM. Para assegurar que os domínios equivalentes foram amostrados de todas as repetições, o tecido foi recolhido a partir de primórdios foliares, ao mesmo estágio de desenvolvimento e os domínios microdissecadas foram medidos em relação a pontos de referência morfológicas, tais como a lígula emergente (Figura 2). Sabe-se que alguns genes são expressos em um gradiente a partir da ponta para a base da folha. Ao medir domínios precisos, devido à variação de amostragem a partir de diferentes locais ao longo do eixo proximal-distal folha foi mantido a um mínimo (Figura 3A). Por microdissecting domínios do mesmo tamanho, devido a variação diferencial de diluição de transcritos específicos de células também foi reduzida (Figura 3B). secções laterais longitudinais do ápice filmagem foram usadas para todos os microdissecações. Estes são secções que são perpendiculares ao eixo nervura central-margem (Figura 4). Usando apenas as seções que incluem a SAM assegura que as regiões laterais equivalentes deprimórdios foliares são analisados.
Em amostras processadas e seccionados para LM, o primeiro sinal morfológico de crescimento ligule é um inchaço no lado adaxial devido a divisões celulares periclinais na epiderme adaxial (Figura 1D, Figura 2). Determinou-se que o ligule emergentes poderiam ser identificados de forma confiável no plastocrono 7 primórdios foliares palco. Nós estávamos interessados em genes expressos em toda a região ligule, incluindo a ligule emergentes e as células imediatamente distais que formarão a aurícula. A fim de assegurar que as selecções de tecido equivalente foram feitas, a colisão lígula foi usada como um ponto de referência morfológica e um rectângulo de 100 um centrada na protuberância lígula foi seleccionado para LM (Figura 2A, 2B). rectângulos de dimensão equivalente de pré-lâmina e pré-bainha foram seleccionados a partir dos mesmos primórdios foliares.
As análises de plantas mutantes liguleless apresentada uma diferente challeESL; lg1-R mutantes não formam um ligule, portanto, esse recurso morfológica não poderia ser usado para selecionar a região de LM. Em vez disso, foi determinado o domínio de transcrição acumulação LG1 em primórdios foliares de tipo selvagem, e uma região que abrange este domínio foi definido. Estas análises preliminares foram realizadas em plântulas a partir do mesmo plantação como foram usadas para a análise final, uma vez que o trabalho anterior demonstrou que a localização do PLB varia dependendo das condições de crescimento. A hibridação in situ indicaram que os transcritos LG1 acumulam no PLB de P6 primórdios foliares (Figura 1E). Nós seleccionado um domínio de 400-900 ^ M a partir da base do primórdios foliares, que englobava o domínio de expressão LG1 (rectângulos roxo, Figura 2A) e capturou estas regiões equivalentes do de tipo selvagem e plantas LG1-R. Para minimizar a variação de fundo e de crescimento genéticos condições quando se comparam transcripforam usadas acumulação t em LG1-R e plantas de tipo selvagem, segregando famílias de mutantes e de tipo selvagem irmãos.
Protocolo
NOTA: Fix tecidos para análise histológica, ao mesmo tempo que o tecido é fixado por LM. Examinar secções coradas para características morfológicas que guiarão depois LM. Ao comparar mutante com o tipo selvagem, efectuar hibridização in situ ou imunolocalização de definir o domínio em que o gene de interesse é expresso (neste caso LG1).
1. fixação do tecido e Processamento
- Crescer apartamentos de plântulas de milho com duas semanas de idade, sob condições padrão 6.
- Dissecar ápices vegetativos para as secções laterais (Figura 4).
- mudas Excise logo abaixo da linha do solo.
- Usando uma lâmina de barbear, remover fatias finas a partir da base da haste (cortes 1, Figura 4A) até uma oval de colmo circundado por uma ou duas folhas maduras é visível (Figura 4B).
- Faça outro corte aproximadamente 10 mm acima da base (cortar 2, Figura 4A).Este segmento 10 milímetros irá conter o SAM e primórdios de folhas jovens.
- Vire o segmento de 10 mm, de forma a base está voltada para cima. Fazer dois cortes paralelos ao eixo lateral de modo a que um pedaço de tecido de 2-3 mm de espessura, é obtido (cortes 3 e 4, Figura 4B). Descartar o exterior duas partes e manter a fatia central para fixação e incorporação.
NOTA: folhas exteriores podem ser aparadas e descartado.
- Fixar o tecido e o processo para a incorporação.
- Assegurar que todos os materiais a serem utilizados nos passos subsequentes são realizados a RNase livre. Tratar soluções com pirocarbonato de dietilo (DEPC) (1 ml de DEPC por litro de solução. Incubar durante a noite com agitação ocasional, e autoclave). Coza artigos de vidro em um forno a 200 ° C ou superior durante pelo menos 6 horas e tratar artigos de plástico com uma solução de descontaminação de RNase.
- Dia 1: Imergir fatias de tecido em ~ 10 ml de solução de Farmer (3: 1 etanol: ácido acético) em frasco de vidro em gelo. Depois de todas as amostras foram dissecados, aplicar vacuum para remover as bolhas de ar e de penetração ajuda de fixador. Mantenha sob vácuo, durante 10 min, em seguida, liberar o vácuo lentamente. Substituir fixador e incubar a 4 ° C durante a noite com agitação suave.
- Dia 2: Incubar à seguinte série de soluções, ~ 10 ml cada, 1 h cada, todos com agitação suave; 85% de etanol a 4 ° C, 95% de etanol a 4 ° C, 100% de etanol a 4 ° C, 100% de etanol a 4 ° C, 100% de etanol a 4 ° C, 1: 1 de etanol: xilenos, à temperatura ambiente, 100% xilenos, à temperatura ambiente, 100% xilenos, à temperatura ambiente.
NOTA: xilenos são tóxicas por contato e inalação. Trabalhar em um exaustor e usar luvas apropriadas. - Adicionar tecido inclusão em parafina peletes médias para aproximadamente metade do volume de xilenos e incubar durante a noite à temperatura ambiente com agitação suave.
- Dia 3: Transferir o frasco a 60 ° C forno até derreter os peletes. Decantar solução e substituir por meio de incorporação de tecido derretido fresco. Mudar o meio mais duas vezesdurante o dia.
- Dia 4: Alteração do tecido de meio de inclusão, uma vez de manhã. Voltar para 60 ° C forno até tarde.
- blocos elenco
- Coloque moldes de incorporação na placa quente de estação de incorporação de tecidos. Utilize uma pinça para transferir as amostras de tecido para os moldes embutindo com a superfície de corte voltada para baixo. Encher o molde com parafina derretida e colocar o anel de incorporação na parte superior do molde. Transfira para um prato fria até parafina tem solidificado. Armazenar os blocos de parafina, a 4 ° C num recipiente hermético com gel de sílica.
2. corte e Deslize Preparação
- Cortar 10 mm seções em um micrótomo 25.
- Examinar fitas e escolha seções medianas. secções medianos são aqueles que incluem o SAM, que aparece como uma cúpula de células rodeado por primórdios foliares.
- seções de montagem em lâminas.
- Colocar as lâminas que são adequados para LM (ou RNase livre oucozido) em 42 ° C deslizar mais quente e aplique algumas gotas de solução de etanol a 50% para cobrir o slide.
- Flutuar seções sobre solução de etanol até que as secções têm expandido.
NOTA: Flutuante seções sobre solução de etanol em vez de água mantém RNA em um estado precipitado reduzir a degradação do RNA. - Inclinação lâmina e remover a solução de etanol em excesso por aspiração com uma pipeta de transferência descartável. Use toalhetes sem fiapos para afastar qualquer solução de etanol adicional.
- lâminas seco a 42 ° C durante várias horas ou durante a noite. Loja desliza a 4 ° C num recipiente hermético com gel de sílica.
- Desparafinar desliza no dia da utilização.
- Prepare três frascos de vidro de Coplin contendo; 100% xilenos (xilenos I), 100% xilenos (xilenos II) e 100% de etanol (~ 50 mL de cada solução).
- Usando fórceps limpas para transferir lâminas, mergulhe as lâminas em xilenos I durante 2 min, xilenos II durante 2 min, e 100% de etanol durante 1 min.
- dreno deslizoues sobre toalhetes sem fiapos e secar ao ar à temperatura ambiente.
3. Microdissecção de Blade, lígula e bainha Amostras de plastocrono 7 Folha Primordia
- Garantir os slides no palco do microscópio LM. Use cinco ou seis lâminas para cada repetição, utilizando cinco secções por slide.
NOTA: Tissue utilização comum para uma única réplica é ilustrada na Figura 5. - Examinar as lâminas e identificar as cinco seções mais medianos em cada slide, utilizando o SAM ápice como o ponto de referência central.
NOTA: Este pode ser feito a baixa ampliação, geralmente um objectivo 5X é suficiente. - Usando 10X ou 20X objectivo, identificar a posição do lígula no plastocrono 7 primórdio foliar de cada secção. O ligule será visível como um galo que sobressai da superfície adaxial do primórdio foliar. Marque esta posição usando a ferramenta de desenho do software LM; selecione o ícone do lápis, mova o cursor para o positi apropriadaon e clique e arraste o mouse para desenhar.
NOTA: A 20X objetiva de 10X ou é apropriado para este e os passos subsequentes. Ao utilizar secções laterais, os dois lados de cada primórdio foliar estará presente em cada secção (Figura 2A). - Usando a ferramenta régua e a ferramenta de desenho retangular, mede 100 mm altas retângulos centradas no ligule de cada seção (retângulos vermelhos, Figura 2a, 2b). Estes serão a amostra "Lígula".
- Para usar a ferramenta régua; selecione o ícone de régua, mova o cursor para uma das extremidades do objeto a ser medido, clique e arraste para medir o objeto. O comprimento da régua será mostrado na tela.
- Para desenhar um retângulo; selecione o ícone do retângulo, mova o cursor para um ponto que será um canto do retângulo, clique e arraste para desenhar um retângulo do tamanho apropriado. Como alternativa, selecione a ferramenta de desenho linear e desenhar quatro linhas retas.
- medida 100 uM rectângulos posicionado 50 mm acima e abaixo do rectângulo "Lígula".
NOTA: Estes serão os "Blade" e amostras "Bainha", respectivamente (verde e azul retângulos, Figura 2a, 2b). Com base em nossos dados histológicos, um retângulo mm 100 abrange a região ligule inteiro. tamanho porções equivalentes de lâmina e bainha foram escolhidos para garantir que quantidades semelhantes de tecidos foram coletadas para cada um. Os espaçadores de 50 pM foram usados para garantir que nenhum tecido região lígula inadvertidamente é incluído nas microdissecações lâmina ou bainha.
- Microdissect medida retângulos (Figura 2D - 2F) 7, 8, 9, coletando amostras Lígula, lâminas e bainhas em tubos separados. Utilizar a função de corte a laser para cortar através da secção de tecido ao longo do contorno do domínio seleccionado. Use o catafunção pult para impulsionar o rectângulo de tecido da lâmina e para a tampa do tubo (Figura 2D - 2F).
4. Microdissecção de Blade, lígula e Bainha adaxial da epiderme Amostras de plastocrono 7 Folha Primordia
- Selecionar seções e usar a ferramenta régua para medir 100 mm elevados segmentos centrados no ligule plastocrono 7, como descrito na seção 3 (acima).
- Selecione apenas as células da epiderme adaxial de cada 100 mm alta "Blade" e "bainha" segmento (verde e azul seleções, figura 2C), descrevendo com a ferramenta de desenho. A epiderme é a camada celular externa; o lado adaxial é o mais próximo do SAM.
- Para a amostra "Lígula", selecionar apenas as células da colisão ligule emergindo como descrito na Seção 3.3 (seleção vermelho, Figura 2C).
- regiões Microdissect selecionada, recolhendo Blade, Lígula e Sheath epiderme amostras em tubos separados, como descrito na seção 3.5.
5. Microdissecção de plastocrono 6 Folha Primordia de lg1-R e do tipo selvagem Irmãos
- Crescer segregando famílias de mutante (lg1-R) e as plantas do tipo selvagem.
- Fix e filmagem processo ápices para LM, conforme descrito nas seções 1,2-1,4. Fix do tipo selvagem e ápices vegetativos mutantes em frascos separados, a fim de mantê-los separados. As amostras da mesma plantação deve ser fixados e processados para hibridação in situ.
- Determinar em que LG1 é transcrito em irmãos de tipo selvagem, através da realização de LG1 a hibridação in situ de 6, 26, 27. Posição de acumulação de transcrição lg1 de base do primórdio da folha em várias amostras (Figura 1E) medir.
- Com base em dados de hibridização in situ, escolha porção do primo folhardium que engloba a região em que LG1 é transcrito. Neste caso, 400-900 ^ M a partir da base de plastocrono 6 primórdios foliares (rectângulos roxo, Figura 2A).
- Microdissect selecionada parte da primórdios foliares, como descrito na seção 3.5, recolhendo lg1-R e as amostras de tipo selvagem em tubos separados.
6. Aplique RNA Tampão de Extracção
- Aplicar 50 ul de tampão de extração de RNA para o tecido microdissecção e prosseguir com a extração de RNA. Continue com a extração de RNA, amplificação RNA, construção de biblioteca, sequenciamento e análise bioinformática como descrito na referência 6.
Resultados
Usando o esquema LM delineado na Figura 2, a cerca de 1,000,000-1,500,000 mm 2 de tecido foi recolhida para cada replicar no todo-cell-camadas LM (Figura 5), e 200.000 mm 2 por replicar para a epiderme adaxial LM. Aproximadamente 2 uM 2.500.000 de tecido foi colhido para cada replicar no LM de LG1-R e primórdios foliares de tipo selvagem. Duas rondas de amplificação de ARN lineares produziram quantidades de mi...
Discussão
O delineamento experimental é um fator crítico em experimentos de RNA-seq. As principais considerações são o domínio preciso (s) e o estágio de desenvolvimento (s) a ser analisado, e será feito o que comparações. É fundamental pensar em termos de comparações, uma vez que a saída é normalmente uma lista de genes que são De entre duas ou mais condições. Tal como acontece com todas as experiências, é importante para alterar apenas uma variável de cada vez. Por exemplo, quando se comparam diferentes dom...
Divulgações
The authors have nothing to disclose.
Agradecimentos
Os autores agradecem a S. Hake para em curso colaboração e estimular discussões sobre o desenvolvimento ligule. Este trabalho é apoiado pela National Science Foundation Grants MCB 1052051 e IOS-1848478.
Materiais
| Name | Company | Catalog Number | Comments |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials - 22 ml | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 mm x 15 mm x 5 mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60 °C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
Referências
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados