É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Produção, cristalização e determinação da estrutura de
Neste Artigo
Resumo
Proline-proline endopeptidase-1 (PPEP-1) is a secreted metalloprotease and promising drug-target from the human pathogen Clostridium difficile. Here we describe all methods necessary for the production and structure determination of this protein.
Resumo
New therapies are needed to treat Clostridium difficile infections that are a major threat to human health. The C. difficile metalloprotease PPEP-1 is a target for future development of inhibitors to decrease the virulence of the pathogen. To perform biophysical and structural characterization as well as inhibitor screening, large amounts of pure and active protein will be needed. We have developed a protocol for efficient production and purification of PPEP-1 by the use of E. coli as the expression host yielding sufficient amounts and purity of protein for crystallization and structure determination. Additionally, using microseeding, highly intergrown crystals of PPEP-1 can be grown to well-ordered crystals suitable for X-ray diffraction analysis. The methods could also be used to produce other recombinant proteins and to study the structures of other proteins producing intergrown crystals.
Introdução
Clostridium difficile é uma das principais causas de infecções diarreia associada a antibióticos nosocomiais 1. Esta bactéria anaeróbia Gram-positiva são transmitidos através da sua forma de esporos através da via fecal-oral. Na década passada, o novo '' epidemia '' ou '' hipervirulento '' estirpes (por exemplo BI / NAP1 / 027) causou um aumento drástico em novas infecções e taxas de mortalidade na América do Norte e na Europa 2. C. doença -associated difficile (CDAD) é uma ameaça à vida inflamação do cólon com altas taxas de mortalidade 3. Os sintomas variam de 4 a diarreia colite pseudomembranosa 5 e o megacólon tóxico, muitas vezes fatal 6.
Tratamento de CDAD é difícil, pois as estirpes virulentas são multi-resistente ea taxa de recorrência é alta 7. No momento em terapia inclui a antibióticos metronidazol, vancomicina ou fidaxomicina, ou em repetiticasos recorrentes vely transplante de microbiota fecal. Novas estratégias terapêuticas são urgentemente necessários 8. Algum progresso está registada como a terapêutica anticorpo monoclonal Bezlotoxumab, visando C. difficile Toxina B 9, foi recentemente passou com êxito ensaios clínicos fase III e foi apresentado para aprovação com a FDA e EMA. Adicionalmente, novos antibióticos estão a ser testado no momento em diferentes fases de ensaios clínicos 10.
Para desenvolver um tratamento eficaz novos alvos terapêuticos devem ser identificados. O recém-descoberto C. difficile protease endopeptidase-1 prolina-prolina (PPEP-1; CD2830 / Zmp1; EC 3.4.24.89) é um alvo tão promissor, como a falta de PPEP-1 em uma cepa knock-out diminui virulência de C . difficile in vivo 11. PPEP-1 é uma metaloprotease segregada 12,13 clivagem dois adesinas de C. difficile no seu terminal C 13, libertando assim o bacter aderenteIA a partir do epitélio do intestino humano. Portanto, está envolvido na manutenção do equilíbrio entre o fenótipo séssil e móveis de C. difficile. Para desenvolver inibidores selectivos contra PPEP-1 e para entender como ele reconhece seus substratos conhecimento íntimo de sua estrutura tridimensional é indispensável. Nós resolvemos a primeira estrutura cristalina de PPEP-1 sozinho e em complexo com um péptido substrato 14. PPEP-1 é a protease primeiro conhecido que cliva selectivamente ligações peptídicas entre os dois resíduos de prolina 15. Liga-se o substrato de um modo duplamente dobrada e estabiliza através de uma rede alifático-aromático prolongado de resíduos localizados no S-ciclo que abrange o local de protease activa 14. Este modo de ligação de substrato é exclusivo para PPEP-1 e não foi encontrado na proteases humanos até à data. Isto o torna um alvo terapêutico promissor, e fora do alvo efeitos de inibidores muito improvável.
Para desenvolver e tela seletiva PPEP-1 habibitors no futuro, é necessária uma grande quantidade de proteína pura e monodispersas PPEP-1. Além disso, para determinar o modo de ligação dos primeiros inibidores, as estruturas de co-cristal com PPEP-1 terão de ser determinados. Em nossas mãos PPEP-1 produz constantemente cristais solidamente incorpora-. Assim, desenvolvemos um procedimento de optimização para produzir cristais de difracção de qualidade individuais de PPEP-1. Neste protocolo, descrever em detalhes a solução de produção, purificação, cristalização e estrutura da PPEP-1 14. Utilizamos a expressão intracelular em Escherichia coli de uma variante PPEP-1 sem a sequência de sinal de secreção, cromatografia de afinidade e cromatografia de exclusão de tamanho, com a remoção do marcador de purificação, seguido por microdispersão 16 para uma tela de optimização e a determinação da estrutura por meio de zinco-comprimento de onda único de dispersão anómala (zinco-SAD) 17. Este protocolo pode ser adaptado para a produção e a estrutura determinação de outras proteínas (por exemplo, </ Em> metaloproteases) e, em particular, para as proteínas produtoras de cristais solidamente incorpora-. No pedido, DNA plasmídeo da construção (pET28a-NHis-rPPEP-1) e dados de difração pode ser fornecida para finalidades educacionais.
Protocolo
1. Clonagem e construir projeto
- Clonar a sequência de codões optimizados (para E. coli) de C. difficile PPEP-1 sem o péptido de sinal [27-220 aminoácidos, denominado a seguir recombinante PPEP-1 (rPPEP-1) 11] no vector pET28a usando NdeI e locais de restrição Xho I (Figura 1) com um codão de stop no (vector resultante pET28a-NHis-rPPEP-1) extremidade 3 '. Isto produz um terminal-N 6xHis-tagged proteína (NHis-rPPEP-1) com um local de clivagem de trombina, permitindo a remoção da etiqueta durante a purificação (Figura 1). O plasmídeo contem uma cassete de resistência à canamicina para selecção. Os iniciadores utilizados para a clonagem são descritos noutro local 14.
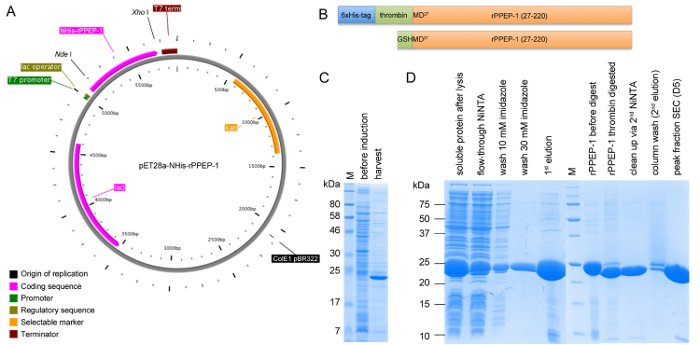
Figura 1: Representação esquemática da construção de pET28a-NHis-rPPEP-1 e SDS-PAGE análise das expression e todos os passos de purificação. (A) Mapa do vetor de NHis-rPPEP-1 clonado em pET28a vetor usando Nde I / Xho I criado com PlasMapper. (B) Representação esquemática do NHis-rPPEP-1 construção (painel superior) a e a construção final depois de trombina-clivagem da 6xHis-tag com o GSHM-sequência adicional resultante a N-terminal (painel inferior) a. A análise de SDS-PAGE (C) da expressão em células BL21 (DE3) de estrelas a 37 ° C durante 4 horas e (D) de amostras de todos os passos de purificação (M: marcador de peso molecular). Por favor clique aqui para ver uma versão maior desta figura.
2. Expressão e Purificação de rPPEP-1
- Expressão de NHis-1-rPPEP
- Adicione-se e autoclave LB (caldo de lisogenia) meio (10 g / L de triptona, 5 g / L de extracto de levedura, 10 g / L de NaCl, ajustÃst a pH 7,5 com NaOH). Suplemento com sulfato de canamicina (50 ug / ml) imediatamente antes da utilização (meio LB / Kan).
- Inocular 200 ml de cultura durante a noite de E. coli recentemente transformada em meio LB / Kan. Crescer durante a noite a 37 ° C com agitação a 220 rpm.
- Na manhã seguinte, verificar a DO600 (densidade óptica a 600 nm comprimento de onda) da cultura durante a noite. Inocular dois 2.8 L perplexo frascos contendo 1 L LB / Kan meio de cada um com a cultura durante a noite para um OD 600 de 0,1. Suplemento com três gotas de emulsão aquosa de silicone para evitar a formação de espuma excessiva. Crescer as células a 37 ° C com agitação a 180 rpm até a DO600 atinge 0,6.
- Retirar uma amostra de pré-indução para a análise de SDS-PAGE (equivalente a 1 ml de uma cultura a OD 600 = 1); adicionar IPTG até à concentração final de 0,5 mM para induzir expressão de NHis-rPPEP-1. Continuar a crescer a 37 ° C / 180 rpm durante 4 h.
- Determinar o OD 600 num di 10xlução e tomar uma amostra colheita (equivalente a 1 ml de uma cultura a OD 600 = 1).
- Recolher as células por centrifugação durante 20 min a 7000 xg e 4 ° C. Para remover os sedimentos celulares meio LB ressuspender residuais a partir de 1 L de cultura em 40 mL de tampão de TBS (solução salina tamponada com Tris: Tris-HCl a 20, pH 7,5, NaCl 200 mM) e transferir para um tubo de centrífuga de 50 ml. Recolher as células por centrifugação durante 10 min a 10000 xg e 4 ° C e armazenar a -80 ° C até à sua utilização. Analisar a expressão (lisados totais e frações solúveis), através de SDS-PAGE 18.
- A purificação de não marcado rPPEP-1
- Aqui amostras de 50 ul de cada passo de purificação para a análise de SDS-PAGE. Ressuspender o sedimento de células a partir de 1 L de cultura em tampão TBS suplementado com 10 ug / ml de ADNasel. Use 5 ml de TBS / ADNasel por g de células.
- Lisar as células por sonicação em gelo / água, utilizando 30% de amplitude durante 15 min (2 s com pulsos pausa de 2 seg). Remover detritos por centrifugation durante 10 min a 10000 xg e 4 ° C e a transferência de sobrenadante para um tubo de ultracentrífuga. Limpar lisado numa ultracentrífuga durante 30 min a 165000 xg e 4 ° C.
- Trabalhar a 4-6 ° C. Usando um sistema de bomba peristáltica ou cromatografia equilibrar 2 ml de ácido (NiNTA) resina de níquel-nitrilotriacético numa coluna de vidro com tampão TBS suplementado com 10 mM de imidazol, pH 7,5. Em alternativa, usar o fluxo de gravidade.
- Ajustar o ligado clarificado com imidazolo 1 M, pH 7,5 a uma concentração final de 10 mM. Aplicar o lisado para a coluna e lava-se passo a passo com tampão TBS suplementado com 10 mM e imidazole 30 mM, respectivamente, até que a absorção de UV a 280 nm tenha atingido a linha de base.
- Elui-se a proteína com tampão de TBS mais imidazol 250 mM. Re-equilibrar a coluna com TBS suplementado com imidazole 10 mM e armazenar durante a noite.
- Determinar a concentração de proteína, quer a 280 nm, utilizando o coeffi extinçãociente de 25900 M-1 cm-1 ou por qualquer outro método (método de Bradford por exemplo 19). Adicionar 2 unidades de trombina por mg de proteína e dialisar a solução de proteína durante a noite a 4 ° C contra um volume 50x de TBS (50x do volume de eluição NiNTA).
NOTA: Tome o espaço em branco correto para a determinação da concentração de proteína, como imidazol absorve fortemente a 280 nm. - Passar a solução de proteína sobre a resina NiNTA equilibrada para remover a proteína não clivada. Em seguida, aplicam-se o mesmo volume de TBS suplementado com imidazole 10 mM a coluna para recuperar toda a proteína clivada. Para limpar a coluna, elui-se toda a proteína restante com imidazole 250 mM. Analisar amostras através de SDS-PAGE (Figura 1).
- Concentra-se a solução de proteína de 4 ml em intervalos de 10 min a 4000 xg e 4 ° C utilizando uma unidade de ultraf iltração centrífuga. Misturar a proteína concentrando depois de cada intervalo para evitar a precipitação e agregação. Neste passo ocasionalLY alguma precipitação é observada para rPPEP-1, apesar do procedimento de mistura.
- Aplicar a proteína concentrada a uma coluna de cromatografia de exclusão de tamanho pré-equilibrada (em tampão de TBS) a 4-6 ° C. Elui-se a coluna com tampão de TBS, recolher fracções de 1 ml e sujeito 5 uL de cada fracção para análise segundo SDS-PAGE. rPPEP-1 elui num único pico correspondendo a um monómero (Figura 2). Ocasionalmente um pico menor de peso molecular maior é observada (pico de frente), que corresponde a um dímero da proteína. O rendimento deve ser em torno de 50 mg de proteína pura por L de cultura. Analisar todas as amostras através de SDS-PAGE 18 (Figura 2).
- Aqui amostras de 50 ul de cada passo de purificação para a análise de SDS-PAGE. Ressuspender o sedimento de células a partir de 1 L de cultura em tampão TBS suplementado com 10 ug / ml de ADNasel. Use 5 ml de TBS / ADNasel por g de células.
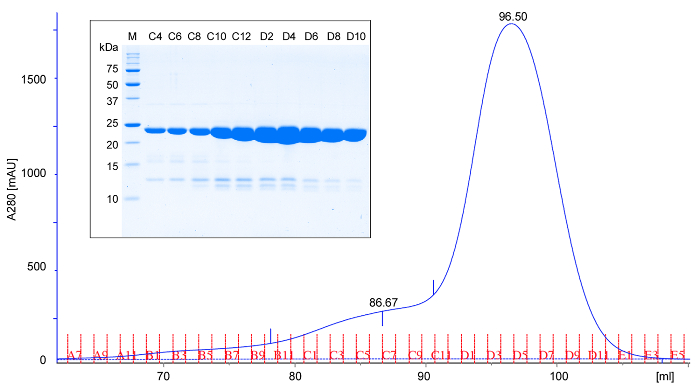
Figura 2: Cromatografia de exclusão de tamanho representativas e análise de SDS-PAGE de rPPEP-1. cromatograma de exclusão por tamanhos (A280; absorvância a 280 nm) de purificado rPPEP-1 não marcada usando um) coluna (16/600 em Tris-HCl, pH 7,5, NaCl 200 mM a 6 ° C. Com base no volume de eluição, rPPEP-1 migra como esperado para uma proteína de 22 kDa, sugerindo que é predominantemente monomérico. Raramente um pico menor aparecem de fachada que corresponde a um dimero. (Inset) a análise de SDS-PAGE das fracções de cromatograf ia de exclusão de tamanho (M; marcador de peso molecular). Toda segunda fracção é aplicada. As bandas fracas abaixo da principal rPPEP-1 banda correspondem aos que ocorrem ocasionalmente pequenas impurezas. Por favor clique aqui para ver uma versão maior desta figura.
3. A cristalização e Crystal Optimization Usando microdispersão
NOTA: rPPEP-1 cristaliza a partir de condições que constantemente produzir cristais altamente solidamente incorpora- não é adequado para análise de difracção de raios-X (Figura 3). Portanto, uma estratégia de optimização (Figura 4) foi desenvolvido para obter cristais de elevada qualidade (Figura 5).
- Triagem inicial de rPPEP-1 utilizando telas comerciais
NOTA: Realizar ensaios de cristalização no formato de gota sentado usando padrão telas disponíveis comercialmente e um robô cristalização.- Concentra-se a proteína purificada a 12 mg / ml utilizando um dispositivo de ultrafiltração centrífuga em intervalos de 5 minutos a 4.000 xg e 4 ° C. Misturar a proteína concentrando depois de cada intervalo para evitar a precipitação e agregação. Determinar a concentração de proteína, quer a 280 nm, utilizando o coeficiente de extinção de 25900 M-1 cm-1 ou por qualquer outro meio (por exemplo, método de Bradford). Equilibrar a proteína a 20 ° C. Limpar todas as partículas de pó e por centrifugação durante 10 min a 16000 xg e 20 ° C.
- Ou usar placas de cristalização já pré-cheias SEAled e armazenado a 4 ° C ou encher os poços das placas de reservatórios com 70 ul de cada condição de cristalização. Equilibrar todas as placas de cristalização de 20 ° C. Trabalhe rapidamente, como os pequenos volumes rapidamente secar. Use uma câmara de umidade em torno da doca do robô, se possível.
NOTA: Use as seguintes telas como um procedimento padrão: SaltRx, índice PEG / Ion, cristal, assistente, PACT ++, JCSG ++. - Configurar a tela pipetando proteína e reservatório para subwells 2-4. O volume da gota é de 300 nl e os rácios (proteína: reservatório) são 200: 100 (subwell 2), 150: 150 (subwell 3) e 100: 200 (subwell 4) (em nl). selar imediatamente a placa e realizado numa câmara a 20 ° C.
- Inspecione bandejas imediatamente após set-up, e depois inspecionar todos os dias durante a primeira semana, seguido por inspeção semanal.
- Co-cristalização da rPPEP-1 com ligantes
- Para a co-cristalização dos complexos de péptido-1-rPPEP substrato misturar rPPEP-1a 24 mg / ml em uma proporção de 1: 1 (v / v) com um excesso molar de 7 vezes de solução de péptido (Ac-EVNPPVPD-NH2) em pó liofilizado solubilizado em tampão TBS), o que dará uma concentração final de 12 mg / ml de proteína de r-PPEP-1 e um excesso molar de 7 vezes de péptido ao longo PPEP-1. Incubar durante 30 min a 20 ° C e limpar todas as partículas de pó e por centrifugação durante 10 min a 16000 xg e 20 ° C. Continuar com a cristalização utilizando o procedimento microdispersão, como descrito para a proteína não ligada r-PPEP-1.

Figura 3: cristais representativos de telas iniciais. cristais desenvolvida intermediariamente de rPPEP-1 a 12 mg / ml cultivadas em condição. (A) de tela de cristal I / 38 (citrato de sódio 1,4 M tribasic desidratar, 0,1 M HEPES pH de sódio a 7,5; 200 nl: 100 nl). / 52 (DIB fosfato de 2,4 M de amônio (B) ecrã SaltRxASIC, 0,1 M de Tris pH 8,5; 100 nl: 200 nl) e (C) (200 nl: 100 nl). (D) de tela SaltRx / 96 (60% v / v Tacsimate pH 7,0, 0,1 M BIS-TRIS propano pH 7,0; 200 nl: 100 nl). A barra de escala = 0,2 mm. índices de volume são sempre proteína: reservatório. Por favor clique aqui para ver uma versão maior desta figura.
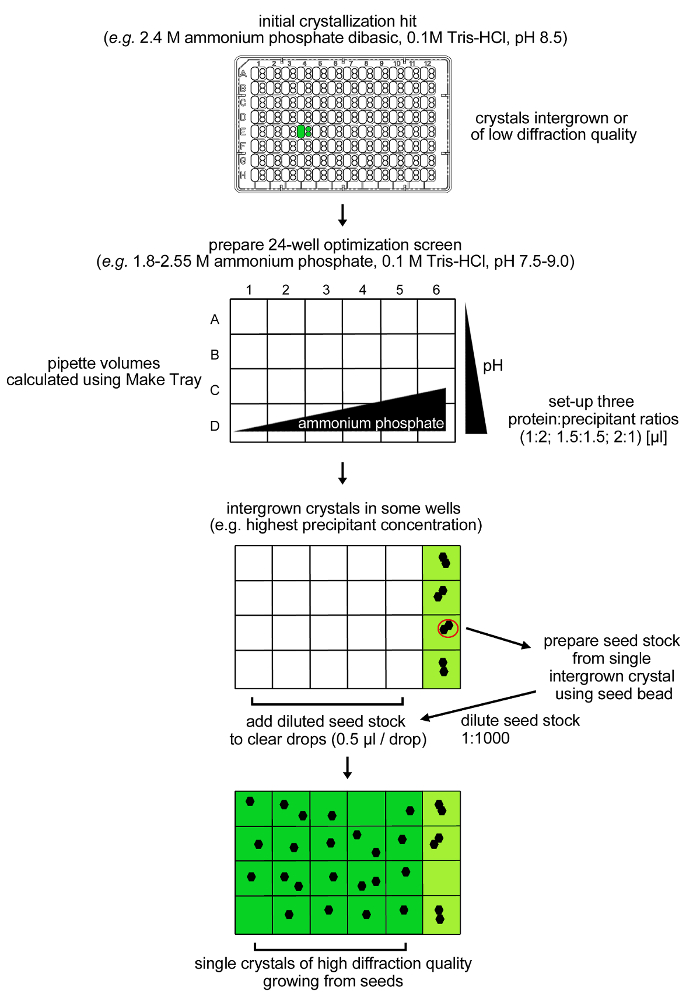
Figura 4: procedimento de otimização para rPPEP-1 cristalização. cristais iniciais de rPPEP-1 a 12 mg / ml de baixa qualidade difração e com várias treliças (desenvolvida intermediariamente) foram reproduzidos em uma tela de otimização 24 condições. Mais uma vez, apenas cristais solidamente incorpora- foram observados em condições contendo fosfato dibásico 2,55 M de amónio. Um estoque de sementes foi preparada a partir de um único cristal desenvolvida intermediariamente e diluído 1: 1000 no mesmo Conditde iões (microdispersão). Um volume de 0,5 ul de estoque de sementes diluída foi adicionada em gotas os restantes claros e únicos cristais cresceram em quase todas as condições. Por favor clique aqui para ver uma versão maior desta figura.
- Otimização de cristal usando microdispersão
NOTA: cristais altamente solidamente incorpora- de rPPEP-1 aparecer depois de dois dias de uma condição que contém 2,4 M de fosfato de amónio dibásico, Tris-HCl 0,1 M, pH 8,5 (tela SaltRx, condição E4, todos os três subwells) (Figura 3). Um procedimento de optimização usando uma tela de grelha em torno da condição inicial combinado com microdispersão foi aplicado (Figura 4).- Prepara-se uma tela de grelha (Figura 4) compreendendo 24 condições com 1,8-2,55 M de fosfato dibásico de amónio (em passos de 0,15 M) e 0,1 M de Tris-HCl pH 7,5-9,0 (em passos de 0,5 unidades de pH) a partir de aprosoluções de reserva comeram (4 M de fosfato de amónio e 1 M de Tris) tampões.
NOTA: Use o applet Bandeja Marca (http://hamptonresearch.com/make_tray.aspx) para calcular os volumes e o esquema de pipetagem para obter 2 ml de cada condição que permite realizar 10 telas de otimização. A solução de partida de fosfato de amónio 4 M é difícil de preparar. Aquece-se a solução durante a agitação para dissolver completamente o pó na água. - Pipetar 200 ul de cada solução tela grade para os poços de uma placa de 24 poços e equilibrar-se a 20 ° C.
- configurar manualmente a placa de cristalização. O volume da gota é de 3 ul e as proporções (proteína: Reservatório) são de 2: 1, 1,5: 1,5 e 1: 2 (em pi). Aqui, utilizar uma pipeta de deslocamento positivo para evitar a formação de bolhas de ar. selar imediatamente a placa e realizado numa câmara a 20 ° C. Evitar a formação de bolhas de ar.
NOTA: Depois de um a quatro dias cristais altamente solidamente incorpora- aparecem nos quatro condições que contêm pho 2,55 M de amóniosphate dibásico e 0,1 M de Tris-HCl pH 7,5-9,0 (Figura 4). Sem cristais são formados nos restantes 20 condições com concentrações de fosfato de amónio abaixo de 2,55 M. O procedimento microdispersão é usado para se obterem cristais individuais de rPPEP-1 nestas condições. - Prepara-se uma colheita da microseed por um único cristal desenvolvida intermediariamente a partir de uma das duas condições com fosfato dibásico de amónio 2,55 M e 0,1 M de Tris-HCl a pH 8,0 ou 8,5. Os cristais pode ser ligado à superfície de plástico. deformação cuidado com o plástico envolvente, com uma agulha de acupuntura ajuda a separar os cristais.
- Transferir 50 ml da respectiva licor-mãe em um tubo de 1,5 ml contendo uma pequena pérola de vidro polido (beads para-sementes). Utilizando uma ansa de nylon montado transferir o cristal em 1 ul da solução mãe colocadas sobre uma lâmina de vidro de cobertura.
- Transferir o líquido contendo o cristal e para dentro do tubo de vórtice a alta velocidade durante 30 segundos. Adicione uma mistura 1:1000 de diluição do stock de sementes para um novo tubo de 1,5 mL contendo a mesma condição recentemente preparado e agitar com vortex vigorosamente durante 5 segundos.
NOTA: Sementes existências pode ser armazenado a -80 ° C para uso posterior.
- Remover a vedação da placa de cobertura 20 com gotas condições claras e pipeta de 0,5 mL do stock de sementes (diluição 1: 1000) nas cavidades. Selar a placa e realizado numa câmara a 20 ° C. Os cristais individuais de alta qualidade de difracção aparecem em 2-7 dias (Figura 5).
- Prepara-se uma tela de grelha (Figura 4) compreendendo 24 condições com 1,8-2,55 M de fosfato dibásico de amónio (em passos de 0,15 M) e 0,1 M de Tris-HCl pH 7,5-9,0 (em passos de 0,5 unidades de pH) a partir de aprosoluções de reserva comeram (4 M de fosfato de amónio e 1 M de Tris) tampões.
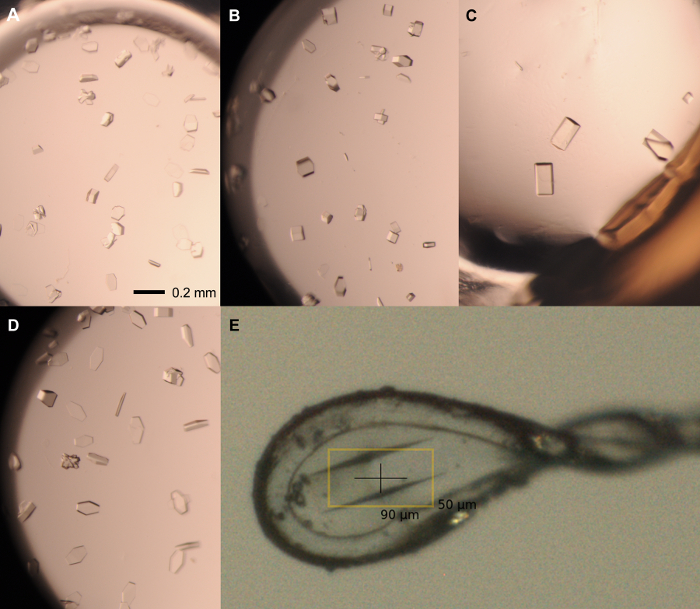
Figura 5: cristais representativos de tela otimização. Os cristais individuais de rPPEP-1 a 12 mg / ml semeado com 1: stock de sementes de 1.000 diluição cultivadas nas seguintes condições: (a) 2,1 M de amónio dibásico de fosfato, 0,1 M de Tris pH 7,5; 1,5 ul: 1,5 ul; (B) De fosfato dibásico de amónio 2,1 M, Tris 0,1 M pH 7,5; 2 ul: 1 mL; (C) 2,25 M de amónio dibásico de fosfato, Tris 0,1 M pH 8; 2 ul: 1 mL; (D) 2,1 M de amónio dibásico de fosfato, Tris 0,1 M pH 8; 1 ul: 2 ul. (E) montado de cristal em malha de nylon de 0,1-0,2 uM, cultivadas em 2,1 M de amónio dibásico de fosfato, 0,1 M de Tris pH 8 (2 mL: 1 mL) e 2,1 M em fosfato de amónio dibásico, 0,1 M de Tris pH 8 protegido-crio, 20% de glicerol. A barra de escala = 0,2 mm (AD). ração de volume são sempre proteína: reservatório. Por favor clique aqui para ver uma versão maior desta figura.
4. cristal de montagem e coleta de dados
NOTA: Para obter a melhor qualidade de cristais de dados de difração deve ser montado no auge da sua qualidade e tamanho. Os cristais podem ser armazenados em azoto líquido até que eles ARe submetido a análise de difracção de raios-X a 100 K. Assim, a condição de que são originários deve ser ajustado para crio condições. rPPEP 1-cristais podem ser crio-protegidas por adição de qualquer glicerol a 20% ou 30% de sacarose (água de substituição de na condição de o crioprotector).
- montagem de cristal
NOTA: Todas as etapas de manipulação de cristal deve ser realizada sob o microscópio estereoscópico.- Escolha o tamanho ideal de loop de nylon para o comprimento máximo dos cristais escolhidas. O eixo mais longo típico de rPPEP-1 cristais é de cerca de 100-200 uM (Figura 5). Prepara-se uma cobertura deslizante e o crio-condições apropriadas (por exemplo, 2,1 M de amónio dibásico de fosfato, Tris 0,1 M, pH 8,0, glicerol a 20%).
- Encha os vasos dewar espuma com azoto líquido, coloque o grampo frasco com um frasco e pré-arrefecer no 800 ml dewar espuma cheio de nitrogênio líquido. Coloque uma luva crio e um suporte de cana-de-crio marcado com um identificador adequadoem 2 L dewar espuma cheio de nitrogênio líquido. Carregar o bastão magnético com um loop de nylon montado.
NOTA: Usar vestuário de protecção (EyeShield / óculos, luvas) quando se trabalha com nitrogênio líquido. objetos quentes mergulhado em nitrogênio líquido pode produzir derrames.
- Encha os vasos dewar espuma com azoto líquido, coloque o grampo frasco com um frasco e pré-arrefecer no 800 ml dewar espuma cheio de nitrogênio líquido. Coloque uma luva crio e um suporte de cana-de-crio marcado com um identificador adequadoem 2 L dewar espuma cheio de nitrogênio líquido. Carregar o bastão magnético com um loop de nylon montado.
- Cortar abrir a fita de vedação com um bisturi afiado e removê-lo com a pinça. Pipetar 1 mL da crio-condição para a lâmina de cobertura (ou, em alternativa, um poço vazio sobre a mesma placa) e remover o cristal da gota pela pesca com o loop de nylon montado (Figura 5). cristais anexados podem ser facilmente retirado do solo por deformar o plástico envolvente, com uma agulha de acupuntura.
- Transferir rapidamente o cristal à gota de crio-condição e deixou-se equilibrar durante 1 segundo. Pesco o cristal, tão rapidamente quanto possível e de imersão por congelação em azoto líquido.
- Quando o azoto líquido em torno do circuito montado pára de ebulição, colocar o laço no frasco.Colocar o frasco no suporte da cana-de-crio e quando carregado com 6 frascos colocar uma manga de crio em torno do suporte. Armazenar os cristais em um tanque cheio com azoto líquido até à sua utilização.
- Escolha o tamanho ideal de loop de nylon para o comprimento máximo dos cristais escolhidas. O eixo mais longo típico de rPPEP-1 cristais é de cerca de 100-200 uM (Figura 5). Prepara-se uma cobertura deslizante e o crio-condições apropriadas (por exemplo, 2,1 M de amónio dibásico de fosfato, Tris 0,1 M, pH 8,0, glicerol a 20%).
- Coleção de dados
NOTA: A coleta de dados pode ser realizado no difratômetro casa, se estiver disponível, ou em uma linha de luz síncrotron. Para dados rPPEP-1 foram coletadas no X06DA beamline da Fonte de Luz suíço, Paul-Scherrer-Institute, Villigen, Suíça usando um detector de contagem híbrida fóton. Os dados originais e todos os arquivos utilizados na determinação da estrutura pode ser fornecido mediante solicitação.- Defina-se o comprimento de onda do feixe de 1,282 ã (9667 keV), que é o raio-x de energia limiar de absorção característico (pico) do elemento de zinco. rPPEP-1 é uma metaloprotease que contém um único zinco por molécula no local activo.
- Recolha de dados a 100 K no modo inverso do feixe em 10 ° cunhas para um total de 270 ° em cada direction. O tempo de exposição é de 0,1 segundos com 0,1 ° de rotação por imagem. Definir a transmissão a 14% (0,14).
- Para recolher um conjunto de dados de alta resolução nativa de um segundo cristal originário da mesma condição de cristalização configurar o comprimento de onda do feixe para 1,00 Å (12.398 keV). Recolha de dados a 100 K. O tempo de exposição é de 0,1 segundos com 0,1 ° de rotação por imagem. Definir a transmissão a 70% (0,7).
5. Determinação Estrutura via Zinc-SAD
NOTA: De modo a determinar a estrutura de uma via rPPEP-zinco-SAD algum conhecimento cristalográfica de base é necessária, bem como os pacotes de software XDS 20, Phenix 21 e o programa coot 22. Para visualização de estruturas é necessário o programa PyMOL 23 ou Chimera 24. Os dados recolhidos no comprimento de onda correspondente ao pico de absorção na extremidade do elemento de zinco pode ser utilizado para um único comprimento de onda disp anómalaersion (SAD) 25 para obter informação de fase que pode ser estendido para todos os átomos de proteína.
- Processamento de dados
- Processar os dois conjuntos de dados de pico (normal e inversa) utilizando o software XDS (alternativamente iMosflm ou HKL3000) no grupo espacial P2 1 2 1 2 1 (grupo espacial 19) que separam companheiros do Friedel (dados anômalos). Os parâmetros da célula unitária deve ser cerca de a, b, c (a) = 43,17, 71,68, 117,70 e α = β = γ (°) = 90. Isto dá dois HKL-arquivos (ficheiros) de reflexão.
- Inspecione o CORRECT.LP arquivo. Use dados até a resolução em que o CC 1/2 é de pelo menos 50%. Escalar juntos os dois conjuntos de dados / arquivos reflexão (HKL-files) usando XSCALE. Inspecione o XSCALE.LP arquivo. Verifique o quanto o sinal anómalo se estende (SigAno) e anote a resolução com uma correlação anômala (anomal Corr) de cerca de 30%, que é de 2 a No caso de os dados utilizados aqui recolhidos para 1,67 Å. Isto é oresolução de corte para o sinal anômalo usado em Phenix Autosol.
- Converter o (escala) HKL-arquivo em um arquivo reflexão CCP4 formato (chamado, por exemplo, peak_anom.mtz) usando XDSCONV criar um subconjunto livre R de 5% e manter os dados anômalos (FRIEDEL'S_LAW = FALSE). Verifique o arquivo mtz de coerência com a mtzdmp programa de inspecionar os parâmetros da unidade célula, o grupo de espaço e a existência do subconjunto livre R (etiqueta FreeRflag) e os dados anômalos (etiquetas DANO / SIGDANO). Prepare também um MTZ-arquivo adicional com XDSCONV sem extrair os dados anômalos (FRIEDEL'S_LAW = TRUE; nomeado, por exemplo, peak_native.mtz) para o refinamento numa fase posterior.
- Solução subestrutura (determinação de fase)
- Executar Phenix Autosol usando o peak_anom.mtz arquivo de reflexão. Selecione pico SAD / MAD como tipo de dados e escolha de 2 sites de zinco (como há duas moléculas por unidade assimétrica). Escolha entre a experi mais exatavalores mental para a f '/ f' 'parâmetros (determinados em uma varredura de fluorescência na linha de luz) ou os valores theotetical f' = -8,245 e f '' = 3,887. Também carregar o ficheiro FASTA contendo a sequência de aminoácidos da proteína cristalizada.
- Definir o limite de resolução para a resolução com uma correlação anômala (anomal Corr) de cerca de 30% (determinado em 5.1.2), neste caso 2 Å e selecione a opção "modelo AutoBuild". Usando as fases dos dois locais de zinco encontrados por Phenix HySS (parte do gasoduto Phenix Autosol) as fases para toda a proteína poderia ser deduzido e o modelo construído (por Phenix RESOLVE) na densidade de elétrons. O melhor modelo é chamado de "overall_best.pdb".
- Construção de modelos, refinamento e validação
- Selecione a opção "modelo AutoBuild" para construir a maior parte do modelo rPPEP-1 automaticamente. Inspecione a densidade de elétrons em 1,0 σ nível de contorno usando o program coot (Figura 6). Deve ser conjuntivo circundante e os átomos do modelo. Idealmente também algumas moléculas de água deve ser construído dentro do modelo (com uma resolução melhor do que 2,5 Å). água bruta (espaço entre as moléculas) não deve conter densidade.
- Verifique se todo o modelo é (todos os aminoácidos incorporados a densidade de elétrons) completos. Se não, construí-los manualmente usando as ferramentas fornecidas pelo Coot. Afinar a estrutura, executando ciclos iterativos de Phenix Refine com 5 refinamento rodadas cada um usando o arquivo de modelo overall_best.pdb, o arquivo peak_native.mtz reflexão e o arquivo de seqüência FASTA; e construção do modelo manual em Coot.
- Validar a qualidade do modelo estrutural com as respectivas ferramentas em coot.
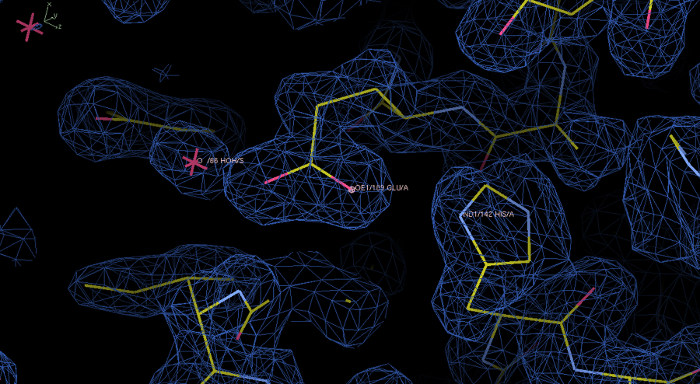
Figura 6: Mapa Experimental densidade de elétrons e modelo do rPPEP-1 após aPhenix Autosol executado. densidade de elétrons em azul em um nível de contorno de 1,0 σ exibido no Coot programa. Neste mapa inicial da densidade de elétrons é bem resolvido e o modelo de construção para a densidade de elétrons. A em zoom mostra os resíduos His142 e Glu189, bem como uma molécula de água. Por favor clique aqui para ver uma versão maior desta figura.
Determinação 6. Estrutura de alta resolução via Substituição Molecular
NOTA: Para obter informações estruturais de alta resolução sobre rPPEP-1 um conjunto de dados nativa é coletado. Em seguida, um processo de substituição molecular, utilizando o software de 26,27 Phaser (dentro do pacote de software Phenix) é empregue utilizando a estrutura determinada por meio de zinco-SAD como um modelo. Este procedimento pode também ser usado mais tarde, quando a resolução de estruturas rPPEP-1 complexadas com moléculas pequenas.
- Para obter uma estrutura cristalina com maior resolução (neste caso até 1,4 Â) processar o conjunto de dados nativa utilizando o software XDS (alternativamente ou iMosflm HKL3000) no grupo espacial P2 1 2 1 2 1 (grupo espacial 19). Os parâmetros da célula unitária deve ser cerca de a, b, c (a) = 43,17, 71,77, 117,80 e α = β = γ (°) = 90. Isto dá uma HKL-ficheiros de arquivo (reflexão).
- Inspecione o CORRECT.LP arquivo. Use dados até a resolução em que o CC 1/2 é de pelo menos 50%. Converter o arquivo HKL em um arquivo de reflexão CCP4 formato (chamado, por exemplo, native.mtz) usando XDSCONV criar um subconjunto livre R de 5%. Verifique o arquivo mtz de coerência com a mtzdmp programa de inspecionar os parâmetros da célula unitária, o grupo de espaço e a existência do subconjunto livre R (etiqueta FreeRflag).
- Prepare o PDB-arquivo que contém o modelo de overall_best.pdb determinada anteriormente e remover todas as moléculas de água e todos os ligantes (ou seja,. o átomo de zinco). Também carregar o ficheiro FASTA contendo a sequência de aminoácidos da proteína cristalizada. Execute Phaser em Phenix usando o native.mtz arquivo de reflexão. Pesquisar por duas moléculas por unidade assimétrica.
- Depois solução estrutura bem-sucedida (TFZ-score maior do que 8; aqui 10.2) inspecionar o modelo (chamado native_phaser.1.pdb) eo mapa de densidade de elétrons em Coot. Construir e aperfeiçoar a estrutura, executando ciclos iterativos de Phenix Refine com 5 refinamento rodadas cada um usando o arquivo de modelo native_phaser.1.pdb, o arquivo de reflexão native.mtz eo arquivo seqüência FASTA; e construção do modelo manual em Coot.
- Validar a qualidade do modelo estrutural com as respectivas ferramentas em coot.
Resultados
rPPEP-1 é sobre-expressa em várias estirpes de E. coli, com o rendimento mais elevado em E. coli BL21 (DE3) Star (Figura 1C). Após o primeiro passo de cromatografia de afinidade NiNTA a 6xHis-tag pode ser clivado com sucesso fora da maior parte da proteína e no segundo passo NiNTA proteína não digerida pode ser completamente separada da proteína digerida com trombina (Figura 1D). Em um S200 16/600 coluna rPPEP-1 não marcado migr...
Discussão
Cristalografia de raios-X ainda é o método mais rápido e mais preciso para determinar tridimensionais estruturas de resolução de quase-atómicas de proteínas 28. No entanto, isso exige o crescimento dos cristais individuais bem ordenadas. Estes são muitas vezes difíceis de obter e estado cristalino é artificial. No entanto, uma comparação de estruturas de proteína determinada por cristalografia de raios-X com os determinados por outros métodos, especialmente RMN, mostra geralmente uma boa concord...
Divulgações
The authors have nothing to disclose.
Agradecimentos
Agradecemos a equipe do X06DA beamline na Fonte de Luz suíço, Paul-Scherrer-Institute, Villigen, Suíça para a sustentação durante a coleta de dados síncrotron. Somos gratos a Monika Gompert para excelente suporte técnico. O projecto foi apoiado pela Universidade de Colónia e conceder INST 216 / 682-1 fugg do Conselho de Investigação alemã. A bolsa de doutoramento da Escola de Pós-Graduação Internacional em Saúde Desenvolvimento e Doença de CP é reconhecido. A investigação conducente a estes resultados foi financiada pelo Sétimo Programa-Quadro da Comunidade Europeia (FP7 / 2007-2013) ao abrigo do contrato de concessão No. 283570 (BioStruct-X).
Materiais
| Name | Company | Catalog Number | Comments |
| Genes / Vectors / cell strains | |||
| pET28a vector | Merck-Millipore | 69864 | Thrombin cleavable N-terminal His-tag |
| E. coli strain BL21 (DE3) Star | ThermoFisher Scientific | C601003 | RNase H deficient |
| Codon-optimized gene (for E. coli) of PPEP-1 (CD630_28300) | Geneart (Thermo Fisher Scientific) | custom | amino acids 27-220 |
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Yeast extract | any | ||
| Tryptone | any | ||
| Antifoam B | Sigma-Aldrich | A5757 | aqueous-silicone emulsion |
| Agar | any | ||
| Kanamycin | any | ||
| IPTG | AppliChem | A1008 | |
| Tris-HCl | AppliChem | A1087 | Buffer grade |
| NaCl | any | Buffer grade | |
| DNaseI | AppliChem | A3778 | |
| Imidazole | AppliChem | A1073 | Buffer grade |
| Thrombin | Sigma-Aldrich | T4648 | |
| Ammonium phosphate dibasic | Sigma-Aldrich | 215996 | |
| Glycerol 100% | any | purest grade | |
| Sucrose | Sigma-Aldrich | 84097 | |
| Liquid nitrogen | any | for storage and cryocooling of crystals | |
| Name | Company | Catalog Number | Comments |
| Equipment (general) | |||
| Shaking incubator | any | providing temperatures of 20 °C - 37 °C | |
| Glassware | any | baffled Erlenmeyer flasks (50 ml - 2.8 L) | |
| Centrifuge for large culture volumes | any | centrifuge for processing volumes up to 12 L | |
| Sonicator Vibra-Cell VCX500 | Sonics | SO-VCX500 | or any other sonicator / cell disruptor |
| Ultracentrifuge | any | centrifuge providing speeds up to 150,000 x g | |
| NiNTA Superflow resin | Qiagen | ||
| Empty Glass Econo-Column | Bio-Rad | 7371007 | or any other empty glass or plastic column |
| Size exclusion chromatography column HiLoad Superdex 200 16/600 | GE Healthcare | 28989335 | |
| Chromatography system Äkta Purifier | GE Healthcare | 28406264 | or any other chromatography system |
| Dialysis tubing Spectra/Por 3 | Spectrum Labs | 132724 | |
| Dialysis tubing closures | Spectrum Labs | 132738 | |
| Ultrafiltration units (concentrators) 10,000 NWCO | any | ||
| UV-Vis spectrophotometer | any | ||
| Name | Company | Catalog Number | Comments |
| Equipment (crystallography) | |||
| Low volume pipette 0.1-10 µl | any | ||
| Positive displacement pipette Microman M10 | Gilson | F148501 | |
| Crystallization robot | any | ||
| 96-well crystallization plates TTP IQ with three protein wells | TTP | 4150-05810 | or any other 96-well crystallization plate |
| 24-well CombiClover Junior Plate | Jena Bioscience | EB-CJR | |
| Crystal Clear Sealing Tape | Hampton Research | HR3-511 | |
| Siliconized Glass Cover Slides | Hampton Research | HR3-225 | |
| Commercial crystallization screens: SaltRx, Index, PEG/Ion, Crystal | Hampton Research | diverse | |
| Commercial crystallization screens: Wizard, PACT++, JCSG++ | Jena Bioscience | diverse | |
| JBS Beads-for-Seeds | Jena Bioscience | CO-501 | |
| CrystalCap SPINE HT (nylon loops) | Hampton Research | diverse | loop sizes 0.025 mm - 0.5 mm |
| CrystalCap Vial | Hampton Research | HR4-904 | |
| Cryogenic Foam Dewar 800 ml | Hampton Research | HR4-673 | |
| Cryogenic Foam Dewar 2 L | Hampton Research | HR4-675 | |
| Vial Clamp, Straight | Hampton Research | HR4-670 | |
| CrystalWand Magnetic, Straight | Hampton Research | HR4-729 | |
| CryoCane 6 Vial Holder | Hampton Research | HR4-711 | |
| CryoSleeve | Hampton Research | HR4-708 | |
| CryoCane Color Coder - White | Hampton Research | HR4-713 | |
| Scalpel | any | ||
| Straight microforcep | any | for manipulation of sealing tape. etc. | |
| Acupuncture needle | any | e.g. from a pharmacy | |
| Stereo microscope | any | for inspection of crystallization plates and crystal mounting, magnification up to 160X |
Referências
- Bouza, E. Consequences of Clostridium difficile infection: understanding the healthcare burden. Clin Microbiol Infect. 18 (Suppl 6), 5-12 (2012).
- O'Connor, J. R., Johnson, S., Gerding, D. N. Clostridium difficile infection caused by the epidemic BI/NAP1/027 strain. Gastroenterology. 136 (6), 1913-1924 (2009).
- Mitchell, B. G., Gardner, A. Mortality and Clostridium difficile infection: a review. Antimicrob Resist Infect Control. 1 (1), (2012).
- George, W. L., Sutter, V. L., Finegold, S. M. Antimicrobial agent-induced diarrhea--a bacterial disease. J Infect Dis. 136 (6), 822-828 (1977).
- George, R. H., et al. Identification of Clostridium difficile as a cause of pseudomembranous colitis. Br Med J. 1 (6114), 695 (1978).
- Bartlett, J. G. Narrative review: the new epidemic of Clostridium difficile-associated enteric disease. Ann Intern Med. 145 (10), 758-764 (2006).
- Kelly, C. P., LaMont, J. T. Clostridium difficile--more difficult than ever. N Engl J Med. 359 (18), 1932-1940 (2008).
- Ünal, C. M., Steinert, M. Novel therapeutic strategies for Clostridium difficile infections. Expert Opin Ther Targets. 20 (3), 269-285 (2016).
- Kelly, C. P., et al. The Monoclonal Antibody, Bezlotoxumab Targeting C. difficile Toxin B Shows Efficacy in Preventing Recurrent C. difficile Infection (CDI) in Patients at High Risk of Recurrence or of CDI-Related Adverse Outcomes. Gastroenterology. 150 (4), S122 (2016).
- Tsutsumi, L. S., Owusu, Y. B., Hurdle, J. G., Sun, D. Progress in the discovery of treatments for C. difficile infection: A clinical and medicinal chemistry review. Curr Top Med Chem. 14 (1), 152-175 (2014).
- Hensbergen, P. J., et al. Clostridium difficile secreted Pro-Pro endopeptidase PPEP-1 (ZMP1/CD2830) modulates adhesion through cleavage of the collagen binding protein CD2831. FEBS Lett. 589 (24), 3952-3958 (2015).
- Cafardi, V., et al. Identification of a novel zinc metalloprotease through a global analysis of Clostridium difficile extracellular proteins. PLoS One. 8 (11), e81306 (2013).
- Hensbergen, P. J., et al. A novel secreted metalloprotease (CD2830) from Clostridium difficile cleaves specific proline sequences in LPXTG cell surface proteins. Mol Cell Proteomics. 13 (5), 1231-1244 (2014).
- Schacherl, M., Pichlo, C., Neundorf, I., Baumann, U. Structural Basis of Proline-Proline Peptide Bond Specificity of the Metalloprotease Zmp1 Implicated in Motility of Clostridium difficile. Structure. 23 (9), 1632-1642 (2015).
- Rawlings, N. D., Waller, M., Barrett, A. J., Bateman, A. MEROPS: the database of proteolytic enzymes, their substrates and inhibitors. Nucleic Acids Res. 42 (Release 10.0), D503-D509 (2014).
- Bergfors, T. Seeds to crystals. J Struct Biol. 142 (1), 66-76 (2003).
- Dauter, Z., Dauter, M., Dodson, E. Jolly SAD. Acta Crystallogr D Biol Crystallogr. 58 (Pt 3), 494-506 (2002).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Kabsch, W. XDS. Acta Crystallogr D Biol Crystallogr. 66 (Pt 2), 125-132 (2010).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallogr D Biol Crystallogr. 66, 213-221 (2010).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 486-501 (2010).
- . . The PyMOL Molecular Graphics System. , (2002).
- Pettersen, E. F., et al. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25 (13), 1605-1612 (2004).
- Wang, B. C. Resolution of phase ambiguity in macromolecular crystallography. Methods Enzymol. 115, 90-112 (1985).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. Phaser crystallographic software. J Appl Crystallogr. 40 (Pt 4), 658-674 (2007).
- Zwart, P. H., et al. Automated structure solution with the PHENIX suite. Methods Mol Biol. 426, 419-435 (2008).
- Zheng, H., Handing, K. B., Zimmerman, M. D., Shabalin, I. G., Almo, S. C., Minor, W. X-ray crystallography over the past decade for novel drug discovery - where are we heading next?. Expert Opin Drug Discov. 10 (9), 975-989 (2015).
- Rubino, J. T., et al. Structural characterization of zinc-bound Zmp1, a zinc-dependent metalloprotease secreted by Clostridium difficile. J Biol Inorg Chem. 21 (2), 185-196 (2016).
- Carson, M., Johnson, D. H., McDonald, H., Brouillette, C., Delucas, L. J. His-tag impact on structure. Acta Crystallogr D Biol Crystallogr. 63 (Pt 3), 295-301 (2007).
- Gasteiger, E., Walker, J. M., et al. Protein Identification and Analysis Tools on the ExPASy Server. The Proteomics Protocols Handbook. , 571-607 (2005).
- Dummler, A., Lawrence, A. M., de Marco, A. Simplified screening for the detection of soluble fusion constructs expressed in E. coli using a modular set of vectors. Microb Cell Fact. 4, 34 (2005).
- Stura, E. A., Wilson, I. A. Applications of the streak seeding technique in protein crystallization. J Crys Growth. 110 (1), 270-282 (1991).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados