É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Mapear o sítio de ligação de um Aptamer no ATP Usando MicroScale termoforese
Neste Artigo
Resumo
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Resumo
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introdução
A interacção entre as moléculas é a base da natureza. Assim, os cientistas em diversas áreas de pesquisa básica e aplicada tentar entender os princípios fundamentais de interacções moleculares de diferentes tipos. MicroScale termoforese (MST) permite aos cientistas realizar a caracterização rápida, precisa e de custo eficiente e de qualidade controlada de interações moleculares em solução, com uma livre escolha de buffers. Já existem mais de 1.000 publicações utilizando MST, a partir de 2016 sozinho, descrevendo diferentes tipos de análises, incluindo rastreios de bibliotecas, a ligação validações de eventos, ensaios de competição, e experimentos com múltiplos parceiros de ligação 1-8. Em geral, o MST permite o estudo dos parâmetros de ligação clássicos, tais como a afinidade de ligação (pM para mM), estequiometria, e termodinâmica, de qualquer tipo de interacção molecular. Uma grande vantagem de MST é a possibilidade de estudar eventos de ligação independentes do tamanho dos parceiros de interacção. mesmo chalinteracções entre lenging aptâmeros de ácidos nucleicos pequenos (15-30 nt) e alvos, tais como pequenas moléculas, drogas, antibióticos, ou metabolitos podem ser quantificados.
Atuais tecnologias state-of-the-art para caracterizar as interações aptâmero-alvo são ou lab-intenso e altamente complexa ou não quantificar aptâmero-pequena molécula interações 9,10. Superfície Plasmon Resonance (SPR) à base de ensaios de 11,12 e abordagens calorimétricos verdadeiramente livre de rótulo, como Calorimetria isotérmica de titulação (ITC) 13-15, eluição isocrática 16, o equilíbrio infiltração 17,18, em linha sondagem 19, distribuição do gel mudar ensaios, stopped- fluxo de fluorescência espectroscopia 20,21, anisotropia de fluorescência (FA) 22,23, uma única molécula de fluorescência de imagem 24,25, e Bio-camada de interferometria (BLI) 26 também são ou imprecisa ou incompatível com a molécula do aptâmero-small interacções. Outros principal questões de estes métodos são de baixa sensibilidade, o consumo elevado de amostras, imobilização, as limitações de transporte de massa em superfícies, e / ou restrições de buffer. Apenas algumas destas tecnologias oferecem controles integrados para efeitos de agregação e de adsorção.
MST representa uma ferramenta poderosa para os cientistas para superar esta limitação para estudar as interacções entre os aptâmeros e pequenas moléculas 27-29, bem como outros objectivos, tais como proteínas de 30-33. A tecnologia baseia-se no movimento de moléculas através de gradientes de temperatura. Este movimento dirigido, chamado "termoforese," depende do tamanho, carga, e camada de hidratação da molécula 34,35. A ligação de um ligando para a molécula irá alterar directamente, pelo menos, um destes parâmetros, o que resulta em uma mobilidade alterada thermophoretic. Ligantes com tamanhos pequenos podem não ter um impacto considerável em termos de mudança de tamanho do não ligado ao estado ligado, mas eles podem ter dr efeitos AMATIC sobre a camada de hidratação e / ou carga. As mudanças no movimento das moléculas após thermophoretic interacções com o parceiro de ligação permite a quantificação dos parâmetros de ligação de base 2,7,34,36,37.
Tal como representado na Figura 1A, o dispositivo consiste MST de um laser infravermelho focalizada sobre a amostra, dentro dos capilares de vidro utilizando a mesma óptica como para a detecção de fluorescência. O movimento thermophoretic de proteínas através da fluorescência intrínseca dos resíduos de triptofano 6 ou de um parceiro de interacção 3,8 marcado por fluorescência podem ser monitorizadas enquanto o laser estabelece um gradiente de temperatura (AT de 2-6 ° C). A diferença de temperatura, resultando em espaço, AT, conduz à depleção ou da acumulação de moléculas na zona de temperatura elevada, que pode ser quantificado pelo coeficiente Soret (S T):
g "/>
c representa quente a concentração na região aquecida, e c frio é a concentração na região fria inicial.
Como mostrado na Figura 1B, uma típica MST os resultados da experiência em um perfil de movimento MST (traço do tempo), que consiste em diferentes fases, que podem ser separadas pelos respectivos prazos. A fluorescência é medida inicial nos primeiros 5 s na ausência do gradiente de temperatura para definir a fluorescência a partir preciso e para verificar a fotobranqueamento ou photoenhancement. O salto de temperatura (T-Jump) representa a fase em que a fluorescência mudanças antes do movimento thermophoretic. Essa redução inicial na fluorescência depende de mudanças dependentes do calor de fl uorophore rendimento quântico. A fase termoforese segue, em que as reduções de fluorescência (ou aumento), devido ao movimento das moléculas de thermophoretic até que a distribuição em estado estacionário é atingido.O TJump reversa e concomitante difusão de moléculas de volta uorescent fl pode ser observado como indicado na figura 1B após o laser é desligado. De modo a aceder aos parâmetros de ligação de base, diferentes razões molares dos parceiros de interacção são analisados e comparados. Tipicamente, 16 diferentes proporções são estudadas numa experiência MST, enquanto que a molécula visível óptico é mantido constante e é fornecido com uma quantidade crescente de ligando não marcado. A interacção entre os dois parceiros de ligação induz alterações na termoforese, e, assim, na fluorescência normalizada, F norma, que é calculada como se segue:
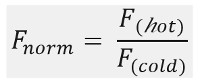
F quente e fria F representam em média intensidades fluorescência na de fi nidas pontos no tempo dos traços do MST. As afinidades de ligação (Kd ou valores de EC50) pode ser calculada por Curve encaixe (Figura 1C).
No geral, o MST é uma ferramenta poderosa para estudar interações moleculares de qualquer tipo. Este manuscrito oferece um protocolo para caracterizar a interação desafio entre a pequena molécula de trifosfato de adenosina (ATP; 0,5 kDa) e do 25-nt curta ssDNA aptâmero DH25.42 (7,9 kDa). Ao longo do manuscrito, o local de ligação do aptâmero na molécula de ATP é mapeado para baixo para o grupo de adenina do ATP.
Protocolo
1. Preparação de Trabalho Aptamer a Bolsa
- Seguir as instruções do fabricante e dissolve-se o oligonucleótido (5-Cy5-CCTG GGGGAGTATTGCGGAGGAAGG-3, a sequência de referência 18) em água, alcançando uma concentração final de 100 uM.
- Preparar a solução de aptâmero de trabalho por diluição da oligonucleótido a 200 nm com tampão de ligação (Tris 20 mM, pH 7,6; NaCl 300 mM; MgCl2 5 mM; 0,01% Tween 20).
- Incubar a mistura durante 2 min a 90 ° C, deixar a amostra arrefecer imediatamente para baixo sobre o gelo, e usar a amostra à temperatura ambiente.
2. Preparação da série de diluição Ligand
- Para cada ligando (trifosfato de adenosina (ATP), difosfato de adenosina (ADP), monofosfato de adenosina (AMP), adenina, guanosina trifosfato (GTP), trifosfato de citosina (CTP), desoxiadenosina-trifosfato (dATP), e s-adenosilmetionina (SAM) ; estoque 10 mM cada), preparar uma diluti série de 16 passosem em 200 ul tubos micro reação.
NOTA: A centrifugação de ações ligando por 5 min a 14.000 xg pode ajudar a remover agregados. Baixo volume, tubos de reacção de ligação baixas são recomendadas para evitar a adsorção de moléculas com as paredes do tubo. - Comece com uma concentração máxima de, pelo menos, 50 vezes mais elevada do que a afinidade estimada e reduzir a concentração do ligando em 50% em cada passo de diluição.
NOTA: A ferramenta concentração localizador implementado no software de controle simula dados de ligação e ajuda a encontrar o intervalo de concentração correta para a série de diluição. - Encher 20 ul de estoque o ligando (10 mM) no tubo 1. Adicionar 10 uL de tampão de ligação em tubos de reacção aptâmero micro 2-16.
- Transferência de 10 ml de tubo 1 para o tubo 2 e misturar bem, pipetando para cima e para baixo várias vezes. Transferir 10 mL para o próximo tubo e repetir esta diluição para os tubos restantes.
- Descartar o excesso de 10 ul a partir do último tubo. Evitar qualquer befeitos de diluição pode ser prejudicado. O tampão no tubo 1 e em tubos de 2-16 deve ser idêntica.
3. Preparação da Mistura final Reacção
- Preparar as reacções de ligação individuais com um volume de 20 ul (10 ul de solução de trabalho do aptâmero + 10 ul de diluição do respectivo ligando) para minimizar erros de pipetagem. Um volume de apenas 4 l é su fi ciente para encher o capilar.
- Adicionar 10 ul da solução de trabalho de 200 nM de aptâmero de 10 ul de cada diluição de ligando e misturar bem por pipetagem para cima e para baixo várias vezes.
- Incubar as amostras durante 5 minutos a temperatura ambiente e encher as amostras padrão em capilares por meio de imersão dos capilares para a amostra. tempos de incubação mais longos podem ser necessários para algumas interacções; no entanto, 5 minutos é adequado para a maioria. Toque os capilares apenas sobre os lados, não na parte do meio, em que a medição óptica irá ser feita.
- Coloque os capilares Onto thbandeja capilar e e iniciar o dispositivo MST.
4. Iniciar o dispositivo MST
NOTA: O aparelho oferece dois pacotes de software pré-instalado, o '' "software para a configuração técnica das condições experimentais e de controle '' análise de" software para a interpretação dos dados produzidos.
- Antes de colocar a bandeja do capilar para o dispositivo MST, inicie o software de controle e ajustar a temperatura desejada geral, selecionando '' permitem controle de temperatura manual "no '' controle de temperatura" menu suspenso. Ajustar a temperatura para 25 ° C no presente modo.
NOTA: Os instrumentos MST pode ser 22 a 45 ° C de temperatura controlada. - Aguarde até que a temperatura para atingir o nível esperado e, em seguida, colocar a bandeja do capilar para o dispositivo MST.
- Ajuste o canal de LED para '' vermelho "para corantes Cy5 e ajustar a potência de LED para ganhar uma fl uorescence sinal de 300 a 1000 unidades de fluorescência a do dispositivo com um sensor de MST padrão. 25% de energia LED é utilizado neste estudo.
NOTA: 6.000 a 18.000 unidades de fluorescência são recomendados para o MST com um sensor de alta sensibilidade.
5. Capilar de digitalização
- Realize uma verificação capilar para verificar aspectos diferentes de qualidade da amostra, escolhendo a posição capilar no software "Control" e clicar em "Start Scan cap" antes de iniciar a medição MST.
- Inspecione a varredura capilar para fluorescência realce / têmpera e efeitos degola (em forma de U ou achatadas picos) no software.
NOTA: Mais detalhes sobre a detecção e tratamento de fluorescência e efeitos degola pode ser encontrada na discussão.
6. Medição MST
NOTA: Antes de iniciar a medição MST, certifique-se de excluir degola efeitos, realce / efeitos de têmpera, ouerros de pipetagem, e garantir que a digitalização capilar indica que o sinal de fluorescência é su fi ciente. Para mais detalhes, veja a discussão.
- Atribuir os concentrações de ligando a partir da série de diluição para a respectiva posição capilar no software "" controle "Considere o passo de diluição da mistura do aptâmero e o ligando. (1: 1).
- Digite a maior concentração de ligando (5 mM) para capilar # 1, selecione o tipo de diluição correta (aqui, 1: 1), clique sobre a concentração máxima e use a função arrastar para atribuir automaticamente as concentrações restantes capilares # 2- 16. A concentração mais baixa é de 152,6 nm.
- Introduza a concentração de aptâmero uorescente FL (aqui, 100 nM) na respectiva secção do software de controlo.
- Use as configurações padrão, que detectam a fluorescência durante 5 segundos, registrar o MST durante 30 segundos, e registrar a fluorescência por mais 5 segundos após a inativação do laser t o monitorar a difusão volta das moléculas.
- Ajustar a potência do laser para 20% na respectiva secção do software de controlo.
NOTA: A fim de receber a melhor relação sinal-ruído e para evitar os efeitos não específicos, uma potência de laser de 20-40% é recomendada. Em casos específicos, a potência do laser maior pode ser necessária para obter uma boa separação de moléculas não ligadas e ligados. - Salve o experimento depois de selecionar a pasta de destino e iniciar a medição do MST, premindo o botão '' de medição Iniciar MST ".
NOTA: O arquivo .ntp será gerado na pasta de destino. Usando esta configuração, uma medida dura 10-15 min. - Repetir o procedimento experimental pelo menos duas vezes para uma determinação mais precisa do valor de CE 50.
NOTA: Para testar a reprodutibilidade técnica, os mesmos capilares podem ser digitalizados várias vezes (repete técnicas).
7. Análise de Dados MST
nt "> NOTA: O software de análise permite a análise de dados sobre a fl y durante a medição O software de análise de traça os traços de tempo MST e mudanças no normalizada fluorescência (norma F) versus a concentração do ligando 37..- Inicie o software de análise de MST (Análise MO.Affinity) e carregar o arquivo .ntp da pasta de destino. Selecione "MST" como o tipo de análise no menu de seleção de dados.
Nota: Em caso de efeitos de fluorescência dependente do ligando, a fluorescência inicial pode ser escolhida para análise. - Adicionar o respectivo prazo (s) técnica ou biológica com uma nova análise por arrastar-e-soltar ou pressionando o botão "+" abaixo do respectivo prazo experimental.
- Pressione o botão informações abaixo do respectivo prazo experimental para obter informações sobre as propriedades do experimento, os traços do MST, varredura capilar, forma capilar, fluorescência inicial e taxa de branqueamento.
Nota: Estes dados brutos podem alpor isso ser inspecionados em etapas posteriores da análise. - Inspecione visualmente os vestígios do MST para efeitos de agregação e precipitação, visível como colisões e picos.
NOTA: Para mais informações sobre a detecção e tratamento dos efeitos de agregação, leia a discussão. - inspecionar visualmente a varredura capilar e a sobreposição de forma capilar para efeitos de adsorção, visível como picos achatadas ou em forma de U. inspecionar visualmente a varredura capilar e a fluorescência inicial para efeitos de fluorescência. inspecionar visualmente a taxa de branqueamento para efeitos fotodegradação.
- Alternar para o modo de dose-resposta e altere a configuração de análise para o modo "expert" pressionando o respectivo botão. Selecione "T-Jump", como a estratégia de avaliação MST.
- Selecione o modelo de "Hill" para a curva fi tting. Os parâmetros de ligação será automaticamente calculado. Normalizar os dados, escolhendo o respectivo tipo de normalização no menu "comparar os resultados". exportar odados tanto como um .xls ou .pdf.
NOTA: A tabela abaixo do gráfico de ligação resume os parâmetros obrigatórios calculados.
Resultados
Neste estudo, o MST foi aplicado para caracterizar o local de ligação do aptâmero ADN DH25.42 18 em ATP. Em contraste com outros estudos que caracterizam a interacção de ATP ou ATP-imitando as moléculas pequenas com as proteínas marcadas aleatoriamente com um ou mais fluoróforos 38-40, este estudo inclui uma versão marcada do aptâmero 7,9 kDa ADNcs com uma molécula de Cy5 na extremidade 5' . Diferentes derivados de ATP e de moléculas relacionadas, to...
Discussão
controles de qualidade:
Inespecífica colagem / adsorção do material da amostra para as superfícies, bem como os efeitos de agregação, ter uma influência dramática na qualidade dos dados de afinidade. No entanto, apenas algumas tecnologias state-of-the-art oferecem opções precisas e rápidas para monitorar e evitar esses efeitos. MST oferece controlos de qualidade integrados que detectam e ajudar a ultrapassar estes problemas, permitindo a optimização gradual da configuração técni...
Divulgações
CE e TS são funcionários da 2bind GmbH, que fornece serviços de análise biofísicos. taxas de publicação do video-artigo são pagos por 2bind GmbH.
Agradecimentos
Os autores não têm confirmações.
Materiais
| Name | Company | Catalog Number | Comments |
| Aptamer binding buffer | 20 mM Tris pH 7.6; 300 mM NaCl; 5 mM MgCl2; 0.01% Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT- ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at - 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at - 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at - 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at - 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at - 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at - 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at - 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at - 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
Referências
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochemistry. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochemistry. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochemistry. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5'-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados