Method Article
Um de alta capacidade: Plataforma celular Microarray para Análise correlativa da diferenciação celular e forças de tração
Neste Artigo
Resumo
diferenciação celular é regulada por uma série de fatores do microambiente, incluindo ambas as propriedades da composição da matriz e material de substrato. Descrevemos aqui uma técnica utilizando microarrays de células em conjunto com microscopia de força de tracção para avaliar tanto a diferenciação de células e as interacções célula-substrato biomecânicas como uma função do contexto microambiental.
Resumo
microarrays celulares microfabricated, que consistem em combinações impressa contato de biomoléculas em uma superfície hidrogel elástico, fornecer um alto throughput sistema rigidamente controlado, projetado para medir o impacto de sinais bioquímicos dispostas na diferenciação celular. Esforços recentes utilizando microarrays de células têm demonstrado a sua utilidade para estudos combinatórios em que muitos fatores do microambiente são apresentados em paralelo. No entanto, estes esforços têm focado principalmente na investigação dos efeitos dos sinais bioquímicos sobre as respostas celulares. Aqui, apresentamos uma plataforma de microarray célula com propriedades materiais ajustáveis para avaliar tanto a diferenciação celular por interações de imunofluorescência e célula-substrato biomecânica por microscopia de força de tração. Para fazer isso, nós desenvolvemos dois formatos diferentes, utilizando hidrogéis de poliacrilamida de diferentes módulo de Young fabricado em ambos lâminas de microscópio ou placas de Petri com fundo de vidro. Nós fornecemos bespráticas t e solução de problemas para a fabricação de microarrays nesses substratos de hidrogel, a cultura de células subsequente sobre microarrays, e aquisição de dados. Esta plataforma está bem adequada para uso em investigações de processos biológicos para os quais tanto bioquímicos (por exemplo, a composição da matriz extracelular) e biofísico (por exemplo, rigidez) do substrato pistas podem desempenhar significativa, intersectando papéis.
Introdução
As interacções entre células vizinhas e factores micro-ambientais mediar uma grande variedade de processos biológicos ao longo do desenvolvimento, homeostase e doenças patogénese 1, 2, 3, 4. Estas interacções incluem microambiente entrega de factores solúveis para as células, a ligação de células-matriz, e interacções célula-célula através da ligação ligando-receptor. Para além das considerações bioquímicas acima, os parâmetros biofísicas, tais como as propriedades mecânicas do substrato (por exemplo, o módulo de Young, a porosidade) e forma da célula, e mecanotransdução jusante associada têm cada vez mais ganhou reconhecimento como os principais mediadores da diferenciação celular da 5, 6, 7, 8, 9 , 10. Sinais resultantes dessas interações microambiente servem como entradas para redes de genes e vias de sinalização. Além disso, esses componentes da célula-intrínseca também fornecer feedback para o microambiente via fatores secretados e enzimas que degradam a matriz, completando um ciclo de co-regulação complexa entre programas genéticos de células-intrínseca e fatores do microambiente de células-extrínseca 5, 11, 12.
A utilização de sistemas de engenharia para a apresentação controlada de factores micro-ambientais mostrou-se útil numa variedade de contextos diferentes 13, 14, 15. Sistemas microfabricados em particular, têm facilitado modelação espacial precisa de proteínas e células, bem como a análise altamente paralelizado através miniaturização 13, 16, 17, 18, 19, 20, 21, 22. Microarrays celulares representam um tal sistema microfabricado em que combinações de biomoléculas são para um hidrogel de poliacrilamida substrato elástico 23, 24, 25 impressos-contacto. A inclusão de componentes de células-adesivo (nomeadamente proteínas da matriz) permite a adesão sustentada cultura de células e em microarrays, que é frequentemente seguida de análise a jusante através de imunocitoquímica e repórteres fluorescentes. Microarrays celulares foram produtiva orientada para alcançar uma melhor compreensão do fenótipo das células do fígado 23, 26, diferenciação neural precursor 27, Mammardecisões y progenitor destino 28, células-tronco embrionárias de manutenção / diferenciação 23, 29, 30, metástase de câncer de pulmão 31 e resposta terapêutica em pacientes com melanoma 32. Nós demonstramos recentemente, a utilização de microarranjos de células para definir o papel de matriz extracelular (ECM) na composição de proteína especificação endoderme 33, fígado progenitoras diferenciação 34, 35, e de tumor de pulmão de células 36 a resposta à droga. Nestes trabalhos, temos nos concentrado em estender as capacidades combinatórias da plataforma matriz e explorar as interseções de sinalização celular-intrínseca com composição da matriz extracelular e biomecânica. Além disso, temos implementado leituras biofísicos nesta plataforma de matriz para fornecer a capacidade de quantitativamente caracteterize o papel da contractilidade celular na diferenciação processa 35. Para fazer isso, integramos microscopia de força de tração (TFM) com microarrays de células para permitir a avaliação de alto rendimento de tração gerada por células. TFM é um método amplamente utilizado para medir forças de tração gerada por células e forneceu insights significativos sobre a coordenação de uma única célula e função em nível de tecido com a composição e biomecânica do microambiente local, 37, 38, 39, 40. Assim, combinando TFM com microarrays celulares fornece um sistema de alto rendimento para medir principais parâmetros biofísicos, fisiologicamente relevantes.
A plataforma de microarray celular aqui descrita consiste em quatro seções: a fabricação de substratos de poliacrilamida, fabricação de matrizes, cultura de células e leitura de ensaio e análise de dados. VejoA Figura 1 para um resumo esquemático dos primeiros três secções experimentais; veja a Figura 2 para um resumo esquemático da secção final com foco em análise de dados de imunofluorescência. A fim de adaptar a plataforma de microarray celular para estudos de interações-substrato celular biomecânicos, foram utilizados substratos de poliacrilamida de módulo ajustável de Young, mas porosidade semelhante, por Wen et al. 41. Para habilitar medições TFM de forças exercidas por células no seu substrato, foi implementado um formato de placa de Petri com fundo de vidro, além do microscópio de vidro grosso lâminas freqüentemente utilizada por outros grupos. Assim, esta plataforma de microarray célula é capaz de medições paralelas de diferenciação celular, através de imunofluorescência em lâminas de microscópio e as forças geradas por celulares via TFM em pratos com fundo de vidro separados. Temos também aplicadas várias melhorias para a abordagem analítica utilizada com microarrays celulares. specifically, em vez de paramétrico Z-pontuação da intensidade global ilha, medir a intensidade de uma única célula e aplicar a normalização quantil, a fim de explicar as distribuições não-normais e com mais precisão descrever o comportamento celular. Acreditamos que essas melhorias fornecem uma utilidade particular para com investigações de processos biológicos em que sinais bioquímicos e biofísicos jogar, papéis de interseção significativos. Além disso, os nossos melhorias analíticos permitir a aplicação de microarrays de células para estudos de uma série de funções celulares para os quais de uma única célula e comportamento de nível população divergem.
Protocolo
1. Fabricação de poliacrilamida Substratos
- substratos de vidro limpas - quer lâminas de microscópio padrão para imunofluorescência endpoint ou com fundo de vidro 35 mm pratos de Petri para a TFM - a fim de assegurar o ótimo poliacrilamida fabricação e integridade hidrogel durante a cultura de células. Alternativamente, o uso pré-limpas substratos de vidro.
- Mergulhar em substratos de vidro de 0,25% v / v de Triton X-100 em água destilada (dH2O). Coloque substratos num agitador orbital durante 30 minutos.
- Remover Triton solução X-100 e enxaguar substratos 5 vezes com substratos dH 2 O. Deixe imersas na lavagem final e coloque em um agitador orbital durante 30 minutos.
- Remover dH2O e mergulhe substratos em acetona. Coloque substratos num agitador orbital durante 30 minutos.
- Remover a acetona e mergulhe substratos em metanol. Coloque substratos num agitador orbital durante 30 minutos.
- Remover o metanol e lavar substratos 5 vezes com dH 2 substratos O. imerso em NaOH 0,05 N e coloque em um agitador orbital durante 1 h.
CUIDADO: NaOH é muito cáustico e pode causar queimaduras na pele e lesões oculares graves. Usar luvas de protecção, roupas e óculos de protecção. - Remover e lavar uma solução de NaOH substratos 5 vezes com dH2O Uso de ar comprimido filtrado para secar substratos e cozer a 110 ° C sobre uma placa quente até que seco (5 - 15 minutos). substratos limpos podem ser armazenados à temperatura ambiente indefinidamente.
- Silanizar substratos de vidro limpo, a fim de garantir a fixação do hidrogel de poliacrilamida.
- Imergir substratos de vidro limpas na preparada de fresco de 2% v / v de metacrilato de 3- (trimetoxissilil) propilo (3-TPM) em etanol. Coloque substratos num agitador orbital durante 30 minutos.
CUIDADO: 3-TPM é um combustível líquido. Manter longe do calor, faíscas, chamas e superfícies quentes e utilizar apenas em uma coifa química. - Remoção da solução de 3-TPM e mergulhar substratos em ethanol. Coloque substratos num agitador orbital por 5 min.
- Use ar comprimido filtrado para secar os substratos e cozer a 110 ° C sobre uma placa quente até que seco (5 - 15 minutos). substratos silanizado pode ser armazenado à temperatura ambiente durante até um mês.
- Imergir substratos de vidro limpas na preparada de fresco de 2% v / v de metacrilato de 3- (trimetoxissilil) propilo (3-TPM) em etanol. Coloque substratos num agitador orbital durante 30 minutos.
- Opção 1: Fabricar hidrogéis de poliacrilamida em lâminas de microscópio silanizadas para imunofluorescência endpoint.
- Prepara-se uma solução de pré-polímero em dH2O com o / percentagem de bisacrilamida desejado acrilamida (w / v), proporção de fabricar substratos com módulos de 4 kPa (4% de acrilamida, 0,4% de bisacrilamida), 13 kPa (6% de acrilamida, 0,45 de Young % bisacrilamida), ou 30 kPa (8% acrilamida, 0,55% bisacrilamida) e porosidade semelhante, por Wen et ai. 41. solução Vortex até claro e filtro com uma seringa de 0,2 mm. As soluções de pré-polímero pode ser armazenado a 4 ° C durante 3 meses.
ATENÇÃO: A exposição à acrilamida ou bisacrylamide pode resultar em toxicidade aguda, Neurotoxicity, e irritação. Usar luvas de protecção, roupas e óculos de protecção. - Prepara-se uma solução de foto-iniciador de 20% w / v de Irgacure 2959 em metanol. Esta solução fotoiniciador não pode ser armazenado e deve ser preparado fresco de cada vez.
- Misturar o pré-polímero e soluções de fotoiniciador numa proporção de 9:: 1 (fotoiniciador pré-polímero). desgaseifica opcionalmente com uma câmara de vácuo durante 15 min para remover as bolhas.
- Colocar as lâminas silanizadas para um tabuleiro de secagem de vidro e pipetar 100 mL de 9: 1 de pré-polímero: solução de foto-iniciador para cada lâmina. Gentilmente cobrir cada lâmina com uma lamela de 22 mm × 60, evitando a criação de bolhas. Note-se que a lamela impede que a inibição da reacção de polimerização por oxigénio.
- Tabuleiro de secagem lugar num reticulador de UV e expor as lâminas a 365 nm UV A durante 10 min (4 W / m 2). Otimizar o tempo de polimerização, conforme necessário. Exposições mais longas dificuldade risco de retirar a lamela devido à overpolymerization. exposições mais curtas runderpolymerization isk e baixa estabilidade hidrogel.
- Mergulhe hidrogéis em dH2O por 5 min. Remover lamelas com uma navalha, tomando cuidado para não danificar os hidrogéis polimerizado.
- Deixar hidrogeles em dH2O à temperatura ambiente durante 1-3 d, mudando dH2O diária. Desidratar hidrogéis a 50 ° C em uma placa quente até secar (15 - 30 min) e armazenar em temperatura ambiente por até 3 meses.
- Prepara-se uma solução de pré-polímero em dH2O com o / percentagem de bisacrilamida desejado acrilamida (w / v), proporção de fabricar substratos com módulos de 4 kPa (4% de acrilamida, 0,4% de bisacrilamida), 13 kPa (6% de acrilamida, 0,45 de Young % bisacrilamida), ou 30 kPa (8% acrilamida, 0,55% bisacrilamida) e porosidade semelhante, por Wen et ai. 41. solução Vortex até claro e filtro com uma seringa de 0,2 mm. As soluções de pré-polímero pode ser armazenado a 4 ° C durante 3 meses.
- Opção 2: Fabricar hidrogéis de poliacrilamida contendo talão fluorescentes sobre com fundo de vidro placas de Petri de 35 mm silanizadas para avaliação ao vivo de interações-substrato celular utilizando TFM.
- Sonicar uma solução stock de 1 uM esferas fluorescentes durante 15 minutos para dispersar os agregados.
- Prepara-se uma solução de pré-polímero em dH2O com o / percentagem de bisacrilamida desejado acrilamida (w / v), proporção de fabricar substratos com módulos de 4 kPa (4% de acrilamida, 0,4% de bisacrilamida), 13 kPa (6% de acrilamida, 0 de Young0,45% bisacrilamida), ou 30 kPa (8% acrilamida, 0,55% bisacrilamida) e porosidade semelhante, por Wen et ai. 41. solução Vortex até claro e filtro com uma seringa de 0,2 mm. As soluções de pré-polímero pode ser armazenado a 4 ° C durante 3 meses.
ATENÇÃO: A exposição à acrilamida ou bisacrylamide pode resultar em toxicidade aguda, neurotoxicidade e irritação. Usar luvas de protecção, roupas e óculos de protecção. - Adicionar pérolas fluorescentes para a solução de pré-polímero a uma concentração final de 0,2% v / v e agitar com vortex para misturar.
- Prepara-se uma solução de foto-iniciador de 20% w / v de Irgacure 2959 em metanol. Esta solução fotoiniciador não pode ser armazenado e deve ser preparado fresco de cada vez.
- Misturar as soluções de pré-polímero / grânulo e fotoiniciadores em uma mistura 9: 1 (pré-polímero / grânulo: fotoiniciador) proporção. desgaseifica opcionalmente com uma câmara de vácuo durante 15 min para remover as bolhas.
- Coloque silanizadas mm com fundo de vidro pratos de Petri 35 em uma bandeja de secagem de vidro e pipet 20 mL de 9: solução de foto-iniciador para o centro de cada prato: um pré-polímero / grânulo. Gentilmente cobrir cada lâmina com uma lamela circular 12 mm enquanto evitando a criação de bolhas. Note-se que a lamela impede que a inibição da reacção de polimerização por oxigénio.
- A fim de distribuir as partículas fluorescentes à superfície do hidrogel, inverter os pratos e deixar à temperatura ambiente durante 20 min, por Knoll et ai. 42.
- Enquanto ainda invertido, expor pratos a 365 nm UV A durante 10 min (4 W / m 2). Otimizar o tempo de polimerização, conforme necessário. Exposições mais longas dificuldade risco de retirar a lamela devido à overpolymerization. Shorter underpolymerization exposições de risco e baixa estabilidade hidrogel.
- Imergir hidrogeles em 0,1 M de 4- (2-hidroxietil) -1-piperazinoetanossulfónico (HEPES) Tampão e deixar à temperatura ambiente durante a noite no escuro. Remova cuidadosamente lamelas com uma navalha, tomando cuidado para não danificar o polymehidrogéis rizados.
- Desidratar hidrogéis a 50 ° C sobre uma placa quente até que a seco (15 - 30 min). Os hidrogéis podem ser armazenados à temperatura ambiente no escuro durante 3 meses.
2. Fabricação de Arrays
- Prepare buffers para imprimir biomoléculas. Use o buffer de impressão adequado para as biomoléculas de interesse. factor de crescimento (GF) tampão de impressão é em geral, adequados para outras classes de moléculas, tais como ligandos de célula-célula.
- Para preparar 2 × tampão de ECM impressão proteína, adicionar 164 mg de acetato de sódio e 37,2 mg de ácido etilenodiaminotetracético (EDTA) a 6 mL de dH2O Vortex e incubar a 37 ° C para dissolver completamente. Após solubilização, adicionar 50 ul de pré-aqueceu-se Triton X-100 e 4 mL de glicerol. Vortex e incubar a 37 ° C, para solubilizar novamente. Adicionar 40 - 80 mL de ácido acético glacial, titulando-se a ajustar o pH para 4,8. 2 × tampão de ECM impressão de proteína pode ser armazenada a 4° C durante 1 mês.
CUIDADO: O ácido acético é inflamável e corrosivo. Usar luvas de protecção, roupas e óculos de protecção. - Para preparar 2 × tampão de impressão GF, adicionar 105,5 mg de acetato de sódio e 37,2 mg de EDTA a 6 ml de solução salina tamponada com fosfato (PBS). Vortex e incubar a 37 ° C para dissolver completamente. Após solubilização, adiciona-se 100 mg de 3 - [(3-colamidopropil) dimetilamónio] -1-propanossulfonato (CHAPS) e 4 mL de glicerol. 2 × tampão de impressão GF proteína pode ser armazenada a 4 ° C durante 1 mês.
- Para preparar 2 × tampão de ECM impressão proteína, adicionar 164 mg de acetato de sódio e 37,2 mg de ácido etilenodiaminotetracético (EDTA) a 6 mL de dH2O Vortex e incubar a 37 ° C para dissolver completamente. Após solubilização, adicionar 50 ul de pré-aqueceu-se Triton X-100 e 4 mL de glicerol. Vortex e incubar a 37 ° C, para solubilizar novamente. Adicionar 40 - 80 mL de ácido acético glacial, titulando-se a ajustar o pH para 4,8. 2 × tampão de ECM impressão de proteína pode ser armazenada a 4° C durante 1 mês.
- Prepare placa de origem.
- Em uma de 384 poços de microplacas de fundo em V, combinar volumes iguais de 2 × tampão de impressão com cada solução biomolécula com o dobro da concentração alvo.
NOTA: Uma concentração alvo apropriada de proteínas de ECM mais comuns é de 250 ug / mL enquanto que as concentrações alvo de outros tipos de factores dispostas variam dependendo da retenção no hidrogel e a função biológica. O volume total em cada poço pode serA partir de 5 mL e não é necessário mais de 15 mL. Para além das combinações de biomoléculas de interesse, incluem um marcador fluorescente dispostas de modo a facilitar a análise de imagem a jusante. Use de dextrano conjugado com rodamina (2,5 mg / mL). - Misture cada poço completamente por pipetagem, tomando cuidado para não gerar bolhas. Centrifugar a microplaca fonte durante 1 min a 100 x g. Fabricar micromatrizes utilizando placas de origem preparadas no mesmo dia e armazenados a 4 ° C, até a fabricação de microarray.
- Em uma de 384 poços de microplacas de fundo em V, combinar volumes iguais de 2 × tampão de impressão com cada solução biomolécula com o dobro da concentração alvo.
- pins limpos de acordo com as instruções do fabricante antes de cada execução microarray fabricação. Coloque pinos limpas diretamente no cabeçote do microarrayer.
- Prepare microarrayer e programa usando o software do fabricante. Embora os passos que se seguem são, em parte específica para o microarrayer particular utilizado aqui, a operação da maior parte das microarrayers é semelhante.
- Ligar o aparelho humidificador, ajustar o ponto de ajuste a 65% RH (sem-condensing) e aguarde até que o reômetro corresponde ao set point. Coloque placa de origem no adaptador apropriado.
- Desidratar hidrogel substratos a 50 ° C durante 15 min e colocar no adaptador apropriado. O microarrayer tem adaptadores para ambas as lâminas de microscópio e microplacas. Para arraying 35 mm com fundo de vidro placas de Petri, coloque os pratos em uma microplaca de 6 poços e coloque a microplaca no adaptador microplaca na arrayer.
- Ajustar os parâmetros do programa para refletir com precisão o layout da placa de fonte, design matriz, e o formato desejado (por exemplo, lâmina de microscópio ou microplaca contendo 35 mm placas de Petri). Incluem as etapas de lavagem, utilizando tanto sulfóxido de água e dimetil (DMSO) entre cada condição, a fim de evitar o reporte e a contaminação cruzada.
- Comece matriz fabricação, verificando nada menos do que uma vez numa hora em que a umidade não caiu abaixo de 65% RH (sem condensação) e que os pinos não estão entupidos. Se a humidity caiu inesperadamente, pausar arraying para encher o humidificador e limpar tubos associados de condensação. Se os pinos estão entupidos, pausar arraying para limpar os pinos ou não substituir com pinos pré-limpos. Note-se que é possível matriz múltiplos tipos de biomoléculas sequencialmente nos mesmos substratos fornecidos tempo de secagem suficiente (isto é, 4 h até durante a noite).
- Quando o programa estiver concluído, coloque matrizes fabricadas em uma caixa de slides ou microplacas coberto com folha de alumínio à temperatura ambiente e 65% RH (sem condensação) durante a noite. Note-se que pode ser necessário para avaliar a qualidade e retenção matriz usando manchas de proteína geral ou imunofluorescência; veja Brafman et al. para mais detalhes 25.
3. celular Cultura e Ensaio de Leitura
- O dia após a fabricação, coloque substratos dispostos em 4 câmaras pratos (slides de microscópio) ou 6 microplacas (placas de Petri) e mergulhar em 1%v / v de penicilina / estreptomicina, em PBS; usar 4 mL de slides e 3 mL de pratos. Expor à radiação UV C durante 30 min. Troca de penicilina / estreptomicina solução para o meio de cultura celular.
- Coletar e contagem de células. Semente para matrizes em 500 × 03-2 outubro × 10 6 células / matriz em 4 ml por lâmina de microscópio e 3 mL por placa de Petri de 35 milímetros. Incubar as culturas de matriz a 37 ° C e 5% de CO2 durante 2-24 h ou até que a formação de ilhas de células bem povoadas. Ajustar a densidade de semeadura e do tempo como necessário para as suas células e aplicação em particular. Underseeding (ou seja, de baixa densidade ou tempo de envio) pode resultar na população pobre da matriz e resultados biológicos distorcidas. Overseeding (ou seja, alta densidade ou época de semeadura) pode resultar na integridade matriz reduzida devido ao descolamento ilha.
- Depois de permitir que para a formação de ilhas de células, culturas de matriz lavar duas vezes com meio de cultura de células pré-aquecida; mais uma vez usar 4 mL de slides e 3 mL de pratos. optionally adicionar controlos e tratamentos apropriados (por exemplo, inibidores de moléculas pequenas, factores de crescimento, etc.) de interesse do sistema biológico. Mudar os meios das matrizes de cada 1-2 d a fim de manter a concentração de todos os tratamentos. Avaliar a expressão do marcador celular e função das células por imunofluorescência ou célula-substrato interações por TFM dentro de 1-5 d de iniciar culturas de matriz - ver Opção 1 e Opção 2 abaixo.
- Opção 1: Execute imunofluorescência endpoint. Note-se que a imunofluorescência de algumas proteínas podem necessitar de permeabilização mais rigorosa utilizando-se metanol, etanol, ou HCl. Devido ao potencial de danos ao matrizes, avaliar e otimizar cada protocolo permeabilização antes do uso em experimentos em larga escala.
- Aspirar o meio de cultura celular a partir de lâminas de matriz em 4 pratos câmaras e adicionar 4 mL / corrediça de recém-preparado de 4% v / v de paraformaldeído (PFA) em PBS. Incubar durante 15 minutos à temperatura ambiente.
CUIDADO: Exposure a PFA pode resultar em toxicidade aguda e também pode irritar ou corroer pele em contato. Usar luvas de protecção, roupas e proteção para os olhos e utilizar apenas em uma coifa química. - Aspirar solução PFA e lave cada lâmina 3 vezes com 4 ml de PBS. Neste ponto, as lâminas podem ser armazenadas a 4 ° C durante 1 semana. É aconselhável, no entanto, continuar através de imunomarcação e montagem no mesmo dia que a fixação, a fim de assegurar a integridade da matriz.
- Aspirar PBS e adicionar 4 mL / corrediça de 0,25% v / v de Triton X-100 em PBS. Incubar durante 10 minutos à temperatura ambiente.
- Aspirar o Triton X-100 a solução e lava-se cada lâmina 3 vezes com 4 ml de PBS. Adicionar 4 ml / corrediça de 5% v / v de soro combinado com a espécie de anticorpo secundário (por exemplo, soro de burro para anticorpos secundários de burro) em PBS e incuba-se à temperatura ambiente durante 1 h.
- Completamente remover a solução de bloqueio de cada slide. Adicionar 500? L / corrediça de anticorpo primário diluído em 5% v / v de soro em PBS. Este volume é suficiente para cobrir matrizes para ambas as incubações de 1 h à temperatura ambiente, bem como as incubações durante a noite a 4 ° C.
- Lavar cada lâmina de matriz 3 vezes com 4 ml de PBS. Cuidadosamente remover a lavagem final e adicionar 500 mL / corrediça do anticorpo secundário apropriado diluído em 5% v / v de soro em PBS.
- Lavar cada lâmina de matriz 3 vezes com 4 ml de PBS. Lavar brevemente com dH2O antes de retirar cuidadosamente corrediças da disposição da solução usando uma pinça. Use um tecido de laboratório para pavio ou seco dH residual 2 O.
- Pipetar 100 L de solução com DAPI montagem em toda a corrediça enquanto confirma visualmente a cobertura completa de toda a matriz.
- Coloque um mm lamela de 22 x 60 sobre a lâmina de montar. Selar as bordas da lamela com unhas polonês claro. Loja no escuro a 4 ° C até imagiologia, o mais tardar, no dia seguinte.
- Imagem matrizes inteiras usando quer um scanner de microarray ou invertida equ microscópio fluorescenteed com um palco robótico. Scanners microarray fornecer leitura mais rápido, mas pode exigir Cy3- ou Cy5 compatíveis com fluoróforos e são muitas vezes de resolução limitada a fim de células individuais (ou seja, 1 - 10 m). microscópios fluorescentes proporcionam a opção de utilizar uma variedade de canais fluorescente e maior resolução (<1 um, ~ 100 vezes no total), mas proporcionar leitura mais lento, dependendo da qualidade do palco robótico e ampliação / objectivo.
- Guardar as imagens capturadas de matrizes inteiras de qualquer método como arquivos TIFF, a fim de evitar a compressão de dados ou perda associada a outros formatos de arquivo (por exemplo, JPG).
- Aspirar o meio de cultura celular a partir de lâminas de matriz em 4 pratos câmaras e adicionar 4 mL / corrediça de recém-preparado de 4% v / v de paraformaldeído (PFA) em PBS. Incubar durante 15 minutos à temperatura ambiente.
- Opção 2: Realizar a avaliação ao vivo de interações-substrato celular utilizando TFM.
- Prepara-se uma solução de 1% v / v de albumina de soro bovino (BSA) e 1% v / v de sulfato de dodecilo de sódio (SDS) em PBS para dissociar as células a partir de substratos durante TFM.
- Mova 35 mm placas de Petri contendo as culturas de matriz paraum (CO 37 ° C, 5% 2) incubados, microscópio de fluorescência invertido, com uma fase robótico para medições TFM.
- Em um prato, marcar as posições (coordenada X, coordenada Y) e se concentrar aviões (coordenada Z) de ilhas de células individuais usando microscopia de contraste de fase.
- Mudar para a microscopia de fluorescência vermelha extrema para visualizar as contas. Voltar para cada uma das posições gravados na etapa anterior e corrigir a coordenada Z do plano de foco de modo que apenas a primeira camada de esferas abaixo da ilha celular está em foco. Salvar as novas coordenadas e proceder à imagem automatizado de todas as ilhas celulares para capturar contraste de fase pré-dissociação e imagens fluorescentes vermelhas distantes.
- Adiciona-se cuidadosamente 150 ul de solução de BSA / SDS para o prato e esperar cinco minutos para permitir a dissociação de células completas a partir do substrato; monitorar dissociação celular utilizando microscopia de contraste de fase.
- Após as ilhas de células foram dissociadas a partir do substrato, de regresso para tele marcou posições e verificar que a primeira camada de contas ainda estão em foco. Se estes grânulos são fora do plano devido à deformação induzida por tracção gerada por células, em seguida, corrigir a coordenada Z do plano de foco de modo que eles são de novo em foco. Save the Z-coordenadas corrigidas e repita imagem automatizado de todas as ilhas para capturar pós-dissociação imagens fluorescentes muito vermelhas.
- Repita os passos 3.5.1 - 3.5.4 para os pratos restantes.
4. Análise de Dados
- Análise de dados de imunofluorescência.
- Processo de imagens adquiridas matriz. Dividir imagens de matriz de compósitos em arquivos contendo canais individuais (ou seja, vermelho, azul ou verde) e converter em imagens TIFF de 8 bits 43, 44. Aplicar binning (por exemplo, 2 × 2 ou 4 × 4) para reduzir o tamanho da imagem para ~ 32 megapixels por canal para reduzir os requisitos de memória durante a análise de uma única célula a jusante da entire imagens de matriz. Veja arquivo de código suplementar intitulado "array_processing.ijm" para uma implementação macro ImageJ dessas etapas de processamento de matriz.
- Observe as coordenadas em pixels do canto superior esquerdo, inferior esquerdo, e marcadores de rodamina conjugada inferior direito dextrano ou condições dispostas. Use essas coordenadas para girar as imagens de 8 bits TIFF para ser perfeitamente vertical e, depois, para anotar de saída a partir da análise de uma única célula com as condições dispostas específicos. Ver Suplementar arquivos de código intitulada "rb_array_rotater.ijm", "rg_array_rotater.ijm", "rgb_array_rotater.ijm", e "array_gridding.ijm" para implementações destas matriz rotativa e gridding passos.
- Realizar análise de uma única célula de binned, rodado imagens TIFF de 8 bits em CellProfiler (versão 2.1.1) 45 utilizando os seguintes módulos: IdentifyPrimaryObjects, IdentifySecondaryObjects e MeasureObjectIntensity. IdentifyPrimaryObjects identifica núcleos, IdentifySecondaryObjects identifica immunolabels associados com cada núcleo da célula, e fornece MeasureObjectIntensity quantificações para ambos os marcadores nucleares e immunolabels.
- dados de uma única célula de saída de todas as três módulos como um arquivo CSV por canal usando o módulo ExportToSpreadsheet para facilitar a análise posterior a jusante. Veja Suplementar arquivos de código intitulado "b_array_image_analysis.cppipe", "gb_array_image_analysis.cppipe", "rb_array_image_analysis.cppipe", e "rgb_array_image_analysis.cppipe" para as condutas CellProfiler execução estes passos para conjuntos de imagens que contêm canais vermelho, verde ou azul.
- Para transformar os dados para explicar a variabilidade experimental e distribuições de uma única célula não-Gauss, aplicar a normalização quantil por replicar biológica 46. Este processo gera uma distribuição compartilhada entre repetições e permite comparações imparciais sobre mudanças na intensidade immunolabel. Além disso, UNLIKE Z-pontuação e outros métodos paramétricos, normalização quantil é não-paramétrica e não assume uma distribuição particular de dados, permitindo a mais representativa análises de comportamento de uma única célula como uma função da condição vestida.
- Traçar dados e interpretar. Dependendo do sistema biológico e hipótese, calcular e definir uma ou mais das seguintes medidas do conjunto para cada condição arrayed:
- Calcular e traçar células por ilha como uma medida combinada da adesão e a sobrevivência durante o decurso da experiência.
- Calcular e traçar a intensidade immunolabel normalizou-quantil como uma medida de destino ou função celular.
- Calcular e representar graficamente a percentagem de células positivas para um immunolabel tal como determinado por intensidade superior a um limiar constante, geralmente 2 DP acima da intensidade média de controlo negativo.
- Alternativamente, as distribuições de enredo de intensidade immunolabel, a fim de examinar e categorizar o comportamento de uma única célulacomo uma função da condição vestida. Essas distribuições podem ainda ser caracterizados utilizando medidas de tendência central (média, mediana, moda) e variação (variância, coeficiente de variação, o fator de Fano) e métodos de teste de hipóteses, tais como o teste de Kolmogorov-Smirnov.
- Análise de dados TFM. Aqui é descrito um método que incorpora um algoritmo previamente desenvolvido por Butler et ai. e Wang et al. 40, 47.
- Use ImageJ batch converter as imagens para arquivos TIFF de 8 bits. Aplicar uma média de arrumação de pixel (por exemplo, 2 x 2) para reduzir o custo computacional e o tempo de análise a jusante. Como algoritmos para TFM têm-se centrado principalmente na análise de uma única célula, o grande interface de substrato celular das ilhas (~ 17,5 × 10 3 uM 2) em comparação com a interface de substrato de células de uma única célula (75? M 2) necessiTates o passo binning.
- Introduza o contraste de fase capturado e imagens fluorescentes vermelhas distantes (ambos pré-dissociação e pós-dissociação) em um ambiente de programação científica, tais como MATLAB e processo usando os algoritmos anteriormente desenvolvidos de Butler et al. e Wang et al. 40, 47.
- Selecione três regiões distantes da ilha celular. Estas regiões são usadas para explicar os deslocamentos devido à imagem ou deriva da amostra.
- Fornecer o factor para converter de pixels para micrómetros (por exemplo, pixels 0,454 / iM), o módulo de Young do substrato (por exemplo, 13 kPa), e o coeficiente de Poisson (por exemplo, 0,48 para os geles de poliacrilamida descrito aqui).
- Para cada ilha, desenhar um limite em torno da periferia para definir as restrições geométricas; todas as forças externas desta fronteira são zerados. Este sistema restrita é razoável dado o grande distance (ou seja, 450 mm) entre as ilhas.
- Calcular a tensão de tração root mean square eo momento contrátil para cada ilha. O momento contráctil é uma medida da tensão residual em toda a ilha celular e foi mostrado para reflectir a força das interacções célula-célula 48. Para cada condição vestiu, os valores médios de raiz quadrada média mais de múltiplas ilhas e repetições biológicas e calcular associado variância para testes de hipóteses. É também possível calcular a média da distribuição de tensões ou momentos ao longo de muitas ilhas para fornecer um mapa representativo de ambas as medidas como uma função da geometria, por exemplo, a distância a partir do centro da ilha.
Resultados
Usando esta plataforma, investigou-se a papel de ambos os sinais bioquímicos e biofísicos na especificação de destino progenitoras do fígado 34, 35. Ligandos / Notch L-conjugado de proteína A mostrou uma melhor retenção e o agrupamento em que o hidrogel de poliacrilamida (Figura 3A) e foram, além disso, capaz de dirigir a diferenciação de células progenitoras do fígado para um destino celular ducto biliar (Figura 3B). Usando a análise de uma única célula, que quantificada a resposta aos ligandos de entalhe para as proteínas da MEC colagénio I, colagénio III, colagénio IV, fibronectina e laminina (Figura 3C), considerando que a resposta de células progenitoras do fígado para o ligando depende também da contexto ECM. Última, utilizamos shRNA knockdown para gerar as progenitoras do fígado sem os ligantes DLL1 e Jag1. A resposta ao ligando Notch dispostas variou de acordo com o presence de qualquer ligando, o que confirma que a capacidade de resposta ao ligando das células-extrínseca é também uma função da expressão de ligando de células-intrínseca (Figura 3D). Além disso, observou-se uma subpopulação distinta de duplo positivo (ALB + / + OPN) em células do knockdown DLL1 (Figura 3D). Juntos, esses resultados representativos mostram: (1) as capacidades combinatórias do formato de matriz, como exemplificado pelo emparelhamento de vários vestiu proteínas da MEC e ligantes Notch com o knockdown de ligantes individuais; (2) a funcionalidade da dispostas não só proteínas ECM, mas também dispostas ligando-célula através de proteína A / G-conjugação mediada; e (3) a implementação de nossa análise de uma única célula e sua capacidade de discernir subpopulações únicas.
Observou-se também que a diferenciação de células progenitoras hepáticas é dependente tanto a rigidez do substrato e a composição de ECM (Figura 4A ong>), especificamente encontrar que o colagénio IV é favorável à diferenciação em ambos os substratos macios e duros, enquanto fibronectina suporta apenas a diferenciação em substratos rígidos (Figura 4B). Mapas de calor representativas das medições da TFM sugeriu que o estresse de tração sustentado a baixa rigidez substrato sobre o colágeno IV promoveu a diferenciação em células do ducto biliar (Figura 4C), um achado confirmado por valores médios root mean square (Figura 4D). Juntos, esses resultados representativos mostram: (1) a integração bem sucedida de TFM com microarrays de células em substratos com uma rigidez ajustável para avaliar tanto o fenótipo celular e o estresse de tração; (2) a coordenação do destino das células progenitoras do fígado, com tanto a composição da matriz e a rigidez do substrato; e (3) a implementação da nossa análise e típicos perfis de estresse de tração TFM em microarrays celulares.
e 1 "src =" / files / ftp_upload / 55362 / 55362fig1.jpg "/>
Figura 1: Visão esquemática que mostra os primeiros três seções experimentais. Na Seção 1, substratos de vidro são limpos e silanizada para facilitar a fabricação de hidrogéis de poliacrilamida. Na secção 2, as combinações de biomoléculas de interesse são preparados de uma fonte de microplacas de 384 poços. A arrayer robótico é então carregado com pinos limpas, a microplaca fonte, e os hidrogéis de poliacrilamida e inicializado, fabricação de matrizes nos hidrogéis. No ponto 3, as células são semeadas sobre os domínios dispostas e deixadas a aderir, após o qual o protocolo de cultura de interesse é realizada. No ponto final, as células ou são fixadas por imunocitoquímica / imunofluorescência ou analisados por meio de TFM. As barras de escala são 75 uM. Por favor clique aqui para ver uma versão maior desta figura.
dentro-page = "1"> 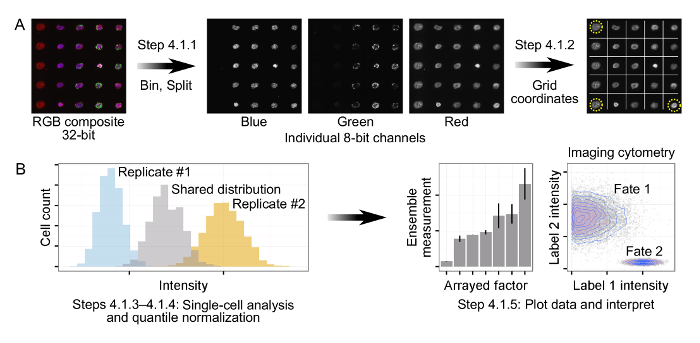
Figura 2: Processamento e Análise de imunofluorescência dados de matrizes. (A) de Azulejo, imagens RGB compostas de 32 bits são primeiro binned e depois dividido em canais de 8 bits individuais. Usando uma combinação de marcadores fluorescentes dispostas e ilhas de células, três cantos da matriz são identificadas para permitir a orientação automatizado e gridding das matrizes. (B) dados de uma única célula é gerado para cada canal dos arrays de entrada. A fim de explicar o desvio experimental, a normalização quantil é aplicado por replicar biológica, produzindo uma única distribuição compartilhada entre todas as repetições. Quantile de dados normalizado é posteriormente plotados e interpretada por cálculo das medições do conjunto (por exemplo, células / ilha, intensidade média, as células percentuais positivos para uma etiqueta) ou análise direta de distribuições de uma única célula.m / files / ftp_upload / 55362 / 55362fig2large.jpg "target =" _ blank "> Clique aqui para ver uma versão maior desta figura.
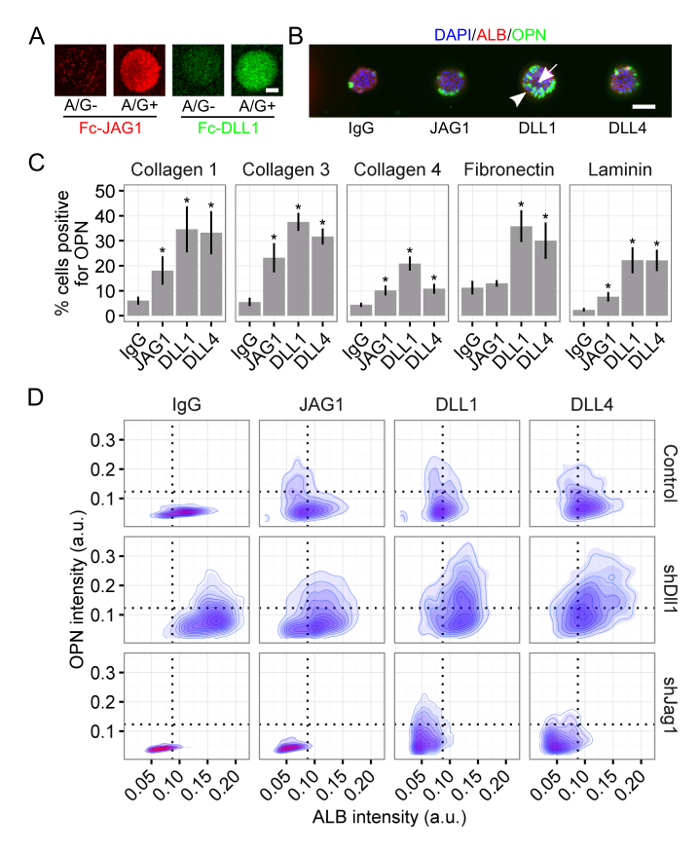
Figura 3: Apresentação Notch Ligando medeia fígado Progenitor diferenciação. (A) Notch Fc recombinante ligantes Jagged-1 (JAG1) e Delta-like 1 (DLL1) apresentaram maior retenção e clustering quando vestida com Proteína A / G. barra de escala é de 50 mm. Progenitores (B) do fígado diferenciadas em células do ducto biliar mediante a apresentação com ligando de Notch. 4 ', 6-diamidino-2-fenilindole (DAPI) é um marcador nuclear, albumina (ALB) é um marcador de célula hepática, e osteopontina (OPN) é um marcador de células do ducto biliar. barra de escala é de 150 mm. (C) Quantificação da percentagem de células positivas para OPN para os ligandos Notch JAG1, DLL1, e semelhante a delta 4 (Dll4) sobre as proteínas da MEC colagénio I, collagen III, colágeno IV, fibronectina e laminina. -Testes T de Student foram realizados contra a IgG de controlo para cada ligando Notch vestida dentro de cada proteína ECM com valores P indicada para P <0,05 (*). (D) Imagem citometria de ALB e OPN para as células no colagénio III apresentados com os ligantes Notch JAG1, DLL1 e DLL4. Progenitoras do fígado sem a Notch ligantes DLL1 e Jag1 (ie, shDll1 e shJag1) foram gerados usando shRNA knockdown. Os dados em (C) apresentados como média ± SEM Esta figura foi modificado a partir do Kaylan et ai. 34. Por favor clique aqui para ver uma versão maior desta figura.
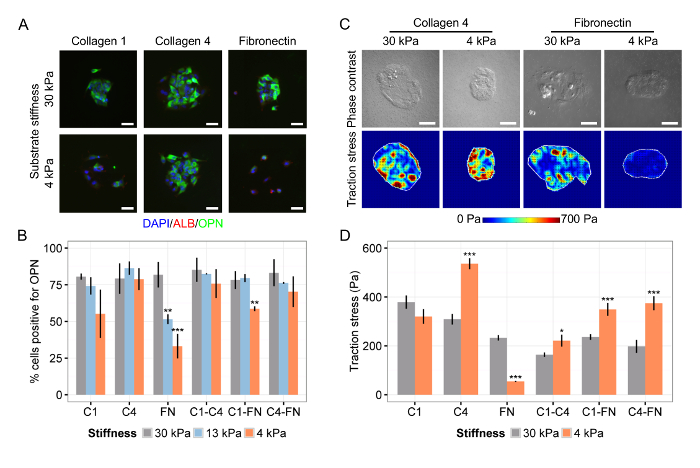
Figura 4: composição da matriz e do substrato Rigidez Coordinate fígado Progenitor diferenciação. (A) a diferenciação de células progenitoras do fígado contra células biliares é dependente tanto a composição ECM e rigidez substrato. DAPI é um marcador nuclear, ALB é um marcador de célula hepática, e o OPN é um marcador de células do ducto biliar. (B) Quantificação da percentagem de células positivas para OPN em substratos de módulo de Young de 30 kPa, 13 kPa, e 4 kPa para o colagénio I (C1), colagénio IV (C4), a fibronectina (FN), e todas as duas combinações diferentes de essas proteínas da MEC. (C) o estresse de tração celular é dependente tanto rigidez substrato e composição ECM. (D) A quantificação dos valores de raiz quadrada média de estresse de tração em substratos do módulo de Young de 30 kPa e 4 kPa para colágeno I (C1), colágeno IV (C4), fibronectina (FN), e todos bidirecionais combinações desses proteínas da MEC. Em (B) e (D), os dados foram apresentados como média ± do SEM e aluno -Testes tForam realizadas contra 30 kPa para cada combinação de ECM com valores P indicada para P <0,05 (*), P <0,01 (**), e P <0,001 (***). As barras de escala são 50 uM. Este valor foi modificado a partir Kourouklis et al. 35. Por favor clique aqui para ver uma versão maior desta figura.
| Seção | Problema | Causas potenciais | Solução |
| 1. Fabricação de poliacrilamida substrato. | Lamela não pode ser removida do hidrogel. | Overpolymerization. | Reduzir o tempo de polimerização para <10 minutos (4 W / m 2). Verifique se crossli UVsaída nker está dentro do intervalo esperado. |
| Pobre de polimerização de poliacrilamida hidrogel. | Underpolymerization. | Aumentar o tempo de polimerização para> 10 minutos (4 W / m 2). Verifique se a saída de reticulação UV está dentro do intervalo esperado. | |
| hidrogeles de poliacrilamida são danificadas após a remoção da lamela. | hidrogéis de poliacrilamida macios são fáceis de danificar. | Observamos diminuição de rendimento hidrogel de fabricação (~ 50%) para o mais macio (ou seja, 4 kPa) hidrogéis em particular. Lidar com hidrogéis suave e aumentar o número de partida para alcançar o rendimento desejado. | |
| 2. Fabricação de matrizes. | morfologia local pobre ou inconsistente. | função umidificador inconsistente. | Verifique se umidificador e re�etro um funcional ao longo de cada tiragem e manter 65% RH. |
| Pinos preso no cabeçote de impressão ou obstruçãoGED. | Limpar a cabeça de impressão para permitir o movimento do pino livre. pins limpar cuidadosamente antes ou depois de cada impressão para remover agregados de canais pinos. | ||
| 3. Cultura de Células e Execução de Ensaio. | descolamento celular ou morte em matrizes após a fixação inicial. | Overseeding e proliferação excessiva. | Reduzir a densidade de sementeira inicial e tempo. Use "manutenção" ou mídia "diferenciação" durante a cultura matriz para reduzir a proliferação celular. |
| Liberação de monômero acrilamida tóxicos do hidrogel. | Soak hidrogeles em dH2O durante pelo menos 3 d para permitir a difusão / libertação de monómero de acrilamida e reduzir a toxicidade celular. | ||
| Células não anexar a matrizes. | Underseeding. | Aumentar a densidade de sementeira inicial e tempo. Use um tipo de célula mais fortemente aderente. | |
| Pobre deposição dematriz ou condição biomolécula. | Pins limpas de partículas e agregados, confirmar parâmetros de impressão, e avaliar manchas de marcadores fluorescentes, por exemplo, dextrano rodamina conjugada. | ||
| A especificidade das interacções célula-matriz. | Diferentes tipos de células aderem especificamente para alguns, mas não outras proteínas de ECM. Testar várias proteínas ECM diferentes, com suas células. | ||
| matriz de armazenamento abaixo do ideal após a fabricação. | Recomendamos armazenar matrizes fabricadas durante a noite a 65% de umidade relativa e temperatura ambiente, em parte para evitar mudanças de fase durante o congelamento. A adesão celular é sensível a ambas humidade, temperatura, e tempo de armazenamento; certifique-se estes parâmetros são consistentes / otimizado para suas experiências. | ||
| Destacamento de hidrogel a partir de substrato de vidro durante a cultura de células. | Pobre limpeza slide e silanização. | Substitua soluções de trabalho para a limpeza de slides esilanização. | |
| hidrogel Overdehydrated. | Não deixe hidrogéis de desidratação em uma chapa quente por mais de 15-30 min. | ||
| 4. Análise de Dados. | Alta variabilidade entre os pontos em duplicado e slides. | A variabilidade na fabricação matriz. | Verifique se os pinos e cabeçote de impressão estão limpos. Confirmar função do umidificador. Visualizar e quantificar local e matriz qualidade utilizando marcadores fluorescentes. matrizes loja como recomendado acima. |
Tabela 1: Solução de problemas.
Discussão
Nas nossas experiências, verificou-se que as deficiências mais comuns estão relacionados com a qualidade das matrizes fabricadas e mal caracterizado resposta no sistema biológico de interesse. Nós nos referimos ao leitor a Tabela 1 para os modos de falha comuns em experimentos de microarranjos celular e passos de resolução de problemas associados. Em relação à qualidade de matrizes em particular, recomendamos o seguinte. Confirmar a qualidade técnica e robustez do arraying programas, parâmetros e buffers usando moléculas fluorescente etiquetado como dextrano rodamina conjugada. pins completamente limpas antes ou depois predispor acordo com as instruções do fabricante e ainda verificar visualmente que os canais de pinos são livre de detritos usando um microscópio de luz. Confirmar retenção biomolécula vestida usando manchas de proteínas gerais ou imunomarcação. Note-se que as biomoléculas com um peso molecular abaixo de 70 kDa, não são frequentemente retidas em que o hidrogel 23, sup> 31. Validar células-funcionalidade biomolécula vestiu o uso de vários tipos de células. Note-se que apenas as células aderentes são compatíveis com matrizes; Além disso, a aderência a matrizes é dependente de ambas as propriedades específicas de células (por exemplo, o perfil de expressão de integrina) e as proteínas da MEC seleccionados.
Devido ao espaço limitado, que não forneceram um extenso tratamento de projeto matriz, layout, e fabricação aqui e remeter o leitor a trabalhos anteriores 23, 25. Geralmente usamos 100 subarrays pontos (150 de diâmetro mm local, 450 mm de distância de centro a centro), composto por 10-20 condições de biomoléculas exclusivos (ie, 5-10 pontos / condição). O número de submatrizes em uma matriz varia, dependendo do número de condições de biomoléculas de interesse, o que pode ser confortavelmente dimensionada até 1280 em uma lâmina de microscópio de 25 x 75 mm (~ 6.400 pontos em 64 submatrizes)refex "> 25, 31 Os parâmetros acima irão variar ainda mais, dependendo do tamanho padrão de interesse;. pinos capazes de gerar padrões de 75-450 | iM são facilmente disponíveis.
experimentos de matriz são melhor complementados pela validação de alta pontuação condições dispostas de interesse usando outros formatos cultura, leituras de ensaio e sistemas de modelos biológicos. Especificamente, recomendamos validar ainda mais os efeitos de selecionar condições dispostas utilizando culturas a granel em conjunto com técnicas de biologia molecular padrão (por exemplo, qRT-PCR, immunoblotting) ou TFM padrão. A manipulação genética (por exemplo, a sobre-expressão ou knockdown) do factor de interesse num sistema de modelo biológico adequado pode servir também para confirmar os efeitos observados em matrizes. Em modelos animais in vivo representam um outro meio de validação e foram recentemente utilizados, por exemplo, para confirmar o papel central da galectina-3 e galectina-8 emo cancro do pulmão metastático nicho, como inicialmente identificado através de microarray de células 31, 49.
Uma série de outros métodos têm sido utilizados para sondar regulação microambiental de funções celulares, incluindo uma variedade de dois-dimensional sistemas microfabricated 18, 50, 51, 52, 53, 54, 55 e tridimensionais sistemas de biomateriais engenharia 56, 57, 58 , 59, 60, 61. Em comparação com outros métodos, as vantagens particulares da plataforma de microarray de células descritas aqui consistem em: (1) saída de atécentenas ou milhares de diferentes combinações de factores, permitindo a análise dos efeitos de interacção; (2) acessíveis, de imagem e análise automatizada; (3) integração de ambas as leituras bioquímicas e biofísicas com apresentação controlada de factores dispostas; (4) capacidade de variar as propriedades do material de substrato; e (5) a análise de uma única célula de alto teor de destino e a função da célula.
Em resumo, a combinação de microarrays de células com TFM em substratos de rigidez do substrato sintonizável permite a caracterização completa de ambos os sinais bioquímicos e biofísicos. Tal como aqui apresentado, esta plataforma é generalizável e pode ser facilmente aplicado a uma variedade de tipos de células aderentes e contextos de tecido para uma melhor compreensão da regulação microambiental combinatória de diferenciação celular e mecanotransdução.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Nós reconhecemos Austin Cyphersmith e Mayandi Sivaguru (Carl R. Woese Instituto de Biologia genômica, Universidade de Illinois em Urbana-Champaign) para assistência com microscopia e para generosamente acomodar tela e captura de vídeo no centro de microscopia.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | Pall Corporation | 4433 | Match with appropriately-sized Luer lock plastic syringes. |
| 100 × penicillin–streptomycin solution | Fisher Scientific | SV30010 | |
| 22 × 60 mm coverglasses | Electron Microscopy Sciences | 63765 | |
| 3-(trimethoxysilyl)propyl methacrylate (3-TPM) | Sigma-Aldrich | 440159 | Store under inert gas per manufacturer's instructions. Exposure of 3-TPM to air could compromise silanization of glass substrates. CAUTION: 3-TPM is a combustible liquid. Keep away from heat, sparks, open flames, and hot surfaces and use only in a chemical fume hood. |
| 3-[(3-Cholamidopropyl)dimethylammonio]-1-propanesulfonate hydrate (CHAPS) | Sigma-Aldrich | C3023 | |
| 35 mm glass-bottom Petri dishes | Cell E&G | GBD00002-200 | 13 mm well consisting of #1.5 coverglass. Enables TFM and live-cell imaging. |
| 384-well polypropylene V-bottom microplate, non-sterile | USA Scientific | 1823-8400 | |
| 6-well polystyrene microplates | Fisher Scientific | 08-772-1B | 35 mm glass-bottom Petri dishes fit into wells of microplate, easing array fabrication. |
| Acetone | Sigma-Aldrich | 179973 | |
| Acrylamide | Sigma-Aldrich | A3553 | CAUTION: Exposure to acrylamide can result in acute toxicity and irritation. Wear protective gloves, clothing, and eye protection. |
| Collagen I, rat tail | EMD Millipore | 08-115MI | |
| Collagen III, human | EMD Millipore | CC054 | |
| Collagen IV, human | EMD Millipore | CC076 | |
| Crosslinker, 365 nm | UVP | CL-1000 | |
| Dextran, rhodamine B-conjugated, 70 kDa | ThermoFisher Scientific | D1841 | Used as a marker for array location. |
| Dimethyl sulfoxide | Fisher Scientific | BP231 | |
| Dulbecco's phosphate-buffered saline (PBS) | Fisher Scientific (HyClone) | SH3001302 | |
| Ethyl alcohol | Decon Labs | 2701 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | ED | |
| Fc-recombinant DLL1, mouse | R&D Systems | 5026-DL-050 | |
| Fc-recombinant DLL4, mouse | AdipoGen | AG-40A-0145-C050 | |
| Fc-recombinant JAG1, rat | R&D Systems | 599-JG-100 | |
| Fibronectin, human | Sigma-Aldrich | F2006 | |
| Fluorescent microscope, inverted | Zeiss | Axiovert 200M | Ensure microscope is equipped with a robotic stage for both automated fluorescent imaging and TFM. Environmental control (i.e., 37 °C and 5% CO2) is highly advisable for TFM. |
| Fluoromount G with DAPI | SouthernBiotech | 0100-20 | |
| Glacial acetic acid | Sigma-Aldrich | 695092 | CAUTION: Acetic acid is flammable and corrosive. Wear protective gloves, clothing, and eye protection. |
| Glycerol | Sigma-Aldrich | M6145 | |
| Irgacure 2959 | BASF Corporation | 55047962 | |
| Laminin, mouse | EMD Millipore | CC095 | |
| Methanol | Sigma-Aldrich | 179957 | |
| Microarray scanner | GenePix | 4000B | Fluorophores must be Cy3- or Cy5-compatible. |
| Microarrayer | Digilab | OmniGrid Micro | Other microarrayers of similar or greater capability can readily be substituted. |
| Microscope slides, 25 × 75 mm | Sigma-Aldrich | CLS294775X25 | ~0.9 – 1.1 mm thickness. |
| N,N′-Methylenebisacrylamide (bisacrylamide) | Sigma-Aldrich | M7279 | CAUTION: Exposure to acrylamide can result in acute toxicity and irritation. Wear protective gloves, clothing, and eye protection. |
| Paraformaldehyde (PFA), 16% v/v | Electron Microscopy Sciences | RT15710 | Prepare PFA fresh (do not store) for optimal fixation. CAUTION: Exposure to PFA can result in acute toxicity and can also irritate or corrode skin on contact. Wear protective gloves, clothing, and eye protection and use only in a chemical fume hood. |
| Protein A/G, recombinant | ThermoFisher Scientific | 21186 | |
| Pyrex drying tray, 2,000 mL | Fisher Scientific | 15-242B | |
| Rectangular 4-chambered culture dish | Fisher Scientific (Nunc) | 12-565-495 | For cell culture on arrayed microscope slides. |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | 415413 | CAUTION: NaOH is highly caustic and can cause severe skin burns and eye damage. Wear protective gloves, clothing, and eye protection. |
| Stealth pin for arraying | ArrayIt | SMP3 | Clean pins after each array run using the instructions of the manufacturer. Produces 150 micron domains; purchase other pin sizes (75–450 microns) as suited to your particular application. |
| Triton X-100 | Sigma-Aldrich | X100 |
Referências
- Joyce, J. A., Pollard, J. W. Microenvironmental regulation of metastasis. Nat Rev Cancer. 9 (4), 239-252 (2009).
- Hsu, Y. C., Fuchs, E. A family business: stem cell progeny join the niche to regulate homeostasis. Nat Rev Mol Cell Biol. 13 (2), 103-114 (2012).
- Whiteside, T. L. The tumor microenvironment and its role in promoting tumor growth. Oncogene. 27 (45), 5904-5912 (2008).
- Jones, D. L., Wagers, A. J. No place like home: anatomy and function of the stem cell niche. Nat Rev Mol Cell Biol. 9 (1), 11-21 (2008).
- Discher, D. E., Mooney, D. J., Zandstra, P. W. Growth factors, matrices, and forces combine and control stem cells. Science. 324 (5935), 1673-1677 (2009).
- Trappmann, B., et al. Extracellular-matrix tethering regulates stem-cell fate. Nat Mater. 11 (7), 642-649 (2012).
- Ivanovska, I. L., Shin, J. W., Swift, J., Discher, D. E. Stem cell mechanobiology: diverse lessons from bone marrow. Trends Cell Biol. 25 (9), 523-532 (2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Chaudhuri, O., et al. Hydrogels with tunable stress relaxation regulate stem cell fate and activity. Nat Mater. 15 (3), 326-334 (2016).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Dev Cell. 6 (4), 483-495 (2004).
- Legate, K. R., Wickstrom, S. A., Fassler, R. Genetic and cell biological analysis of integrin outside-in signaling. Genes Dev. 23 (4), 397-418 (2009).
- Kessenbrock, K., Plaks, V., Werb, Z. Matrix metalloproteinases: regulators of the tumor microenvironment. Cell. 141 (1), 52-67 (2010).
- Underhill, G. H. Stem cell bioengineering at the interface of systems-based models and high-throughput platforms. Wiley Interdiscip Rev Syst Biol Med. 4 (6), 525-545 (2012).
- Underhill, G. H., Galie, P., Chen, C. S., Bhatia, S. N. Bioengineering methods for analysis of cells in vitro. Annu Rev Cell Dev Biol. 28, 385-410 (2012).
- Zorlutuna, P., et al. Microfabricated biomaterials for engineering 3D tissues. Adv Mater. 24 (14), 1782-1804 (2012).
- Ruiz, S. A., Chen, C. S. Microcontact printing: A tool to pattern. Soft Matter. 3 (2), 168-177 (2007).
- Guillotin, B., Guillemot, F. Cell patterning technologies for organotypic tissue fabrication. Trends Biotechnol. 29 (4), 183-190 (2011).
- Théry, M. Micropatterning as a tool to decipher cell morphogenesis and functions. J. Cell Sci. 123 (Pt 24), 4201-4213 (2010).
- Ranga, A., Lutolf, M. P. High-throughput approaches for the analysis of extrinsic regulators of stem cell fate. Curr. Opin. Cell Biol. 24 (2), 236-244 (2012).
- Kobel, S., Lutolf, M. High-throughput methods to define complex stem cell niches. Biotechniques. 48 (4), ix-xxii (2010).
- Fernandes, T. G., Diogo, M. M., Clark, D. S., Dordick, J. S., Cabral, J. M. S. High-throughput cellular microarray platforms: applications in drug discovery, toxicology and stem cell research. Trends Biotechnol. 27 (6), 342-349 (2009).
- Montanez-Sauri, S. I., Beebe, D. J., Sung, K. E. Microscale screening systems for 3D cellular microenvironments: platforms, advances, and challenges. Cell Mol Life Sci. 72 (2), 237-249 (2015).
- Flaim, C. J., Chien, S., Bhatia, S. N. An extracellular matrix microarray for probing cellular differentiation. Nat Methods. 2 (2), 119-125 (2005).
- Underhill, G. H., Flaim, C. J., Bhatia, S. N., Parekkadan, B., Yarmush, M. . Methods in Bioengineering: Stem Cell Bioengineering Artech House Methods in Bioengineering. , 63-73 (2009).
- Brafman, D. A., Chien, S., Willert, K. Arrayed cellular microenvironments for identifying culture and differentiation conditions for stem, primary and rare cell populations. Nat Protoc. 7 (4), 703-717 (2012).
- Brafman, D. A., et al. Investigating the role of the extracellular environment in modulating hepatic stellate cell biology with arrayed combinatorial microenvironments. Integr Biol. 1 (8-9), 513-524 (2009).
- Soen, Y., Mori, A., Palmer, T. D., Brown, P. O. Exploring the regulation of human neural precursor cell differentiation using arrays of signaling microenvironments. Mol Syst Biol. 2, 37 (2006).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integr Biol. 1 (1), 70-79 (2009).
- Anderson, D. G., Levenberg, S., Langer, R. Nanoliter-scale synthesis of arrayed biomaterials and application to human embryonic stem cells. Nat Biotechnol. 22 (7), 863-866 (2004).
- Brafman, D. A., Shah, K. D., Fellner, T., Chien, S., Willert, K. Defining long-term maintenance conditions of human embryonic stem cells with arrayed cellular microenvironment technology. Stem Cells Dev. 18 (8), 1141-1154 (2009).
- Reticker-Flynn, N. E., et al. A combinatorial extracellular matrix platform identifies cell-extracellular matrix interactions that correlate with metastasis. Nat Commun. 3, 1122 (2012).
- Wood, K. C., et al. MicroSCALE screening reveals genetic modifiers of therapeutic response in melanoma. Sci Signal. 5 (224), rs4 (2012).
- Braga Malta, D. F., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kaylan, K. B., Ermilova, V., Yada, R. C., Underhill, G. H. Combinatorial microenvironmental regulation of liver progenitor differentiation by Notch ligands, TGFbeta, and extracellular matrix. Sci Rep. 6 (23490), 23490 (2016).
- Kourouklis, A. P., Kaylan, K. B., Underhill, G. H. Substrate stiffness and matrix composition coordinately control the differentiation of liver progenitor cells. Biomaterials. 99, 82-94 (2016).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integr. Biol. , (2016).
- Mann, C., Leckband, D. Measuring Traction Forces in Long-Term Cell Cultures. Cellular and Molecular Bioengineering. 3 (1), 40-49 (2010).
- Heisenberg, C. P., Bellaiche, Y. Forces in tissue morphogenesis and patterning. Cell. 153 (5), 948-962 (2013).
- Schwarz, U. S., Soine, J. R. Traction force microscopy on soft elastic substrates: A guide to recent computational advances. Biochim Biophys Acta. 1853 (11 Pt B), 3095-3104 (2015).
- Butler, J. P., Tolic-Norrelykke, I. M., Fabry, B., Fredberg, J. J. Traction fields, moments, and strain energy that cells exert on their surroundings. American Journal of Physiology-Cell Physiology. 282 (3), C595-C605 (2002).
- Wen, J. H., et al. Interplay of matrix stiffness and protein tethering in stem cell differentiation. Nat Mater. 13 (10), 979-987 (2014).
- Knoll, S. G., Ali, M. Y., Saif, M. T. A novel method for localizing reporter fluorescent beads near the cell culture surface for traction force microscopy. J Vis Exp. (91), e51873 (2014).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Bolstad, B. M., Irizarry, R. A., Astrand, M., Speed, T. P. A comparison of normalization methods for high density oligonucleotide array data based on variance and bias. Bioinformatics. 19 (2), 185-193 (2003).
- Wang, N., et al. Cell prestress. I. Stiffness and prestress are closely associated in adherent contractile cells. Am J Physiol Cell Physiol. 282 (3), C606-C616 (2002).
- Krishnan, R., et al. Substrate stiffening promotes endothelial monolayer disruption through enhanced physical forces. Am J Physiol Cell Physiol. 300 (1), C146-C154 (2011).
- Reticker-Flynn, N. E., Bhatia, S. N. Aberrant glycosylation promotes lung cancer metastasis through adhesion to galectins in the metastatic niche. Cancer Discov. 5 (2), 168-181 (2015).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric control of cell life and death. Science. 276 (5317), 1425-1428 (1997).
- Kilian, K. A., Bugarija, B., Lahn, B. T., Mrksich, M. Geometric cues for directing the differentiation of mesenchymal stem cells. Proc Natl Acad Sci USA. 107 (11), 4872 (2010).
- Nelson, C. M., Chen, C. S. Cell-cell signaling by direct contact increases cell proliferation via a PI3K-dependent signal. FEBS Lett. 514 (2-3), 238-242 (2002).
- Hui, E. E., Bhatia, S. N. Micromechanical control of cell-cell interactions. Proc. Natl. Acad. Sci. U. S. A. 104 (14), 5722-5726 (2007).
- Lutolf, M. P., Blau, H. M. Artificial stem cell niches. Adv Mater. 21 (32-33), 3255-3268 (2009).
- Gobaa, S., et al. Artificial niche microarrays for probing single stem cell fate in high throughput. Nat. Methods. 8 (11), 949-955 (2011).
- DeForest, C. A., Anseth, K. S. Cytocompatible click-based hydrogels with dynamically tunable properties through orthogonal photoconjugation and photocleavage reactions. Nat Chem. 3 (12), 925-931 (2011).
- Nelson, C. M., VanDuijn, M. M., Inman, J. L., Fletcher, D. A., Bissell, M. J. Tissue geometry determines sites of mammary branching morphogenesis in organotypic cultures. Sci. STKE. 314 (5797), 298 (2006).
- Liu Tsang, V., et al. Fabrication of 3D hepatic tissues by additive photopatterning of cellular hydrogels. FASEB J. 21 (3), 790-801 (2007).
- Albrecht, D. R., Underhill, G. H., Mendelson, A., Bhatia, S. N. Multiphase electropatterning of cells and biomaterials. Lab. Chip. 7 (6), 702-709 (2007).
- Chan, V., Zorlutuna, P., Jeong, J. H., Kong, H., Bashir, R. Three-dimensional photopatterning of hydrogels using stereolithography for long-term cell encapsulation. Lab. Chip. 10 (16), 2062-2070 (2010).
- Boghaert, E., et al. Host epithelial geometry regulates breast cancer cell invasiveness. Proc. Natl. Acad. Sci. U. S. A. 109 (48), 19632-19637 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados