Method Article
Avaliação da contaminação de DNA em amostras de RNA com base no ADN ribossómico
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para rastreamento genômica contaminação de DNA (gDNA) em amostras de RNA. O método apresentado utiliza primers específicos para a região de espaçador transcrito interno (ITS) de genes ribossomais de DNA (rDNA). O método é adequado para detecção fiável e sensível de contaminação de DNA na maioria dos eucariontes e procariontes.
Resumo
Um método amplamente utilizado para a quantificação das alterações de expressão de gene e abundâncias de transcrição é reverso-transcrição do PCR quantitativo em tempo real (RT-qPCR). Fornece resultados precisos, sensíveis, confiáveis e reprodutíveis. Vários fatores podem afetar a sensibilidade e especificidade do RT-qPCR. DNA genômico residual (gDNA) contaminar amostras do RNA é um deles. Na análise da expressão de gene, amplificação não-específica, devido à contaminação de gDNA vai superestimar a abundância de níveis de transcrição e pode afetar os resultados de RT-qPCR. Geralmente, gDNA é detectado por qRT-PCR usar primer pares recozimento para regiões intergênicas ou um intrão do gene de interesse. Infelizmente, as anotações de intron/exon ainda não são conhecidas por todos os genes do vertebrado, bactérias, Protista, fungos, plantas e espécies de invertebrados metazoários.
Aqui nós apresentamos um protocolo para a deteção de gDNA contaminação no ARN amostras usando o ADN ribossómico (rDNA)-com base em cartilhas. O método baseia-se as características únicas do rDNA: sua natureza multigene, sequências altamente conservadas e alta frequência no genoma. Também como um estudo de caso, um conjunto exclusivo de primers foram projetados com base na região conservada de ADN ribossómico (rDNA) pertencente à família Poaceae. A universalidade desses pares da primeira demão foi testada pelo derretimento curva análise e agarose electroforese do gel. Embora nosso método explica como as primeiras demão rDNA-based podem ser aplicadas para o ensaio de contaminação gDNA pertencente à família Poaceae, poderia ser facilmente usado para outras espécies de procarionte e Eukaryota
Introdução
Explorar o Regulamento transcriptional de interessante moda gene ou redes de sinalização é essencial para compreender os mecanismos moleculares complexos envolvidas em eventos biológicos1. Atualmente, a análise de qPCR é o mais amplamente utilizado abordagem para gene expressão estudos que podem direcionar o DNA (genoma) ou RNA (a transcriptoma) que permitem a análise methylome e transcriptoma, respectivamente. Transcrição reversa (RT), seguida por qPCR é amplamente utilizada para a análise do transcriptoma que medem os níveis de expressão de gene em várias áreas de pesquisa biológica2. Em comparação com outros métodos como a hibridação Northern tradicional, deteção específicas de tecido via in situ da hibridação, ensaios da proteção ribonuclease (RPA) e semi-RT-PCR, a precisão, conveniência, velocidade e ampla faixa dinâmica de ensaios baseados em qPCR são altamente notável3,4. Há vários fatores importantes que devem ser considerados para uma quantificação confiável de RNA mensageiro (mRNA), incluindo a qualidade e a quantidade de RNA a partir de material. Além disso, amplificação não-específica, a eficiência de RT-qPCR e eficiência da PCR tem que ser considerado5,6.
A presença de gDNA é um problema inerente durante a extração do RNA devido, em parte, as propriedades físicas e químicas similares de DNA e RNA7. Por causa da identidade de sequência dos gDNA e DNA complementar (cDNA) derivado das amostras de mRNA, amplificação não-específica pode ocorrer, que irão influenciar a precisão dos resultados de RT-qPCR. O restante gDNA levará a superestimação da abundância de mRNA alvo de análise de expressão de gene8.
Basicamente, o amplicon inespecíficas principalmente surge da formação da primeira demão-dímero ou amplificação de fundo inespecíficos devido gDNA, ambos os quais podem ser avaliados usando amostras de controlo apropriadas. Tais amostras são nenhum controle modelo (NTC) e nenhum controle de transcriptase reversa (NRT), respectivamente. Desde que os níveis de contaminação gDNA nas amostras estudadas são diferentes e a sensibilidade em direção gDNA difere grandemente entre os genes analisados, os controles NRT são necessários para cada par de amostra/ensaio. Embora isto aumenta substancialmente o custo e o trabalho em estudos de perfil de RT-qPCR, esses controles são necessários7,9.
Métodos alternativos de lidar com a contaminação gDNA incluem o uso de pares da primeira demão, recozimento para regiões intergênicas ou um intrão do gene de interesse10e o uso de primers que flanqueiam um intrão grande ou abrangem uma junção exon-exon, ou seja os recozimento sites estão ausentes no mRNA maduro sequência1,4. No entanto, as anotações de intron/exon para todos os genes de muitos vertebrado, bactérias, Protista, fungos, plantas e espécies de invertebrados metazoários são conhecidas ainda. Além disso, muitos organismos eucariotas têm os pseudogenes derivados de eventos de duplicação. Além disso, o projeto da primeira demão do outro lado os intrões não garante não-amplificação de gDNA. Como a cromatina acessibilidade das regiões genômicas para DNase que varia, é recomendável desenhar pares diferentes cartilha visando diferentes cromossomos10.
Os genomas de organismos eucariotas podem abranger até mil cópias de genes rDNA codificação subunidades ribossomais necessárias para formação de ribossomas. Estes genes rDNA organizam-se frequentemente em matrizes simples ou em tandem repetição11. Policistrônico rRNAs (Figura 1), incluindo a grande subunidade (LSU) e uma pequena subunidade (SSU) são trascritos pela RNA polimerase eu (pol RNA eu). Os pre-rRNAs resultantes são tratados posteriormente, eliminando as duas regiões de espaçador transcrito interno ITS1 e ITS2. Como produtos finais, três maduras rRNAs, 17-18S rRNA (SSU), 5.8S e 25-28S rRNA (LSU) são gerados12. rDNA genes são típicos representantes de uma família multigene com sequências altamente conservadas. Eles ocorrem com frequência no genoma e estão potencialmente presentes em mais de um local cromossômico13. O processamento do rRNA e a degradação dos espaçadores transcritos são um processo rápido no nucléolo. Devido ao alto grau de repetitividade, a proporção do número de cópia genômica e premolecules de RNA detectáveis no seu estado inalterado é baixa em comparação com as sequências de baixo-cópia intrão e precursores unspliced. Estas características fazem do rDNA genes bem adequados para detecção confiável e altamente sensível de contaminação gDNA na maioria dos eucariontes e procariontes3.
Aqui um novel procedimento para detecção de contaminação gDNA em amostras do RNA é descrito. Um conjunto de primers universais com base na sequência de rDNA conservada é apresentado para gDNA ensaios em diversas espécies de Poaceae. A especificidade e a universalidade dos primers propostos foram testados usando o DNA como um modelo de análise da curva de derreter. Nosso protocolo não só é aplicável para Poaceae, mas pode também facilmente ser adaptado para outras espécies procariotas e eucariotas.
Protocolo
Nota: Qualquer tecido pode ser usado.
1. ácido nucleico extração
- Colocar 100 mg de amostras de tecido em um tubo de 2,0 mL, adicionar dois grânulos de aço inoxidável de 5mm e homogeneizar o tecido a 25-30 Hz para 30 s (duração de homogeneização e frequência, dependendo do tipo de tecido) para RNA e DNA.
- Isole o RNA total de acordo com as instruções do fabricante.
- Isole o DNA total de acordo com as instruções do fabricante.
- Controle a pureza e a quantidade de amostras de RNA medindo a absorbância em 260 e 280 nm.
- Controle a pureza e a quantidade das amostras de DNA por medir a absorvância a 260 e 280 nm.
Nota: Embora ácidos nucleicos absorvem a luz com um comprimento de onda de 260 nm (A260), a absorção de luz no comprimento de onda de 280 (A280) pode ser usada para quantificar a quantidade de proteínas e fenóis presentes na amostra. Portanto, a taxa de A260/A280 nm pode ser usada para avaliar a pureza do DNA e do RNA extraído de uma amostra. A260/280 valores no intervalo de ≥1.8 e > 2.0 são geralmente considerados como "puro" para DNA e RNA, respectivamente. Valores mais baixos de A260/280 podem indicar contaminação por proteínas ou produtos químicos orgânicos. - Teste a qualidade do DNA, executando uma eletroforese em gel de agarose 0.7%. Preparar o gel e executar em EDTA-tampão TRIS-bórico 1x (TBE: 89 mM Tris, o ácido bórico 89 mM e 2 mM EDTA) a 100 V para 30 min. alta qualidade gDNA aparece como um acentuado, alto peso molecular (HMW) banda com sem manchas no intervalo de moléculas de baixo peso molecular (HBPM).
- Verifique o RNA isolado por quantidade, pureza e integridade sob desnaturação condições por uma eletroforese em gel de agarose guanidina tiocianato (GTC) ou por um chip de eletroforese capilar, de acordo com as instruções do fabricante.
- Prepare o gel do GTC, acrescentando 5 mM GTC para um padrão 1 gel de agarose a 1% de x TBE após refrigerar o agar para 60 ° C.
Nota: GTC é tóxico, então dispense-lo em uma coifa e usar equipamento de protecção adequado. - Preparar RNA desnaturação amortecedor do carregamento: 95% formamida, 10mm EDTA pH 8.0, azul de bromofenol 0,1% 0,1% cyanole de xileno e brometo de etídio 10 µ l.
Nota: Formamida e ethidium brometo são tóxico e deve ser dispensado em uma coifa. - Carga 1-5 µ g de RNA total no ARN desnaturação amortecedor do carregamento, calor a mistura por 5 min a 70 ° C, coloque-o no gelo antes de carregá-lo em um gel e então separar o RNA em gel GTC a 100 V por 45 min. carga de DNA ou RNA marcador de peso molecular como um padrão ao lado a amostra de RNA.
- Manchar o gel com brometo de etídio e visualizar as bandas utilizando sistemas de captura de imagem sob luz ultravioleta. Em eucariontes, intacto RNA total executado em condições de desnaturação irá mostrar pelo menos duas nítidas e claras rRNA bandas (28S e 18S) com uma proporção de 2:1 intensidade.
- Prepare o gel do GTC, acrescentando 5 mM GTC para um padrão 1 gel de agarose a 1% de x TBE após refrigerar o agar para 60 ° C.
- Remover vestígios de gDNA por tratamento com DNase (DNase eu RNase-livre). Adicionar a um tubo no volume total de 10 µ l de RNase-livre: 0.1 - 1 µ g de RNA total, uma unidade de DNase I e 1 µ l de reação 10x de buffer com MgCl2. Incubar a mistura por 30 min a 37 ° C. Terminar a reação adicionando 1 µ l de 50 mM de EDTA e incubando a 65 ° C por 10 min.
- Remova traços de RNA gDNA extratos usando livre de DNase RNase A, de acordo com o protocolo do fabricante. Adicionar 5 µ l de RNase 10mg/mL para o DNA total e incubar a 37 ° C para extratos de armazenamento de RNA e DNA de 1 h. a-80 ° C.
2. primer Design de rDNA região para gDNA ensaio
Nota: A sequência de rDNA completos contém duas regiões (ITS1 e ITS2), que são removidas na molécula de RNA ribossomal maduro por uma série de segmentações de endonucleolytic e em seguida degradados (Figura 1).
- Recupere a sequência de nucleotídeos do rDNA de NCBI (http://www.ncbi.nlm.nih.gov) para as espécies de interesse. A melhor palavra-chave para pesquisar o banco de dados é "espaçador transcrito interno."
- Entrada a sequência de nucleótidos de destino em uma pesquisa BLASTn para encontrar regiões do espaçador transcrito interno (ITS), SSU e LSU conservadas regiões.
- Selecione as primeiras demão que também flanquear uma sequência de ITSs ou que amplificar sequências de ITSs que não estão presentes no rRNA maduro.
- Projetar primers flanqueando as sequências de ITS1 ou ITS2: alinhar as regiões conservadas de várias espécies de ClustalW. Projetar primers específicos para sua região de acompanhamento após a análise de táxons específicos/entre espécies com software AlleleID. O primer dois pares de amplificação SSU-5.8S e 5.8S-LSU amplicons pode ser projetada com base nas regiões de flanqueamento de ITS1 e ITS2, respectivamente. Porque estes amplicons são abrangendo toda a região ITS, o amplicon comprimento será aumentado pelo menos 300 bp em amplicons de gDNA. Este aumento reduz a sensibilidade.
- Flanqueando o ITS1: Selecione SSU e 5.8S sequência de RNA ribossomal. Primers selecionados para Poaceae são: SSU, SF: CGTAACAAGGTTTCCGTAGGTG, r: GGTTCACGGGATTCTGCAAT. Este par de primer (SF: r: ré e frente) amplifica a região parcial da SSU, o longa-metragem de ITS1 e região parcial de 5.8S rDNA.
- Flanqueando a ITS2: Selecione 5.8S e sequência LSU. Primers selecionados para Poaceae são: sequência de consenso f: ATTGCAGAATCCCGTGAACC LSU, LR: TGCTTAAAYTCAGCGGGTAGYC. Este par de primer amplifica a região parcial de 5.8S, o longa-metragem de ITS2 e região parcial da LSU (Figura 1).
Nota: No caso do projeto da primeira demão, com base em ITSs flanqueando a região, as áreas altamente conservadas da SSU, 5.8S e LSU foram identificados. Os primers para diante e reversos de 5.8S rRNA foram projetados com base em um motivo conservado em plantas de floração14. Os primers para diante e reversos foram projetados com base no SSU e LSU conservadas regiões em Poaceae, respectivamente. A divergência de SSU e LSU primers para cada espécie é dado na tabela 1.
- Primers para amplificar uma sequência de ITSs: neste protocolo, iniciadores do projeto ITS1 baseiam no Aeluropus sua sequência (número de CNPJ NCBI: 110873). Para as primeiras demão, usar: para a frente: GGTATGGCGTCAAGGAACACT, reverso: ATAGCATCGCTGCAAGAGGT. De acordo com os amplicons gerados pelo primer pares no silico, o tamanho deve variar de 60 a 200 bp. Este também é o tamanho recomendado para análise de qPCR.
- Projetar primers flanqueando as sequências de ITS1 ou ITS2: alinhar as regiões conservadas de várias espécies de ClustalW. Projetar primers específicos para sua região de acompanhamento após a análise de táxons específicos/entre espécies com software AlleleID. O primer dois pares de amplificação SSU-5.8S e 5.8S-LSU amplicons pode ser projetada com base nas regiões de flanqueamento de ITS1 e ITS2, respectivamente. Porque estes amplicons são abrangendo toda a região ITS, o amplicon comprimento será aumentado pelo menos 300 bp em amplicons de gDNA. Este aumento reduz a sensibilidade.
- Escolher as primeiras demão ao considerar estas recomendações: conteúdo GC: 40-60%, o comprimento da primeira demão: 18-23 base, comprimento do produto do PCR: 60-160 bp (especialmente para sua primeira demão), temperatura (Tm) de fusão: 60 ° C, o Tm final para as duas primeiras demão não diferem mais de 5 ° C e o prim ERS não são complementares a mesmos ou parceiro primeiras demão.
- Verificar o número de especificidade e cópia da primeira demão. Execute em silico análise da sequência de cartilha selecionados pelo programa primeira demão-explosão (https://www.ncbi.nlm.nih.gov/tools/primer-blast/).
- Abra a página de submissão de Primer-BLAST. Insira ambas as sequências de cartilha na seção de parâmetros da primeira demão do formulário. Na especificidade de par de primer verificar a seção de parâmetros, digite um nome do organismo (ou nome de grupo de organismo) e selecione o banco de dados do genoma. Essas configurações dão a especificidade informação sobre parâmetros de sequência e primer de destino incluindo o comprimento do produto, posição no cromossomo e copiar o número.
3. executar qPCR passo para validação de Primers baseados em rDNA com modelos de DNA
Nota: A funcionalidade dos primers projetados deve ser validada através da realização de qPCR usando gDNA como um modelo. Para executar várias reações paralelas e reduzir erros de pipetagem, recomenda-se a preparação de uma mistura de mestre. Prepare uma mistura de mestre, um volume equivalente ao número total da mistura de reação mais ~ 10%.
- Prepare uma mistura de mestre pela mistura de todos os componentes de reação, exceto o modelo de DNA em um tubo de reação de PCR. Upscale como necessário de uma reação para preparar a mistura de mestre: 5 µ l SYBR green (SYBR) mestre mistura (2x), 0,3 µ l da primeira demão (0,3 µM cada de primer para diante e reverso) e ajustar o volume final de 10 µ l com água livre de RNase. Use aproximadamente ≤200 ng em 1 µ l de gDNA de modelo para análise.
Nota: Descongelar, montar e manter todos os reagentes, componentes e misturas de reação no gelo. - O mix de mestre em uma placa de 96 poços óptica de alíquota. Pipetar 1 µ l de gDNA para cada poço e em seguida, cubra-o com placa óptica película de vedação. Girar e colocar em cycler.
- Executar o ensaio de qPCR em um termociclador em tempo real, sob as seguintes condições: 10 min a 95 ° C seguido de 40 ciclos de 95 ° C por 15 s e 60 ° C, durante 1 min. aquisição de dados executar o recozimento 60 ° C/extensão step.
- Após o procedimento de amplificação, submeter todas as reações de PCR para uma derretimento análise de curva com medição contínua da fluorescência de 55 ° C e 95 ° C. Normalmente, colete um ponto de dados cada ciclo por um aumento gradual da temperatura de 0,5 º C por ciclo.
Nota: Inclua pelo menos 2 controles não-modelo (NTC) para cada mistura de mestre de par de primer. Realize todos os ensaios pelo menos três repetições. - Confirme a especificidade da primeira demão, através da análise de curva de derreter. Analise as curvas com o limiar único ciclo e método de curva subtraídos.
Nota: A aparência de um pico afiado individual indica um uniforme amplicon individual. Produtos de dímero da primeira demão podem aparecer como picos individuais a baixas temperaturas. - Valide o tamanho de cada amplicon por eletroforese em gel de agarose.
- Preparar um gel de agarose a 3% misturando 3 g agarose com 100 mL de tampão TBE (TBE: 89 mM Tris, o ácido bórico 89 mM e 2 mM EDTA).
- Misture 5-10 µ l de produto PCR com DNA e 1-2 µ l de 6 x amortecedor do carregamento. Carregar o produto do PCR ao lado de uma escada de DNA em gel de agarose a 3%. Realize uma separação eletroforética em 1x do EDTA-tampão TRIS-bórico em 100 V por 45 min.
- Manchar o gel com brometo de etídio ou qualquer outro agente intercalante e visualizar as bandas utilizando sistemas de captura de imagem sob luz ultravioleta.
Nota: Agentes intercalante de DNA (por exemplo, brometo de etídio) são cancerígenos e devem ser manuseados com cuidado e separadamente dispensados. O surgimento de uma única banda afiada (em relação ao tamanho e sem primer-dímero ou amplificação artificial base) confirma a especificidade do amplicon.
4. procedimento do ensaio de contaminação com modelos de RNA de gDNA
Nota: Após o tratamento com DNase, o RNA purified amostra é testada por iniciadores específicos do rDNA. Devido o processamento do recurso de ITSs intrão-como quando estas regiões são utilizadas para amplificação, nenhum sinal de amplificação deve ser detectado em amostras de RNA DNA-livre. Com base nisso, se for detectado um sinal de amplificação em qPCR ou uma banda no gel do agarose observado com o tamanho esperado (estimado em silico análise), que isto deve ser devido a contaminação gDNA. As etapas executadas nesta seção, são semelhantes a seção 3, exceto que o cDNA de todas as amostras é usado como modelo em vez de gDNA.
- Prepare uma mistura de mestre pela mistura de todos os componentes de reação exceto modelo de RNA em um tubo de reação de PCR. Combinação de mistura para uma reação de mestre: 5 µ l SYBR mestre mistura (2x), 0,3 µ l da primeira demão (0,3 µM cada de mistura de cartilha frente e verso) e ajustar o volume final de 10 µ l com água livre de RNase. Use aproximadamente 500 ng modelo RNA em volume de 1 µ l para análise.
- A mestre mistura em uma placa de 96 poços óptica de alíquota. Pipetar 1 RNA µ l de cada poço e depois cobri-lo pela ótica placa película de vedação. Centrífuga e lugar em cycler.
Nota: Inclua pelo menos dois controles NTC e dois controles gDNA positivo para cada ensaio. Realize todos os ensaios em três repetições de técnicas. - Executar o ensaio de qPCR em um termociclador em tempo real, sob as seguintes condições: 10 min a 95 ° C seguido de 40 ciclos de 95 ° C por 15 s e 60 ° C, durante 1 min. aquisição de dados executar o recozimento 60 ° C/extensão step.
- Após o procedimento de amplificação, submeter todas as reações de PCR para uma derretimento análise de curva com medição contínua da fluorescência de 55 ° C e 95 ° C. Geralmente, colete um ponto de dados cada ciclo por um aumento gradual da temperatura de 0,5 º C por ciclo.
- Verifique todos os produtos PCR executando em agarose a 3% da electroforese do gel.
Nota: A aparência de qualquer banda ou pico na reação de NTC provavelmente está relacionada a formação da primeira demão-dímero que geralmente é vista em baixas temperaturas na curva de fusão, enquanto a presença de qualquer banda ou pico em amostras do RNA é o resultado de contaminação gDNA. Recomenda-se primeiro testar todas as amostras de RNA por cartilhas rDNA-baseado, e em seguida amostras contaminadas não-DNA são usadas para aplicações a jusante, tais como a síntese do cDNA, análise da expressão de gene, etc.
5. passo RT-PCR para síntese de cDNA e qPCR análise
- Descongelar o DNase-RNA e os reagentes de síntese do cDNA tratados à temperatura ambiente. Após o descongelamento, spin para baixo os reagentes. Adicionar 1 µ g de RNA e 1 µ l de Oligo (dT) 18 cartilha para um tubo de nuclease-livre. Ajustar o volume total de 12 µ l com água livre de RNase, misture delicadamente e em seguida armazenar no gelo.
- Derreta estruturas secundárias do modelo de RNA incubando a reação a 65 ° C por 5 min. Spin para baixo e fixe o frasco no gelo.
- Prepare a mistura de reação mestre (volume final de 20 µ l de cada reação) do seguinte modo: 1 µ l de transcriptase reversa (200 U / µ l), 4 µ l de tampão de reação (5x), 1 µ l de inibidor de RNase (20 U / µ l) e 2 µ l de dNTP Mix (10 mM). Misture delicadamente e deixe esfriar o frasco no gelo. Adicione 19 µ l para o tubo preparado contendo o RNA.
- Incubar a reação de 60 min a 42 ° C e então incubar a 70 ° C por 5 min finalizar a atividade da transcriptase reversa. Coloque as reações de RT no gelo e proceder à análise de expressão do gene por procedimento de rotina qPCR (conforme explicado na seção 3 e 4).
Resultados
Propomos o uso de primers rDNA-baseado para validar a ausência de contaminação gDNA em amostras do RNA do tecido foliar. O fluxograma da qPCR análise e gDNA contaminação do ensaio é mostrado na Figura 2. O protocolo apresentado, duas estratégias complementares foram usadas para o projeto da primeira demão do rDNA-based: cartilhas 1) espécie-específicos foram seleccionadas a partir de sequências de ITSs e primeiras demão 2) universal foram selecionadas de ITSs flanqueiam regiões. Para prova de conceito, nós projetamos primers específicos para Aeluropus littoralis e primers universais com base em espécies de Poaceae, como determinado no protocolo. O 5.8S primers para diante e reversos foram selecionados com base em um motivo de 14 pares de base (PB) conservada que mostra semelhança entre plantas, briófitas e várias ordens de algas e fungos14. As características das primeiras demão projetadas são dadas na tabela 2. A universalidade da SSU, 5.8S e a primeira demão LSU foram verificadas por BLASTn, e da primeira demão homologia os resultados são apresentados na Figura 3 , como um logotipo do motivo. A lista de espécies que constam a análise de homologia, bem como as primeiras demão divergentes para cada espécie são dadas tabela 1. Especificidade da primeira demão foi cheque por Primer-BLAST. Para espécies onde a sequência do genoma inteiro está disponível, estimou-se a localização cromossômica dos genes rDNA. Por exemplo, em Oryza sativa e Arabidopsis thaliana, rDNA genes situam em dois cromossomos diferentes e em Zea mays em três diferentes cromossomas.
qPCR validação de primers baseados em rDNA foi realizada com a análise da curva de ITS1 e ITS2-flanco amplicons usando o DNA como um modelo de fusão. Conforme apresentado na Figura 4 e Figura 5, especificidade cartilha foi confirmada experimentalmente pela observação de um único pico afiado com sem formação de cartilha-dímero em diferentes espécies de Poaceae, incluindo Triticum aestivum, Hordeum vulgare, Oryza sativae nas dicotiledôneas Medicago sativa, Cucumis sativus, Nicotiana tabacum, chamada de Trifolium, Vicia fabae Arabidopsis thaliana. O teste ainda mais os produtos de amplificação por separação eletroforética tamanho mostrou uma banda única. Como esperado, as bandas derivado de amostras de diferentes espécies variadas em tamanho (figura 6A e 6B). Curiosamente, o uso dos primers universais projetado especificamente para as três espécies de Poaceae não são apenas úteis para outras espécies de Poaceae, mas também para outras espécies de plantas como a. thaliana e para um fungos endofíticos viz. Piriformospora indica.
A validade do primer específico projetado (ITS1) também foi confirmada por qPCR em a. littoralis usando gDNA como modelo. Observou-se um único pico com sem formação de dímero da primeira demão. Surpreendentemente, a primeira demão a. littoralis ITS1 (como o primer específico) gerou uma única banda afiada não só na . littoralis, mas também para todas as outras espécies testadas com exceção de Nicotiana tabacum e chamada de Trifolium, que produziu duas bandas (Figura 6). O ensaio de contaminação gDNA foi realizado por primers ITS ou ITS-acompanhamento em todas as amostras de RNA. Uma representação esquemática da placa de amplificação no ensaio gDNA contaminação e a interpretação dos resultados é apresentado na Figura 7.
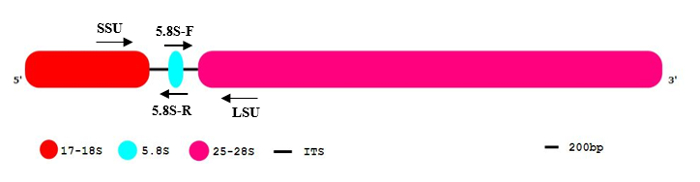
Figura 1: O padrão geral de organização de sequência de rDNA eucarióticas.
O segmento de rDNA eucariota contém 17-18 (vermelho), 5.8S (azul) e 25-28S rRNA (rosa). Os espaçadores transcritos internos (ITS) são indicados como linhas pretas. 5´and 3´ indicar a orientação da molécula de DNA. Clique aqui para ver uma versão maior desta figura.
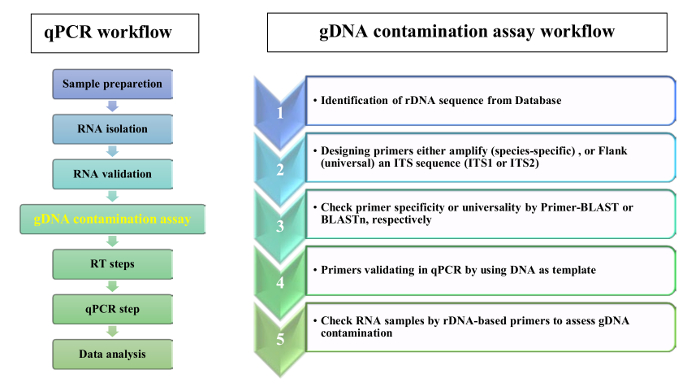
Figura 2: fluxo de trabalho para um ensaio de contaminação RT-qPCR e gDNA. Clique aqui para ver uma versão maior desta figura.
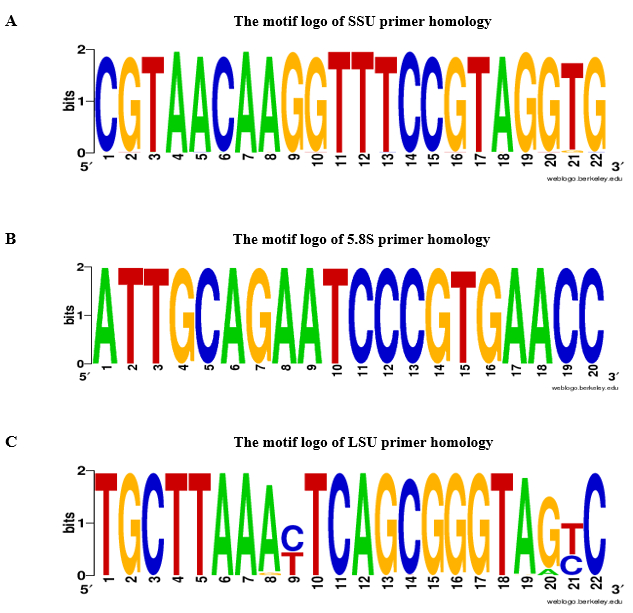
Figura 3: logotipo do motivo da SSU r., B. 5.8S e C. LSU cartilha homologia. Para SSU, 5.8S, e iniciadores da LSU, o logotipo do motivo foi construído pelo BLASTn baseado em 2.000 registros de planta verde (número do CNPJ NCBI: 33090) com um corte e valor ≤ 10-10. A-adenina, T-timina, G-guanina, C-citosina. Clique aqui para ver uma versão maior desta figura.
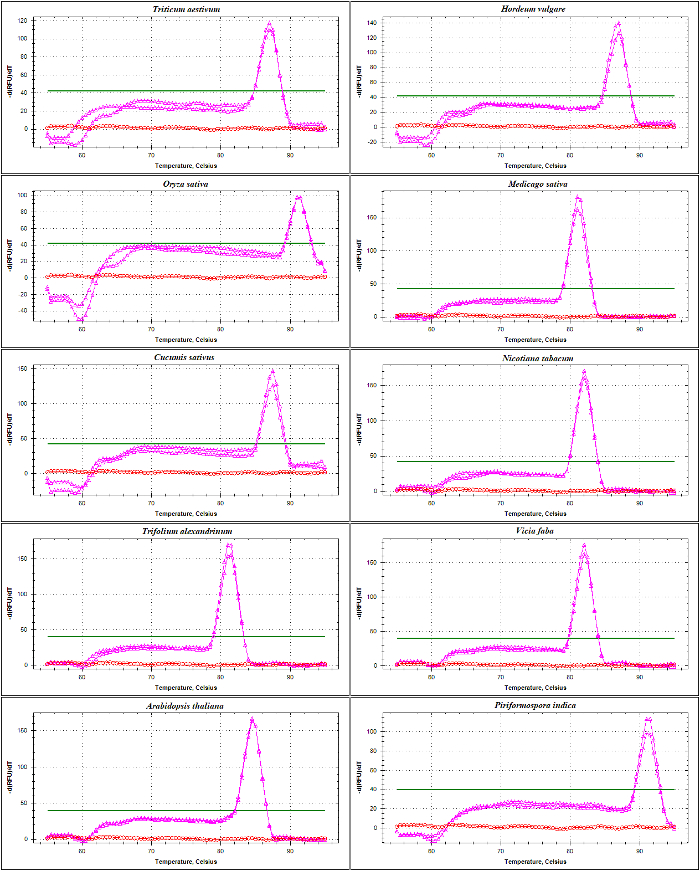
Figura 4: O derretimento análise da curva de ITS1-flanqueando amplicon em diferentes espécies.
Este amplicon, amplificado por SSU e 5.8S-primeiras demão R, contém parte da sequência da região de codificação de 17-18, a sequência inteira de ITS1 e sequência parcial de 5.8S. São mostradas as curvas de fusão de amplicons gerados (rosa) e NTC (vermelho) de Triticum aestivum, Hordeum vulgare, Oryza sativa, Medicago truncatula, Cucumis sativus, Nicotiana tabacum, Trifolium chamada, Vicia faba, Arabidopsis thaliana e Piriformospora indica. A linha plana em negrito indica o limite de linha de base. Clique aqui para ver uma versão maior desta figura.
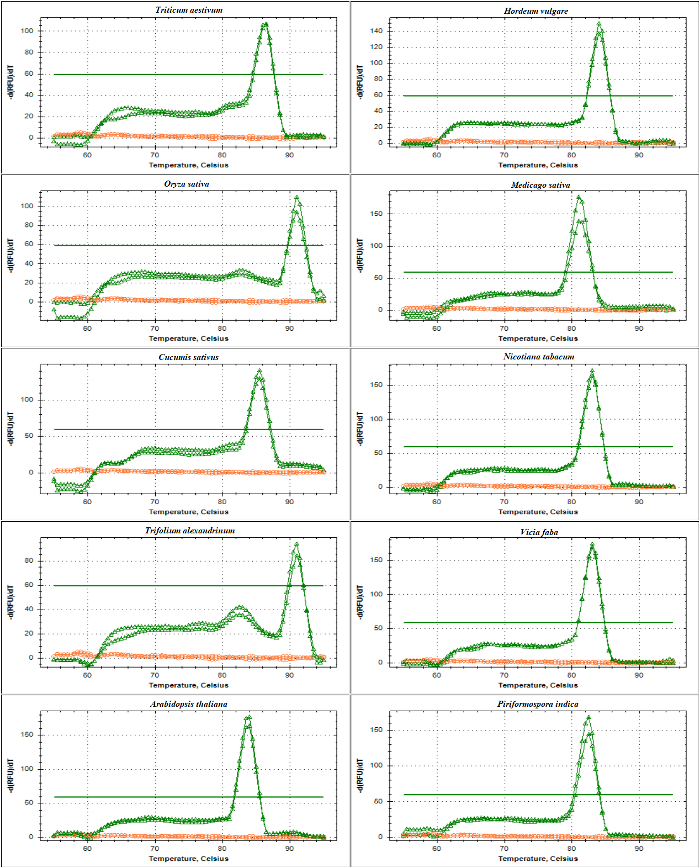
Figura 5: O derretimento análise da curva de amplicons ITS2-acompanhamento em diferentes espécies.
Este amplicon é gerada pelo uso de 5.8S-F e LSU cartilhas. O amplicon descrito contém sequências de parte de 5,8 S, a sequência inteira de ITS2 e uma sequência parcial de 25-28S. São mostradas as curvas de fusão de amplicons (verde) e NTC (vermelho), gerado a partir de Triticum aestivum, Hordeum vulgare, Oryza sativa, Medicago truncatula, Cucumis sativus, Nicotiana tabacum, Trifolium chamada, Vicia faba, Arabidopsis thaliana e Piriformospora indica. Clique aqui para ver uma versão maior desta figura.
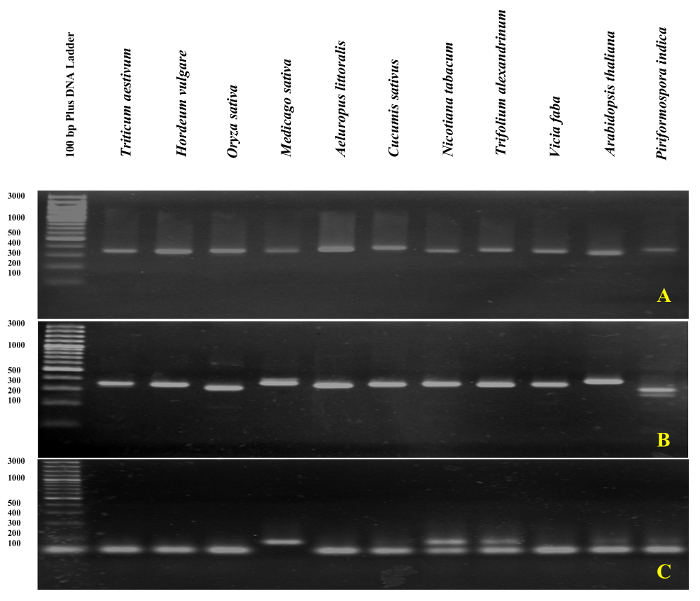
Figura 6: Análise do gel do Agarose de produto à base de rDNA PCR.
O amplicon de ITS1-flancos (A), ITS2-flancos (B) e ITS1 (C) foram executados em gel de agarose a 3%. Clique aqui para ver uma versão maior desta figura.

Figura 7: Intron-como características de ITSs podem ser consideradas para projetar primers que podem detectar a contaminação gDNA.
Qualquer pico ou a banda com o tamanho esperado em análise qPCR indicar gDNA contaminação da amostra de RNA. UNK: amostra desconhecida, pos: controle positivo, NTC: controle de não-modelo. Clique aqui para ver uma versão maior desta figura.
| Primeira demão | Gênero | ID de CNPJ | Espécies | Primeira demão divergente | ||
| SSU | Arabidopsis | 3701 | kamchatica, thaliana e lyrata | - | ||
| Vicia | 3904 | villosa, americana, unijuga, amoenane, amurensis, craccamal, pseudo-orobus, multicaulis, japonica, ramuliflora e faba | ||||
| Trifolium | 3898 | chamada, montanum, resupinatum e repens | - | |||
| Nicotiana | 4085 | tabacum, benthamiana, otophora, picilla, bigelovii, palmeri, tomentosiformis, tomentosa, digluta, kawakamii, clevelandii, nesophila, solanifolia, cordifolia, debneyi, arentsii, thyrsiflora, wigandioides, undulata, glutinosa, noctiflora, petunioides, obtusifolia, miersii, pauciflora, atenuar, acuminata linearis, alata, sylvestris, rustica e suaveolens | - | |||
| Cucumis | 3655 | anguria, melo e sativus | CGTAACAAGGTTTCCGTAGGKG | |||
| Aeluropus | 110873 | - | Não há cartilha encontrada | |||
| Medicago | 3877 | sativa, lupulina, pamphylica, lunata, rostrada, sulcada e truncatula | - | |||
| Oryza | 4597 | sativa, glumipatula, rufipogon barthii glaberrima punctate, longistaminata, meridionalis, nivara, meridionalis e longistaminata | - | |||
| Triticum | 4564 | aestivum, urartu e monococcum | - | |||
| Hordeum | 4512 | vulgare, bulbosum, marinum, brevisubulatum e bogdanii | - | |||
| LSU | Arabidopsis | 3701 | Pétrea, thaliana e lyrata | TGCTTAAACTCAGCGGGTAATC | ||
| Vicia | 3904 | sylvatica, tetrasperma, sativa, hirsuto, sepium, parviflora, cracca, lathyroides, orobus, orobus, bithynica e faba | TGCTTAAATTCAGCGGGTAGCC | |||
| Trifolium | 3898 | pretensão, nigrescens, resupinatum occidentale, subterraneum, strictum, ochroleucon, glomeratum, squamosum, ornithopodioides e repens | TGCTTAAATTCAGCGGGTAGCC | |||
| Nicotiana | 4085 | tabacum, benthamiana, otophora, picilla, bigelovii, palmeri, tomentosiformis, tomentosa, digluta, kawakamii, clevelandii, nesophila, solanifolia, cordifolia, debneyi, arentsii, thyrsiflora, wigandioides, undulata, glutinosa, noctiflora, petunioides, obtusifolia, miersii, pauciflora, atenuar, acuminata linearis, alata, sylvestris e suaveolens | TGCTTAAACTCAGCGGGTAGTC | |||
| Cucumis | 3655 | Melo, ritchiei e javanica | TGCTTAAACTCAGCGGGTAGTC | |||
| Aeluropus | 110873 | lagopoide, pungens e littoralis | TGCTTAAATTCAGCGGGTAATC | |||
| Medicago | 3877 | ruthenica, sativa, lupulina, goma-arábica, polymorpha e mínimos | TGCTTAAATTCAGCGGGTAGCC | |||
| pamphylica, lunata, rostrada e sulcada | TGCTTAAACTCAGCGGGTAGTC | |||||
| Oryza | 4597 | sativa, glumipatula, rufipogon, barthiial, glaberrima, australiensis, officinalis australiensis, ridleyi, malampuzhaensis, alta, nivara, rufipogon, meridionalis e longistaminata | TGCTTAAACTCAGCGGGTAGTC | |||
| Triticum | 4564 | aestivum, spelta, turgidum, dicoccoides, petropavlovskyi, urartu e monococcum | TGCTTAAACTCAGCGGGTAGTC | |||
| Hordeum | 4512 | vulgare, bulbosum, murinum, secalinum, brevisubulatum e bogdanii | TGCTTAAACTCAGCGGGTAGTC | |||
| Um primer degenerado é definido como sistema IUPAC para a nomenclatura de nucleotídeo | ||||||
Tabela 1: Lista de espécies consideradas para escolher as primeiras demão rDNA-based.
Sítio de ligação SSU em comparação com o sítio de ligação de LSU mostrou mais alta homologia de sequência sobre dado gênero.
| Comprimento do amplicon | Área de amplificação | Sequência de | Nome da primeira demão | Amplicon | |
| 332 - 405 bp | Sequência parcial de SSU, toda a sequência de ITS1 e sequência parcial de 5.8S | CGTAACAAGGTTTCCGTAGGTG | SSU | ITS1-flancos | |
| GGTTCACGGGATTCTGCAAT | 5.8S-R | ||||
| 318 - 361 bp | Sequência parcial de 5.8S, toda a sequência de ITS2 e sequência parcial de LSU | ATTGCAGAATCCCGTGAACC | 5.8S-F | ITS2-flancos | |
| TGCTTAAAYTCAGCGGGTAGYC | LSU | ||||
| 100 - 200 bp | ITS1 | GGTATGGCGTCAAGGAACACT | ITS1-F | ITS1 | |
| ATAGCATCGCTGCAAGAGGT | ITS1-R | ||||
Tabela 2: Primer de sequências.
Discussão
Análise de expressão genética por PCR quantitativo tem sido amplamente aplicada nos últimos anos. A principal vantagem deste método rápido, econômico e automatizado é seu resultado preciso e exato. No entanto, obter benefícios ideais destas vantagens exige uma compreensão clara da configuração dos parâmetros utilizados para o experimento de qPCR. Para receber um resultado confiável em análise de expressão de gene qPCR, é necessário evitar a amplificação inespecífica que surge a partir da primeira demão-dímero ou gDNA contaminação da amostra de RNA3,15. Espera-se que os níveis de transcrição do RNA vão ser sobrestimados sob gDNA contaminação8. Aqui, as características únicas de um gene de rDNA foi considerado para um ensaio de contaminação gDNA em amostras de RNA.
As propriedades básicas do rDNA utilizados neste protocolo: Ribosomal genes consistem o dois ITSs, ITS1 e ITS2 e os três genes rRNA codificação, 17-18, 5.8S e 25-28S subunidade12. As duas regiões ITS não são parte da sequência de codificação das subunidades ribossomais. Eles são removidos pelo menos três actividades enzimáticas para processar o precursor para amadurecer rRNA: uma atividade endonuclease, helicase e do exonuclease. Como o RNA ribossomal é transcrito como um transcrito policistrônico, um produto primário contendo o STI está certamente presente. O processamento é muito rápido e tem lugar no nucléolo, e a quantidade de moléculas detectáveis precursor contendo o ITS está abaixo do limite de detecção do método qPCR. Portanto, quando ITS1 ou ITS2 são amplificados pelo seu flanqueando as primeiras demão, sem amplificação pode ser detectada em amostras de RNA a menos gDNA contaminação está presente. O número de rDNA genes no genoma de organismos eucariotas, estimava-se incluir até um mil cópias, que estão dispostas em matrizes simples ou em tandem no cromossomos11. Neste protocolo, propomos uma maneira alternativa, em vez de NRT, para detectar a contaminação gDNA, que é usada em cada reação/ensaio.
Benefícios e limitações no que diz respeito a métodos existentes: NRT é normalmente usado para testar se a amostra preparada do RNA é limpa ou contaminada por gDNA. Desde gDNA contaminação não é distribuída uniformemente entre diferentes amostras de RNA, e a sensibilidade de reação para gDNA é significativamente afetada pelos genes analisados, NRT controles são necessários para cada par de amostra/ensaio7,15. Isso adicionará substancialmente o custo e trabalho ao manusear muitas amostras simultaneamente3,9. Outros métodos alternativos, documentados na literatura incluem o uso de primers específicos de intron para a detecção de gDNA, ou projetar primers que flanqueiam um intrão ou abrangem uma junção exon-exon. As limitações destes métodos decorrem a indisponibilidade de informações de sequência intrão anotação incompleta da estrutura intron/exon e a ausência de intrões de genes ou pseudogenes de interesse1,4,10 . Devido à evolução, genes rDNA existirem como famílias multigene e altamente conservada do gene. Eles são muito abundantes no genoma e presente em diferentes cromossomos13. Em comparação com outros genes codificação ou nonconding, os genes rDNA mostram o melhor ajuste para a deteção de contaminação gDNA. Em análises comparativas de transcriptomic, a normalização dos dados qPCR pelo calibrador rRNA não é recomendada para algumas questões, tais como diferenças no cDNA preparação (polyA escorva vs escorva hexâmero aleatório), grandes diferenças em abundância entre rRNA e mRNA , e biogênese diferente que pode gerar enganosa resulta10,16. No entanto, os problemas que só mencionamos são uma vantagem para o ensaio de contaminação gDNA. Por exemplo, no que diz respeito a maior abundância local alvo no genoma e localização em diferentes cromossomas, as primeiras demão rDNA-based melhorar significativamente a sensibilidade de detecção de gDNA em comparação com os métodos existentes.
Versatilidade de rDNA-based para outro organismo: rDNA genes são uma família bem estudado gene identificada na maioria dos organismos. O método proposto baseia rDNA representa um sistema simples, altamente sensível e econômico para os ensaios de contaminação gDNA que pode ser facilmente adaptado para outros organismos procariotas e eucariotas (protocolo 2 - 5). Como um estudo de caso, demonstramos aqui a utilidade desse método em algumas espécies de Poceae (Figura 4 e Figura 5). Os primers utilizados mostram uma alta taxa de transferência para outras espécies de Poceae, devido à estrutura altamente conservada de rDNA subunidades entre espécies. Esta questão se torna ainda mais importante quando informações suficientes de sequência genômica não estão disponíveis para o projeto da primeira demão. Assim, as primeiras demão ITS-flanqueando projetadas para uma espécie podem ser usadas em uma espécie relacionada. Além disso, o 5.8S-primeiras demão de F/R foram escolhidas com base em um motivo conservado que mostra alta similaridade na maioria das plantas floríferas14. Embora técnicas de sequenciamento de alto throughput permanentemente aumentam o número de genomas conhecidos, a anotação de intron-exon da maioria dos organismos não é concluída, e assim muitas vezes não é possível projetar primers para abranger uma fronteira exon-exon. Nosso método explica rDNA-baseado como primeiras demão pode ser aplicada para o ensaio de contaminação gDNA na análise de qPCR de procariotas e eucariotas com o objetivo de eliminar os controles NRT caros em cada combinação de ensaio/primer.
Divulgações
Os autores têm sem interesses financeiros concorrentes.
Agradecimentos
Esta pesquisa foi apoiada pela genética e agrícola Instituto de biotecnologia de Mazandaran (GABIT), Ciências Agrárias Sari e Universidade de recursos naturais (SANRU). O grupo de Júnior pesquisa genômica de estresse abiótico foi financiado por IZN (Centro Interdisciplinar de pesquisa de planta de colheita, Halle (Saale), Alemanha. Agradecemos a Rhonda Meyer pela leitura crítica do manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| Maxima SYBR Green / ROX qPCR Master Mix (2X) | Thermo Scientific | K0221 | |
| TissueLyser II | QIAGEN | 85300 | |
| RevertAid H Minus First Strand cDNA Synthesis Kit | Thermo Scientific | K1631 | |
| GeneRuler 100 bp Plus DNA Ladder | Thermo Scientific | SM0321 | |
| 96 well WHT/CLR | Bio-Rad | HSP9601 | |
| Microseal B film | Bio-Rad | MJ-0558 | |
| Low tube strip CLR | Bio-Rad | TLS0801 | |
| Flat cap strips | Bio-Rad | TCS0803 | |
| NanoDrop 2000 | Peqlab | ND-2000 | |
| RNaseZAP | Ambion | 9780 | |
| Centrifuge | Eppendorf | 5810 R | |
| Agilent RNA 6000 Nano Kit | Agilent Technologies | 5067-1511 | |
| 2100 Electrophoresis Bioanalyzer | Agilent Technologies | G2939AA | |
| RNase A, DNase and Protease-free | Thermo Scientific | EN0531 | |
| DNase I, RNase-free | Thermo Scientific | EN0523 | |
| TRIZOL Reagent | Ambion | 15596026 | |
| CFX96 Touch Real-Time PCR Detection System | BIO RAD | 1855195 | |
| PCR tube, 0.2 mL, RNase-free | Stratagene | Z376426 | |
| Guanidine thiocyanate for molecular biology | Sigma-Aldrich | G9277 | |
| Agarose - Nucleic Acid Electrophoresis | Sigma-Aldrich | A9414 | |
| Boric Acid for molecular biology | AppliChem | A2940 | |
| bromophenol blue | AppliChem | A2331 | |
| ethidium bromide | AppliChem | A1151 | |
| Gel documentation system | BIO RAD | Gel Doc 2000 |
Referências
- Bustin, S. A., Nolan, T. Pitfalls of quantitative real-time reverse-transcription polymerase chain reaction. J Biomol Tech. 15 (3), 155-166 (2004).
- Gutierrez, L., Mauriat, M., Pelloux, J., Bellini, C., Van Wuytswinkel, O. Towards a systematic validation of references in real-time RT-PCR. Plant Cell. 20 (7), 1734-1735 (2008).
- Hashemi, S. H., Nematzadeh, G., Ahmadian, G., Yamchi, A., Kuhlmann, M. Identification and validation of Aeluropus littoralis reference genes for Quantitative Real-Time PCR Normalization. J Biol Res (Thessalon). 23 (1), 18(2016).
- Bustin, S. A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. J. Mol. Endocrinol. 25 (2), 169-193 (2000).
- Andersen, C. L., Jensen, J. L., Orntoft, T. F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res. 64 (15), 5245-5250 (2004).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clin Chem. 55 (4), 611-622 (2009).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40 (7), e51(2012).
- Galiveti, C. R., Rozhdestvensky, T. S., Brosius, J., Lehrach, H., Konthur, Z. Application of housekeeping npcRNAs for quantitative expression analysis of human transcriptome by real-time PCR. RNA. 16 (2), 450-461 (2010).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40, e51(2012).
- Caldana, C., Scheible, W. R., Mueller-Roeber, B., Ruzicic, S. A quantitative RT-PCR platform for high-throughput expression profiling of 2500 rice transcription factors. Plant Methods. 3 (1), 7(2007).
- Lawrence, R. J., Pikaard, C. S. Perspectives Chromatin Turn Ons and Turn Offs of Ribosomal RNA Genes. Cell Cycle. 3 (7), 880(2004).
- Boisvert, F. M., van Koningsbruggen, S., Navascues, J., Lamond, A. I. The multifunctional nucleolus. Nat Rev Mol Cell Biol. 8 (7), 574-585 (2007).
- Alvarez, I., Wendel, J. F. Ribosomal ITS sequences and plant phylogenetic inference. Mol Phylogenet Evol. 29 (3), 417-434 (2003).
- Jobes, D. V., Thien, L. B. A conserved motif in the 5.8 S ribosomal RNA (rRNA) gene is a useful diagnostic marker for plant internal transcribed spacer (ITS) sequences. Plant Mol Biol Report. 15 (4), 326-334 (1997).
- Padhi, B. K., Singh, M., Huang, N., Pelletier, G. A PCR-based approach to assess genomic DNA contamination in RNA: Application to rat RNA samples. Anal Biochem. 494, 49-51 (2016).
- Dheda, K., et al. Validation of housekeeping genes for normalizing RNA expression in real-time PCR. Biotechniques. 37 (1), 112-114 (2004).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados