É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Atuador baseado em células musculares cardíacas e auto-estabilizante Biorobot - Parte 2
Neste Artigo
Resumo
Neste estudo, um atuador biológico e um biorobot de natação auto-estabilizante com braços elastoméricos em cantilever funcionalizados são semeados com cardiomiócitos, cultivados e caracterizados por suas propriedades bioquímicas e biomecânicas ao longo do tempo.
Resumo
Nos últimos anos, foram desenvolvidos dispositivos híbridos que consistem numa célula viva ou componente de tecido integrado com uma espinha dorsal mecânica sintética. Estes dispositivos, chamados biorobots, são alimentados apenas pela força gerada a partir da atividade contrátil do componente vivo e, devido às suas muitas vantagens inerentes, poderia ser uma alternativa aos robôs convencionais totalmente artificiais. Aqui, descrevemos os métodos para semear e caracterizar um atuador biológico e um biorobot que foi projetado, fabricado e funcionalizado na primeira parte deste artigo em duas partes. O actuador biológico fabricado e os dispositivos biorobot compostos por uma base de polidimetilsiloxano (PDMS) e um cantilever de película fina foram funcionalizados para ligação de células com fibronectina. Após a funcionalização, os cardiomiócitos de ratos neonatais foram semeados no braço cantilever PDMS a uma densidade elevada, resultando numa folha de células confluentes. Os dispositivos foram visualizados todos os dias eo movimento do cantiBraços de alavanca foi analisada. No segundo dia após a semeadura, observamos a flexão dos braços em consola devido às forças exercidas pelas células durante as contrações espontâneas. Após análise quantitativa da dobra em cantilever, observou-se um aumento gradual no stress superficial exercido pelas células à medida que amadureciam ao longo do tempo. Da mesma forma, observamos o movimento do biorobot devido à atuação do braço cantilever PDMS, que atuou como uma aleta. Após a quantificação dos perfis de natação dos dispositivos, foram observados vários modos de propulsão, que foram influenciados pelo ângulo de repouso da barbatana. A direção do movimento e a freqüência de batimento também foram determinadas pelo ângulo de repouso da barbatana, e observou-se uma velocidade máxima de natação de 142 μm / s. Neste artigo, descrevemos o procedimento para preencher os dispositivos fabricados com cardiomiócitos, bem como para a avaliação do atuador biológico e atividade biorobot.
Introdução
Biorobots são dispositivos baseados em células vivas que são incorporados dentro de um backbone mecânico que é geralmente composto de materiais macios, elásticos, como PDMS ou hidrogéis 1 . As células sofrem contrações rítmicas, espontaneamente ou em resposta a estímulos, e assim funcionam como um atuador. A energia gerada a partir da célula de contração drives vários biorobots. As células do coração de mamíferos (cardiomiócitos) e as células do músculo esquelético são frequentemente utilizadas para a actuação biorobot devido às suas propriedades contrácteis. Além de cardiomiócitos e células musculares esqueléticas, foram utilizados outros tipos de células, tais como tecidos musculares de insectos 2 e tecidos musculares explantados 3 . Os tecidos musculares de insetos permitem a operação de atuadores biológicos à temperatura ambiente.
A função eo desempenho de um biorobot são determinados principalmente pela força e consistência do atuador biológico ( ie. Células musculares), enquanto que a estrutura principal mecânica determina os mecanismos de locomoção, estabilidade e potência. Uma vez que estes dispositivos são exclusivamente movidos por forças geradas por células, não existem poluentes químicos ou ruídos operacionais. Portanto, eles formam uma alternativa de energia eficiente para outros robôs convencionais. Várias fontes da literatura discutiram os diferentes métodos para integrar células vivas e tecidos em biorobots 1 , 4 , 5 . Os avanços na microfabricação e técnicas de engenharia de tecidos permitiram o desenvolvimento de biorobôs que podem andar, segurar, nadar ou bombear 5 , 6 . Em geral, as células são cultivadas directamente sobre o esqueleto mecânico (polimérico) como uma folha de células confluentes ou são moldadas em estruturas de actuação tridimensionais dentro de andaimes tais como anéis e tiras. Na maioria das vezes, biorobots sãoFabricadas utilizando folhas de cardiomiócitos 6 , 7 , uma vez que estas células têm uma capacidade inata para exibir uma contracção espontânea sem estímulos externos. Por outro lado, os relatos sobre as folhas de células do músculo esquelético são limitados devido à sua necessidade de estímulos para iniciar as contrações in vitro , a fim de iniciar a despolarização da membrana 8 .
Este protocolo descreve, em primeiro lugar, como semear cardiomiócitos num actuador biológico funcionalizado feito de um cantilever PDMS fino. Em seguida, descreve em detalhes a semeadura e análise dos perfis de natação. O cantilever é funcionalizado com uma proteína adesiva celular tal como a fibronectina e é semeado confluentemente com cardiomiócitos. À medida que as células semeadas no dispositivo se contraem, elas fazem com que o cantilever se dobre e assim actue como um actuador. Ao longo do tempo, à medida que as células se amadurecem, rastreamos as mudanças no estresse superficial no dispositivo, analisando vídeosDobra em cantilever. O actuador biológico desenvolvido aqui pode ser utilizado para determinar as propriedades contrácteis de qualquer tipo de célula, tal como os fibroblastos ou células de caules pluripotentes induzidas, à medida que sofrem diferenciação.
Grande parte da pesquisa anterior sobre biorobôs foi focada no desenvolvimento de atuadores biológicos, enquanto a otimização da arquitetura biorobot e capacidades funcionais foram largamente negligenciadas. Recentemente, alguns estudos têm demonstrado a implementação de modos de natação em biorobôs que são inspirados pela natureza. Por exemplo, os biorobots de natação com movimento baseado em flagelos 6 , propulsão de água-viva 9 e raios bio-híbridos 4 foram projetados. Ao contrário de outros trabalhos na literatura, aqui nos concentramos em variar as propriedades da espinha dorsal mecânica para criar uma estrutura de auto-estabilização. O biorobot desenvolvido neste estudo é capaz de manter um passo constante, roll, e imMersion como ele nada. Estes parâmetros podem ser modificados variando a espessura de cada composto de base. Os passos de fabricação envolvidos no desenvolvimento do atuador PDMS, o biorobot submergível e a funcionalização do dispositivo são descritos detalhadamente na Parte 1 deste artigo em duas partes, bem como em nosso recente trabalho 7. A técnica desenvolvida aqui pode pavimentar o Para o desenvolvimento de novos, altamente eficientes biorobots para várias aplicações, tais como entrega de carga.
O processo de isolamento seguido neste estudo é semelhante ao processo descrito em um trabalho anterior 10 , bem como em trabalhos recentemente publicados 7 . Os métodos de microfabricação utilizados para fabricar os actuadores PDMS e os dispositivos biorobot são descritos em pormenor na Parte 1 deste manuscrito de duas partes. A seção de protocolo deste manuscrito descreve os passos envolvidos na semeadura de cardiomiócitos no PDMS fabricado aE o biorobot após a sua funcionalização com proteínas adesivas celulares.
Protocolo
Todos os procedimentos aqui descritos foram realizados utilizando um protocolo aprovado e de acordo com os regulamentos do Comitê Institucional de Cuidados e Uso de Animais da Universidade de Notre Dame.
1. Semente e Cultura de Células
- Antes de iniciar, prepare os itens necessários: um pequeno funil, pipetas e meio de Eagle modificado Dulbecco morno (DMEM) suplementado com 10% de soro fetal bovino (FBS) e 1% de antibiótico penicilina (DMEM completo).
- Tome frascos T-25 juntamente com o dispositivo funcionalizado (atuador biológico ou biorobot) dentro dele. Consulte a seção 4 da Parte 1 deste manuscrito em duas partes para obter detalhes sobre a preparação do dispositivo, a funcionalização e o armazenamento antes da semeadura celular.
- Prepare um funil, que pode ser feito rolando uma pequena folha quadrada de plástico. Coloque-o sobre o atuador biológico ou biorobot dentro do frasco T-25. Ajuste o diâmetro da extremidade mais larga para ajustar o dispositivo inteiroE a altura de modo a que encaixe confortavelmente quando o topo do balão é apertado.
- Para os biorobôs, use um íman para manter o dispositivo no lugar no fundo do frasco durante todo o processo de semeadura.
Nota: Aqui, um único ímã de disco de neodímio (1,26 "de diâmetro) foi usado, mas qualquer ímã de tamanho e força semelhantes também pode ser usado para manter o biorobot com a base compósita magnética de níquel-PDMS. - UV esterilizar a folha de plástico por pelo menos 30 min antes da utilização.
- Para os biorobôs, use um íman para manter o dispositivo no lugar no fundo do frasco durante todo o processo de semeadura.
- Certifique-se de que não haja um grande espaço entre a base do funil eo balão.
- Ressuspender os cardiomiócitos em DMEM completo a uma densidade de 1,6 x 10 7 células / mL e deixar cair lentamente 400 μL de suspensão para o dispositivo através do funil. Use um hemocitômetro ou qualquer outro contador de células para determinar o número de células obtidas.
- Lentamente mover o sistema de volta para a incubadora sem perturbar o dispositivo eo funil witHin. Cultura durante 24 h a 37 ° C.
- Após o período de incubação, retire lentamente o funil, lave cuidadosamente a amostra com PBS, e reabasteça o balão com 10 mL de DMEM fresco completo.
Nota: Para os biorobôs, remova o íman para que o dispositivo flutue.
2. Caracterização bioquímica
- Ensaio de fluxo de cálcio
Nota: O ensaio de fluxo de cálcio é realizado para avaliar a interconectividade das células. O procedimento para carregar as células com o corante fluorescente, específico do ião cálcio, segue o processo descrito num protocolo previamente estabelecido 11 .- Primeiro, prepare os materiais requeridos, éster de fluo-4-acetometil (AM) de cálcio, poliol surfactante não iónico (ver Tabela de Materiais ) e solução de sal de Tyrode.
- Usando pinças longas, transferir suavemente o dispositivo do frasco de cultura para uma placa de Petri de 35 mm com 2 mL de solução salina de Tyrode Ção.
- Em um tubo de centrífuga separado, tomar 1 mL de solução de Tyrode quente (aquecida a 37 ° C) e adicionar 3-5 μL de corante de cálcio fluo-4 AM (concentração de trabalho: 3-5 μM) e uma parte igual de surfactante não iónico Poliol (concentração de trabalho: 0,2%). Substituir a solução da amostra por uma solução quente de Tyrode suplementada com o corante indicador de cálcio, fluo-4 e 0,2% de surfactante não iónico. Incubar durante 25 - 30 min a 37 ° C.
- Remover a solução de corante e lavar cuidadosamente a amostra com a solução fresca de Tyrode. Re-incubar a amostra em 2 mL de DMEM fresco completo durante mais 30 min a 37 ° C antes da imagem.
Nota: Os resultados deste ensaio e vídeo relacionados são fornecidos no trabalho publicado anteriormente 7 .
- Imunofluorescência
Nota: A imunocoloração dupla de todas as amostras foi realizada seguindo o protocolo previamente estabelecido> 12.- Primeiro, preparar 10% de soro de cabra (GS) em solução salina tamponada com fosfato (PBS), paraformaldeído a 4% (PFA) em diH2O, 0,1% de detergente de lise celular (ver Tabela de Materiais ) em PBS, anticorpos primários Anticorpos monoclonais de murganho monoclonal cardíaco troponina I e anticorpos monoclonais anti-coelho conexinas 43), anticorpos secundários (conjugado Alexa 594 e conjugado de cabra anti-IgG de rato anti-IgG de rato (Alexa 488) e DAPI.
Cuidado: Paraformaldeído é cancerígeno. - Remover a amostra de interesse do balão e lavá-la suavemente duas vezes com PBS. Consulte a seção 4 na Parte 1 deste manuscrito em duas partes para obter detalhes sobre a preparação da amostra e a sua funcionalização.
- Adicione uma gotícula de PBS em uma pequena lamela (diâmetro: 12 mm ou 15 mm). Segure delicadamente a base do dispositivo com uma pinça e corte os braços PDMS finos (cantilever, Figura 1 ) usando tesouras da extremidade onde se conecta ao topo da baseE. Transfira os braços cantilever para a gotícula com o lado aderente à célula voltado para cima. A gota de PBS impedirá que as células seque.
- Fixar as amostras com PFA a 4% e realizar a imunomarcação dupla das amostras, como descrito anteriormente 12 .
- Após a imunocoloração, montar as amostras em uma lâmina de vidro limpo usando anti-fade montagem reagente e reservado, sem ser perturbado, no escuro por 24 h.
- Repita o procedimento para todas as amostras.
Nota: Os resultados deste ensaio e as imagens relacionadas são discutidos em profundidade no trabalho previamente publicado 7 .
- Primeiro, preparar 10% de soro de cabra (GS) em solução salina tamponada com fosfato (PBS), paraformaldeído a 4% (PFA) em diH2O, 0,1% de detergente de lise celular (ver Tabela de Materiais ) em PBS, anticorpos primários Anticorpos monoclonais de murganho monoclonal cardíaco troponina I e anticorpos monoclonais anti-coelho conexinas 43), anticorpos secundários (conjugado Alexa 594 e conjugado de cabra anti-IgG de rato anti-IgG de rato (Alexa 488) e DAPI.
3. Imagens
- Colocar o frasco T-25 ereto numa incubadora de CO 2 e preparar o sistema de imagem dentro da incubadora. Grave o dispositivo usando uma câmera (consulte a Tabela de Materiais ) com uma lente de zoom (consulte a Tabela de Materiais ). Para uma fonte de luz,Use uma faixa de luzes LED.
Nota: Uma tira de LEDs de luz branca foi usada aqui, mas qualquer LED normal também funcionará. - Conecte a câmera a um sistema operacional e abra o software específico da câmera (consulte a Tabela de Materiais ). Clique na imagem da câmera embaixo da guia "Arquivo", no painel superior, para abrir todas as opções da câmera e escolha a câmera correta.
- Escolha "live" na lista de guias no painel superior dentro do software.
- Coloque manualmente a imagem em foco, ajustando o mostrador da lente. Selecione "Cortar para a região de interesse (ROI)" no painel superior. Em seguida, desenhe manualmente um retângulo na moldura de vídeo, encerrando o dispositivo de atuador biológico e os braços em consola, para marcar o ROI.
Nota: Escolher um ROI adequado minimiza o tamanho dos ficheiros de imagem.- No caso de biorobots, capturar toda a tela, a fim de gravar o movimento de natação do dispositivo.
NOTA: Não há necessidade de desenhar um ROI para os biorobôs
- No caso de biorobots, capturar toda a tela, a fim de gravar o movimento de natação do dispositivo.
- Antes de iniciar a gravação, selecione "Configurações da câmera" em uma das guias no painel superior da tela. Defina a taxa de quadros ajustando a relação de exposição e pixel da imagem ao vivo, deslizando a barra para cada um ou inserindo manualmente os valores. Defina as taxas de quadros para cerca de 30 ± 2 fps.
Nota: Alterar a relação de exposição e pixel altera o brilho eo contraste da imagem ao vivo. - Clique no botão "Gravar" do painel superior do software para começar a gravar os vídeos dos atuadores com resolução de 1.000 x 1.000 pixels para precisamente 30 s. Repita o processo para todas as amostras.
4. Análise de imagem dos atuadores biológicos em uma base estacionária
- Analise as imagens usando um software de programação ( por exemplo, Matlab) executando o script personalizado. Veja a Tabela de Materiais eo Arquivo Suplementar para mais detalhes.
Nota: O script exibe cada quadro dos vídeos gravados, recebe a entrada do mouse do usuário para registrar a coordenação dos pontos do cantilever nas imagens, calcula o diâmetro e o centro do círculo que passa os pontos introduzidos por meio do encaixe pelo menos quadrado e Exporta todos os dados introduzidos e calculados para utilização posterior.- Abra o software de programação clicando no ícone. Clique em "Arquivo" -> "Abrir" na barra de menu na parte superior e selecione o arquivo de script .m para análise de imagem. Verifique se as imagens TIFF gravadas estão na mesma pasta que o arquivo .m. Clique em "Executar" para executar o script.
Nota: Uma tela interativa aparecerá para alterar. - Pressione "play" para iniciar o programa real. Clique no botão "abrir" e localize o arquivo TIFF que vai ser analisado.
- Clique no botão "base & #34; Botão e, em seguida, clique no ponto onde o cantilever atribui à base no topo; Pressione Enter. Isto irá colocar um marcador quadrado na imagem para cada quadro para denotar a localização da base cantilever.
- Clique no botão "Escala" e, em seguida, clique manualmente em uma borda do grânulo de vidro. Traga o ponteiro do mouse para o lado oposto da conta de vidro e pressione "Enter".
Nota: Isto deve traçar uma linha que mede o diâmetro do cordão de vidro. Uma vez que o rebordo de vidro tem 3 mm de diâmetro, isto irá relacionar 3 mm com os pixels apresentados. - Clique no botão "Analisar". Clique ao longo do cantilever a uma curta distância do primeiro marcador quadrado que representa a base cantilever.
- Clique no botão "Analisar". Em seguida, clique ao longo do cantilever a uma curta distância do primeiro marcador quadrado que representa a base cantilever. Continue a clicar ao longo do cantilever, incluindo a ponta, e pressione enter quandofeito. Isto colocará um "x" em cada ponto clicado no cantilever.
Nota: Com base na coordenação do marcador quadrado e nos marcadores x, o centro e o diâmetro de um círculo serão calculados usando uma função de encaixe pelo menos quadrada (consulte o arquivo suplementar anexado para o script utilizado). O círculo, que passa os marcadores x eo criador de quadrados, será sobreposto automaticamente à imagem. - Verifique se o círculo sobreposto traça corretamente o perfil do cantilever.
Nota: Quando o cantilever é muito plano, é difícil julgar se o perfil do cantilever é traçado corretamente. Consulte a Figura 3 . - Clique no botão "próximo quadro". Isto irá mudar para o próximo quadro no ficheiro TIFF. A base ea escala já estão definidas a partir da etapa anterior.
- Repita as etapas 4.1.5 até 4.1.7 até que todos os quadros no arquivo TIFF tenham sido concluídos. Uma vez que todos os quadros foram proceSsed, clique no botão "Exportar".
Nota: Isso fará um arquivo de planilha com o nome do arquivo TIFF para o cantilever que acabou de ser analisado. Edite o nome do arquivo para incluir qual lado (esquerdo ou direito) do cantilever foi analisado.
- Abra o software de programação clicando no ícone. Clique em "Arquivo" -> "Abrir" na barra de menu na parte superior e selecione o arquivo de script .m para análise de imagem. Verifique se as imagens TIFF gravadas estão na mesma pasta que o arquivo .m. Clique em "Executar" para executar o script.
- Calculando o estresse na planilha.
- Calcule a tensão superficial "σ" no cantilever usando a seguinte equação:
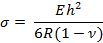
Onde E, R, v e h são o módulo de Young, o raio de curvatura, a razão de Poisson ea espessura em consola, respectivamente.
Nota: A espessura do cantilever pode ser alterada para alterar a sensibilidade. Neste estudo, os valores foram os seguintes: E = 750 kPa, v = 0,49 e h = 25 μm 13 , 14 . - Calcular a tensão superficial utilizando a equação do passo 4.2.1. Abra o .Xls arquivo de planilha. Observe que a saída tem várias colunas mostrando primeiro as coordenadas x e y de base e círculo e, em seguida, o raio de curvatura. Calcule as coordenadas xey de cada ponto clicado com base nestes.
Nota: A representação gráfica das tensões sobre os quadros de imagem de intervalo de tempo mostra mudanças na força exercida sobre o cantilever ao longo do tempo. As depressões mostram o estresse sobre o cantilever durante o relaxamento dos cardiomiócitos ou a tensão estática exercida sobre o cantilever devido às forças de tração celular. Os picos mostram o estresse dinâmico no cantilever, que foi exercido pelo batimento dos cardiomiócitos. Este valor corresponde à quantidade máxima de força gerada pela contracção dos cardiomiócitos.
- Calcule a tensão superficial "σ" no cantilever usando a seguinte equação:
5. Análise de Natação Biorobots
- Registre a posição do biorobot usando o software de análise de imagens.
Nota: Consulte a lista de Materiais para o softwareÉ seção.- Abra o software de análise de imagem ( por exemplo, ImageJ). Pressione "Arquivo" e "Abrir" e selecione o arquivo de vídeo biorobot natação. Clique em "OK" e deixe o programa carregar o arquivo. Abra o software da folha de cálculo.
- No vídeo biorobot carregado, encontre uma referência de dimensões conhecidas ( por exemplo, o rebordo de vidro de 3 mm de diâmetro que foi incorporado no atuador biológico).
Nota: Qualquer objeto com dimensão conhecida funcionará. Isso determinará as proporções de pixel para comprimento em cada vídeo. - Use a ferramenta "Straight" para desenhar uma linha em toda a pérola de vidro. Clique em "Analisar" e selecione "Definir escala". Defina o campo "Distância conhecida" para "3.000 μm" e clique em "Ok".
Nota: Isto irá definir as coordenadas x e y para micrômetros. - Escolha um ponto no dispositivo que não oscila entre quadros para agir como um marcador.
Nota:Recomenda-se escolher um canto da base. - Aponte para o ponto selecionado em 5.1.4 no primeiro quadro. Registre as coordenadas x e y na planilha.
- Volte para a janela do software de análise de imagem e pressione a tecla de seta para a direita para mudar para a próxima moldura. Aponte novamente para o marcador (a partir do passo 5.1.4) e registre as coordenadas x e y na planilha.
- Repita o passo 5.1.6 para todos os quadros.
- Calcule os parâmetros de natação do biorobot usando a planilha de coordenadas 7 .
- Calcule o período entre quadros a partir da taxa de quadros conhecida de cada vídeo.
- Calcule a mudança nas coordenadas x e y entre quadros para encontrar a distância movida, incluindo a distância total.
- Calcule a amplitude de contração da mudança máxima ao longo do eixo y. Determine a frequência de batimento para cada biorobot do inverso do período entre duas contrações.
- CCalcular a velocidade de natação de cada dispositivo a partir do tempo total e da distância movida na direção x.
- Repita o passo 5.2 para cada vídeo biorobot que foi analisado.
- Normalize cada parâmetro medido.
Nota: Normalize todos os valores para melhor visualizar as diferenças. Este protocolo demonstra normalização com relação a um biorobot de modo horizontal com contrações de baixa frequência (LF horizontal) ( Figura 4 ) 7 .
6. Análise da Expressão de Proteína
Nota: As amostras montadas preparadas nos passos 2.2.4 e 2.2.5 foram visualizadas utilizando um microscópio confocal. As imagens foram adquiridas em amplificação 20X, 40X e 60X sequencialmente em três canais simultaneamente: 460 nm, 488 nm e 594 nm. Um conjunto de 5 imagens foram capturadas a uma ampliação de 40X, a partir de posições diferentes para cada amostra, e cada canal foi guardado como um indivíduo.Arquivo. O ajuste de exposição foi determinado pela ampliação do objectivo utilizado e foi fixado constante para todas as capturas nessa ampliação.
- Abra o software de análise de imagem e selecione "Arquivo" -> "Abrir" para carregar as imagens.
- Desenhe um polígono retangular na moldura da imagem para marcar o ROI. Selecione "Analisar" -> "Medir" para medir a intensidade fluorescente média.
- Repita o passo 6.2 para coletar medições de intensidade de todas as amostras e calcular a respectiva intensidade média para cada condição.
Nota: Aqui, uma condição diferente refere-se a diferentes pontos de tempo, como em, dia 1, dia 2 e até dia 6. - Exporte os resultados em uma planilha para análise estatística adicional e para a produção de gráficos de dados.
Resultados
O atuador biológico feito de um cantilever PDMS fino (25 μm de espessura) e cardiomiócitos constitui o núcleo do biorobot de natação, conforme mostrado na esquemática e captura de tela dos dispositivos na Figura 1 . As células começam a apresentar contracções após 24 h em cultura e a flexão dos braços em consola foi observada até ao dia 2. O perfil lateral do dispositivo foi registado todos os dias e o stress superficial foi quantificado a partir da dobra ...
Discussão
O procedimento descrito aqui descreve um método de sementeira bem sucedido para actuadores e biorobots baseados em PDMS, o que facilita a ligação de cardiomiócitos. Além disso, foi descrito o processo de aquisição de imagem e a subsequente análise que caracteriza o comportamento das células eo desempenho dos dispositivos.
Observamos a contração espontânea de células nos braços cantilever após 24 h; A intensidade das contrações continuou a aumentar de forma constante ao longo...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
MT Holley é apoiado pelo programa Graduate Fellows da Louisiana Board of Regents, e C. Danielson é apoiado pelo Programa de Professores do Instituto Médico Howard Hughes. Este estudo é apoiado pelo NSF Grant No: 1530884.
Materiais
| Name | Company | Catalog Number | Comments |
| Chemicals and reagents | |||
| Cardiomyocytes (primary cardiac cells) | Charles River | NA | Isolated from 2-day old neonatal Sprague Dawley rats |
| Dulbecco’s modified eagle’s media (DMEM) | Hyclone Laboratories | 16750-074 | with 4500 mg/L glucose, 4.0 mM L-glutamine, and 110 mg/L sodium pyruvate |
| Fetalclone III serum | Hyclone industries, GE | 16777-240 | Fetal bovin serum (FBS) |
| Dulbecco’s phosphate buffer (PBS) | Sigma-Aldrich | D1408-100ML | |
| Penicillin-G sodium salt | Sigma-Aldrich | P3032 | |
| Goat serum | Sigma-Aldrich | G9023 | |
| 4,6-diamidino-2-phenylindole dihydrocholride powder (DAPI) | Sigma-Aldrich | D9542 | |
| Fibronectin from bovine plasma | Sigma-Aldrich | F1141 | Solution (1 mg/ml) |
| Calcein-AM and ethidium homodimer-1 kit (Live/Dead Assay) | Molecular Probes | L3224 | |
| Calcium Fluo-4, AM | Molecular Probes | F14217 | calcium indicator dye |
| Tyrodes salt solution | Sigma-Aldrich | T2397 | buffer solution |
| Pluronic F-127 | Molecular Probes | P3000MP | nonionic surfactant-20 % solution in Dimethylsiloxane (DMSO) |
| 16% Parafomaldehyde | Electron microscopy | 15710 | Caution: Irritant and combustible |
| Triton x-100 | Sigma-Aldrich | X-100 100 mL | cell lyses detergent, (4-(1,1,3,3-Tetramethylbutyl)phenyl-polyethylene glycol, t-Octylphenoxypolyethoxyethanol, Polyethylene glycol tert-octylphenyl ether) |
| ProLong gold antifade reagent | Molecular Probes | P10144 | Mounting agent |
| Alexa Fluor 594 Phalloidin | Molecular Probes | A12381 | Actin filament marker |
| Goat anti-rabbit IgG (H+L) secondary antibody, Alexa Fluor 594 conjugate | Molecular Probes | A-11012 | |
| pha | Molecular Probes | A-11001 | |
| Anti-connexin 43 antibody | Abcam | ab11370 | Gap junction marker |
| Anti-cardiac troponin I antibody | Abcam | ab10231 | Contractile protein |
| 16% EM grade paraformaldehyde solution | Electron microscopy | 100503-916 | |
| Polydimethylsiloxane (PDMS) | Elsevier | Sylgard 184 | |
| Materials and Equipment | |||
| Camera | Thor Labs | DCC1545M | |
| LED light strip | NA | NA | Any white LED without spectrum emission |
| Confocal microscope | Nikkon C2 | NA | Confocal microscope with three filter set. |
| Zooming lens | Infinity | Model# 252120 | |
| Software | |||
| Matlab | Mathworks | NA | Used in Section 4) for biological actuator analysis. |
| Image J | National Institute of Health | NA | Java-based image processing software. Used in Section 5) for biorobot analysis. Free Image Processing and Analysis software in java. (https://imagej.nih.gov/ij/) |
| Thor Cam | Thor Labs | NA | Camera operating software |
Referências
- Feinberg, A. W. Biological Soft Robotics. Annu. Rev. Biomed. Eng. 17, 243-265 (2015).
- Akiyama, Y., et al. Room Temperature Operable Autonomously Moving Bio-Microrobot Powered by Insect Dorsal Vessel Tissue. PLOS ONE. 7, 38274 (2012).
- Herr, H., Dennis, R. G. A swimming robot actuated by living muscle tissue. J. NeuroEng Rehabil. 1, 6 (2004).
- Park, S., et al. Phototactic guidance of a tissue-engineered soft-robotic ray. Science. 353 (6295), 158-162 (2016).
- Cvetkovic, C., et al. Three-dimensionally printed biological machines powered by skeletal muscle. Proc. Natl. Acad. Sci. 111, 10125-10130 (2014).
- Williams, B. J., Anand, S. V., Rajagopalan, J., Saif, M. T. A. A self-propelled biohybrid swimmer at low Reynolds number. Nat. Commun. 5, (2014).
- Holley, M. T., Nagarajan, N., Danielson, C., Zorlutuna, P., Park, K. Development and characterization of muscle-based actuators for self-stabilizing swimming biorobots. Lab. Chip. 16, 3473-3484 (2016).
- Hopkins, P. M. Skeletal muscle physiology. Contin Educ Anaesth Crit Care Pain. 6, 1-6 (2006).
- Nawroth, J., et al. A tissue-engineered jellyfish with biomimetic propulsion. Nat Biotechnol. 30 (8), 729-797 (2012).
- Ehler, E., Moore-Morris, T., Lange, S. Isolation and Culture of Neonatal Mouse Cardiomyocytes. J. Vis. Exp. JoVE. (79), e50154 (2013).
- Bers, D. M. Calcium Fluxes Involved in Control of Cardiac Myocyte Contraction. Circ. Res. 87, 275-281 (2000).
- Shin, S. R., et al. Carbon-Nanotube-Embedded Hydrogel Sheets for Engineering Cardiac Constructs and Biological actuators. ACS Nano. 7, 2369-2380 (2013).
- Park, J., et al. Real-Time Measurement of the Contractile Forces of Self-Organized Cardiomyocytes on Hybrid Biopolymer Microcantilevers. Anal. Chem. 77, 6571-6580 (2005).
- Tamayo, J., et al. Quantification of the surface stress in microcantilever biosensors: revisiting Stoney's equation. Nanotechnology. 23, 475702 (2012).
- Nunes, S. S., et al. Biowire: a platform for maturation of human pluripotent stem cell-derived cardiomyocytes. Nat. Methods. 10, 781-787 (2013).
- Louch, W. E., Sheehan, K. A., Wolska, B. M. Methods in Cardiomyocyte Isolation, Culture, and Gene Transfer. J. Mol. Cell. Cardiol. 51, 288-298 (2011).
- Alford, P. W., Feinberg, A. W., Sheehy, S. P., Parker, K. K. Biohybrid thin films for measuring contractility in engineered cardiovascular muscle. Biomaterials. 31, 3613-3621 (2010).
- Sfakiotakis, M., Lane, D. M., Davies, J. B. C. Review of fish swimming modes for aquatic locomotion. IEEE J. Ocean. Eng. 24, 237-252 (1999).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados