É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Predicando silenciamento de genes através do controle Spatiotemporal da liberação de siRNA de Nanocarriers poliméricos com fotos
Neste Artigo
Resumo
Apresentamos um novo método que usa copolímeros de blocos foto-responsivos para um controle spatiotemporal mais eficiente do silenciamento gênico sem efeitos detectáveis fora do alvo. Além disso, as mudanças na expressão gênica podem ser previstas usando ensaios diretos de liberação de siRNA e modelagem cinética simples.
Resumo
São necessários novos materiais e métodos para controlar melhor a ligação e a liberação de ácidos nucleicos para uma ampla gama de aplicações que requerem uma regulação precisa da atividade gênica. Em particular, os novos materiais responsivos aos estímulos com controle espaciotemporal melhorado sobre a expressão gênica destravam plataformas traduzíveis em tecnologias de descoberta de drogas e medicina regenerativa. Além disso, uma capacidade aprimorada para controlar a liberação de ácido nucleico a partir de materiais permitiria o desenvolvimento de métodos simplificados para prever a eficácia do nanocarrier a priori , levando a triagem acelerada dos veículos de entrega. Aqui, apresentamos um protocolo para prever a eficiência de silenciamento gênico e alcançar o controle espaciotemporal sobre a expressão gênica através de um sistema nanocarrier modificador fotossensível modular. O pequeno ARN de interferência (siRNA) é complexado com mPEG- b- poli (metacrilato de 5- (3- (amino) propoxi) -2-nitrobenzilo) (mPEG- b- P (APNBMA)) paraRm estabilidade nanocarriers que podem ser controlados com luz para facilitar sintonizável, on / off siRNA libertação. Descrevemos dois ensaios complementares que utilizam espectroscopia de correlação de fluorescência e eletroforese em gel para a quantificação precisa da liberação de siRNA a partir de soluções que imitam ambientes intracelulares. As informações obtidas a partir desses ensaios foram incorporadas em um modelo cinético simples de RNAi (RNAi) para prever as respostas de silenciamento dinâmico a várias condições de fotoestimulação. Por sua vez, essas condições de irradiação otimizadas permitiram o refinamento de um novo protocolo para controle espacial controlando o silenciamento gênico. Este método pode gerar padrões celulares na expressão de genes com resolução célula a célula e sem efeitos detectáveis fora do alvo. Em conjunto, nossa abordagem oferece um método fácil de usar para prever mudanças dinâmicas na expressão gênica e controlar com precisão a atividade de siRNA no espaço e no tempo. Este conjunto de ensaios pode ser facilmente adaptado para testar uma grande variedade de otSeus sistemas responsivos ao estímulo para abordar os principais desafios pertinentes a uma multiplicidade de aplicações em pesquisa biomédica e medicina.
Introdução
Os pequenos ARNs de interferência (siRNAs) medeiam o silenciamento de genes pós-transcrição através de uma via de RNAi catalítica que é altamente específica, potente e adaptada a praticamente qualquer gene alvo 1 . Essas características promissoras permitiram que a terapia com siRNA avançasse em ensaios clínicos em humanos para o tratamento de numerosas doenças, incluindo melanoma metastático e hemofilia 2 , 3 . No entanto, persistem persistentes problemas de entrega que prejudicaram a tradução 4 . Em particular, os veículos de entrega devem permanecer estáveis e proteger siRNAs de degradação extracelular, mas também liberar a carga útil no citoplasma 5 . Além disso, muitas aplicações de RNAi requerem métodos aprimorados para regular o silenciamento de genes no espaço e no tempo 6 , o que reduzirá os efeitos colaterais na terapêutica com siRNA 7 e possibilitará transformar aDvances em aplicações que vão desde microarrays celulares para a descoberta de drogas 8 até a modulação de respostas celulares em andaimes regenerativos 9 . Esses desafios destacam a necessidade de novos materiais e métodos para controlar melhor a ligação e a liberação nos nanocarriers de siRNA.
Uma das estratégias mais promissoras para controlar a liberação de siRNA e melhorar a regulação espaciotemporal é o uso de materiais sensíveis ao estímulo 10 . Por exemplo, uma grande variedade de biomateriais foram projetados com afinidade de ligação de ácido nucleico variável em resposta ao potencial redox ou pH alterado, ou campos magnéticos aplicados, ultra-som ou luz 11 . Embora muitos desses sistemas demonstrem um controle melhorado sobre a atividade do ácido nucleico, o uso da luz como gatilho é particularmente vantajoso devido à sua resposta temporal instantânea, resolução espacial precisa e facilidade de afinação 12. Além disso, o potencial das tecnologias sensíveis à foto para a regulação da expressão gênica foi demonstrado por sistemas de regulação induzível induciável e sistemas reguladores optogênicos de última geração; No entanto, esses sistemas sofrem de inúmeros desafios, incluindo capacidades limitadas para regular genes endógenos, preocupações de segurança, como imunogenicidade e dificuldades na entrega de conjuntos de componentes múltiplos 13 , 14 , 15 . Photo-responsive siRNA nanocarriers são ideais para superar essas desvantagens e proporcionar uma abordagem mais simples e mais robusta para spatiotemporally modular a expressão de genes 16 , 17 , 18 . Infelizmente, métodos para prever com precisão a resposta resultante da redução de proteína permanecem difíceis.
Um desafio fundamental é que as avaliações quantitativas da liberação de siRNA sãoRaro 19 , 20 , e mesmo quando essas avaliações são realizadas, elas não foram acopladas a análises da dinâmica de rotação de siRNA / proteína. Tanto a quantidade de siRNA liberada quanto a persistência / vida são determinantes importantes da dinâmica de silenciamento de genes resultante; Daí, a falta de tal informação é uma grande desconexão que impede uma previsão precisa de dose-resposta no RNAi 21 . Dirigir-se a este desafio agilizaria a formulação das relações estrutura-função apropriadas em nanocarriers e melhor informará os projetos de biomateriais 22 . Além disso, tais abordagens permitiriam o desenvolvimento de protocolos de dosagem de siARN mais eficazes. Na tentativa de entender a resposta dinamizante ao silenciamento, vários grupos investigaram modelos matemáticos de RNAi 23 , 24 , 25 . Esses quadros foramBem sucedido em fornecer informações sobre as mudanças mediadas pelo siRNA na expressão gênica e na identificação das etapas de limitação de taxa 26 . No entanto, esses modelos foram aplicados apenas aos sistemas comerciais de administração de genes ( por exemplo , Lipofectamina e polietilenimina (PEI)) que não são capazes de liberação de siRNA controlada e a complexidade dos modelos limitou severamente sua utilidade 27 . Essas deficiências destacam uma necessidade insatisfeita de novos materiais capazes de obter uma versão de siRNA sintonizável, combinada com modelos cinéticos preditivos simplificados e fáceis de usar.
Nosso método aborda todos esses desafios através da integração de uma plataforma de nanocarrier sensível à luz com métodos acoplados para quantificar siRNA e dinâmica de RNAi de modelo. Em particular, o lançamento de siRNA precisamente controlado da nossa plataforma 28 é monitorado por dois métodos complementares para quantificar com precisão encapsulado vs.SiRNA ligado. Os dados experimentais desses ensaios são inseridos em um modelo cinético simples para prever as eficiências de silenciamento de genes a priori 29 . Finalmente, a natureza on / off dos nanocarriers é facilmente explorada para gerar padrões de células na expressão gênica com controle espacial na escala de comprimento celular. Assim, este método fornece um método facilmente adaptável para controlar e prever o silenciamento de genes em uma variedade de aplicações que se beneficiarão da regulação espaciotemporal do comportamento celular.
Protocolo
1. Formulação de siRNA Nanocarriers
- Prepare soluções separadas de siRNA e mPEG- b- P (APNBMA) com volumes iguais diluídos em tampão 20 mM de ácido 4- (2-hidroxietil) piperazina-1-etanossulfónico (HEPES) a pH 6,0.
- Adicionar siRNA a uma concentração de 32 μg / mL a 20 mM de solução de HEPES.
NOTA: O siRNA foi uma sequência de controle negativa universal não-direcionada; No entanto, o siRNA pode ser projetado para direcionar qualquer gene de interesse. - Dissolver os polímeros mPEG- b- P (APNBMA) em uma solução de 20 mM de HEPES. Adicione uma quantidade apropriada de mPEG- b- P (APNBMA) para fazer uma solução de 220 μg / mL de modo que a relação N / P (N, grupos de amina em mPEG- b- P (APNBMA); P, grupos de fosfato em siRNA) É 4.
NOTA: O protocolo sintético para os polímeros mPEG- b- P (APNBMA) é relatado em outro lugar 30 .
- Adicionar siRNA a uma concentração de 32 μg / mL a 20 mM de solução de HEPES.
- Adicione a solução mPEG- b- P (APNBMA), gota a gota, a um volume igualE da solução de siRNA enquanto mistura suavemente em uma máquina vortex. Continue a vórtice durante 30 s após a adição de polímero. Incubar as amostras no escuro a temperatura ambiente durante 30 min.
2. Medindo a liberação de siRNA usando eletroforese em gel
- Formule os nanocarriers de acordo com as etapas 1.1-1.2 e dimensione os volumes para acomodar o número de amostras desejadas.
- Misture o nanocarrier com dodecilsulfato de sódio (SDS).
- Prepare uma solução de SDS de 1 mg / ml em água. Alíquota a quantidade de solução SDS necessária para produzir soluções com uma relação S / P (S, grupos sulfato em SDS, P, grupos fosfato em siRNA) de 15.
NOTA: Se a solução de polyplex contiver 1 μg de siRNA, devem ser adicionados 13 μg de SDS para atingir uma relação S / P de 15. - Adicione a solução SDS a cada solução de nanocarrier gota a gota enquanto se misturam suavemente em uma máquina vortex. Continue a vórtice por 30 s após a adição do SDS.
- Centrifugar as amostras a 3.000 xg por 5 s. Incubar as amostras no escuro a temperatura ambiente durante 30 min.
- Prepare uma solução de SDS de 1 mg / ml em água. Alíquota a quantidade de solução SDS necessária para produzir soluções com uma relação S / P (S, grupos sulfato em SDS, P, grupos fosfato em siRNA) de 15.
- Calibre e ajuste um laser UV com um filtro de 365 nm para uma intensidade de 200 W / m. Certifique-se de que a intensidade da luz seja medida a partir da localização em que o fundo da solução de amostra será assentado.
- Coloque a solução nanocarrier / SDS em uma câmara de vidro composta por lâminas de vidro separadas por uma junta de borracha.
- Prelavar as lâminas de vidro em uma solução de etanol / água 7: 3 (v / v) em água e secar completamente. Corte um buraco (~ 2 x 3 cm de retângulo) em uma junta de borracha. Coloque a junta de borracha em uma lâmina de vidro.
- Pipetar a solução nanocarrier / SDS no slide de vidro dentro do orifício da junta de borracha. Carregue um excesso de solução (20 μL excedente) no deslize de vidro evitando o contato com a junta de borracha.
NOTA: Alguns líquidos serão perdidos durante as etapas subsequentes. - Coloque o segundo copoDeslize na parte superior da junta deslizante. Para evitar a geração de bolhas de ar, coloque uma extremidade do slide em primeiro lugar e, em seguida, abaixe lentamente a outra extremidade.
- Anexe clipes de ligação a cada lado da câmara de vidro para mantê-lo fechado.
- Irradiate as amostras para o período de tempo desejado ( por exemplo , 0-60 min) usando o laser UV com um filtro de 365 nm com uma intensidade de 200 W / m. Remova os clipes da pasta e abra a câmara.
- Pipetar 25 μL das amostras de nanocarrier / SDS irradiadas em um tubo de microcentrífuga. Incubar as soluções no escuro à temperatura ambiente durante 30 min.
- Prepare um gel de agarose a 2% em peso pré-corado com 0,5 ug / mL de brometo de etidio em solução tampão Tris / Borate / EDTA (TBE) de acordo com os protocolos padrão 31 . Prepare um tampão de carga composto de 3: 7 (v / v) de glicerol / água.
- Adicione 5 μL da solução tampão de carga a 25 μL de cada amostra nanocarrier / SDS. Incube as amostras no escuro emTemperatura ambiente por 10 min.
- Carregue 30 μL de cada amostra nanocarrier / SDS no gel de agarose a 2%. Execute o gel no escuro a 100 V por 30 min. Imagine o gel usando um sistema de imagem em gel com filtros de brometo de etidio. Salve os arquivos de imagem em gel e avance para a etapa 2.10 para a quantificação da intensidade da banda. Certifique-se de que as intensidades da banda são suficientemente brilhantes para visualizar claramente, mas não muito brilhantes, que os sinais estão saturados.
- Quantifique as intensidades da banda usando o software ImageJ disponível publicamente 32 .
- Usando a ferramenta ROI do ImageJ, determine a intensidade de fluorescência das bandas de siRNA gratuitas em cada faixa, desenhando um retângulo ao redor de cada banda. Trace as curvas de intensidade de cada pista e integre a área sob as curvas, desenhando uma linha horizontal através das curvas de intensidade e clicando na varinha de rastreamento dentro das áreas incluídas.
- Calcule a intensidade relativa de cada pista dividindo a área sob o comandoRve de cada amostra pela área sob a curva do controle positivo de siRNA (sem mPEG- b -P (APNBMA) adicionado e sem SDS adicionado). Relate a porcentagem de siRNA liberada como a intensidade da banda normalizada de cada amostra.
3. Medindo a liberação de siRNA usando a espectroscopia de correlação de fluorescência (FCS)
- Obtenha siRNA marcado com um único fluoróforo na extremidade 5 'da cadeia sensora.
NOTA: O siRNA pode ser comprado pré-recozido com os rótulos conjugados no local desejado. O fluoróforo deve ser foto-estável e absorver / emitir entre 450 e 750 nm para evitar a extinção da luz UV e transferência de energia com mPEG- b- P (APNBMA). - Formule os nanocarriers de acordo com as etapas 1.1-1.2 usando o siRNA marcado. Dimensione os volumes para acomodar o número de amostras desejadas.
- Incubar as soluções em SDS e irradiar durante o período desejado de acordo com as etapas 2.2-2.6.
- PrepararCâmara de amostra FCS.
- Lave uma lâmina de vidro com uma solução de etanol / água 7: 3 (v / v) e secar completamente o vidro usando uma limpeza e uma corrente de ar.
- Remova os pedaços de papel de um espaçador adesivo de dupla face para expor o espaçador adesivo de dupla face. Anexe o espaçador a uma lamínula de vidro.
- Pipetar a solução de nanocarrier / SDS no deslizamento da cobertura no meio do orifício do espaçador adesivo.
- Coloque o slide de vidro em cima da lamínula. Pressione o escorregador de vidro para garantir que o escorregador de vidro e a lamínula estejam bem unidos e formem uma vedação.
- Use um microscópio confocal para medições de FCS 33 . Use um objetivo de apochromat de imersão em água 40X com uma abertura numérica de 1,2. Use o canal laser de excitação apropriado (488 nm) para coletar pelo menos 30 medidas de 10 s cada por amostra 34 . Certifique-se de que a intensidade do laser eo alinhamento do detector permanecemO mesmo para cada amostra.
- Além das amostras experimentais, controle de medidas, incluindo: uma amostra em branco sem siARN marcado; E uma amostra de siRNA grátis com siRNA marcado, mas sem mPEG- b- P (APNBMA).
- Analise os dados usando o software específico do FCS. Identifique a taxa de contagem de linha de base de cada amostra, determinando a taxa de contagem estável durante um período em que não há nanocarriers passando pelo volume confocal 29 .
- Subtrair a taxa de contagem da amostra em branco de cada valor da taxa de contagem da base da amostra. Normalize os valores resultantes para o controle de siRNA gratuito para calcular a porcentagem de siRNA livre 35 .
4. Modelagem cinética para prever o silenciamento de genes
- Crie scripts em uma linguagem de programação matemática usando o conjunto simples de equações diferenciais ordinárias para prever o silenciamento de genes 29.
NOTA: Os scripts podem ser disponibilizados mediante solicitação.- Escreva o conjunto de equações diferenciais ordinárias como:
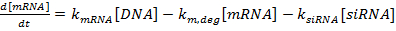 (1)
(1)  (2)
(2) 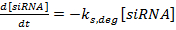 (3)
(3)
NOTA: Para as equações 1-3, os termos k mRNA , k siRNA e k prot são as constantes de taxa para a produção de mRNA, siRNA e proteína, respectivamente. Os termos k m, deg , k s, deg e k p, deg são as constantes de velocidade para a degradação de mRNA, siARN e proteína, respectivamente. As constantes da taxa de degradação são calculadas com base nas semi-vidas dos componentes e as constantes da taxa de produção são adequadas para garantir que os valores do mRNA e da estabilidade da proteína sejam atingidos na ausência oSIRRNA.- Determine as meias-vidas do mRNA e da proteína para o (s) gene (s) de interesse, seja experimentalmente como descrito na referência 36 ou da literatura (ver Discussão ). Além disso, determine o tempo de duplicação para a linha celular. Insira esses valores nas expressões da taxa de degradação apropriada.
- Sintonize as constantes de taxa de produção para que os níveis de expressão de genes permaneçam estáveis a um valor normalizado de 100 se nenhum siARN for introduzido. Especificamente, definir [siRNA] para zero e variar os valores das constantes de taxa de produção k mRNA , k siRNA e k prot até [mRNA] e [proteína] permanecerem dentro de 1% do valor normalizado inicial de 100% durante a duração de A simulação.
- Usando as quantidades relativas de siRNA liberado da eletroforese em gel previamente descrita e dos ensaios FCS como estimativas, ajuste a concentração relativa inicial de siRNA no script. Especificamente, variar [siRNA] para ser proporcional à quantidade relativa de siRNA liberado, com um valor de 100 correspondente à quantidade máxima 29 .
- Escreva o conjunto de equações diferenciais ordinárias como:
5. Cultura celular e entrega in vitro de siRNA
- Cultura NIH / 3T3 fibroblastos embrionários murinos de acordo com os protocolos do fornecedor.
- Crescer as células em meio de crescimento (Meio de Eagle Modificado de Dulbecco (DMEM) suplementado com 10% de soro de bovino fetal inactivado pelo calor e 1% de penicilina-estreptomicina). Manter as células a 37 ° C em um ambiente humidificado com 5% de CO 2 .
- Semeie as células em placas tratadas com cultura de tecido de 6 poços.
- Siga o procedimento de subcultivação recomendado do fornecedor. Contar as células usando um hemocitômetro. Diluir as células em meios de crescimento suplementados a uma concentração de 75.000 células / mL.
- Adicionar 2 mL de suspensão celular (75.000 célulasS / mL) para cada poço da placa de 6 poços. Deixe as células aderir e recuperar durante 24 h na incubadora.
- Prepare as células para transfecção lavando com solução salina tamponada com fosfato (PBS) e adicionando 1,5 ml de meio de transfecção sem soro e antibiótico (ver Tabela de Materiais ) a cada poço.
- Formule os nanocarriers siRNA de acordo com as etapas 1.1-1.2. Adicione 25 μL de solução de nanocarrier contendo 30 pmol de siRNA para cada poço. Agrida levemente a mídia para cima e para baixo para misturar. Coloque as células na incubadora por 3 h.
- Remova o meio de transfecção e lave cada poço com PBS. Adicione 1 mL de meio de crescimento suplementado e coloque as células na incubadora para recuperar durante 30 min.
- Para preparar as células para tratamento com um fotoestimulante, remova os meios de crescimento suplementados. Adicione 1 mL de meio de transfecção (sem vermelho fenol) a cada poço.
NOTA: Certifique-se de que a mídia de transfecção não contém vermelho de fenol. - Calibre e ajuste um laser UV com um filtro de 365 nm para uma intensidade de 200 W / m. Certifique-se de que a intensidade da luz seja medida a partir da localização em que o fundo da placa celular será assentado.
- Coloque as células em uma placa quente ajustada para 37 ° C. Remova a tampa da placa das células. Irradiate as células de acima da placa durante o tempo desejado (até 20 min) usando o laser UV com um filtro de 365 nm com uma intensidade de 200 W m -2 .
- Remova o meio de transfecção e adicione 2 mL de meio de crescimento suplementado. Coloque na incubadora até análise posterior ( por exemplo , 24 h para qPCR e 48 h para Western blot).
- Medir mudanças na expressão de genes usando uma variedade de técnicas, tais como Western Blot 37 e qPCR. 38 Para genes com sinais visíveis, como GFP, use microscopia de fluorescência 29 .
NOTA: Estas técnicas são sugeridas devido à sua facilidade de uso e precisãoNa quantificação da expressão gênica
- Medir mudanças na expressão de genes usando uma variedade de técnicas, tais como Western Blot 37 e qPCR. 38 Para genes com sinais visíveis, como GFP, use microscopia de fluorescência 29 .
6. Controlando o silenciamento de genes de forma Spatiotemporal
- Cultura, semente e células transfectadas de acordo com as etapas 5.1-5.7.
- Prepare uma fotomátema que bloqueie completamente a luz de 365 nm e minimiza as reflexões.
NOTA: Neste caso, foram utilizados fragmentos de papel de alumínio de 10 x 10 cm e papel de construção preto para bloquear a luz e reduzir as reflexões, respectivamente. A folha de alumínio e o papel de construção foram colados para formar uma única unidade.- Corte / soco / máquina a forma desejada na fotomáscara. Por exemplo, use uma lâmina afiada e um perfurador para formar um padrão de linha reta (~ 5 cm de comprimento) e um padrão circular (~ 7 mm de diâmetro) na fotomáscara, respectivamente.
- Cola a fotomask no fundo da placa de 6 poços com o padrão centrado sob o poço contendo as células com o lado anti-reflexo ( por exemplo , preto cPapel de impressão) de frente para a placa. Certifique-se de que a cola não está colocada perto da borda (dentro de ~ 3 mm) do padrão.
- Configure dois suportes de anel a aproximadamente 25 cm de distância e prenda uma plataforma a cada suporte de anel para que as plataformas sejam de igual altura. Suspenda a placa da célula entre os dois suportes, descansando a placa sobre as plataformas. Certifique-se de que a placa esteja nivelada.
- Irradiar as células de abaixo da amostra durante o tempo desejado (até 20 min) usando o laser UV com um filtro de 365 nm com uma intensidade de 200 W / m 2 .
- Remova o meio de transfecção e adicione 2 mL de meio de crescimento suplementado. Coloque na incubadora para recuperar por pelo menos 24 h. Imagem das células usando microscopia de fluorescência como descrito 29 .
Resultados
Após a formulação dos nanocarriers, os ensaios de libertação de siRNA foram conduzidos para informar as condições de irradiação a serem utilizadas nas transfecções in vitro . Várias doses de luz foram aplicadas para determinar a porcentagem de siRNA que foi liberado. O primeiro ensaio utilizou eletroforese em gel para separar as moléculas de siRNA gratuitas das moléculas de siRNA ainda complexas / associadas ao polímero. Nanocarriers que não foram tratados com lu...
Discussão
Existem alguns passos no método que são particularmente críticos. Ao formular os nanocarriers, a ordem de adição de componentes e velocidade de mistura são dois parâmetros importantes que influenciam a eficácia 39 . Este protocolo requer que o componente catiónico, mPEG- b- P (APNBMA), seja adicionado ao componente aniónico, siARN, de forma gota a gota enquanto se agita com vortex. Dependendo do volume total da formulação, este processo de mistura leva 3-6 s. Para testar se o...
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Os autores agradecem ao Instituto Nacional de Ciências Médicas Gerais dos Institutos Nacionais de Saúde (NIH) pelo apoio financeiro através de um Prêmio de Desenvolvimento Institucional (IDeA) sob o número de concessão P20GM103541, bem como o número de concessão P20GM10344615. As declarações aqui contidas não refletem as opiniões do NIH. Também reconhecemos o Delaware Biotechnology Institute (DBI) eo Delaware Economic Development Office (DEDO) para apoio financeiro através do prêmio Bioscience Center for Advanced Technology (Bioscience CAT) (12A00448).
Materiais
| Name | Company | Catalog Number | Comments |
| siRNA | Sigma-Aldrich | SIC001 | non-targeted, universal negative control |
| mPEG-b-P(APNBMA) | synthesized in our lab | N/A | photo-responsive polymer |
| HEPES | Fisher Scientific | BP310-100 | |
| sodium dodecyl sulfate | Sigma-Aldrich | 436143 | |
| rubber gasket | McMaster-Carr | 3788T21 | 0.5 mL thick |
| UV laser | Excelitas Technologies | Omnicure S2000 | collimating lens and 365 nm filter used |
| agarose | Fisher Scientific | BP160-100 | |
| ethidium bromide | Fisher Scientific | BP1302-10 | |
| siRNA labelled with Dy547 | GE Healthcare Dharmacon, Inc. | custom order | fluorophore conjugated to 5’ end of sense strand |
| microscope slide | Fisher Scientific | 12-550-A3 | pre-cleaned glass |
| Secure-Seal Spacer | Life Technologies | S24735 | double-sided adhesive |
| LSM 780 | Carl Zeiss | N/A | confocal microscope |
| ZEN 2010 | Carl Zeiss | N/A | FCS analysis software |
| MATLAB | MathWorks | N/A | programming language |
| NIH/3T3 cells | ATCC | ATCC CRL-1658 | |
| DMEM | Mediatech | 10-013-CV | growth media |
| fetal bovine serum | Mediatech | 35-011-CV | heat-inactivated |
| penicillin-streptomycin | Mediatech | 30-002-CI | |
| 6-well plates | Fisher Scientific | 08-772-1B | |
| Opti-MEM | Life Technologies | 11058021 | transfection media |
Referências
- Forbes, D. C., Peppas, N. A. Oral delivery of small RNA and DNA. J Control Release. 162 (2), 438-445 (2012).
- Davis, M. E., et al. Evidence of RNAi in humans from systemically administered siRNA via targeted nanoparticles. Nature. 464 (7291), 1067-1070 (2010).
- Bouchie, A. Companies in footrace to deliver RNAi. Nat Biotechnol. 30 (12), 1154-1157 (2012).
- Burke, P. A., Pun, S. H., Reineke, T. M. Advancing Polymeric Delivery Systems Amidst a Nucleic Acid Therapy Renaissance. ACS Macro Lett. 2 (10), 928-934 (2013).
- Gooding, M., Browne, L. P., Quinteiro, F. M., Selwood, D. L. siRNA Delivery: From Lipids to Cell-penetrating Peptides and Their Mimics. Chem Biol Drug Des. 80 (6), 787-809 (2012).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nat Chem Biol. 10 (3), 196-202 (2014).
- Wang, X., Chen, X., Yang, Y. Spatiotemporal control of gene expression by a light-switchable transgene system. Nat Methods. 9 (3), 266-269 (2012).
- Ziauddin, J., Sabatini, D. M. Microarrays of cells expressing defined cDNAs. Nature. 411 (6833), 107-110 (2001).
- Saltzman, W. M., Olbricht, W. L. Building drug delivery into tissue engineering. Nat Rev Drug Discov. 1 (3), 177-186 (2002).
- Mura, S., Nicolas, J., Couvreur, P. Stimuli-responsive nanocarriers for drug delivery. Nat Mater. 12 (11), 991-1003 (2013).
- Shim, M. S., Kwon, Y. J. Stimuli-responsive polymers and nanomaterials for gene delivery and imaging applications. Adv Drug Delivery Rev. 64 (11), 1046-1058 (2012).
- Kelley, E. G., Albert, J. N. L., Sullivan, M. O., Epps, T. H. Stimuli-responsive copolymer solution and surface assemblies for biomedical applications. Chem Soc Rev. 42 (17), 7057-7071 (2013).
- Weber, W., Fussenegger, M. Emerging biomedical applications of synthetic biology. Nat Rev Genet. 13 (1), 21-35 (2012).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Shimizu-Sato, S., Huq, E., Tepperman, J. M., Quail, P. H. A light-switchable gene promoter system. Nat Biotechnol. 20 (10), 1041-1044 (2002).
- Huschka, R., et al. Gene Silencing by Gold Nanoshell-Mediated Delivery and Laser-Triggered Release of Antisense Oligonucleotide and siRNA. ACS Nano. 6 (9), 7681-7691 (2012).
- Li, H. -. J., Wang, H. -. X., Sun, C. -. Y., Du, J. -. Z., Wang, J. Shell-detachable nanoparticles based on a light-responsive amphiphile for enhanced siRNA delivery. R Soc Chem Adv. 4 (4), 1961-1964 (2014).
- Braun, G. B., et al. Laser-Activated Gene Silencing via Gold Nanoshell-siRNA Conjugates. ACS Nano. 3 (7), 2007-2015 (2009).
- Gilleron, J., et al. Image-based analysis of lipid nanoparticle-mediated siRNA delivery, intracellular trafficking and endosomal escape. Nat Biotechnol. 31 (7), 638-646 (2013).
- Wittrup, A., et al. Visualizing lipid-formulated siRNA release from endosomes and target gene knockdown. Nat Biotechnol. 33 (8), 870-876 (2015).
- Roth, C. M. Quantitative measurements and rational materials design for intracellular delivery of oligonucleotides. Biotechnol Prog. 24 (1), 23-28 (2008).
- Mao, S., et al. Influence of polyethylene glycol chain length on the physicochemical and biological properties of poly(ethylene imine)-graft-poly(ethylene glycol) block copolymer/SiRNA polyplexes. Bioconjugate Chem. 17 (5), 1209-1218 (2006).
- Raab, R. M., Stephanopoulos, G. Dynamics of gene silencing by RNA interference. Biotechnol Bioeng. 88 (1), 121-132 (2004).
- Cuccato, G., et al. Modeling RNA interference in mammalian cells. BMC Systems Biology. 5, 1 (2011).
- Varga, C. M., Hong, K., Lauffenburger, D. A. Quantitative analysis of synthetic gene delivery vector design properties. Mol Ther. 4 (5), 438-446 (2001).
- Chen, H. H., et al. Quantitative comparison of intracellular unpacking kinetics of polyplexes by a model constructed from quantum Dot-FRET. Mol Ther. 16 (2), 324-332 (2008).
- Bartlett, D. W., Davis, M. E. Insights into the kinetics of siRNA-mediated gene silencing from live-cell and live-animal bioluminescent imaging. Nucleic Acids Res. 34 (1), 322-333 (2006).
- Foster, A. A., Greco, C. T., Green, M. D., Epps, T. H., Sullivan, M. O. Light-Mediated Activation of siRNA Release in Diblock Copolymer Assemblies for Controlled Gene Silencing. Adv Healthc Mater. 4 (5), 760-770 (2015).
- Greco, C. T., Epps, T. H., Sullivan, M. O. Mechanistic Design of Polymer Nanocarriers to Spatiotemporally Control Gene Silencing. ACS Biomater Sci Eng. 2 (9), 1582-1594 (2016).
- Green, M. D., et al. Catch and release: photocleavable cationic diblock copolymers as a potential platform for nucleic acid delivery. Polym Chem. 5 (19), 5535-5541 (2014).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Marquer, C., Leveque-Fort, S., Potier, M. C. Determination of Lipid Raft Partitioning of Fluorescently-tagged Probes in Living Cells by Fluorescence Correlation Spectroscopy (FCS). J Vis Exp. (62), (2012).
- Staaf, E., Bagawath-Singh, S., Johansson, S. Molecular Diffusion in Plasma Membranes of Primary Lymphocytes Measured by Fluorescence Correlation Spectroscopy. J Vis Exp. (120), e54756 (2017).
- Buyens, K., et al. Monitoring the disassembly of siRNA polyplexes in serum is crucial for predicting their biological efficacy. J Control Release. 141 (1), 38-41 (2010).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764-768 (2011).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. Proc Natl Acad. Sci U S A. 76 (9), 4350-4354 (1979).
- Wittwer, C. T., Herrmann, M. G., Moss, A. A., Rasmussen, R. P. Continuous Fluorescence Monitoring of Rapid Cycle DNA Amplification. Biotechniques. 54 (6), 314-320 (2013).
- Cho, S. K., Dang, C., Wang, X., Ragan, R., Kwon, Y. J. Mixing-sequence-dependent nucleic acid complexation and gene transfer efficiency by polyethylenimine. Biomater Sci. 3 (7), 1124-1133 (2015).
- Liu, Y. M., Reineke, T. M. Poly(glycoamidoamine)s for gene delivery: Stability of polyplexes and efficacy with cardiomyoblast cells. Bioconjugate Chem. 17 (1), 101-108 (2006).
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Greco, C. T., Muir, V. G., Epps, T. H., Sullivan, M. O. Efficient tuning of siRNA dose response by combining mixed polymer nanocarriers with simple kinetic modeling. Acta Biomater. 50, 407-416 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados