É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Modulação Reversível Mediada por Luz do Caminho Protein Quinaso Ativado por Mitogênio durante a Diferenciação Celular e Xenopus Desenvolvimento embrionário
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este protocolo descreve uma estratégia optogenética para modular a atividade da proteína quinase ativada por mitógenos (MAPK) durante a diferenciação celular e o desenvolvimento embrionário de Xenopus . Este método permite a ativação reversível da via de sinalização MAPK na cultura de células de mamíferos e em organismos vivos multicelulares, como embriões Xenopus , com alta resolução espacial e temporal.
Resumo
A atividade da cinase é crucial para uma infinidade de funções celulares, incluindo proliferação celular, diferenciação, migração e apoptose. Durante o desenvolvimento embrionário precoce, a atividade quinasa é altamente dinâmica e generalizada em todo o embrião. As abordagens farmacológicas e genéticas são comumente usadas para sondar atividades de quinase. Infelizmente, é desafiador alcançar uma resolução espacial e temporal superior usando essas estratégias. Além disso, não é viável controlar a atividade quinasa de forma reversível em células vivas e organismos multicelulares. Essa limitação continua a ser um gargalo para alcançar uma compreensão quantitativa da atividade quinasa durante o desenvolvimento e a diferenciação. Este trabalho apresenta uma estratégia optogenética que tira proveito de um sistema bicistrônico contendo proteínas fotoativáveis Arabidopsis thaliana cryptochrome 2 (CRY2) e o domínio N-terminal da híbrida-hélice-hélice básica interativa criptocromo (CIBN). ReversiA ativação da ativação da via de sinalização da proteína quinase ativada por mitogeno (MAPK) é conseguida através da translocação da proteína mediada por luz em células vivas. Esta abordagem pode ser aplicada a culturas de células de mamíferos e embriões de vertebrados vivos. Este sistema bicistrônico pode ser generalizado para controlar a atividade de outras quinases com mecanismos de ativação semelhantes e pode ser aplicado a outros sistemas modelo.
Introdução
Os fatores de crescimento estão envolvidos em um amplo espectro de funções celulares, incluindo proliferação, diferenciação, migração e apoptose, e desempenham papéis fundamentais em muitos eventos biológicos, incluindo desenvolvimento embrionário, envelhecimento e regulação do estado mental 1 , 2 , 3 , 4 , 5 . Muitos fatores de crescimento indicam através de cascatas de sinalização intracelular complexas. Esses eventos de sinalização são freqüentemente operados por fosforilação protéica reversível de forma regulada com precisão 6 , 7 . Assim, uma compreensão dos resultados de sinalização das proteínas quinases, que são responsáveis pela fosforilação de proteínas, é fundamentalmente importante.
Diferentes fatores de crescimento agem através de uma rede de sinalização intracelular bastante comum, embora estimulem distInct celular respostas 8 , 9 . Os mediadores intracelulares comuns das tirosina cinases receptoras incluem Ras, Raf, cinase regulada por sinal extracelular (ERK), proteína quinase activada por mitogeno (MAPK) / ERK quinase (MEK), phosphoinositide 3-kinase (PI3K), Akt e fosfolipase C gama (PLCγ) 10 , 11 . A evidência de acumulação sugere que a diversidade e a especificidade da sinalização dependem da regulação espacial e temporal da atividade de sinalização 12 . Por exemplo, em células de feocromocitoma de ratos (PC12), a estimulação do fator de crescimento epidérmico (EGF), que resulta na proliferação celular, ativa a via ERK 9 transitória. Por outro lado, a estimulação com o fator de crescimento nervoso (NGF), que leva à diferenciação celular, ativa a via ERK de forma sustentada 9 , 13 . Em culturaNos neurônios do hipocampo, a sinalização transitória por fator neurotrófico derivado do cérebro (BDNF) promove a proliferação primária dos neurites, enquanto a sinalização sustentada leva ao aumento da ramificação dos neurites 14 . Durante o desenvolvimento embrionário precoce, a atividade ERK fosforilada é temporariamente dinâmica e está disseminada em todo o embrião 6 . Uma tela genética recente durante a embriogênese precoce de Xenopus mostrou que as cascatas de sinalização de ERK e Akt, duas vias do factor de crescimento primário a jusante, exibiam os perfis de ativação específicos do estágio 7 . Assim, uma compreensão dos resultados da sinalização da quinase requer ferramentas que possam investigar as características espaciais e temporais da atividade quinasa com resolução suficiente.
As abordagens experimentais convencionais para investigar a natureza dinâmica da transdução de sinal durante o desenvolvimento não possuem a resolução espacial e temporal desejável. Por exemplo, as abordagens farmacológicas utilizamMoléculas histológicas ou biológicas para estimular ou suprimir a transdução de sinal em células e tecidos. A natureza difusiva dessas moléculas pequenas torna desafiador restringir sua ação a uma região específica de interesse 15 . As abordagens genéticas ( por exemplo, transgênese, sistema Cre-Lox ou mutagênese) geralmente levam à ativação ou à repressão irreversível da expressão do gene alvo ou da atividade protéica 16 , 17 , 18 . O sistema Tet-On / Tet-Off 19 oferece um controle temporal melhorado da transcrição genética, mas não possui controle espacial rigoroso porque depende da difusão da tetraciclina. Desenvolvimentos recentes na dimerização de proteína induzida quimicamente 20 ou foto-uncaging 21 , 22 , 23 , 24 aumentaram bastanteO controle temporal das redes de sinalização. O controle espacial, no entanto, continua desafiando devido à natureza difusiva dos produtos químicos enjaulados.
As recentes abordagens optogenéticas emergentes, que aproveitam o poder da luz para controlar as interações proteína-proteína, permitem a modulação de caminhos de sinalização com alta precisão espaciotemporal e reversibilidade. Pouco depois do seu sucesso inicial no controle do disparo neuronal 25 , 26 , 27 , a optogenética foi estendida para controlar outros processos celulares, como transcrição de genes, tradução, migração celular, diferenciação e apoptose 28 , 29 , 30 , 31 , 32 , 33 , 34 . Uma estratégia usando a pO pino proteico de hotoactividade da proteína Arabidopsis thaliana cryptochrome 2 (CRY2) e o domínio N-terminal da hélice-laço-hélice básica (CIBN) que interage criptocromo foi recentemente desenvolvido para controlar a atividade da quinase Raf1 em células de mamíferos e embriões Xenopus 35 . CRY2 liga-se a CIBN após a estimulação de luz azul e o complexo de proteína CRY2 / CIBN dissocia espontaneamente no escuro 34 . A luz azul excita o cofactor CRY2, o dinucleótido de flavina adenina (FAD), o que leva a uma mudança conformacional em CRY2 e sua subsequente ligação à CIBN. Os mutantes de CRY2 deficientes em flavina (D387A) podem ser produzidos através de mutações no bolso de ligação ao FAD: o mutante CRY2 W374A liga-se a CIBN independentemente da luz, enquanto que o mutante CRY2 D387A não se liga à CIBN sob o azul Estimulação luminosa 36 , 37 . O sistema optogenético descrito iN este protocolo usa CRY2 e CIBN de tipo selvagem para induzir ativação Raf1 mediada por translocação de proteínas em células vivas. Sabe-se que o recrutamento de membranas de Raf1 aumenta sua atividade 38 . Neste sistema, um módulo CIBN em tandem é ancorado na membrana plasmática e CRY2-mCherry é fundido com o N-terminal de Raf1 35 . Na ausência de luz azul, CRY2-mCherry-Raf1 permanece no citoplasma e Raf1 está inativo. A estimulação de luz azul induz a ligação CRY2-CIBN e recruta Raf1 para a membrana plasmática, onde Raf1 é ativado. A ativação do Raf estimula uma cascata de sinalização Raf / MEK / ERK. As proteínas de fusão CRY2 e CIBN são codificadas em um sistema genético bicistrônico. Essa estratégia pode ser generalizada para controlar outras quinases, como Akt, cujo estado de ativação também pode ser ativado pela translocação de proteínas nas células 39 . Este trabalho apresenta protocolos detalhados para implementar esta estratégia optogenética em cultu de células de mamíferosRes e organismos multicelulares.
Access restricted. Please log in or start a trial to view this content.
Protocolo
A pesquisa com animais foi conduzida de acordo com as diretrizes estabelecidas pelo Comitê de Uso e Cuidados de Animais Institucionais de Illinois (IACUC) e pelo Departamento de Recursos Animais da Universidade de Illinois (DAR).
1. Indução optogenética da localização de proteínas na cultura celular BHK21 de mamífero
NOTA: As etapas 1.1-1.3 fornecem um método para montar uma câmara de cultura celular para imagens com objetivos de alta ampliação ( por exemplo, 63X ou 100X), que tipicamente têm curtas distâncias de trabalho. Esses objetivos exigem uma lamínula de vidro fina ( por exemplo, # 1,5, espessura de 170 um) como o substrato de imagem. Alternativamente, pode usar-se um prato de cultura de células de fundo de vidro. Nesse caso, as etapas 1.1-1.3 podem ser ignoradas.
- Limpando lâminas de vidro
- Coloque 30 lamelas de vidro em um porta-lamínulas.
- Em um copo de plástico de 600 mL, adicione 20 g de detergente e 400 mL de água quente da torneira. PlacE o copo em um agitador magnético e mexa até dissolver todo o detergente. Remova a espuma com papel de seda.
- Use o fundo de uma placa de Petri de 100 mm x 15 mm como um espaçador da barra de agitação e como uma superfície para o porta-lamínulas. Para manter o fluxo adequado durante o processo de limpeza, faça 3-4 furos de drenagem na placa de Petri.
- Para fazer os buracos de drenagem, aquecer um ferro de solda e queimar através do plástico em um capuz ventilado.
CUIDADO: Não toque no ferro de solda aquecido com as mãos nuas. - Coloque o suporte da lamínula na placa de Petri no copo. Mexa durante 2 h até a noite à temperatura ambiente.
- Lave três vezes com 500 mL de água DI num copo de 1000 mL. A solução de detergente pode ser reutilizada.
- Pegue lamínulas individuais usando fórceps. Mergulhe os lamínulos em etanol à prova de 190 (95%) durante 1 min.
- Seque os lamínulas dentro de um capô estéril. Armazene os lamínulas em um recipiente de plástico estéril até o uso.
- Fabricação de lâminas revestidas de poli-L-lisina (PLL)
- Faça um tampão de borato 0,1 M dissolvendo 1,24 g de ácido bórico e 1,9 g de tetraborato dissódico em 400 mL de água. Ajuste o pH para 8,5 com NaOH 10 M.
- Dissolver PLL no tampão borato até uma concentração final de 1 mg / mL. Alíquota da solução PLL em tubos de centrífuga estéril de 50 ml.
- Adicione 50 mL de solução de revestimento PLL a um prato de cultura de tecido de 10 cm. Coloque o prato em uma incubadora de 37 ° C para aquecer a solução PLL.
- Abra o recipiente de lamínula (etapa 1.1.8) em um capô estéril.
- Mergulhe os cobertores limpos, um a um, na solução pré-aquecida de PLL no prato de cultura de tecidos. Evite a formação de bolhas para imergir todas as superfícies.
- Coloque o prato com a lamínula em uma incubadora de CO 2 de 37 ° durante a noite. Retire cada lamínula e coloque-o em um suporte de lamínula estéril. Enxaguar três vezes com água ultrapura autoclavada (18,2 MΩ e #183; cm de resistividade) num copo de 1000 mL em um capuz estéril.
- Seque os lamínulas no suporte da lamínula na capa estéril. Armazene os lamínulas em um recipiente de plástico estéril até o uso.
- Montagem de câmaras de cultura celular de polidimetilsiloxano (PDMS)
- Em um recipiente de plástico, pesa 40 g de base de PDMS e adicione 4 g de agente de cura para atingir uma proporção de 10: 1 p / p de PDMS e agente de cura. Misture o PDMS / agente de cura agitando com uma ponta de pipeta durante 2 min. Alternativamente, use um misturador centrífugo.
- Use um copo com um diâmetro de 4 polegadas como modelo para fazer um suporte com um pedaço de papel alumínio. Coloque o suporte de alumínio em uma placa de Petri de 15 cm. Coloque uma bolacha de silicone de 4 polegadas dentro deste suporte de alumínio.
- Despeje a solução de PDMS misturada no suporte. Use uma vara de vidro fino para tocar a borda da bolacha suavemente. Remova as bolhas de ar presas sob a bolacha.
- Insira a placa de Petri de 15 cm em um chambe de vácuoR para desgastar a solução PDMS. Continue a desgaseificar por cerca de 20 min, até que não sejam geradas bolhas. Enquanto isso, pré-aqueça um forno de convecção a 65 ° C.
- Coloque cuidadosamente a placa de Petri de 15 cm no forno. Incubar durante 2 h.
- Retire a placa do forno. Use uma haste de vidro para tocar suavemente a borda do PDMS e assegurar uma reticulação completa. Caso contrário, incuba mais, até o PDMS ser sólido e não pegajoso.
- Retire a folha de alumínio da placa e descasque-a da bolacha de silício. Partindo da borda, remova com cuidado o PDMS curado da bolacha de silício.
CUIDADO: Evite aplicar muita força, pois pode quebrar a bolacha. - Corte o PDMS em peças retangulares de 20 mm x 30 mm para caber a lamela de 24 mm x 40 mm com uma lâmina de barbear.
- Use uma lâmina em forma de cinzel para cortar uma abertura retangular de 10 mm x 20 mm do centro de cada peça PDMS.
- Limpe as câmaras PDMS usando o protocolo de detergente descrito no passo 1.1.
- Secar as câmaras PDMS em uma capa limpa. Coloque as câmaras secas em um recipiente autoclavável coberto com papel alumínio.
- Autoclave o recipiente usando gravidade (121 ° C, 15 psi) por 30 min e armazene o recipiente à temperatura ambiente.
- Cultivo e transfecção de células BHK21
- Faça 500 mL de meio para cultura de células BHK21 misturando 445 mL de DMEM com 50 mL de FBS e 5 mL de Penn-Strep-Glutamina 100 × (10 000 U / mL de penicilina, 10 mg / mL de estreptomicina e 29,2 mg / mL de L -glutamina). Certifique-se de que a concentração final do FBS seja de 10%.
- Alíquota 10 mL de meio não dependente de HEPES CO 2 para imagens de células vivas.
NOTA: O meio independente de CO 2 suporta o crescimento celular sem uma incubadora de CO 2 e é ideal para células de imagem em condições atmosféricas. Consulte a Lista de Materiais para obter informações detalhadas. Além da linha celular BHK21, outros tipos de células de mamíferos também devemResultados semelhantes. - Monte um dispositivo de cultura de células, colocando uma câmara PDMS esterilizada em autoclave (etapa 1.3) em uma lamínula estéril revestida com PLL (passo 1.2) em uma capa estéril.
- Coloque cada dispositivo em uma placa de Petri estéril de 60 mm.
- Use 0,5 mL de tripsina a 0,25% para separar as células de um poço de uma placa de cultura de tecido de 12 poços. Contagem de densidade celular com um hemocitómetro. Placa 20 000 células BHK21 na câmara (cerca de 10 000 células / cm 2 ) com 200 μL de meio de cultura de células.
- Prepare o plasmídeo de DNA com um kit de preparação de plasmídeo (veja a Lista de Materiais).
NOTA: O design e a funcionalidade do CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX são mostrados na Figura 1A - 1B . - Vinte e quatro (24) h após o revestimento celular, transfiram as células com 50-100 ng de plasmídeo CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX, de acordo com o protocolo do fabricante.
- Três (3) horas após a transfecção, mude o mEdium para 200 μL de meio de cultura fresco (passo 1.4.1) e permitir que as células se recuperem durante a noite incubando a cultura em uma incubadora de CO 2 de 37 ° C e 5%.
- Fluorescência de imagens de células vivas
- Ligue um computador, fonte de luz, microscópio e câmera. Use um microscópio de fluorescência confocal (equipado com um objetivo de imersão em óleo de 100X) para o resto do protocolo.
NOTA: Um microscópio de epifluorescência invertido também pode ser usado. - Substitua o meio de cultura celular por 200 μL de meio independente de CO 2 .
- Configure o protocolo de aquisição de dados antes de colocar as células no microscópio. Use um canal FITC de excitação de 488 nm para estimulação optogenética. Use um canal TRITC de excitação de 561 nm para localizar células transfectadas e rastrear a localização celular da proteína marcada com mCherry.
- Defina o ganho dos canais FITC e TRITC como 120 e 200, respectivamente. Use um tempo de pixel-moradia de 2.21; s e tamanho de 512 x 512 pixels.
- Medir a potência da luz de 488 nm, colocando um medidor de energia perto da janela objetiva. Uma potência total de 2 μW (cerca de 10 000 W / cm 2 no foco) é suficiente para induzir associação CIBN-CRY2PHR.
- Configure uma aquisição de timestamp com um intervalo de 5 s e um tempo de aquisição total de 2 min.
- Aplique material apropriado de correspondência de índice (óleo de imersão) na janela do objetivo. Use o modo de contraste de fase para focar as células na superfície da lamínula.
- Localize uma célula transfectada sob luz de 561 nm movendo o estágio do microscópio.
CUIDADO: Evite usar luz azul durante esta etapa, pois ativará a associação entre proteínas fotoativáveis. - Uma vez que uma célula transfectada está localizada, inicie a aquisição de dados.
NOTA: as células transfectadas com sucesso devem mostrar fluorescência nos canais FITC (de 2CIBN-GFP-CaaX) e TRITC (de CRY2-mCherry-Raf1) ( Figura 1C-D ). - Grave uma série de imagens marcadas no horário nos canais FITC e TRITC ( Figura 1D ).
- Para sondar a dissociação espontânea do complexo de proteínas CRY2-CIBN, registre outro timestamp com o canal Txred sozinho durante 20 min, com um intervalo de 30 s.
- Salve a imagem marcada no horário para análise de dados.
- Ligue um computador, fonte de luz, microscópio e câmera. Use um microscópio de fluorescência confocal (equipado com um objetivo de imersão em óleo de 100X) para o resto do protocolo.
- Análise de imagem
- Abra as imagens com um software de análise de imagem que pode extrair a intensidade de uma imagem 40 .
- Selecione dois instantâneos representativos: um antes da exposição à luz azul e a outra exposição pós-luz azul.
- Desenhe uma linha através da célula que abaixa o fundo, a membrana plasmática e o citoplasma. Projeto a intensidade ao longo desta linha. Salve os valores e traçá-los para comparar a diferença antes e depois da exposição à luz ( Figura 1D ).
- Para analisar a cinética da associação de proteínas, selecioneT uma região representativa de interesse (ROI) na membrana plasmática, um ROI no citoplasma e um ROI em segundo plano.
- Adquirir as intensidades médias da membrana plasmática (I PM ), do citoplasma (I CYT ) e do fundo (I BKD ) para a pilha de imagens.
- Calcule a proporção de intensidade de membrana / citosolic para cada imagem usando a seguinte equação:
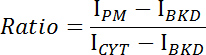
- Traçar a relação versus tempo e determinar a cinética de ligação ou dissociação de CRY2-CIBN ( Figura 1E- F ).
2. Construção de uma matriz de LED para estimulação de luz a longo prazo em uma incubadora de CO 2
NOTA: O esquema geral da configuração experimental é mostrado na Figura 2A .
- Faça a matriz LED inserindo 12 LEDs azuis em dois panosDs e conectando resistores limitadores de corrente.
NOTA: com uma fonte de alimentação de 30 V, quatro LEDs podem ser conectados em série e seu brilho pode ser controlado por um resistor de limitação de corrente. - Coloque as placas de pão em uma caixa de alumínio.
NOTA: A altura da caixa de alumínio deve ser de 2 pol. Esta altura é ideal para iluminar uma placa de cultura de tecidos de 12 poços, porque o tamanho do ponto de luz divergente é o mesmo que um único poço. - Use dois fios metálicos para conectar a fonte de alimentação. Certifique-se de que o comprimento do fio é suficiente quando a caixa de luz é colocada dentro de uma incubadora de CO 2 .
- Use um difusor de luz transparente como a capa da caixa de luz ( Figura 2C ).
- Calibre a saída de energia de cada LED em uma faixa de entradas de tensão. Use uma potência de 0,2 mW / cm 2 para o ensaio de diferenciação de células PC12 de 24 h. Use uma potência de 5 mW / cm 2 para embriões ou explantes Xenopus ao vivoEnsaios.
3. Indução optogenética da diferenciação celular PC12
- Cultivo celular e transfecção
- Faça 500 mL de meio para uma cultura de células de feocromocitoma de ratos (PC12) misturando 407,5 mL de F12K com 75 mL de soro de cavalo + 12,5 mL de FBS + 5 mL de 100 × Penn-Strep-Glutamina (concentrações finais de soro de cavalo e FBS : 15% e 2,5%, respectivamente). Faça um meio de baixo teor de soro misturando 1 volume de meio completo em 99 volumes de meio F12K.
- Placa de células PC12 em uma placa de 12 poços com uma densidade de 300.000 células / poço ou 75.000 células / cm2.
- Transfecte as células com CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX 24 h após o revestimento celular (semelhante ao passo 1.4.7). Use 1,2 μg de DNA para cada poço. Permitir que as células se recuperem durante a noite em meio de cultura numa incubadora a 37 ° C suplementada com 5% de CO 2 . Verifique a eficiência de transfecção 16 h após a transfecção.
NOTA: Uma transfecção de 30 a 50%A ciência deve ser alcançada para garantir uma contagem de células suficiente. - Mude o meio médio para baixo do soro 24 h após a transfecção. Use 1 mL de meio por poço de uma placa de 12 poços.
- Diferenciação de células PC12 induzida por luz
- Coloque a matriz LED em uma incubadora de CO 2 . Conecte-o à fonte de alimentação usando um par de fios. Defina a potência do LED para 0.2 mW / cm 2 . Coloque a placa de 12 poços contendo células PC12 transfectadas (passo 3.1.3) na janela da matriz LED. Aplique iluminação de luz contínua durante 24 h em uma incubadora a 37 ° C suplementada com 5% de CO 2 .
- Imagens de células vivas de fluorescência
- Siga as etapas 1.5.1-1.5.4 para microscópio e preparação da amostra.
- Configure a aquisição de dados de instantâneo único. Use 200 ms para o canal GFP e Txred.
- Capture imagens das células transfectadas nos canais GFP e Txred. Gravar cerca de 200 celÉ por cada condição. Salve os arquivos para análise de dados.
- Análise de imagem
- Contar a porcentagem de células diferenciadas sobre o número total de células transfectadas.
- Use qualquer módulo de contagem de células no software de análise de imagem para contar as células.
- Contar manualmente as células diferenciadas.
NOTA: As células diferenciadas são definidas como aquelas em que pelo menos um neurite é discernivelmente mais longo do que o corpo celular ( Figura 3A- 3B ). - Repita o passo 3.4.3 com células indiferenciadas.
- Calcule o índice de diferenciação usando a seguinte equação:
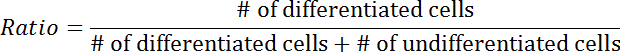
4. Controle optogenético da atividade do quinases em embriões Xenopus
- Preparação de buffers
- Prepare 1 L de 1x Marced Modified Ringer (MMR): 100 mMNaCl, 2 mM de KCl, 1 mM de MgCl2, 2 mM de CaCl2 e 5 mM de HEPES; PH 7,5.
- Preparação de mRNA
- Digerir o DNA do plasmídeo CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX (2 μg) com 1 μL de ApaI (unidade 50) a 37 ° C durante 2 h.
- DNA linearizado com precipitado em 60 μL de etanol a 100%. Girar a 12 000 × g durante 5 minutos à temperatura ambiente para sedimentar o DNA. Retire cuidadosamente o sobrenadante e lave o sedimento novamente com 60 μL de etanol a 70%. Gire a 12,000 × g por mais 5 minutos à temperatura ambiente. Retire cuidadosamente o sobrenadante e ressuspenda o sedimento de DNA em 6 μL de H2O livre de RNase.
- Realize a transcrição in vitro incubando 1 μg de DNA linearizado com 2 μL de ARN polimerase SP6 fornecida, uma mistura de ribonucleótidos (ATP 10 mM, CTP e UTP, GTP 2 mM e análogo de tampão 8 mM) e 20 μL de nuclease- Água livre à temperatura ambiente durante 1 h.
- Remova o DModelo NA por digestão com DNase I a 37 ° C durante pelo menos 15 minutos e purificar o ARN sintetizado utilizando uma coluna de rotação de sílica-membrana.
- Pare a reação e precipite o RNA adicionando 30 μL de água livre de nuclease e 30 μL de solução de precipitação de LiCl. Misture bem e relaxe durante 30 min a -20 ° C.
- Centrifugar a 4 ° C durante 15 minutos a 12 000 × g para sedimentar o ARN.
- Remova cuidadosamente o sobrenadante. Lavar o ARN uma vez com 1 mL de etanol a 70% e ressuspender em 40 μL de água livre de nuclease.
- Carregue 40 μL de RNA numa coluna de rotação. Centrifugar a 4 ° C durante 1 min a 12 000 × g para remover os nucleotídeos e tampas não incorporados.
- Colher Xenopus embriões
- Siga o protocolo descrito por Sive et al. 41 para obter os embriões Xenopus .
- Microinjeção de mRNA
- FabricarAs agulhas para microinjecção puxando os capilares de vidro com um extractor capilar. Defina o programa de puxar para: calor = 355, força de tração = 80, velocidade = 50 e tempo = 100.
- Remova o revestimento de geléia dos embriões tratando-os com 3% de cisteína (diluída em 0.2x MMR).
- Transfira os embriões para 3% de polissucrose e solução de MMR 0,5x para microinjecção. Injecte 500 pg a 1 ng de ARN CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX em cada embrião.
NOTA: A injeção de uma dose superior a 1 ng resulta em ativação independente da luz azul da sinalização MAPK.
- Estimulação optogenética da atividade quinasa
- Cultura de embriões microinjetados em 3% de polissucrose, solução de MMR 0,5x à temperatura ambiente até atingir o estádio da gastrula média (estágio 12). Em seguida, cultura os embriões na solução de MMR 0,2x.
- Transfira os embriões ou explantes para uma placa de 12 poços. Coloque a placa de 12 poços na matriz de LED construída na casa (etapa 2.5) para azul-lighTratamento de t (475 nm).
- Coloque um espelho na parte superior da placa de 12 poços para garantir a iluminação azul-clara dos embriões ou explantes.
- Ajuste a potência da luz azul para 5 mW / cm 2 .
NOTA: O tratamento com luz azul pode ser realizado a qualquer momento desejado, em solução de polissonfa a 3%, solução de MMX 0,5x ou solução de MMR 0,2x. - Colher os embriões em qualquer momento desejado. Use-os para análise histológica, Western-blot ou de expressão de genes.
Access restricted. Please log in or start a trial to view this content.
Resultados
Expressão raatiométrica de pares de proteínas fotoativáveis: a Figura 1A mostra o projeto de uma construção optogenética bicistrônica, CRY2-mCherry-Raf1-P2A-CIBN-CIBN-GFP-CaaX (referido como CRY2-2A-2CIBN), com base no teschovirum porcino- 1 péptido 2A (P2A), que mostra a maior eficiência de picos de ribossoma entre as linhas celulares de mamíferos 42 . No trabalho anterior, determinou-se que a razão ideal para CIBN-GFP-Ca...
Access restricted. Please log in or start a trial to view this content.
Discussão
Ao construir a caixa de luz, a potência dos LEDs individuais deve ser medida. Com base na experiência anterior, a potência pode variar entre LEDs individuais devido a variação de fabricação. Selecione um conjunto de LEDs que tenham uma saída de energia dentro de 10% uns dos outros. O número de LEDs, o resistor de limitação de corrente e a entrada de energia podem ser modificados para diferentes tipos de recipientes de cultura celular ( por exemplo, uma placa de 6 poços ou de 24 poços). Uma ilumina?...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Este trabalho foi apoiado pela Universidade de Illinois em Urbana-Champaign (UIUC) e os Institutos Nacionais de Saúde (NIGMS R01GM111816).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Glass coverslip | VWR | 48393 230 | Substrate for live cell imaging |
| Coverslip holder | Newcomer Supply | 6817B | Holder for coverslips |
| Detergent | ThermoFisher | 16 000 104 | For cleaning coverslips |
| Boric acid | Sigma-Aldrich | B6768-500G | For making PLL buffer |
| Disodium tetraborate | Sigma-Aldrich | 71996-250G | For making PLL buffer |
| Plastic beaker | Nalgene | 1201-1000 | For cleaning coverslips |
| Sodium hydroxide | Sigma-Aldrich | 221465-2.5KG | For adjust pH |
| Poly-L-lysine hydrobromide | Sigma-Aldrich | P1274-500MG | For coating coverslip |
| Diethylpyrocarbonate (DEPC)-Treated Water | ThermoFisher Scientific | 750024 | For DNA preparation |
| Cover Glass Forceps | Ted Pella | 5645 | Cover glass handling |
| Tissue cutlure dish | Thermofisher | 12565321 | Cell culture dish |
| Sterile centrifuge tubes | ThermoFisher | 12-565-271 | Buffer storage |
| Transfection Reagent | ThermoFisher | R0534 | Transfection |
| CO2-independent medium | ThermoFisher | 18045088 | For live cell imaging |
| Polydimethylsiloxane (PDMS) | Ellsworth Adhesives | 184 SIL ELAST KIT 0.5KG | Form make cell chamber |
| Plasmid Maxiprep kit | Qiagen | 12965 | Plasmid preparation |
| DMEM medium | ThermoFisher | 11965-084 | Cell culturing medium component |
| F12K medium | ThermoFisher | 21127022 | Cell culturing medium component |
| Horse serum | ThermoFisher | 16050122 | Cell culturing medium component |
| Fetal Bovine Serum | Signa-Aldrich | 12303C-500 mL | Cell culturing medium component |
| Penicillin-Streptomycin-Glutamine | ThermoFisher | 10378016 | Cell culturing medium component |
| Trypsin (0.25%), phenol red | ThermoFisher Scientific | 15050065 | For mammalian cell dissociation |
| Agarose | Fisher Scientific | BP1356-100 | For DNA preparation |
| Ficoll PM400 | GE Heathcare Life Sciences | 17-5442-02 | For embryo buffer |
| L-Cysteine hydrochloride monohydrate | Sigma-Aldrich | 1.02839.0025 | Oocyte preparation |
| ApaI | ThermoFisher | FD1414 | For linearization of plasmids |
| Dnase I | ThermoFisher | AM2222 | For removing DNA template in the in vitro transcription assay |
| Index-match materials (immersion oil) | Thorlabs | MOIL-20LN | For matching the index between sample substrate and objective |
| Blue LED | Adafruit | 301 | Light source for optogenetic stimulation |
| Resistor kit | Amazon | EPC-103 | current-limiting resistor |
| Aluminum boxes | BUD Industries | AC-401 | light box |
| BreadBoard | Jekewin | 837654333686 | For making LED array |
| Hook up Wire | Electronix Express | 27WK22SLD25 | For making LED array |
| Relay Module | Jbtek | SRD-05VDC-SL-C | For intermittent light control |
| DC Power Supply | TMS | DCPowerSupply-LW-(PS-305D) | Power supply for LED |
| Silicon Power Head | Thorlabs | S121C | For light intensity measurement |
| Power meter | Thorlabs | PM100D | For light intensity measurement |
| Microscope | Leica Biosystems | DMI8 | For live cell imaging |
| BioSafety Cabinet | ThermoFisher | 1300 Series A2 | For mammalian cell handling |
| CO2 incubator | ThermoFisher | Isotemp | For mammalian cell culturing |
| Stereo microscope | Leica | M60 | For embryo micro-manipulation |
| Microinjector | Narishige | IM300 | For embryo microinjection |
| Micropipette puller | Sutter Instruments | P87 | Needle puller |
| in vitro transcription kit | ThermoFisher | AM1340 | For in vitro transcription. The kit includes nuclease-free water, SP6 RNA Polymerase, ribonucleotide mixture, cap analog, lithium choride precipitation solution, and spin column |
| RNA purfication kit | Qiagen | 74104 | Silica-membrane spin column for purification of synthesized RNA |
| Convection oven | MTI corporation | EQ-DHG-9015 | PDMS curing |
| Centrifugal mixer and teflon container | THINKY | AR310 | For mixing PDMS |
| Silicon wafer | UniversityWafer | 452 | Base for making PDMS devices |
| Blade | Techni Edge | 01-801 | For cutting PDMS |
| Capillary glass | Sutter Instruments | BF100-58-10 | For fabrication of injecting needles. |
Referências
- Schlessinger, J., Ullrich, A. Growth factor signaling by receptor tyrosine kinases. Neuron. 9 (3), 383-391 (1992).
- Thisse, B., Thisse, C. Functions and regulations of fibroblast growth factor signaling during embryonic development. Dev Biol. 287 (2), 390-402 (2005).
- Perrimon, N., Pitsouli, C., Shilo, B. Z. Signaling mechanisms controlling cell fate and embryonic patterning. Cold Spring Harb Perspect Biol. 4 (8), a005975(2012).
- Salles, F. H., et al. Mental disorders, functional impairment, and nerve growth factor. Psychol Res Behav Manag. 10, 9-15 (2017).
- Basson, M. A. Signaling in cell differentiation and morphogenesis. Cold Spring Harb Perspect Biol. 4 (6), (2012).
- Schohl, A., Fagotto, F. beta-catenin, MAPK and Smad signaling during early Xenopus development. Development. 129 (1), 37-52 (2002).
- Zhang, S. W., Li, J. J., Lea, R., Amaya, E., Dorey, K. A Functional Genome-Wide In Vivo Screen Identifies New Regulators of Signalling Pathways during Early Xenopus Embryogenesis. PloS one. 8 (11), e79469(2013).
- Sweeney, C., et al. Growth factor-specific signaling pathway stimulation and gene expression mediated by ErbB receptors. J Biol Chem. 276 (25), 22685-22698 (2001).
- Marshall, C. J. Specificity of receptor tyrosine kinase signaling: transient versus sustained extracellular signal-regulated kinase activation. Cell. 80 (2), 179-185 (1995).
- Vandergeer, P., Hunter, T., Lindberg, R. A. Receptor Protein-Tyrosine Kinases and Their Signal-Transduction Pathways. Annu Rev Cell Biol. 10, 251-337 (1994).
- Lemmon, M. A., Schlessinger, J. Cell signaling by receptor tyrosine kinases. Cell. 141 (7), 1117-1134 (2010).
- Hunter, T. Signaling--2000 and beyond. Cell. 100 (1), 113-127 (2000).
- Qiu, M. S., Green, S. H. PC12 cell neuronal differentiation is associated with prolonged p21ras activity and consequent prolonged ERK activity. Neuron. 9 (4), 705-717 (1992).
- Ji, Y., et al. Acute and gradual increases in BDNF concentration elicit distinct signaling and functions in neurons. Nat Neurosci. 13 (3), 302-309 (2010).
- Luby-Phelps, K. Cytoarchitecture and physical properties of cytoplasm: volume, viscosity, diffusion, intracellular surface area. Int Rev Cytol. 192, 189-221 (2000).
- Sauer, B. Inducible gene targeting in mice using the Cre/lox system. Methods-a Companion to Methods in Enzymology. 14 (4), 381-392 (1998).
- Ling, M. M., Robinson, B. H. Approaches to DNA mutagenesis: an overview. Anal Biochem. 254 (2), 157-178 (1997).
- Gama Sosa, M. A., De Gasperi, R., Elder, G. A. Animal transgenesis: an overview. Brain Struct Funct. 214 (2-3), 91-109 (2010).
- Wanka, F., et al. Tet-on, or Tet-off, that is the question: Advanced conditional gene expression in Aspergillus. Fungal Genet Biol. 89, 72-83 (2016).
- Karginov, A. V., Ding, F., Kota, P., Dokholyan, N. V., Hahn, K. M. Engineered allosteric activation of kinases in living cells. Nat Biotechnol. 28 (7), 743-747 (2010).
- Liu, Q. Y., Deiters, A. Optochemical Control of Deoxyoligonucleotide Function via a Nucleobase-Caging Approach. Accounts of Chemical Research. 47 (1), 45-55 (2014).
- Arbely, E., Torres-Kolbus, J., Deiters, A., Chin, J. W. Photocontrol of tyrosine phosphorylation in mammalian cells via genetic encoding of photocaged tyrosine. J Am Chem Soc. 134 (29), 11912-11915 (2012).
- Gautier, A., Deiters, A., Chin, J. W. Light-activated kinases enable temporal dissection of signaling networks in living cells. J Am Chem Soc. 133 (7), 2124-2127 (2011).
- Nguyen, D. P., et al. Genetic Encoding of Photocaged Cysteine Allows Photoactivation of TEV Protease in Live Mammalian Cells. J Am Chem Soc. 136 (6), 2240-2243 (2014).
- Deisseroth, K. Optogenetics. Nat Methods. 8 (1), 26-29 (2011).
- Boyden, E. S., Zhang, F., Bamberg, E., Nagel, G., Deisseroth, K. Millisecond-timescale, genetically targeted optical control of neural activity. Nat Neurosci. 8 (9), 1263-1268 (2005).
- Banghart, M., Borges, K., Isacoff, E., Trauner, D., Kramer, R. H. Light-activated ion channels for remote control of neuronal firing. Nat Neurosci. 7 (12), 1381-1386 (2004).
- Zhang, K., Cui, B. Optogenetic control of intracellular signaling pathways. Trends in Biotechnology. 33 (2), 92-100 (2015).
- Tischer, D., Weiner, O. D. Illuminating cell signalling with optogenetic tools. Nat Rev Mol Cell Bio. 15 (8), 551-558 (2014).
- Kim, B., Lin, M. Z. Optobiology: optical control of biological processes via protein engineering. Biochemical Society Transactions. 41 (5), 1183-1188 (2013).
- Tucker, C. L. Manipulating cellular processes using optical control of protein-protein interactions. Prog Brain Res. 196, 95-117 (2012).
- Toettcher, J. E., Gong, D. Q., Lim, W. A., Weiner, O. D. Light Control of Plasma Membrane Recruitment Using the Phy-Pif System. Method Enzymol. 497, 409-423 (2011).
- Zoltowski, B. D., Gardner, K. H. Tripping the light fantastic: blue-light photoreceptors as examples of environmentally modulated protein-protein interactions. Biochemistry. 50 (1), 4-16 (2011).
- Kennedy, M. J., et al. Rapid blue-light-mediated induction of protein interactions in living cells. Nat Methods. 7 (12), 973-975 (2010).
- Krishnamurthy, V. V., et al. Reversible optogenetic control of kinase activity during differentiation and embryonic development. Development. 143 (21), 4085-4094 (2016).
- Liu, H., et al. Photoexcited CRY2 Interacts with CIB1 to Regulate Transcription and Floral Initiation in Arabidopsis. Science. 322 (5907), 1535-1539 (2008).
- Li, X., et al. Arabidopsis cryptochrome 2 (CRY2) functions by the photoactivation mechanism distinct from the tryptophan (trp) triad-dependent photoreduction. Proc Natl Acad Sci U S A. 108 (51), 20844-20849 (2011).
- Leevers, S. J., Paterson, H. F., Marshall, C. J. Requirement for Ras in Raf Activation Is Overcome by Targeting Raf to the Plasma-Membrane. Nature. 369 (6479), 411-414 (1994).
- Kohn, A. D., Takeuchi, F., Roth, R. A. Akt, a pleckstrin homology domain containing kinase, is activated primarily by phosphorylation. J Biol Chem. 271 (36), 21920-21926 (1996).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sive, H. L., Grainger, R. M., Harland, R. M. Early Development of Xenopus laevis: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. (2000).
- Kim, J. H., et al. High cleavage efficiency of a 2A peptide derived from porcine teschovirus-1 in human cell lines, zebrafish and mice. PloS one. 6 (4), e18556(2011).
- Ishimura, A., et al. Oncogenic Met receptor induces ectopic structures in Xenopus embryos. Oncogene. 25 (31), 4286-4299 (2006).
- Zhang, K., et al. Light-Mediated Kinetic Control Reveals the Temporal Effect of the Raf/MEK/ERK Pathway in PC12 Cell Neurite Outgrowth. PloS one. 9 (3), e92917(2014).
- Mohanty, S. K., Lakshminarayananan, V. Optical Techniques in Optogenetics. J Mod Opt. 62 (12), 949-970 (2015).
- Taslimi, A., et al. Optimized second-generation CRY2-CIB dimerizers and photoactivatable Cre recombinase. Nature chemical biology. 12 (6), 425-430 (2016).
- Kawano, F., Suzuki, H., Furuya, A., Sato, M. Engineered pairs of distinct photoswitches for optogenetic control of cellular proteins. Nat Commun. 6, 6256(2015).
- Wang, H., et al. LOVTRAP: an optogenetic system for photoinduced protein dissociation. Nat Methods. 13 (9), 755-758 (2016).
- Chang, K. Y., et al. Light-inducible receptor tyrosine kinases that regulate neurotrophin signalling. Nat Commun. 5, 4057(2014).
- Boulina, M., Samarajeewa, H., Baker, J. D., Kim, M. D., Chiba, A. Live imaging of multicolor-labeled cells in Drosophila. Development. 140 (7), 1605-1613 (2013).
- Liu, H., Gomez, G., Lin, S., Lin, C. Optogenetic control of transcription in zebrafish. PLoS One. 7 (11), e50738(2012).
- Buckley, C. E., et al. Reversible Optogenetic Control of Subcellular Protein Localization in a Live Vertebrate Embryo. Dev Cell. 36 (1), 117-126 (2016).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature chemical biology. 10 (3), 196-202 (2014).
- Beyer, H. M., et al. Red Light-Regulated Reversible Nuclear Localization of Proteins in Mammalian Cells and Zebrafish. ACS Synth Biol. 4 (9), 951-958 (2015).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados