Method Article
Caracterização bioquímica e estrutural do carboidrato transporte proteína-substrato-SP0092
Neste Artigo
Resumo
É apresentado um protocolo simplificado para realizar uma extensa Caracterização bioquímica e estrutural de uma proteína de ligação do substrato de hidrato de carbono do Streptococcus pneumoniae .
Resumo
Desenvolvimento de novos agentes antimicrobianos e de vacinas para Streptococcus pneumoniae (pneumococo) são necessárias para deter a ascensão rápida em cepas resistentes de múltiplas. Proteínas de ligação de substrato de carboidratos (a) representam metas viáveis para o desenvolvimento de vacinas à base de proteínas e novos agentes antimicrobianos devido à sua localização extracelular e a centralidade da importação de hidrato de carbono para o metabolismo pneumocócica, respectivamente. Descrito aqui é um protocolo integrado Racionalizado para realizar uma caracterização abrangente de SP0092, que pode ser estendido para outros hidratos de carbono a partir do pneumococo e outras bactérias. Este procedimento pode ajudar o design baseado em estrutura de inibidores para esta classe de proteínas. Apresentado na primeira parte deste manuscrito são protocolos para análise bioquímica, por meio de ensaio térmico turno, multi ângulo de espalhamento de luz (MALS) e cromatografia de exclusão (SEC), do tamanho que otimizam a estabilidade e homogeneidade da amostra direcionado para ensaios de cristalização e assim aumentar a probabilidade de sucesso. A segunda parte deste procedimento descreve a caracterização dos cristais SBP usando uma trajetória de síncrotron de difração anômala ajustáveis, comprimento de onda e protocolos de coleta de dados para dados de medição que podem ser usados para resolver a proteína cristalizada estrutura.
Introdução
S. pneumoniae (pneumococo) é uma bactéria Gram-positiva que residem assintomaticamente em vias aéreas superiores do trato respiratório humano com a capacidade de migrar para nichos normalmente estéril causando otite média, pneumonia, sepse, septicemia, e meningite,1,2. Além disso, a infecção pneumocócica é a principal causa de pneumonia adquirida na Comunidade, que contribui para um fardo clínico e económico mundial3,4. Cepas resistentes aos antibióticos de S. pneumoniae espalharam-se pelo globo e embora um sete-valente e a vacina conjugada de proteína pneumocócica 13-valente ajudaram a reduzem a taxa de resistência aos antimicrobianos, variedades de substituição da uso de vacina surgiram e levaram a demanda crescente de investigação para o desenvolvimento de novos tratamentos para a doença pneumocócica5,6,7,8.
O pneumococo depende de açúcares importados de host como um carbono fonte9,10; com efeito, que dedica 30% de seu maquinário de importação para o transporte de carboidratos diferentes 3211,12,13. Estes importadores incluem pelo menos oito transportadores ABC13. Em transportadores ABC, a extracelular a desempenhar um papel fundamental na determinação da especificidade para o ligante e apresentá-lo para o transportador de integral de membrana para a absorção na célula. A representa os alvos válidos para a concepção de novas vacinas e antimicrobianos porque eles são proteínas de superfície e seu papel vital em processos celulares.
Caracterização de proteína alvo e descrição detalhada das características estruturais, como ligante bolsos e flexibilidade entre domínios, fornecem uma ferramenta útil para desenho de drogas baseada em estrutura14,15. Cristalografia de raios x é o método de escolha para a caracterização estrutural de proteínas em perto de resolução atômica, mas o processo de cristalização é imprevisível, demorado e não sempre bem sucedido. Métodos sistemáticos têm melhorado a taxa de sucesso e fatores importantes são a qualidade da amostra e a estabilidade. A taxa de sucesso de cristalização é influenciada pelas propriedades químicas de proteína e metodologia de preparação de amostra. O efeito destes pode ser avaliado e informado pela caracterização bioquímica16,17.
Uma outra complicação para design baseado em estrutura é o problema de fase cristalográfica, que deve ser abordado. Como estruturas de proteína mais se tornaram disponíveis, muitas estruturas podem ser resolvidas pelo método de substituição molecular, que exige uma estrutura homóloga18. Como a apresenta uma estrutura de domínio flexível, substituição molecular também pode revelar-se desafiador19. Se um modelo estrutural que é suficientemente semelhante à proteína alvo não estiver disponível, um número de técnicas pode ser usado para obter a progressiva experimental20. Entre estes, o método de dispersão anômala Single-comprimento de onda (SAD) surgiu como a principal técnica e tem sido extensivamente usado para resolver o problema de fase21. O uso do método triste tem sido ainda mais avançado com melhorias em hardware e software, bem como estratégias de coleta de dados para permitir a detecção e o uso de sinais fracos de anômalos para eliminação gradual de22,23, 24. Além disso, avanços em métodos diretos para solução de estruturas de macromoléculas, que, no passado, necessários dados de difração de resolução atômica, agora pode ser utilizado, por exemplo, combinando conhecimento estereoquímica conforme implementado no programa ARCIMOLDO25. Um recurso útil de métodos para resolver o problema da fase em cristalografia é dada por Taylor26.
Aqui nós apresentamos um protocolo Racionalizado para a caracterização do transporte de carboidratos SBP, SP0092 de S. pneumoniae, integrar técnicas bioquímicas e estruturais (Figura 1). Este protocolo passo a passo fornece um exemplo útil caso de teste de estratégias para melhorar a taxa de sucesso de estudos estruturais na apresentação, em geral, que são encontrados em todos os reinos da vida. Em particular, o protocolo destaca a importância de caracterizar o estado oligoméricos mais estável da proteína em solução em um método rápido e eficaz e permite a identificação das melhores espécies para acompanhar para experimentos de cristalização. Embora existam mais de 500 estruturas SBP relatadas na Protein Data Bank27, substituição molecular pode ser um desafio devido à natureza flexível inerente as dois domínios α/β, que são ligadas por uma dobradiça região19. Assim, a segunda parte do protocolo descreve como usar o método triste para eliminação de íons metálicos acoplados, que é comum na apresentação, bem como a incorporação de selenometionina e uso do selênio (Se) na fase triste.
Protocolo
Nota: A sequência de código para o qual é eliminado e o peptídeo sinal é clonada no vetor pOPINF seguindo um protocolo padrão de na fusão; a proteína nativa é expresso como uma fusão de sua marca em células de Escherichia coli BL21 Rosetta 28 , 29. a selenometionina rotulada variante é expresso seguindo métodos padrão de acordo com o fabricante de 30. A recombinação SBP é purificado como anteriormente descrito 31 , 32.
1. Caracterização bioquímica
- térmica deslocar ensaio
- 48 preparar soluções tampão com pH variando de 4.0 para 9,5 e concentração de NaCl de 0 a 0,5 M e dispense 40 µ l em uma placa de 96 poços, conforme descrito no Tabela 1 33.
- Adicionar para cada bem 5 µ l da solução de SBP na concentração de 1-2 mg/mL.
- Adicionar para cada bem 5 µ l de 20 x mancha fluorescente para proteínas (ver Tabela de materiais).
- Misturar as soluções pipetando, selar a placa usando uma película adesiva transparente e girar por 2 min a 112 x g, à temperatura ambiente.
- Executar o experimento em uma máquina em tempo real: definir uma rampa de temperatura de 3 ° C/min de 4 a 99 ° C com um tempo de espera 10 s e ler a fluorescência cada 0,5 ° C com excitação em 483 nm e emissão em 568 nm.
- Analisar as curvas de emissão de fluorescência para cada condição de buffer, identificando a temperatura de fusão (T m) como o mínimo da derivada.
Nota: Este procedimento dá uma boa indicação da melhor solução-tampão para ajudar na melhoria da estabilidade da proteína. Escolha a faixa de pH e concentração de sal com base na condição de reserva com a mais alta T m e adicionar 2,5% (v/v) de glicerol e 0,5 mM de tris(2-carboxyethyl) fosfina (TCEP) para definir a solução-tampão para as próximas etapas (SEC-tampão).
- MALS e analítico SEC
- realizar uma lavagem de duas colunas volume da coluna de filtração de gel pré embalados com água filtrada libertos (usar uma coluna de 24 mL de peso molecular de vasta gama).
- Conectar-se a coluna para o detector de espalhamento de luz e equilibrar a coluna com o determinado SEC-buffer de ensaio térmico turno.
- Injetar 100 µ l de SBP purificada em 5 mg/mL para o equilibrado SEC coluna e fluxo uma coluna volume do SEC-buffer.
- Verificar o cromatograma de eluição para único ou vários picos e examinar os dados de espalhamento usando o software de análise, obtendo a massa molar e o índice de polidispersividade para todas as espécies.
Nota: Para cada pico correspondendo a uma espécie oligoméricas, é benéfico verificar a polidispersividade índice calculado como massa molar ponderada na massa molar/massa ponderada em número de moléculas (Mw/Mn). Um valor de polidispersividade próximo de 1 indica um pico monodispersas.
Nota: Uma vez que todas as espécies de oligomerização são definidas, é útil estudar a sua dependência da concentração de proteína, como umas concentrações mais elevadas podem favorecer a maior formação oligómero. - Executar várias execuções de SEC analíticas; injetar 100 µ l de SBP purificado a aumentar as concentrações (0,1 - 10 mg/mL) na coluna de filtração de gel equilibrado (usar uma coluna de 5 mL de peso molecular de vasta gama) e um volume de coluna do SEC-buffer de fluxo de cada vez.
- Verificar o cromatograma de eluição para único ou vários picos e analisar as intensidades de absorbância em 280 nm das curvas correspondentes a diferentes concentrações de proteína de partida. Se as intensidades relativas dos picos diferentes permanecem constantes, nenhuma conversão inter entre as espécies oligoméricas está presente. Variação nas intensidades relativas às diferentes concentrações é uma indicação da interconversão entre diferentes espécies oligoméricas que é dependentes da concentração de proteína.
Nota: Esta etapa de caracterização é fundamental para definir quais espécies são mais adequadas para a cristalização. Com efeito, monodispersas distintas espécies são mais propensas a cristalizar-se eles estão em um estado constante oligoméricos numa concentração definidas.
2. Preparação de proteína e cristalização
- SEC Preparative
- após um volume de uma coluna de lavagem com água filtrada desgaseificada, equilibrar com SEC-buffer a coluna de filtração de gel de pré-embalados preparativa (120 mL de peso molecular uma variedade coluna).
- Injetar 15 mL de SBP purificado no 5-50 mg/mL para o volume de uma coluna coluna e fluxo da filtração do gel de equilibrada do SEC-buffer.
- Recolher a coluna de passagem em frações de 1 mL.
- Piscina as frações correspondentes a um pico único monodisperso; girar a amostra em filtros de centrifugação 15ml a 1.500 g e quantificar a concentração até se atingir a concentração desejada (normalmente de 50-100 mg/mL para SP0092).
- Cristalização
- determinar o intervalo de concentração ideal para o experimento de cristalização usando o teste de pré-cristalização conforme descrito pelo fabricante:
- Dispense 0,5 - 1,0 mL de cada um do teste da cristalização de quatro soluções em um reservatório diferente de uma sessão de 24-bem larga placa cristalização.
- Prepara as gotas de cristalização misturando 1 µ l de solução de proteína com 1 µ l de solução de reservatório, selar a placa e incubar à temperatura ambiente para não menos de 1 h (resultados mais confiáveis são obtidos após incubação durante a noite).
- Verificar a qualidade da gota para cada concentração, usando um microscópio de ampliação de 10x.
Nota: Testar a concentração da proteína diferente pelo menos três: (I) a maioria das gotas restantes clara indica que a proteína é demasiado diluída para cristalização; (II) a presença de precipitado pesado na maioria das gotas implica uma concentração demasiado elevada; e (III) uma equilibrada ocorrência de gotas claras e precipitado (melhor se luz) é uma boa indicação de que a concentração testada é favorável para cristalização.
- Preparar a queda de 96 poços sessão placas de cristalização; dispensar 100 µ l de solução de cristalização comerciais diferentes em cada reservatório bem 34 , 35 , 36 , 37.
- nL 100 dispensar da amostra de proteína na concentração definida anteriormente (etapa 1.1.6) em todos os poços de queda de cristalização usando um sistema robótico de cristalização.
- NL 100 dispensar do reservatório de diferentes soluções para a cristalização correspondente gota poços para misturar com proteína dispensada em passo 2.2.3. A placa de cristalização para evitar a evaporação e habilitar o equilibrio da gota cristalização com o reservatório do selo.
- Verificar as gotas periodicamente (inicialmente cada 1-2 dias, depois todas as semanas) usando um x 10 (pelo menos) microscópio de ampliação para avaliar o crescimento e a formação de cristais.
Nota: Um sistema automatizado de imagens para cristalização de proteínas pode ser usada para as gotas visualização e captura.
- determinar o intervalo de concentração ideal para o experimento de cristalização usando o teste de pré-cristalização conforme descrito pelo fabricante:
- cristal montagem
- Prepare a solução crioprotetoras adicionando 25% (v/v) de glicerol (concentração final) à condição de cristalização (substituindo assim os 25% de água na inicial mistura) 38.
- Preencher uma espuma dewar com nitrogênio líquido e coloque a parte de compartimento de amostra de uma uni-puck no dewar. Deixe-a esfriar a temperatura do nitrogênio líquido.
- Corte e remover a vedação a fita da placa de cristalização durante a queda, onde os cristais são formados.
- Colocar uma gota de 1 µ l de solução crioprotetoras sobre um coverslide posicionado nas proximidades da gota alvo.
- Transferir o cristal selecionado da lista suspensa original para a gota de solução crioprotetoras usando um nylon cryo-loop sobre uma base de coluna padrão montada em uma varinha magnética 39 , 40.
- Rapidamente transferir o cristal da lista suspensa crioprotetoras para nitrogênio líquido, colocando o loop para a primeira posição vazia do porta-amostras uni-disco.
- Repita (desde o passo corte de fita da selagem) até que todos os cristais desejados são colhidos e armazenados no porta-amostras uni-disco.
- Lugar o uni-disco de base sobre o uni-disco usando a varinha de disco e leve o disco para a trajetória (sob condições de nitrogênio líquido).
Atenção: A temperatura extremamente baixa! - Usar o cryo-pinça e o disco ferramenta de carregamento de dewar para carregar a uni-puck(s) para o robô de trocador de amostra beamline dewar. Porta-amostras uni-disco irá desanexar da base tampa deixando os titulares de laço de amostra na posição vertical no robô amostra dewar e assim exposto para o nitrogênio líquido e acessível para o robô de trocador de amostra.
- Caracterização de cristal
Nota: os cristais colhidos em seguida podem ser rastreados para qualidade de difração usando ou uma fonte de raio-x do laboratório ou em uma fonte de raio x radiação síncrotron (SR). Beamlines macromolecular cristalografia (MX) fornecem uma fonte de energia ajustáveis para explorar difração anômala para solução de estrutura. Na seção a seguir, propõe-se uma série de instruções de trabalho, em consonância com os recursos experimentais de MX de fonte de luz de diamante de Luz Síncroton, mas estas orientações também podem ser adaptadas para MX de luz síncroton em outros os síncrotrons em todo o mundo.- Como primeiro passo, é útil confirmar ou identificar anômala para disseminar no cristal. Isto pode ser conseguido convenientemente medindo um espectro de fluorescência de raio-x de cristal. Primeiro verifique se a energia do raio-x da trajetória está definida alta o suficiente para excitar todos os elementos que normalmente esperados para cristais macromoleculares (14 keV ou superior).
- Usar o software de controle de trajetória para selecionar a amostra a ser montado pelo robô trocador amostra; o loop será automaticamente centralizado no feixe de raios-x e a centralização de cristal pode ser confirmado usando manualmente o software de controle de trajetória.
- Gravar um espectro de fluorescência de raios-x usando o software de controle de trajetória: o usuário seleciona um tempo de exposição e inicia a medição (configuração do espectro de fluorescência/fluorescência, → executar, Figura 2A). O software de controle de trajetória automaticamente posiciona o raio-x, detector de fluorescência no lugar e determina o incidente mínimo fluxo de raio-x para obter um sinal legível sobre o detector de fluorescência. O espectro adquirido é então analisado em uma forma automatizada, com picos de emissão equipados de ocorrência natural elementos biológicos. Essas rotinas estão disponíveis em uma trajetória controle software interface gráfica do usuário (GUI) - GDA (www.opengda.org) em nossa empresa e os síncrotrons durante todo Europa 41 , 42.
- Se elementos adequados são identificados que podem ser exploradas por um experimento de difração anômala, executar uma verificação de energia de borda de absorção raio-x para determinar o comprimento de onda ideal para coleta de dados de difração anômala de cristal: sobre a trajetória software de controle, selecione o elemento e iniciar a digitalização (configuração de varredura de fluorescência/fluorescência, → nome de átomo, → executar, Figura 2B).
Nota: Para uma experiência única de difração anômala, o pico da borda de absorção é usado para maximizar a dispersão anômala. Considerações experimentais para a realização de um experimento de difração anômala de onda multi tem sido anteriormente detalhada 43. - Para determinar os parâmetros de célula unidade cristal, simetria e limite de difração, mede três padrões de difração de raios x em intervalos de 45°, utilizando o método de oscilação (coleta de dados/triagem → executar atual, Figura 2). O software de controle de trajetória fornece o usuário com uma escolha de ângulo de oscilação e tempo de exposição e a capacidade de definir a porcentagem de transmissão do feixe de raios-x. Para um rastreio padrão cristal, um ângulo de oscilação de 0,5 °, um tempo de exposição de 0,5 s e 5% transmissão de feixe de raios-x são recomendados. As imagens de difração medido são automaticamente analisadas pelo pipeline EDNA e retornar um conjunto de estratégias para a coleção de um conjunto de dados completo 22.
- Raio x coleta de dados
Nota: O usuário pode selecionar para coletar dados em modo de oscilação padrão ou feixe inversa, que permite que os companheiros de Friedel ser gravado perto no tempo e dose de raios-x e com aproximadamente o mesmo comportamento de absorção permitindo uma medição mais precisa das diferenças anômalas. O último é útil especialmente se esperam-se pequenas diferenças anômalas e/ou as amostras são radiação sensível ao realizar um experimento de difração anômala. Considerações sobre as melhores estratégias de coleta de dados para usar foram exaustivamente analisados 22 , 23 , 44 , 45 , 46 , 47.- usando o software de controle de trajetória, importe os parâmetros de coleta de dados como sugerido pelo programa de estratégia que irá fornecer:
i. Iniciar posição do eixo de rotação ω
ii. Largura de oscilação de rotação do eixo ω ângulo para cada imagem de difração
iii. Tempo de exposição para cada imagem de difração
iv. Número de imagens para um conjunto de dados completo (indiretamente definindo o ângulo de rotação total ω-eixo para a coleta de dados inteira)
v. percentagem de atenuação do feixe de raios-x para evitar excesso de exposição e radiação danos
Nota: em nossa instituição, para executando a coleta de dados, os encanamentos de software para processamento automatizado dos dados recolhidos estão bem estabelecidos: dados (i) a difração são reduzidos (indexado, integrada e dimensionado) pelo pipeline xia2, que gera arquivos de mtz de reflexão para o usuário como entrada para eliminação e estrutura de solução/refinamento 48. (ii) quando um significativo sinal anômalo é detectado durante a análise de dados, um primeira estrutura automatizada rápida solução pipeline (fast_ep) usando SHELX tenta resolver a subestrutura do átomo pesado por experimental gradual, fornecendo um mapa de densidade de elétrons em fases sempre que possível. Um segundo mineroduto de solução mais abrangente estrutura compromete-se automaticamente tenta resolver e construir o structuestiver usando software independente suites 49 , 50 ,, 51 , 52; em casos onde é bem sucedido, o usuário será fornecido com um mapa de densidade modelo e elétron inicial. Isso fornece a base para o usuário completar o requinte e validar o modelo com os pacotes de software cristalográficas de escolha.
- usando o software de controle de trajetória, importe os parâmetros de coleta de dados como sugerido pelo programa de estratégia que irá fornecer:
Resultados
Este protocolo integrado provou-se ser bem sucedido com quatro (dois publicados e dois inéditos estruturas) de carboidratos seis objectivos de proteína vinculativos de pneumococo analisaram até à data,32,53. Nesta seção, apresentamos a caracterização bioquímica e estrutural de SP0092 como resultado representativo para orientar estudos estruturais da apresentação em geral.
Após o SP0092 SBP foi expressa e purificado conforme definido anteriormente32, a proteína purificada foi analisada para estabilidade de buffer usando um ensaio de mudança térmica: SP0092 exibe um aumento Tm em pH 6,5 e na presença de NaCl no 0 - 0,2 M intervalo de concentração (Figura 3A). Neste contexto, a solução-tampão para as etapas a seguir foi definida como: pH MES de 0,02 M 6.5, 0,2 M NaCl, 2,5% (v/v) glicerol, 0.5 mM TCEP. Absoluta é a massa molar dos Estados diferentes oligomerização de SP0092 foi medida por MALS acoplado ao SEC medir um peso molecular de 187.2, 140.8, 97.0 e 49.4 kDa, correspondente à espécie tetramérica, trimeric, dimérica e monomérico, respectivamente ( Figura 3B). A análise do perfil do SEC em diluições diferentes proteínas revelou que a oligomerização é desencadeada pela concentração de proteína aumentada, sugerindo que os oligómeros maiores são mais estáveis em concentrações mais elevadas do que costuma ser usada no cristalização. Com efeito, as espécies oligoméricas maiores purificadas direcionado para ensaios de cristalização, cristais de proteína com sucesso produzido enquanto o monómero de espécies que não.
O otimizado cristais obtidos do nativo e metionina-Se rotulado de formas de SP0092 foram caracterizadas por difração de raios x. Medição da fluorescência de raio-x destes cristais revelou em ambos os casos picos de emissão para o Zn, sendo ligado à proteína, enquanto Se foi detectado apenas para os cristais de metionina-Se conforme o esperado. Mais tarde, exames de absorção de raio-x nas bordas Se e Zn foram realizadas, que forneceu dados experimentais diretos para ajustar o comprimento de onda do raio x incidente às respectivas bordas raio x absorção de Zn ou Se apresentam os cristais para maximizar o sinal anômalo obtidos a partir dos dados resultantes (Figura 4A–B).
Depois de medir três padrões de difração na baixa transmissão, obteve-se um conjunto completo de dados anômalo usando a estratégia de coleta de dados sugerida por EDNA (Figura 4). O sinal anômalo presente aciona o pipeline progressiva automatizado para a trajetória para determinar a estrutura da sub, e baseado em fases iniciais experimentais derivadas, produz inicial mapas e modelos, que podem ser refinados e validados (Figura 4 ).
Em resumo, possíveis armadilhas da técnica estão centradas principalmente na qualidade e disponibilidade de cristal. Otimização das condições de reserva, para melhorar a estabilidade da proteína, bem como a identificação do estado oligoméricas mais adequada da proteína para usar, quando mais do que um oligómero é identificado em solução, pode reduzir o risco de falha para a cristalização palco. Explorar o uso de íons metálicos acoplados identificadas numa fase precoce pode acelerar a solução da estrutura e evitar a produção desnecessária de selenometionina rotulada proteína quando métodos de substituição molecular falharem.
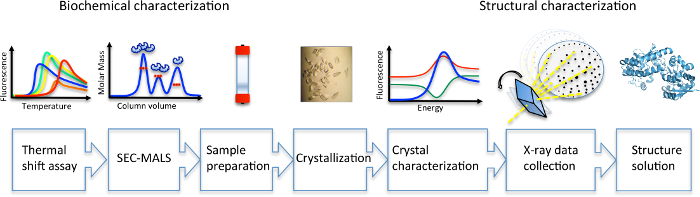
Figura 1. Diagrama de fluxo de trabalho para a caracterização bioquímica e estrutural de carboidratos a. Clique aqui para ver uma versão maior desta figura.
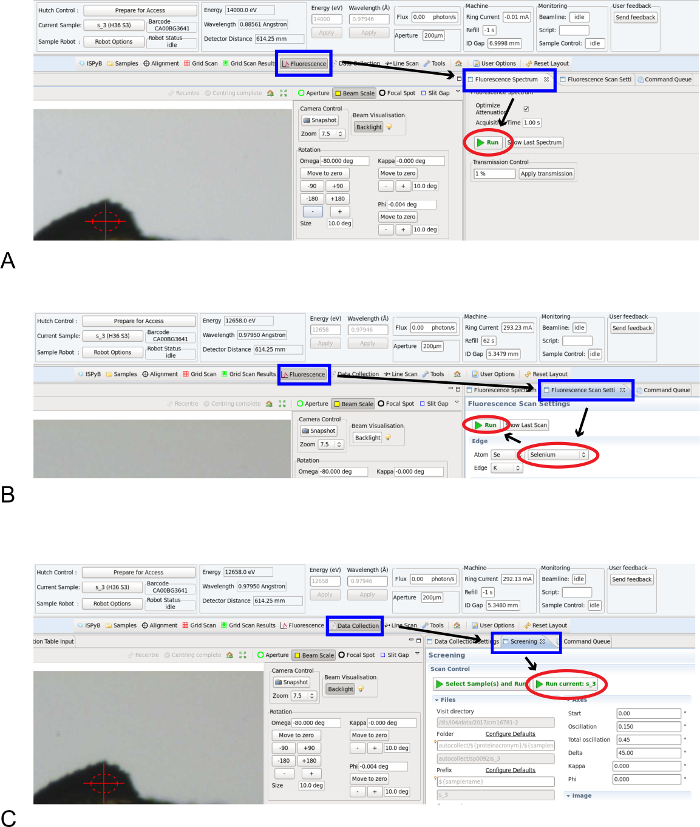
Figura 2. Caracterização de cristal em GDA. (A) Screenshot da guia de controle fluorescência de raio-x do software de controle de trajetória GDA. (B) imagem de raio-x energia borda-digitalização controle guia no GDA. (C) Screenshot da coleção cristal triagem guia dados no GDA. Clique aqui para ver uma versão maior desta figura.

Figura 3. Caracterização bioquímica de SP009239-491. Gráfico de superfície 3D (A) plotar a temperatura de fusão de SP009239-491 como uma função do pH e da concentração de NaCl da solução-tampão. (B) s e MALS resultados para SP009239-491. Absorção em 280 nm é mostrado em azul. As massas molares dos Estados oligomerização diferentes são mostradas em vermelho. Painel (B) foi modificado de32. Clique aqui para ver uma versão maior desta figura.
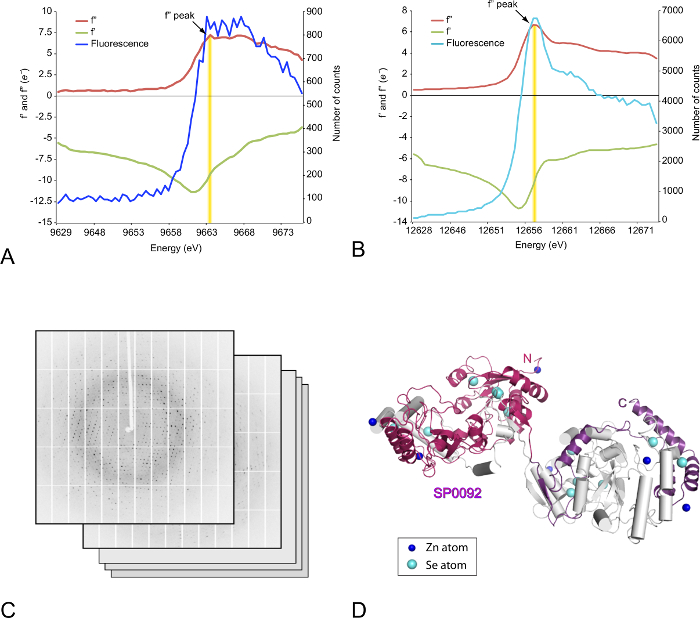
Figura 4. Caracterização estrutural de SP009239-491. (A) e energia de absorção de raios-x (B) varredura na borda para SP009239-491 cristais Zn e Se. A fluorescência medida do Zn e Se contendo cristais é mostrada em azul e ciano, o calculado f' e f "dispersão anômala frações são em verde e vermelho, respectivamente. (C) exemplos de padrões de difração de raios x coletados de cristais de SP009239-491 . (D) Cartoon representação da estrutura de dímero de SP009239-491 . Um protomer é colorido branco e o magenta (resíduos 39-366) e violeta (resíduos 367-491), e os átomos de dispersão anômala são mostrados como bolas azuis e ciana para Zn e Se, respectivamente. Clique aqui para ver uma versão maior desta figura.
| 0,1 M ácido cítrico pH 4.0 | 0,1 M ácido cítrico pH 4.5 | 0,1 pH de fosfato M 5.0 | 0.1 pH de citrato de M 5.5 | 0,1 M Bis-tris pH 6 | 0,1 M pH de ADA 6.5 | 0,1 M MOPS pH 7 | 0,1 M pH HEPES 7.5 | 0.1 pH 8.0 do Imidazole de M | 0,1 M Tris pH 8,5 | 0,1 M pH CHES 9 | 0,1 M pH CHES 9.5 | |
| NaCl 0 M | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL |
Tabela 1. Composição de tampão para ensaio térmico de turno.
Discussão
Neste artigo, descrevemos e validar um protocolo integrado para Caracterização bioquímica e estrutural de carboidratos apresentação com uma ênfase específica em proteínas de S. pneumoniae. No entanto, isto pode ser usado como um procedimento padrão para a análise de outra a partir de organismos diferentes e até mesmo outras proteínas solúveis não relacionadas.
A primeira parte do protocolo está focada em fornecer informações bioquímicas na estabilidade da proteína e estrutura quaternária, que pode ser explorada na preparação de amostras da proteína para cristalização. Na seção ensaios térmicos turno, descrevemos apenas o pH e as variações de concentração de NaCl para manter a natureza geral deste procedimento. Apesar disso, muitas outras condições do tampão podem ser testadas de forma semelhante, por exemplo, incluindo qualquer composto químico utilizado como aditivo estabilizador: em particular, a ligand(s) real que ligam para um específico SBP é notavelmente eficaz em aumentar a Tm por alguns graus Celsius31. Em alguns casos, as curvas de desnaturação podem ser mal definidas devido ao baixo sinal ou um fundo de fluorescência de alta, que é causado pela agregação de proteínas ou desdobramento parcial. Para evitar isso, uma titulação de proteína: tintura pode ser realizada para otimizar as curvas de desnaturação pouco claras. Se nenhuma melhoria é obtida, triagem de vários aditivos que podem melhorar a estabilidade da proteína é aconselhável, e telas adequadas tem sido descrito anteriormente54.
Normalmente, a maioria é monomérica em seu ambiente natural, mas como mostrado aqui multimerization pode ocorrer nas altas concentrações utilizadas em experimentos de cristalização, assim é a caracterização do comportamento de oligomerização fornecida por MALS e SEC essencial para avaliar o estado de oligomerização monodisperso estável mais favorável para a cristalização. No entanto, é difícil prever o efeito de diferentes produtos químicos incluídos na condição de cristalização no comportamento de oligomerização das proteínas. Se o exame da SEC e MALS mostra extensa agregação da amostra da proteína, recomendamos o seguinte para reduzir a probabilidade disso acontecer: usar amostra de proteína fresca (não congelar-descongelada) e expandir a análise de estabilização realizada com térmico ensaios, testes possíveis aditivos e detergentes suaves como um último recurso, para minimizar a agregação de turno. Neste trabalho, apresentamos as diretrizes básicas para cristalização com rastreio de cristalização comercial de matriz esparsa de alta produtividade para manter a natureza geral do presente protocolo. No entanto, a obtenção de cristais de proteína de difração de raios x de alta resolução pode precisar iterativo de ajuste fino para otimizar as condições de cristalização em relação a concentração precipitant, pH, adição de aditivos químicos, temperaturas diferentes, e outros fatores de mudança dinâmica de equilíbrio entre o cristal gota e reservatório16,17.
A segunda parte do protocolo descreve a caracterização os cristais de proteínas a fim de definir a melhor estratégia para coleta de dados de difração de raios x com um foco específico sobre a aquisição de dados anômalos para fase triste. Mesmo se a manter uma arquitetura geral semelhante (e existem muitas estruturas 3D depositadas potencialmente utilizável como modelos de partida), eliminação destas proteínas pelo método de substituição molecular não é sempre simples por causa da variabilidade do elementos de estrutura secundária e a flexibilidade intrínseca destas proteínas. Assim, propomos o método triste e destacar que estas proteínas podem já ter intrinsecamente ligado metais ou fato inespecificas de metais das condições de reserva de cristalização, que podem fornecer uma gama de elementos diffracting anômalas como um passo padrão em nosso protocolo geral.
Em conclusão, este protocolo define um padrão de fluxo de trabalho guiado de procedimentos que permitam a descrição detalhada das características bioquímicas e estruturais que pode ser explorada para aumentar a taxa de sucesso de determinação de estrutura, bem como acelerar a caracterização estrutural de apresentação em geral.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Reconhecemos a OPPF-UK para assistência em clonagem, Gemma Harris para SEC-shoppings e os cientistas de luz síncroton I03 e I04 na fonte de luz de diamante.
Materiais
| Name | Company | Catalog Number | Comments |
| SelenoMethionine Medium Complete | Molecular Dimensions | MD12-500 | Based on a synthetic M9 minimal media supplemented with glucose, vitamins and amino acids with the exception of L-methionine. Other equivalent products are commercially available by other companies. |
| MicroAmp Optical 96-Well plate | Applied Biosystems | 4306737 | The Applied Biosystems MicroAmp Optical 96-Well Reaction Plate with Barcode is optimized to provide unmatched temperature accuracy and uniformity for fast, efficient PCR amplification. This plate, constructed from a single rigid piece of polypropylene in a 96-well format, is compatible with Applied Biosystems® 96-Well Real-Time PCR systems and thermal cyclers. |
| SYPRO Orange | Molecular Probes | S6651 | SYPRO Orange Protein Gel Stain is a sensitive, ready-to-use fluorescent stain for proteins in 1D gels. Quite universal and well-established protein dye for hydrophobic regions. |
| MicroAmp Optical Adhesive film | Applied Biosystems | 4311971 | The Applied Biosystems MicroAmp Optical Adhesive Film reduces the chance of well-to-well contamination and sample evaporation when applied to a microplate. It is ideal for optical measurement, because it gives low background. |
| 7500 Fast Real-time PCR System | Applied Biosystems | 4362143 | The Applied Biosystems 7500 Fast Real-Time PCR System enables standard 96-well format high speed thermal cycling, significantly reducing your run time for quantitative real-time PCR applications, delivering results in about 30 minutes. The Upgrade Kit is available to upgrade a standard 7500 Real-Time PCR System to the Fast Configuration via a field service installation. Other RT-PCR machine can be used. Data export not easy for the old data analysis software. |
| Superdex 200 increase 10/300 GL column | GE Healthcare | 28990944 | Superdex 200 Increase 10/300 GL is a versatile, prepacked column for size exclusion chromatography in small-scale (mg) preparative purification as well as for characterization and analysis of proteins with molecular weights between 10 000 and 600 000, such as antibodies. Optimal separation ideal for high resolution biophysical techniques. |
| DAWN HELEOS II | Wyatt | DAWN HELEOS II is the premier Multi-Angle static Light Scattering (MALS) detector for absolute characterization of the molar mass and size of macromolecules and nanoparticles in solution. The DAWN offers the highest sensitivity, the widest range of molecular weight, size and concentration, and the largest selection of configurations and optional modules for enhanced capabiliites. Other MALS detecting systems from other companies apart from Wyatt have not been tested, so no additional feedback can be provided. | |
| Superdex 200 5/150 GL | GE Healthcare | 28906561 | Superdex 200 are prepacked size exclusion chromatography columns for high-resolution small-scale preparative and analytical separations of biomolecules. Superdex 200 has a separation range for molecules with molecular weights between 10 000 and 600 000. The peak separation is not as optimal as for the "increase" version but this model is ideal for the standard day by day use. |
| HiLoad 16/600 Superdex 200 pg | GE Healthcare | 28989335 | HiLoad 16/600 Superdex 200 prep grade are prepacked XK columns designed for high-resolution preparative gel filtration chromatography. |
| 24 Well "Big" Sitting Drop Crystallization Plate | MiTeGen | XQ-P-24S-A | The 24 well "big" crystallization plate is used mainly for protein crystal screening by sitting drop vapor diffusion techniques, and for crystallization condition optimization. It has quite large reservoir well and sample container, which favor manual handling and big sized protein crystal growth. In addition, its flat surfaces are easily to be sealed by transparent tape or cover slips. |
| PCT Pre-Crystallization Test | Hampton | HR2-140 | The PCT Pre-Crystallization Test is used to determine the appropriate protein concentration for crystallization screening. |
| 96 Well CrystalQuick | Greiner bio-one | 6098xx | Square-well plates have three crystallization wells per reservoir, making it possible to test 288 samples per plate. Generally used only for the inital screening because their squared edge make crystals fishing difficult. |
| Uni-Puck | Molecular Dimensions | MD7-601 | The Universal V1-Puck (Uni-puck) is a sample pin storage and shipping container for use with the majority of automated sample mounting systems worldwide - includes the ACTOR, SAM and CATS systems amongst others (Diamond, Soleil, SPring-8, Photon Factory, CLSI and across the USA) |
| Standard Foam Dewar | Molecular Dimensions | MD7-35 | 5.7" diameter by 2.8" deep. 800mL capacity. |
| Mounted CryoLoop - 20 micron | Hampton | HR4-955 | Mounted CryoLoops with 20 micron diameter nylon. These nylon loops are bonded to hollow, stainless steel MicroTubes™ that are used to mount, freeze, and secure the crystal during cryocrystallographic procedures and X-ray data collection. Different sizes exist and they can be adapted in lenght. They are quite versatile tools. |
| CryoWand | Molecular Dimensions | MD7-411 | |
| Puck dewar loading tool | Molecular Dimensions | MD7-607 | This tool is used to separate uni-pucks to load them into the robot dewar. It consists of two pieces: a Teflon tube part and a metal rod part. |
Referências
- Austrian, R. Prevention of pneumococcal infection by immunization with capsular polysaccharides of Streptococcus pneumoniae: current status of polyvalent vaccines. J Infect Dis. 136, S38-S42 (1977).
- Bogaert, D., De Groot, R., Hermans, P. W. Streptococcus pneumoniae colonisation: the key to pneumococcal disease. Lancet Infect Dis. 4 (3), 144-154 (2004).
- van Mens, S. P., et al. Longitudinal analysis of pneumococcal antibodies during community-acquired pneumonia reveals a much higher involvement of Streptococcus pneumoniae than estimated by conventional methods alone. Clin Vaccine Immunol. 18 (5), 796-801 (2011).
- Weycker, D., Strutton, D., Edelsberg, J., Sato, R., Jackson, L. A. Clinical and economic burden of pneumococcal disease in older US adults. Vaccine. 28 (31), 4955-4960 (2010).
- Doern, G. V. Antimicrobial use and the emergence of antimicrobial resistance with Streptococcus pneumoniae in the United States. Clin Infect Dis. 33, S187-S192 (2001).
- Vasoo, S., et al. Increasing antibiotic resistance in Streptococcus pneumoniae colonizing children attending day-care centres in Singapore. Respirology. 16 (8), 1241-1248 (2011).
- Black, S., et al. Efficacy, safety and immunogenicity of heptavalent pneumococcal conjugate vaccine in children. Northern California Kaiser Permanente Vaccine Study Center Group. Pediatr Infect Dis J. 19 (3), 187-195 (2000).
- Hanage, W. P., et al. Diversity and antibiotic resistance among nonvaccine serotypes of Streptococcus pneumoniae carriage isolates in the post-heptavalent conjugate vaccine era. J Infect Dis. 195 (3), 347-352 (2007).
- Burnaugh, A. M., Frantz, L. J., King, S. J. Growth of Streptococcus pneumoniae on human glycoconjugates is dependent upon the sequential activity of bacterial exoglycosidases. J Bacteriol. 190 (1), 221-230 (2008).
- King, S. J. Pneumococcal modification of host sugars: a major contributor to colonization of the human airway?. Mol Oral Microbiol. 25 (1), 15-24 (2010).
- Tettelin, H., et al. Complete genome sequence of a virulent isolate of Streptococcus pneumoniae. Science. 293 (5529), 498-506 (2001).
- Buckwalter, C. M. Pneumococcal carbohydrate transport: food for thought. Trends Microbiol. 20 (11), 517-522 (2012).
- Bidossi, A., et al. A functional genomics approach to establish the complement of carbohydrate transporters in Streptococcus pneumoniae. PLoS One. 7 (3), e33320 (2012).
- Zheng, X., Gan, L., Wang, E., Wang, J. Pocket-based drug design: exploring pocket space. AAPS J. 15 (1), 228-241 (2013).
- Singh, S., Malik, B. K., Sharma, D. K. Molecular drug targets and structure based drug design: A holistic approach. Bioinformation. 1 (8), 314-320 (2006).
- Dessau, M. A., Modis, Y. Protein crystallization for X-ray crystallography. J Vis Exp. (47), (2011).
- Bergfors, T. . Protein crystallization: techniques, strategies, and tips. A laboratory manual. , (1999).
- Scapin, G. Molecular replacement then and now. Acta Crystallogr D Biol Crystallogr. 69 (Pt 11), 2266-2275 (2013).
- Mao, B., Pear, M. R., McCammon, J. A., Quiocho, F. A. Hinge-bending in L-arabinose-binding protein. The "Venus's-flytrap" model. J Biol Chem. 257 (3), 1131-1133 (1982).
- McCoy, A. J., Read, R. J. Experimental phasing: best practice and pitfalls. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 458-469 (2010).
- Dodson, E. Is it jolly SAD?. Acta Crystallogr D Biol Crystallogr. 59 (Pt 11), 1958-1965 (2003).
- Incardona, M. F., et al. EDNA: a framework for plugin-based applications applied to X-ray experiment online data analysis. J Synchrotron Radiat. 16 (Pt 6), 872-879 (2009).
- Popov, A. N., Bourenkov, G. P. Choice of data-collection parameters based on statistic modelling. Acta Crystallogr D Biol Crystallogr. 59 (Pt 7), 1145-1153 (2003).
- Terwilliger, T. C., et al. Can I solve my structure by SAD phasing? Planning an experiment, scaling data and evaluating the useful anomalous correlation and anomalous signal. Acta Crystallogr D Struct Biol. 72 (Pt 3), 359-374 (2016).
- Millan, C., Sammito, M., Uson, I. Macromolecular ab initio phasing enforcing secondary and tertiary structure. IUCrJ. 2 (Pt 1), 95-105 (2015).
- Taylor, G. L. Introduction to phasing. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 325-338 (2010).
- Berman, H. M., et al. The Protein Data Bank. Nucleic Acids Res. 28 (1), 235-242 (2000).
- Bird, L. E., et al. Green fluorescent protein-based expression screening of membrane proteins in Escherichia coli. J Vis Exp. (95), e52357 (2015).
- Berrow, N. S., et al. A versatile ligation-independent cloning method suitable for high-throughput expression screening applications. Nucleic Acids Res. 35 (6), e45 (2007).
- Hendrickson, W. A., Horton, J. R., LeMaster, D. M. Selenomethionyl proteins produced for analysis by multiwavelength anomalous diffraction (MAD): a vehicle for direct determination of three-dimensional structure. EMBO J. 9 (5), 1665-1672 (1990).
- Culurgioni, S., Harris, G., Singh, A. K., King, S. J., Walsh, M. A. Structural Basis for Regulation and Specificity of Fructooligosaccharide Import in Streptococcus pneumoniae. Structure. , (2016).
- Culurgioni, S., Tang, M., Walsh, M. A. Structural characterization of the Streptococcus pneumoniae carbohydrate substrate-binding protein SP0092. Acta Crystallogr F Struct Biol Commun. 73 (Pt 1), 54-61 (2017).
- Ericsson, U. B., Hallberg, B. M., Detitta, G. T., Dekker, N., Nordlund, P. Thermofluor-based high-throughput stability optimization of proteins for structural studies. Anal Biochem. 357 (2), 289-298 (2006).
- Wooh, J. W., Kidd, R. D., Martin, J. L., Kobe, B. Comparison of three commercial sparse-matrix crystallization screens. Acta Crystallogr D Biol Crystallogr. 59 (Pt 4), 769-772 (2003).
- Gorrec, F. Protein crystallization screens developed at the MRC Laboratory of Molecular Biology. Drug Discov Today. 21 (5), 819-825 (2016).
- Jancarik, J., Scott, W. G., Milligan, D. L., Koshland, D. E., Kim, S. H. Crystallization and preliminary X-ray diffraction study of the ligand-binding domain of the bacterial chemotaxis-mediating aspartate receptor of Salmonella typhimurium. J Mol Biol. 221 (1), 31-34 (1991).
- Newman, J., et al. Towards rationalization of crystallization screening for small- to medium-sized academic laboratories: the PACT/JCSG+ strategy. Acta Crystallogr D Biol Crystallogr. 61 (Pt 10), 1426-1431 (2005).
- Garman, E. F., Mitchell, E. P. Glycerol concentrations required for cryoprotection of 50 typical protein crystallization solutions. Journal of Applied Crystallography. 29 (5), 584-587 (1996).
- Teng, T. -. Y. Mounting of crystals for macromolecular crystallography in a free-standing thin film. Journal of Applied Crystallography. 23 (5), 387-391 (1990).
- Cipriani, F., et al. Automation of sample mounting for macromolecular crystallography. Acta Crystallogr D Biol Crystallogr. 62 (Pt 10), 1251-1259 (2006).
- Gabadinho, J., et al. MxCuBE: a synchrotron beamline control environment customized for macromolecular crystallography experiments. J Synchrotron Radiat. 17 (5), 700-707 (2010).
- Leonard, G. A., et al. Online collection and analysis of X-ray fluorescence spectra on the macromolecular crystallography beamlines of the ESRF. Journal of Applied Crystallography. 42 (2), 333-335 (2009).
- Walsh, M. A., Evans, G., Sanishvili, R., Dementieva, I., Joachimiak, A. MAD data collection - current trends. Acta Crystallogr D Biol Crystallogr. 55 (Pt 10), 1726-1732 (1999).
- Dauter, Z. Data-collection strategies. Acta Crystallogr D Biol Crystallogr. 55 (Pt 10), 1703-1717 (1999).
- Finke, A. D., et al. Advanced Crystallographic Data Collection Protocols for Experimental Phasing. Methods Mol Biol. 1320, 175-191 (2016).
- Gonzalez, A. Optimizing data collection for structure determination. Acta Crystallogr D Biol Crystallogr. 59 (Pt 11), 1935-1942 (2003).
- Leslie, A. G. The integration of macromolecular diffraction data. Acta Crystallogr D Biol Crystallogr. 62 (Pt 1), 48-57 (2006).
- Winter, G., Lobley, C. M., Prince, S. M. Decision making in xia2. Acta Crystallogr D Biol Crystallogr. 69 (Pt 7), 1260-1273 (2013).
- Sheldrick, G. M. Experimental phasing with SHELXC/D/E: combining chain tracing with density modification. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 479-485 (2010).
- Skubak, P., Pannu, N. S. Automatic protein structure solution from weak X-ray data. Nat Commun. 4, 2777 (2013).
- Terwilliger, T. C., et al. Decision-making in structure solution using Bayesian estimates of map quality: the PHENIX AutoSol wizard. Acta Crystallogr D Biol Crystallogr. 65 (Pt 6), 582-601 (2009).
- Vonrhein, C., Blanc, E., Roversi, P., Bricogne, G. Automated structure solution with autoSHARP. Methods Mol Biol. 364, 215-230 (2007).
- Culurgioni, S., Harris, G., Singh, A. K., King, S. J., Walsh, M. A. Structural Basis for Regulation and Specificity of Fructooligosaccharide Import in Streptococcus pneumoniae. Structure. 25 (1), 79-93 (2017).
- Boivin, S., Kozak, S., Meijers, R. Optimization of protein purification and characterization using Thermofluor screens. Protein Expression and Purification. 91 (2), 192-206 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados