É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Elevado-throughput sequenciamento paralela à medida Fitness de Leptospira interrogans Transposon inserção mutantes durante a Golden sírio Hamster infecção
Neste Artigo
Resumo
Aqui descrevemos uma técnica que combina o mutagenesis transposon com sequenciamento de alto rendimento para identificar e quantificar os mutantes leptospiral de transposon em tecidos após um desafio de hamsters. Este protocolo pode ser usado para mutantes de tela para a sobrevivência e disseminação em animais e também pode ser aplicado aos estudos em vitro .
Resumo
Este manuscrito, descrevemos um transposon sequenciamento (Tn-Seq) técnica para identificar e quantificar os mutantes de Leptospira interrogans alterados em fitness durante a infecção de hamsters sírios dourados. TN-Seq combina o mutagenesis aleatório transposon com o poder da tecnologia de sequenciamento de alta produtividade. Animais são desafiados com uma piscina de mutantes transposon (pool de entrada), seguida de colheita de sangue e tecidos alguns dias mais tarde para identificar e quantificar o número de mutantes em cada órgão (piscinas de saída). As piscinas de saída são comparadas com o pool de entrada para avaliar a aptidão na vivo de cada mutante. Esta abordagem permite a triagem de uma grande piscina de mutantes em um número limitado de animais. Com pequenas modificações, este protocolo pode ser executado com qualquer modelo animal de leptospirose, modelos de hospedeiro reservatório tais como ratos e modelos de infecção aguda, tais como os hamsters, bem como estudos em vitro . TN-Seq fornece uma ferramenta poderosa para a tela para mutantes com defeitos de aptidão in vivo e in vitro .
Introdução
Identificação de genes de virulência para algumas bactérias, tais como Leptospira spp., é difícil devido ao número limitado de ferramentas genéticas disponíveis. Uma abordagem comumente utilizada é a criação de uma colecção de mutantes por mutagênese aleatória transposon, seguido da identificação do local de inserção em cada mutante e virulência testes de mutantes transposon individuais em um modelo animal. Essa abordagem é demorado, caro e requer um grande número de animais.
Quando o mutagenesis aleatório foi desenvolvido para o patógeno Leptospira interrogans, genes envolvidos na virulência foram identificados pelo teste mutantes individuais em um modelo animal1. Os mutantes foram selecionados com base em critérios como suas potenciais funções na sinalização ou motilidade ou sua membrana externa prevista ou superfície local. Como a maioria dos leptospiral genes codificam proteínas hipotéticas de função desconhecida2, selecionando os mutantes baseiam em limites esses critérios a capacidade de detectar genes de virulência leptospiral romance.
Mais recentemente, piscinas de mutantes de transposon L. interrogans foram projectadas para infecciosidade no hamster e mouse modelos3. Cada animal foi desafiado com uma piscina de até 10 mutantes. Infecciosidade de um mutante foi marcada como positivo se foi detectado por PCR das culturas obtidos de sangue e rim. Teste de PCR foi trabalhoso porque necessitava de uma reação de PCR individual para cada mutante na piscina. Porque a frequência de cada mutante nas culturas não foi quantificada, a abordagem foi inclinada para identificação de mutantes altamente atenuadas.
Descrevemos um transposon sequenciamento (Tn-Seq) técnica, como uma estratégia para a tela de forma mais eficiente para genes de virulência. TN-seq consiste da criação de uma biblioteca de mutantes por mutagénese transposon seguido por sequenciamento paralelo maciço4,5,6. Brevemente, transposon mutantes são agrupados, inoculados em animais e posteriormente recuperados de diferentes órgãos (piscinas de saída). O DNA das piscinas saída é extraído e digerido com enzimas de restrição ou distorcido pelo sonication. São realizadas duas rodadas de PCR direcionamento das junções dos locais de inserção de transposon. Esta etapa permite a adição dos adaptadores necessários para o sequenciamento. Os produtos resultantes da PCR são analisados por sequenciamento de alto rendimento para identificar o local de inserção do transposon de cada mutante da piscina junto com a sua abundância relativa, que é comparada com a composição inicial do grupo de mutantes.
A principal vantagem desta abordagem é a capacidade de tela simultaneamente um grande número de mutantes com um pequeno número de animais. TN-Seq não exige o conhecimento prévio dos locais de inserção do transposon que aumenta as chances de descobrir novos Leptospira-genes específicos envolvidos na virulência com menos tempo e maior eficiência. Porque leptospiral fardo nos tecidos é relativamente alto em roedores modelos suscetíveis a letal infecção (tipicamente 104 108 bactérias/g de tecido)7,8,9 , bem como em hospedeiros reservatório 10,11, os tecidos podem ser analisados diretamente sem a necessidade de cultura, reduzindo preconceitos devido ao crescimento em vitro .
Em estudos de Tn-Seq com a maioria dos patógenos bacterianos descritos até à data, a alta frequência de mutagênese insercional permitido infecção com grandes piscinas contendo mutantes coletivamente tendo múltiplas inserções espaçadas transposon dentro de cada gene4 ,12,13,14. TN-Seq também foi desenvolvido para uma bactéria, para o qual a frequência de mutagênese é muito inferior6. Com Leptospira, uma biblioteca de mutantes transposon pode ser gerada introduzindo o transposon em um plasmídeo sequência por conjugação, como descrito por Slamti et al.15. No entanto, a frequência de mutagénese transposon de L. interrogans é baixa. Quando o transposon Himar1 foi introduzido em um plasmídeo conjugativo, a frequência de transconjugant foi relatada para ser apenas 8,5 x 10-8 por beneficiário da pilha com a cepa de Lai de L. interrogans16 e é provável que seja da mesma forma pobre com a maioria das outras cepas de L. interrogans. O protocolo descrito aqui é em parte com base no que desenvolvi para Borrelia burgdorferi, em que a frequência de mutagênese insercional transposon é também baixa6.
Para a nossa experiência piloto com o protocolo17, realizamos mutagênese transposon com L. interrogans sorovar Manilae estirpe L495 por causa do sucesso de outros grupos em isolamento de mutantes de inserção de transposon na estirpe juntamente com a sua baixa DL50 (dose letal) para virulência1. Nós selecionados 42 mutantes por Tn-Seq e identificou vários candidatos mutantes defeituosos em virulência, incluindo dois com inserções em um gene de adenilato ciclase de candidato. O teste individual dos dois mutantes em hamsters confirmou que eles estavam deficientes em virulência17.
Protocolo
Cuidado: Cepas patogênicas de Leptospira spp. devem ser tratadas no âmbito de procedimentos de contenção do nível de biossegurança 2 (BSL-2). Equipamento de protecção adequado (EPI) deve ser usado. Uma armário de biossegurança classe II deve ser utilizada para todas as manipulações de patogenicidade Leptospira spp..
1. criação do Transposon Mutant biblioteca15
-
Transferência do transposon para Leptospira spp. por conjugação (Figura 1)
- Inocule um volume de cultura exponencial-fase de patogenicidade Leptospira spp, correspondente a 107 células em 10 mL de Ellinghausen McCullough-Johnson-Harris médio (EMJH)18,19. Incube a 30 ° C, com 150 rpm, agitando até que a densidade atinge 2-8 x 108 células/mL.
Nota: O tempo de duplicação das leptospiras patogênicas é de 12 a 24 h, dependendo da estirpe. - Inocular 50 µ l do doador Escherichia coli estirpe β216320 carregando a sequência transposon plasmídeo (pCjTKS2)16 em 5 mL de caldo Luria (LB) para suplementado com 0,3 mM de 2,6-diaminopimelicacid (DAP), 50 µ g/mL de canamicina (Km) e 50 µ g/mL de espectinomicina (Spc) e lugar durante a noite em uma incubadora de 37 ° C em 255 rpm.
- Inocule 60 µ l de células de Escherichia coli em 3 mL de EMJH suplementado com 0,3 mM de DAP (EMJH + DAP). Incube a 37 ° C, a agitação de 255 rpm para 3-4 h até uma OD600nm≈ 0,3.
- Para montar a unidade de filtração (Figura 1), coloque a base sobre uma tubuladura lateral 125 mL frasco Erlenmeyer, coloque um filtro de acetato-celulose (tamanho de poros 0,1 mm; diâmetro 25mm) sobre o base e prenda o funil na base. Ligue a unidade de filtração para um sistema de vácuo.
- Adicione 5 mL de Leptospira spp. cultura e 0,5 mL de cultura de e. coli dentro do funil. O líquido através do filtro de vácuo.
- Transferi o filtro com a superfície de bactérias, virada para cima para um EMJH + DAPplate. Incube a 30 ° C durante a noite com o filtro virado para cima.
- Coloque o filtro em um tubo de 15 mL contendo 1 mL de EMJH e vórtice para 10 s para liberar as bactérias em meios de comunicação. Espalhou-se 200 µ l da suspensão em 5 EMJH platescontaining 50 µ g/mL de Km usando contas de vidro estéril de 1 mm de 10-15 ou um espalhador estéril descartável. Embrulhe as placas com parafilm e incube-os de cabeça para baixo a 30 ° C para 3 a 4 semanas até que colônias são visíveis.
- Transferência de colônias individualmente em 3 mL de EMJH contendo 50 µ g/mL de Km (EMJH + Km) a 30° C, sob agitação a 150 rpm por 7 a 10 dias até que a cultura atinge uma densidade de ≈ 108/mL.
Nota: As culturas podem ser armazenadas a-80 ° C ou em nitrogênio líquido (com 4% de glicerol).
- Inocule um volume de cultura exponencial-fase de patogenicidade Leptospira spp, correspondente a 107 células em 10 mL de Ellinghausen McCullough-Johnson-Harris médio (EMJH)18,19. Incube a 30 ° C, com 150 rpm, agitando até que a densidade atinge 2-8 x 108 células/mL.
-
Identificação do local de inserção do transposon pelo PCR aninhado (Figura 2)
- Lyse 50 µ l de cada mutante transposon em cadeia da polimerase por incubação a 95 ° C por 15 min.
Nota: DNA pode ser purificado em vez disso, usando um kit de extração de DNA. - Prepare a mistura PCR com primers Deg1 e Tnk1 (tabela 3), de acordo com a tabela 1. Transferência de 23,7 µ l da mistura para cada tubo PCR e adicionar 1,3 µ l de células lisadas. Execute o programa: 95° C por 5 min; 40 ciclos: 95 ° C por 15 s, 40 ° C por 1 min, 72 ° C por 2 min; 72 ° C por 10 min.
- Fazer a mistura PCR com primers Tag e TnkN1 (tabela 3), de acordo com a tabela 2. Transferência de 24,2 µ l da mistura para cada tubo PCR e adicionar 0,8 µ l de reação de PCR #1. Execute o programa: 95 ° C por 5 min; 35 ciclos: 95 ° C por 15 s, 55 ° C por 30 s, 72 ° C por 2 min; 72 ° C por 10 min.
- Execute 3 µ l de produtos do PCR em um gel de agarose 1% com 1 buffer X Tris-Acetato-EDTA (TAE) em 10-15 V/cm (Figura 2B).
- Purifica os produtos de PCR das amostras positivas, usando um kit de purificação de PCR. Eluir o DNA com o volume mais baixo permitido pelo kit para maximizar a concentração de DNA recuperado.
- Envie produtos PCR purificados por Sanger sequenciamento usando o primer TnkN1 (tabela 3).
- Identifica os locais de inserção comparando a sequência resultante com a sequência do genoma da estirpe parental pela análise BLASTN (http://blast.ncbi.nlm.nih.gov/) ou usando o SpiroScope banco de dados (http://www.genoscope.cns.fr/agc/mage)21.
- Confirme o local de inserção do transposon por PCR utilizando primers recozimento para as sequências flanqueando do anfitrião.
Nota: O transposon aumenta o tamanho da sequência de tipo selvagem de ≈ 2 kb.
- Lyse 50 µ l de cada mutante transposon em cadeia da polimerase por incubação a 95 ° C por 15 min.
2. animal experimento (Figura 3)
-
Cultura de Leptospira mutantes
- Cresce individualmente cada mutante transposon selecionado em 10 mL de EMJH + Km a 30 ° C, a agitação a 150 rpm para uma densidade de7-108 as leptospiras/10ml.
- Conte as leptospiras pela microscopia de campo escuro com um contador de Petroff-Hausser ou como descrito por Miller23.
- Dilua cada cultura EMJH para a mesma densidade, por exemplo 106 células/mL.
- Para montar a entrada piscina, misture as culturas diluídas em volumes iguais.
Nota: Incluir controles na piscina entrada adicionando os mutantes com defeitos de aptidão conhecido como loa2217,24 e mutantes com fitness inalterado como ligB17,25, respectivamente. Piscinas de mutantes podem ser armazenadas com 4% de glicerol a-80 ° C ou em nitrogênio líquido.
-
Desafio
Nota: Os métodos descritos aqui foram aprovados pelo Comitê de uso (protocolo #09018-14) e veteranos assuntos maior Los Angeles institucional Cuidado Animal.- Injecte intraperitonealmente 1ml da entrada piscina para cada animal com uma seringa de insulina U-100 com 26 x ½" agulha.
Nota: No experimento piloto, 8 animais foram desafiados com 1 mL de entrada piscina, ou seja, 106 bactérias total17. A infecção foi autorizada a prosseguir por 4 dias antes da eutanásia. - Coletar 10 mL de entrada piscina e girar para 20 min no x 3.220 g. Remova cuidadosamente o sobrenadante sem perturbar o sedimento. Armazene o centrifugado em - 80˚C até o uso (passo 3.1.3).
- Monitore os animais diariamente, até que eles são finalizados no ponto de extremidade predeterminado. Pesar os hamsters diariamente e procurar por critérios de ponto de extremidade: perda de apetite, marcha ou respirar, dificuldade, prostração, peles de babados, ou perda de > 10% do peso máximo atingido.
- Injecte intraperitonealmente 1ml da entrada piscina para cada animal com uma seringa de insulina U-100 com 26 x ½" agulha.
-
Experimento in vitro
- No dia do desafio, inocule 3 frascos de 25 mL de EMJH + Km com 5 mL de entrada piscina. Cultivar culturas a 30 ° C, sob agitação a 150 rpm.
- Conte as leptospiras diariamente usando um dos métodos da secção 2.1.2. Quando a densidade atinge ≈ 1 x 108/mL, spin para baixo cada suspensão por 20 min a 3.220 x g.
- Armazene as pelotas de célula a-80 ° C até o uso.
-
Colheita e armazenamento dos tecidos
- Eutanásia dos animais por inalação de isoflurano, seguida por toracotomia bilateral26.
- Imediatamente colete 1 a 2 mL de sangue por punção cardíaca com uma seringa de 3 mL e uma agulha 25 x 5/8". Transferi o sangue no tubo contendo EDTA. Misture por inversão, 5 a 6 vezes.
- Colete um rim e ⅓ para ½ do lobo mediano ventral do fígado em cryotubes.
- Loja de tecidos a-80 ° C até o uso.
3. construção de bibliotecas genômicas para arranjar em sequência do elevado-throughput (Figura 4)
-
Extração de DNA
- Extração de DNA do sangue.
- Transferi 100 µ l de sangue do tubo EDTA para um tubo de microcentrifugadora.
- Purifica DNA usando um kit de extração de DNA. Siga as instruções do fabricante.
- Extração de DNA de tecidos.
- Usando bisturis e tesouras, entre 50 a 80 mg de cada órgão em pedaços pequenos (1 mm x 1 mm) com dados e transferi-los para um tubo seco estéril de tampa de rosca. Medir o peso do tecido com uma balança de precisão.
- Adicione 500 µ l de PBS estéril no tubo.
- Homogeneizar as amostras usando o disruptor por 1 min em 5 movimentos por segundo.
- Calcule o volume correspondente a 25 mg de tecido utilizando a seguinte equação:
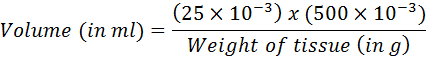
- Transferi o volume calculado para o kit de extração de DNA. Prosseguir com a purificação de DNA, seguindo as instruções do fabricante.
- Extração de DNA das culturas entrada de piscina e in vitro .
- Descongele o pelotas bacterianas em temperatura ambiente por 5-10 min.
- Prosseguir com a extração de DNA, seguindo as instruções que acompanham o kit.
- DNA de loja a-80 ° C até o uso.
- Extração de DNA do sangue.
-
O DNA de corte (Figura 5)
- Transferi 50 µ l de DNA extraído em um tubo de microcentrifugadora de 1,5 mL.
- Coloque os tubos dentro do rack do chifre sonicador copo cheio de água fria (4 ° C).
- Executar o sonicador por 3 min a 80% de intensidade com 10 s no pulso e 5 s do pulso.
Atenção: Usar aquecedores de orelha ou tampões para os ouvidos para proteger a audição. - Executar 2,5 µ l do cortado o DNA em um gel de agarose 2% para confirmar que a maioria do DNA é < 600 bp em tamanho.
-
Adição da cauda-C
- Medir a concentração de DNA com um Espectrofotômetro de pequeno volume.
- Calcular o volume correspondente a 500 ng de DNA usando a seguinte equação:
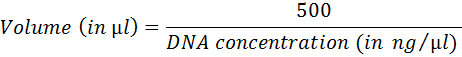
- Prepare a reação de rejeito (tabela 4). Incubar a 1 h a 37 ° C; inativar a 75 ° C por 20 min.
Nota: Para amostras que exigem ajuste para um volume superior a 14,5 µ l, aumentar o volume final de reação de 40 µ l e scale-up os componentes restantes em conformidade. - Limpe as amostras com um kit de purificação de PCR. Eluir o DNA com 12 µ l de tampão de eluição.
-
PCR aninhado
- PCR #1
- Prepare a mistura PCR de acordo com tabelas 3 e 5. Transferência de 22 µ l do mix biblioteca para cada tubo PCR e adicione 3 µ l de DNA purificado. Proceda da mesma forma com a mistura de controle.
Nota: Primer TnkN317 é específico para o transposon e cartilha olj3766 é específico para o C-cauda. A mistura de controle está faltando o primer TnkN3, que visa especificamente o transposon. - Execute o seguinte programa: 95 ° C por 2 min; 24 ciclos: 95 ° C por 30 s, 60 ° C por 30 s, 72 ° C por 2 min; 72 ° C por 2 min.
- Prepare a mistura PCR de acordo com tabelas 3 e 5. Transferência de 22 µ l do mix biblioteca para cada tubo PCR e adicione 3 µ l de DNA purificado. Proceda da mesma forma com a mistura de controle.
- #2 DE PCR.
- Prepare a mistura PCR segunda de acordo com tabelas 3 e 6. Transferência de 49 µ l do mix biblioteca para cada tubo PCR e adicione 1 µ l de reação de PCR #1 biblioteca. Transferência de 24,5 µ l do mix controle para cada tubo PCR e adicione 0,5 µ l de reação de PCR #1 controle.
Nota: PMargent2 Primer é específico para o transposon, e as primeiras demão IP6 conter código de barras do seis-base-par e reconhecidas pela plataforma de próxima geração de sequenciamento de sequências específicas. - Execute o seguinte programa: 95 ° C por 2 min; 18 ciclos: 95 ° C por 30 s, 60 ° C por 30 s, 72 ° C por 2 min; 72 ° C por 2 min.
- Prepare a mistura PCR segunda de acordo com tabelas 3 e 6. Transferência de 49 µ l do mix biblioteca para cada tubo PCR e adicione 1 µ l de reação de PCR #1 biblioteca. Transferência de 24,5 µ l do mix controle para cada tubo PCR e adicione 0,5 µ l de reação de PCR #1 controle.
- Execute 3 µ l em um gel de agarose 2%. A biblioteca deve mostrar um esfregaço com a maioria do sinal entre 200 a 600 bp (Figura 6) e sem amplificação para a reação de controle.
- PCR #1
-
Purificação de produtos PCR.
Nota: Limpe as bibliotecas genômicas com um kit de purificação de PCR seguindo as instruções do fabricante. Eluir o DNA com 30 µ l de tampão de eluição. - Medir a concentração de DNA usando um dados. Médias 2 a 3 leituras.
- Calcule o volume de cada biblioteca equivalente a 15 ou 20 ng usando a equação abaixo:
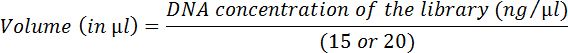
-
Misturar todas as bibliotecas juntos de acordo com os cálculos anteriores. Determine a concentração molar do DNA com equação do seguinte site:
http://www.molbiol.edu.ru/ENG/scripts/01_07.html
Cuidado: Requisitos de volume e concentração de DNA dependem da plataforma de sequenciamento.
4. elevado-throughput sequenciamento e análise de dados
- Sequenciamento
- Bibliotecas de sequência como 64 leituras de único-extremidade bp usando o sequenciamento personalizado cartilha pMargent3 e o primer de sequenciamento comercial padrão. A plataforma de sequenciamento irá fornecer-lhe FastQ arquivos com todas as leituras de sequenciamento.
- Análise com o software Galaxy
- Baixe o arquivo da sequência do genoma
- Na homepage SpiroScope banco de dados (consulte a etapa 1.2.7 para link), selecione o organismo usado para o experimento e clique em "LOAD em navegador do GENOMA".
- Na barra de ferramentas (perto do topo da página inicial), selecione "busca/exportação > Baixar dados", na linha de "Sequência (fasta)", clique em "Genoma" para baixar a sequência.
- Abra o arquivo com o bloco de notas (PC) ou TextEdit (Mac) e renomear o cromossomo (por exemplo, "Cristo"). Manter o formato fasta.
- Siga os mesmos passos para baixar a sequência do cromossoma II. Combine ambas as sequências de cromossomos em um arquivo. txt simples copiando e colando.
Nota: Cromossomo sequências também podem ser baixadas do site do NCBI (https://www.ncbi.nlm.nih.gov) e combinadas em um arquivo único. txt.
- Upload de arquivos para o servidor da galáxia
Nota: A galáxia é uma fonte aberta, plataforma baseada na web para o gerenciamento de dados intensivos bioinformática fluxos de trabalho27,28,29,30 e pode ser acessada em https://usegalaxy.org/.- No menu ferramentas, selecione "obter dados > Enviar arquivo do seu computador". Arraste e solte os arquivos de .fastq gerados pela plataforma de sequenciamento para a janela, em seguida, clique em "Iniciar".
- Siga as mesmas etapas para carregar o arquivo. txt de sequência de genoma de Leptospira .
- Noivo lê
- Selecione "NGS: QC e manipulação > FASTQ Groomer" do menu ferramentas.
- Ao lado de arquivo ao noivo, selecione as bibliotecas carregadas na etapa 4.2.2. Em tipo de escores de qualidade de FASTQ de entrada, selecione o sistema de sequenciamento adequado. Para opções avançadas, deixe esconder Advanced Office selecionado.
- Clique em "Executar".
- Remover artefatos de sequenciamento
- Selecione "NGS: QC e manipulação > remover artefatos de sequenciamento". Ao lado da biblioteca para filtrar, selecione os arquivos preparados gerados na etapa 4.2.3. Clique em "Executar".
- Remover sequências C-cauda
Nota: Repita estes passos uma vez ou duas vezes para garantir que todos os C-caudas são removidos.- Selecione "NGS: QC e manipulação > Clip sequências de adaptador".
- Selecione ou digite o seguinte:
Biblioteca de clip: selecione os arquivos gerados na etapa 4.2.4.
Comprimento de sequência mínima: 15
Fonte: digite a sequência personalizada
Digite a sequência de recorte personalizado: CCCCCCC
Digite o valor diferente de zero para manter o adaptador sequências e bases de x que seguem-lo: 0
Descarte de sequências com desconhecidos (N) bases: Sim
Opções de saída: saída sequências recortadas e não-cortada - Clique em "Executar".
- Remover sequências de adaptador
- Selecione "NGS: QC e manipulação > Clip sequências de adaptador".
- Selecione ou digite o seguinte:
- Biblioteca de clip: selecione os arquivos gerados na etapa 4.2.5.
- Comprimento de sequência mínima: 15
- Fonte: digite a sequência personalizada
- Digite a sequência de recorte personalizado: CGTATGCCGTCTTCTGCTTG
- Digite o valor diferente de zero para manter o adaptador sequências e bases de x que seguem-lo: 0
- Descarte de sequências com desconhecidos (N) bases: Sim
- Opções de saída: saída sequências recortadas e não-cortada
- Clique em "Executar".
- Leituras de filtro com base em sua qualidade
- Selecione "NGS: QC e manipulação > filtro de qualidade".
Nota: Esta ferramenta seleciona leituras com base em resultados de qualidade. - Selecione o seguinte:
Biblioteca para filtrar: selecione os arquivos gerados na etapa 4.2.6.
Valor de corte de qualidade: 20
Por cento das bases em sequência que deve ter qualidade igual a/superior valor de Cut-off: 95 - Clique em "Executar".
Nota: Com essas configurações, leituras mais curtas do que 20 nucleotídeos ou com um índice de qualidade de 20 ou menos para 95% dos ciclos são descartadas. Adaptar-se as configurações de rigor a sua experiência.
- Selecione "NGS: QC e manipulação > filtro de qualidade".
- Mapa lê32
- Selecione "NGS: mapeamento > Bowtie2"32.
- Selecione o seguinte nos campos na janela principal:
É esta biblioteca única ou a par: single-final
Arquivo FASTQ: selecione a biblioteca filtrada pela qualidade da etapa 4.2.7.
Escrever leituras desalinhadas (no formato fastq) para separar os arquivo (s): não
Escrever leituras alinhadas (no formato fastq) para separar os arquivo (s): não
Você irá selecionar um genoma de referência de sua história ou usar um índice interno?: Use um genoma da história e construir o índice
Selecione o genoma de referência: selecione o arquivo de genome.txt de Leptospira carregado na etapa 4.2.2.
Conjunto ler informações de grupos?: não definida
Modo de análise selecione: 1: configuração padrão somente
Você quer usar predefinições?: não, é só usar padrões
Salve as estatísticas de mapeamento de bowtie2 para a história: não
Parâmetros do recurso de trabalho: usar parâmetros de recurso de trabalho padrão - Clique em "Executar" para alinhar leituras para o genoma.
- Converter arquivos
- Selecione "NGS: SAMtools > BAM BAM-para-SAM converter SAM".
- Selecione o seguinte:
Arquivo de BAM para converter: selecione mapeado biblioteca da etapa 4.2.8.
Opções de cabeçalho: incluir cabeçalho na saída de SAM (-h) - Clique em "Executar".
- Converter arquivos
- Selecione "NGS: SAMtools > converter SAM em intervalo".
- Selecione o seguinte:
Selecione o conjunto de dados a converter: selecione o arquivo de biblioteca mapeada SAM gerado na etapa 4.2.9.
Imprimir tudo?: Sim - Clique em "Executar".
- Tipo de leituras
- Selecione "filtrar e classificar > classificar dados em ordem crescente ou decrescente".
- Selecione o seguinte:
Conjunto de dados tipo: selecione o arquivo de intervalo gerado na etapa 4.2.10.
na coluna: 2
com sabor: ordem numérica
tudo em: ordem crescente - Clique em "Executar".
- Selecione lê correspondência cromossomo eu
- Selecione "filtrar e classificar > selecione as linhas que correspondem a uma expressão".
- Selecione o seguinte:
Selecione as linhas de: selecione o arquivo com leituras classificadas gerado na etapa 4.2.11.
isso: correspondência
o padrão: digite o nome de cromossomo eu como determinado na etapa 4.2.1.3 (por exemplo, "Cristo"). - Clique em "Executar".
- Selecione lê correspondência cromossomo II
Prossiga seguindo passo 4.2.12. - Grupo lê de acordo com os sites de inserções no cromossomo eu
- Selecione "unir, subtrair e grupo > agrupar dados por uma coluna e executar operação agregada em outras colunas".
- Selecione o seguinte:
Selecionar dados: selecione o arquivo resultante da etapa 4.2.12.
Agrupar por coluna: 2
Ignorar o caso enquanto agrupamento?: não
Ignorar as linhas que começam com esses personagens: Ø
Operação > + insert operação
Tipo: contagem
Na coluna: 2
Arredondar o resultado para o inteiro mais próximo: N - Clique em "Executar".
- Grupo lê de acordo com os locais de inserção no cromossomo II
Prossiga seguindo passo 4.2.14. - Locais de introdução da espécie no cromossomo eu
- Selecione "filtrar e classificar > classificar dados em ordem crescente ou decrescente".
- Selecione o seguinte:
Conjunto de tipo de dados: Selecione o arquivo da etapa 4.2.14.
na coluna: 1
com sabor: ordem numérica
tudo em: ordem crescente - Clique em "Executar".
- Locais de inserção tipo no cromossomo II
Prossiga seguindo passo 4.2.16.
- Baixe o arquivo da sequência do genoma
- Análise estatística
- Transferi os dados de galáxia em arquivos de planilha, copiando e colando as duas colunas de passos 4.2.16. e 4.2.17. em um arquivo do Excel.
Nota: A primeira coluna é a coordenada de nucleotídeo transposon local de introdução, e a segunda coluna é o número de leituras em cada local de inserção. - Identifica o gene abrigando o transposon usando a coordenada de nucleótidos fornecida na tabela.
Nota: por exemplo, um transposon inserido no nucleotídeo #30718 do cromossomo é localizado no gene LIC10024 , que se estende por nucleotídeos em cromossomo 29263-31539 eu. - Calcule as frequências relativas (F) para cada mutante em cada tecido e na piscina entrada seguinte a equação abaixo:
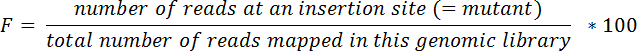
- Calcule índices de entrada/saída (R) para cada mutante e tecido, usando a equação abaixo:
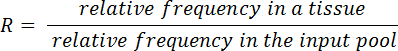
- Teste de Wilcoxon rank-assinado
- Normalize todos os rácios de entrada/saída, definindo a relação mediana para cada tecido em cada animal para 1.0. Uma razão de 1.0 é neutra, > 1.0 é desvantajoso, e < 1.0 é vantajoso33.
- Comparar os rácios de saída/entrada para 1.0 (aptidão neutro) usando o teste de Wilcoxon rank com valores de P < 0,05 considerado estatisticamente significativo.
- Transferi os dados de galáxia em arquivos de planilha, copiando e colando as duas colunas de passos 4.2.16. e 4.2.17. em um arquivo do Excel.
Resultados
Criação de uma biblioteca de mutantes transposon em L. interrogans por conjugação requer uma unidade de filtração, como mostrado na Figura 1. Recuperamos a 100-200 transconjugants de cada acasalamento.
O local de inserção do transposon é identificado em cada mutante pelo sequenciamento do produto do PCR gerado pelo PCR semi-aleatório que tem como alvo o fim do transposon e acolhim...
Discussão
Embora os resultados de nossa experiência piloto para hamster desafiada intraperitonealmente com 42 L. interrogans mutantes são apresentados17, esperamos que os maiores piscinas de mutantes podem ser rastreadas por Tn-Seq Porque a frequência de transconjugants é baixa (100-200 transconjugants/acasalamento), vários acasalamentos são necessários para gerar um número suficiente de mutantes para grandes experiências de Tn-Seq. Manter um grande número de mutantes em culturas líquida...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi apoiado por um prêmio de mérito de assuntos de veteranos (a D.A.H.) e um Instituto Nacional de saúde conceder R01 AI 034431 (a D.A.H.).
Materiais
| Name | Company | Catalog Number | Comments |
| Kanamycin sulfate from Streptomyces kanamyceticus | Sigma-Aldrich | K4000 | |
| 2,6-diaminopimelic acid | Sigma-Aldrich | D1377 | |
| Spectinomycin dihydrochloride pentahydrate | Sigma-Aldrich | S4014 | |
| Axio Lab A1 microscope with a darkfieldcondenser | Zeiss | 490950-001-000 | |
| DNeasy blood and tissue kit | Qiagen | 69504/69506 | |
| MinElute PCR Purification | Qiagen | 28004/28006 | |
| QIAquick PCR purification kit | Qiagen | 28104/28106 | |
| Model 505 Sonic Dismembrator | Fisher Scientific | FB-505 | |
| 2.5" Cup horn | Fisher Scientific | FB-4625 | |
| Bead Ruptor 24 | Omni International | 19-010 | Step 3.1.2.4 |
| Terminal deoxynucleotidyl transferase | Promega | M828C | |
| Master mix Phusion | Thermo Scientific | F531 | Preparation of genomic libraries, step 3.4. |
| DreamTaq Master Mix | Thermo Scientific | K9011/K9012 | Identification of the transposon insertion site, step 1.2. |
| dCTP | Thermo Scientific | R0151 | |
| ddCTP | Affymetrix/ USBProducts | 77112 | |
| T100 Thermal cycler | BioRad | 1861096 | |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | step 3.6. |
| Qubit dsDNA HS assay kit | Invitrogen | Q32851/Q32854 | step 3.6. |
| Qubit assay tubes | life technologies | Q32856 | step 3.6. |
| PBS pH 7.2 (1X) | Gibco | 20012-027 20012-050 | |
| Disposable scalpel No10 | Feather | 2975#10 | |
| Plastic K2 EDTA 2 ml tubes | BD vacutainer | 367841 | |
| syringe U-100 with 26G x ½” needle | BD vacutainer | 329652 | IP challenge, step 2.2.1. |
| 3 mL Luer-Lok tip syringe | BD vacutainer | 309657 | Cardiac puncture, step 2.4.2. |
| 25G X 5/8” needle | BD vacutainer | 305901 | Cardiac puncture, step 2.4.2. |
| 25 mm fritted glass base with stopper | EMD Millipore | XX1002502 | Filtration unit system, step 1.1.7. |
| 25 mm aluminum spring clamp | EMD Millipore | XX1002503 | Filtration unit system, step 1.1.7. |
| 15 ml borosilcate glass funnel | EMD Millipore | XX1002514 | Filtration unit system, step 1.1.7. |
| 125 ml side-arm Erlenmeyer flask | EMD Millipore | XX1002505 | Filtration unit system, step 1.1.7. |
| Acetate-cellulose filter VVPP (pore size 0.1 mm; diameter 25 mm) | EMD Millipore | VVLP02500 |
Referências
- Murray, G. L., et al. Genome-wide transposon mutagenesis in pathogenic Leptospira species. Infect Immun. 77 (2), 810-816 (2009).
- Adler, B., Lo, M., Seemann, T., Murray, G. L. Pathogenesis of leptospirosis: the influence of genomics. Vet Microbiol. 153 (1-2), 73-81 (2011).
- Marcsisin, R. A., et al. Use of a high-throughput screen to identify Leptospira mutants unable to colonize the carrier host or cause disease in the acute model of infection. J Med Microbiol. 62, 1601-1608 (2013).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat Methods. 6 (10), 767-772 (2009).
- van Opijnen, T., Lazinski, D. W., Camilli, A. Genome-wide fitness and genetic interactions determined by Tn-seq, a high-throughput massively parallel sequencing method for microorganisms. Curr Protoc Microbiol. 36, 1E.3.1-1E.3.24 (2015).
- Troy, E. B., et al. Understanding barriers to Borrelia burgdorferi dissemination during infection using massively parallel sequencing. Infect Immun. 81 (7), 2347-2357 (2013).
- Wunder, E. A., et al. Real-time PCR reveals rapid dissemination of Leptospira interrogans after intraperitoneal and conjunctival inoculation of hamsters. Infect Immun. 84 (7), 2105-2115 (2016).
- Lourdault, K., Aviat, F., Picardeau, M. Use of quantitative real-time PCR for studying the dissemination of Leptospira interrogans in the guinea pig infection model of leptospirosis. J Med Microbiol. 58, 648-655 (2009).
- Coutinho, M. L., et al. Kinetics of Leptospira interrogans infection in hamsters after intradermal and subcutaneous challenge. PLoS Negl Trop Dis. 8 (11), e3307 (2014).
- Athanazio, D. A., et al. Rattus norvegicus as a model for persistent renal colonization by pathogenic Leptospira interrogans. Acta Trop. 105 (2), 176-180 (2008).
- Ratet, G., et al. Live imaging of bioluminescent Leptospira interrogans in mice reveals renal colonization as a stealth escape from the blood defenses and antibiotics. PLoS Negl Trop Dis. 8 (12), e3359 (2014).
- Gutierrez, M. G., Yoder-Himes, D. R., Warawa, J. M. Comprehensive identification of virulence factors required for respiratory melioidosis using Tn-seq mutagenesis. Front Cell Infect Microbiol. 5 (78), (2015).
- Gawronski, J. D., Wong, S. M., Giannoukos, G., Ward, D. V., Akerley, B. J. Tracking insertion mutants within libraries by deep sequencing and a genome-wide screen for Haemophilus genes required in the lung. Proc Natl Acad Sci U S A. 106 (38), 16422-16427 (2009).
- Gallagher, L. A., Shendure, J., Manoil, C. Genome-scale identification of resistance functions in Pseudomonas aeruginosa using Tn-seq. MBio. 2 (1), (2011).
- Slamti, L., Picardeau, M. Construction of a library of random mutants in the spirochete Leptospira biflexa using a mariner transposon. Methods Mol Biol. 859, (2012).
- Picardeau, M. Conjugative transfer between Escherichia coli and Leptospira spp. as a new genetic tool. Appl Environ Microbiol. 74 (1), 319-322 (2008).
- Lourdault, K., Matsunaga, J., Haake, D. A. High-throughput parallel sequencing to measure fitness of Leptospira interrogans transposon insertion mutants during acute infection. PLoS Negl Trop Dis. 10 (11), e0005117 (2016).
- Ellinghausen, H. C., McCullough, W. G. Nutrition of Leptospirapomona and growth of 13 other serotypes: Fractionation of oleic albumin complex and a medium of bovine albumin and polysorbate 80. Am J Vet Res. 26, 45-51 (1965).
- Johnson, R. C., Harris, V. G. Differentiation of pathogenic and saprophytic letospires. I. Growth at low temperatures. J Bacteriol. 94 (1), 27-31 (1967).
- Demarre, G., et al. A new family of mobilizable suicide plasmids based on broad host range R388 plasmid (IncW) and RP4 plasmid (IncPa) conjugative machineries and their cognate Escherichia coli host strains. Res Microbiol. 156 (2), 245-255 (2005).
- Vallenet, D., et al. MicroScope: a platform for microbial genome annotation and comparative genomics. Database (Oxford). , bap021 (2009).
- Goodman, A. L., Wu, M., Gordon, J. I. Identifying microbial fitness determinants by insertion sequencing using genome-wide transposon mutant libraries. Nat. Protocols. 6 (12), 1969-1980 (2011).
- Miller, J. . Spirochetes in body fluids and tissues manual of investigative methods. , 22-23 (1971).
- Ristow, P., et al. The OmpA-like protein Loa22 is essential for leptospiral virulence. PLoS Pathog. 3 (7), (2007).
- Croda, J., et al. Targeted mutagenesis in pathogenic Leptospira species: disruption of the LigB gene does not affect virulence in animal models of leptospirosis. Infect Immun. 76 (12), 5826-5833 (2008).
- Haake, D. A. Hamster model of leptospirosis. Curr Protoc Microbiol. 12, 2 (2006).
- Giardine, B., et al. Galaxy: a platform for interactive large-scale genome analysis. Genome Res. 15 (10), 1451-1455 (2005).
- Goecks, J., Nekrutenko, A., Taylor, J., Galaxy, T. Galaxy: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences. Genome Biol. 11 (8), R86 (2010).
- Blankenberg, D., et al. Galaxy: a web-based genome analysis tool for experimentalists. Curr Protoc Mol Biol. (Chapter 19), 11-21 (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2016 update. Nucleic Acids Res. 8 (44), (2016).
- McCoy, K. M., Antonio, M. L., van Opijnen, T. MAGenTA; a Galaxy implemented tool for complete Tn-Seq analysis and data visualization. Bioinformatics. , 1367 (2017).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10 (3), R25 (2009).
- Kamp, H. D., Patimalla-Dipali, B., Lazinski, D. W., Wallace-Gadsden, F., Camilli, A. Gene fitness landscapes of Vibrio cholerae at important stages of its life cycle. PLoS Pathog. 9 (12), e1003800 (2013).
- Poggi, D., Oliveira de Giuseppe, P., Picardeau, M. Antibiotic resistance markers for genetic manipulations of Leptospira spp. Appl Environ Microbiol. 76 (14), 4882-4885 (2010).
- van Opijnen, T., Camilli, A. Transposon insertion sequencing: a new tool for systems-level analysis of microorganisms. Nat Rev Microbiol. 11 (7), 435-442 (2013).
- Pappas, C. J., Picardeau, M. Control of gene expression in Leptospira spp. by Transcription activator-like effectors demonstrates a potential role for LigA and LigB in Leptospira interrogans virulence. Appl Environ Microbiol. 81 (22), 7888-7892 (2015).
- Koskiniemi, S., Sun, S., Berg, O. G., Andersson, D. I. Selection-driven gene loss in bacteria. PLoS Genet. 8 (6), e1002787 (2012).
- Troy, E. B., et al. Global Tn-seq analysis of carbohydrate utilization and vertebrate infectivity of Borrelia burgdorferi. Mol Microbiol. 101 (6), 1003-1023 (2016).
- Ramsey, M. E., et al. A high-throughput genetic screen identifies previously uncharacterized Borrelia burgdorferi genes important for resistance against reactive oxygen and nitrogen species. PLoS Negl Trop Dis. 13 (2), e1006225 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados