É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Avaliação In Vitro o dano de DNA usando ensaio cometa
Neste Artigo
Resumo
O ensaio cometa é um método eficiente para detectar danos do DNA incluindo single e double-stranded DNA quebra. Descrevemos o cometa alcalino e neutro de ensaios para medir o dano de DNA em células cancerosas para avaliar o efeito terapêutico da quimioterapia.
Resumo
Dano do ADN é um fenômeno comum para cada célula durante sua vida útil e é definido como uma alteração da estrutura química do DNA genômico. Terapias de câncer, tais como o rádio e quimioterapia, apresentar a enorme quantidade de danos adicionais de DNA, levando à detenção do ciclo celular e apoptose para limitar a progressão do câncer. Avaliação quantitativa do dano do ADN durante a terapia de câncer experimental é um passo fundamental para justificar a eficácia de um agente genotóxico. Neste estudo, focamos em um ensaio de electroforese única célula, também conhecido como o ensaio cometa, que pode quantificar single e double-strand DNA quebras em vitro. O ensaio cometa é um método de quantificação de dano de DNA que é eficiente e fácil de executar, e tem exigências de tempo/orçamento baixas e alta reprodutibilidade. Aqui, destacamos o utilitário do ensaio cometa para estudos pré-clínicos, avaliando o efeito genotóxico de terapia de combinação olaparib/temozolomide U251 células de glioma.
Introdução
O ensaio cometa foi primeiro desenvolvido por Ostling e Johanson, em 1984, demonstrando que a migração do DNA fragmentos dos núcleos sob uma condição neutra1. A técnica foi desenvolvida mais tarde por Singh et al, mostrando que uma condição alcalina aumentou substancialmente a especificidade e a reprodutibilidade do ensaio2. Desde então, o ensaio cometa neutro é usado principalmente para detectar quebras de DNA dupla-hélice, Considerando que o ensaio cometa alcalino é mais sensível para pequenas quantidades de dano do ADN, incluindo único e quebra de DNA de fita dupla, alcaloide-labile sites, DNA-DNA ou Cross-linking da ADN-proteína e quebras de single-strand DNA associadas com incompleta excisão reparar sites3,4. Ambos os ensaios permitem a visualização de DNA fragmentado e fornecem uma maneira simples de avaliar quantitativamente o dano do ADN. O ensaio cometa é considerado como um método sensível para os estudos toxicológicos in vitro e em vivo genéticos e é aplicável a áreas de investigação diferentes, tais como a seleção precoce de drogas-candidato, monitoramento ambiental, a biomonitorização humana, e fundamental Pesquisar no dano do ADN e reparar5.
O princípio do ensaio é que sob um campo elétrico, DNA fragmentado migra para fora do corpo do nucleoide (também conhecido como o "cabeça de cometa") e forma uma mancha de DNA no gel do agarose (também conhecido como a "cauda de cometa"). Com coloração de nucleótidos, a extensão dos danos do DNA pode ser quantificada pela análise "cometas", formados por electroforese esta única célula. Cálculo do momento de cauda mais pode ajudar a comparar o dano do ADN entre diferentes grupos experimentais. Em comparação com métodos tradicionais de detecção de danos do DNA, o ensaio cometa é direta, sensível, barata e relativamente simples.
Radioterapia e agentes quimioterápicos são estratégias comuns para o tratamento de câncer, gerando único filamento e quebras de DNA de fita dupla em cromossomos6. O avanço recente em inibidores de reparação do ADN permite um mais eficaz efeito genotóxico pela quimioterapia de combinação e, portanto, potencialmente reduz os efeitos secundários sistêmicos tais como anemia, infecção e medula óssea supressão7, 8. neste estudo, mostramos a investigação de um inibidor de poli (ADP-ribose) polimerase (PARP), olaparib (Ola)9. PARP é uma proteína nuclear abundante e é responsável pelo reparo de excisão de base de DNA, formando um polímero de poli (ADP-ribose)10. Temozolomide (TMZ) é um Agente alquilante disponível por via oral e tem sido amplamente utilizada para o tratamento do paciente de glioma. Usando o ensaio cometa para quantificar os danos do DNA, demonstramos que combina olaparib com temozolomide profundamente aumenta o dano de DNA em células de glioma, que sugere a terapia combinada de olaparib/temozolomide é uma estratégia eficaz para tratar de glioma, comparado com o temozolomide sozinho11.
Protocolo
1. preparar reagentes
- 1X PBS
- diluir 100 mL x 10 PBS com 900 mL dH 2 O e ajustar o pH para 7,4 usando um medidor de pH. Armazenar em temperatura ambiente.
- Solução de lise (LS)
- preparar 2,5 M NaCl, EDTA dissódico de 100 mM, 10 mM Tris base e 200mm NaOH em 900 mL dH 2 O; comumente leva cerca de 20 min para permitir a mistura dissolver totalmente. Ajuste o pH a 10 usando um medidor de pH. Adicionar sarcosinate de Laurilsulfato de sódio 1% e 1% Triton X-100 e ajustar o volume final de 1.000 mL. Legal a 4 ° C, durante pelo menos 30 min antes do uso.
- Solução de eletroforese alcalina (AES), pH > 13
- preparar dissódico NaOH e 1 mM 200 mM EDTA em 800 mL dH 2 O. ajustar o pH e certifique-se que é pH > 13. Ajuste o volume final de 1.000 mL. Fazer fresco antes do utilizar e refrigerar a 4 ° C, durante pelo menos 30 min antes do uso.
- Sollution electroforese neutro (NES)
- preparar 1.000 mL de tampão de eletroforese neutro misturando 100 mM Tris base e 300mm de acetato de sódio para 1.000 mL dH 2 O. ajustar o pH a 9.0 com ácido acético glacial. Legal a 4 ° C, durante pelo menos 30 min antes do uso.
- Solução de precipitação de DNA (DPS)
- preparação de ações de acetato de amónio de 7,5 M 10 mL. Para 50 mL de solução de precipitação de DNA, mistura de acetato de amónio 6,7 mL 7,5 M com 43,3 mL de etanol 95%. Armazenar em temperatura ambiente.
- Solução de coloração
- Adicionar 1 µ l 10.000 x mancha verde fluorescente de ácidos nucleicos (por exemplo, SYBR Green) em tampão de 30 mL de Tris-EDTA (10 mM Tris-HCl, EDTA dissódico de 1 mM, pH 7,4) e loja em 4 ° C. proteger da luz.
- 1% baixa fusão agarose
- derreter 1% baixa fusão ponto agarose (1 g em 100 mL dH 2 O) no microondas. Agite a agarose cada 15-20 s para certificar-se que a agarose é completamente derretido. Coloque o agarose em banho de água a 37 ° C pelo menos 20 min antes do uso.
- Pontas de pipetas pre-quente
- cortar as extremidades estreitas de pontas de pipetas P200 por 3 mm e quente a 37 ° C antes de pipetagem agarose.
2. Preparar os Slides cometa
- , deslize o revestimento
- Melt agarose a 1% (1 g em 100 mL dH 2 O) no microondas durante 2-3 minutos ou até que a agarose é completamente derretido. Mergulhar as vidro corrediças do microscópio para a agarose e limpar um lado da lâmina utilizando um cotão.
- Colocar os slides sobre uma superfície plana para secar ao ar livre ou aqueça a 50 ° C para secagem mais rápida; um filme de agarose transparente deve ser formado após a secagem. Coloque as lâminas revestidas em 37 ° C antes do uso.
- Preparação de suspensões celulares único
- cultura e tratar as células de glioma
- cultura as células U251 MG em meio DMEM-Ham F-12 suplementado com 10% FBS, 100 U/mL penicilina e estreptomicina 10 de µ g/mL em 37 & # 176; C com 5% de CO 2.
- Digerir as células usando a tripsina 1 mL por 3 min e neutralizar a tripsina usando meio DMEM-Ham F-12 com FBS. Coletar em tubo de 15 mL, giram a 300 x g, durante 4 min, Aspire o meio e suspender as células em 2 x 10 5 células/mL de 1X PBS.
Nota: A amostra de célula deve ser preparada imediatamente antes de iniciar o ensaio e todas as amostras devem ser manuseadas em um ambiente escuro ou esmaecido para evitar danos no DNA da luz. - Combinar a suspensão de células com agarose a 1% derretido baixo ponto de fusão (a 37 ° C) na proporção de 01:10 (v/v), misture suavemente pipetando para cima e para baixo e imediatamente distribuir 30 µ l de uma lâmina. Use o lado da ponta da pipeta para espalhar a mistura para garantir a formação de uma camada fina de agarose/célula.
- Coloque a corrediça plana a 4 ° C, no escuro por 10 min., aumentando o tempo de coagulação para 30 min melhora a aderência das amostras em ambientes de alta umidade.
- Imergir o slide em 4 ° C LS no escuro por 1h durante a noite.
- cultura e tratar as células de glioma
3. Única célula eletroforese
- prosseguir com alcalino (passo 3.2) ou cometa neutro (passo 3.3) do ensaio
- para ensaio cometa alcalina
- gentilmente remover slides de LS, escorra o excesso reserva e delicadamente mergulhe na AES por 1h no 4 ° C para permitir o desenrolar de DNA. Manter os slides no escuro.
- Adicionar pre-refrigerados AES na bandeja de slide de electroforese, não deve exceder 0,5 cm acima os slides (isso depende do tamanho das unidades de eletroforese), coloque os slides dentro e cubra com uma tampa. Definir a tensão de alimentação para 1 V/cm (o comprimento entre os eletrodos) e executada por 30 min em 4 ° C.
- Solução de electroforese em excesso de dreno de slide. Mergulhe delicadamente slides duas vezes na dH 2 O por 5 min à temperatura ambiente.
- Delicadamente mergulhe slides em etanol a 70% por 5 min à temperatura ambiente. Avance para o passo 4.
- Para ensaio cometa neutro
- Remover suavemente os slides de LS, escorra o excesso tampão e delicadamente mergulhe no NES por 30 min em 4 ° ° C. conservar o slide no escuro. Tampão de eletroforese neutro previamente refrigerados
- Add na bandeja de slide de electroforese, não deve exceder 0,5 cm acima slides (isso depende do tamanho das unidades de eletroforese), coloque os slides dentro e cubra com uma tampa. Definir a tensão de alimentação para 1 V/cm (o comprimento entre os eletrodos) e executada por 45 min em 4 ° C.
- Buffer excesso de drenagem das lâminas de. Mergulhe delicadamente slides em DPS por 30 min à temperatura ambiente.
- Delicadamente mergulhe os slides em etanol a 70% durante 30 min à temperatura ambiente. Avance para o passo 4.
4. Mancha de Slides cometa
- seco slides a 37 ° C por 10-15 min no escuro.
- Lugar 50-100 µ l verde fluorescente ácido nucleico manchando a solução em cada secas agarose e mancha durante 15 minutos à temperatura ambiente no escuro.
- Lavar os slides brevemente em dH 2 O e secar completamente a 37 ° C, no escuro. Proceder à análise e aquisição de imagens.
5. Aquisição e análise de imagens
Nota: A visualização e a quantificação de quebras de DNA baseiam-se na microscopia de epifluorescência e o software do ensaio cometa (ver Tabela de materiais) 12 .
- Coloque os slides sobre o microscópio com um suporte de slide. Certifique-se que o gel de agarose é voltado para a lente objetiva. Aleatoriamente, capturam imagens de slides cometa manchada usando um microscópio de fluorescência com uma lente objetiva de 10x. Evitar as bordas e as áreas ao redor de quaisquer bolhas de ar.
- , Certifique-se de cada cauda de cometa é distribuída horizontalmente. Cabeças de cometa devem se originam da esquerda e a cauda da direita.
- Salvar cada imagem em um formato binário de TIF com mancha brilhante de DNA e fundo escuro. Carregar imagens para o software usando o " selecione arquivos para analisar " botão, que está localizado no lado esquerdo da barra de ferramentas. Uma janela de exibição de imagem deve aparecer ( Figura 1).
- Desenhar um quadro de medição na tela e ajustar seu tamanho em conformidade com o cometa da célula. Clique o " ajustar " o botão para configurar o limite da cabeça, cometa, cauda de acordo com a imagem e, em seguida, clique o " começar as medições " botão ( Figura 1).
- , Selecione uma célula usando o frame e ativar a medição clicando com o mouse sobre o " do ensaio cometa " botão; uma intensidade imagem aparece no " perfis " janela com os parâmetros de medição selecionado. Os resultados podem ser salvos clicando o " armazenar resultado " botão ( Figura 1).
Nota: O software calcula os parâmetros, incluindo o comprimento da cauda de cometa, a porcentagem de DNA cauda, o momento de cauda (TM) e o momento de cauda verde-oliva (OTM). Os momentos de cauda são calculados por fórmulas como segue:

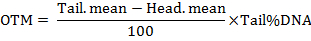
- analisar pelo menos 50 células por tratamento.
Resultados
O presente protocolo descreve um fluxo de trabalho passo a passo para a execução do ensaio cometa e análise de dados (Figura 1). Resultados de ensaios os cometa alcalino e neutro, mostrou que a cauda do cometa de células U251 tratados com doxorrubicina (1 µM, 20 h) era maior e tinha maior intensidade de DNA, sugerindo um acúmulo substancial de DNA fragmentado devido à quimioterapia (Figura 2).
Discussão
O ensaio cometa é uma ferramenta eficiente para medir intervalos de single e double-strand DNA a nível celular. O ensaio foi amplamente aplicado como um "padrão ouro" em estudos sobre a genotoxicidade e biomonitorização13, variando de ligações cruzadas base lesões, DNA, desenvolvimento de drogas e locais sensíveis do alcaloide. No presente estudo, mostramos dois protocolos distintos de passo a passo para ensaios de cometa alcalino e neutro, respectivamente. Combinando a electroforese úni...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Esta pesquisa foi apoiada pelo programa de pesquisa Intramural do NIH, ICN e CCR. Todos os autores receberam Intramural bolsa de pesquisa de NIH, ICN e CCR.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 10x PBS(Ca++, Mg++ free) | TEKnova | P0196 | |
| NaCl | Sigma | S5886 | |
| EDTA | TEKnova | E0308 | |
| Trizma base | Sigma | T1503 | |
| NaOH | Sigma | 72068 | |
| Sodium lauryl sarcosinate | Sigma | L7414 | |
| Triton X-100 | Sigma | 93443 | |
| Sodium acetate | Sigma | 32318 | |
| Glacial acetic acid | Sigma | 695092 | |
| Ammonium acetate | Sigma | A1542 | |
| SYBR Green | Invitrogen | S33102 | |
| Low melting point agarose | Invitrogen | 16520 | |
| Agarose | Invitrogen | 16500 | |
| 95% ethanol | WARNER-GRAHAM | #64-17-5 | |
| Trypsin | GIBICO | 25300-054 | |
| Name | Company | Catalog Number | Comments |
| Consumables | |||
| Glass tissue slides | ELECTRON MICROSCOPY SCIENCES | 63422-11 | |
| Kimwipes | KIMberly-Clark | ||
| 1.5 mL Microcentrifuge Tubes | DENVILLE | ||
| Pipette Tips | SHARP | ||
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| Microwave | Avanti | ||
| Waterbath | PRECISION | ||
| Horizontal electrophoresis chamber | TREVIGEN | Cometassay ES II | |
| Power supply | Bio-Rad | ||
| Incubator | Quincy Lab | Model 12-140E | |
| Fluorescent microscope | Zeiss | LSM700 | |
| Micropipettor | Eppendorf |
Referências
- Ostling, O., Johanson, K. J. Microelectrophoretic study of radiation-induced DNA damages in individual mammalian cells. Biochem Biophys Res Commun. 123 (1), 291-298 (1984).
- Singh, N. P., McCoy, M. T., Tice, R. R., Schneider, E. L. A simple technique for quantitation of low levels of DNA damage in individual cells. Exp Cell Res. 175 (1), 184-191 (1988).
- Tice, R. R., et al. Single cell gel/comet assay: guidelines for in vitro and in vivo genetic toxicology testing. Environ Mol Mutagen. 35 (3), 206-221 (2000).
- Shah, A. J., Lakkad, B. C., Rao, M. V. Genotoxicity in lead treated human lymphocytes evaluated by micronucleus and comet assays. Indian J Exp Biol. 54 (8), 502-508 (2016).
- Azqueta, A., Collins, A. R. The essential comet assay: a comprehensive guide to measuring DNA damage and repair. Arch Toxicol. 87 (6), 949-968 (2013).
- Goldstein, M., Kastan, M. B. The DNA damage response: implications for tumor responses to radiation and chemotherapy. Annu Rev Med. 66, 129-143 (2015).
- Gavande, N. S., et al. DNA repair targeted therapy: The past or future of cancer treatment?. Pharmacol Ther. 160, 65-83 (2016).
- Torgovnick, A., Schumacher, B. DNA repair mechanisms in cancer development and therapy. Front Genet. 6, 157 (2015).
- Weston, V. J., et al. The PARP inhibitor olaparib induces significant killing of ATM-deficient lymphoid tumor cells in vitro and in vivo. Blood. 116 (22), 4578-4587 (2010).
- Brown, J. S., O'Carrigan, B., Jackson, S. P., Yap, T. A. Targeting DNA Repair in Cancer: Beyond PARP Inhibitors. Cancer Discov. 7 (1), 20-37 (2017).
- Lu, Y., et al. Chemosensitivity of IDH1-Mutated Gliomas Due to an Impairment in PARP1-Mediated DNA Repair. Cancer Res. 77 (7), 1709-1718 (2017).
- Konca, K., et al. A cross-platform public domain PC image-analysis program for the comet assay. Mutat Res. 534 (1-2), 15-20 (2003).
- Valverde, M., Rojas, E. Environmental and occupational biomonitoring using the Comet assay. Mutat Res. 681 (1), 93-109 (2009).
- Collins, A. R. The comet assay for DNA damage and repair: principles, applications, and limitations. Mol Biotechnol. 26 (3), 249-261 (2004).
- Karbaschi, M., Cooke, M. S. Novel method for the high-throughput processing of slides for the comet assay. Sci Rep. 4, 7200 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados