É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Aplicação da microscopia de velocidade de alta velocidade super-resolução no cílio primário ao vivo
Neste Artigo
Resumo
Recentemente nós mapeamos locais espaciais tridimensionais (3D) de rotas de transporte para várias proteínas se dentro cílios primários em células vivas. Aqui este detalhes de papel a instalação experimental, o processo de amostras biológicas e as análises de dados para a fluorescência de super-resolução 3D imagem abordagem recém aplicadas em viver cílios primários.
Resumo
O cílio primário é uma protrusão do microtubule-baseado na superfície de muitas células eucarióticas e contém um único complemento de proteínas que funcionam criticamente em motilidade celular e sinalização. Desde que cílios são incapazes de sintetizar suas próprias proteínas, cerca de 200 proteínas ciliares originais precisam ser traficadas entre o citosol e cílios primários. No entanto, ainda é um desafio técnico para mapear tridimensional (3D) locais, das vias de transporte para estas proteínas em cílios primários ao vivo devido às limitações do atualmente existente técnicas. Para conquistar o desafio, recentemente desenvolvemos e empregou uma microscopia 3D de alta velocidade virtual Super-resolução, denominada microscopia difração sub (velocidade) de single-point da borda-excitação, para determinar a localização espacial 3D das vias de transporte para ambos citosólico e proteínas de membrana em cílios primários de células vivas. Neste artigo, vamos demonstrar a configuração detalhada da microscopia de velocidade, a preparação de celulas que expressam proteínas ciliares fluorescência-proteína-labeled, o acompanhamento em tempo real do único-molécula de proteínas individuais no cílio ao vivo e a realização de densidade de probabilidade espacial 3D mapas de rotas de transporte para proteínas ciliares.
Introdução
Como afirmado por Ernst Abbe em 1873, a resolução da microscopia de luz convencional foi crida para ser limitada a aproximadamente 200 nm devido à difração de luz do objectivo1,2. Atualmente, as técnicas de microscopia de luz de super-resolução quebrar essa limitação e permitam a captura de imagens dinâmicas, com resolução de difração sub (< 200 nm). As técnicas geralmente caem em duas categorias amplas: estimulada abordagens de microscopia com base de depleção (STED) emissão, que geram o volume de iluminação sub difração devido à resposta óptica não-linear de fluorophores em amostras3; e (palma), microscopia de luz fotoativo e microscopia de reconstrução óptica estocástico (Tempestade)-com base em técnicas de super-resolução, que utilizam funções matemáticas para localizar os centroides de fluorophores e então reconstituir essas centroides para formar imagens de super-resolução4,5. Atualmente, devido a configuração óptica relativamente uncomplicated, PALM e tempestade são extensivamente empregados ativando apenas um pequeno subconjunto de fluorophores em cada quadro de um vídeo longo de uma preparação biológica. Isto permite a localização mais exata por 2D montagem Gaussian da mancha fluorescente, denominado a função de ponto de espalhar (PSF), das proteínas fluorescente-etiquetadas em cada quadro do vídeo. A localização 2D de cada molécula fluorescente-etiquetadas então pode ser sobreposta a um único avião de imagens para produzir uma imagem de super-resolução da preparação biológica1,2. Enquanto estes localização único-molécula, abordagens de super-resolução para microscopia certamente revolucionaram como imagem latente de amostras biológicas foi executada, ainda existem desafios a serem vencidos. Por exemplo, tempestade e PALM podem alcançar seus melhores resoluções espaciais após a fixação de amostras biológicas e, portanto, apresentar uma representação estática das proteínas fluorescente-etiquetadas, que é uma limitação similar de microscopia eletrônica. Além disso, para atingir a alta resolução espacial para cada proteína fluorescente-etiquetadas em células vivas, amostras devem ser fotografadas no framerates há muito tempo que são incapazes de capturar a dinâmica da proteína. Portanto, é necessário superar estes obstáculos técnicos principais.
Para obter uma alta resolução spatiotemporal que é adequada para a detecção de proteínas ou RNAs de movimento rápido em células vivas, nós desenvolvemos microscopia de super-resolução velocidade em nosso laboratório (Figura 1)6,7, 8. vários grandes avanços técnicos na microscopia de velocidade anteriormente permitiram-nos controlar com sucesso nucleocytoplasmic transporte de pequenas moléculas, proteínas, mRNA e vírus através de nativo nuclear pore complexos (NPCs)6, 7 , 8. resumidamente, os seguintes recursos da microscopia de velocidade serão usados para rastrear velozes macromoléculas através das estruturas simétrica e rotacionalmente sub-micrônicas em células vivas, tais como NPCs e cílios primários: (1) um inclinado ou uma iluminação vertical PSF permite a excitação de moléculas simples dentro de um volume pequeno de difração-limite no plano focal (Figura 1); (2), o PSF inclinado pode evitar muito fora de foco da fluorescência e, assim, melhorar a relação sinal-ruído. (3), a densidade óptica de 100-500 kW / cm2 no PSF iluminação permite que milhares de fótons para ser coletado de único fluorophores com velocidades de deteção rápida (> 500 Hz). Velocidade de deteção (4), o jejum também reduz o erro de localização espacial único-molécula (< 10 nm) ao determinar as trajetórias espaciais de mover moléculas fluorescentes em células vivas, porque a difusão molecular é um dos principais fatores fazendo com que as imperfeições da único-molécula de localização para a movimentação de moléculas. Algoritmos de transformação (5) bem estabelecidas 2D para 3D permitem-nos fornecer a densidade de probabilidade espacial 3D mapas de rotas de transporte para moléculas no NPC ou o cílio primário. Vale ressaltar que nosso processo de conversão entre o cartesiano e o sistema de coordenação cilíndrico é usado para gerar uma densidade de probabilidade espacial 3D mapa ao invés de 3D único-molécula rastreamento (Figura 2). Anteriormente, os dados de microscopia eletrônica revelaram que o NPC9,10 e o cílio primário11 ambos têm uma estrutura simétrica e rotacionalmente. Em princípio, aleatoriamente, difundir moléculas movendo-se através do NPC ou cílio primário também deve ter distribuições simétricas e rotacionalmente. Como mostrado na Figura 2, um número elevado de difusão aleatoriamente as moléculas no interior do cilindro geraria distribuições simétricas e rotacionalmente com a vista de seção transversal como que em NPC, ainda mais, resultando em um aproximadamente uniforme espacial distribuição dentro de cada sub-região muito pequena entre dois anéis vizinhos(Figura 2). Esta distribuição uniforme leva a que a distribuição espacial ao longo da dimensão θ no sistema cilíndrico é constante. Então as coordenadas 3D (X, R, θ) podem ser simplificadas para ser as coordenadas 2D (R, X, constante). Na verdade, nosso processo de conversão entre os cartesianos e os sistemas cilíndricos é de 2D (X, Y) a 2D (R, X, constante). A constante θ, refere-se a densidade espacial p na Figura 2,E, é calculada utilizando a equação A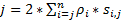 .
.
Em última análise, rastreamento único-molécula tem ampla aplicação em pesquisa biológica, portanto, é natural que uma infinidade de técnicas será desenvolvida para preencher nichos biológicos específicos12,13,14. Tal é o caso com microscopia de velocidade. Anteriormente, quando combinada com um algoritmo de transformação 3D, esta técnica foi desenvolvida para resolver as rotas de transporte 3D de trânsito moléculas através os NPCs, uma estrutura biológica rotacionalmente simétrica e diffraction subtamanho6. Neste trabalho, cílios primários são mostrados para ser organelas excelente modelo também. Cílios primários são organelas cilíndricas, como antena (raio de ~ 125 nm) que o projeto da superfície dos mamíferos mais células15,16,17. Eles são responsáveis por receber os sinais externos e transmitir uma resposta intracelular, geralmente associada com crescimento e metabolismo de15,16. Portanto, fluxo de proteínas estruturais, reciclagem de receptores transmembranares e transmissão dos mensageiros intracelulares são responsabilidades vitais dos cílios primários. Na junção entre os cílios primários e o corpo da célula é uma barreira de seletividade crítica, chamada de zona de transição ou TZ, através do qual todo o transporte desta proteína deve ocorrer11,18,19, 20. Além da função associada do TZ, pelo menos, dois processos de transporte, do transporte e difusão passiva, são pensados para ser responsável pelo movimento da proteína através desta região16,21, 22. do ponto de vista da saúde humana, a perda de cílios primários e desregulamentação subsequente de sinalização a jusante são característica de muitos cancros. Além disso, muitas doenças genéticas, como síndrome de Bardet-Biedl e doença renal policística, estão associadas a proteína defeituosa transporte23. Tanto o tamanho do limite de difração sub e o complexo processo de transporte seletivo de proteína através do TZ fazem os cílios primários um alvo principal para esta técnica. Neste trabalho de métodos, demonstraremos o rastreamento de uma proteína transmembrana ciliar, receptor de somatostatina 3 (SSTR3)24, rotulado externamente com Alexa Fluor 647 e um componente do IFT, IFT2025, rotulado com uma molécula GFP fundida.
Protocolo
1. preparação da pilha NIH-3T3 para microscopia de velocidade do estoque
- 1,5 semanas antes do experimento, recuperar uma cultura fresca de células NIH-3T3 de um estoque congelado descongelar a 37 ° C e transferindo as células para um balão de cultura de células de2 25 cm com 3 mL de modificado águia de Dulbecco médio (DMEM) suplementado com 110 mg/mL piruvato de sódio, glutamina 2 mM, 10% de soro fetal bovino e 1% penicilina/estreptomicina.
- Incube a 37 ° C numa incubadora 5% CO2 células.
- Dividi células na confluência de 80%, de dois em dois dias, pelo menos três vezes antes do dia experimental para garantir a homogeneidade do ciclo celular. Trypsinize células com tripsina 0,25% por 2 min a 37 ° C, Aspire tripsina e substituí-lo com 2 mL do meio. Pipeta repetidamente a médio e romper a conjuntos de células, remova o número desejado de células e trazer o volume total dos meios de comunicação de volta até 3 mL.
Nota: NIH-3T3 foram anteriormente alterado geneticamente para expressar NPHP-4, uma proteína que localiza o TZ26, fundidos no terminal C para mCherry. mCherry é um fluoróforo que pode ser animado com 561 iluminação nm para localizar quantitativamente a barreira de seletividade TZ e orientar os cílios primários. - Dois dias antes do experimento, placa as células em um fundo de vidro 35mm prato na confluência de 60-70% com 1,5 mL do mesmo meio de como passo 1.1 e retornam as células para a incubadora.
- Um dia antes do experimento, transfect quimicamente as células com o plasmídeo desejado. Misturar 500-1000 ng do plasmídeo desejado (veja a Nota abaixo) na proporção de 1: 2.5 com reagente de transfeccao em 0,25 mL de mídia reduzida soro sem antibióticos durante 30 min. aspirado de mídia de 35 milímetros de vidro prato fundo e substituí-lo com o plasmídeo de 0,25 mL / mistura de reagente de transfeccao mais um extra 1,25 mL de mídia reduzida soro sem antibióticos. Mídia reduzida soro serve ao propósito de facilitar um bem sucedida do transfection, induzindo o crescimento de cílios primário, bem como manter as células viva tempo suficiente para realizar o experimento. Retorno as células para a incubadora para o experimento no dia seguinte.
Nota: Ao executar rastreamento único-molécula de IFT20, um plasmídeo que contém que um IFT20 geneticamente modificados fundidas no seu terminal C a GFP é usado25. Ao executar o único-molécula de rastreamento de SSTR3, um plasmídeo que contém um SSTR3 geneticamente modificados fundidas no seu término de N para um domínio de peptídeo (AP) aceitador e C terminal a GFP é usado22. Além de construção de SSTR3, um plasmídeo contendo o ligase biotina BirA deve ser expressa co e os meios de comunicação de transfeccao devem ser suplementados com 10 biotina µM. BirA, em seguida, anexa biotina ao domínio AP de moléculas de AP-SSTR3-GFP recém sintetizados ao nível do PS. Alexa647 conjugada com três dos quatro sites de ligação de biotina na estreptavidina, em média, então podem ser completadas para a mídia antes da imagem latente para etiqueta fluorescente as moléculas de AP-SSTR3-GFP na superfície exterior do celular22,27 . GFP e AlexaFluor647 são utilizados neste método; no entanto, outras sondas fluorescentes podem ser usadas se eles têm da mesma forma, foto-estabilidade elevada e quântica rendimento. - Se usando a construção de SSTR3 marcado externamente, remover a mídia da parte inferior de vidro prato 1 h antes do experimento, lave a célula 5 vezes com 1 mL de tampão fosfato salino (PBS) e adiciona 1 mL de mídia reduzida soro suplementado com 1 µM Alexa647 conjugados streptavidin.
- Não mais que 15 min antes do experimento, remova a mídia do prato fundo vidro e lavar as células transfectadas e etiquetadas 5 vezes com 1 mL de PBS.
- Coloque 1 mL de tampão de imagem (20 mM HEPES, 110mm KOAc, 5mm NaOAc, 2mm MgOAc, 1mm EGTA, pH 7,3) no prato fundo vidro.
Nota: No buffer de imagem, as células são viáveis para não mais do que 3 h. Portanto, apenas a 2 h de experimentos são executadas em cada prato.
2. microscopia de velocidade
Nota: A configuração de microscopia de velocidade inclui um microscópio de fluorescência invertido equipado com um objectivo apocromático de 1,4-at 100 × óleo-imersão, 35 mW 633 nm He-Ne laser, 50 mW estado sólido 488 nm e 561-nm lasers, um ganho de multiplicação em-microplaqueta câmera acoplado-dispositivo de carga e um pacote de software do microscópio para aquisição de dados e processamento (Figura 1). Para a imagem latente de canal individual, GFP, mCherry e Alexa647 estão animado por 488 nm, 561 nm ou lasers de 633 nm, respectivamente. Para rastreamento de molécula, único ponto de iluminação é usada para controlar as moléculas individuais fluorescente-etiquetadas. Para a imagem latente de epifluorescência, uma lente côncava é colocado no caminho da iluminação de laser para expandir o feixe em um campo uniforme da iluminação. A emissão de fluorescência é coletado pelo mesmo objectivo, filtrado por um filtro dicroico (405/488/561/635) e um filtro de emissão (405/488/561/635) e fotografado com a câmera do CCD acima operando a 500 Hz para rastreamento de molécula ou 2 Hz para imagem de epifluorescência.
- Fixe a placa de fundo de vidro para o palco do microscópio e localize uma célula que corretamente expressa as construções desejadas. Uma vez que foi encontrado um celular adequada, alinhe o ponto de NPHP4-mCherry na base dos cílios primários com a localização no plano de imagem que corresponde à iluminação de único ponto do laser.
- Capturar uma imagem de epifluorescência de NPHP4-mCherry e IFT20-GFP ou AP-SSTR3-GFP usando a função de "Snap" na aba "Câmera" da janela "Controles de foco", se usando o pacote de software de microscopia digital (ver Tabela de materiais).
Nota: Estas imagens atuará como uma referência para os locais da molécula subsequentes. - Uma vez que as imagens de referência são obtidas, localmente reduza a concentração de moléculas simples rotuladas. Foto-descora o TZ com 1 iluminação de laser mW por 20 s ou até que a intensidade da fluorescência é próximo de fluorescência de fundo.
Nota: Quando a concentração exacta pode ser controlada, 0,1-1 nM rotulado moléculas simples são usados. - Para preparar para rastreamento de molécula, reduza a potência de iluminação do laser de ~0.15 mW para moléculas simples rotulado com GFP ou ~0.5 mW para moléculas rotulado com Alexa647.
- Assim que a potência do laser e parâmetros de imagem são conjunto, ganho máximo e intensificação e taxa de quadros de 2 ms, para a imagem latente de molécula, envolver-se o laser de iluminação adequada e registrar não-foto, rotulado de moléculas simples como eles são transportados através da região de foto do TZ clicando no botão de "Stream" na aba "Câmera" da janela "Controles de foco".
Nota: Não mais de 2 min de vídeo deve ser capturada para minimizar os efeitos da deriva ciliar para um nível insignificante. - Depois de capturar o vídeo única molécula, processe os vídeos usando um algoritmo de ajuste Gaussian 2D, tais como vislumbre pelo laboratório Gelles, que localiza precisamente do centroide de excitação do cada molécula PSF em uma abrangente área de interesse (AOI).
- Selecione todos os locais da molécula com precisão < 10 nm e corrigir o centro de cílios com base na distribuição da molécula locais equipados com uma função gaussiana 2D.
Nota: Usando 2D para o algoritmo de transformação 3D, as rotas de transporte 3D de rotas IFT20-GFP e AP-SSTR3 são claramente mostradas na membrana ciliar ou axonemal ciliar, respectivamente.
3. 2D para 3D transformação
- Uma vez que várias mil localizações para o trânsito de moléculas (sinal-ruído ratio > 11) no cílio são coletadas, selecione eixo longo do cílio como a X-dimensão. Faça um histograma de dimensão Y dos locais e obter as somas bin em incrementos de 10 nm.
Nota: A 2D para 3D transformação pode ser avaliada pela mão ou qualquer software ou linguagem de programação. Os autores têm implementado com sucesso a transformação em Matlab e Python 2.7.
Resultados
Esta seção demonstra os dados obtidos da realização de microscopia de velocidade para o TZ de cílios primários para estudar a rota de transporte de SSTR3 ligado por um vinculador externo de nm ~ 15 de Alexa647(Figura 3). Serve o duplo objectivo de verificar o algoritmo de transformação 3D. Alexa647 deve apenas rótulo a superfície externa do cílio primário e, portanto, a rota de transporte 3D deve revelar uma rota de transporte de ...
Discussão
Este protocolo descreve a aplicação da microscopia de velocidade para o cílio primário, uma organela celular de sinalização que é altamente dependente do transporte eficiente de proteínas. Microscopia de velocidade pode fornecer alta resolução locais (< 10 nm) para moléculas fluorescente-etiquetadas como passam através da iluminação de ponto único centralizada sobre o TZ. Anteriormente tiver sido aplicado para estudar a proteína tráfico através do NPC6,
Divulgações
Os autores declaram não há conflitos de interesse.
Agradecimentos
Agradecemos o Dr. Kristen Verhey (Universidade de Michigan, Ann Arbor) e Dr. Gregory Pazour (Universidade de Massachusetts Medical School) para fornecer alguns plasmídeos. O projeto foi apoiado por concessões do National Institutes of Health (NIH GM097037, GM116204 e GM122552 para W.Y.).
Materiais
| Name | Company | Catalog Number | Comments |
| 25 cm2 tissue culture dish | Corning | VV-01936-00 | |
| Penicillin/streptomycin | ThermoFisher | 15140122 | |
| Fetal bovine serum | ThermoFisher | 10438018 | |
| DMEM | ThermoFisher | 10566-016 | |
| OPTIMEM | ThermoFisher | 31985062 | |
| Trypsin | ThermoFisher | 25300054 | |
| Phosphate buffered saline | Sigma-Aldrich | P3813-1PAK | |
| Transit LT1 | Mirus | MIR 2300 | |
| 35 mm glass bottom dish | MatTek | P35GCOL-0-14-C | |
| AlexaFluor 647-conjugated streptavidin | ThermoFisher | S21374 | |
| Biotin | Sigma-Aldrich | B4501-100MG | |
| 633 nm He-Ne laser | Melles Griot | 25-LHP-928-249 | |
| 561 nm solid state laser | Coherent | OBIS 561-50 LS | |
| 488 nm solid state laser | Coherent | 1185053 | |
| Inverted fluorescence microscope | Olympus | IX81 | |
| 1.4-NA 100× oil-immersion apochromatic objective | Olympus | UPLSAPO 100× | |
| On-chip multiplication gain charge-coupled-device camera | Roper Scientific | Cascade 128+ | |
| Dichroic filter | Semrock | Di01- R405/488/561/635-25x36 | |
| Emission filter | Semrock | NF01-405/488/561/635-25X5.0 | |
| Slidebook 6.0 | Intelligent Imaging Innovations | digital microscopy software |
Referências
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annu Rev Biochem. 78, 993-1016 (2009).
- Leung, B. O., Chou, K. C. Review of super-resolution fluorescence microscopy for biology. Appl Spectrosc. 65, 967-980 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440, 935-939 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3, 793-796 (2006).
- Ma, J., Yang, W. Three-dimensional distribution of transient interactions in the nuclear pore complex obtained from single-molecule snapshots. Proc Natl Acad Sci USA. 107, 7305-7310 (2010).
- Ma, J., Goryaynov, A., Sarma, A., Yang, W. Self-regulated viscous channel in the nuclear pore complex. Proc Natl Acad Sci USA. 109, 7326-7331 (2012).
- Ma, J., et al. High-resolution three-dimensional mapping of mRNA export through the nuclear pore. Nat Comm. 4, (2013).
- Akey, C. W., Radermacher, M. Architecture of the Xenopus nuclear pore complex revealed by three-dimensional cryo-electron microscopy. J Cell Biol. 122, 1-19 (1993).
- Akey, C. W. Interactions and structure of the nuclear pore complex revealed by cryo-electron microscopy. J Cell Biol. 109, 955-970 (1989).
- Czarnecki, P. G., Shah, J. V. The ciliary transition zone: from morphology and molecules to medicine. Trends Cell Biol. 22, 201-210 (2012).
- Elf, J., Li, G. -. W., Xie, X. S. Probing transcription factor dynamics at the single-molecule level in a living cell. Science. 316, 1191-1194 (2007).
- Anzalone, A., Annibale, P., Gratton, E. 3D orbital tracking in a modified two-photon microscope: an application to the tracking of intracellular vesicles. J Vis Exp. , (2014).
- Ritter, J. G., Veith, R., Veenendaal, A., Siebrasse, J. P., Kubitscheck, U. Light sheet microscopy for single molecule tracking in living tissue. PloS one. 5, 11639 (2010).
- Marshall, W. F., Nonaka, S. Cilia: tuning in to the cell's antenna. Curr Biol. 16, 604-614 (2006).
- Scholey, J. M., Anderson, K. V. Intraflagellar transport and cilium-based signaling. Cell. 125, 439-442 (2006).
- Yang, T. T., et al. Superresolution pattern recognition reveals the architectural map of the ciliary transition zone. Sci Rep. 5, 14096 (2015).
- Craige, B., et al. CEP290 tethers flagellar transition zone microtubules to the membrane and regulates flagellar protein content. J Cell Biol. 190, 927-940 (2010).
- Kee, H. L., et al. A size-exclusion permeability barrier and nucleoporins characterize a ciliary pore complex that regulates transport into cilia. Nat Cell Biol. 14, 431-437 (2012).
- Najafi, M., Maza, N. A., Calvert, P. D. Steric volume exclusion sets soluble protein concentrations in photoreceptor sensory cilia. Proc Natl Acad Sci USA. 109, 203-208 (2012).
- Nachury, M. V., Seeley, E. S., Jin, H. Trafficking to the ciliary membrane: how to get across the periciliary diffusion barrier. Annu Rev Cell Dev Biol. 26, 59-87 (2010).
- Ye, F., et al. Single molecule imaging reveals a major role for diffusion in the exploration of ciliary space by signaling receptors. Elife. 2, 00654 (2013).
- Ross, A. J., et al. Disruption of Bardet-Biedl syndrome ciliary proteins perturbs planar cell polarity in vertebrates. Nat Genetics. 37, 1135-1140 (2005).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. Neuroscience. 89, 909-926 (1999).
- Follit, J. A., Tuft, R. A., Fogarty, K. E., Pazour, G. J. The intraflagellar transport protein IFT20 is associated with the Golgi complex and is required for cilia assembly. Mol Biol Cell. 17, 3781-3792 (2006).
- Awata, J., et al. NPHP4 controls ciliary trafficking of membrane proteins and large soluble proteins at the transition zone. J Cell Sci. 127, 4714-4727 (2014).
- Howarth, M., Ting, A. Y. Imaging proteins in live mammalian cells with biotin ligase and monovalent streptavidin. Nat Protoc. 3, 534-545 (2008).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy. Science. 319, 810-813 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados