É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Replicação do requisitado, biblioteca não redundante de estirpe Pseudomonas aeruginosa PA14 Transposon mutantes de inserção
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Pseudomonas aeruginosa infecção causa morbidade significativa em hosts vulneráveis. A biblioteca de mutantes de inserção transposon não redundante da estirpe de p. aeruginosa PA14, designado como PA14NR definida, facilita a análise da funcionalidade do gene em inúmeros processos. Apresentado aqui é um protocolo para gerar cópias de alta qualidade da biblioteca mutante PA14NR definido.
Resumo
Pseudomonas aeruginosa é uma bactéria Gram-negativa fenotipicamente e Leishmania diversificada e adaptável onipresente em ambientes humanos. P. aeruginosa é capaz de formar biofilmes, desenvolver resistência aos antibióticos, produzem fatores de virulência e evoluir rapidamente no decurso de uma infecção crônica. Assim, p. aeruginosa pode causar tanto aguda e crônica, difícil de tratar infecções, resultando em morbidade significativa em certas populações de pacientes. Estirpe de p. aeruginosa PA14 é um humano isolado clínico com uma estrutura de genoma conservada que infecta uma variedade de hospedeiros mamíferos e nonvertebrate fazendo PA14 uma estirpe atraente para estudar este patógeno. Em 2006, uma biblioteca de mutantes de inserção do transposon não redundante contendo 5.459 mutantes correspondente previsto 4.596 PA14 genes foi gerada. Desde então, a distribuição da biblioteca PA14 permitiu a comunidade de pesquisa compreender melhor a função de genes individuais e complexos caminhos de p. aeruginosa. Manutenção da integridade da biblioteca através do processo de replicação requer técnicas adequado manuseio e precisas. Para o efeito, este manuscrito apresenta protocolos que descrevem detalhadamente as etapas envolvidas em replicação de biblioteca, biblioteca de controle de qualidade e armazenamento adequado dos mutantes individuais.
Introdução
Pseudomonas aeruginosa é uma bactéria Gram-negativa fenotipicamente e Leishmania diversificada e adaptável presente no solo, água e ambientes mais humanos, bem como a microflora da pele. Comparado a muitas espécies bacterianas, p. aeruginosa tem um genoma relativamente grande de 5.5-7 Mbp com conteúdo de alto G + C (% de 65-67). Além disso, uma parte significativa dos seus genes estão envolvida na adaptabilidade metabólica e fazem parte de redes de regulação, permitindo uma grande flexibilidade em resposta ao estresse ambiental1. P. aeruginosa expressa uma infinidade de fatores de virulência, apresenta tendência para a forma de biofilmes, possui a habilidade de coordenar as respostas através do quórum de múltiplos caminhos de detecção e exibe uma notável capacidade de desenvolver resistência aos antibióticos e tolerância de2,3,4,5,6,7,8. Esses atributos apresentam desafios significativos no tratamento de infecções causadas por p. aeruginosa.
Crônica infecções de p. aeruginosa podem ocorrer em vários Estados da doença. Fibrose cística (CF), uma doença genética causada pela mutação do gene da Fibrose cística regulador de condutância transmembrana (CFTR) , resulta em secreções infectadas, inspissated dentro das vias aéreas, bronquiectasias progressiva e, finalmente, morte de insuficiência respiratória9. Pela idade adulta, a maioria dos pacientes com FC é cronicamente infectada com p. aeruginosa, que desempenha um papel fundamental na morbilidade e mortalidade associadas a esta doença10. Além disso, pacientes com lesões de queimadura grave11, traqueostomia12, as artroplastias13ou permanência de cateteres14 correm o risco de infecção p. aeruginosa , relacionada com a capacidade das bactérias para formar biofilmes e fuga de acolhimento respostas inflamatórias15. Além disso, colonização ocorre sem concorrência depois uma população tolerante ou resistente aos antibiótico multi é selecionada através do tratamento antimicrobiano de largo espectro, sequencial12,16,17 , 18. melhor compreensão da patogênese de p. aeruginosa terá implicações significativas para inúmeros Estados de doença.
Vários isolados clínicos de p. aeruginosa , incluindo cepas PAO1, PA103, PA14 e PAK, têm sido muito estudados para investigar diferentes características de p. aeruginosa patogênese. Estirpe PA14 é uma clínica isolada que pertence a um dos grupos clonal mais comum no mundo19,20 e não tem sido extensivamente passada no laboratório. PA14is altamente virulento em vertebrados modelos de infecção, com uma notável endotoxina perfil21, pili estrutura22, patogenicidade dos consoles23, tipo sistema do secretion de III (TTSS), citotoxicidade para mamíferos células24 e perfis em antibióticos de resistência e persistência25. Além disso, PA14 também é altamente virulento em numerosos sistemas de modelo do patógeno-hospedeiro, incluindo folha de planta infiltração modelos26,27, infecçãoCaenorhabditis elegans modelos28, 29, inseto modelos30,31, bem como pneumonia rato modelos32,33 e34modelos de queimadura da pele.
Bibliotecas de todo o genoma mutantes são coleções de mutantes isogénicas nos genes não essenciais que constituem ferramentas muito poderosas para compreender a biologia de um organismo, permitindo que a análise da função do gene em escala genômica. Dois perto de-saturação transposon inserção mutante bibliotecas construídas em p. aeruginosa estão atualmente disponíveis para distribuição. Os locais de inserção dos transposões foram determinados para ambas as bibliotecas. Essas bibliotecas não redundante chamadas facilitam estudos de todo o genoma de cepas bacterianas, diminuindo consideravelmente o tempo e custo envolvidos na triagem descaracterizada mutantes transposon aleatório. P. aeruginosa PAO1 transposon mutant biblioteca, construída no MPAO1 isolar de estirpe PAO1 usando transposões ISphoA/ ha e élacZ/ ha35, é comissariada pelo laboratório Manoil, Universidade de Washington. A biblioteca consiste em uma coleção de 9.437 mutantes transposon sequência-verificado que oferece cobertura ampla do genoma e inclui dois mutantes para a maioria dos genes36. Informações sobre a p. aeruginosa PAO1 transposon mutant biblioteca estão disponíveis para o site do laboratório Manoil público, acessível pela internet em http://www.gs.washington.edu/labs/manoil/libraryindex.htm. A p. aeruginosa estirpe PA14 não redundante transposon inserção mutante biblioteca (conjunto PA14NR) construída em tensão PA14 usando transposões MAR2xT7 e TnphoA37 atualmente é distribuída pelo departamento de Pediatria hospital geral de Massachusetts. O conjunto PA14NR é composto por uma coleção de mais de 5.800 mutantes com inserções de transposon único em genes não essenciais37. Detalhes sobre a construção do Set PA14NR são descritos no http://pa14.mgh.harvard.edu/cgi-bin/pa14/home.cgi?section=NR_LIB local público, acessível pela internet, que também contém uma variedade de ferramentas de busca on-line para facilitar o uso da PA14NR Conjunto.
O conjunto original de PA14NR composta por 5.459 mutantes, selecionados a partir de uma biblioteca abrangente de aproximadamente 34.000 transposon aleatório inserção mutantes, que correspondem a 4.596 genes PA14 previstos, representando 77% do PA14 previu todos os genes37. Desde a construção da biblioteca em 2006 foram adicionados novos mutantes, e atualmente o conjunto de PA14NR inclui mais de 5.800 mutantes38 que representam aproximadamente 4.600 PA14 genes. A maioria dos mutantes transposon PA14 foram gerada no selvagem tipo fundo37. Pormenores relativos a cada membro da biblioteca mutante, incluindo a base genética, estão disponíveis através de pesquisa de banco de dados on-line ou baixando a planilha de biblioteca não redundante, ambos os recursos disponíveis no site PA14 (http:// PA14.MGH.Harvard.edu/cgi-bin/PA14/Home.cgi). A maioria dos mutantes foram criada usando o MAR2xT7 transposon (MrT7), com um pequeno conjunto criado usando o TnPhoA (phoA) transposon37. Cada transposon tem uma gaveta de resistência aos antibióticos, o que permite a seleção de mutantes usando gentamicina (MrT7) ou canamicina (phoA). O conjunto de PA14NR de mutantes é armazenado em sessenta e três placas de 96 poços e inclui controle de 96 poços adicionais duas placas, que consistem de tipo selvagem PA14 inoculadas e não inoculados poços intercalados em um padrão predefinido. O formato da placa de 96 poços emparelhado com as ferramentas de busca on-line muito facilita o desenvolvimento personalizado de ensaios que permitem aos usuários facilmente identificar genes associados com fenótipos mutantes de triagem. As ferramentas de busca on-line também facilitam a busca e seleção de mutantes de relevantes adicionais necessários para estudos adicionais.
As bibliotecas de mutante de transposon PA14 e PAO1 são muito importantes recursos globais para a comunidade científica, e eles se complementam na validação da função dos genes desconhecidos e os caminhos deste patógeno bacteriano. Coincidentemente, desde a construção das bibliotecas de mutação transposon PAO1 e PA14, análise de sequenciamento de DNA completo-genoma dos muitos isolados de p. aeruginosa mostrou que PAO1 e PA14 pertencem a diferentes subclados principais do P. aeruginosa 7,de filogenia39,40,41. Porque isolados clínicos de p. aeruginosa são encontrados distribuídos em toda a filogenia, o fato de que PAO1 e PA14 pertencem a diferentes p. aeruginosa subgrupos aumenta o valor das duas bibliotecas de mutação transposon para comparativo estudos.
Publicações descrevendo a construção e seleção de bactérias mutantes bibliotecas, incluindo p. aeruginosa bibliotecas35,,37,42, estão prontamente disponíveis na literatura. No entanto, o melhor de nosso conhecimento, não há protocolos publicados descrevendo os procedimentos detalhados e técnicas usadas para replicação, manutenção e validação das bactérias mutantes bibliotecas estão disponíveis.
A metodologia descrita nesta publicação descreve um conjunto de três protocolos que facilitam o uso e manutenção do PA14NR Set. O primeiro protocolo descreve a replicação da biblioteca como recomendado aos destinatários da PA14NR Set. O segundo protocolo inclui diretrizes para estrias, crescendo e armazenar individuais mutantes identificados usando o conjunto de PA14NR. O terceiro protocolo descreve técnicas de controle de qualidade, incluindo a amplificação por PCR de fragmentos de mutantes transposon e posterior sequenciamento para confirmar identidade mutante. Este conjunto de protocolos também pode ser adaptado para a replicação e a manutenção de outras bibliotecas mutantes bacterianas ou coleções. A replicação de bactérias mutantes bibliotecas ou coleções é altamente recomendada para preservar a integridade da cópia do"mestre" (cópia original recebida). Replicação de várias cópias do conjunto de PA14NR para uso em laboratório de rotina minimiza a probabilidade de contaminação interwell da cópia mestre.
Protocolo
Atenção: Utilize padrão BSL-2 medidas de segurança ao manusear a p. aeruginosa, um patógeno humano. Se você é um indivíduo imunocomprometido ou tem qualquer condição médica que aumenta sua susceptibilidade à infecção bacteriana, tome cuidado especial ao trabalhar com P. aeruginosa. Consulte o escritório de biossegurança na sua instituição e obter a aprovação do seu médico antes de trabalhar com o o PA14 NR definido ou bibliotecas mutantes de patógenos bacterianos.

Figura 1: visão geral da replicação i: protocolo do PA14NR definido. Dia 1: Replicar congeladas culturas mutantes de "cópia master" de PA14NR definida em meios de ágar LB e crescer mutantes durante a noite a 37 ° C. Dia 2: Transferência de crescimento mutante de media LB ágar para profundo bem blocos contendo líquido caldo LB, crescer durante a noite a 37 ° C, com agitação a 950 rpm. Dia 3: Misturar culturas LB durante a noite com glicerol e, em seguida, transferir para placas de 96 poços destino para armazenamento a longo prazo. Lugar de 96 poços chapas plana no congelador-80 ° C. Clique aqui para ver uma versão maior desta figura.
Figura 2: recomendado instalação. Esterilidade e suave fluxo de trabalho devem ser mantidas através do uso de precauções apropriadas. Clique aqui para ver uma versão maior desta figura.
1. protocolo i: replicação de PA14NR o conjunto
Nota: A replicação da biblioteca pode ser alcançada dividindo-se o conjunto de PA14NR nas quatro subconjuntos de dezesseis placas cada que podem ser processados em quatro semanas consecutivas. Geração de 1 a 6 cópias adere ao fluxo de trabalho semanal, descritas na tabela 1, enquanto a geração de mais de 6 cópias segue o fluxo de trabalho semanal, descrito na tabela 2. Para gerar 12 cópias do conjunto de PA14NR, de inocular o mesmo subconjunto de PA14NR em meio LB líquido no dia 2 e novamente no dia 3 (a partir do mesmo conjunto de placas de ágar replicada em 1 dia) e transferência pernoite culturas mutantes em chapas de cópia no dia 3 e dia 4 respectivamente.
| Dia 0 | Dia 1 | Dia 2 | Dia 3 |
| Dia da preparação | Crescimento de mutantes PA14NR definido em meios de ágar LB | Crescimento de mutantes PA14NR definida em meio líquido LB | Transferência de culturas mutantes PA14NR definido para placas de destino |
Tabela 1: cronograma para replicação de 1-6 cópias do conjunto de PA14NR de. Replicação de um pequeno número de cópias pode aderir a um fluxo de trabalho semanal.
| Dia 0 | Dia 1 | Dia 2 | Dia 3 | Dia 4 |
| Dia da preparação | Crescimento de mutantes PA14NR definido no ágar LB | Crescer de mutantes PA14NR definida em meio líquido LB | Transferência de culturas mutantes de dia 3 para gerar 1 conjunto de 6 cópias do conjunto de PA14NR | Transferência de culturas mutantes dia 4 para gerar 2º conjunto de 6 cópias do conjunto de PA14NR |
| Crescimento de mutantes PA14NR definida em meio líquido LB |
Tabela 2: cronograma para replicação de até 12 cópias do conjunto de PA14NR de. Replicação de um maior número de cópias exigirá camadas dentro de um fluxo de trabalho semanal.
- Dia 1: Crescimento de PA14NR conjunto mestre cópia em placas de ágar LB
- Use luvas, jaleco e máscara para manipular o conjunto de PA14NR.
- Limpar a bancada e limpe a superfície com etanol a 70%.
- Preparar LB ágar mídia43 e esterilizar em autoclave pela mídia legal de 25 min. a 55 ° C na água do banho e acrescentar ou gentamicina 15 µ g/mL, para mutantes contendo inserções de transposon MAR2xT7 , ou 200 canamicina µ g/mL, para mutantes contendo TnphoA inserções.
- Despeje fundido LB ágar em placas retangulares usando aproximadamente 60 mL de mídia por placa. Chapas secas em uma capa estéril para aproximadamente 1 h antes de usam. Certifique-se que não há nenhuma condensação de água na superfície do ágar. Armazenar as placas a 4 ° C, se necessário.
- Criar um campo estéril com um bico de Bunsen na bancada de etanol-limpou e configurar recipientes com soluções adequadas para a esterilização de pino replicator (consulte a etapa 1.1.9). Uso abrir recipientes plásticos (5.25" L x 4,25" W x 1.75 "H tamanho aproximado) para esterilização de pino replicator. Recipientes de plástico de autoclave antes de usar.
Nota: O calor da chama de Bunsen cria uma convecção atual, que se aquece o espaço acima da chama e elevadores de partículas no ar e longe do ar mais frio por baixo, mantendo a área de trabalho estéril. - Retire um máximo de quatro placas de mestre PA14NR conjunto do freezer-80 ° C (para evitar descongelamento desnecessário) e coloque-os em gelo seco em panela de gelo 4L.
- Pegue a placa de 96 poços mestre para ser replicado do gelo seco e colocá-lo no banco para permitir breve degelo, que só vai levar o tempo necessário para a esterilização dos pinos replicator (aproximadamente 3-4 min) (consulte a etapa 1.1.8). Marca a posição da A1 coordenada na placa de ágar antes do carimbo. Alinhe a placa mestra e placa de ágar com a coordenada de A1 de ambas as placas no canto superior esquerdo.
- Esterilize o replicador "pinos", seguindo os passos descritos abaixo. Torneira replicator ligeiramente depois de cada passo para remover o excesso de líquido.
- Mergulhe os pinos em 250 mL de 0,3-0,5% de hipoclorito de sódio (10% de alvejante doméstico) por 30 segundos. Minimize o contato com solução de hipoclorito de sódio, pois pode levar a danos de pinos. Mergulhe os pinos em 250 mL estéril ddH2O ou água ultrapura esterilizada durante 10 segundos, em seguida, em 250 mL de etanol a 70% por 30 segundos, em seguida, em 250 mL de etanol a 95% por 2 min.
- Chama esterilizar pinos replicator segurando replicator perpendicularmente para a chama do bico de Bunsen, lentamente se aproximando chama até etanol inflama e, em seguida, retirá-la imediatamente do fogo. Chama apaga-se uma vez todas as queimaduras do etanol fora. Uma tampa ou recipiente similar manter por perto para sufocar as chamas de etanol, se necessário.
Nota: Muito cuidado quando trabalhar com etanol perto de uma chama. Não segure replicator diretamente sobre a chama. - Legais pinos fixando-se em um prato retangular estéril não utilizado que contém a mídia LB ágar durante 30 segundos.
- Brevemente alumínio quente do mestre PA14NR definir a placa do selo com a mão antes descascando. Fazer isso enquanto pinos replicador de refrigeração. Remova o selo de alumínio com cuidado para evitar que o selo retoque a placa. Use uma pinça Retire todos os restos do selo de alumínio.
- Inserir replicator pinos placa mestra, empurrando suavemente e replicator balançando em todas as quatro direções para garantir que os pinos fazem contato com culturas bacterianas congeladas em cada um dos 96-poços. Preste atenção extra para poços localizados na borda externa da placa empurrando pinos replicator contra culturas congeladas nos poços localizados na borda da placa.
- Coloque delicadamente pinos replicator na superfície da placa de ágar. Mova o replicador em um ligeiro movimento circular de forma minigramados de aproximadamente 4-5 mm para cada estirpe mutante. Evite a possibilidade de que o minigramados podem sobrepor-se, para evitar a contaminação cruzada.
- PA14NR conjunto placa mestre do selo com um novo selo de alumínio estéril. Não toque o lado adesivo do selo de alumínio em qualquer ponto para evitar a contaminação. Certifique-se de que cada poço e as bordas da placa são completamente seladas usando um rolo de chapa. Placa de retorno de 96 poços de gelo seco.
- Repita o procedimento para cada placa mestra.
- Limpe todas as superfícies de trabalho com etanol a 70% após manusear o PA14NR definido.
- Transferência replicados placas de ágar para 37 ° C incubadora e incubar durante uma noite.
- Dia 2: Crescimento da cópia mestre de PA14NR definido no líquido caldo LB
- Prepare LB caldo líquido43 contendo gentamicina ou 15 µ g/mL, para mutantes contendo inserções de transposon MAR2xT7 , ou 200 canamicina µ g/mL, para mutantes contendo inserções dephoA Tn.
- Limpar a capa do fluxo laminar de equipamentos desnecessários e ligue o ventilador exaustor para um mínimo de 10 min antes de iniciar o trabalho. Limpe as superfícies de capuz e todos os itens colocados no bairro usando etanol a 70%.
- Preencha blocos de poço profundo 2ml com 525 µ l de caldo líquido de LB contendo antibióticos apropriados no capô de fluxo laminar, usando uma pipeta eletrônica de 12 canais 50-1200 µ l. Em seguida, transferi blocos de fundo bem cheio de mídia para etanol-limpou a bancada. Reutilize dicas desde que sejam respeitadas as estéreis.
- Use luvas, jaleco e máscara para lidar com PA14NR definido.
- Limpar a bancada e limpe a superfície com etanol a 70%.
- Traz placas de ágar com cepas mutantes crescidas durante a noite para a bancada.
- Criar um campo estéril com um bico de Bunsen na bancada de etanol-limpou e configurar recipientes com soluções adequadas para a esterilização de pino replicator (ver próximo passo).
- Esterilize os pinos"replicador" seguindo os passos descritos abaixo. Torneira replicator ligeiramente depois de cada imersão para remover o excesso de líquido.
- Mergulhe os pinos em 250 mL de 0,3-0,5% de hipoclorito de sódio por 30 segundos. Minimize o contato de pino replicador com solução de hipoclorito de sódio, pois pode levar a danos de pinos. Em seguida, mergulhe os pinos em 250 mL estéril ddH2O ou água ultrapura durante 10 segundos, em seguida, em 250 mL de etanol a 70% por 30 segundos e, em seguida, em 250 mL de etanol a 95% por 2 min.
- Chama esterilizar pinos replicator e, em seguida, pinos legais fixando em chapa Retangular não utilizada que contém a mídia LB ágar durante 30 segundos.
Nota: Muito cuidado quando trabalhar com etanol perto de uma chama (ver 1.1.8).
- Gentilmente colocar pinos replicator em placa de ágar contendo o crescimento de mutantes e verifique se os pinos estão em contacto com todos os 96 mutantes na placa de ágar e, em seguida, mergulhe os pinos contendo líquido caldo LB de poços profundos. Evite tocar os lados dos poços com os pinos.
- Selo do bloco poço profundo com uma membrana de vedação respirável estéril. Use um rolo de placa para garantir que cada indivíduo bem está devidamente selado.
- Repita o procedimento para cada placa de ágar.
- Limpe todas as superfícies de trabalho com etanol a 70% após manusear o PA14NR definido.
- Cultivar culturas líquidas inoculadas para 15-16 h a 37° C a 950 rpm usando um misturador de alta velocidade, se disponível.
Nota: Se não houver um misturador de alta velocidade, incubação de poço profundo blocos em shaker a 250-300 rpm é viável. No entanto, há uma maior chance de colônia pequenas variantes (VCEs)25 emergentes sob condições de baixa oxigenação. Portanto, é altamente aconselhável manter incubação vezes abaixo dos 15-16 h quando crescer culturas em poço profundo blocos em velocidades mais baixas do agitador. Proliferação indesejada de VCEs em poços mutantes pode alterar fenótipos mutantes ao usar a biblioteca para executar telas genéticas.
Alguns poços em conjunto o PA14NR contenham mutantes crescendo/não-crescimento lento, falta de clones, ou contenham mídia não inoculada. A localização desses poços foi gravada e pode ser encontrada no arquivo suplementar PA14NR definir informações de poços incluído com esta publicação.
- Dia 3: Transferência de PA14NR conjunto de culturas durante a noite em placas de destino
- Limpar o exaustor de fluxo laminar e ligue o ventilador exaustor para um mínimo de 10 min antes de iniciar o trabalho. Limpe as superfícies de capuz e todos os itens colocados no bairro usando etanol a 70%.
- Imprimir etiquetas adesivas à prova d'água (ver arquivo suplementar PA14NR definir rótulos para modelo). Remover 96 poços chapas de plástico enrole dentro de uma capa de fluxo laminar. Peel-off placa rotular e colocá-lo na borda do prato está mais próximo de A1 H1 aos poços da placa de destino. Ligeiramente, levante a tampa da placa de destino e coloque a etiqueta na borda inferior para exibir o rótulo, quando a placa é coberta com a tampa.
- 3,5 L de glicerol de 60% (v/v) de preparar e esterilizar 20 min em autoclave.
- Use luvas, jaleco e máscara para manipular o conjunto de PA14NR. Remova blocos de poço profundo do agitador de alta velocidade ou agitador regular.
- Transferir blocos de poços profundos para fluxo de laminar estéril hood e remova cuidadosamente a membrana respirável de selagem. Substitua com selo de alumínio. Desativação da rotação dos blocos bem profundo com velocidade muito baixa para coletar condensação (30 segundos a 50-150 x g, então desacelerar quicky tendo centrífuga freio no).
- Transferir blocos de fundo-bem volta para hood estéril e usando uma pipeta eletrônica de 50-1200 µ l 12 canais e dicas filtradas estéril adicionem 525 µ l de mistura de caldo líquido de glicerol/LB (partes iguais LB caldo e 60% glicerol solução líquida) a cada poço. Misture delicadamente pipetagem 300 µ l acima e para baixo 3 vezes com pipeta eletrônica. Toque em dicas para o lado do bem antes de ejetar dicas para evitar gotejamento. Dicas de ejetar e continuar com a próxima linha até terminar com todo bloco poço profundo.
- Usando o 12 canais eletrônica repetitiva pipeta e dicas filtradas, para evitar a contaminação interwell durante alíquotas, puxe 900 µ l de cultura mutante e dispense 150 µ l em cada placas de 96 poços de destino para gerar 6 cópias da placa da biblioteca. Prevenir gotejamentos tocando a parede dos poços com dicas antes de iniciar a transferência da cultura em placas de destino. Se não repetir pipeta está disponível, use uma pipeta multicanal para dispensar 150 µ l em cada uma das seis placas 96 poços, usando a técnica descrita acima para evitar gotejamento.
- Use selos de alumínio estéril para cobrir as placas e usar um rolo de placa completamente selo placa bordas e todos os poços. Certifique-se de não cobrir o identificador de etiqueta com o selo de alumínio. Não agite as placas, como a cultura pode salpicar nos lados dos poços ou no selo de alumínio.
- Remova placas seladas de placas capô e coloque um plano de superfície plana em congelador a-80 ° C.
- Limpe todas as superfícies de trabalho com etanol a 70% após a biblioteca de manipulação.
- Execute verificações de controlo de qualidade após a replicação de biblioteca e depois de usar a biblioteca para executar telas genéticas (ver protocolo III).
2. Protocolo II: Manipulação e armazenamento de mutantes individuais do conjunto de PA14NR
-
Dia 1: Mutante de raia de interesse
- Identificar a localização do mutante de interesse através do conjunto PA14NR link http://pa14.mgh.harvard.edu/cgi-bin/pa14/search.cgi?searchType=SEARCH_PLATE_POSITIONS ou usando o arquivo Library.xls não redundante baixado o link http:// PA14.MGH.Harvard.edu/cgi-bin/PA14/downloads.cgi). Tome nota do antibiótico necessário para seleccionar todos os mutantes particular (gentamicina ou canamicina).
- Preparar a mídia de ágar LB, esterilizar em autoclave por 20-25 min e fixe a 55 ° C em banho-maria. Adicione 15 gentamicina µ g/mL para mutantes contendo inserções de transposon MAR2xT7 e 200 µ g/mL canamicina para mutantes contendo inserções de transposon TnphoA . Despeje a media de ágar LB em placas (placas redondas ou retangulares são adequadas). Chapas secas na capa estéril para cerca de 30 min a 1 h antes de usam.
- Autoclave todos plasticware e suprimentos não esterilizados antes do uso.
- Use luvas, jaleco e máscara para manipular o conjunto de PA14NR. Limpar a bancada e limpe a superfície com álcool 70% antes de trabalhar com o conjunto de PA14NR.
- Crie um campo estéril com um bico de Bunsen.
- Remover placa de 96 poços de PA14NR conjunto com mutante de interesse do congelador-80 ° C e coloque-o em gelo seco, leve o recipiente de gelo seco para o banco e brevemente colocar placa de 96 poços em cima do banco para permitir ligeira de descongelamento (aproximadamente 1-2 min).
Nota: Manter um registro de todos os PA14NR conjunto 96 placas boas acessado para mutantes individuais de raia, como mais acesso às placas de biblioteca está correlacionado com um maior risco de contaminação interwell. - Selo de alumínio quente com a mão antes descascando, tendo o cuidado de evitar que o selo retoque a placa. Use uma pinça Retire todos os restos do selo de alumínio.
- Localize o mutante de interesse sobre a placa de 96 poços. Usar vara de madeira estéril ou ponta da pipeta estéril para escolherem uma pequena quantidade da cultura congelada o indivíduo bem contendo o mutante de interesse.
- Raia congelada cultura em placa de ágar para colônias mutantes única como segue: delicadamente espalhar bactérias sobre uma secção da placa para criar a raia 1, usando um fresca, estéril de madeira vara ou pipeta de ponta, arraste através de raia 1, espalhando as bactérias uma segunda seção de a placa, para criar a raia 2. Usando um terceiro estéril de madeira vara ou pipeta ponta, arraste através de raia 2, espalhando as bactérias a última seção da placa, para criar a raia 3.
- Placa de fonte do selo com um novo selo de alumínio estéril. Não toque o lado adesivo do selo de alumínio em qualquer ponto para evitar a contaminação. Certifique-se de que cada bem e as bordas da placa são completamente seladas usando um rolo de chapa. Placa de retorno de 96 poços de gelo seco e, em seguida, congelador a-80 ° C.
- Incube a placa de ágar em incubadora 37 ° C durante a noite.
- Limpe todas as superfícies de trabalho com etanol a 70% após a biblioteca de manipulação.
-
Dia 2: Crescimento de mutantes de interesse em caldo líquido LB
- Prepare o caldo LB líquido contendo 15 gentamicina µ g/mL ou canamicina de 200 µ g/mL, conforme a inserção de transposon.
- Use luvas, jaleco e máscara para lidar com a p. aeruginosa.
- Limpar a bancada e limpe a superfície com etanol a 70%. Crie um campo estéril com um bico de Bunsen.
- Transferência de 3-5 mL de caldo LB com antibiótico apropriado em tubo de cultura estéril com tampa.
- Usando um aplicador estéril ou uma ponta de pipeta estéril, escolher uma única colônia da estirpe mutante e inoculá-lo em LB mídia.
- Incube as culturas de líquido de LB a 37 ° C em uma coqueteleira a 225-250 rpm, durante a noite.
- Limpe todas as superfícies de trabalho com etanol a 70% após a manipulação de p. aeruginosa.
-
Dia 3: Armazenar mutante de interesse no freezer-80 ° C
- Cryovial de etiqueta com o nome de mutantes, antibióticos adicionados ao caldo LB e data de armazenamento.
- Preparar 500 mL de 50% (v/v) de glicerol e esterilizar em autoclave.
- Use luvas, jaleco e máscara para lidar com a p. aeruginosa.
- Limpar o banco e/ou capa de inundação laminar e limpe a superfície com etanol a 70%.
- Retire o tubo contendo a cultura mutante do agitador.
- Prepare um recipiente pequeno com gelo seco.
- Use uma capa de fluxo laminar ou criar um campo estéril na bancada etanol-limpou com bico de Bunsen.
- Adicionar quantidades iguais de cultura bacteriana e glicerol 50% para o cryovial etiquetado usando condições estéreis e misture suavemente com pipeta (volume final 1-2 mL/frasco dependendo do tamanho do cryovials usado). Lugar cryovial em gelo seco para congelamento rápido.
- Coloque cryovial na caixa rotulada no freezer-80 ° C.
- Limpe todas as superfícies de trabalho com etanol a 70% após a manipulação de p. aeruginosa.
3. protocolo III: controle de qualidade do conjunto de PA14NR
- Selecione um conjunto aleatório de mutantes recém replicadas placas para detectar possível contaminação interwell (teste de 30-40 mutantes é recomendado).
Nota: Em casos onde é necessário confirmar a identidade de um mutante sendo usado para a caracterização de um gene específico, é recomendável executar amplificações do PCR usando as primeiras demão gene-específico destinadas a sequência conhecida do gene que contém a inserção de Transposon. Embora mais desafiador, os benefícios do uso arbitrário iniciadores de PCR, ao invés de iniciadores de PCR do gene-específico quando amplyfing DNA fragmentos de mutantes transposon incluem a facilidade de confirmação mutante em grande escala e a capacidade de detectar a presença de potencial contaminantes. Para efeitos de controlo de qualidade, não é necessário obter dados de sequenciamento de PCR de alta qualidade para todos aleatoriamente escolhidas mutantes, contanto que um número suficiente de mutantes é selecionado para avaliar a taxa de erro. - Siga o "Protocolo II" para a raia e crescer cepas mutantes.
- Autoclave todos plasticware e suprimentos não esterilizados antes do uso.
- Isole o DNA genômico por método preferido. Para a análise descrita neste trabalho, um kit de isolamento de DNA genômico foi utilizado seguindo o protocolo do fabricante. Várias cepas mutantes podem ser analisadas simultaneamente.
- Medir a concentração de DNA genômica usando um spectrophotometer microvolume. Ajuste a concentração de DNA genômica de aproximadamente 100 ng / µ l.
- Use o DNA genômico como modelo para executar "Reação de PCR1", conforme descrito na etapa 1 da tabela 3, gerando fragmentos de Arb1 PCR (Figura 3). Primers para esta etapa estão listados na tabela 4.
- Adicionar 0,5 µ l de 10x buffer para 5 µ l de reação PCR1 a carregar, carregar em um gel de agarose 1,5 a 2% e execute o gel em 80-150 V.
Nota: Um comprimento de fragmento específico ou um intervalo de fragmento específico não é esperado, e mais do que uma banda pode estar presente como o primer arbitrário pode vincular a vários locais. - Usar o DNA de reação de PCR1 como modelo para executar "PCR2 reação", conforme descrito na etapa 2 da tabela 3, gerando fragmentos de PCR Arb2 (Figura 3). Primers para esta etapa estão listados na tabela 4.
- Adicionar 0,5 µ l de 10x buffer para 5 µ l de reação PCR2 a carregar, carregar em um gel de agarose 1,5 a 2% e funcione o gel em 80-150 V.
Nota: Um comprimento de fragmento específico ou um intervalo de fragmento específico não é esperado, e mais do que uma banda pode estar presente como o primer arbitrário pode vincular a vários locais. - Envie PCR2 reação juntamente com a cartilha de transposon específicas apropriada para sequenciamento.
- Analisar resultados de sequenciamento byBLASTing-los contra o genoma completo de PA14 usando o link de explosão fornecidos no site da biblioteca de PA14NR (http://pa14.mgh.harvard.edu/cgi-bin/pa14/blast.cgi) ou explodindo-os diretamente contra a sequência do gene específico de o mutante de interesse.
Nota: Informações sobre mutantes selecionadas podem ser encontradas pesquisando o site PA14NR definido (pesquisa http://pa14.mgh.harvard.edu/cgi-bin/pa14/search.cgi?searchType=SEARCH_PLATE_POSITIONS ou baixar o Library.xls não redundante do arquivo de http:/ o link / pa14.mgh.harvard.edu/cgi-bin/pa14/downloads.cgi).
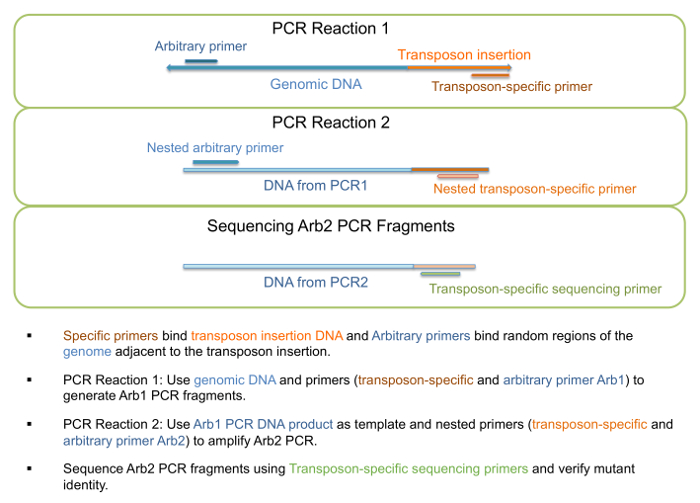
Figura 3: PCR amplificação e sequenciamento de mutantes de inserção de transposon. Visão esquemática das etapas envolvidas na amplificação por PCR e sequenciamento para verificação de identidade mutante. Clique aqui para ver uma versão maior desta figura.
| Afinação de reação de PCR | |
| Etapa 1 | Passo 2 |
| Reação de PCR1 : | PCR2 reação: |
| Água 23.25 µ l (grau de Biologia Molecular) | 19.15 µ l água (grau de Biologia Molecular) |
| Polymerase de Taq 3µL 10x Buffer | 5 µ l 10 x tampão da Taq polimerase |
| 0.5µL polymerase de Taq | 0,6 µ l Taq polimerase |
| 0,625 µ l 20 µM primer Arb1D (tabela 4) | 0,625 µ l 20 µM primer Arb2A (tabela 4) |
| Primeira demão de transposon-específica 0,625 µ l 20 µM (PMFLGM. 3a GB ou Tn5Ext) (tabela 4) | primeira demão de Transposon-específica 0,625 mL 20 µM (PMFLGM. 2a GB ou Tn5Int2) (tabela 4) |
| 1 µ l 10mm dNTPs | 1 µ l 10mm dNTPs |
| 1 µ l DNA genômico, 100 ng | Reação de PCR1 de 5 µ l |
| Volume de reação final de 30 µ l | Volume de reação final de 30 µ l |
| Configurações de reação de PCR | |
| PCR1 Thermocycler condições: | PCR2 Thermocycler condições: |
| 95 ° C – 2 min | 95 ° C – 2 min |
| Repita 5 ciclos: | Repita 30 ciclos: |
| 95 ° C – 30 s | 95 ° C – 30 s |
| 30 ° C – 1 min | 54 ° C – 30 s |
| 72 ° C – 1 min | 72 ° C – 1,5 min |
| Repita 30 ciclos: | 72 ° C – 10 min |
| 95 ° C – 30 s | 4 ° C – Hold |
| 45 ° C – 30 s | |
| 72 ° C – 1 min | |
| 72 ° C – 10 min | |
| 4° C – Hold |
Tabela 3: PCR reação set-up e thermocycler condições usadas para PCR arbitrária. Reações de PCR arbitrárias são executadas sequencialmente, e fragmentos gerados durante a reação de PCR1 são usados como modelo em PCR2 reação. Configurações específicas thermocycler são usadas para cada conjunto de reações.
| Nome da primeira demão | Sequência da primeira demão |
| MAR2xT7 específicos Transposon Primers | |
| PMFLGM. GB-3a | TACAGTTTACGAACCGAACAGGC |
| PMFLGM. GB-2a | TGTCAACTGGGTTCGTGCCTTCATCCG |
| MAR2xT7 Transposon sequenciamento Primer | |
| PMFLGM. GB-4a | GACCGAGATAGGGTTGAGTG |
| Primers dephoA específicas do Transposon TN | |
| Tn5Ext | GAACGTTACCATGTTAGGAGGTC |
| Tn5Int2 | GGAGGTCACATGGAAGTCAGATCCTGG |
| TNphoA Transposon Sequencing Primer | |
| Tn5Int | CGGGAAAGGTTCCGTTCAGGACGC |
| Primers arbitrários | |
| ARB1D | GGCCAGGCCTGCAGATGATGNNNNNNNNNNGTAT |
| ARB2A | GGCCAGGCCTGCAGATGATG |
Tabela 4: lista dos Primers usados no controle de qualidade Experimentos. Primers utilizados para amplificação por PCR e sequenciamento de mutantes de inserção de transposon para confirmar identidade mutante.
Resultados
Doze novas cópias da PA14NR Set foram replicadas usando protocolo I e uma avaliação de controle de qualidade das cópias do novas gerado foi realizado utilizando protocolo III.
PA14NR conjunto placas mutantes, juntamente com as placas de controle, que consistem de tipo selvagem PA14 inoculado e não inoculadom poços intercalados em um padrão predefinido (Figura 4A), foram replicadas na sequênc...
Discussão
O P. aeruginosa Conjunto de PA14NR é um recurso valioso para a comunidade científica. De acordo com o conjunto de dados de março de 2017 de banco de dados de indicadores essenciais da ciência de Clarivate do Analytics, et al . Liberati (2006) 37, que descreve a construção de conjunto de PA14NR, é classificado no top 1% das publicações de microbiologia. Google Scholar relata mais de 600 citações da Liberati et al manuscrito original (2006) a partir de ...
Divulgações
Os autores relatam que não há conflitos de interesses financeiros. Eliana Drenkard e Frederick Ausubel participaram da criação biblioteca mutante PA14 transposon não redundante. Bryan Hurley e Lael Yonker atualmente casa e distribuem a biblioteca mutante como parte do departamento de Pediatria do hospital geral de Massachusetts.
Agradecimentos
Gostaríamos de agradecer a Lisa Philpotts da MGH Treadwell Biblioteca Virtual para sua orientação na busca de dados. Este trabalho foi financiado pela Fundação de fibrose cística (YONKER16G0 e HURLEY16G0) e NIH NIAID (BPH e ADE: R01 A1095338).
Materiais
| Name | Company | Catalog Number | Comments |
| Materials for Library Replication | |||
| Sterile 96-well Tissue-culture treated, case of 50 | Corning Life Sciences | 353072 | via Fisher Scientific |
| Sterile 96 Well Clear V-Bottom 2000μL Deep Well Plates, case of 25 | Corning Life Sciences | 3960 | via Fisher Scientific |
| Nunc OmniTray (rectangular plates), case of 60 | Thermo Scientific Rochester | 242811 | via Fisher Scientific |
| Rectangular Ice Pan, Midi (4L) | Corning Life Sciences | 432104 | via Fisher Scientific |
| Secure-Gard Cone Mask, case of 300 | Cardinal Health | AT7509 | via Fisher Scientific |
| AluminaSeal, pack of 100 | Diversified Biotech | ALUM-100 | via Fisher Scientific |
| Breathe-Easy membrane, pack of 100 | Diversified Biotech | BEM-1 | via Sigma-Aldrich |
| Sterile, individually wrapped, 50mL Solution Trough/Reagent Reservoir, case of 100 | Sorenson | S50100 | via Westnet Incorporated |
| Plate roller | VWR | 60941-118 | via VWR |
| Cryo Laser Labels - CRYOLAZRTAG 2.64" x 0.277", pack of 16 sheets | GA International | RCL-11T1-WH | via Labtag.com (template for printing also available from Labtag.com) |
| 96-well replicator | V & P Scientific, Inc. | Custom 407C, 3.18mm pin diameter, 57mm long | via V & P Scientific, Inc. |
| Multitron Pro, 3mm Shaking incubator | Infors HT | l10003P | via Infors HT |
| Picus 12 Channel 50-1200μL Electronic Pipette | Sartorius | 735491PR | via Sartorius |
| Filter Tips 50-1200μL, pack of 960 | Biohit | 14-559-512 | via Fisher Scientific; use electronic multichannel-compatible tips |
| Dry Ice | User-specific vendor | ||
| Materials for Individual Mutant Storage | |||
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 05-408-130 | via Fisher Scientific |
| Pipettes (P1000, P200, P20, P2) | Gilson | F167370 | via Gilson |
| Materials for Quality Control PCR | |||
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 05-408-130 | via Fisher Scientific |
| NanoDrop | Thermo Scientific | ND-2000 | via ThermoFisher |
| PCR Thermocycler | |||
| Omnistrips PCR Tubes with domed lids | Thermo Scientific | AB0404 | via Fisher Scientific |
| ART Barrier low-retention pipette tips (10 uL, 100 uL, 1000 uL) | Molecular BioProducts, Inc. | Z676543 (10 uL), Z676713 (100 uL), Z676802 (1000 uL) | via Sigma-Aldrich |
| Pipettes (P1000, P200, P20, P2) | Gilson | F167370 | via Gilson |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 05-408-130 | via Fisher Scientific |
| MasterPure DNA Purification Kit | Epicentre | MCD85201 | via Epicentre Technologies Corp |
| GeneRuler 1 kb Plus DNA Ladder, ready-to-use | Thermo Scientific | SM1333 | via ThermoFisher |
| RediLoad Loading Buffer | Invitrogen | 750026 | via ThermoFisher |
| Chemicals | |||
| Chemicals for Library and Individual Mutant Storage | |||
| Glycerol MB Grade, 1L | Sigma Aldrich | G5516 | via Sigma-Aldrich |
| LB Broth | Per 1L dH2O: 10g tryptone, 5g yeast extract, 5g NaCl, 1ml 1N NaOH (Current Protocols in Molecular Biology. Wiley, 1994.) | ||
| Tryptone | Sigma Aldrich | T7293 | via Sigma-Aldrich |
| Yeast Extract | Sigma Aldrich | Y1625 | via Sigma-Aldrich |
| Sodium Chloride | Sigma Aldrich | S7653 | via Sigma-Aldrich |
| Sodium Hydroxide | Sigma Aldrich | S8045 | via Sigma-Aldrich |
| LB agar | See preparation above, add 15g Bacto Agar | ||
| Bacto Agar | Sigma Aldrich | A5306 | via Sigma-Aldrich |
| Gentamicin sulfate, 10g | BioReagent | 1405-41-0 | via Sigma-Aldrich |
| Kanamycin sulfate | Gibco | 11815024 | via ThermoFisher |
| Ethanol, 190 proof | Decon | 04-355-221 | via Fisher Scientific |
| Chemicals for Quality Control PCR | |||
| Primers | User-preferred vendor | See primers listed in Table 3 | |
| Corning cellgro Molecular Biology Grade Water | Corning | 46000CV | via Fisher Scientific |
| Taq Polymerase Buffer | Invitrogen | 10342020 | via ThermoFisher |
| Taq DNA Polymerase, recombinant | Invitrogen | 10342020 | via ThermoFisher |
| dNTPs | Invitrogen | 10297018 | via ThermoFisher |
| Agarose | Sigma | A9539 | via Sigma-Aldrich |
Referências
- Moradali, M. F., Ghods, S., Rehm, B. H. Pseudomonas aeruginosa lifestyle: A paradigm for adaptation, survival, and persistence. Front Cell Infect Microbiol. 7, 39 (2017).
- Bleves, S., et al. Protein secretion systems in Pseudomonas aeruginosa: A wealth of pathogenic weapons. Int J Med Microbiol. 300 (8), 534-543 (2010).
- Breidenstein, E. B., de la Fuente-Nunez, C., Hancock, R. E. Pseudomonas aeruginosa: all roads lead to resistance. Trends Microbiol. 19 (8), 419-426 (2011).
- Flynn, K. M., et al. Evolution of ecological diversity in biofilms of Pseudomonas aeruginosa by altered cyclic diguanylate signaling. J Bacteriol. 198 (19), 2608-2618 (2016).
- Hazan, R., Maura, D., Que, Y. A., Rahme, L. G. Assessing Pseudomonas aeruginosa persister/antibiotic tolerant cells. Methods Mol Biol. 1149, 699-707 (2014).
- Klockgether, J., et al. Genome diversity of Pseudomonas aeruginosa PAO1 laboratory strains. J Bacteriol. 192 (4), 1113-1121 (2010).
- Mathee, K., et al. Dynamics of Pseudomonas aeruginosa genome evolution. Proc Natl Acad Sci U S A. 105 (8), 3100-3105 (2008).
- Taylor, P. K., Yeung, A. T., Hancock, R. E. Antibiotic resistance in Pseudomonas aeruginosa biofilms: towards the development of novel anti-biofilm therapies. J Biotechnol. 191, 121-130 (2014).
- Flume, P. A., Van Devanter, D. R. State of progress in treating cystic fibrosis respiratory disease. BMC Med. 10, 88 (2012).
- . Cystic Fibrosis Foundation Patient Registry 2015 Annual Data Report Available from: https://www.cff.org/Our-Research/CF-Patient-Registry/2015-Patient-Registry-Annual-Data-Report.pdf (2016)
- Church, D., Elsayed, S., Reid, O., Winston, B., Lindsay, R. Burn wound infections. Clin Microbiol Rev. 19 (2), 403-434 (2006).
- Sadikot, R. T., Blackwell, T. S., Christman, J., Prince, A. Pathogen-host interactions in Pseudomonas aeruginosa pneumonia. Am J Respir Crit Care Med. 171 (11), 1209-1223 (2005).
- Song, Z., et al. Prosthesis infections after orthopedic joint replacement: the possible role of bacterial biofilms. Orthop Rev (Pavia). 5 (2), 65-71 (2013).
- National Nosocomial Infections Surveillance, S. National Nosocomial Infections Surveillance (NNIS) System Report, data summary from January 1992 through June 2004, issued October 2004. Am J Infect Control. 32 (8), 470-485 (2004).
- Cohen, T. S., Parker, D., Prince, A. . Pseudomonas aeruginosa Host Immune Evasion. 7, 3-23 (2014).
- Fernandes, A., Dias, M. The microbiological profiles of infected prosthetic implants with an emphasis on the organisms which form biofilms. J Clin Diagn Res. 7 (2), 219-223 (2013).
- Khosravi, A. D., Ahmadi, F., Salmanzadeh, S., Dashtbozorg, A., Montazeri, E. A. Study of Bacteria Isolated from Orthopedic Implant Infections and their Antimicrobial Susceptibility Pattern. Res J of Microbiol. 4 (4), 6 (2009).
- Roemhild, R., Barbosa, C., Beardmore, R. E., Jansen, G., Schulenburg, H. Temporal variation in antibiotic environments slows down resistance evolution in pathogenic Pseudomonas aeruginosa. Evol Appl. 8 (10), 945-955 (2015).
- Fischer, S., et al. Intraclonal genome diversity of the major Pseudomonas aeruginosa clones C and PA14. Environ Microbiol Rep. 8 (2), 227-234 (2016).
- Wiehlmann, L., et al. Population structure of Pseudomonas aeruginosa. Proc Natl Acad Sci U S A. 104 (19), 8101-8106 (2007).
- Lam, J. S., Taylor, V. L., Islam, S. T., Hao, Y., Kocincova, D. Genetic and functional diversity of Pseudomonas aeruginosa lipopolysaccharide. Front Microbiol. 2, 118 (2011).
- Choi, J. Y., et al. Identification of virulence genes in a pathogenic strain of Pseudomonas aeruginosa by representational difference analysis. J Bacteriol. 184 (4), 952-961 (2002).
- He, J., et al. The broad host range pathogen Pseudomonas aeruginosa strain PA14 carries two pathogenicity islands harboring plant and animal virulence genes. Proc Natl Acad Sci U S A. 101 (8), 2530-2535 (2004).
- Mikkelsen, H., McMullan, R., Filloux, A. The Pseudomonas aeruginosa reference strain PA14 displays increased virulence due to a mutation in ladS. PLoS One. 6 (12), e29113 (2011).
- Drenkard, E., Ausubel, F. M. Pseudomonas biofilm formation and antibiotic resistance are linked to phenotypic variation. Nature. 416 (6882), 740-743 (2002).
- Rahme, L. G., et al. Common virulence factors for bacterial pathogenicity in plants and animals. Science. 268 (5219), 1899-1902 (1995).
- Rahme, L. G., et al. Use of model plant hosts to identify Pseudomonas aeruginosa virulence factors. Proc Natl Acad Sci U S A. 94 (24), 13245-13250 (1997).
- Kirienko, N. V., Cezairliyan, B. O., Ausubel, F. M., Powell, J. R. Pseudomonas aeruginosa PA14 pathogenesis in Caenorhabditis elegans. Methods Mol Biol. 1149, 653-669 (2014).
- Mahajan-Miklos, S., Tan, M. W., Rahme, L. G., Ausubel, F. M. Molecular mechanisms of bacterial virulence elucidated using a Pseudomonas aeruginosa-Caenorhabditis elegans pathogenesis model. Cell. 96 (1), 47-56 (1999).
- Limmer, S., et al. Pseudomonas aeruginosa RhlR is required to neutralize the cellular immune response in a Drosophila melanogaster oral infection model. Proc Natl Acad Sci U S A. 108 (42), 17378-17383 (2011).
- Miyata, S., Casey, M., Frank, D. W., Ausubel, F. M., Drenkard, E. Use of the Galleria mellonella caterpillar as a model host to study the role of the type III secretion system in Pseudomonas aeruginosa pathogenesis. Infect Immun. 71 (5), 2404-2413 (2003).
- Coleman, F. T., et al. Hypersusceptibility of cystic fibrosis mice to chronic Pseudomonas aeruginosa oropharyngeal colonization and lung infection. Proc Natl Acad Sci U S A. 100 (4), 1949-1954 (2003).
- Pazos, M. A., et al. Pseudomonas aeruginosa ExoU augments neutrophil transepithelial migration. PLoS Pathog. 13 (8), e1006548 (2017).
- Maura, D., Hazan, R., Kitao, T., Ballok, A. E., Rahme, L. G. Evidence for direct control of virulence and defense gene circuits by the Pseudomonas aeruginosa quorum sensing regulator, MvfR. Sci Rep. 6, 34083 (2016).
- Jacobs, M. A., et al. Comprehensive transposon mutant library of Pseudomonas aeruginosa. Proc Natl Acad Sci U S A. 100 (24), 14339-14344 (2003).
- Held, K., Ramage, E., Jacobs, M., Gallagher, L., Manoil, C. Sequence-verified two-allele transposon mutant library for Pseudomonas aeruginosa PAO1. J Bacteriol. 194 (23), 6387-6389 (2012).
- Liberati, N. T., et al. An ordered, nonredundant library of Pseudomonas aeruginosa strain PA14 transposon insertion mutants. Proc Natl Acad Sci U S A. 103 (8), 2833-2838 (2006).
- Feinbaum, R. L., et al. Genome-wide identification of Pseudomonas aeruginosa virulence-related genes using a Caenorhabditis elegans infection model. PLoS Pathog. 8 (7), e1002813 (2012).
- Stewart, L., et al. Draft genomes of 12 host-adapted and environmental isolates of Pseudomonas aeruginosa and their positions in the core genome phylogeny. Pathog Dis. 71 (1), 20-25 (2014).
- Thrane, S. W., et al. The widespread multidrug-resistant serotype O12 Pseudomonas aeruginosa clone emerged through concomitant horizontal transfer of serotype antigen and antibiotic resistance gene clusters. MBio. 6 (5), e01396-e01315 (2015).
- van Belkum, A., et al. Phylogenetic Distribution of CRISPR-Cas Systems in Antibiotic-Resistant Pseudomonas aeruginosa. MBio. 6 (6), e01796-e01715 (2015).
- Lewenza, S., et al. Construction of a mini-Tn5-luxCDABE mutant library in Pseudomonas aeruginosa PAO1: a tool for identifying differentially regulated genes. Genome Res. 15 (4), 583-589 (2005).
- . . Current Protocols in Molecular Biology. , (1994).
- Breidenstein, E. B., Khaira, B. K., Wiegand, I., Overhage, J., Hancock, R. E. Complex ciprofloxacin resistome revealed by screening a Pseudomonas aeruginosa mutant library for altered susceptibility. Antimicrob Agents Chemother. 52 (12), 4486-4491 (2008).
- Musken, M., Di Fiore, S., Dotsch, A., Fischer, R., Haussler, S. Genetic determinants of Pseudomonas aeruginosa biofilm establishment. Microbiology. 156 (Pt 2), 431-441 (2010).
- Schurek, K. N., et al. Novel genetic determinants of low-level aminoglycoside resistance in Pseudomonas aeruginosa. Antimicrob Agents Chemother. 52 (12), 4213-4219 (2008).
- Oumeraci, T., et al. Comprehensive MALDI-TOF biotyping of the non-redundant Harvard Pseudomonas aeruginosa PA14 transposon insertion mutant library. PLoS One. 10 (2), e0117144 (2015).
- Yeung, A. T., et al. Swarming of Pseudomonas aeruginosa is controlled by a broad spectrum of transcriptional regulators, including MetR. J Bacteriol. 191 (18), 5592-5602 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados