Method Article
Digitalização de slides automatizado e segmentação em tecidos fluorescente-rotulado, usando um sistema de análise de Widefield alto teor
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui descrevemos um protocolo para a segmentação automática de tecidos fluorescente etiquetadas em slides utilizando um sistema de análise de alto teor widefield (WHCAS). Este protocolo tem aplicações abrangentes em qualquer campo que envolve a quantificação de marcadores fluorescentes em tecidos biológicos, incluindo as ciências biológicas, engenharia médica e Ciências da saúde.
Resumo
Automatizado de digitalização de slides e segmentação dos tecidos fluorescente-etiquetadas é a maneira mais eficiente de analisar toda slides ou grandes cortes histologicos. Infelizmente, muitos investigadores gastam grandes quantidades de tempo e recursos, desenvolvimento e otimização de fluxos de trabalho que só são relevantes para suas próprias experiências. Neste artigo, descrevemos um protocolo que pode ser usado por aqueles com acesso a um sistema de análise de alto teor widefield (WHCAS) para a imagem de qualquer tecido de slide-montado, com opções de personalização em módulos pré-construídos encontrados no software associado. Não originalmente destinados à digitalização de slides, os passos detalhados neste artigo possibilitam adquirir slide Digitalizando imagens na WHCAS que podem ser importados para o software associado. Neste exemplo, a segmentação automática de slides do tumor de cérebro é demonstrada, mas a segmentação automática de qualquer marcador fluorescente-etiquetadas nuclear ou citoplasmática é possível. Além disso, há uma variedade de outros módulos de software quantitativa, incluindo ensaios para a localização da proteína/translocação, proliferação celular/viabilidade/apoptose e angiogênese que pode ser executados. Esta técnica vai poupar esforço e tempo de pesquisadores e criar um protocolo automatizado para análise de slide.
Introdução
A quantificação exata e precisa dos tecidos fluorescente-etiquetadas em slides é uma técnica altamente procurada em muitos campos científicos. No entanto, pesquisadores muitas vezes manualmente contam espécimes ou gastam uma quantidade considerável de tempo desenvolvendo esotéricas técnicas automatizadas para conseguir isso. Neste documento, nós fornecemos um protocolo para o slide automatizado digitalização e quantificação de células usando uma WHCAS e seu software associado, com células do sistema imunológico inatas nas seções de tumor de cérebro humano congelado como exemplo. Os softwares associados oferece uma ampla gama de built-in módulos personalizáveis do axônio consequência contando para diferenciação de célula tipos1,2,3,4,5, 6. o objetivo desse método é proporcionar a investigadores com um protocolo de início ao fim, facilmente reproduzível para adquirir imagens de e dosar entidades fluorescente-etiquetadas em quaisquer tecidos slide-montado.
Neste protocolo, o WHCAS é usado principalmente para placas de imagem de para a posterior análise do software associado mesmo que um adaptador de slide e o básico para deslizar a digitalização7 estava disponível. Era proibitivo para slides imagem porque a cuidado calibração espacial da área de aquisição, a seleção de periódicos apropriados, a criação de loadouts adaptados e uma ligação com os representantes do produto foram necessárias. No mais vasto corpo de literatura, em vez de comprar uma imagem de slide dedicada e análise aparelhos8, um relatório anterior tecnológico com acesso a este software contornado a aquisição de imagens de slides no WHCAS no total9. Realização de análise de imagem ou de aquisição de imagem em plataformas diferentes requer trabalho extra para garantir que cada um é compatível com o outro.
A capacidade de usar o WHCAS e seu software para captura de imagem gostaria de evitar as complicações desnecessárias de procura ou desenvolver um alien de fluxo de trabalho para essas ferramentas. Neste artigo, os passos necessários para criar uma varredura de visão baixa ampliação e as correspondentes imagens de alta ampliação, tratando o slide como um prato, e as análises subsequentes usando o módulo de segmentação de comprimento de onda multi célula marcando que permitem a redefinição de objetivos do WHCAS. Este protocolo prontamente utilizável fornece uma vantagem sobre técnicas alternativas porque não há nenhuma necessidade de desenvolver algoritmos ou várias etapas contando protocolos10,11 , uma vez que as imagens são adquiridas no WHCAS. Este protocolo reduz o tempo necessário para otimizar uma técnica de quantificação, é mais preciso12 e eficiente que a contagem manual e maximiza o uso da WHCAS. Este protocolo pode ser fácil e amplamente usado desde que permite que a imagem e a análise de quaisquer tecidos fluorescente-etiquetadas em slides.
Protocolo
Tumor espécimes foram obtidos conforme o protocolo aprovado pelo comitê local revisão institucional Conselho e ética e conduzidos de acordo com as normas nacionais. O WHCAS e seu software associado, usado neste artigo estão listados na Tabela de materiais.
1. importação de periódicos
- Abra o software associado.
- Baixe a suite diário indicada na Tabela de materiais.
- Importar a suite de diário para um diretório apropriado clicando no menu principal, indo para o jornal, selecionando Importação Journal Suite...e clicando em Import.
2. criando configurações para a visualização da verificação de aquisição
- Na caixa de diálogo Configuração de placa de aquisição , ir na guia objetivo e câmera 4 Select X como a ampliação, 1 como a câmera binning e 2 que o ganho.
- Entrada as configurações na figura 1A , sob a guia da placa – Slidescanning.
Nota: Essas configurações foram criadas para permitir que o usuário visualizar e navegar até 3 slides. Cada slide foi dividida em 3 poços adjacentes para a navegação fácil e 'ao vivo' visualização de fatias de tecidos ou células. - Na guia de locais a visitar , encha os poços uniformemente com os sites.
- Clique em Editar placa inferior configurações... e digite os valores exibidos na figura 1B.
- Sob a guia de Aquisição Loop , insira o comprimento de onda desejado (qualquer mancha nuclear neste exemplo) para a verificação de pré-visualização. Para o melhor contraste e baseados em imagem com foco, use o sinal fluorescente mais brilhante (por exemplo, muitas vezes uma mancha nuclear).
Nota: Mancha os tecidos com uma mancha brilhante como uma mancha nuclear é recomendado já que este fornece um alto contraste em comparação com o plano de fundo. Não só vai esta ajuda na localidade de amostra, mas quando mais de um fluoróforo na seguinte aquisição alta magnificação de imagem, a cor mais brilhante será usada para basear o deslocamento da imagem latente das outras cores. - Ativar Correção sombreamento no modo de aquisição de placa para costurar imagens.
- Salvar essas configurações como a configuração.
3. criar configurações para aquisição de Slide em alta ampliação
- Na caixa de diálogo Configuração de placa de aquisição , ir na guia objetivo e câmera selecionar 40 X como a ampliação, 1 como a câmera binning (para a mais alta resolução digital) e 2 que o ganho (para aumentar o sinal e o brilho das imagens).
Nota: Uma configuração de ampliação objetiva inferior pode ser usada, mas os autores encontraram 40 X ser a configuração ideal para a análise da microglia humana e macrófagos no tecido do tumor cerebral usando o módulo de Comprimento de onda multi célula marcando . - Certifique-se de todas as configurações para as abas placa – Slidescanning e Editar placa inferior configurações... são o mesmo que para configuração A (consulte a etapa 2).
- Se as imagens obtidas não são nítidas, mude a profundidade bem, altura de placae Placa inferior configurações para ajustar o foco. Se ainda existe um problema com o foco, escolha Laser com recuperação de imagem na seção Opções de focagem automática .
- Sob a guia de Aquisição Loop , insira o número de comprimentos de onda presentes na amostra. Verifique as opções habilitar baseados em laser com foco e habilitar baseada em imagem com foco (para recuperação de aquisição ou laser) .
- Foco automático na guia, selecione foco na placa e bem fundo.
- Na guia de periódicos , selecione as opções conforme indicado na Figura 2, garantindo a impedir hardware assíncrono move também é selecionado.
- Salvar essas configurações como configuração B.
4. colocar o Slide no sistema de análise Widefield alto teor
- Pressione F4 para obter o Menu principal. Selecione o Slide a digitalização. Clique em porta aberta – ejetar Slide e inserir slides e o adaptador slide (que pode conter até três slides) para o WHCAS (Figura 3A e 3B). Oriente o slide com a lamínula voltada para baixo e a etiqueta no lado do entalhe no adaptador slide (Figura 3A).
Nota: Os slides usados neste experimento foram padrão 25 x 75 x 1 slides mm. - Clique em Fechar porta – Slide de carga.
5. adquirir uma varredura de visualização
- Sob o título de seleção , selecione a placa de aquisição e controle... e Instalação de placa de aquisição... e carregar a configuração A.
- Sob a guia de poços de visita , mova-se para o bem que contém a amostra, usando o botão direito do mouse no mouse. Sob a guia de locais a visitar , mova para locais diferentes com a captura de imagem Live até que as células estão localizadas. Concentre-se na amostra ou usar o foco automáticomanualmente. Use o Snap para capturar a imagem.
Nota: A amostra é mais fácil para localizar com mais sites e encaixando os sites para o bem (ver passo 2.3). - No menu principal, selecione a Visualização de Slides.
- Na caixa de diálogo que aparece automaticamente, selecione a opção refletindo slides quantas vão ser digitalizados. Digitalizar um slide de cada vez. Imprensa Okey. Selecione 4 X como a ampliação. Imprensa Okey. Selecione a lamela para baixo (0,17 mm) como a orientação do slide (uso uma correção colar se uma espessura diferente da lamela é usada). Imprensa Okey. Clique em continuar.
Nota: Se o usuário deseja adquirir varreduras de visualização para 2 ou mais slides, cada verificação prévia tem que ser aberto individualmente antes da aquisição da imagem correspondente alta ampliação. - Sob Carga digitalizar slides configuração, certifique-se de que caixas de aquisição ajustes e calibrações são verificadas. Pressione Cancelar. Quando a caixa de Configurações de deslize de verificação reaparece, selecione continuar.
- Quando aparece a caixa de diálogo Digitalizar slides , ajuste as configurações conforme sugerido na Figura 4A-4 C. Selecione fechar quando terminar.
Nota: Aumentando a câmera binning e diminuindo o tempo de exposição da câmera irá resultar em uma varredura mais rápida, embora em uma faixa dinâmica e menor resolução digital, respectivamente, portanto, diminuindo a qualidade da imagem. Sombreamento de correção irá garantir uma intensidade uniforme através de um único campo de visão. Girando sobre Hardware e imagem Autofocus assegurará a verificação está em foco, mas retardará a digitalização. No interesse da economia de tempo, é recomendável ter sombreamento correção e Hardware e imagem Autofocus fora enquanto adquirindo a digitalização ampliação baixa, mas para ter essas opções ativada para a etapa 6 durante a geração de imagens de alta ampliação. A resolução da digitalização ampliação baixa não afetará a resolução da digitalização ampliação elevada. - Selecione um diretório para o slide de digitalização de dados. Clique Okey. Digite um nome para o arquivo. Clique Okey.
- Caixa de diálogo Desenhar região aparecerá automaticamente. Use a ferramenta de região retangular (Figura 5A) para selecionar a região de varredura de visualização inteira como mostrado na Figura 5B. Clique em continuar.
- Aparecerá a caixa de diálogo Instalação completa . Selecione continuar.
Nota: A digitalização de slides de visualização vai começar. - Quando a visualização é concluída, será exibida a caixa de diálogo Verificação completa . Selecione continuar. Não feche a janela de verificação de visualização (neste exemplo, a janela Digitalizar DAPI ; consulte a Figura 6).
Nota: A configuração e o processo de aquisição de varredura de baixa ampliação levará cerca de 20 min. O tempo de aquisição de imagem posterior alta ampliação dependerá do número de sites escolhido assim como os tempos de exposição para cada canal.
6. alta ampliação digitalização
- Carregar configuração B.
- Determine quais poços são para ser fotografada. Sob a guia de poços de visita , mova-se para o bem que contém a amostra, usando o botão direito do mouse no mouse.
Nota: A técnica é demonstrada aqui por imagem bem 1. - Sob a guia de locais a visitar , mova para diferentes sites com o recurso Live até as células ou tecidos estão localizado. Concentre-se na amostra ou usar o foco automáticomanualmente. Use o Snap para capturar a imagem.
Nota: Para ajudar a localizar a amostra em uma ampliação alta, use a caixa de diálogo placa de aquisição e controle para ajustar o tamanho do passo em qualquer lugar de 5 a 100 µm para encontrar a amostra se Autofocus não pode. - W1 DAPI na guia, clique em Auto expor. Assegurar que a imagem da Placa de aquisição Snap - DAPI é nítida.
- No menu principal, clique no jornal para revelar o suspenso. Selecione executar diário....
- Clique duas vezes no jornal Deslize região aquisição instalação para iniciá-lo. Quando aparece a caixa de diálogo de Configuração deslize aquisição de região , selecione continuar.
- Quando instruído, selecione corretamente o DAPI Scan para selecionar a imagem de slide de baixa ampliação. Imprensa Okey. Da mesma forma, destaca-se a placa de aquisição Snap - DAPI quando pediu para receber a bola de aquisição de placa. Clique no Okey.
- Quando aparece a caixa Create ou regiões de carga , use a ferramenta região retangular para selecionar (a) região (ões) de interesse em examinar o DAPI (Figura 6). Pressione continuar.
Nota: A área máxima que pode ser selecionada por 40 X é 45 colunas por 35 linhas. Se várias regiões de interesse são selecionadas, as regiões serão redimensionadas para a caixa com a maior área. As caixas podem ser reposicionadas após o redimensionamento. - Selecione não sob a caixa de diálogo de Regiões de carga .
Nota: Selecionando Sim carrega previamente salvo regiões para lotes de slides com a mesma região de locais de interesse. - Usando a ferramenta de localização , destacar a maior região de interesse e pressione continuar sob a caixa de diálogo Selecionar região . Quando aparece a caixa de regiões de confirmar , selecione continuar. Decida se a região precisa ser salvo quando aparece a caixa de diálogo Salvar regiões .
- A próxima caixa de diálogo, Espaçamento de Site, permite ao usuário escolher o mosaico, que resulta em uma imagem sem sobreposição, ou sobreposição de 10%. Escolha uma opção e clique em Okey. Os autores escolheu mosaico.
- Três caixas de diálogo irão aparecer agora. Caixa de Instalação completa pode ser demitida, pressionando a continuar. Abre a caixa de Configuração de Site de aquisição por 40 X (Figura 7A) por agora desde que faz claro quantas colunas e linhas precisam ser digitado na caixa de Configuração de placa de aquisição (Figura 7B). Abre a caixa de WellPositions (Figura 7) que aparece no canto inferior esquerdo para a duração da imagem.
- Na caixa de Configuração de placa de aquisição , em poços de visita, o mesmo número de poços, como regiões de interesse previamente selecionado deve ser realçado (Figura 7).
Nota: Os autores desenvolveram este protocolo para um máximo de nove regiões de interesse por slide. Se houver mais, simplesmente aumente o número de colunas ou linhas na aba placa - SlideScanning para refletir o número de regiões de interesse. Esses poços não representam espacialmente a localização das regiões de interesse de qualquer maneira, mas o número apropriado deve ser realçado (botão esquerdo). - Na caixa de Configuração de placa de aquisição , vá para a guia de locais a visitar insira sugerido de colunas e linhas (Figura 7B). Lembre-se de clicar em sites de telha.
- Para eliminar as conjecturas de encontrar os sites que contém a amostra, pressione a Placa de adquirir sob a guia Resumo para posicionar o estágio na região pre-selecionada de interesse. Uma vez que a câmera panelas sobre um site que contém células, pressione Cancelar no canto inferior direito para impedir a aquisição de imagem. Certifique-se de fase está agora localizado sobre a amostra.
- Otimize as configurações de comprimento de onda para garantir que um foco e uma imagem de alta gama dinâmica é obtido. Quando satisfeito, clique na guia Resumo e Adquirir a placa.
7. análise de imagens
- Selecione o módulo desejado e personalizado.
Nota: Os autores escolheu o módulo Multi-Wavelength célula marcando para demonstrar uma segmentação automatizada de slides de tumor no cérebro. - Execute a análise de todos os sites. Se os parâmetros de análise são as mesmas para muitos slides, a análise pode ser incluída na caixa de Configuração de placa de aquisição sob a guia Após a aquisição .
- Quando a análise estiver concluída, exclua qualquer sites de má qualidade de imagem para não influenciar os resultados. Desde que o objetivo desta análise é quantificar microglia e macrófagos em células de parênquima do tumor, selecione a região de interesse dentro de bordas a amostra tumor. Além disso, exclua os sites que estão fora de foco e/ou contem tecido dobras ou bolhas e lágrimas no tecido. Para uma demonstração, veja os Resultados do representante. Como alternativa, usar a Pontuação de foco Laser gerado para cada site, dependendo da amplitude da reflexão do laser como um limiar abaixo quais os sites que estão a ser excluído (o limite é personalizável e depende de parâmetros como o tempo de exposição e a tipo de mídia usado).
Nota: As imagens de alta ampliação tornam possível excluir especificamente estruturas, tais como os grandes vasos e áreas de necrose, se necessário. Também é possível incluir apenas as áreas de interesse, tais como aquelas que são hipercelular. Desta forma, a especificidade e a exatidão das análises podem ser aumentados.
Resultados
As imagens podem ser visualizadas dentro do software WHCAS. A Figura 8 mostra as miniaturas de alta magnificação de todos os sites dentro da região definida de interesse. Examine cada site para identificar os que deveriam ser excluídos da análise (exemplos são mostrados em baixa e alta ampliação na Figura 8 e Figura 9, respectivamente). Por exemplo, Site 149 está fora de foco (Figura 9A), Site 219 tem bolhas (Figura 9B) e 54 Site contém uma dobra (Figura 9) e devem ser excluídos. É típico para excluir a 10-15% de todos os sites de criação da imagem. No exemplo fornecido, 15,3% dos sites foram excluídos (25/300 tinha bolhas, 17/300 estavam fora de foco, 3/300 foram dobradas e 1/300 estava na borda). Figura 10A é uma imagem representativa escolhida para análise (sua miniatura de baixa ampliação correspondente é mostrada na Figura 8). Aqui, as sobreposições correspondentes geradas pelo módulo de comprimento de onda multi célula marcando demonstram os resultados da segmentação automatizada executada no Site 59, personalizado às especificações dos autores (largura mínima de núcleos = 2,5 µm, largura máxima = 7,5 µm, e intensidade acima de fundo local = 35 graylevels; A largura mínima das células CD11b-positivo = 4 µm, largura máxima = 18 µm, manchado área mínima = 15 µm2e intensidade acima de fundo local = 310 graylevels; e a largura mínima das células CD45-positivo = 4 µm, largura máxima = 18 µm, manchado área mínima = 15 µm2e intensidade acima de fundo local = 50 graylevels). Após a segmentação, os dados quantitativos da proporção de células coloração positiva para cada marcador (6,2 ± 5,1% e ± 3,8% de 2.1 de CD11b+ e CD45+ células, respectivamente), ambos os marcadores (3,5 ± 2,1% CD11b+CD45+ células), sem marcadores (86,6 ± 9,0% CD11b–CD45 células– ; Figura 10B), significa manchada área (40,0 ± 9,2 µm e 36.7 ± 7,6 µm de CD11b+ e CD45+ células, respectivamente; Figura 10) e significa a intensidade da fluorescência (408,9 ± 40,3 fluorescência relativa unidades e 373,9 ± 38.1 fluorescência relativo de CD11b+ e CD45+ células, respectivamente; Figura 10) pode ser obtido.
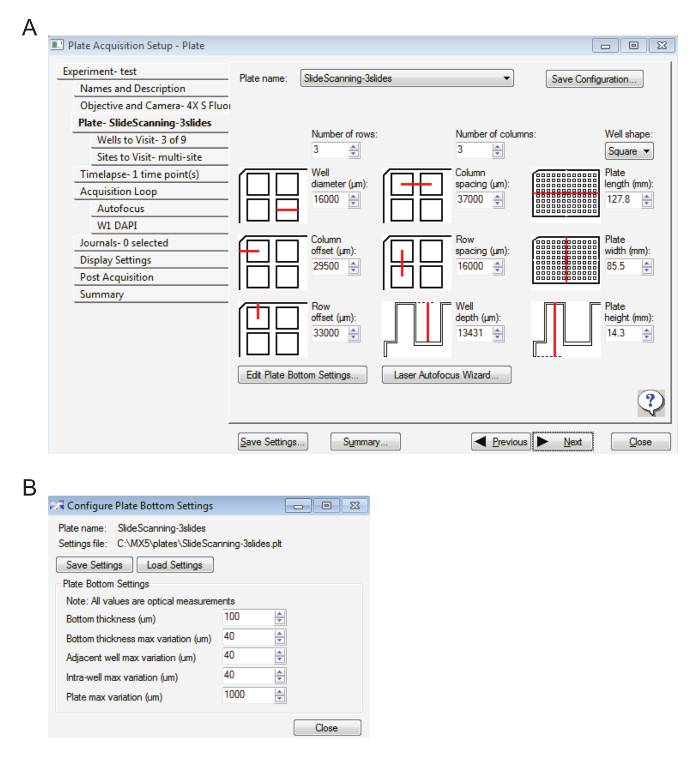
Figura 1: configurações para aquisição de slide em baixa ampliação. A. esta imagem exibe as configurações da placa. B. aqui, as configurações de fundo de prato são exibidas. Clique aqui para ver uma versão maior desta figura.
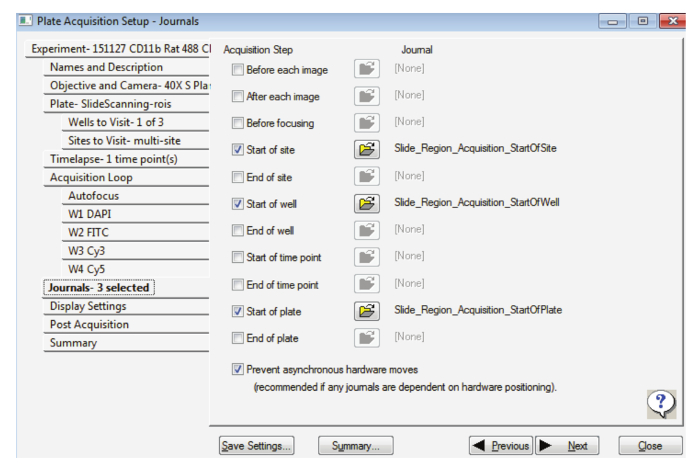
Figura 2: configurações para aquisição de slide em alta ampliação. Esta figura mostra a seleção dos periódicos adequados. Clique aqui para ver uma versão maior desta figura.
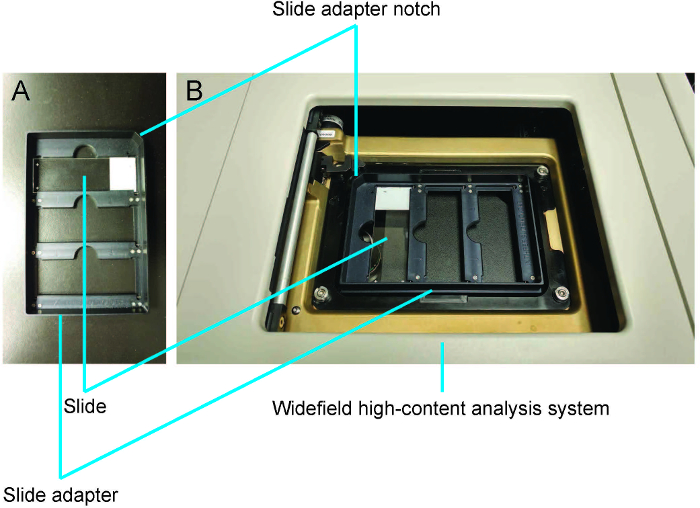
Figura 3: demonstração de como carregar slides usando o adaptador slide. A. O slide é colocado a lamela para baixo com a etiqueta no lado do entalhe de adaptador de slide. B. O adaptador de slide é carregado no sistema de análise de alto teor widefield. Clique aqui para ver uma versão maior desta figura.
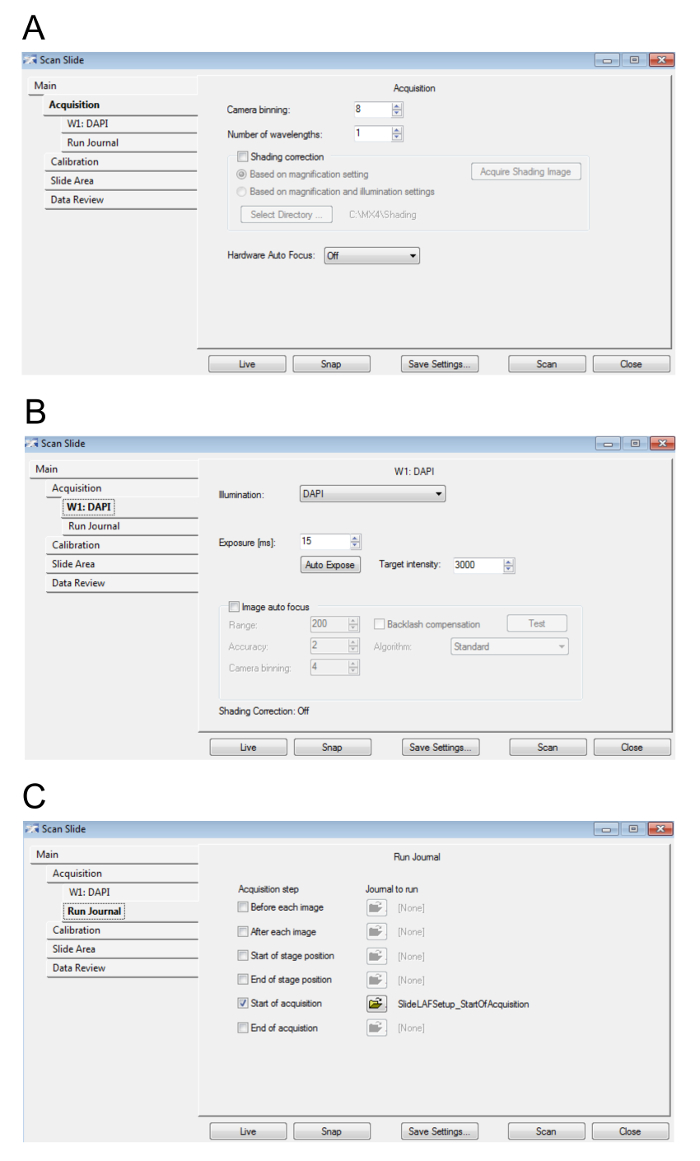
Figura 4: configurações de visualização slide. A. esta figura exibe os parâmetros para a aquisição. B. aqui, os valores para as configurações de comprimento de onda de Hoescht/DAPI são exibidos. C. esta imagem mostra as configurações do diário. Clique aqui para ver uma versão maior desta figura.
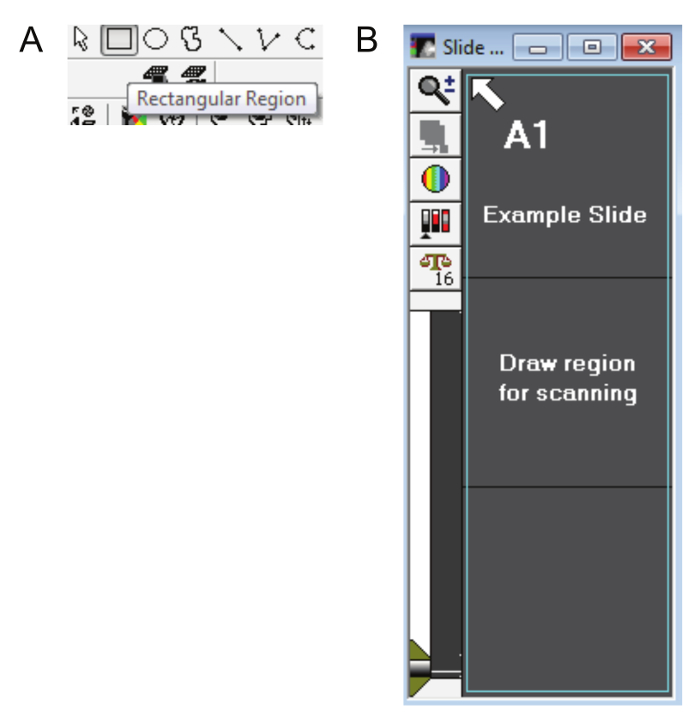
Figura 5: desenho de uma região do slide. A. A ferramenta região retangular (outras ferramentas de desenho não podem ser usadas) é usado para selecionar a área de varredura de visualização e criar regiões de interesse na digitalização DAPI. B. O contorno da cerceta nesta figura mostra como toda a área do slide (incluindo o rótulo) deve ser seleccionada. Clique aqui para ver uma versão maior desta figura.

Figura 6: criação de regiões de interesse sobre a verificação de pré-visualização. A visualização ou DAPI scan representa a área do slide inteiro. Neste exemplo, existem três seções de tecido de cérebro serial (sólidos brancos retangulares contornos). A área acima estas seções representa os rótulos circulares no slide. Arranjos de seção diferente não limitará a seleção subsequente de regiões de interesse. A região de interesse neste exemplo particular é mostrada com um contorno retangular branco tracejado. O scalebar = 1 mm. clique aqui para ver uma versão maior desta figura.
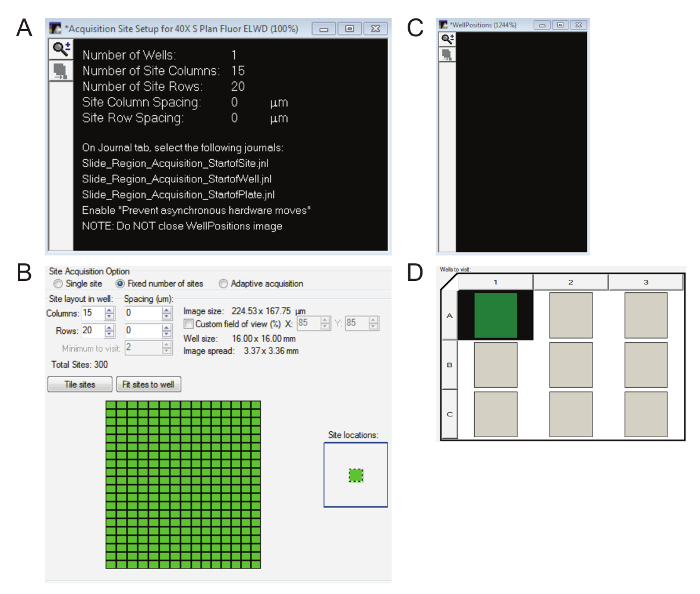
Figura 7: windows essencial para alta magnificação de imagem aquisição. A. janela de configuração de Site a aquisição irá mostrar quantas colunas e linhas são dentro da região ou regiões de interesse. B. assegurar o número correto de colunas e linhas indicadas na Figura 7A são inseridas na caixa configuração de aquisição da placa e os sites estão lado a lado. C. é necessário manter a janela de WellPositions aberto para a duração da aquisição da imagem, mesmo que seja em branco. D. deve salientar-se o número apropriado de regiões de interesse. Clique aqui para ver uma versão maior desta figura.

Figura 8: imagem representativa da região de interesse. Cada site pode ser exibido em uma miniatura composta da região de interesse. Os sites representativos que foram excluídos (marcados com caixas vermelhas) e um exemplo de um site incluído (marcado com uma caixa verde) é mostrado em uma maior ampliação. Recomenda-se examinar cada site para garantir que é de qualidade suficiente para a análise. O scalebar = 1 mm. clique aqui para ver uma versão maior desta figura.
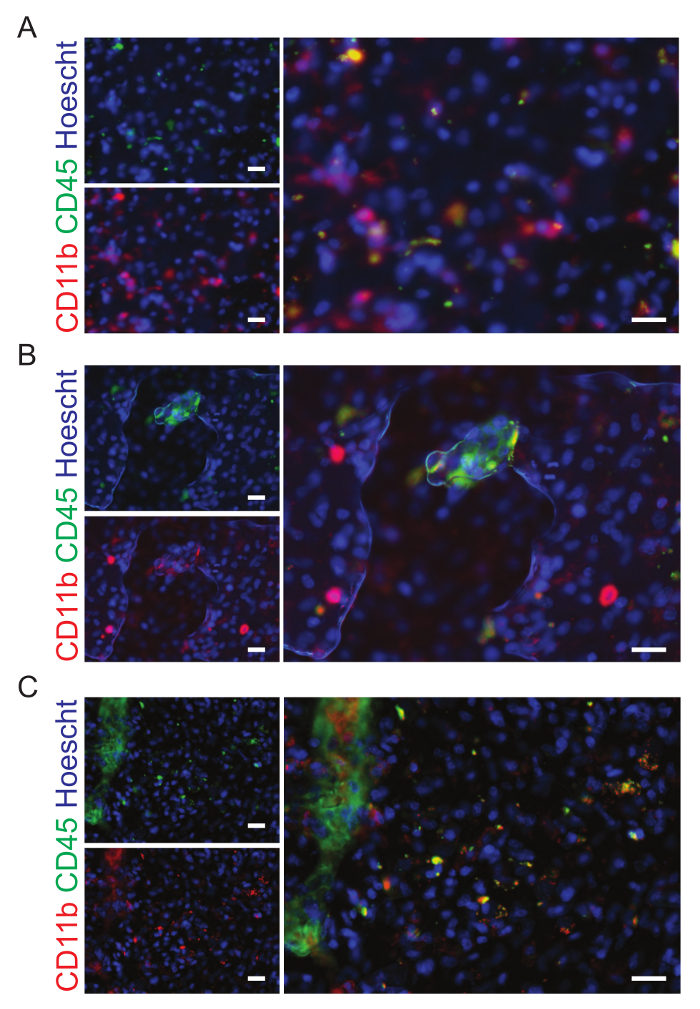
Figura 9: exemplos de sites que devem ser excluídos da análise. A. as seções do tumor de cérebro humano tem sido manchadas com CD11b (verde) e CD45 (vermelho), marcadores de micróglia e macrófagos. Esta imagem está fora de foco e foi excluída da análise. B. as bolhas estão presentes nesta imagem que foi excluído da análise final. C. Este site foi excluído da análise por causa da dobra do tecido. As barras de escala são 20 µm. clique aqui para ver uma versão maior desta figura.
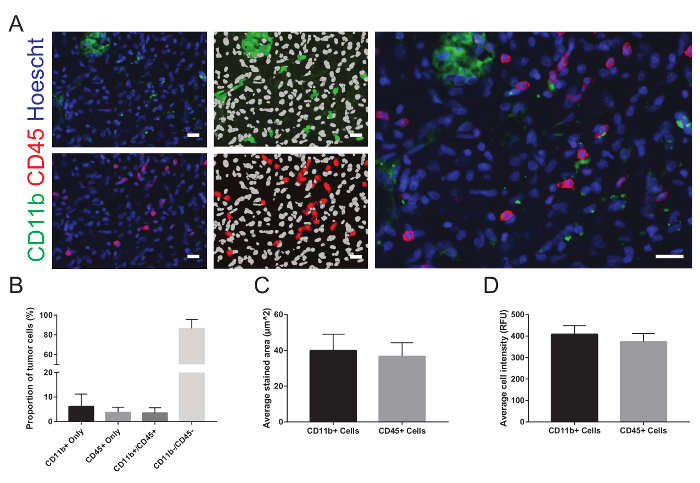
Figura 10: imagens representativas e análise. A. cada imagem é exibida ao lado as sobreposições que representa os resultados da segmentação automatizada. Na sobreposição, núcleos são exibidos em branco e células coradas positivamente são mostradas em verde para CD11b e vermelho para CD45. Depois excluindo os sites de inadequada qualidade da análise e combinando os resultados de todos os restantes locais, B. as proporções médias do número total de células em cada site que mancharam positiva para cada marcador e co rotulada para ambos os marcadores, C. a média manchadas de área e D. a intensidade média da célula são representados graficamente. RFU = unidades de fluorescência relativo. Os dados são expressos como média ± desvio-padrão. As barras de escala são 20 µm. clique aqui para ver uma versão maior desta figura.
Discussão
Um comum problema impedindo a eficiência da pesquisa em ciências biológicas é o desenvolvimento de protocolos para a quantificação de imparcial, exata e precisa dos tecidos fluorescente-etiquetadas e suas estruturas dentro. Uma quantidade significativa de tempo e esforço é dirigida no sentido de encontrar maneiras de analisar lâminas de tecido, uma vez que eles têm sido fotografados. Muitos métodos existentes fornecem algoritmos para que os usuários recriar dentro de programas12,13,14. Esses métodos são aceitáveis, mas a importância deste relatório é que ele permite que o usuário facilmente e rapidamente estabelecer uma aquisição de imagem holística e protocolo de análise, se o usuário tem acesso a um WHCAS. Ensaios que diferenciam os tipos de células e quantificar várias estruturas e processos, análise de ciclo celular e translocação nuclear, por exemplo, já estão disponíveis no software associado.
A configuração envolve alguns passos críticos. Em primeiro lugar, os parâmetros espaciais do slide são definidos como se fosse um prato. Em segundo lugar, um exame de visão baixa ampliação é criado de que regiões de interesse são selecionadas para a imagem latente de alta ampliação. Por último, sites que afetam a precisão e a precisão das análises subsequentes são excluídos. A maior limitação dessa técnica é que sua aplicabilidade depende se o usuário tem acesso a um WHCAS. No entanto, com a crescente necessidade de sistemas de análise de alto teor, muitas instituições estão fornecendo estas para seus pesquisadores para permanecer competitiva15. Solução de problemas é necessário mais comumente quando cortes histologicos não têm a mesma espessura. Se várias regiões de interesse são selecionadas, alguns estarão em foco, enquanto os outros não vão. Idealmente, durante o corte, o usuário cuidaria para criar amostras homogêneas. No entanto, se as amostras são inconsistentes, o foco sobre o comprimento de onda nuclear mancha (ou fluoróforo mais brilhante do usuário), que é usada para foco, pode ser reajustado para cada região fora do foco de interesse e até mesmo para cada campo de visão. Como os outros comprimentos de onda são simplesmente offset do comprimento de onda de mancha nuclear, apenas neste comprimento de onda precisa de reajuste.
Neste relatório, detalhamos como digitalizar e analisar slides usando um WHCAS e software associado. O módulo de comprimento de onda multi célula marcando permite ao usuário contar automaticamente qualquer marcadores nucleares ou citoplasmáticos que são rotulados fluorescente. Após ajustar as configurações de foco e definição das características celulares, tais como largura e área para personalizar o módulo no tecido cuja imagem, não há necessidade de intervenção do usuário obter a imagem de slides e dados quantitativos. Até três slides pode ser fotografada em um momento e várias regiões de interesse podem ser definidas. Esta função permite protocolo WHCAS os usuários que precisam analisar slides aproveitar personalizável, multiuso, automatizado de fluxos de trabalho que não exigem pouca ou nenhuma otimização e podem ser aplicados em todos os projetos no futuro que envolvem a análise histológica do tecido.
Divulgações
Os autores têm sem interesses financeiros dos produtos descritos neste manuscrito e têm mais nada para divulgar.
Agradecimentos
Este projeto foi financiado por uma concessão do institutos canadenses de pesquisa em saúde e Alberta inova - saúde soluções/Alberta Cancer Foundation. Os autores desejam reconhecer a unidade de regeneração na instalação de núcleo de neurobiologia para o uso de seus equipamentos, o trabalho de Paula Gedraitis na construção da Fundação sobre a qual foi viabilizada slides digitalização sobre o WHCAS e o criador dos produtos mencionados neste artigo, dispositivos moleculares.
Materiais
| Name | Company | Catalog Number | Comments |
| ImageXpress MicroXLS | Molecular Devices | NA | Apparatus for image acquisition |
| MetaXpress 5.1 | Molecular Devices | NA | Associated software for ImageXpress MicroXL (runs on a PC with the Windows operating system). |
| Slide adapter | Molecular Devices | NA | Metal slide holder that fits into ImageXpress MicroXL |
| Slide_Region_Acquisition_revA.jzp | Molecular Devices | NA | The journal can be obtained from metamorph.moleculardevices.com/forum/showthread.php?tid=218&highlight=slide or from contacting a Molecular Devices representative |
| Slide_Region_Acquisition _Setup.JNL | Molecular Devices | NA | Select this journal in Step 6.6. |
Referências
- Hua, Y., Shun, T. Y., Strock, C. J., Johnston, P. A. High-content positional biosensor screening assay for compounds to prevent or disrupt androgen receptor and transcriptional intermediary factor 2 protein-protein interactions. Assay and Drug Development Technologies. 12 (7), 395-418 (2014).
- Rishal, I., et al. WIS-NeuroMath enables versatile high throughput analyses of neuronal processes. Developmental Neurobiology. 73 (3), 247-256 (2013).
- Kanungo, J., Lantz, S., Paule, M. G. In vivo imaging and quantitative analysis of changes in axon length using transgenic zebrafish embryos. Neurotoxicology and Teratology. 33 (6), 618-623 (2011).
- Schurmann, C., et al. Analyzing illumina gene expression microarray data from different tissues: methodological aspects of data analysis in the MetaXpress Consortium. PLoS ONE. 7 (12), e50938 (2012).
- Vogt, A., Codore, H., Day, B. W., Hukriede, N. A., Tsang, M. Development of automated imaging and analysis for zebrafish chemical screens. Journal of Visualized Experiments. (40), e1900 (2010).
- Bravo-San Pedro, J. M., et al. High-throughput quantification of GFP-LC3+ dots by automated fluorescence microscopy. Methods in Enzymology. , 71-86 (2017).
- Varga, V. S., et al. Automated multichannel fluorescent whole slide imaging and its application for cytometry. Cytometry Part A. 75 (12), 1020-1030 (2009).
- Narayan, P. J., et al. Assessing fibrinogen extravasation into Alzheimer's disease brain using high-content screening of brain tissue microarrays. Journal of Neuroscience Methods. 247, 41-49 (2015).
- Du, Y., Budman, H. M., Duever, T. A. Segmentation and quantitative analysis of apoptosis of Chinese hamster ovary cells from fluorescence microscopy images. Microscopy and Microanalysis. 23 (3), 569-583 (2017).
- Mueller, J. L., et al. Quantitative segmentation of fluorescence microscopy images of heterogeneous tissue: application to the detection of residual disease in tumor margins. PLoS One. 8 (6), e66198 (2013).
- Donnelly, D. J., Gensel, J. C., Ankeny, D. P., van Rooijen, N., Popovich, P. G. An efficient and reproducible method for quantifying macrophages in different experimental models of central nervous system pathology. Journal of Neuroscience Methods. 181 (1), 36-44 (2009).
- Kozlowski, C., Weimer, R. M. An automated method to quantify microglia morphology and application to monitor activation state longitudinally in vivo. PLoS One. 7 (2), e31814 (2012).
- Fish, K. N., Sweet, R. A., Deo, A. J., Lewis, D. A. An automated segmentation methodology for quantifying immunoreactive puncta number and fluorescence intensity in tissue sections. Brain Research. 1240, 62-72 (2008).
- Zock, J. M. Applications of high content screening in life science research. Combinatorial Chemistry & High Throughput Screening. 12 (9), 870-876 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados