Method Article
Uma plataforma Microfluidic de ultra alta taxa de transferência para o sequenciamento do genoma de célula única
Neste Artigo
Resumo
Sequenciamento de célula única revela a heterogeneidade genotípica em sistemas biológicos, mas as tecnologias atuais não têm a produtividade necessária para a criação de perfil profundo de função e composição da Comunidade. Aqui, descrevemos um fluxo de trabalho microfluidic para sequenciamento > 50.000 monocelulares genomas de diversas populações de células.
Resumo
Tecnologias de sequenciamento sofreram uma mudança de paradigma de em massa de célula única resolução em resposta a um entendimento em evolução do papel da heterogeneidade celular em sistemas biológicos. No entanto, sequenciamento de célula única de grandes populações tem sido dificultado por limitações no processamento de genomas para sequenciamento. Neste trabalho, descrevemos um método de sequenciamento de genoma de célula única (SiC-seq) que usa gota microfluídica para isolar, amplificar e código de barras os genomas de células únicas. Encapsulamento de célula em microgels permite a purificação compartimentada e tagmentation do DNA, enquanto uma fusão microfluidic eficientemente pares cada genoma com um código de barras exclusivo do oligonucleotide de célula única, permitindo > 50.000 células únicas a ser sequenciado por execução. Os dados de sequenciamento é demultiplexed por código de barras, gerando grupos de leituras provenientes de células únicas. Como um método de alta produtividade e baixa-viés de sequenciamento de célula única, SiC-seq permitirá uma ampla gama de estudos genômicos dirigidos a populações de diversas células.
Introdução
O genoma serve como um modelo de identidade celular e função, que contém a totalidade de um organismo da codificação de potencial. Uma compreensão da biologia celular no nível do genoma pode explicar a diversidade fenotípica observada dentro de populações de células heterogêneas. Esta heterogeneidade é aparente em sistemas biológicos e tem grandes implicações para a saúde humana e doença. Por exemplo, variações de número cópia genética entre as células do tumor estão ligadas à evolução e propagação do câncer1,2. Em infecções bacterianas, Ilhas de patogenicidade presentes em uma pequena fração dos genomas podem ser transferidas horizontalmente em provocar a proliferação de bactérias resistentes a antibióticos3,4. Um desafio principal no estudo de genomas a nível de célula única é as baixas quantidades de DNA disponível, bem como a necessidade de analisar milhares de células para a completa diversidade de genótipos da amostra. Por estas razões, limitações no throughput experimental tem dificultado a eficácia dos estudos de célula única, resultados para as células mais abundantes de polarização. Técnicas de isolamento de célula única como fluxo classificação5,6, pinça óptica7, embedment em granel géis8e microfluídica9 são capazes de processar centenas de células para sequenciamento; no entanto, isso representa apenas uma pequena fração da maioria das amostras. Um método para o sequenciamento do genoma de célula única com substancialmente maior throughput permitiria mais profunda e mais completa caracterização das populações de células, assim, elucidar o papel da diversidade genotípica dentro dessas comunidades.
Gotícula microfluídica permite a manipulação da elevado-produção de células e reagentes biológicos dentro de milhões de reatores picoliter-escala. Até à data, microdroplet tecnologias têm sido usadas para estudar os padrões de expressão diferencial entre células de tecidos heterogêneos de11,10,12, profundamente sequenciar moléculas longo13,14 ,15e conduta imunoprecipitação de cromatina sequenciamento (ChiP-seq) análises em células únicas16. Com efeito, microdroplets são capazes de operações de elevado-throughput, compartimentadas, tornando-os passíveis de aplicações em genómica de célula única. O desenvolvimento desta tecnologia apresenta seus próprios desafios tecnológicos exclusivos, no entanto. Células devem ser lysed, purificadas e amplificadas com viés mínima, para populações de células uniformemente amostra. Além disso, ao contrário de polyadenylated transcrições de mRNA em células de mamíferos, não há nenhum motivo molecular comparável no genoma para facilitar a captura do ácido nucleico alvo. Por estas razões, o sequenciamento do genoma de célula única tem sido difícil de implementar em plataformas microdroplet.
Neste trabalho, nós fornecemos um protocolo detalhado de nossa abordagem anteriormente relatados monocelulares microfluidic capaz de sequenciamento de genomas de dezenas de milhares de células em uma única experiência de17. Com esta tecnologia, chamada SiC-seq, células bacterianas são encapsuladas em hidrogel mícron-escala e lysed individualmente, tagmented e fundiu-se com um microdroplet que contém um código de barras do oligonucleotide exclusivo, que é emendado no DNA genômico da célula através de um sobreposição de simples extensão cadeia da polimerase (PCR). O hidrogel serve como recipientes isolados em que DNA genômico de alto peso molecular é estericamente envolto, permitindo que as moléculas menores tais como detergentes e enzimas líticas para acessar e purificar DNA barcoding18antes de. Este protocolo processos > 50.000 células únicas em questão de horas, resultando em uma biblioteca de código de barras pronta para sequenciamento. Após o sequenciamento, as leituras são demultiplexed de acordo com sua sequência de célula única de código de barras, resultando em um conjunto de dados composto por milhões de leituras, cada um com um índice de celular.
Protocolo
1. fabricação de dispositivos microfluídicos
- Preparar o microfluidic desenhos de máscara usando o software de desenho assistido por computador (CAD) (fornecido como. DWG; Ver Arquivos suplementares). Tenho esses desenhos impressos pelo vendedor com 10 µm resolução sobre um filme de placa de circuito.
Nota: Para dispositivos microfluídicos em várias camadas, as máscaras correspondentes contêm marcas de alinhamento. -
Para cada dispositivo, fabrica o molde mestre SU-8 (figura 1A) como segue.
- Prepare uma bolacha de silicone diâmetro 3 em derramando-se aproximadamente 1 mL de fotorresiste 3025 SU-8 no centro da bolacha. Fixe a bolacha sobre o aplicador girar chuck aplicando sucção.
- Consulte a tabela 1 para obter uma listagem das espessuras camada e girar a velocidades para cada dispositivo. Para todos os dispositivos, começar o revestimento de rotação com 30 s a 500 rpm, seguido por 30 s a velocidade indicada.
- Retire o wafer de silício-SU-8 do aplicador girar e macio Asse-o em uma chapa que defina a 135 ° C por 30 min. permitir a bolacha arrefecer à temperatura ambiente após o cozimento.
- Expor o wafer de silício-SU-8 com a máscara de microfluidic apropriado sob um colimado 190 mW, 365 nanômetro UV LED por 3 min.
- Após a exposição, difícil asse a bolacha sobre uma chapa definida a 135 ° C por 1 min. Após esta etapa de cozimento, permita a bolacha arrefecer à temperatura ambiente.
- Para um dispositivo de camada única microfluídicos, pule para a etapa 1.2.7. Para um dispositivo microfluidic multi-camadas, repita as etapas 1.2.1 - 1.2.5 para a segunda camada do fotorresiste (figura 1B).
- Após o primeiro Asse difícil por um dispositivo de camada única (ou a segunda Asse difícil por um dispositivo de múltiplas camado), desenvolva a bolacha por imersão em um banho de propileno glicol mono-metil éter acetato de (PGMEA) por 30 min.
- Após o desenvolvimento do wafer, use uma garrafa de esguicho que contém PGMEA para lavar a bolacha. Enxague a bolacha com uma garrafa de esguicho que contém isopropanol antes de colocá-lo em uma placa de 135 ° C por 1 min secar.
- Coloque a bolacha (doravante referida como o mestre) em uma placa de Petri para fundição com polydimethylsiloxane (PDMS).
-
Com o mestre preparado na etapa 1.2, proceda para executar a fabricação de dispositivo com um casting de PDMS.
- Prepare o PDMS, combinando um base de silicone com um agente de cura em uma proporção de 11:1 em massa. Misture a base de silicone e agente de cura à mão com uma vara do stir.
- Desgaseifica o PDMS colocá-lo em uma câmara de desgaseificação e aplicando um vácuo. Permitir que o PDMS desgaseificar até que as bolhas de ar não são mais visíveis (tipicamente 30 min).
- Com cuidado, despeje o PDMS desgaseificado sobre o mestre, para uma espessura de PDMS-camada final de aproximadamente 5 mm. Degas o PDMS novamente para garantir a remoção de quaisquer bolhas de ar.
- Após a desgaseificação, Asse o PDMS e o mestre a 80 ° C para 80 min.
- Cuidadosamente excisar a laje PDMS curada com o mestre cozido usando uma lâmina de barbear. Certifique-se de que todos os cortes são em cima da pastilha de silício.
Nota: Quaisquer cortes feitos fora da pastilha de silício podem resultar em um lábio impedindo uma ligação uniforme. - Socar as entradas e saídas usando um soco de biópsia de 0,75 mm. Remova o pó e os vadios PDMS usando uma fita de embalagem no lado do recurso do dispositivo.
- Antes de plasma tratando o dispositivo, limpe uma lâmina de vidro de 50 x 75 mm enxaguá-lo com isopropanol e secá-lo.
- Para o tratamento de plasma, coloque o slide de laje e vidro PDMS para o bonder de plasma com os recursos voltados para cima. Realize o tratamento de plasma usando 1 mbar O plasma de2 para 1 min. Bond o dispositivo para o vidro deslizar, trazendo a exposta, ou face para cima, dos lados juntos.
- Após o tratamento de plasma, Asse o dispositivo a 80 ° C por 40 min.
- Finalmente, injete fluido de tratamento de superfície de vidro em uma das entradas para processar os canais microfluídicos hidrofóbicas. Certifique-se de todos os canais estão completamente inundados com a solução e repita a injeção para cada dropmaker. Asse o dispositivo Tratado a 80 ° C durante 10 minutos evaporar o solvente em excesso.
2. encapsulamento de células em Agarose Microgels
Nota: Ver Fig. 2A.
- Prepare-se 1 mL de agarose 3% w/v de temperatura de baixo ponto de fusão em tampão Tris-EDTA (TE) 1x. Manter a solução de agarose em um bloco de calor de 90 ° C até imediatamente antes de carregar a seringa.
-
Prepare a suspensão de eritrócitos.
Nota: Este protocolo e seus dispositivos microfluídicos associado foram validados para trabalhar com as células bacterianas, ou de um estoque congelado ou fresca preparação. Células de mamíferos, dependendo do tipo de célula, podem exigir um ajuste das dimensões microfluidic canal para acomodar os maiores tamanhos de célula.- Ressuspender as células em 1 mL de tampão fosfato salino (PBS).
- Conte as células em um hemocytometer ou classificando de fluxo. Para 25 µm microgels com uma taxa de encapsulação de célula alvo de 1 em cada 10, prepare-se 1 mL de suspensão de células em uma concentração final de 2,4 x 107 células/mL.
- Girar as células para baixo a 3.000 x g, durante 3 min. Aspire o sobrenadante e ressuspender as células em 1 mL de 17% v/v densidade gradiente médio (ver Tabela de materiais) em PBS. Mantê-lo no gelo até o carregamento de seringa.
- Carregar uma seringa de 3 mL com óleo fluorado (HFE) que contém um surfactante de glicol (PEG-PFPE) 2% w/w perfluoropoliéter-polietileno, ajustá-lo com uma agulha 27G e colocá-lo em uma bomba de seringa.
Nota: Caber todas as seringas com agulha 27G para o microfluidic etapas neste protocolo. Para reduzir o risco de picada acidental, mantenha tampas em todas as agulhas até começa a operação da bomba. - Carregar a suspensão de células e agarose derretido em seringas de 1 mL, ambos com agulhas de calibre 27-se e colocá-las em bombas de seringa.
- Mantenha a seringa de agarose e bomba quente com um pequeno aquecedor para impedir que a agarose coagulando na seringa e tubulação de entrada. Definido o aquecedor para alto e posicioná-lo para que a superfície de aquecimento é de aproximadamente 10 cm de distância da seringa. Certifique-se de que a temperatura medida na seringa é aproximadamente 80 ° C.
Nota: Os usuários são aconselhados a manter o aquecimento à distância recomendada do aparato bombeamento para reduzir o risco de danificar o equipamento, incluindo a fusão da tubulação. -
Gere 25 µm microgel gotas usando o dispositivo de dropmaking de fluxo de co.
Nota: Ver Figura 3A para um dispositivo esquemático indicando a localização das entradas de reagente e tomada.- Ligar as agulhas de seringa dispositivo microfluidic nas entradas usando pedaços de tubos de polietileno (PE). Antes de inserir os tubos no aparelho, primeiro as bombas para retirar o ar da linha.
- Conectar-se à saída de um pedaço de tubo e coloque a extremidade livre em um tubo de coleta de 15 mL.
- Use as seguintes taxas de fluxo (recomendado) para dropmaking: 800 µ l/h para o HFE 2% w/w PFPE-PEG; 200 µ l/h para a suspensão de eritrócitos em PBS; e 200 µ l/h para o 3% w/v agarose.
- Após o dropmaking, coloque o tubo de coleta a 4 ° C por 30 min garantir a completa gelificação da agarose.
3. quebrando e lavar o Microgels de Agarose
- Remova a camada mais baixa do óleo do tubo da coleção, usando uma seringa de 3 mL, munida de uma agulha 20g, tomando cuidado para não perturbar a camada superior das gotas de agarose.
-
Quebre as emulsões com perfluorooctanol (PFO).
- Adicione 1 mL de 10% v/v PFO em HFE para as gotas de agarose. Pipeta esta solução acima e para baixo por 1 min revestir completamente as emulsões.
Nota: Para todos a microgel lave passos, pipetar as soluções com uma ponta de 1.000 µ l. A suspensão microgel deve aparecer homogêneo e livre de grupos depois de pipetagem-lo. - Gire o tubo cônico a 2.000 x g por 1 min coletar o microgels de agarose. Remover o sobrenadante PFO/HFE por aspiração; os microgels agora está livres de sua camada de surfactante e aparecerá claras.
- Adicione 1 mL de 10% v/v PFO em HFE para as gotas de agarose. Pipeta esta solução acima e para baixo por 1 min revestir completamente as emulsões.
-
Lave o microgels com um surfactante em hexano.
Cuidado: Hexano é um solvente orgânico volátil, e a lavagem na etapa 3.3 deve efectuar-se em uma coifa.- Adicionar 2 mL de 1% v/v sorbitano monooleato não iónico surfactante (ver Tabela de materiais) em hexano para o microgels de agarose. Pipeta e descer 10x para misturar, garantindo a completa separação do microgel pelota.
- Gire o tubo a 1.000 x g por 1 min coletar o microgels. Aspire o sobrenadante para remover a solução de surfactante/hexano.
- Repeti a lavagem de surfactante/hexano.
-
Lave o microgels em um buffer aquoso para remover qualquer solvente orgânico residual.
- Adicionar 5 mL de tampão de TET [detergente não iônico etoxilato 0,1% v/v Jobling (ver Tabela de materiais) em 1 x TE] ao tubo cónico. Pipeta e descer 10x para misturar.
- Gire o tubo cônico a 2.000 x g por 2 min coletar o microgels. Aspire o sobrenadante para remover o tampão de TET.
- Repita o TET lavar 2x.
- Adicione 5 mL de tampão de TE x 1 para o tubo cônico. Pipeta e descer 10x para misturar.
- Gire o tubo cônico a 2.000 x g por 2 min coletar o microgels. Aspire o sobrenadante para remover o tampão TE.
- Repeti a lavagem de TE.
- Verifique se o encapsulamento de célula no microgels sob um microscópio com uma ampliação de 400 X manchando uma alíquota de 10 µ l de géis com 1 x do ácido nucleico mancha (ver Tabela de materiais).
Nota: Microgels aparecerá clara sob o canal transparente enquanto célula que DNA irá fluorescem sob o canal da GFP (497/520 nm de excitação/emissão comprimentos de onda). Uma sobreposição de canais transparentes e fluorescentes mostrará as células no microgels, como indicado na Figura 4A.
4. a Lise das células em Agarose através das enzimas líticas
- Prepare-se 1 mL de uma enzima lítica cocktail usando 800 µ l de tampão TE (1x), 2 µ l da enzima lítica de levedura (5 estoque U / µ l, final de 10 U/mL), 30 µ l de ditiotreitol (estoque de 1 M, final de 30mm), 60 mg de lisozima (pó liofilizado), 15 µ l de EDTA (0,5 M estoque final 7,5 mM), 2 µ l de mutanolysin (estoque de 100 U / µ l, final de 200 U/mL), 2 µ l de lysostaphin (estoque de 10 U / µ l, final de 20 U/mL) e 30 µ l de NaCl (estoque de 1 M, final de 30mm).
- Adicione TE adicional para trazer o volume de 1 mL. Misture a solução vortexing.
- Adicione o inteiro 1 mL da solução de enzima lítica a não mais do que 1 mL do microgels lavado. Misture por pipetagem x 10. Incubar a mistura a 37 ° C para > 2h em um shaker (o máximo é de incubação durante a noite).
5. detergente baseada Microgel tratamento
-
Após a Lise através das enzimas líticas (etapa 4), lave o microgels.
- Spin para baixo a microgels a 2.000 x g por 2 min e aspirar o sobrenadante. As gotas aparecerá branco opaco devido a detritos celulares dispersado na pelota.
- Ressuspender o microgels em 5 mL de tampão Tris-HCl de 10 mM e pipetar acima e para baixo 10 x para misturar.
- Girar a mistura para baixo a 2.000 x g por 2 min e, em seguida, remover o sobrenadante por aspiração.
-
Realizar um tratamento de detergente sobre o microgels.
- Resuspenda o gel em um tampão de lise de lítio Dodecil sulfato de sódio (tampas) (0,5% p/v tampas em 20 mM Tris-HCl) e 60 µ l de EDTA 0,5 M até um volume final de 3 mL.
- Adicione 5 µ l de uma enzima Proteinase K (estoque de 800 U/mL). Pipeta e descer 10x para misturar, então incubar a mistura a 42 ° C em um bloco de calor por 1h solubilizar as membranas celulares e digerir as proteínas.
-
Após o tratamento de detergente, lave o microgels.
- Gire o tubo cônico com o microgels para baixo a 2.000 x g por 2 min. Retire o sobrenadante por aspiração.
- Lave o microgels com 10 mL de Polissorbato de 2% v/v 20 na água. Pipeta e descer 10x para misturar.
- Girar o tubo cônico para baixo a 2.000 x g por 2 min e, em seguida, remover o sobrenadante por aspiração.
- Lave o microgels com 10 mL de etanol a 100% para inativar qualquer enzima residual. Pipeta e descer 10x para misturar.
- Girar o tubo cônico para baixo a 2.000 x g por 2 min e, em seguida, remover o sobrenadante por aspiração.
- Lave o microgels com 10 mL de Polissorbato de 0.02% v/v 20 na água. Pipeta e descer 10x para misturar.
- Girar o tubo cônico para baixo a 2.000 x g por 2 min e, em seguida, remover o sobrenadante por aspiração.
- Repetir o polissorbato 20 lavar 3x. Passe a solução através de um filtro de célula de 100 µm antes a última lavagem para retirar qualquer grandes aglomerações.
- Ressuspender o microgels em 5 mL de 10 mM de tampão Tris-HCl para prevenir a degradação do DNA. O microgels pode ser armazenado a 4 ° C por até 1 semana antes do tagmentation (passo 7).
6. gerando gotículas de código de barras por PCR Digital
- Prepare um estoque de 500-pM de barra da primeira demão (tabela 2) em um tampão de TE em um tubo de baixa vincular 1x. Antes de cada utilização, diluir a primeira demão para um estoque de trabalho das 13:00 e aquecê-lo a 70 ° C por 1 min em um bloco de calor.
- Preparar uma mistura de reação de PCR de 150 µ l usando 75 µ l do mestre de alta-fidelidade começo quente mistura (veja Tabela de materiais) (2 x), 42 µ l de água do PCR-série, 3 µ l de primer DNA_BAR (10 µM de estoque, final de 0,2 µM), 3 µ l de P7_BAR primer (10 estoque µM Final de 0,2 µM), 6 µ l da diluição de código de barras (estoque de 13:00, final 40 fM), 6 µ l de polissorbato 20 (50% v/v estoque, 2% final) e 15 µ l de PEG 6K (50% w/v estoque, 5% final). Misture por pipetagem e descer x 10.
- Preparar uma seringa de HFE-apoiado pelo desenho 200 µ l de HFE óleo em uma seringa e ajustá-lo com uma agulha. Anexar uma seção da tubulação do PE para a agulha e prime a linha com a mão. Inserir a extremidade do tubo de PE a solução de alvo e cuidadosamente desenhar todos os 150 µ l da mistura do PCR para a tubulação do PE e seringa. Carrega a seringa em uma bomba de seringa.
- Carregar uma seringa de 1 mL com óleo fluorado (HFE) que contém um surfactante de glicol (PEG-PFPE) 2% w/w perfluoropoliéter-polietileno, ajustá-lo com uma agulha e colocá-lo em uma bomba de seringa.
-
Gere 25 µm gotas de código de barras usando o dispositivo de dropmaking de fluxo de co.
Nota: Ver Figura 3A para um dispositivo esquemático indicando a localização das entradas de reagente e tomada.- Conecte a entrada de células de um dispositivo de dropmaker de fluxo de co com um pequeno pedaço de solda de chumbo.
- Conecte as seringas carregadas com HFE e PCR mix microfluidic nas entradas dispositivo usando pedaços de tubos de PE, usando a entrada de Agarose derretido para o código de barras mistura PCR. Antes de inserir os tubos no aparelho, primeiro as bombas para retirar o ar da linha.
- Conectar-se à saída de um pedaço de tubo e coloque a extremidade livre em um tubo PCR de 0,2 mL. Use as seguintes taxas de fluxo (recomendado) para dropmaking: 600 µ l/h para o HFE 2% w/w PFPE-PEG e 200 µ l/h para a mistura PCR. Recolha as gotas para os tubos de PCR com cerca de 50 µ l de gotas em cada tubo.
- Após o dropmaking, Retire cuidadosamente a camada inferior de óleo HFE das emulsões usando pontas de pipeta de gel de carregamento e substituí-lo com óleo fluorado FC-40, que contém um surfactante de PFPE-PEG 5% w/w. Ciclo térmico com o seguinte protocolo: 98 ° C por 3 min, 40 x de (98 ° C, durante 10 s, 62 ° C, durante 20 s, 72 ° C por 20 s), 72 ° C por 5 min e mantenha a 12 ° C.
Nota: As gotas de um ciclo térmico podem ser armazenadas a 4 ° C por até 1 dia. -
Verifique se a taxa de amplificação e encapsulamento de código de barras.
- Preparar um ácido nucleico de 1 x mancha (ver Tabela de materiais) em HFE com um surfactante de PFPE-PEG 2% w/w; a mancha é marginalmente miscível em HFE e irá ligar para o DNA nas gotas.
- Adicione 1 µ l de emulsão de um ciclo térmico de código de barras para 10 µ l de óleo de coloração. Incube-os por 5 min à temperatura ambiente.
- Gotículas de imagem por microscopia fluorescente (GFP channel, comprimentos de onda de excitação/emissão do 497/520 nm) em uma ampliação X 200 e contar a taxa de encapsulamento de código de barras. Note que o sinal será discreto: as gotículas contendo códigos de barras amplificados serão fluorescem brilhantemente, Considerando que as gotas vazias aparecerá escuras (Figura 4B).
7. Tagmentation do DNA genômico em gotas
Nota: Ver Figura 2B.
- Preparar 500 µ l de solução de tagmentation usando os reagentes de uma preparação de biblioteca de sequenciamento de próxima geração kit (veja a Tabela de materiais). Use 7 µ l da enzima tagmentation, 250 µ l de tampão de tagmentation e 243 µ l de água da PCR-classe. Misture-os num Vortex e girar a mistura para baixo a recolher. Carregar esta solução em uma seringa de 1ml lastreados em óleo HFE e ajustá-lo com uma agulha.
-
Prepare o microgels para a reinjeção.
- Girar o tubo microgel por 2 min a 2.000 x g e aspirar o sobrenadante. 200 µ l de géis de transferência para o topo de uma seringa de HFE-apoiado com uma carregamento de gel de ponta da pipeta e selar o bocal com um pequeno pedaço de fita.
- Usando um adaptador 3D-impresso centrífuga (ver suplementar arquivos 1 e 2), girar a microgel seringa para 3 min a 3.000 x g.
- Retire o líquido sobrenadante da seringa utilizando uma gel de carregamento ponta da pipeta. Empurre a camada microgel à base do bico da seringa. Encaixe a seringa com uma agulha.
- Carregar uma seringa de 3 mL com óleo fluorado (HFE) que contém um surfactante de glicol (PEG-PFPE) 2% w/w perfluoropoliéter-polietileno, ajustá-lo com uma agulha e colocá-lo em uma bomba de seringa.
- Re-encapsule o microgels em gotículas contendo reagentes tagmentation.
Nota: Ver Figura 3B para um dispositivo esquemático indicando a localização das entradas de reagente e tomada.- Conecte as seringas contendo HFE, mistura de tagmentation e microgels para as entradas de dispositivo microfluidic usando pedaços de tubos de PE. Antes de inserir os tubos no aparelho, primeiro as bombas para retirar o ar da linha.
- Conectar-se à saída de um pedaço de tubo e coloque a extremidade livre em uma seringa de 1 mL vazia com o êmbolo desenhado para a linha de 1 mL.
- Use as seguintes taxas de fluxo (recomendado) para dropmaking: 2.000 µ l/h para o HFE 2% w/w PFPE-PEG, 200 µ l/h para o microgels e 500 µ l/h para a mistura de tagmentation.
- Verifique se a taxa de encapsulação microgel sob um microscópio de luz em uma ampliação X 400. Cerca de 80-90% das gotas deve conter um microgel, conforme mostrado na Figura 4.
- Encaixar a seringa contendo as emulsões tagmentation com uma agulha e incube-lo na posição vertical em um bloco de calor ou forno por 1h a 55 ° C para fragmentar o DNA genômico.
8. célula única Barcoding Microfluidic dupla fusão
Nota: Consulte a Figura 2.
- Prepare as gotas de código de barras para a fusão, substituindo a fração de óleo FC-40 com HFE 2% w/w PFPE-PEG. Com cuidado transferir estas gotas em uma seringa de 1 mL, ajustá-lo com uma agulha e colocá-lo em uma bomba de seringa.
- Carrega a seringa de gotículas microgel incubada e tagmented em uma bomba de seringa.
- Prepare 500 µ l de mistura PCR. Adicionar os reagentes na seguinte ordem para impedir a formação de precipitados: 140 µ l de água do PCR-grau 10 µ l de P5_DNA primer (10 µM de estoque, final de 0,2 µM), 10 µ l de P7_BAR primer (10 µM de estoque, final de 0,2 µM), 50 µ l de PEG 6K (estoque 50% w/v 5% final), 50 µ l de polissorbato 20 (50% v/v estoque, 5% final), 250 µ l de Taq Master Mix (2x) (ver Tabela de materiais), 10 µ l de polimerase isotérmico (ver Tabela de materiais) e 10 µ l de tampão de neutralização (ver Tabela de materiais). Misturá-los pipetando acima e para baixo 10 x e girar a mistura para baixo para coletá-lo. Carregar esta solução em uma seringa de 1 mL lastreados em HFE, ajustá-lo com uma agulha e colocá-lo em uma bomba de seringa.
- Carga três 3 mL seringas com óleo fluorado (HFE) contendo tensoativo 2% w/w perfluoropoliéter-polietileno glicol (PEG-PFPE) se encaixa cada um com uma agulha e colocá-los em bombas de seringa.
- Carregar três seringas de 1 mL com 2 M de NaCl, se cada um com uma agulha e reserve-os.
-
Mesclar as gotas de código de barras, tagmented gotas de genoma, e PCR mix usando o dispositivo de fusão dupla.
Nota: Consulte a Figura 5 para um dispositivo esquemático indicando a localização do reagente e eletrodo de entradas e saída.- Conecte as 3 seringas de NaCl a 2 entradas de eletrodo e pedaços de inletusing único fosso de tubos de PE. Para os eletrodos, aplica pressão para as seringas manualmente até que os eletrodos são completamente preenchidos com solução salina. Depois de encher os eléctrodos, aplique pressão manual a seringa do fosso até o fosso é preenchido. Conecte a extremidade livre do fosso com um pequeno pedaço de solda de chumbo.
- Conecte as 3 seringas HFE montadas em bombas para as 2 entradas de óleo de espaçador e pedaços de inletusing de óleo de dropmaking de tubos de PE. Antes de inserir os tubos no aparelho, primeiro as bombas para retirar o ar da linha.
- Conecte a seringa de mistura PCR, microgel gotas seringa e código de barras cai seringa para suas respectivas entradas usando tubos de PE. Atirar em todos os tubos de reinjeção da gota com uma arma antiestático antes de conectá-los para as agulhas de seringa; o tratamento antiestático reduz o risco de coalescência da gota induzida por cargas estáticas ao tubo de PE.
- Ligue um pedaço de tubo de PE à tomada e coloque a extremidade livre em um tubo PCR de 0,2 mL.
- Conecte a agulha da seringa eletrodo para um inversor fluorescente de catodo frio usando um grampo de jacaré. Conjunto de alimentação do Inversor DC para 2 V.
- Executar o dispositivo duplo fusão com as taxas de fluxo recomendada: 300 µ l/h para as gotas de microgel tagmented, 100 µ l/h para as gotas de código de barras, 1500 µ l/h para o HFE 2% w/w PFPE-PEG (óleo dropmaking), 600 µ l/h para a amplificação de misturar, 200 µ l/h para o HFE 2% w/w PFPE-PEG ( óleo de espaçador de código de barras) e 700 µ l/h para o HFE 2% w/w PFPE-PEG (microgel óleo de espaçador). Recolha as gotas em cadeia da polimerase com aproximadamente 50 µ l de emulsão em cada tubo.
- Antes a ciclagem térmica, Retire cuidadosamente a camada inferior de óleo HFE das emulsões usando pontas de pipeta de gel de carregamento e substituí-los com óleo fluorado FC-40, que contém um surfactante de PFPE-PEG 5% w/w. Ciclo térmico com o seguinte protocolo: 65 ° C por 5 min, 95 ° C por 2 min, 30 x (95 ° C por 15 s, 60 ° C por 1 min, 72 ° C por 1 min), 72 ° C por 5 min e, em seguida, mantenha a 12 ° C.
-
Recupere o código de barras DNA as gotas de um ciclo térmico.
- Juntar as gotas em um tubo de microcentrifugadora e quebrar as emulsões usando 20 µ l de FOP. Vórtice-os por 10 s para misturar.
- Gire o tubo a 10.000 x g por 1 min fracionar a mistura aquoso (topo) e fases de óleo (fundo). Cuidadosamente retire a camada aquosa superior do tubo usando uma pipeta e transferi-lo para um novo tubo de microcentrifugadora. Descarte a fase óleo.
- Purificar o produto PCR de código de barras em uma coluna de rotação de acordo com o protocolo do fabricante e Elui-lo em 20 µ l de tampão de TE x 1.
- Prossiga para o sequenciamento realizado e análise conforme as etapas 9 e 10.
9. Biblioteca preparação e sequenciamento
- Prepare-se a biblioteca de célula única para o sequenciamento seguindo protocolos do fabricante para a seleção de tamanho de fragmento e quantificação.
- Sequência da biblioteca emparelhado-final com química padrão para leitura de 1 e 2 de leitura. Usar o personalizado índice 1 primer I7_READ (tabela 2) para um índice de 15-bp ler, correspondente ao código de barras de célula única.
10. análise de dados de célula única
Nota: Os scripts de Python personalizada para controle de qualidade e análise preliminar dos dados de SiC-seq podem ser baixados de https://www.github.com/AbateLab/SiC-seq.
- Execute o script "barcodeCleanup.py" para executar o controle de qualidade sobre as leituras de código de barras e exportar os dados de célula única para um banco de dados SQLite. Para um experimento de controle, use este script com o "-Alinhar" sinalizador definido para alinhar as leituras de genomas de referência conhecida.
- Analisar a pureza dos grupos de código de barras (para um experimento de controle), usando o script "purity.py" e confirmar os valores de alta pureza consistentes com Figura 6B.
Resultados
O fluxo de trabalho experimental de SiC-seq contém 3 dispositivos microfluídicos PDMS fabricados usando um procedimento de litografia macia (Figura 1). Um fluxo de co dropmaker (Figura 3A) gera 25 µm de gotículas digital de código de barras para a rotulagem de DNA genômico com um identificador exclusivo de célula única. Os oligonucleotides barcode consistem de uma sequência de degenerado bp 15 ladeado por alças PCR para a amplificação (tabela 2, BAR da primeira demão). Os códigos de barras são diluídos a uma concentração de femtomolar para alcançar o encapsulamento único-molécula, e todas as gotas recebem s de código de barras ou 0 ou 1. As gotículas contendo um código de barras são amplificadas, gerando muitas cópias de amplicons de dupla-hélice de código de barras. Uma mancha de ácido nucleico é usada para verificar a amplificação bem-sucedida e quantificar a taxa de encapsulamento dos fragmentos de código de barras (Figura 4B). Os microgels são geradas pela co fluxo uma suspensão de células bacterianas e um gel de agarose fundido com vazões iguais (Figura 2A). A agarose é preparada em duas vezes a concentração final desejada, como o processo de dropmaking de fluxo de co efetivamente dilui as soluções aquosas por um fator de 2. Como a agarose esfria, ela se solidifica em um diâmetro de 25 µm microgel ocupando o volume esférico de gota.
Uma série de etapas de lavagem e lise purifica o DNA genômico de alto peso molecular no microgels (Figura 2B). Depois de quebrar as emulsões, lavagens aquosas são realizadas em grandes volumes para diluir para rastreamento de solventes orgânicos, que podem inibir os tratamentos enzimáticos a jusante. Os microgels lavados são observados sob um microscópio para verificar a taxa de encapsulação de célula (Figura 4A). Um coquetel de enzimas com ampla atividade lítica é adicionado à suspensão microgel para digerir as paredes celulares de bactérias e micróbios eucariotas19. Um segundo tratamento com Proteinase K e detergente degrada as proteínas e solubiliza os restos celulares.
Tagmentation de DNA purificado é realizado em gotas para evitar contaminação potencial resultante da difusão de fragmentos de DNA de pequenas tagmented entre os microgels18. Um dispositivo de encapsulamento da gota (Figura 3B) compartimentaliza cada microgel com uma enzima de buffer e tagmentation, que simultaneamente fragmentos de DNA dupla-hélice enquanto também "marcação"-com um pré-carregado do oligonucleotide20. Os microgels são carregados para as gotas como partículas perto-embalado, conseguindo taxas de encapsulamento se aproximando 1 microgel para cada gota com poucas parelhas21 (Figura 4).
Na etapa final do fluxo de trabalho microfluidic (Figura 2), um dispositivo executa uma operação de fusão dupla combinação 1 gota de código de barras, 1 gota contendo microgel e a mistura de amplificação em um processo de duas etapas controladas. Primeiro, uma gota contendo o reagente PCR é emparelhada e fundiu-se com uma gota de código de barras na região mostrada em amarelo (Figura 5). Água salgados eletrodos no canal microfluidic produzem um gradiente de alto campo elétrico que desencadeia a fusão da gota. De forma semelhante, a primeira gota mesclada é emparelhada com um droplet microgel e fundiu-se uma segunda vez na região mostrada em vermelho. As gotículas são coletadas e térmica ciclada fora do chip em uma extensão de single-sobreposição (SOE) do PCR. As extremidades complementares sobreposição de código de barras e o DNA genômico de tagmented permitam fusão e amplificação exponencial das construções de código de barras somente corretamente.
Os dados de sequenciamento é primeiro filtrados por uma qualidade de leitura e analisados, em seguida, agrupando-se as leituras de acordo com sua sequência de código de barras de célula única 15-bp. Para um grupo de código de barras ser considerado válido, ele deve conter um número mínimo de leituras; Este limiarização limita a análise de células com uma quantidade útil de dados de sequenciamento e remove o código de barras do PCR-mutante "órfãos" do conjunto de dados. Neste exemplo executar, o mínimo é definido como 7,5 kbps por grupo (50 leituras de 150 bp cada). Um histograma dos Condes de código de barras contra o tamanho do grupo mostra que uma parcela significativa dos grupos de código de barras válido é um pouco acima do tamanho-limite (figura 6A).
Em um experimento de controle onde a composição da comunidade microbiana é conhecida, a pureza e abundância relativa métricas são usadas para avaliar a qualidade de um SiC-seq executar. Aqui, uma comunidade 10-célula sintética, consistindo de 3 bactérias Gram-negativas, 5 bactérias gram-positivas e 2 leveduras é analisada. A pureza de um grupo determinado de código de barras é definida como o número de leituras de mapeamento de genoma mais comuns no grupo dividido pelo número total de leituras do grupo. A grande maioria dos grupos de código de barras têm purezas maiores que 0,95 (Figura 6B). Abundância relativa dos tipos de célula é calculada contando o lê cru e contando os grupos de código de barras, onde os grupos são atribuídos um tipo de célula correspondente ao consenso de suas leituras de membro (Figura 6). A abundância de leituras e grupos de código de barras track em proporções aproximadamente iguais, indicando que as populações de células estão sendo amostradas tal que certas espécies não estão contidas no código de barras desproporcionalmente pequenos ou grandes grupos. Plotando a cobertura agregada de todos os grupos de código de barras de uma única espécie indica uma alta cobertura em todo o genoma inteiro, com poucos ou nenhum regiões de abandono (Figura 7). A uniformidade da cobertura pode ser verificada com uma distribuição de frequência dos valores normalizados de cobertura, com a maioria dos valores centrados em torno da média (Figura 7, de encastrar).
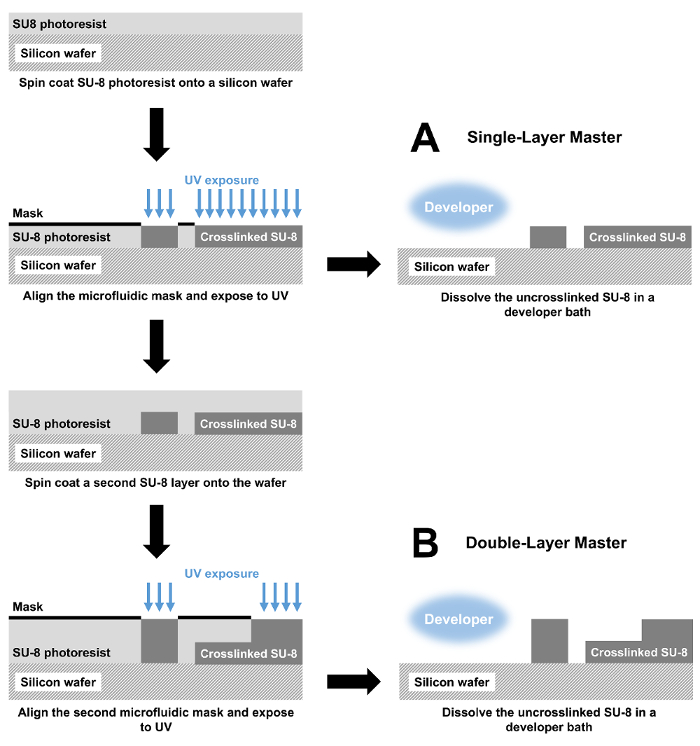
Figura 1 : Fabricação de dispositivos microfluídicos por fotolitografia. Mestre (A) moldes com uma altura de único recurso são fabricados pela spin, uma camada de fotorresiste SU-8 em um wafer de silício. O fotorresiste é então modelado com uma máscara fotolitográfica e luz UV, ligando o exposto SU-8. Finalmente, filmes SU-8 é dissolvida em um banho de desenvolvedor. O molde resultante é usado para converter PDMS que é ligado a uma lâmina de vidro para produzir o dispositivo microfluidic completa. (B) para um dispositivo de camada dupla, a fabricação da mesma forma começa com etapas de revestimento e exposição de rotação. Essas etapas são repetidas, em seguida, criar um dispositivo de duas camadas. Clique aqui para ver uma versão maior desta figura.
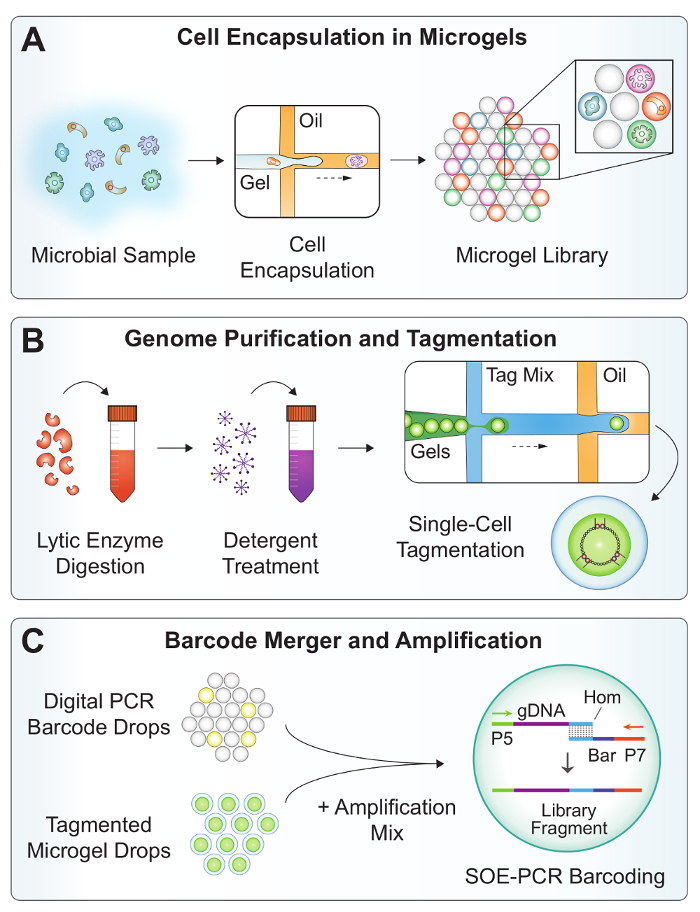
Figura 2: Visão geral do fluxo de trabalho SiC-seq. (A) A suspensão microbiana é co fluiu com agarose derretido em um dispositivo de dropmaker para encapsular células únicas em microgels. (B) os microgels estão sujeitos a uma série de lavagens para purificar o DNA genômico bacteriano. Líticas enzimas digerem as paredes celulares de bactérias gram-positivas e leveduras, e detergente solubiliza os restos celulares. Os microgels são re-encapsulados em gotas para o tagmentation reduzir a contaminação cruzada. (C) a fusão microfluidic combina um código de barras digital do PCR, um genoma de microgel tagmented e uma mistura de amplificação em uma taxa > 1 kHz. Fora do chip SOE-PCR emendas a um código de barras único de célula única no genoma tagmented e seletivamente amplifica totalmente construções de código de barras. Clique aqui para ver uma versão maior desta figura.
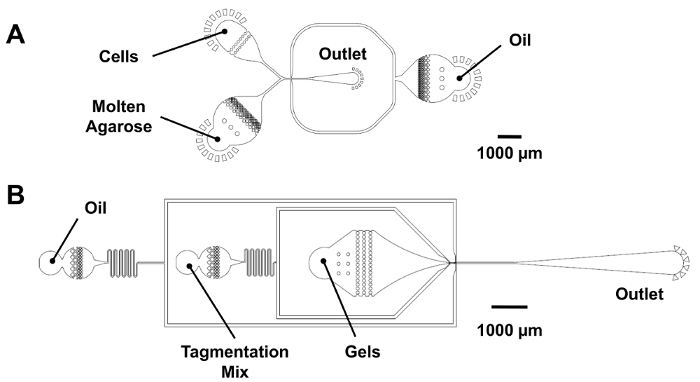
Figura 3: Dispositivos microfluídicos para re-encapsulamento de dropmaking e microgel. (A), este painel mostra um fluxo de co dropmaker (25 µm de altura de recurso). Células e agarose fundido são introduzidos no aparelho com vazões iguais para produzir 25 µm gotas em uma junção de µm µm x 25 25. Para o dropmaking de código de barras digitais, a entrada da célula está conectada, e uma mistura PCR é introduzida na entrada de agarose. (B), este painel mostra um dispositivo de re-encapsulamento microgel (25 µm de altura de recurso). As microgels fluir para uma entrada em forma de funil para manter sua ordenação fechar-embalados e receber um volume de mistura de tagmentation antes do re-encapsulamento em um entroncamento de µm 25 µm x 30. Clique aqui para ver uma versão maior desta figura.

Figura 4 : Micrografias de gotículas e microgels. (A), este painel mostra lavada 25 µm microgels antes da lise enzimática. As bactérias são coradas fluorescentemente para a quantificação da taxa de encapsulação. Estatísticas de carregamento de Poisson ditam que as células devem ser encapsulados em uma taxa de 1 em cada 10 gotas ou menos para minimizar a frequência de eventos de múltiplo-encapsulamento. (B) este painel mostra uma imagem de microscopia de fluorescência de 25 µm de código de barras digital gotículas tratados com uma mancha de ácido nucleico. As gotículas contendo fragmentos amplificados barcode produzem um sinal forte fluorescência. (C) este painel mostra microgels re-encapsulado em gotas de 50 µm. A embalagem-perto do microgels permite taxas de encapsulamento se aproximando 1 gel por gota com poucas parelhas. Clique aqui para ver uma versão maior desta figura.
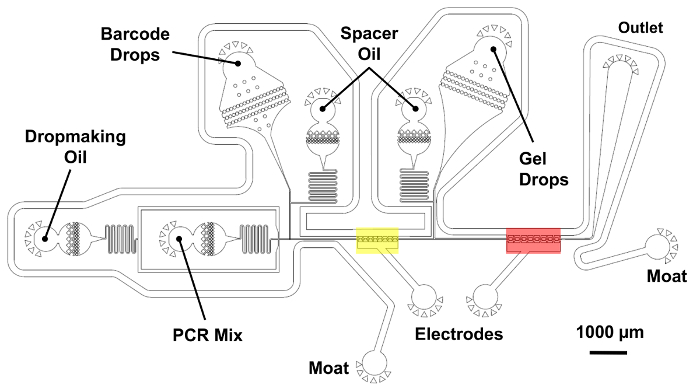
Figura 5 : Dispositivo de fusão dupla Microfluidic para código de barras do genoma de célula única. Uma operação de fusão de duas etapas de pares gotículas de código de barras com genomas de tagmented em uma alta produtividade. Primeiro, uma gota da mistura PCR é gerada e fundiu-se com uma gota de código de barras na região mostrada em amarelo usando eletrodos de água salgados. Em seguida, uma gota contendo um microgel é introduzida e fundiu-se uma segunda vez na região mostrada em vermelho. Entradas de óleo permitem um controle preciso do afastamento entre as gotas de reinjected. A câmara de reinjeção de código de barras e seu óleo espaçador são colocados na camada mais curta de 25 µm, sombreada em azul. Todas as outras características do dispositivo pertencem à camada mais espessa com 45 µm de altura total. Clique aqui para ver uma versão maior desta figura.
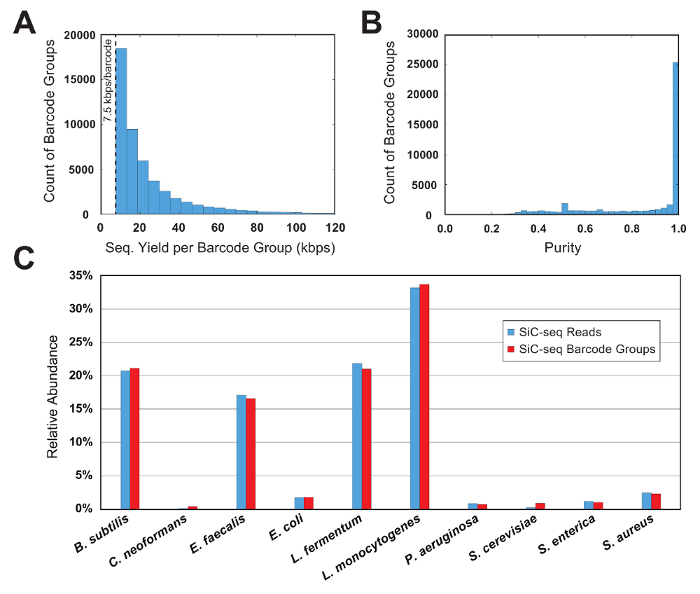
Figura 6 : Métricas de grupo de código de barras para uma comunidade microbiana sintética de 10 células. (A), este painel mostra a distribuição de código de barras de tamanhos de grupo. O número de grupos de um determinado tamanho diminui exponencialmente conforme o tamanho do grupo aumenta. Um limiar mínimo de 7,5 kbps por grupo limita a análise para grupos com uma quantidade suficiente de informações e remove a sequência de PCR-mutante "órfãos". (B), este painel mostra a distribuição do código de barras purezas do grupo. A grande maioria (> 90%) dos grupos são de altíssima pureza (> 95%). (C) este painel mostra a abundância relativa de 10 espécies calculado no nível de grupo de leitura e de código de barras. O 2 contagem métodos produzem resultados semelhantes, indicando que os tamanhos de grupo de código de barras são consistentes em toda a espécie. Clique aqui para ver uma versão maior desta figura.
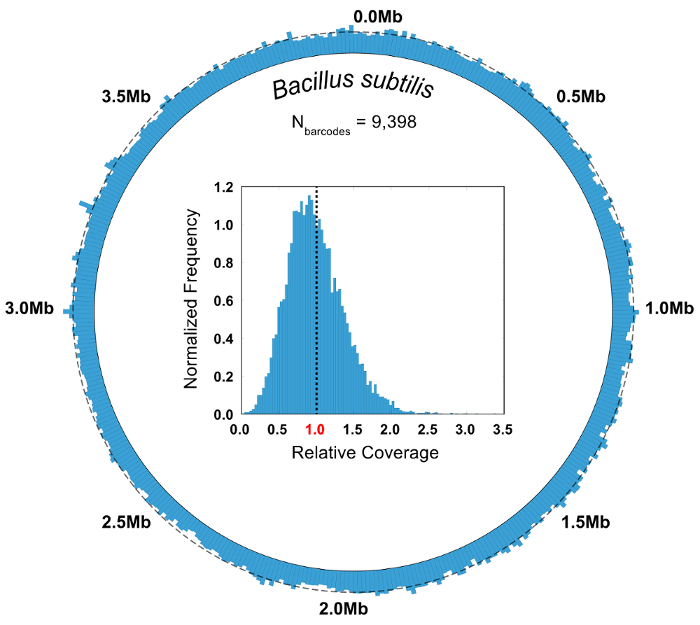
Figura 7 : Agregar cobertura genômica de Bacillus subtilis grupos de código de barras. As leituras de todos os grupos de código de barras de mapeamento para a bactéria b. subtilis (N = 9.398) são agrupados e analisados em conjunto. Um mapa de cobertura circular ilustra a uniformidade da cobertura do SiC-seq diz: com regiões não observáveis desistente. Uma linha tracejada ao redor da circunferência indica a cobertura média (5,55 x). O histograma de inserção das frequências relativa cobertura mostra que a maioria das bases estão cobertas de profundidade perto da média de todo o genoma, representado pela linha tracejada. Clique aqui para ver uma versão maior desta figura.
| Dispositivo | 1 altura de camada (µm) | 1ª camada Spin velocidade (rpm) | Altura de camada 2 (µm) | 2ª camada Spin velocidade (rpm) |
| Dropmaker de fluxo de co | 25 | 4000 | N/A | N/A |
| Re-encapsulante gel | 25 | 4000 | N/A | N/A |
| Fusão dupla | 25 | 4000 | 20 | 5000 |
Tabela 1: parâmetros de fabricação de dispositivo Microfluidic. Esta tabela mostra uma listagem dos dispositivos microfluídicos utilizados na SiC-seq fluxo de trabalho com suas velocidades necessárias para girar fotorresiste revestimento (com base nas especificações do fabricante para 3025 SU-8).
| Rótulo | Sequência (5' > 3') | ||||
| BAR | GCAGCTGGCGTAATAGCGAGTACAATCTGCTCTGATGCCGCATAGNNNNNNNNNNNNNNNTAAGCCAGCCCCGACACT | ||||
| DNA_BAR | CTGTCTCTTATACACATCTCCGAGCCCACGAGACGTGTCGGGGCTGGCTTA | ||||
| P7_BAR | CAAGCAGAAGACGGCATACGAGATCAGCTGGCGTAATAGCG | ||||
| P5_DNA | AATGATACGGCGACCACCGAGATCTACACTCGTCGGCAGCGTC | ||||
| I7_READ | GCCCACGAGACGTGTCGGGGCTGGCTTA | ||||
Tabela 2: Primer de sequências.
Arquivo suplementar 1: Clique aqui para baixar este arquivo.
Suplementar arquivo 2: Clique aqui para baixar este arquivo.
Discussão
O fluxo de trabalho do SiC-seq microfluidic produz dados de sequenciamento do genoma de célula única de milhares de células bacterianas. Códigos de barras Digitas emendados para os genomas de células encapsuladas microgel permitem a em silico deconvolução de dados NGS em grupos de leituras de código de barras, provenientes da mesma célula. Um experimento de controle com uma comunidade microbiana de composição conhecida é necessário para avaliar a pureza dos grupos de código de barras. Uma grande fração dos grupos de baixa pureza indica que a taxa de encapsulação de célula é muito alta ou que não há contaminação gota significativa ocorrendo durante as etapas de processamento microfluidic. De acordo com estatísticas de Poisson, o código de barras e células devem ser encapsuladas em uma proporção de alvo de 1 partícula para cada 10 gotas limitar a taxa de vários eventos de encapsulamento para menos de 5% de todas as gotas de não-vazio. Uma taxa de encapsulação superior isto aumenta as taxas de parelhas exponencialmente, para a verificação da relação de encapsulamento durante o processo de dropmaking é de importância crítica. Os usuários devem ser particularmente cautelosos com o encapsulamento de várias células em uma única microgel porque lê a partir de células diferentes, compartilhando a mesma sequência de código de barras não pode ser bioinformatically separado. No caso 1 celular recebe 2 códigos de barras diferentes, a pureza do grupo de código de barras é afetada, embora as métricas de abundância são distorcidas quando contando por sequência de código de barras.
Contaminação cruzada da gota também pode surgir devido às condições de fusão de qualidade inferior. Durante uma operação bem sucedida, o dispositivo de fusão microfluidic (Figura 5) botija pode emparelhar-se 1 gota de código de barras com 1 microgel e um volume de reagente PCR. Taxas de fluxo non-ideal irão resultar em uma gota de emparelhamento em rácios incorretos: 1 código de barras pode ser emparelhado com 2 microgels, por exemplo. Todos os caudais constantes do protocolo se destinam a ser estimativas e podem precisar ser ajustada dependendo ligeiras variações em tamanhos de geometria e da gota do dispositivo. Os usuários com acesso a câmeras com capacidade de gravação de alta velocidade (> 10.000 quadros/s) deve verificar se a fusão de gotículas correto no início e ao longo da operação microfluidic. Os usuários sem acesso a uma câmera de alta velocidade podem coletar um pequeno volume de saída mesclada e medir manualmente os tamanhos de gotículas sob um microscópio. O tamanho da gota deve ser uniforme: um excesso de códigos de barras não mesclados ou gotas microgel indica que as taxas de reinjeção devem ser reduzidas em conformidade.
Diversos gerais devem ser tomadas precauções ao manusear microgels e microdroplets para preservar a sua integridade. Microgels, embora mecanicamente robusto, deve ser suficientemente arrefecido antes da quebra e etapas para garantir a completa gelificação de lavagem. Microgels não-esférica são uma indicação de que a agarose não foi dado tempo suficiente para solidificar. Quando microgels de lavagem, gire as suspensões para baixo na velocidade necessária para evitar uma perda de produto. Agarose hidrogel tem um índice de refração estreita correspondência da água e pode ser difíceis de ver em um tubo22, então os usuários cuidadosamente devem identificar o limite de gel-líquido antes da aspiração. Gotículas de água em óleo são suscetíveis a coalescência pela acumulação de forças estáticas23 luvas de laboratório e tubulação. Por este motivo, recomendamos carregar as seringas de reinjeção de gotículas com as próprias mãos e tratar todas as linhas de reinjeção com uma arma antiestática antes da escorva da bomba. As gotas se consolidou grandes podem ser removidas girando lentamente as emulsões em uma seringa e aspirar manualmente as gotas maiores, que se acumulam perto do topo, devido à sua maior força de empuxo.
SiC-seq é a primeira tecnologia para demonstrar o sequenciamento do genoma de célula única de > 50.000 células bacterianas. Esta plataforma oferece vantagens significativas na taxa de transferência sobre as abordagens existentes e permite uma amostragem mais profunda das comunidades microbianas heterogêneas. Até à data, microfluidic tecnologias para o sequenciamento do genoma de célula única têm empregado microchambers9 e micropoços24 para isolamento de célula e amplificação, mas com a quantidade no intervalo de apenas dezenas a centenas de células. O fluxo de classificação de células únicas em wellplates5,6 não requer nenhuma instrumentação especializada microfluidic mas possui uma taxa de transferência baixa da mesma forma. Dado que as amostras de solo e água do ambiente comumente tem diversidades alfa de > 1.000 da espécie nível25,26, SiC-seq é altamente vantajosa em virtude de sua capacidade de experimentar um número muito maior de organismos. O fluxo de trabalho de SiC-seq é adaptável às entradas de célula de laboratório de cultura, o ambiente natural ou um hospedeiro vivo. Uma amostra de células só precisa estar em uma suspensão aquosa e livre de partículas grandes (> 10 µm) para ser apropriado para microfluidic encapsulamento. Por exemplo, o método tem sido anteriormente aplicado a uma amostra de água do mar usando uma série de lavagem e filtragem de etapas para pré-processar as células antes de encapsulamento17.
O protocolo de SiC-seq gera uma quantidade relativamente escassa de dados de sequenciamento de cada célula única e pode não ser adequado para todas as aplicações. Alguns algoritmos de Bioinformática como montagem do genoma de novo ou variante de nucleotídeo único (SNV) chamando exigem maiores profundidades de cobertura para trabalhar eficazmente. Em vez disso, grupos de código de barras podem ser agrupado em silico por taxonômica binning métodos27 para que os algoritmos podem ser aplicados em conjuntos maiores de leituras. A relativamente baixa eficiência global de código de barras do fluxo de trabalho SiC-seq também pode apresentar desafios em casos onde a disponibilidade do exemplo de entrada é baixa. SiC-seq baseia-se em uma etapa de encapsulamento de Poisson-distribuído o código de barras, pois cerca de 10% das células receber um código de barras molecular e são amplificados durante a etapa de preparação final da biblioteca. Enquanto isto é comparável a outros regimes de barcoding baseada em microdroplet10, os usuários que trabalham com amostras de células preciosas podem ter dificuldade em alcançar o rendimento adequado biblioteca de sequenciamento e talvez seja necessário aumentar o número de ciclos PCR na final etapa de amplificação. Outra solução potencial para usuários com conhecimentos microfluídicos é classificar gotículas de código de barras positivas após a etapa PCR digital, trazendo assim a eficiência global do código de barras para > 85%28.
Uma direção futura potencial para SiC-seq tecnologia é adaptar o fluxo de trabalho para uso com células de mamíferos, pavimentando o caminho para novos estudos clínicos de célula única. Como exemplo, uma análise da variação número cópia entre único câncer células maio ainda mais nossa compreensão do papel da heterogeneidade em câncer patologia2. Alternativamente, integrar SiC-seq com os métodos existentes para sondar e enriquecer as sequências de DNA de interesse29 permitiria o sequenciamento de célula única orientado de subpopulações ou raras linhagens de células. Com amostras ambientais, genes de dentro de uma via metabólica conhecida poderiam ser alvejados e analisados contextualmente, ao lado de vizinhos genes para identificar novas ilhas genômicas. De dentro de um ambiente do hospedeiro humano, amostras de bactérias patogênicas de baixo-título podem ser isoladas e sequenciado no nível de célula única para examinar mais de perto suas origens genotípicas de virulência.
Divulgações
Patentes relativas a este fluxo de trabalho podem ser licenciado para Bio da missão, da qual Adam R. Abate é uma acionista.
Agradecimentos
Este trabalho foi financiado pela National Science Foundation através de um prémio de carreira (número de concessão DBI-1253293); o National Institutes of Health (NIH) (concessão números HG007233-01, 01-R01-EB019453, 1R21HG007233, DP2-AR068129-01, R01-HG008978); e defesa avançada projetos Agência vivendo fundições programa de pesquisa (contrato número HR0011-12-C-0065, N66001-12-C-4211, HR0011-12-C-0066).
Materiais
| Name | Company | Catalog Number | Comments |
| 3" silicon wafers, P type, virgin test grade | University Wafers | 447 | |
| SU-8 3025 photoresist | Microchem | 17030192 | |
| Spin coater | Specialty Coating Systems | G3P-8 | |
| Photomasks | CadArt Servcies | (custom) | See Supplemental Files for mask designs |
| PGMEA developer | Sigma-Aldrich | 484431 | |
| Isopropanol | Sigma-Aldrich | 109827 | |
| Sylgard 184 silicone elastomer kit | Krayden | 4019862 | |
| Degassing chamber | Bel-Art | 42025 | |
| 0.75 mm biopsy punch | World Precision Instruments | 504529 | |
| Glass microscope slides (75 mm x 50 mm) | Corning | 294775X50 | |
| Aquapel (hydrophobic glass treatment) | Pittsburgh Glass Works | 47100 | |
| PE-2 polyethylene tubing | Scientific Commodities | B31695-PE/2 | |
| 1 mL syringes | BD | 309628 | |
| 27 gauge needles | BD | 305109 | |
| Syringe pump | New Era Pump Systems | NE-501 | |
| Novec HFE-7500 fluorinated oil (HFE) | 3M | 98-0212-2928-5 | |
| FC-40 fluorinated oil | Sigma-Aldrich | F9755 | |
| PEG-PFPE surfactant | Ran Biotechnologies | 008-FluoroSurfactant | |
| Space heater | Lasko | CD09250 | |
| Agarose, low gelling temperature | Sigma-Aldrich | a9414 | |
| TE (10X) | Rockland | mb-007 | |
| PBS 1X, pH 7.4 | E&K Scientific Products | EK-65083 | |
| OptiPrep (density gradient medium) | Sigma-Aldrich | d1556 | |
| 1H,1H,2H-Perfluoro-1-Octanol (PFO) | Sigma-Aldrich | 370533 | |
| Span 80 (sorbitane monooleate) | Sigma-Aldrich | s6760 | |
| Hexane | Sigma-Aldrich | 139386 | |
| Tween 20 (polysorbate 20) | Sigma-Aldrich | p2287 | |
| Lysozyme Type IV | MP Biomedicals | 195303 | |
| Mutanolysin | Sigma-Aldrich | M9901 | |
| Zymolyase (yeast lytic enzyme) | Zymo Research | e1004 | |
| Lysostaphin | Sigma-Aldrich | L7386 | |
| Sodium chloride | Sigma-Aldrich | S9888 | |
| EDTA | Sigma-Aldrich | E6758 | |
| Tris-HCl, pH 7.5, 1M | Invitrogen | 15567-027 | |
| Dithiothreitol (DTT) | Teknova | d9750 | |
| Lithium dodecyl sulfate | Sigma-Aldrich | L9781 | |
| Proteinase K | New England Biosciences | P8107S | |
| Ethanol, 200 Proof (100%) | Koptec | V1001 | |
| SYBR Green I (nucleic acid stain) | Invitrogen | S7563 | |
| PEG 6k | Sigma-Aldrich | 81260 | |
| Triton X-100 (octylphenol ethoxylate) | Sigma-Aldrich | t8787 | |
| Nextera DNA Library Prep Kit | Illumina | FC-121-1030 | |
| Phusion Hot Start Flex Master Mix (High-Fidelity Hot Start Master Mix) | New England Biosciences | m05365 | |
| Platinum Multiplex PCR Master Mix (Taq Master Mix) | Applied Biosystems | 4464263 | |
| Warmstart 2.0 Bst Polymerase (isothermal polymerase) | New England Biosciences | m0538m | |
| NT buffer from Nextera XT kit (neutralization buffer) | Illumina | FC-131-1024 | |
| Cold cathode fluorescent inverter | (custom) | (custom) | |
| DC power supply | Mastech | HY1503D | |
| Zerostat 3 anti-static gun | Milty | 5036694022153 | |
| 3D-printed centrifuge syringe holder | (custom) | (custom) | See Supplemental Files for 3D print file |
| Zymo DNA Clean & Concentrator-5 | Zymo Research | D4003 |
Referências
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Ni, X., et al. Reproducible copy number variation patterns among single circulating tumor cells of lung cancer patients. Proceedings of the National Academy of Sciences of the United States of America. 110 (52), 21083-21088 (2013).
- Schmidt, H., Hensel, M. Pathogenicity islands in bacterial pathogenesis. Clinical Microbiology Reviews. 17, 14-56 (2004).
- Martínez, J. L., Baquero, F. Interactions among strategies associated with bacterial infection: pathogenicity epidemicity, and antibiotic resistance. Clinical Microbiology Reviews. 15 (4), 647-679 (2002).
- Rinke, C., et al. Obtaining genomes from uncultivated environmental microorganisms using FACS-based single-cell genomics. Nature Protocols. 9 (5), 1038-1048 (2014).
- Rinke, C., et al. Insights into the phylogeny and coding potential of microbial dark matter. Nature. 499 (7459), 431-437 (2013).
- Zhang, H., Liu, K. K. Optical tweezers for single cells. Journal of the Royal Society Interface. 5 (24), 671-690 (2008).
- Xu, L., Brito, I. L., Alm, E. J., Blainey, P. C. Virtual microfluidics for digital quantification and single-cell sequencing. Nature Methods. 13 (9), 759-762 (2016).
- Gawad, C., Koh, W., Quake, S. R. Dissecting the clonal origins of childhood acute lymphoblastic leukemia by single-cell genomics. Proceedings of the National Academy of Sciences of the United States of America. 111 (50), 17947-17952 (2014).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161 (5), 1202-1214 (2015).
- Klein, A. M., et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell. 161 (5), 1187-1201 (2015).
- Rotem, A., et al. High-throughput single-cell labeling (Hi-SCL) for RNA-Seq using drop-based microfluidics. PLoS One. 10 (5), 1-14 (2015).
- Amini, S., et al. Haplotype-resolved whole-genome sequencing by contiguity-preserving transposition and combinatorial indexing. Nature Reviews Genetics. 46 (12), 1343-1349 (2014).
- Zheng, G. X. Y., et al. Haplotyping germline and cancer genomes with high-throughput linked-read sequencing. Nature Biotechnology. 34 (3), 303-311 (2016).
- Lan, F., Haliburton, J. R., Yuan, A., Abate, A. R. Droplet barcoding for massively parallel single-molecule deep sequencing. Nature Communications. 7, 11784 (2016).
- Rotem, A., et al. Single-cell ChIP-seq reveals cell subpopulations defined by chromatin state. Nature Biotechnology. 33 (11), 1165-1172 (2015).
- Lan, F., Demaree, B., Ahmed, N., Abate, A. R. Single-cell genome sequencing at ultra-high-throughput with microfluidic droplet barcoding. Nature Biotechnology. 35 (7), 640-646 (2017).
- Novak, R., et al. Single-cell multiplex gene detection and sequencing with microfluidically generated agarose emulsions. Angewandte Chemie Internation Edition. 50 (2), 390-395 (2011).
- Gill, C., Van De Wijgert, J. H. H. M., Blow, F., Darby, A. C. Evaluation of lysis methods for the extraction of bacterial DNA for analysis of the vaginal microbiota. PLoS One. 11 (9), 1-16 (2016).
- Picelli, S., et al. Tn5 transposase and tagmentation procedures for massively scaled sequencing projects. Genome Research. 24 (12), 2033-2040 (2014).
- Abate, A. R., Chen, C. H., Agresti, J. J., Weitz, D. A. Beating Poisson encapsulation statistics using close-packed ordering. Lab on a Chip. 9 (18), 2628 (2009).
- Jain, A., Yang, A. H. J., Erickson, D. Gel-based optical waveguides with live cell encapsulation and integrated microfluidics. Optic Letters. 37 (9), 1472 (2012).
- Karbaschi, M., Shahi, P., Abate, A. R. Rapid chemical-free breaking of microfluidic emulsions with a hand-held antistatic gun. Biomicrofluidics. 11 (4), 1-6 (2017).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nature Biotechnology. 31 (12), 1126-1132 (2013).
- Chao, Y., et al. Metagenomic analysis reveals significant changes of microbial compositions and protective functions during drinking water treatment. Scientific Reports. 3 (1), 3550 (2013).
- Fierer, N., et al. Cross-biome metagenomic analyses of soil microbial communities and their functional attributes. Proceedings of the National Academy of Sciences of the United States of America. 109 (52), 21390-21395 (2012).
- Mande, S. S., Mohammed, M. H., Ghosh, T. S. Classification of metagenomic sequences: methods and challenges. Briefings in Bioinformatics. 13 (6), 669-681 (2012).
- Eastburn, D. J., et al. Microfluidic droplet enrichment for targeted sequencing. Nucleic Acids Research. 43 (13), e86 (2015).
- Clark, I. C., Abate, A. R. Finding a helix in a haystack: nucleic acid cytometry with droplet microfluidics. Lab on a Chip. 17 (12), 2032-2045 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados