É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Selecionando vários subconjuntos de biomarcador com Performances de classificação binária eficaz da mesma forma
Neste Artigo
Resumo
Algoritmos existentes geram uma solução para um dataset de deteção de biomarcador. Este protocolo demonstra a existência de múltiplas soluções similarmente eficazes e apresenta um software de fácil utilização para ajudar os investigadores biomédicos investigar seus conjuntos de dados para o desafio proposto. Cientistas de computador também podem fornecer esta funcionalidade em seu biomarcador algoritmos de detecção.
Resumo
Detecção de biomarcador é uma das mais importantes perguntas biomédicas para pesquisadores do elevado-throughput 'omics', e quase todos os algoritmos de detecção da biomarcador existentes geram um subconjunto de biomarcador com a medição de desempenho otimizado para um determinado conjunto de dados . No entanto, um estudo recente demonstrou a existência de vários subconjuntos de biomarcador com performances de classificação da mesma forma eficaz ou mesmo idênticas. Este protocolo apresenta uma metodologia simples e direta para a detecção de subconjuntos de biomarcador com performances de classificação binária, melhores do que um limite definido pelo usuário. O protocolo consiste de preparação de dados e carregamento, sumarização de informações de base, ajuste de parâmetro, triagem de biomarcador, resultado visualização e interpretação, anotações de gene biomarcador e exportação de resultado e visualização por qualidade de publicação. O biomarcador proposta estratégia de rastreio é intuitiva e demonstra uma regra geral para o desenvolvimento de algoritmos de detecção de biomarcador. Uma interface gráfica de fácil utilização (GUI) foi desenvolvida utilizando a linguagem de programação Python, permitindo que os pesquisadores biomédicos ter acesso direto aos seus resultados. O código-fonte e manual de kSolutionVis podem ser baixados de http://www.healthinformaticslab.org/supp/resources.php.
Introdução
Classificação binária, um dos mais comumente investigados e desafiadoras dados mineração problemas na área biomédica, é usado para construir um modelo de classificação treinado em dois grupos de amostras com a mais exata discriminação potência1, 2 , 3 , 4 , 5 , 6 , 7. no entanto, o grande volume de dados gerado no campo biomédico tem o inerente "grande p pequeno n" paradigma, com o número de características geralmente muito maiores do que o número de amostras6,8,9. Portanto, os pesquisadores biomédicos tem que reduzir a dimensão do recurso antes de utilizar os algoritmos de classificação para evitar o overfitting problema8,9. Biomarcadores de diagnóstico são definidos como um subconjunto das características detectadas, separando os pacientes de uma determinada doença de controle saudável amostras10,11. Os pacientes geralmente são definidos como as amostras positivas, e os controles saudáveis são definidos como as amostras negativas12.
Estudos recentes têm sugerido que existe mais de uma solução com performances de classificação idêntica ou similarmente eficaz para um conjunto de dados biomédicos5. Quase todos os algoritmos de seleção de recurso são algoritmos determinísticos, produzindo apenas uma solução para o mesmo conjunto de dados. Algoritmos genéticos simultaneamente podem gerar múltiplas soluções com desempenhos semelhantes, mas eles ainda tentam selecionar uma solução com a melhor função de aptidão como a saída para um determinado conjunto de dados13,14.
Algoritmos de seleção recurso aproximadamente podem ser agrupados como filtros ou invólucros12. Um algoritmo de filtragem escolhe as características dek top - ranking por sua associação significativa individual com os rótulos de binary classe com base na suposição de que dispõe são independentes do outro15,16,17 . Embora esta hipótese não prende verdadeira para quase todos os datasets de mundo real, a regra de filtragem heurística executa bem em muitos casos, por exemplo, o algoritmo mRMR (redundância de mínimo e máximo de relevância), o Wilcoxon teste baseado recurso de filtragem (WRank) algoritmo e o enredo ROC (característica de funcionamento do receptor), com base em algoritmo de filtragem (ROCRank). Flavia, é um algoritmo de filtragem eficiente porque calcula o problema combinatório de estimativa com uma série de problemas muito menores, comparando com o algoritmo de seleção de dependência máxima característica, cada uma das quais envolve apenas duas variáveis, e Portanto usa emparelhadas probabilidades de articulação, que são mais robustos18,19. No entanto, mRMR pode subestimar a utilidade de algumas características, como ele não mede as interações entre características que podem aumentar a relevância e assim perde algumas combinações de recurso que são individualmente inúteis, mas são úteis apenas quando combinado. O algoritmo de WRank calcula uma pontuação não-paramétricos de discriminativa como um recurso é entre duas classes de amostras e é conhecido por sua robustez para outliers20,21. Além disso, o algoritmo de ROCRank avalia como significativo a área sob o ROC curva (AUC) de um determinado recurso é para o desempenho de classificação binária investigadas22,23.
Por outro lado, um wrapper avalia desempenho do classificador pré-definidos de um subconjunto de determinado recurso, iterativamente, gerado por uma regra heurística e cria o subconjunto de recurso com o melhor desempenho de medição24. Um wrapper geralmente supera um filtro no desempenho classificação mas corre mais lento25. Por exemplo, o algoritmo de27 26,floresta aleatório regularizada (RRF) usa uma regra gananciosa, avaliando as características em um subconjunto de dados de treinamento em cada nó da floresta aleatório, pontuações de importância cujos recursos são avaliadas pelo índice de Gini . A escolha de um novo recurso será penalizada se o seu ganho de informações não melhorar isso das características escolhidas. Além disso, a análise de previsão de Microarrays (PAM)28,algoritmo de29 , também é um algoritmo de invólucro, calcula um centroide para cada um dos rótulos de classe e então seleciona recursos para encolher os centroides de gene em direção geral centroide da classe. PAM é robusta para características periféricas.
Várias soluções com o desempenho superior de classificação podem ser necessárias para qualquer determinado conjunto de dados. Em primeiro lugar, o objetivo da otimização de um algoritmo determinístico é definido por uma fórmula matemática, por exemplo, taxa de erro mínimo30, que não é necessariamente ideal para amostras biológicas. Em segundo lugar, um conjunto de dados pode ter soluções significativamente diferentes, de múltiplo, com performances similares de eficazes ou mesmo idênticas. Quase todos os algoritmos de seleção de recurso existente selecionará uma dessas soluções como a saída de31.
Este estudo irá introduzir um protocolo analítico de informática para gerar múltiplas soluções de seleção recurso com performances semelhantes para qualquer conjunto de dados de determinada classificação binária. Considerando que os pesquisadores mais biomédicos não estão familiarizados com técnicas de informáticos ou computador codificação, uma interface gráfica de fácil utilização (GUI) foi desenvolvida para facilitar a análise rápida de conjuntos de dados biomédicos classificação binária. O protocolo analítico consiste de carregamento de dados e resumindo, ajustar o parâmetro, execução de pipeline e interpretações do resultado. Com um simples clique, o pesquisador é capaz de gerar o biomarcador subconjuntos e parcelas de visualização de qualidade de publicação. O protocolo foi testado usando o transcriptomes de dois datasets de classificação binária de leucemia linfoblástica aguda (ALL), ou seja, ALL1 e ALL212. Os conjuntos de dados de ALL1 e ALL2 foram baixados do Broad Institute genoma análise centro de dados, disponível em http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi. ALL1 contém 128 amostras com 12.625 características. Estas amostras, 95 são células B todos e 33 são células T todos. ALL2 inclui 100 amostras com 12.625 características também. Estas amostras, há 65 pacientes que sofreram recaídas e 35 pacientes que não tinham. ALL1 foi um conjunto de dados de fácil classificação binária, com uma precisão mínima de quatro filtros e quatro invólucros sendo 96,7% e 6 dos algoritmos de seleção de 8 recurso atingir 100%12. Enquanto ALL2 foi um conjunto de dados mais difícil, com os algoritmos de seleção acima 8 recurso atingir não é melhor que a precisão de 83,7%12. Esta maior precisão foi alcançado com 56 características detectadas pelo algoritmo do invólucro, seleção de recurso baseado em correlação (CFS).
Protocolo
Nota: O protocolo seguinte descreve os detalhes do procedimento analítico informática e pseudo códigos dos módulos principais. O sistema de análise automática foi desenvolvido usando Python versão 3.6.0 e os pandas de módulos Python, abc, numpy, scipy, sklearn, sys, PyQt5, sys, mRMR, matemática e matplotlib. Os materiais utilizados neste estudo são listados na Tabela de materiais.
1. preparar a matriz de dados e rótulos de classe
- Prepare o arquivo da matriz de dados como um arquivo delimitado por tabulação ou vírgula da matriz, conforme ilustrado na figura 1A.
Nota: Cada linha tem todos os valores de um recurso, e o primeiro item é o nome do recurso. Uma característica é uma ID de probeset para o conjunto de dados de microarray-baseado transcriptoma ou pode ser outro valor ID como um resíduo de cisteína com seu valor de metilação em um dataset methylomic. Cada coluna dá os valores de recurso de uma determinada amostra, com o primeiro item, sendo o nome de amostra. Uma linha é separada em colunas por uma guia (figura 1B) ou uma vírgula (Figura 1). Um arquivo delimitado por tabulação matriz é reconhecido pelo TSV de extensão de arquivo e um arquivo delimitado por vírgula matriz tem a extensão. csv. Este arquivo pode ser gerado por salvar uma matriz como também o formato TSV ou CSV de software como o Microsoft Excel. A matriz de dados também pode ser gerada por computador codificação. - Prepare o arquivo de rótulo de classe como um delimitado por tabulação ou vírgula arquivo matriz (Figura 1), semelhante ao arquivo da matriz de dados.
Nota: A primeira coluna dá os nomes de amostra, e o rótulo de classe de cada amostra é dada na coluna intitulada classe. Compatibilidade máxima é considerada no processo de codificação, para que as colunas adicionais podem ser adicionadas. O arquivo de rótulo de classe pode ser formatado como um arquivo TSV ou CSV. Os nomes na coluna de classe podem ser quaisquer termos, e pode haver mais de duas classes de amostras. O usuário pode escolher quaisquer duas das classes para a análise a seguir.
2. carregar a matriz de dados e rótulos de classe
- Carrega os rótulos de matriz e classe de dados para o software. Clique no botão de matriz de dados de carga para escolher o arquivo de matriz de dados especificado pelo usuário. Clique no botão etiquetas de classe de carga para escolher o arquivo de rótulo de classe correspondente.
Nota: Depois de ambos os arquivos são carregados, kSolutionVis irá realizar uma rotina tela da compatibilidade entre os dois arquivos. - Resuma as características e as amostras do arquivo de dados de matriz. Estime o tamanho do arquivo de dados de matriz.
- Resuma as amostras e as classes do arquivo de rótulo de classe. Estime o tamanho do arquivo de rótulo de classe.
- Teste se cada amostra da matriz de dados tem um rótulo de classe. Resuma os números das amostras com os rótulos de classe.
3. resuma e exibir as estatísticas de base do conjunto de dados
- Clique no botão resumir, sem qualquer palavra-chave especificada de entrada, e o software irá exibir 20 recursos indexados e os nomes de recursos correspondente.
Nota: Os usuários precisam especificar o nome do recurso que desejam encontrar para ver suas estatísticas de base e a distribuição do valor correspondente entre todas as amostras de entrada. - Fornece uma palavra-chave, por exemplo, "1000_at", na caixa de texto recurso localizar um recurso específico para ser resumido. Clique no botão resumir para obter as estatísticas de base para esse determinado recurso.
Nota: A palavra-chave pode aparecer em qualquer lugar nos nomes de recurso de destino, facilitando o processo de pesquisa para os usuários. - Clique no botão resumir para encontrar mais de um recurso com a determinada palavra-chave e em seguida, especificar o ID de recurso exclusivo para prosseguir com o passo acima de resumir uma característica particular.
4. determine as etiquetas de classe e o número de características de ranking
- Escolha os nomes das classes de negativo ("N (95)") e positivos ("P (33)") na caixa dropdown Classe positivos e Negativos de classe, conforme mostrado na Figura 2 (médio).
Nota: É aconselhável para escolher que um dataset de classificação binária balanceada, ou seja, a diferença entre o número de amostras positivas e negativas é mínimo. O número de amostras é também dada entre parênteses após o nome de cada rótulo de classe nas duas caixas dropdown. - Escolha 10 como o número de características de ranking (parâmetro pTopX) na caixa de lista suspensa Top_X (?) para uma tela completa do recurso-subconjunto.
Nota: O software automaticamente classifica todos os recursos pelo P-valor calculado por um t-teste de cada recurso, comparando as classes positivas e negativas. Um recurso com uma menor P-valor tem um poder melhor discriminar entre as duas classes de amostras. O módulo de rastreio abrangente é computacionalmente intensivo. O parâmetro pTopX é 10 por padrão. Os usuários podem alterar este parâmetro na faixa de 10 a 50, até encontrarem satisfazendo apresentam subconjuntos com performances de boa classificação.
5. ajustar parâmetros do sistema para diferentes desempenhos
- Escolha a medição de desempenho (pMeasurement), precisão (Acc) na caixa de lista suspensa Acc/bAcc (?) para o classificador selecionado extrema aprendizagem máquina (ELM). Outra opção deste parâmetro é a medição precisão equilibrada (bAcc).
Nota: Deixe o TP, FN, TN, e FP ser os números positivos verdadeiros, falsos negativos, verdadeiros negativos e falsos positivos, respectivamente. A medição Acc é definida como (TP+TN)/(TP+FN+TN+FP), que funciona melhor em um conjunto equilibrado de dados6. Mas um classificador otimizado para Acc tende a atribuir todas as amostras à classe negativo se o número de amostras negativas é muito maior do que os positivos. A bAcc é definido como (Sn + Sp) / 2, onde Sn = TP/(TP+FN) e Sp = TN/(TN+FP) são as taxas corretamente previstas para o positivo e negativo amostras, respectivamente. Portanto, bAcc normaliza as performances de previsão sobre as duas classes e pode levar a um desempenho equilibrado previsão sobre duas classes desbalanceadas. ACC é a escolha de padrão de pMeasurement. O software usa o classificador ELM por padrão para calcular as performances de classificação. O usuário também pode escolher um classificador de SVM (Support Vector Machine), KNN (k vizinho mais próximo), árvore de decisão ou Naïve Bayes. - Escolha o valor de corte 0.70 (parâmetro pCutoff) para a medição de desempenho especificado na caixa de entrada pCutoff:.
Nota: Tanto Acc e bAcc variam entre 0 e 1, e o usuário pode especificar um valor pCutoff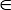 [0, 1] como o corte para exibir as soluções correspondentes. O software realiza um recurso abrangente-subconjunto de rastreio, e uma escolha apropriada de pCutoff fará a visualização em 3D mais intuitiva e explícita. O valor padrão para pCutoff é 0,70.
[0, 1] como o corte para exibir as soluções correspondentes. O software realiza um recurso abrangente-subconjunto de rastreio, e uma escolha apropriada de pCutoff fará a visualização em 3D mais intuitiva e explícita. O valor padrão para pCutoff é 0,70.
6. Execute o Pipeline e produzir os resultados visualizados interativos
- Clique no botão Analyze para executar o pipeline e gerar as parcelas de visualização, como mostrado na Figura 2 (abaixo).
Nota: A tabela à esquerda dá todos os subconjuntos de recurso e seus pMeasurement calculado pela estratégia de validação cruzada 10 vezes do classificador ELM, conforme descrito previamente5. Dois gráficos de dispersão 3D e parcelas de duas linhas são geradas para o procedimento de rastreio de recurso-subconjunto com as atuais configurações de parâmetro. - Escolha 0.70 como o valor padrão de pMeasurement corte (parâmetro piCutoff, caixa de entrada valor) e 10 como o padrão do número de subconjuntos de recurso melhores (parâmetro piFSNum).
Nota: O gasoduto é executado usando os parâmetros pTopX, pMeasurement e pCutoff. O recurso detectado subconjuntos podem ser ainda mais selecionados usando o corte piCutoff, porém piCutoff não pode ser menor do que pCutoff. Portanto, piCutoff é inicializado como pCutoff e apenas os subconjuntos de recurso com a medição de desempenho ≥ piCutoff vão ser visualizados. O valor padrão de piCutoff é pCutoff. Às vezes o kSolutionVis detecta muitas soluções e apenas os melhores piFSNum (padrão: 10) subconjuntos de recurso vão ser visualizados. Se o número de subconjuntos de característica detectado pelo software for menor que piFSNum, todos os subconjuntos de recurso vão ser visualizados. - Coletar e interpretar as características detectadas pelo software, conforme mostrado na Figura 3.
Nota: A tabela na caixa da esquerda mostra os subconjuntos de recurso detectados e suas medições de desempenho. Os nomes das três primeiras colunas são "F1", "F2" e "F3". As três características em cada subconjunto de recurso são dadas em sua ordem de classificação em uma linha (F1 < F2 < F3). A última coluna dá a medição de desempenho (Acc ou bAcc) de cada subconjunto de recurso, e seu nome de coluna (Acc ou bAcc) é o valor do pMeasurement.
7. interpretar o 3D Scatter Plots-Visualizar e interpretar os subconjuntos de recurso com Performances de classificação binária similarmente eficaz usando gráficos de dispersão 3D
- Clique no botão Analyze para gerar o 3D de dispersão dos subconjuntos de 10 recurso top com os melhores desempenhos de classificação (Acc ou bAcc) detectado pelo software, conforme mostrado na Figura 3 (caixa central). Classificar os três recursos em um subconjunto de recurso em ordem crescente de suas fileiras e use as fileiras das três características como os eixos de F1/F2/F3, ou seja, F1 < F2 < F3.
Nota: A cor de um ponto representa o desempenho de classificação binária do subconjunto correspondente recurso. Um dataset pode ter vários subconjuntos de recurso com medições de desempenho eficaz da mesma forma. Portanto, um interativo e simplificado de dispersão é necessário. - Altere o valor de 0,70 na caixa de entrada pCutoff: e clique no botão Analyze para gerar o gráfico de dispersão 3D dos subconjuntos de recurso com o desempenho medição ≥ piCutoff, como pode ser visto na Figura 3 (caixa direita). Clique no botão de ajuste de 3D para abrir uma nova janela para ajustar manualmente os ângulos de visão de 3D o gráfico de dispersão.
Nota: Cada subconjunto de recurso é representado por um ponto da mesma forma como acima. O gráfico de dispersão 3D foi gerado no ângulo padrão. Para facilitar a visualização em 3D e tuning, outra janela será aberta clicando no botão de ajuste de 3D. - Clique no botão reduzir para reduzir a redundância de subconjuntos a característica detectado.
Nota: Se os usuários desejam mais selecionar os trigêmeos de recurso e minimizar a redundância de subconjuntos a característica, o software também oferece essa função usando o algoritmo de seleção de recurso mRMR. Após clicar no botão reduzir , kSolutionVis irá remover esses recursos redundantes em trigêmeos o recurso e regenerar a tabela e os dois dispersam parcelas mencionadas acima. As características removidas de trigêmeos o recurso serão substituídas pela palavra chave na tabela. Os valores de None no eixo F1/F2/F3 irão ser denotados como o valor de piFSNum (o intervalo entre o valor normal de F1/F2/F3 é [1, top_x]). Portanto, os pontos que incluem um valor nenhum podem parecer ser parcelas de pontos de "outlier" em 3D. Os terrenos 3D manualmente ajustáveis podem ser encontrados em "Sintonia Manual dos terrenos 3D dot" no material complementar.
8. encontrar o Gene anotações e suas associações com doenças humanas
Nota: Os passos 8 a 10 irão ilustrar como anotar um gene do nível de sequência de DNA e proteína. Em primeiro lugar, o símbolo do gene de cada ID de biomarcador das etapas acima será recuperado do banco de dados de DAVID32, e depois de dois servidores web representante serão usados para analisar este símbolo de gene dos níveis de DNA e proteínas, respectivamente. O servidor GeneCard fornece uma abrangente anotação funcional de um símbolo de determinado gene e a herança mendeliana Online no banco de dados do homem (OMIM) fornece a curadoria mais abrangente das associações de doença genética. O servidor UniProtKB é um dos mais completo banco de dados da proteína, e o servidor sistema de predição baseada em grupo (GPS) prevê a sinalização fosforilação para obter uma lista muito grande de quinases.
- Copie e cole o link de web da base de dados de DAVID em um navegador da web e abra a página web deste banco de dados. Clique no link Gene ID conversão visto na Figura 4A e entrada o recurso IDs 38319_at/38147_at/33238_at do primeiro subconjunto de biomarcador de dataset ALL1 (Figura 4B). Clique no link Lista de Gene e clique em Enviar a lista , como mostrado na Figura 4B. Recuperar as anotações de interesse e clique em Mostrar lista de Gene (Figura 4). Obter a lista de símbolos de gene (Figura 4).
Nota: Os símbolos de gene obtidos aqui serão usados para ainda mais funcionais anotações nas próximas etapas. - Copie e cole o link de web da base de dados de cartões de Gene em um navegador da web e abra a página web deste banco de dados. Procurar o nome de um gene CD3D na caixa de entrada de consulta de banco de dados e encontrar as anotações deste gene de Gene cartões33,34, conforme mostrado na tabela 1 e Figura 5A.
Nota: Cartões de Gene é uma base de conhecimento abrangente de gene, fornecendo a nomenclatura, genômica, proteômica, Localização subcellular e vias envolvidas e outros módulos funcionais. Ele também fornece links externos para vários outros bancos de dados biomédicos como Apo/PDB_REDO35, Entrez Gene36, OMIM37e UniProtKB38. Se o nome de recurso não é um símbolo de gene padrão, use o banco de dados ENSEMBL para convertê-lo39. CD3D é o nome do gene do Receptor de células T T3 Delta cadeia. - Copie e cole o link de web de banco de dados OMIM em um navegador da web e abra a página web deste banco de dados. Nome de um gene CD3D de busca e encontrar as anotações deste gene do banco de dados OMIM37, conforme mostrado na tabela 1 e Figura 5B.
Nota: OMIM serve agora como uma das fontes mais abrangentes e autoritária de conexões de genes humanos com doenças hereditárias. OMIM foi iniciado pelo Dr. Victor A. McKusick para catalogar as mutações genéticas associadas a doença40. OMIM agora, abrange 15.000 genes humanos e mais de 8.500 fenótipos, a partir de dezembro de 1st 2017.
9. anotar as proteínas codificadas e as modificações borne-translational
- Copie e cole o link de web de banco de dados UniProtKB em um navegador da web e abra a página web deste banco de dados. Busca o nome de um gene CD3D na caixa de entrada de consulta de UniProtKB e encontrar as anotações deste gene do banco de dados38, conforme mostrado na tabela 1 e Figura 5.
Nota: UniProtKB recolhe uma fonte rica de anotações para proteínas, incluindo tanto informações funcionais e nomenclatura. Este banco de dados também fornece links externos para outros bancos de dados amplamente utilizados, incluindo PDB/PDB_REDO35, OMIM37e Pfam41. - Copie e cole o link da web do servidor de web do GPS em um navegador da web e abra a página web do servidor web. Recuperar a sequência da proteína codificada pelo gene biomarcador CD3D o banco de dados de UniProtKB38 e prever resíduos de modificação pós-traducional (PTM) da proteína usando a ferramenta on-line GPS, conforme mostrado na tabela 1 e Figura 5.
Nota: Um sistema biológico é dinâmico e complicado, e os bancos de dados existentes coletam apenas informações conhecidas. Portanto, ferramentas on-line de previsão biomédica, bem como programas off-line podem proporcionar evidência útil para complementar um mecanismo hipotético. GPS foi desenvolvido e melhorado por mais de 12 anos7,42 e pode ser usada para prever a resíduos de uma proteína PTM em uma sequência de determinado peptídeo43,44. Ferramentas também estão disponíveis para vários tópicos de pesquisa, incluindo a previsão de uma proteína Localização subcellular45 e transcrição fator vinculação motivos 46 entre outros.
10. anotar seus módulos funcionais enriquecidos e interações da proteína-proteína
- Copie e cole o link da web do servidor web String em um navegador da web e abra a página web do servidor web. Pesquisar a lista para os genes CD3D e P53 e encontrar suas propriedades orquestradas usando o banco de dados String47. O mesmo procedimento pode ser realizado usando outro servidor web, DAVID32.
Nota: Além das anotações acima mencionadas para genes individuais, existem muitas ferramentas de informática em grande escala disponíveis para investigar as propriedades de um grupo de genes. Um estudo recente demonstrou que os genes marcadores individualmente ruim podem constituir um conjunto muito melhor gene5. Portanto, vale o custo computacional para triagem de biomarcadores mais complicados. O banco de dados de sequência de caracteres pode visualizar as conexões de interação conhecidas ou previstas, e o servidor de David pode detectar os módulos funcionais com fenótipo-associações significativas no consultado genes47,32. Várias outras ferramentas de análise de informática em grande escala também estão disponíveis.
11. exportar os subconjuntos de biomarcador gerado e as parcelas de visualização
- Exporte os subconjuntos de biomarcador detectado como um arquivo de texto de TSV ou CSV para uma análise mais aprofundada. Clique no botão exportar a tabela sob a tabela de todos os subconjuntos de biomarcador detectado e escolher qual formato de texto para salvar como.
- Exporte as parcelas de visualização como um arquivo de imagem. Clique no botão salvar em cada parcela e escolher qual formato de imagem para salvar como.
Nota: O software suporta o formato de pixel. png e o vetor formato SVG. As imagens de pixel são bons para a exibição na tela do computador, enquanto as imagens vetoriais podem ser convertidas em qualquer resolução necessária para fins de publicação do jornal.
Resultados
O objetivo do fluxo de trabalho (Figura 6) é detectar vários subconjuntos de biomarcador com eficiência similar para um dataset de classificação binária. Todo o processo é ilustrado por dois conjuntos de dados exemplo ALL1 e ALL2, extraído de uma detecção de biomarcador recentemente publicado estudo12,,48. Um usuário pode instalar kSolutionVis, seguindo as instruções nos materiais supleme...
Discussão
Este estudo apresenta um protocolo de deteção e caracterização de biomarcador de solução multi fácil-à-siga para um dataset classificação binário especificado pelo usuário. O software coloca ênfase na facilidade de uso e interfaces flexíveis de importação/exportação para vários formatos de arquivo, permitindo que um pesquisador biomédico investigar seu dataset facilmente usando a GUI do software. Este estudo também destaca a necessidade de gerar mais de uma solução com performances de modelagem sim...
Divulgações
Temos sem conflitos de interesse relacionados a este relatório.
Agradecimentos
Este trabalho foi financiado pelo programa de investigação estratégica prioridade da Academia Chinesa de Ciências (XDB13040400) e a concessão de inicialização da Universidade de Jilin. Revisores anônimos e usuários testes biomédicos foram apreciados por seus comentários construtivos em melhorar a usabilidade e funcionalidade do kSolutionVis.
Materiais
| Name | Company | Catalog Number | Comments |
| Hardware | |||
| laptop | Lenovo | X1 carbon | Any computer works. Recommended minimum configuration: 1GB extra hard disk space, 1 GB memory, 2.0MHz CPU |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Python 3.0 | WingWare | Wing Personal | Any python programming and running environments support Python version 3.0 or above |
Referências
- Heckerman, D., et al. Genetic variants associated with physical performance and anthropometry in old age: a genome-wide association study in the ilSIRENTE cohort. Scientific Reports. 7, 15879 (2017).
- Li, Z., et al. Genome-wide association analysis identifies 30 new susceptibility loci for schizophrenia. Nature Genetics. 49, 1576-1583 (2017).
- Winkler, T. W., et al. Quality control and conduct of genome-wide association meta-analyses. Nature Protocols. 9, 1192-1212 (2014).
- Harrison, R. N. S., et al. Development of multivariable models to predict change in Body Mass Index within a clinical trial population of psychotic individuals. Scientific Reports. 7, 14738 (2017).
- Liu, J., et al. Multiple similarly-well solutions exist for biomedical feature selection and classification problems. Scientific Reports. 7, 12830 (2017).
- Ye, Y., Zhang, R., Zheng, W., Liu, S., Zhou, F. RIFS: a randomly restarted incremental feature selection algorithm. Scientific Reports. 7, 13013 (2017).
- Zhou, F. F., Xue, Y., Chen, G. L., Yao, X. GPS: a novel group-based phosphorylation predicting and scoring method. Biochemical and Biophysical Research Communications. 325, 1443-1448 (2004).
- Sanchez, B. N., Wu, M., Song, P. X., Wang, W. Study design in high-dimensional classification analysis. Biostatistics. 17, 722-736 (2016).
- Shujie, M. A., Carroll, R. J., Liang, H., Xu, S. Estimation and Inference in Generalized Additive Coefficient Models for Nonlinear Interactions with High-Dimensional Covariates. Annals of Statistics. 43, 2102-2131 (2015).
- Li, J. H., et al. MiR-205 as a promising biomarker in the diagnosis and prognosis of lung cancer. Oncotarget. 8, 91938-91949 (2017).
- Lyskjaer, I., Rasmussen, M. H., Andersen, C. L. Putting a brake on stress signaling: miR-625-3p as a biomarker for choice of therapy in colorectal cancer. Epigenomics. 8, 1449-1452 (2016).
- Ge, R., et al. McTwo: a two-step feature selection algorithm based on maximal information coefficient. BMC Bioinformatics. 17, 142 (2016).
- Tumuluru, J. S., McCulloch, R. Application of Hybrid Genetic Algorithm Routine in Optimizing Food and Bioengineering Processes. Foods. 5, (2016).
- Gen, M., Cheng, R., Lin, L. . Network models and optimization: Multiobjective genetic algorithm approach. , (2008).
- Radovic, M., Ghalwash, M., Filipovic, N., Obradovic, Z. Minimum redundancy maximum relevance feature selection approach for temporal gene expression data. BMC Bioinformatics. 18, 9 (2017).
- Ciuculete, D. M., et al. A methylome-wide mQTL analysis reveals associations of methylation sites with GAD1 and HDAC3 SNPs and a general psychiatric risk score. Translational Psychiatry. 7, e1002 (2017).
- Lin, H., et al. Methylome-wide Association Study of Atrial Fibrillation in Framingham Heart Study. Scientific Reports. 7, 40377 (2017).
- Wang, S., Li, J., Yuan, F., Huang, T., Cai, Y. D. Computational method for distinguishing lysine acetylation, sumoylation, and ubiquitination using the random forest algorithm with a feature selection procedure. combinatorial chemistry & high throughput screening. , (2017).
- Zhang, Q., et al. Predicting Citrullination Sites in Protein Sequences Using mRMR Method and Random Forest Algorithm. combinatorial chemistry & high throughput screening. 20, 164-173 (2017).
- Cuena-Lombrana, A., Fois, M., Fenu, G., Cogoni, D., Bacchetta, G. The impact of climatic variations on the reproductive success of Gentiana lutea L. in a Mediterranean mountain area. International journal of biometeorology. , (2018).
- Coghe, G., et al. Fatigue, as measured using the Modified Fatigue Impact Scale, is a predictor of processing speed improvement induced by exercise in patients with multiple sclerosis: data from a randomized controlled trial. Journal of Neurology. , (2018).
- Hong, H., et al. Applying genetic algorithms to set the optimal combination of forest fire related variables and model forest fire susceptibility based on data mining models. The case of Dayu County, China. Science of the Total Environment. 630, 1044-1056 (2018).
- Borges, D. L., et al. Photoanthropometric face iridial proportions for age estimation: An investigation using features selected via a joint mutual information criterion. Forensic Science International. 284, 9-14 (2018).
- Kohavi, R., John, G. H. Wrappers for feature subset selection. Artificial intelligence. 97, 273-324 (1997).
- Yu, L., Liu, H. Efficient feature selection via analysis of relevance and redundancy. Journal of machine learning research. 5, 1205-1224 (2004).
- Wexler, R. B., Martirez, J. M. P., Rappe, A. M. Chemical Pressure-Driven Enhancement of the Hydrogen Evolving Activity of Ni2P from Nonmetal Surface Doping Interpreted via Machine Learning. Journal of American Chemical Society. , (2018).
- Wijaya, S. H., Batubara, I., Nishioka, T., Altaf-Ul-Amin, M., Kanaya, S. Metabolomic Studies of Indonesian Jamu Medicines: Prediction of Jamu Efficacy and Identification of Important Metabolites. Molecular Informatics. 36, (2017).
- Shangkuan, W. C., et al. Risk analysis of colorectal cancer incidence by gene expression analysis. PeerJ. 5, e3003 (2017).
- Chu, C. M., et al. Gene expression profiling of colorectal tumors and normal mucosa by microarrays meta-analysis using prediction analysis of microarray, artificial neural network, classification, and regression trees. Disease Markers. , 634123 (2014).
- Fleuret, F. Fast binary feature selection with conditional mutual information. Journal of Machine Learning Research. 5, 1531-1555 (2004).
- Pacheco, J., Alfaro, E., Casado, S., Gámez, M., García, N. A GRASP method for building classification trees. Expert Systems with Applications. 39, 3241-3248 (2012).
- Jiao, X., et al. DAVID-WS: a stateful web service to facilitate gene/protein list analysis. Bioinformatics. 28, 1805-1806 (2012).
- Rappaport, N., et al. Rational confederation of genes and diseases: NGS interpretation via GeneCards, MalaCards and VarElect. Biomedical Engineering OnLine. 16, 72 (2017).
- Rebhan, M., Chalifa-Caspi, V., Prilusky, J., Lancet, D. GeneCards: integrating information about genes, proteins and diseases. Trends in Genet. 13, 163 (1997).
- Joosten, R. P., Long, F., Murshudov, G. N., Perrakis, A. The PDB_REDO server for macromolecular structure model optimization. IUCrJ. 1, 213-220 (2014).
- Maglott, D., Ostell, J., Pruitt, K. D., Tatusova, T. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Research. 39, D52-D57 (2011).
- Amberger, J. S., Bocchini, C. A., Schiettecatte, F., Scott, A. F., Hamosh, A. OMIM.org: Online Mendelian Inheritance in Man (OMIM(R)), an online catalog of human genes and genetic disorders. Nucleic Acids Research. 43, D789-D798 (2015).
- Boutet, E., et al. the Manually Annotated Section of the UniProt KnowledgeBase: How to Use the Entry View. Methods in Molecular Biology. 1374, 23-54 (2016).
- Zerbino, D. R., et al. Ensembl 2018. Nucleic Acids Res. , (2017).
- McKusick, V. A., Amberger, J. S. The morbid anatomy of the human genome: chromosomal location of mutations causing disease. Journal of Medical Genetics. 30, 1-26 (1993).
- Finn, R. D., et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Research. 44, D279-D285 (2016).
- Xue, Y., et al. GPS: a comprehensive www server for phosphorylation sites prediction. Nucleic Acids Research. 33, W184-W187 (2005).
- Deng, W., et al. GPS-PAIL: prediction of lysine acetyltransferase-specific modification sites from protein sequences. Scientific Reports. 6, 39787 (2016).
- Zhao, Q., et al. GPS-SUMO: a tool for the prediction of sumoylation sites and SUMO-interaction motifs. Nucleic Acids Research. 42, W325-W330 (2014).
- Wan, S., Duan, Y., Zou, Q. HPSLPred: An Ensemble Multi-Label Classifier for Human Protein Subcellular Location Prediction with Imbalanced Source. Proteomics. 17, (2017).
- Zhang, H., Zhu, L., Huang, D. S. WSMD: weakly-supervised motif discovery in transcription factor ChIP-seq data. Scientific Reports. 7, 3217 (2017).
- Szklarczyk, D., et al. STRING v10: protein-protein interaction networks, integrated over the tree of life. Nucleic Acids Research. 43, D447-D452 (2015).
- Chiaretti, S., et al. Gene expression profile of adult T-cell acute lymphocytic leukemia identifies distinct subsets of patients with different response to therapy and survival. Blood. 103, 2771-2778 (2004).
- Rowley, J. D., et al. Mapping chromosome band 11q23 in human acute leukemia with biotinylated probes: identification of 11q23 translocation breakpoints with a yeast artificial chromosome. Proceedings of the National Academy of Sciences of the United States of America. 87, 9358-9362 (1990).
- Rabbitts, T. H., et al. The chromosomal location of T-cell receptor genes and a T cell rearranging gene: possible correlation with specific translocations in human T cell leukaemia. Embo Journal. 4, 1461-1465 (1985).
- Yin, L., et al. SH2D1A mutation analysis for diagnosis of XLP in typical and atypical patients. Human Genetics. 105, 501-505 (1999).
- Brandau, O., et al. Epstein-Barr virus-negative boys with non-Hodgkin lymphoma are mutated in the SH2D1A gene, as are patients with X-linked lymphoproliferative disease (XLP). Human Molecular Genetics. 8, 2407-2413 (1999).
- Burnett, R. C., Thirman, M. J., Rowley, J. D., Diaz, M. O. Molecular analysis of the T-cell acute lymphoblastic leukemia-associated t(1;7)(p34;q34) that fuses LCK and TCRB. Blood. 84, 1232-1236 (1994).
- Taylor, G. M., et al. Genetic susceptibility to childhood common acute lymphoblastic leukaemia is associated with polymorphic peptide-binding pocket profiles in HLA-DPB1*0201. Human Molecular Genetics. 11, 1585-1597 (2002).
- Wadia, P. P., et al. Antibodies specifically target AML antigen NuSAP1 after allogeneic bone marrow transplantation. Blood. 115, 2077-2087 (2010).
- Wilson, D. M., et al. 3rd et al. Hex1: a new human Rad2 nuclease family member with homology to yeast exonuclease 1. Nucleic Acids Research. 26, 3762-3768 (1998).
- O'Sullivan, R. J., et al. Rapid induction of alternative lengthening of telomeres by depletion of the histone chaperone ASF1. Nature Structural & Molecular Biology. 21, 167-174 (2014).
- Lee-Sherick, A. B., et al. Aberrant Mer receptor tyrosine kinase expression contributes to leukemogenesis in acute myeloid leukemia. Oncogene. 32, 5359-5368 (2013).
- Guyon, I., Elisseeff, A. An introduction to variable and feature selection. Journal of machine learning research. 3, 1157-1182 (2003).
- John, G. H., Kohavi, R., Pfleger, K. . Machine learning: proceedings of the eleventh international conference. , 121-129 (1994).
- Jain, A., Zongker, D. Feature selection: Evaluation, application, and small sample performance. IEEE transactions on pattern analysis and machine intelligence. 19, 153-158 (1997).
- Taylor, S. L., Kim, K. A jackknife and voting classifier approach to feature selection and classification. Cancer Informatics. 10, 133-147 (2011).
- Andresen, K., et al. Novel target genes and a valid biomarker panel identified for cholangiocarcinoma. Epigenetics. 7, 1249-1257 (2012).
- Guo, P., et al. Gene expression profile based classification models of psoriasis. Genomics. 103, 48-55 (2014).
- Xie, J., Wang, C. Using support vector machines with a novel hybrid feature selection method for diagnosis of erythemato-squamous diseases. Expert Systems with Applications. 38, 5809-5815 (2011).
- Zou, Q., Zeng, J., Cao, L., Ji, R. A novel features ranking metric with application to scalable visual and bioinformatics data classification. Neurocomputing. 173, 346-354 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados