Method Article
Dissecação do Enhancer função usando interferência Multiplex Enhancer CRISPR-baseado em linhas de células
Neste Artigo
Resumo
Este protocolo descreve as etapas necessárias para projetar e executar a segmentação multiplexada de realçadores com a proteína de fusão desativação CASCUDO-SID4X-dCas9, também conhecido como interferência de realçador (Enhancer-i). Este protocolo permite a identificação de potenciadores que regulam a expressão gênica e facilita a dissecação das relações entre potenciadores de regulação de um gene alvo comum.
Resumo
Potenciadores de múltiplas vezes regulam um determinado gene, no entanto, para a maioria dos genes, ainda não está claro quais realçadores são necessários para a expressão do gene, e como estes potenciadores combinam para produzir uma resposta transcricional. Como milhões de realçadores foram identificados, ferramentas de alta produtividade são necessários para determinar a função de potenciador na escala do genoma. Métodos atuais para estudar a função potenciador incluem fazer genéticas exclusões usando Cas9 nuclease-proficiente, mas é difícil de estudar os efeitos combinatórios de realçadores múltiplos usando esta técnica, como várias linhas sucessivas clonal de célula devem ser gerados. Aqui, apresentamos Enhancer-i, um método baseado em interferência CRISPR que permite o interrogatório funcional de potenciadores de múltiplos simultaneamente em seus loci endógena. Potenciador-i empreg dois domínios repressivos fundidos a nuclease deficiente Cas9, SID e CASCUDO, para alcançar a desativação do realçador através de deacetilação de histona em loci alvo. Este protocolo utiliza transfection transiente de guia RNAs para habilitar transitória inactivação das regiões alvo e é particularmente eficaz em bloquear inducible transcriptional respostas aos estímulos em configurações de cultura de tecidos. Potenciador-i é altamente específica, tanto em seu direcionamento genômica e seus efeitos na expressão gênica global. Resultados obtidos com este protocolo ajudam a compreender se um intensificador está a contribuir para a expressão do gene, a magnitude da contribuição e como a contribuição é afectada por outros potenciadores nas proximidades.
Introdução
Em grande escala sequenciamento projetos como ENCODE1, roteiro Epigenomics2e FANTOM3 identificaram milhões de realçadores putativos dentro do genoma humano em centenas de tipos de células. Estima-se que cada promotor associa-se com uma média de 4,9 potenciadores e cada potenciador de contatos uma média de 2,4 genes3, sugerindo que a expressão gênica é muitas vezes o resultado da integração de múltiplas interações reguladoras distribuídas. Um desafio significativo remanescente é definir não só individuais como potenciadores de contribuam para a expressão do gene, mas como eles se combinam para afetar a expressão. Abordagens genéticas são comumente usadas para identificar as relações entre potenciadores em organismos-modelo de drosófila4 para ratos5. No entanto, estas experiências são morosas e de baixo rendimento para o estudo de múltiplas potenciadores de múltiplos genes.
Uma abordagem para estudar a função potenciador em grande escala envolve ensaios repórter massivamente paralelo. Estes ensaios permitem a exibição simultânea de milhares de sequências de DNA por sua capacidade de conduzir a expressão de um gene de repórter6. Enquanto estes ensaios têm mostrado que a sequência de DNA sozinho pode ser suficiente para transmitir o gene Regulamento informações7, eles vêm com as advertências de sendo executada fora do contexto de cromatina nativa e com um promotor heterólogo. Além disso, o tamanho da sequência de DNA, sendo analisado em ensaios de repórter maciçamente paralelo é geralmente basepairs menos de 200, que podem excluir a sequência relevante circundante. Importante, como repórter ensaios apenas medem a atividade de uma sequência de cada vez, eles não levam em conta as relações complexas que podem existir entre potenciadores. Assim, enquanto repórter maciçamente paralelo ensaios podem ser informativos sobre a atividade intrínseca de uma sequência de DNA, eles não necessariamente nos informar sobre a função dessa sequência de DNA no contexto do genoma.
Recentemente desenvolvidos CRISPR/Cas9 ferramentas8 facilitaram o estudo da regulação genética, já que permitem a exclusão de realçadores no locus endógeno. No entanto, excluir potenciadores de múltiplos simultaneamente pode levar à instabilidade genômica e é demorado para gerar exclusões potenciador sucessivas em uma linha única célula. Além disso, nova sequência genômica é criada no site da exclusão após o reparo, e essa sequência pode ganhar função reguladora. Uma versão alternativa do Cas9 foi desenvolvida especificamente para modular a expressão gênica, contando com fusões de ativação9,10 ou reprimir11,12 domínios de forma deficiente nuclease das Cas9 (dCas9). Estas proteínas de fusão são ideais para estudar simultaneamente múltiplos loci, como eles não fisicamente alterar a sequência de DNA e em vez disso, modulam epigenética para interrogar uma região reguladora. A fusão repressiva mais amplamente utilizada é CASCUDO, que recruta o complexo de repressor co KAP1, promovendo a deposição da repressão-associado histona H3 lisina 9 trimethylation (H3K9me3)13. dCas9-CASCUDO, também conhecido como CRISPR interferência14, tem sido usada para alvo e tela potenciadores individuais por suas contribuições para o de15,de expressão do gene16; no entanto, isso não foi otimizado para segmentação de várias regiões simultaneamente. Uma versão do multiplex interferência CRISPR para realçadores, mosaico-seq17, usa única célula RNA-seq como uma leitura, mas esta tecnologia é caro e indicado apenas para o estudo de genes altamente expressos devido à baixa sensibilidade de uma única célula RNA-Seq
Temos procurado desenvolver um método baseado em interferência de CRISPR para dissecar função combinatória potenciador dentro do contexto de uma resposta transcricional de estrogênio. Cerca de metade dos genes hormona-responsivo contêm 2 ou mais potenciadores vinculados pela Alfa do receptor de estrógeno (ER) nas proximidades de18, sugerindo que potenciadores de múltiplas podem ser participantes em resposta ao estrogênio e compreender que a lógica de regulamentação exigiria segmentação potenciadores de múltiplos simultaneamente. Como os estudos iniciais usando interferência CRISPR em promotores sugeriram que nem todos os promotores são igualmente responsivos a repressão mediada por CASCUDO19, nós raciocinou que a adição de um domínio repressivo distinta para dCas9 pode facilitar a desativação de potenciadores de diversas. Escolhemos o interagindo na Sin3a domínio de Mad1 (SID)20 como ele leva para o recrutamento de histona deacetilases21, que removem grupos acetila em histonas que estão associados com a atividade transcricional. Importante, o domínio de SID foi eficaz em reduzir a expressão de gene quando fundido dCas922 e contos23, e Sin3a tem demonstrado ser um potente repressivo co-fator em uma variedade de potenciador sequência contextos24. Costumávamos a alvo 10 diferentes realçadores vinculados a ER SID4x-dCas9-CASCUDO (Enhancer-i) e identificar os locais obrigatórios de ER (ERBS) que são necessários para a resposta transcricional de estrogênio em 4 genes18. Também escolhemos as combinações de potenciadores para identificar os sites que cooperam na produção da resposta transcricional de estrogênio. Achamos que até 50 sites pode potencialmente ser alvo simultaneamente com as mudanças de expressão de gene detectável. Usando o ChIP-seq e RNA-seq, demonstrámos que Enhancer-i é uma técnica altamente específica para estudar potenciadores de múltiplos simultaneamente.
Este protocolo, descreveremos as etapas envolvidas na realização de Enhancer-i, uma técnica flexível que permite o estudo funcional de potenciadores de múltiplos simultaneamente em uma configuração de cultura de tecidos. Potenciador-i está altamente correlacionada com deleção genética mas fornece desativação transiente que é dependente de histona deacetilases (HDACs). Entregando a guia RNAs através de transfecção transiente em oposição a integração estável através de vetores virais, este protocolo evita deposição e potencial de disseminação de H3K9me3. Este protocolo detalhes guia RNA design e clonagem através de montagem de Gibson, o transfection de guia RNAs usando lipofection, e a análise resultante da expressão do gene alterado por qPCR. Nós também incluímos os métodos para avaliar a especificidade do Enhancer-i visando ao nível do genoma e transcriptoma. Enquanto esta técnica foi desenvolvida para estudar a regulação gênica por ER vinculado potenciadores em linhas de células cancerosas humanas, é aplicável para a dissecação de qualquer realçador de mamíferos.
Protocolo
1. geração de célula linhas estàvel expressando SID4X-dCas9-CASCUDO
Nota: A transfeccao condições e concentrações de drogas aqui apresentadas foram otimizadas para células de Ishikawa, uma linhagem de células de câncer de endométrio, cultivadas em meios RPMI 1640, suplementados com 10% FBS e 1% penicilina/estreptomicina (RPMI completa). Outras linhas de células podem exigir diferentes do transfection condições e concentrações de drogas. Os usuários também podem executar experimentos de transfecção transiente no selvagem-tipo pilhas, em vez de gerar uma linha de celular estável, com um plasmídeo expressando SID4X-dCas9-CASCUDO juntamente com RNA guia expressando plasmídeos; no entanto, resultados de transfections transitórias podem ser difícil de reproduzir como SID4X-dCas9-CASCUDO níveis podem variar por transfecção.
- Células de Ishikawa em pelo menos 2 poços de uma placa de 6 na confluência de 30-50% (aproximadamente 300.000 Ishikawa células) em 3 mL de RPMI completa da placa.
- Aspire os meios de comunicação das células. Lave as células uma vez com PBS 1x (pH 7,4). Aspirar a PBS e adicionar tripsina (4 mL para um prato de 10 cm ou 5 mL para um balão de T-75).
- Incube as celulas por ~ 5 min a 37 ° C, verificando a cada 2 min para células desanexadas e agitando suavemente o navio.
- Uma vez que as células têm destacado, pipete células trypsinized e descer algumas vezes e pipeta suavemente para o lado do navio para liberar quaisquer células anexadas.
- Transferir as células para um tubo cônico de 15 mL e girar as células para baixo por 5 min em x 250g.
- Aspirar a tripsina e ressuspender as células em 5-10 mL de mídia. Use uma pipeta P1000 para dissociar aglomerados de células, se necessário.
- Contar as células e determinar o volume necessário para placa ~ 300.000 células por poço em um volume total de 3 mL. Adicione as células 2 separados poços de uma placa de 6. Encha cada um bem para 3 mL com RPMI completa.
- Agite suavemente a placa cada 5 min nos primeiro 15 min após chapeamento para garantir que as células são distribuídas uniformemente no prato. Use um microscópio para garantir que as células têm dispersos do meio do poço.
- Dentro de 24 h de chapeamento, execute os seguintes transfections usando um reagente de transfeccao apropriado para a linhagem de células de interesse. Para células de Ishikawa, use o procedimento conforme descrito abaixo.
Nota: Este protocolo pressupõe a utilização de reagentes de transfecção baseada em lipossomas catiônicos. Electroporation fornece um método alternativo para tipos de células que são muito sensíveis a estes reagentes ou que apresentam eficiência de transfeccao baixa com lipofection. Condições de transfeccao devem ser otimizadas para a linhagem de células de interesse antes de tentar a experimentos Enhancer-i.- Em um tubo de Eppendorf de 1,7 mL, diluir 2,5 μg do plasmídeo SID4X-dCas9-CASCUDO e 800 ng do plasmídeo expressando uma proteína fluorescente em media serum-free, tal que o volume final no tubo é 155 µ l e a concentração final de plasmídeo é 0,020 µ g / µ l.
- Em outro tubo, dilua 3,3 µ g de um plasmídeo que não contém uma fita de resistência de neomicina, tais como pCMV-GFP, em media serum-free de modo que o volume final no tubo é 155 µ l e a concentração final de plasmídeo é 0,020 µ g / µ l.
- Vórtice cada tubo brevemente e spin para baixo usando uma microcentrífuga.
- Adicione 9,9 µ l de reagente de transfeccao (Tabela de materiais) para cada tubo. Misture vortexing brevemente a baixa velocidade. Spin os tubos para baixo com uma microcentrífuga.
- Incube os tubos à temperatura ambiente pelo menos 5 min, mas não mais de 20 min.
- Na biossegurança do armário, adicione 150 µ l da mistura preparada DNA: reagente gota a gota a um poço a placa de 6. Repita para o outro tubo de mistura de DNA: reagente preparado. Misturar as placas agitando suavemente e devolve a chapa para a incubadora.
- A transfeccao de post do dia 2, alterar a mídia e complementar com G418 para uma concentração final de 600 ng/μL. Esta concentração pode precisar de ser otimizado para o tipo de célula.
- Alterar a mídia RPMI completa e complementar com G418 todos os dias para 2-4 semanas até que as células transfectada de controle estão mortas e os poços contendo o SID4X-dCas9-CASCUDO se tornar confluentes. A quantidade exata de tempo necessário para as células recuperar dependerá o tempo de duplicação das células.
- Quando as células se tornam confluentes, passagem de um navio de T-25 ou T-75 em completar RPMI com uma dose baixa de G418 (300 ng/μL de células de Ishikawa). Durante esta passagem, fazer 2 alíquotas de ~ 100.000 células cada (aproximadamente 1/10th de uma placa de 6) para 2 tubos de Eppendorf separado 1,7 mL para isolamento de RNA e DNA, respectivamente. Girar estes tubos para baixo (5 min, 250g de x), remover tripsina pipetando e congelar os tubos a-20 ° C para uso futuro.
- Isolar o DNA genômico, usando kits comercialmente disponíveis e realizar PCR usando o "pAC95_PCR" ou "SID4X_PCR" as primeiras demão (tabela 1) para verificar a presença da proteína de fusão dentro da linha da célula. Use o DNA genômico extraído da linha parental como um controle negativo e SID4x-dCas9-CASCUDO Plasmídeo como controle positivo. Use uma mistura de mestre do polymerase de alta fidelidade com 50-100 ng de DNA genômico e as seguintes condições de ciclismo: 98 ° C por 30 s, 25 ciclos de (98 ° C, durante 10 s, 58 ° C por 30 s, 72 ° C por 2 min), 72 ° C por 5 min , segure a 4 ° C.
- Para verificar a expressão da proteína de fusão no nível do RNA, execute qPCR com RNA extraído de linhagem celular usando kits comercialmente disponíveis. Use os primers "dCas9_qPCR" (tabela 1) e o protocolo de uma etapa qPCR fornecidas na etapa 6.3 do presente protocolo.
- Para verificar a expressão do nível de proteína da fusão, realizar um Western mancha na lysates de linhagem celular. Usar antibandeira ou anti-HA anticorpos para detectar a proteína de fusão.
2. guia do RNA Design
Nota: Este protocolo é projetado para uso com o RNA de guia U6 clonagem vetor criado pelo laboratório de igreja e estão disponível em Addgene (Addgene 41824). Para criar uma versão deste vetor contendo resistência puromicina que permitiu a mesma estratégia de clonagem como 41824, mudamos o local múltiplo do clonagem do vetor para o vetor pGL3-U6-sgRNA-PGK-puromicina (Addgene 51133). Addgene 41824 ou nossa versão com puromicina (Addgene 106404) são compatíveis com a estratégia de clonagem apresentada abaixo.
- Obter basepairs 600-900 da sequência de DNA, para cada região reguladora de interesse. Use locais de ligação do fator de transcrição e/ou acessibilidade de cromatina para orientação sobre como definir a região de interesse (Figura 2A).
Nota: Enquanto o exemplo na Figura 2A apresenta potenciadores de montante e a jusante, é também possível alvo localizados dentro os intrões dos elementos reguladores. - Coloque todas as sequências obtidas em um arquivo de texto simples usando o formato FASTA.
- Identifica pelo menos uma região de controlo negativo que não deverá mudar sobre condições experimentais, tais como um promotor de um gene que não é expressa em linhagem celular de interesse. Obter a sequência de DNA para esta região e adicioná-lo para o arquivo de texto no formato FASTA.
Nota: Nós usamos o guia RNAs alvejando o IL1RN promotor25 como um controle negativo para todas as regiões, temos como alvo. Os usuários também podem selecionar a sequência intergênica próximo a região de interesse que não contenha locais de ligação do fator da transcrição como controlo negativo. No entanto, se múltiplos loci é alvo simultaneamente, uma região de controlo negativo único simplifica delineamento experimental e interpretação dos resultados. Se o alvo enhancer é intrônicas, pode ser útil para uma região intrônicas no mesmo locus que não contém um elemento regulamentar putativo como um controle negativo adicional, como a fusão de dCas9 pode interferir com a transcrição de destino. - Identifica regiões de controle positivo, como os promotores que são os alvos putativos das regiões reguladoras de interesse ou promotores de genes que são altamente transcritas em linhagem celular de interesse. Obter a sequência de DNA para essas regiões e adicioná-lo para o arquivo de texto no formato FASTA.
- Use um programa como o e-batata frita26 (http://www.e-crisp.org/E-CRISP/) sobre as sequências de DNA geradas para encontrar o guia de RNAs com baixos fora-alvos (idealmente 0-3). Guia RNAs consistem de 20 nucleotídeos a montante de um protospacer adjacentes do motivo (PAM), que assume a forma "NGG" para o dCas9 de S. pyogenes.
- No site do e-batata frita, selecione o organismo de interesse usando o menu drop-down. A montagem do genoma aparece à direita do nome da espécie.
- Selecione o botão de entrada é a sequência FASTA . Copie as sequências FASTA de cima e cole-os na caixa de diálogo. Certifique-se de que um cabeçalho FASTA está incluído para cada sequência.
Nota: Até 50 sequências podem ser consultadas simultaneamente. - Selecione o botão de rádio médio e design único no menu drop-down.
- Clique no botão Iniciar pesquisa sgRNA. Vai abrir uma nova aba do navegador, e os resultados serão exibidos. Baixe as sequências de candidato clicando no botão baixar um relatório tabular Excel formatado para todas as sequências de consulta juntos.
- Abra o relatório tabular usando o Excel ou um programa de edição de texto.
- Use o pesquisador de genoma UCSC para sequências de gRNA completos de candidato BLAT (23 basepairs) para o genoma.
- Em um navegador, navegar até o site do UCSC genome navegador (http://genome.ucsc.edu). Sob a seção nossas ferramentas, localize a palavra BLAT e clique sobre ele. A ferramenta de busca BLAT será aberto.
- Use os menus drop-down localizados sob o texto BLAT pesquisa do genoma para selecionar o conjunto de organismo e genoma de interesse.
- Copie as sequências de RNA guia do relatório tabular gerado pelo e-batata frita e cole-os na caixa de diálogo. Verifique se cada sequência tem um único cabeçalho FASTA, clique em Enviar na parte inferior da caixa de diálogo.
Nota: Até 25 sequências podem ser examinadas imediatamente. Na página de Resultados de pesquisa BLAT , alinhamentos de cada guia sequência de RNA aparecerá, com cada linha que representa um alinhamento. Idealmente, deve haver um alinhamento para cada guia de RNA, indicando a exclusividade de que o guia do RNA. - Evite os guias que alinham a vários locais no genoma se possível.
- Para examinar a guia RNA localização e distribuição dentro da região de interesse, clique no link navegador sob a seção ações para um dos RNAs guia consultado. O navegador do genoma irá aparecer e será centralizado na guia selecionado de RNA. Use os botões de Zoom na parte superior da página para visualizar a distribuição de outro guia RNAs identificados por e-batata frita dentro da região de interesse.
- Selecione 4 de preferência não-sobreposição guia sequências de ARN que são distribuídas em toda a região de interesse (Figura 2B). Se a região de interesse exceder 600 bp, considere a adição de 1-2 guias adicionais. Evite guia RNAs com trechos de homopolymeric e extremo conteúdo GC, pois estas características podem dificultar o RNA guia processo de clonagem e reduzir RNA guia direcionamento de eficiência.
- Uma vez que os guias foram selecionados, criar um arquivo que contém o guia completo sequência de RNA (23 nucleotídeos) para cada guia desejado e em seguida, remover o nucleotídeo 5' bem como o PAM (NGG) da extremidade 3'. Este passo facilita a ordenação de oligo.
- Adicionar a seguinte sequência à extremidade 5' da sequência do oligonucleotide: GTGGAAAGGACGAAACACCG.
- Adicionar a seguinte sequência à extremidade 3' da sequência do oligonucleotide: GTTTTAGAGCTAGAAATAGC.
Nota: A sequência final deve ser 59 nucleotídeos há muito tempo e olha como este: GTGGAAAGGACGAAACACCG-alvo (19 nt)-GTTTTAGAGCTAGAAATAGC. - Certifique-se de que cada elemento regulatório a ser voltado com Enhancer-i tem pelo menos 4 oligonucleotides exclusivos projetados para ele. Ordene essas sequências juntamente com os primers "U6_internal" listados na tabela 1.
3. guia do RNA clonagem
Nota: RNA guia clonagem através de montagem de Gibson provou para ser altamente eficiente em nossas mãos, gerando centenas de colônias por placa, com poucas ou nenhumas colônias presentes no único controle de vetor. Tal eficiência é fundamental para manter a complexidade durante a clonagem em pool. Outra vantagem do conjunto de Gibson clonagem é que os usuários não precisam se preocupar com a presença de uma enzima de restrição corta site no guia de RNA estão tentando inserir o vetor de clonagem U6. No entanto, este protocolo pode ser adaptado para tradicional enzima de restrição com base clonagem se desejado.
- Reconstitua os oligos guia RNA em uma concentração final de 100 μM em água ultrapura (RNase-livre, livre de DNase). Deve haver pelo menos 4 oligos RNA de guia separado para cada região de interesse.
- Para cada região reguladora de interesse, crie um pool de todos os oligos correspondentes para a região de interesse. Em um tubo de Eppendorf, combine 5 μL de cada oligo de RNA guia reconstituído individuais para cada região. Misture a piscina bem vortexing, em seguida, remover 1 μL e diluir esta alíquota 1: 200 em água ultrapura.
Nota: Se desejar, estas piscinas segmentando regiões reguladoras individuais podem ainda ser combinadas para gerar uma complexa piscina segmentando regiões múltiplas. Até 50 regiões reguladoras podem ser alvo simultaneamente em um único pool (Figura 3). - Realize um curto PCR com primers U6 para anexar as regiões de homologia os oligos antes da montagem de Gibson. Cerca de 40 bases serão adicionadas para cada oligo, produzindo um produto bp ~ 100 que contém homologia suficiente para o vetor de U6 em ambas as extremidades.
- Para cada guia pool de RNA, configurar um 20 μL do PCR com uma mistura de mestre do polymerase de alta fidelidade e os seguintes componentes: 1 μL de oligo diluídos piscina de passo 3.2, 1 μL de U6 frente primer (10 μM), 1 μL de U6 reverter a primeira demão (10 μM) e até 20 μL de água.
- Incubar em um termociclador com as seguintes condições: 98 ° C por 30 s, 10 ciclos de (98 ° C, durante 10 s, 55 ° C por 30 s, 72 ° C por 2 min), 72 ° C por 5 min e espera a 4 ° C.
- Execute 5 μL da reação em um gel de agarose 1-2% com uma escada de baixo peso molecular. O produto final deve ser basepairs ~ 100 (Figura 2).
- Limpar a reação de extensão com um kit de purificação de DNA baseado em coluna e eluir em 20 μL de tampão de eluição, fornecido no kit.
Nota: Como o produto é curto, evitar o uso do grânulo-baseado limpezas, que são projetadas para excluir fragmentos pequenos, menor que 100 bp. - Quantificar o DNA purificado utilizando um dados ou espectrofotômetro (esperado rendimento é de 10-20 ng / µ l). Inserções de guia do RNA podem ser armazenadas a-20 ° C, ou podem ser usadas imediatamente em montagem de Gibson com um vetor de U6 tornado linear.
- Para preparar o destinatário U6 vetor de clonagem para montagem de Gibson, configure um digest de enzima de restrição. Se muitas reações de montagem Gibson devem ser executadas, configurar vários sumários para assegurar rendimento suficiente do vetor de corte.
- Use 20 unidades de enzima AflII e 1 μg do plasmídeo em uma reação de 20 μL com o buffer de enzima de restrição apropriada. Incube a 37 ° C, durante 1-2 h.
- Limpar o digest com grânulos ou um kit com base em coluna para a purificação de DNA e eluir em 20 μL de tampão de eluição. Quantificar o DNA purificado utilizando um dados ou Espectrofotômetro. As amostras podem ser congeladas a-20 ° C para uso posterior.
- Executar montagem de Gibson sobre o vector preparado e inserir.
- Configure as reações de montagem Gibson no gelo. Uso 50 ng do vetor e 7 ng da inserção em uma reação de 20 μL. Dilua as inserções 01:10 em água ultrapura para facilitar a pipetagem. Configurar uma vetor único Gibson montagem reação, usando 50 ng do vetor e substituindo o folheto com água.
- Incubar as reações de montagem Gibson por 15 min a 50 ° C, seguido de uma espera a 4 ° C.
- Transferi os produtos montados em gelo. Dilua os produtos montados 1:4 em água ultrapura no gelo. Por exemplo, adicione 5 μL de produto de montagem Gibson para 15 μL de água ultrapura.
- Transforme os produtos de montagem Gibson diluídos.
- Descongelar as células competentes de alta eficiência no gelo e fazer 25 alíquotas μL de cada transformação. Se for desejada uma complexa piscina em sítios múltiplos, descongele células suficientes para tubos diferentes para executar várias transformações independentes da mesma piscina complexo gRNA.
- Para cada produto diluído, adicione 1 μL desta diluição para um 1,7 mL tubo Eppendorf contendo 25 μL de células competentes. Misture rapidamente, passando rapidamente o tubo. Incube os tubos em gelo por 30 min.
- Choque de calor das células por 30 s a 42 ° C, depois transferir imediatamente para o gelo por 2 min.
- Adicionar 300 μL SOC mídia (triptona de 2%, 0,5% de extrato de levedura, 10 mM de NaCl, 2,5 mM KCl, 10 mM MgCl2, 10 mM de MgSO4e 20 mM de glicose) e deixar que as células recuperar durante 1 h a 37 ° C, com agitação (300 rpm). Durante este tempo, aqueça as placas de ágar com ampicilina/carbenicilina a 37 ° C numa incubadora. Use um prato para cada transformação.
- Placa 50 μL de células e colocar as placas em uma incubadora de 37 ° C durante a noite. Para as piscinas em sítios individuais, coloque directamente no 3-5 mL de caldo LB (Tabela de materiais) contendo ampicilina/carbenicilina (1 mg/mL) e incubar durante uma noite com agitação a 250 rpm a 37 ° C para minipreps.
- As células da colheita e isolar o DNA.
- Para bibliotecas de RNA guia grande segmentação de vários sites, use um raspador de placa para coletar todas as colônias de cada prato individual em um maxiprep (cultura líquida de 150 mL). Isto pode ser facilitado pela derramando ~ 5 mL de LB com o antibiótico adequado em um tubo falcon de 50ml e raspagem das colônias no tubo. Para bibliotecas individuais em sítios, raspe as placas em um miniprep (cultura líquida de 3-5 mL).
- Incube a estas culturas com o antibiótico adequado para 3-5 h a 37 ° C com agitação a 250 rpm.
- Realize a extração de DNA usando um kit que resulta em preparações livre de endotoxinas.
- Quantificação de DNA usando um dados ou Espectrofotômetro. Plasmídeos podem ser usados imediatamente na transfecção ou armazenados a-20 ° C para uso futuro.
- Para confirmar a presença da sequência de RNA guia dentro do vetor de U6 para pequenas piscinas em sítios único, use Sanger sequenciamento sobre o miniprep preparado com o primer "U6_PCR_R" listados na tabela 1. Devido ao pool de guia RNAs, a sequência de destino 19 basepair gRNA renderá bases misturadas, mas o promotor U6 e andaime de RNA guia rodeiam esta sequência devem ser intactos.
4. a transfeccao de Enhancer-i
Nota: Para o bloqueio bem sucedido de uma resposta de estrogênio usando Enhancer-i nas células de Ishikawa, é necessário privar as células de estrogênio para 5-7 dias antes do transfection, mantendo-os em vermelho de fenol livre RPMI com 10% carvão-despojado FBS e 1% penicilina/estreptomicina. Células devem ser cultivadas nessa mídia durante e após a transfeccao se tentar bloquear uma resposta de estrogênio. Recomendamos o uso de tripsina livre de vermelho de fenol para passagem de células em completa vermelho de fenol livre RPMI.
- O dia antes de transfeccao, placa as células (o selvagem-tipo ou expressando estàvel CASCUDO-SID4X-dCas9) em uma placa de 24 na confluência de 30-50% (~ 60.000 células por poço para células de Ishikawa). Placa de células suficientes tais que transfections podem ser realizadas em duplicata e incluem poços para transfected com guia controle RNAs. Certifique-se de que as células estão distribuídas uniformemente através do poço sacudindo suavemente a placa depois de chapeamento de célula como na etapa 1.1.7.
Nota: Este protocolo pressupõe a utilização de reagentes de transfecção baseada em lipossomas catiônicos. Electroporation fornece um método alternativo para os tipos de células que são muito sensíveis a estes reagentes. Condições de transfeccao devem ser otimizadas para a linhagem de células de interesse antes de tentar a experimentos Enhancer-i. - No dia seguinte, prepare transfections conforme as instruções do reagente de Transfeccao de escolha. Para células de Ishikawa, uso 550 ng do plasmídeo total para cada poço de uma placa de 24. Dilua o plasmídeo para uma concentração final de 0,020 μg/μL em media serum-free (1.1 μg de DNA em 52 μL de volume total para transfecting 2 poços). Use 3 µ l de reagente de Transfeccao para cada 1 μg de DNA, vórtex e incubar conforme descrito na etapa 1.2. Adicione 25 μL da mistura final a cada poço.
Nota: A combinações de alvo de sites, use o mesmo peso do plasmídeo para cada site individual e em seguida, preencher o peso restante com um plasmídeo controle (RNA guia vazia clonagem vetor ou guia RNAs visando uma região de controlo negativo como o IL1RN promotor). Para transfections transitórias, use uma proporção de 3:2 Cas9 plasmídeo de RNA fusão: guia. Plasmídeos contendo repórteres fluorescentes podem ser adicionados para monitorar a eficiência do transfection. - A transfeccao de post de 36h, mudar a mídia usando o vermelho de fenol livre RPMI com 10% carvão-despojado FBS e 1% penicilina/estreptomicina (para células de Ishikawa) e fornecer puromicina (concentração final: 1 μg/mL) e neomicina (concentração final: 300 ng/mL). Se as células são sensíveis ao reagente de transfeccao, a mídia pode ser alterada mais cedo, mas devem ser adicionados os antibióticos não mais cedo do que de Transfeccao de post de 24 h.
Nota: Aguarde pelo menos 24 h após a adição de antibiótico antes da colheita de células. Alterações de expressão devido a Enhancer-i podem ser detectadas como cedo como 48 h post transfeccao e acima de 5 dias postar do transfection. Se trabalhando com células de Ishikawa que foram privadas de estrogênio, executar um 8-h 10 nM 17 β-estradiol (E2) indução do dia após o tratamento antibiótico e em seguida colher células imediatamente.
5. colheita e extração de RNA de células
- Prepare o tampão de Lise com 1% de β-mercaptoetanol (BME). Certifique-se de que não há mais mistura de Lise-BME (300 μL de cada bem sendo colhidas).
- Aspire a mídia usando um aspirador a vácuo.
- Lavar as células uma vez com um volume igual de 1X PBS (500 µ l) e Aspire para remover tanto PBS quanto possível.
- Adicione 300 μL de solução de Lise-BME a cada poço usando uma pipeta multicanal. Adicionar a solução de Lise acima e para baixo de 8 - 10 vezes e transferir para uma placa de fundo ou 1,7 mL Eppendorf tubos no gelo. RNA pode ser extraído imediatamente, ou lysates pode ser congelado a-80 ° C para processamento futuro.
- Para extrair o RNA de lisados, use um kit comercialmente disponível, que inclui um tratamento de DNase. Eluir o menor volume recomendado de água ultrapura (RNase-livre, livre de DNase) ou eluição do buffer e quantificar o RNA. Para pequenas quantidades de amostras, use um dados ou Espectrofotômetro. Para grande número de amostras, use uma sonda fluorescente que detecta o RNA e medida em um leitor de placa. As amostras podem ser congeladas a-80 ° C, antes ou depois da quantificação.
6. quantificar as mudanças de expressão de Gene usando uma etapa qPCR e RNA-seq
- Obter qPCR cartilhas para os genes de interesse e para o gene de pelo menos um serviço de limpeza que é expresso perto do nível de genes alvo e não mudam em condições experimentais. Idealmente, estas primeiras demão vai abranger uma junção exon-exon para evitar a amplificação de DNA genômico.
- Teste estas primeiras demão sobre RNA obtidos a partir da linha de células de interesse. Uso do derretimento análise de curva para verificar a produção de um único produto. Se não é produzido um único produto, teste pares adicionais da primeira demão.
- Para cada amostra de RNA Tratado Enhancer-i e controle guia, identifica quantos genes devem ser analisados na amostra. Este conjunto de genes deve incluir genes das tarefas domésticas, tais como CTCF ou GAPDH.
- Configure as reações qPCR.
- Diluir todas as amostras à mesma concentração na água, tal que 50 ng do RNA total é pipetado facilmente e há bastante diluído RNA para cada reação. Por exemplo, diluir o RNA para ~16.6 ng/μL e usar 3 μL de RNA em cada reação. Manter RNA no gelo durante a criação de misturas de mestre.
- Prepare misturas mestre separadas para cada gene a ser medido usando kits de qPCR comercialmente disponível de uma etapa. Use um volume de reação de 20 μL com 1 μL de cada primer (solução estoque de 10 μM). Configure essas reações no gelo.
- Em uma placa de reação que é apropriada para o termociclador, adicionar seguidas pelo mestre misturas de amostras do RNA. Selar com um aferidor de prato e misture suavemente pela utilização do Vortex ou pipetagem. Brevemente, centrifugar a placa (140 x g durante 60 s) assegurar o líquido está no fundo dos poços.
- Incubar a placa em um termociclador como segue (ou como kit instrui): 48 ° C por 30 min, 95 ° C por 10 min, 40 ciclos de (95 ° C por 15 s, 60 ° C por 1 min).
- Obter valores de Ct para cada gene medido dentro de cada amostra. Use o método comparativo de Ct para identificar alterações na expressão gênica.
- Subtrai o Ct do gene das tarefas domésticas do Ct de cada gene de interesse para cada amostra para gerar valores de Ct normalizados.
- Para as amostras de controlo de tratados, tire uma média dos valores de Ct normalizados para cada gene. Registro base 2-escala prega repressão então pode ser calculado subtraindo-se a amostra de Enhancer-i Tratado normalizada Ct para cada gene do mesmo valor para as amostras de controlo de tratados para o gene correspondente.
- Para determinar as mudanças globais na expressão do gene Enhancer-i no tratamento, prepare as amostras para sequenciamento de RNA usando um kit disponível comercialmente compatíveI com a tecnologia de sequenciamento do usuário. Uso ~ 500 ng do RNA para começar o material e preparar as bibliotecas para pelo menos 2 réplicas biológicas.
7. verificação de direcionamento genômico específico por SID4X-dCas9-CASCUDO usando ChIP-seq
Nota: A proteína de fusão de SID4X-dCas9-CASCUDO contém uma tag epítopo da bandeira e uma marca de epítopo HA, mas com anticorpos anti-FLAG, obtiveram-se melhores resultados para o ChIP-seq. Se desejado, o usuário pode executar experimentos adicionais de ChIP-seq para fatores de transcrição potencialmente afetados pela Enhancer-i, ou H3K27ac, uma marca da atividade de potenciador que é diminuída por Enhancer-i. No entanto, cada ChIP-seq experimento requer 10 x 106 células, tão plano nesse sentido.
- Transfect células com piscinas Enhancer-i.
- Placa 10 x 106 células numa placa de cultura de tecidos de 15 cm. Cada prato representa 1 ChIP-seq experimento para 1 fator de interesse.
- No dia seguinte, transfect as células usando 20 μg de DNA total por prato. Para transfections em linhagens celulares expressando estàvel SID4X-dCas9-CASCUDO, o DNA deve ser uma piscina de plasmídeo de guia RNAs todos os sítios de interesse e opcionalmente um plasmídeo expressando a proteína fluorescente. Para transfections transitórias, use uma proporção de pool fusão RNA da proteína: guia de 3:2 dCas9. Para ChIP-seq de outros TFs ou modificações do histone, execute pelo menos uma transfecção de guia RNA controle adicional em outro prato.
- Trate os pratos com puromicina (1 μg/mL) e neomicina (300 ng/mL) para transfeccao de post de 24-48 h. Espere pelo menos 24 h antes da colheita da cromatina.
- Colheita de cromatina de pratos.
Nota: Para estudar os efeitos do Enhancer-i na ligação genômica de ER em células de Ishikawa, realizar um tratamento de nM E2 1 h 10 pratos transfected com guia controle RNAs e Enhancer-i antes de colher. Para estudar os efeitos do Enhancer-i na H3K27ac, executar um 8 h 10 nM E2 indução em pratos transfected com guia controle RNAs e Enhancer-i antes de colher.- Aplique 500 μL de formol 37% para cada prato (concentração final de 1%). Agite as placas brevemente. Deixe as placas sentar-se à temperatura ambiente por 10 min.
- Adicione 1 mL de glicina 2,5 M (concentração final de 125 mM). Agite as placas brevemente.
- Deite fora a mídia com formaldeído e glicina. Adicione um volume igual (~ 20 mL) de frio 1X PBS.
- Despeje a PBS. Aspirar com um aspirador de vácuo para remover tanto PBS quanto possível. Coloque os pratos no gelo.
- 3-5 ml de frio 1X PBS ou Farnham tampão de lise (pH de tubos de 5mm 8.0, 85 mM KCl, 0,5% NP-40) com 1 x inibidor da protease (adicionado apenas antes de usar) para cada placa. Raspe o prato com um raspador de placa e transferir a solução para um tubo cônico de 15 mL no gelo.
- Cromatina de pelotas por girando para baixo os tubos em uma centrífuga por 5 min a 4 ° C, a 1000 x g descartar o sobrenadante e armazenar as pelotas a-80 ° C para uso futuro ou proceder com protocolo de escolha usando um anticorpo antibandeira ou anticorpos como alvo outro ChIP-seq factores de transcrição ou modificações do histone de interesse (H3K27ac, H3K9me3).
Resultados
A Figura 1 mostra um diagrama de fluxo de trabalho descrito no protocolo. Para determinar as contribuições dos realçadores de ER-limite perto o estrogênio-regulado do gene MMP17, que tem 3 sítios de ligação nas proximidades, definidos pelo ChIP-seq (Figura 2A), guia de RNAs foram projetados para cada região. Para a concepção de guia RNAs, uma janela de bp de 600-900 da sequência que cercam cada ER local obrigatório de interesse foi selecionado e colocar em um programa de design de RNA guia. RNA resultando guia sequências com 0-2 prevista fora do alvo locais foram alinhados para o genoma humano usando BLAT. Quatro não-sobreposição guia RNAs que atravessavam a região definida pelo ChIP-seq e DNaseI hipersensibilidade foram escolhidos para o direcionamento (Figura 2B). Sequência adicional (tabela 1) foi adicionada a cada extremidade para facilitar a jusante de clonagem e o resultante 59 fragmentos de nucleotídeos foram ordenados. À chegada, guia RNAs foram diluídos e agrupados por site e um curto PCR foi realizado para adicionar regiões de homologia antes da montagem de Gibson. A Figura 2 mostra o produto de RNA guia esperado após um curto PCR utilizando primers "U6_internal" (tabela 1), que irão adicionar 20 basepairs de sequência para cada extremidade do basepair 59 guia fragmento de RNA, resultando em uma sequência de basepair ~ 100. Na sequência de montagem de Gibson, estas piscinas guia RNA foram transformadas em bactérias e plasmídeo minipreps foram preparados no dia seguinte. Figura 2D mostra resultados de um experimento de dissecação potenciador, onde múltiplos potenciadores nas proximidades de MMP17 são direcionados em paz e em combinação usando Enhancer-i. Sites direcionados por Enhancer-i são indicados com um hexágono preto. Plasmídeos de RNA guia a indicada em sítios foram transfectados em uma linhagem de células de Ishikawa privado de estrogênio estàvel expressando SID4X-dCas9-CASCUDO. Dois dias depois, a mídia foi alterada e puromicina foi adicionada para enriquecer para células transfectadas. No dia seguinte, as células foram colhidas após um tratamento de estradiol 8 h 10 nM. RNA foi isolado, e um One-Step qPCR foi realizada. Neste exemplo, sites 1 e 2 são necessários para uma completa resposta estrogênica de MMP17, enquanto o site 3 não contribui sob essas condições (Figura 2D, pistas ii-iv). Quando apenas sites de 2 ou 3 são ativos (vi e vii), a resposta de estrogênio é semelhante a quando não locais são ativos (viii), sugerindo que esses sites não podem contribuir de forma independente. Local 1 pode contribuir alguma expressão por si só (v), mas a maior atividade é vista quando sites 1 e 2 são ativos (iv).
Para manipular 10 potenciadores perto de 4 genes diferentes simultaneamente (Figura 3A), complexas piscinas de guia RNAs foram gerados contendo 42 potenciador de guias e 16 guias de promotor. Guia do RNA oligos foram agrupados antes o guia inicial extensão RNA PCR (passo 3.3), e produtos resultantes da PCR foram purificados e combinados com o vetor de clonagem puromicina vazio U6 usando assembly de Gibson. Após a montagem de Gibson, várias transformações independentes foram realizadas e chapeadas. As placas foram raspadas em LB e permitiu a crescer para 2-4 h antes de maxiprep. Figura 3B mostra representativas reduções na expressão gênica por qPCR quando esses pools de RNA guia foram transfectadas em uma linhagem de células de Ishikawa privado de estrogênio estàvel expressando SID4X-dCas9-CASCUDO e tratados conforme descrito acima (Figura 2D). Reduções de Enhancer-i são semelhantes aos obtidos pela segmentação do promotor do gene alvo putativo. A Figura 3 mostra os efeitos de diluição de guia RNAs na redução da resposta estrogênio usando Enhancer-i. Um 01:50 diluição de um pool de guia RNA visando o realçador perto G0S2 ainda produz redução significativa na expressão gênica, sugerindo que o Enhancer-i pode ser usado para atingir até 50 sites de uma só vez. No entanto, a desativação pode ser diluída, indicando que centenas de sites não podem ser alvo simultaneamente, a menos que os métodos de deteção mais sensíveis são empregados.

Figura 1. Protocolo esquemático para dissecação potenciador multiplex usando Enhancer-i. Guia de RNAs (vermelhos e azuis) são projetados usando e-batata frita e selecionado usando o UCSC genome browser. Guia quatro RNAs são escolhidos que abrangem as regiões de interesse (transcrição locais obrigatórios do fator conforme definido pelo ChIP-seq). Oligonucleotídeos de RNA guia que têm sido colocados em pool pela região de interesse (vermelho e azul) passam por um PCR para adicionar regiões de homologia (laranja) antes da montagem de Gibson e transformação. Piscinas de plasmídeo resultante são transfectadas através de lipofection em linhagens celulares expressando estàvel CASCUDO-SID4X-dCas9 ou em células de tipo selvagem, em conjunto com o plasmídeo SID4X-dCas9-CASCUDO. Guia do RNA do plasmídeo piscinas podem transfected individualmente para um site de destino de cada vez, ou em combinação para atingir vários sites simultaneamente. Células transfectadas são tratadas com antibióticos para enriquecer para células contendo guia de RNAs. A transfeccao de post ~ 72 h, as células são colhidas. Os ácidos nucleicos podem ser extraídos para qPCR, RNA-seq ou ChIP-Seq. clique aqui para ver uma versão maior desta figura.
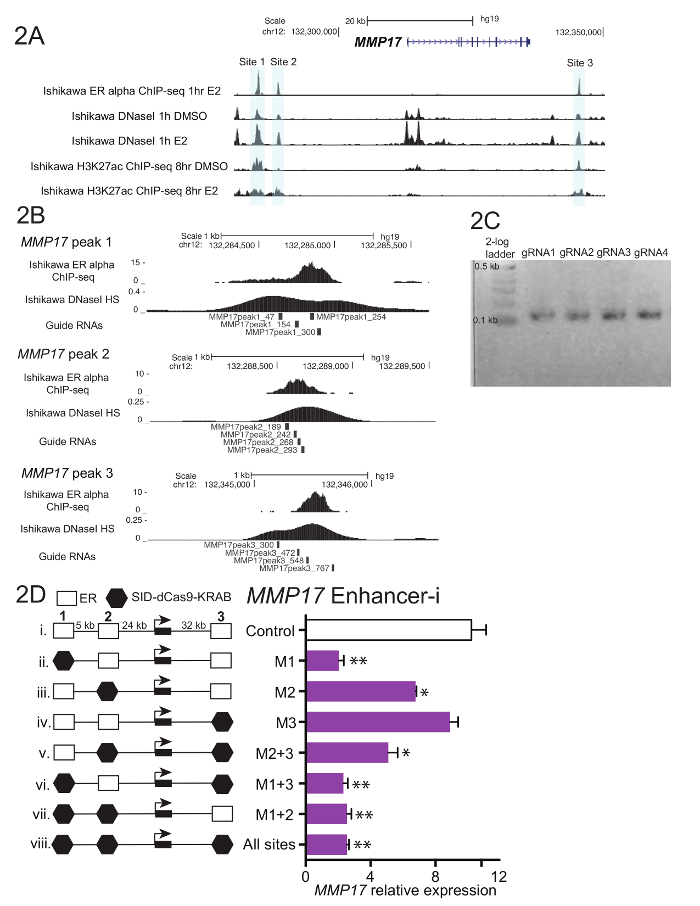
Figura 2. Guia de dissecação de design e potenciador de RNA para MMP17. (A) genoma navegador screenshot dos realçadores alfa-limite ER (cinza) ser alvo perto MMP17. Esta figura foi modificada de Carleton, et al 18. (B) guia de RNA projeta para o 3 vinculação sites18. O sítio de ligação para ER, conforme definido pelo ChIP-seq é o alvo e a telha de RNAs 4 guia em toda esta região. O sinal de sensibilidade DNaseI, que atravessa o sítio de ligação, também pode ser usado para definir a sequência de destino para o guia de design de RNA. Tanto ChIP-seq e dados DNaseI HS foram obtidos de Ishikawa células tratadas com estradiol de nM 10 para sequências de RNA de guia representante (C) h. 1 que estão prontos para a montagem de Gibson, tendo sofrido uma PCR curto para adicionar regiões de homologia. (D) expressão relativa da MMP17 medido através de qPCR seguindo o direcionamento de regiões específicas com Enhancer-i e um tratamento de estradiol h10-8 nM. Expressão é em relação ao nível de MMP17 em células não tratadas com estradiol CTCF e expressão. Guia de controle RNAs alvo o promotor da IL1RN. Todas as barras de erro representam SEM, asteriscos duplos indicam p < 0,01 e único asteriscos indicam p < 0.05 em um teste t pareado. Esta figura foi modificada de Carleton, et al. 18. clique aqui para ver uma versão maior desta figura.
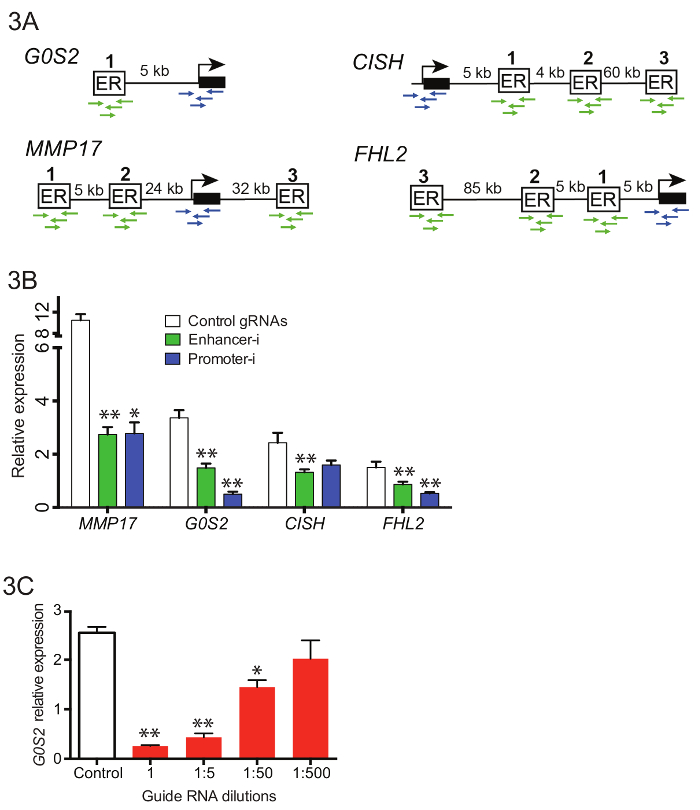
Figura 3. Como alvo múltiplas potenciadores perto genes diferentes simultaneamente com pool Enhancer-i. (A) diagrama esquemático dos sítios de ligação e promotores para ser alvo de Enhancer-i em pool. (B) os efeitos na expressão medida pelo qPCR após tratamento E2 em Ishikawa células transfectadas com piscina de plasmídeo Enhancer-i (verde), piscina de plasmídeo de promotor-i (azul) ou controle gRNAs (branco)18. Observa-se uma redução significativa em todos os genes com Enhancer-i. Esta figura foi modificada de Carleton, et al. 18. (C) os efeitos sobre os níveis de expressão de G0S2 após o tratamento de E2 em Ishikawa células transfectados com diferentes quantidades de guia RNAs segmentação G0S2. Uma redução significativa pode ser vista mesmo com pequenas quantidades de guia do RNA (01:50 diluição), sugerindo que até 50 sites pode alvo simultaneamente. Todas as barras de erro representam SEM, asteriscos duplos indicam p < 0,01 e único asteriscos indicam p < 0.05 em um teste t pareado. Clique aqui para ver uma versão maior desta figura.
| Nome | Sequência de |
| U6_internal_F | TTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCG |
| U6_internal_R | GACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC |
| U6_PCR_F | CCAATTCAGTCGACTGGATCCGGTA |
| U6_PCR_R | AAAAAAAGCACCGACTCGGTGCCA |
| gRNA_qPCR_F | GCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCG |
| gRNA_qPCR_R | AAAAAGCACCGACTCGGTGCC |
| dCas9_qPCR_F | GTGACCGAGGGAATGAGAAA |
| dCas9_qPCR_R | AGCTGCTTCACGGTCACTTT |
| pAC95_PCR_F | AGAAGAGAAAGGTGGAGGCC |
| pAC95_PCR_R | CGTCACCGCATGTTAGAAGG |
| SID4X_PCR_F | CAATAGAAACTGGGCTTGTCG |
| SID4X_PCR_R | TCGTGCTTCTTATCCTCTTCC |
Tabela 1. Primers utilizados para extensão de RNA guia e sequenciamento, qPCR e a detecção da proteína de fusão.
Discussão
Este protocolo descreve um método simples e flexível para dissecar função potenciador no locus genômico endógena sem fisicamente alterar a sequência de DNA. Enquanto similar em conceito a protocolos de interferência CRISPR anteriormente publicados usando dCas9-CASCUDO27, Enhancer-i difere esses protocolos de 3 maneiras. Primeiro, Enhancer-i utiliza o domínio interagindo SIN3A de MAD120 para alcançar a desativação do realçador. Desativação de potenciador pode ser resgatada usando inibidores HDAC, sugerindo que o mecanismo primário de desativação é dependente de HDAC. Ao contrário de interferência CRISPR com dCas9-CASCUDO, Enhancer-i não leva à deposição de H3K9me3. Isto é provavelmente devido ao fato de que Enhancer-i baseia-se na transitória introdução guia RNAs, para o post com as células, sendo colhidas aos 3 dias do transfection. Interferência de CRISPR, observa-se um aumento na H3K9me3 em 7 dias post transdução12. Finalmente, o protocolo de Enhancer-i fornece uma estratégia para vários sites de destino simultaneamente e monitorar a eficiência da segmentação. Em mosaico-seq17, dCas9-CASCUDO é usado para direcionar potenciadores de múltiplos simultaneamente, mas esta técnica se baseia em sequenciamento de RNA monocelulares identificar alterações de expressão, e muitos genes (tais como genes hormona-responsivo) passam despercebidos devido a baixa sensibilidade da célula única RNA-Seq Enhancer-i fornece um método confiável para estudar potenciadores individualmente e em combinação para qualquer gene.
A etapa mais crítica de Enhancer-i é transfeccao, quais deve ser otimizado para a linhagem de células de interesse. Este protocolo se baseia em puromicina enriquecer para células transfectadas, mas é possível que o co transfecting guia RNAs com uma proteína fluorescente e a classificação para as células fluorescentes usando citometria de fluxo pode vir a ser um melhor método de enriquecimento para uma célula tipos. Recomenda-se monitoramento do nível de expressão do guia RNAs e SID4x-dCas9-CASCUDO por qPCR para solucionar problemas e confirmar a transfeccao. Se a guia RNA níveis são baixos (ciclo limite > 30), os usuários também podem considerar guia alternativo estratégias de produção de RNA como em vitro transcrição28. Também é possível que apesar de gRNA altos níveis, guia RNA alvo da proteína SID4x-dCas9-CASCUDO é ineficiente, caso em que selecionando diferentes: guia de sequências de RNA pode ser necessário. Por meio de ChIP-seq a proteína de fusão com cromatina das células tratada Enhancer-i, a eficiência de segmentação pode ser monitorada. Se houver sinal elevado de SID4x-dCas9-CASCUDO na região de interesse, e sem alterações de expressão em seu gene alvo putativo são detectadas, então a região provável não contribui para a expressão de que gene nas condições estudadas.
Uma limitação potencial de Enhancer-i é que efeitos fora do alvo podem acumular-se se há muitos sites são direcionados simultaneamente. No entanto, estratégias de interferência CRISPR por nocaute tem menos fora alvo dos efeitos do RNAi29, especialmente quando é usada uma linha de celular policlonal expressando dCas9-CASCUDO. Enquanto nós vimos o alvo genômica vinculação de SID4X-dCas9-CASCUDO ao direcionamento 10 sites simultaneamente, não identificamos as mudanças de expressão de gene como resultado desses eventos de vinculação. Como alguns intensificadores podem contatar vários promotores e/ou outros potenciadores, é possível que muitos genes podem mudar a expressão sobre o direcionamento de um único potenciador, embora não está claro se esta forma de regulação genética é comum. Para confirmar que as mudanças de expressão observaram são devido ao direcionamento de um específico potenciador e não fora do alvo os efeitos, os usuários podem executar Enhancer-i com dois conjuntos distintos de disjunto guia RNAs direcionamento da mesma região. Além disso, a exclusão genética da região usando Cas9 nuclease-competente pode confirmar ainda mais seus efeitos na expressão gênica.
Como funções de Enhancer-i através da deacetilação de histona, é possível que suas habilidades de desativação são limitadas para potenciadores que têm níveis apreciáveis de acetilação da histona. Há uma variedade de fusões de repressivos alternativos que pode ser mais eficaz no direcionamento específicos potenciadores. Fusões de DNA metiltransferase para dCas9 podem ser usados para reduzir a expressão de gene quando direcionados para distal potenciadores de30, mas essa repressão muitas vezes não é transitória. Outra fusão repressiva usa o domínio de amigo de GATA1 (FOG1), que leva a histona H3 lisina 27 trimethylation e reprime a expressão gênica em níveis semelhantes para dCas9-CASCUDO através de uma variedade de células linhas e promotores31. Curiosamente, adicionar mais cópias de FOG1 para dCas9 reduziu o potencial repressivo em promotores, sugerindo que uma única cópia do domínio SID pode fornecer mais desativação potenciador do que as 4 cópias atualmente utilizados nas Enhancer-i. É possível que alguns loci pode beneficiar de dupla definião de combinações diferentes das fusões de dCas9 acima. Por exemplo, repressão de longo prazo estável pode ser conseguida simultânea transdução de dCas9-DNMT3a e dCas9-CASCUDO32. Maior parte destas fusões repressivas têm sido alvo de um único locus cada vez, e ele ainda não está claro que é mais eficaz na manipulação potenciadores de múltiplos simultaneamente.
Potenciador-i, enquanto um método adequado para estudar combinações de potenciadores de por um punhado de genes, é ainda um pouco limitada na taxa de transferência se o usuário deseja estudar putativos potenciadores para centenas de genes. Futuras aplicações desta técnica irão incorporar tecnologias baseadas em imagem para quantificar vários genes em amostras múltiplas simultaneamente. Importante, essas tecnologias são compatíveis com detecção direta de moléculas de RNA de lisado, eliminando a necessidade de isolamento de RNA demorada. Estas adaptações facilitam o interrogatório de conjuntos maiores de realçadores.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi financiado pelo NIH/tempo R00 HG006922 e HG008974 de R01 NIH/tempo de J.G. e o Instituto de câncer Huntsman. J.B.C. foi apoiada pelo programa de treinamento de NIH em genética T32GM007464.
Materiais
| Name | Company | Catalog Number | Comments |
| ZR 96-well Quick-RNA Kit | Zymo Research | R1053 | |
| Power SYBR Green RNA-to-CT 1-Step | Applied Biosystems | 4389986 | |
| AflII restriction enzyme | NEB | R0520S | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531L | |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | |
| FuGENE HD | Promega | E2312 | |
| DNA Clean & Concentrator Kit | Zymo Research | D4013 | |
| Buffer RLT Plus | Qiagen | 1053393 | |
| b-estradiol | Sigma-Aldrich | E2758 | |
| Human: Ishikawa cells | ECACC | 99040201 | |
| H3K27ac rabbit polyclonal | Active Motif | 39133 | |
| H3K9me3 rabbit polyclonal | Abcam | ab8898 | |
| FLAG mouse monoclonal | Sigma-Aldrich | F1804 | |
| ER alpha rabbit polyclonal | Santa Cruz | sc-544 | |
| pGL3-U6-PGK-Puro plasmid | Addgene | 51133 | Shen et al., 2014 |
| gRNA_cloningVector plasmid | Addgene | 41824 | Mali et al., 2013 |
| AflII U6 puromycin plasmid | Addgene | 106404 | Carleton et al., 2017 |
| SID4X-dCas9-KRAB plasmid | Addgene | 106399 | Carleton et al., 2017 |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-10ML | |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977-023 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985070 | |
| KAPA Stranded mRNA-Seq Kit, with KAPA mRNA Capture Beads | Kapa Biosytems | KK8420 | |
| Pierce Protease and Phosphatase Inhibitor Mini Tablets | ThermoFisher Scientific | A32959 | |
| Formaldehyde solution | Sigma-Aldrich | 252549-25ML | |
| Geneticin Selective Antibiotic (G418 Sulfate) (50 mg/mL) | ThermoFisher Scientific | 10131035 | |
| LB Broth | ThermoFisher Scientific | 10855001 | |
| Quick-DNA Miniprep Kit | Zymo Research | D3020 | |
| Quick-Load Purple 2-Log DNA Ladder | NEB | N0050S |
Referências
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Roadmap Epigenomics, C., et al. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Perry, M. W., Boettiger, A. N., Bothma, J. P., Levine, M. Shadow enhancers foster robustness of Drosophila gastrulation. Curr Biol. 20 (17), 1562-1567 (2010).
- Lam, D. D., et al. Partially redundant enhancers cooperatively maintain Mammalian pomc expression above a critical functional threshold. PLoS Genet. 11 (2), e1004935 (2015).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Savic, D., et al. Promoter-distal RNA polymerase II binding discriminates active from inactive CCAAT/ enhancer-binding protein beta binding sites. Genome Res. 25 (12), 1791-1800 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Cheng, A. W., et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res. 23 (10), 1163-1171 (2013).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol. , (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nat Methods. 12 (5), 401-403 (2015).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nat Methods. 12 (12), 1143-1149 (2015).
- Groner, A. C., et al. KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLoS Genet. 6 (3), e1000869 (2010).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Fulco, C. P., et al. Systematic mapping of functional enhancer-promoter connections with CRISPR interference. Science. 354 (6313), 769-773 (2016).
- Joo, J. Y., Schaukowitch, K., Farbiak, L., Kilaru, G., Kim, T. K. Stimulus-specific combinatorial functionality of neuronal c-fos enhancers. Nat Neurosci. 19 (1), 75-83 (2016).
- Xie, S., Duan, J., Li, B., Zhou, P., Hon, G. C. Multiplexed Engineering and Analysis of Combinatorial Enhancer Activity in Single Cells. Mol Cell. 66 (2), 285-299 (2017).
- Carleton, J. B., Berrett, K. C., Gertz, J. Multiplex Enhancer Interference Reveals Collaborative Control of Gene Regulation by Estrogen Receptor alpha-Bound Enhancers. Cell Syst. 5 (4), 333-344 (2017).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Ayer, D. E., Laherty, C. D., Lawrence, Q. A., Armstrong, A. P., Eisenman, R. N. Mad proteins contain a dominant transcription repression domain. Mol Cell Biol. 16 (10), 5772-5781 (1996).
- Alland, L., et al. Role for N-CoR and histone deacetylase in Sin3-mediated transcriptional repression. Nature. 387 (6628), 49-55 (1997).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Rennoll, S. A., Scott, S. A., Yochum, G. S. Targeted repression of AXIN2 and MYC gene expression using designer TALEs. Biochem Biophys Res Commun. 446 (4), 1120-1125 (2014).
- Stampfel, G., et al. Transcriptional regulators form diverse groups with context-dependent regulatory functions. Nature. 528 (7580), 147-151 (2015).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nat Methods. 10 (10), 973-976 (2013).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nat Methods. 11 (2), 122-123 (2014).
- Parsi, K. M., Hennessy, E., Kearns, N., Maehr, R. Using an Inducible CRISPR-dCas9-KRAB Effector System to Dissect Transcriptional Regulation in Human Embryonic Stem Cells. Methods Mol Biol. , 221-233 (2017).
- Romanienko, P. J., et al. A Vector with a Single Promoter for In Vitro Transcription and Mammalian Cell Expression of CRISPR gRNAs. PLoS One. 11 (2), e0148362 (2016).
- Smith, I., et al. Evaluation of RNAi and CRISPR technologies by large-scale gene expression profiling in the Connectivity Map. PLoS Biol. 15 (11), e2003213 (2017).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Res. 45 (17), 9901-9916 (2017).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados