É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Design e síntese de um Rack de acordeão de DNA reconfigurável
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Descrevemos o protocolo detalhado para projeto, simulação, molhado-laboratório experimentos e análise para um rack de acordeão de DNA reconfigurável de malhas de 6 por 6.
Resumo
Sistemas mecânicos baseados em nanostructure de DNA ou DNA nanomáquinas, que produzem movimento complexo nanoescala em 2D e 3D no nanômetro a resolução ångström, mostram grande potencial em vários campos da nanotecnologia como os reactores moleculares, administração de medicamentos, e sistemas de nanoplasmonic. O rack acordeão reconfigurável de DNA, que coletivamente pode manipular a uma rede de nanoescala 2D ou 3D de elementos, em múltiplos estágios em resposta às entradas do DNA, é descrito. A plataforma tem potencial para aumentar o número de elementos que DNA nanomáquinas podem controlar a partir de alguns elementos para uma escala de rede com múltiplos estágios de reconfiguração.
Este protocolo, descreveremos todo o processo experimental do rack acordeão DNA reconfigurável de malhas de 6 por 6. O protocolo inclui um procedimento de regra e simulação de projeto de estruturas e uma experiência de molhado-laboratório de síntese e reconfiguração. Além disso, a análise de estrutura de temperatura (microscopia eletrônica de transmissão) e FRET (transferência de energia de ressonância de fluorescência) está incluído no protocolo. Os métodos de projeto e simulação romance cobertos neste protocolo ajudará pesquisadores a utilizar o cesto de acordeão de DNA para novas aplicações.
Introdução
Sistemas mecânicos baseados em nanoestruturas de DNA ou ADN nanomáquinas1,2,3,4,5 são exclusivos porque eles produzem movimento complexo nanoescala em 2D e 3D no nanômetros para Ångström-resolução, de acordo com vários biomolecular estímulos2,3,6. Anexar materiais funcionais sobre estas estruturas e controlando as suas posições, estas estruturas podem ser aplicadas a diversas áreas. Por exemplo, as nanomáquinas de DNA têm sido propostas para um reator molecular7, droga entrega8e nanoplasmonic sistemas9,10.
Anteriormente, nós introduzimos o rack acordeão reconfigurável de DNA, que pode manipular a uma rede de nanoescala 2D ou 3D de elementos11 (figura 1A). Ao contrário de outras DNA nanomáquinas que apenas alguns elementos de controle, a plataforma pode coletivamente manipular periodicamente matriz elementos 2D ou 3D em várias fases. Nós antecipamos que uma rede programável reação química e biológica ou um sistema de computação molecular pode ser construído de nosso sistema, aumentando o número de elementos controláveis. O rack de acordeão de DNA é uma estrutura, em que a rede de múltiplos feixes de ADN está ligada a articulações compostas de DNA de cadeia simples (figura 1B). O acordeão rack gerado por vigas do DNA é reconfigurado pelos bloqueios de DNA, que hibridizam à parte pegajosa de vigas e alterar o ângulo entre os feixes de acordo com o comprimento da ponte parte dos bloqueios (estado bloqueado). Além disso, várias etapa reconfiguração é demonstrada adicionando novas fechaduras após a formação do estado livre removendo bloqueios de DNA através da vertente baseada no ponto de apoio deslocamento12,13.
Neste protocolo, descrevemos o todo processo de design e síntese do rack acordeão DNA reconfigurável. O protocolo inclui design, simulação, molhado-laboratório experimentos e análise para a síntese de rack acordeão DNA de 6 por 6 malhas e uma reconfiguração desses. A estrutura coberta no protocolo é o modelo básico da pesquisa anterior11 e 65 nm em 65 nm de tamanho, constituído por 14 vigas. Em termos de projeto e simulação, o projeto estrutural do acordeão rack é diferente do convencional ADN origami14,15 (ou seja, hermeticamente embalados). Portanto, a regra de projeto e simulação molecular foram modificados dos métodos tradicionais. Para demonstrar, mostramos a técnica de desenho usando a abordagem modificada de circunflexo14 e a simulação do acordeão rack usando oxDNA16,17 com scripts adicionais. Finalmente, os dois protocolos de temperatura e FRET para análise de estruturas de cremalheira acordeão configurados são descritos.
Protocolo
1. projeto do 6 por 6 DNA acordeão Rack com circunflexo14
- Baixe e instale o circunflexo 2.0 software14 para projetar um rack de acordeão de DNA (circunflexo 2.5 também está disponível em https://github.com/cadnano/cadnano2.5). Abra o circunflexo14 e clique na Ferramenta de Praça para adicionar uma nova parte com uma malha quadrada.
- O número de cada feixe do acordeão rack e desenhar no painel esquerdo da estrutura do circunflexo14 (Figura 2).
- Clique na ferramenta lápis e desenhar cada feixe no painel Editar direito sobre o circunflexo14. Pausa feixes cada 32 bp, que é para articulações entre vigas adjacentes. Coloque o grampos cruzamentos na mesma posição que as articulações. Use a Ferramenta lápis e a Ferramenta inserir no circunflexo14 deixar as articulações ter adicionais single-stranded cruzamentos.
- Clique na Ferramenta lápis e conectar as articulações. Cada feixe tem sete articulações.
- Gere os cruzamentos de andaime para mesclar os andaimes para o único loop usando o andaime relatado anteriormente roteamento algoritmo11. Não deixe que o domínio de ligação mínima entre vertentes andaime e grampo ser menor que 8 bp (Figura 3).
- Coloque os andaimes que não são usados na montagem nos vértices localizados em lados opostos do acordeão rack, conforme mostrado na Figura 3.
- Clique a quebrar a ferramenta. Quebre os fios de fibras descontínuas aonde circular ou mais de 60 bp.
- Desenha a bloqueio de DNA.
- Clique a quebrar a ferramenta. Pausa 8 bp de uma região de DNA descontínuas para fazer uma parte pegajosa e excluir 8 bp de uma região de DNA descontínuas. Existem 18 partes pegajosas (Figura 1) no rack acordeão 6 por 6.
- Coloque sequências que são inversa complementar às partes pegajosas em ambas as extremidades de fechamento cordões e conectá-los por uma região de ponte, que consiste de cadeias de poli T do comprimento desejado (figura 1B).
- Para a reconfiguração, adicionar 8 bp de sequências de ponto de apoio no final do DNA bloqueia para deslocamento de vertente. A sequência de suporte usada é na tabela 2.
- Prepare-se fios de poli A que são reverse complementar à região da ponte.
- Vertentes do projeto que são reverse complementares para as fechaduras de DNA para o experimento de reconfiguração.
- Clique na Ferramenta de sequência e clique em DNA de andaime. Escolha o cadafalso como padrão M13mp18. Clique na Ferramenta de exportação e salvar a sequência no formato csv (tabela 1).
2. simular a estrutura com o oxDNA
- Baixe e instale o oxDNA16,17. O código-fonte mais recente está disponível em https://sourceforge.net/projects/oxdna/files/.
- Fazer arquivos de configuração inicial do arquivo de14 circunflexo usando python script 'cadnano_interface.py', que é fornecido no pacote oxDNA16,17 . O uso é a seguinte: 'python cadnano_interface.py cadnano_file.json sq'. O arquivo de topologia e o arquivo de configuração agora são gerados.
Nota: O arquivo de topologia inclui quantos fios e nucleotídeos são na estrutura e informações sobre ligações de backbone-espinha dorsal entre nucleotídeos. O arquivo de configuração inclui informações gerais tais como temporais, energia e tamanho da caixa. Informações de orientação como vector de posição, espinha dorsal da base vetorial, vetor normal, velocidade e velocidade angular de nucleotídeos são também incluída (Figura 4). - Altere as informações no arquivo de configuração e topologia de circunflexo14 para fazê-los refletir as informações estruturais reais do acordeão rack. Todas as vigas são arranjadas em paralelo quando os arquivos de topologia e configuração do circunflexo14 são visualizados. No entanto, o acordeão rack é uma estrutura de treliça assim a distância entre nucleotídeos ligados são longe para simulação (Figura 5).
- Girar e mover cada feixe para a estrutura da estrutura desejada. As nove colunas à esquerda no arquivo de configuração são o vector de posição, espinha dorsal da base vetorial e vetor normal (Figura 4). Para girar um feixe, todos de posição, gire vetores base de espinha dorsal e normais, usando a transformação de rotação. Em seguida, mova um feixe alterando o vector de posição para localizá-lo, como mostrado na Figura 5.
- Relaxe a estrutura usando o script fornecido no pacote oxDNA (ver exemplo em $oxDNA/exemplos/RELAX_INITIAL_CONFIGURATION para mais informações).
- Execute a simulação de dinâmica molecular para 10 milhões passos usando o arquivo de configuração relaxados. O uso é a seguinte: '. / oxDNA < entrada >' salvar dados todos os passos de 5000 ou 10000.
- Visualização
Nota: As estruturas foram visualizadas usando cogli.- Baixe e instale a versão mais recente do cogli (https://sourceforge.net/projects/cogli1/).
- Execute o cogli com os arquivos de configuração e topologia de simulação oxDNA. O uso é a seguinte: '. / cogli1 -t < arquivo de topologia >< arquivo de configuração >'.
- Esconda a caixa pressionando b.
3. síntese da estrutura
Nota: O método de síntese é adaptado do anterior protocolo15,18.
- Compre os grampos de DNA projetados de um provedor do oligonucleotide.
- Ajuste a concentração destes grampos de DNA a 100 μM usando água livre de nuclease.
- Cada cadeia de DNA que constitui uma estrutura de 'free estado' em um tubo do pool e ajustar a concentração de 2 μM para cada vertente.
- Bloqueio de DNA piscina vertentes pelo comprimento e número de sites de bloqueio nos tubos e ajustar a concentração de 2 μM para cada vertente. sites de bloqueio 4, 9 e 18 são usados. Adicione fios de A poli, que são complementares à região ponte na mesma concentração.
- Vertentes de piscina que são inverter complementares ao DNA trancar fios pelo comprimento em tubos e ajustam a concentração de 2 μM para cada vertente.
- Preparar a solução de MgCl2 de 300 nM, misturando 70 μL de água livre de nuclease e 30 µ l da solução de2 1 μM MgCl. Prepare uma solução de EDTA Tris de 5x misturando 95 μL de água livre de nuclease e 5 μL de solução de EDTA Tris de 100x.
- Adicione 2 μL de DNA, 1.1 μL de MgCl2 solução de grampo, 2 μL de solução de EDTA Tris, 7.6 μL de água livre de nuclease e 7.3 μL de DNA de andaime de que a concentração é 110 nM tornar 20 μL de caldo misto. Definir a concentração final de cadafalso DNA de 40 nM, grampear o DNA de 200 nM, MgCl2 a 16 mM e solução de Tris-EDTA a 0,5 x.
- Aqueça rapidamente a solução-mãe misturada em um termociclador a 80 ° C e esfriar a 60 ° C, a uma taxa de 4 min por ° C e frio de 60 ° C a 4 ° C, a uma taxa de 40 min por ° C.
4. purificação da estrutura
Nota: As amostras de todas as estruturas foram purificadas antes da análise. Nesta seção, descrevemos o protocolo de purificação de PEG, que é adaptado a partir de literatura anterior19. A amostra também pode ser purificada por eletroforese em gel, conforme descrito em anterior literatura15,18.
- Prepare-se 5m de NaCl e 100 x Tris-EDTA.
- Prepare o buffer de precipitação misturando 150 μL de PEG 8000, 500 μL de 100 x Tris EDTA e 101 μL de 5 M NaCl e 249 μL de água livre de nuclease.
- Prepare o buffer de destino misturando 5.5 μL de solução de 300 nM MgCl2 da secção 3.3, 10 μL de solução de EDTA Tris x 5 da secção 3.3 e 84.5 μL de água livre de nuclease.
- Mix 20 μL da estrutura sintetizada da seção 3 e 20 μL de tampão-precipitação da seção 4.2. Em seguida, girar o estoque misturado a 16000 x g a 4 ° C. Remover o sobrenadante e dissolver a pelota no buffer de destino da seção 4.3.
5. reconfiguração do acordeão Rack de um estado' livre' para um 'estado bloqueado'
- Sintetiza a estrutura sem bloqueios de DNA para a experiência de configuração.
- Prepare o bloqueio de DNA da seção 3.
- Adicione 2 μL de cadeias de ADN bloqueio do comprimento desejado em 20 μL da estrutura sintetizada. Concentração do bloqueio de DNA é cinco vezes maior do que a estrutura.
- Incubar a amostra de 0, 10, 25, 50 ou 100 minutos para ver como a rápida reconfiguração ocorre.
- Para a incubação de 100 minutos, incubar a amostra a 50 ° C durante 30 minutos e deixe esfriar lentamente até 25 ° C, a uma taxa de 0,33 ° C/minuto.
- Para a incubação de 50 minutos, incubar a amostra a 50 ° C por 15 minutos e deixe esfriar lentamente até 25 ° C, a uma taxa de 0,66 ° C/minuto.
- Para a incubação de 25 minutos, incubar a amostra a 50 ° C para 7,5 minutos e deixe esfriar lentamente até 25 ° C, a uma taxa de 1,32 ° C/minuto.
- Para a incubação de 10 minutos, incubar a amostra a 50 ° C por 3 minutos e deixe esfriar lentamente até 25 ° C, a uma taxa de 3,3 ° C por minuto.
- Para a incubação de 0 minutos, armazene a amostra à direita de 4 ° C após fechamentos de DNA é adicionadas.
- Logo após a etapa de fixação, esfria rapidamente a amostra a 4 ° C para evitar a desnaturação indesejada.
6. reconfiguração do acordeão Rack de um estado 'bloqueado' para um 'estado livre'
- Sintetiza a estrutura com bloqueios de DNA do comprimento desejado para a experiência de configuração.
- Prepare o reversos costas complementares da seção 3.
- Adicione 2 μL de cordões que são inverter complementares para as vertentes de bloqueio do comprimento desejado em 20 μL da estrutura sintetizada. Concentração do bloqueio de DNA é cinco vezes maior do que a estrutura.
- Incubar a amostra para 0, 12, 60, 120, 240 minutos para ver como a rápida reconfiguração ocorre.
- Para o 12, 60, 120, incubação de 240 minutos, aqueça rapidamente a amostra a 40 ° C e esfriar lentamente até 20 ° C para o tempo correspondente a cada uma. Logo após a etapa de desmontar, esfria rapidamente a amostra a 4 ° C para evitar a desnaturação indesejada.
- Para a incubação de 0 minutos, armazene a amostra à direita de 4 ° C depois reversos costas complementares são adicionadas.
7. TEM imagem
Nota: O protocolo de imagem TEM foi adaptado do anterior literatura18,20.
- Prepare a solução de NaOH 1,25 M misturando 87,5 μL de água livre de nuclease e 12,5 μL de solução de NaOH 10 M.
- Adicione 1 μL de solução de NaOH 1,25 M para 50 μL de solução de formiato de uranilo a 2%.
- Vórtice a solução durante 3 minutos e centrifugar a velocidade máxima durante 3 minutos. Depositar 3 μL da amostra purificada na grelha TEM brilho-alta por 3 minutos e lavar rapidamente com papel de filtro.
- Deposite 7 μL de solução de formiato de uranilo preparado por 30 segundos e lavar rapidamente com papel de filtro.
- Medir o comprimento e o ângulo da estrutura acordeão fotografada pela TEM.
8. FRET análise
- Use Atto 550 e Atto 647N corante, para que a distância de Förster é 6.5 nm. Substitua grampo 58 e 117 de grampo na tabela 1 fluorescente etiquetados cordões. Então, sintetiza a estrutura com vertentes fluorescente rotulados pelo método descrito na seção 3.
- Medir a concentração da amostra purificada.
- Normalize a amostra de 10 μL 50 nM e carga para as 384 microplacas bem.
- Excitar a amostra com doador e aceptor de corantes em 550 nm e medir o espectro de fluorescência de 570 nm para 800 nm com um dados.
- Medir o espectro de fluorescência da amostra somente doador da mesma forma.
- Excitar as tinturas da amostra a 650 nm e o espectro de fluorescência e medida de 670 nm para 800 nm. Isto é para medir a concentração do aceitador.
- Obter os desvios-padrão, repetindo o mesmo experimento com três amostras, que são sintetizados e purificados separadamente.
- Calcule a eficiência do traste com a relação de um método, conforme descrito pela equação abaixo de21.
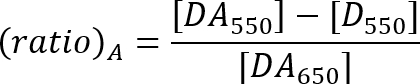
DA550: intensidade de fluorescência de pico aceitador da amostra com doador e aceptor no 550 excitação nm.
D550: intensidade de fluorescência na faixa de emissão aceitador da amostra do doador somente em 550 excitação nm.
DA650: intensidade de fluorescência de pico aceitador da amostra com doador e aceptor na excitação de nm 650.
Resultados
O rack de acordeão de DNA 6 projetado por 6 é simulado a partir do oxDNA16,17 , e os resultados são mostrados na Figura 6. O resultado da simulação, foi confirmado que a estrutura se destina é formada sem distorção da estrutura.
As imagens TEM na Figura 7 são imagens de estruturas configuradas com um co...
Discussão
Este protocolo apresenta todo o processo de design, simulação, síntese e análise do básico 2D DNA acordeão rack. O projeto modificado e regras de simulação têm sido descritas porque a regra do projeto difere do padrão origami de DNA, em que a cremalheira de acordeão de DNA tem nucleotídeos adicionais em crossovers para flexibilidade14,15. A partir disso, esperamos que o protocolo pode acelerar várias pesquisas usando acordeão cremalheiras de DNA. Al...
Divulgações
Os autores não têm nada a divulgar
Agradecimentos
Esta pesquisa foi parcialmente suportada pelo Global Research Center programa de desenvolvimento através do nacional Research Foundation de Korea(NRF) financiado pelo Ministério da ciência e TIC (MSIT) (2015K1A4A3047345) e Nano· Programa de desenvolvimento de tecnologia de material através da nacional Research Foundation de Coreia (NRF) financiado pelo Ministério da ciência e TIC (MSIT) (2012M3A7A9671610). O Instituto de pesquisa de engenharia na Universidade Nacional de Seul desde instalações de pesquisa para este trabalho. Os autores reconhecem gratitude para Yoon Tae-Young (Ciências Biológicas, Universidade Nacional de Seul) em relação a espectroscopia de fluorescência para a análise de traste.
Materiais
| Name | Company | Catalog Number | Comments |
| M13mp18 Single-stranded DNA | NEB | N4040s | |
| 1M MgCl2 Solution | Biosesang | M2001 | |
| Tris-EDTA buffer | Biosesang | T2142 | |
| Nuclease-Free Water | Qiagen | 129114 | |
| 5M Sodium Chloride solution | Biosesang | s2007 | |
| PEG 8000 | Sigma Aldrich | 1546605 | |
| 10N NaOH | Biosesang | S2038 | |
| Uranyl formate | Thomas Science | C993L42 | |
| Thermal cycler C1000 | Biorad | ||
| Nanodropic 2000 | Thermo Fisher Scientific | ||
| TEM (LIBRA 120) | Carl Zeiss | ||
| Fluorometer Enspire 2300 | Perkin-Elmer | ||
| Centrifuge | Labogene | LZ-1580 |
Referências
- Andersen, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459 (7243), 73-76 (2009).
- Cha, T. -. G., et al. Design principles of DNA enzyme based walkers: Translocation kinetics and photo-regulation. Journal of the American Chemical Society. 137 (29), 9429-9437 (2015).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347 (6229), 1446-1452 (2015).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature nanotechnology. 6 (12), 763-772 (2011).
- Li, J., et al. Exploring the speed limit of toehold exchange with a cartwheeling DNA acrobat. Nature Nanotechnology. 1, (2018).
- Krishnan, Y., Simmel, F. C. Nucleic acid based molecular devices. Angewandte Chemie International Edition. 50 (14), 3124-3156 (2011).
- Liu, M., et al. A DNA tweezer-actuated enzyme nanoreactor. Nature communications. 4, 2127 (2013).
- Douglas, S. M., Bachelet, I., Church, G. M. A logic-gated nanorobot for targeted transport of molecular payloads. Science. 335 (6070), 831-834 (2012).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13 (9), 862-866 (2014).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature communications. 6, 8102 (2015).
- Choi, Y., Choi, H., Lee, A. C., Lee, H., Kwon, S. A Reconfigurable DNA Accordion Rack. Angewandte Chemie International Edition. 57 (11), 2811-2815 (2018).
- Chen, H., et al. Understanding the Mechanical Properties of DNA Origami Tiles and Controlling the Kinetics of their Folding and Unfolding Reconfiguration. Journal of the American Chemical Society. 136 (19), 6995-7005 (2014).
- Han, D., Pal, S., Liu, Y., Yan, H. Folding and cutting DNA into reconfigurable topological nanostructures. Nature Nanotechnology. 5 (10), 712-717 (2010).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37 (15), 5001-5006 (2009).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature methods. 8 (3), 221-229 (2011).
- Ouldridge, T. E., Louis, A. A., Doye, J. P. K. DNA Nanotweezers Studied with a Coarse-Grained Model of DNA. Physical Review Letters. 104 (17), 178101 (2010).
- Snodin, B. E. K., et al. Direct Simulation of the Self-Assembly of a Small DNA Origami. ACS Nano. 10 (2), 1724-1737 (2016).
- Amir, Y., Abu-Horowitz, A., Bachelet, I. Folding and Characterization of a Bio-responsive Robot from DNA Origami. Journal of Visualized Experiments. (106), e51272 (2015).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53 (47), 12735-12740 (2014).
- Wei, B., Vhudzijena, M. K., Robaszewski, J., Yin, P. Self-assembly of Complex Two-dimensional Shapes from Single-stranded DNA Tiles. Journal of Visualized Experiments. (99), e52486 (2015).
- Clegg, R. M. Fluorescence resonance energy transfer and nucleic acids. Methods in enzymology. 211, 353-388 (1992).
- Kopperger, E., et al. A self-assembled nanoscale robotic arm controlled by electric fields. Science. 359 (6373), 296-301 (2018).
- Lauback, S., et al. Real-time magnetic actuation of DNA nanodevices via modular integration with stiff micro-levers. Nature Communications. 9 (1), 1446 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados