Method Article
Utilização de proteínas recombinantes de fusão em uma plataforma de ensaio de Protease fluorescente e sua renaturalização em-gel
Neste Artigo
Resumo
Aqui, apresentamos o procedimento detalhado de uma plataforma de ensaio de protease recentemente desenvolvido utilizando o N-terminal hexahistidine/maltose-proteína e fluorescentes substratos recombinantes da proteína-fundido, anexados à superfície do níquel-nitrilotriacético grânulos de agarose magnético ácido. Também é apresentada uma análise subsequente em-gel das amostras de ensaio separados por eletroforese em gel polyacrylamide do sulfato dodecyl de sódio.
Resumo
Proteases são enzimas intensivamente estudadas devido à suas funções essenciais em diversos caminhos biológicos dos organismos vivos e na patogênese; Portanto, eles são alvos de drogas importantes. Nós desenvolvemos uma plataforma de ensaio magnético-agarose-grânulo-baseado para a investigação da atividade proteolítica, que é baseada na utilização de substratos de proteínas recombinantes de fusão. Para demonstrar o uso deste sistema de ensaio, um protocolo é apresentado no exemplo do vírus da imunodeficiência humana tipo 1 (HIV-1) de protease. A plataforma de ensaio apresentado pode ser utilizada com eficiência na caracterização bioquímica de proteases, incluindo medições de atividade enzimática em mutagênese, cinética, inibição ou estudos de especificidade, e pode ser apropriado para alta produtividade rastreio de substrato ou pode ser adaptado a outras enzimas proteolíticas.
Neste sistema de ensaio, os substratos aplicados contém maltose-ligação proteína (MBP) marcas e hexahistidine N-terminal (a6), sítios de clivagem para tabaco etch vírus (TEV) e proteases HIV-1 e uma proteína fluorescente do C-terminal. Os substratos podem ser eficientemente produzidos em células de Escherichia coli e facilmente purificada usando - quelato - grânulos revestidos de níquel (Ni). Durante o ensaio, a clivagem proteolítica de substratos do grânulo-anexado leva à liberação de fragmentos de clivagem fluorescente, que pode ser medido por fluorimetria. Além disso, reações de clivagem podem ser analisadas por eletroforese em gel polyacrylamide do sulfato dodecyl de sódio (SDS-PAGE). Um protocolo para a renaturalização em-gel de componentes de ensaio também é descrito, como renaturalização parcial de proteínas fluorescentes permite sua detecção com base no peso molecular e fluorescência.
Introdução
Enzimas proteolíticas pertencem aos grupos mais intensamente investigado enzima devido a sua importância em vias metabólicas e em aplicações industriais, bem como. Seu papel-chave nas doenças virais, a regulação da coagulação do sangue, câncer e cardiovascular e doenças neurodegenerativas faz proteases alvos proeminentes no campo da descoberta da droga. Portanto, a caracterização detalhada da especificidade de substrato e inibidor de perfilação da protease (PR), de interesse é cruciais e são realizados de preferência por ensaios bioquímicos rápida, eficiente e robusto1,2, 3.
Hoje em dia, a grande maioria dos ensaios in vitro protease aplicado no campo da descoberta de drogas para perfis compostos é homogêneo, fluorescente baseado em peptídeo e elevado-throughput screening (HTS)-plataformas compatíveis4. Além disso, peptídeos etiquetados não são somente apropriados para triagem de biblioteca, mas eles também oferecem ótimas ferramentas para a determinação da enzima parâmetros cinéticos sobre os substratos selecionados. Em outros casos, onde a rotulagem do substrato não é possível, ensaios baseados em separação podem fornecer uma solução possível para avaliar as propriedades cinéticas de reações proteolíticas3.
Geralmente, os ensaios in vitro protease baseiam-se na utilização de dois tipos de substrato: curto peptídeos ou proteínas inteira. Nesses casos, onde a clivagem de sequências peptídicas curtas refletem as propriedades de clivagem suficientemente, são aplicáveis as seguintes abordagens padrão: (i) análise de substratos de proteína padrão tais como insulina oxidado da cadeia B, (ii) teste comercialmente disponíveis substratos de outras proteases, (iii) bibliotecas de peptídeo sintético e fluorescente etiquetadas criadas por química combinatória, ou (iv) de rastreio usando métodos genéticos, por exemplo, biológico exibir tecnologias5, 6. A classificação convencional, além de outras plataformas de romance também estão disponíveis para geração de substrato (por exemplo, a formação do peptídeo derivado de proteome bibliotecas7 ou subtipos especiais de métodos genéticos, como a fusão recombinante substratos à base de proteínas8,9,10,11,.12).
Todos os tipos de substrato acima mencionados e ensaios têm suas próprias vantagens e limitações, e o desenvolvimento de formatos de ensaio combinando e/ou melhorar as vantagens das plataformas conhecidas ainda está em demanda. Aqui descrevemos um protocolo para um ensaio de separação-baseado protease fluorescente, que utiliza substratos recombinantes. Estas proteínas de fusão consistem em seus rótulos MBP fundidos a um local de clivagem de controle do PR do PEV, que é seguido pela sequência substrato de interesse que está directamente ligado a uma proteína fluorescente do C-terminal (FP) (figura 1A) e6 . A clonagem de uma sequência de DNA que codifica para um local de clivagem de interesse dentro da ' clonagem' pode ser executada por uma reação de ligadura único para o plasmídeo de expressão, que tem sido linearizada anteriormente por endonucleases de restrição.
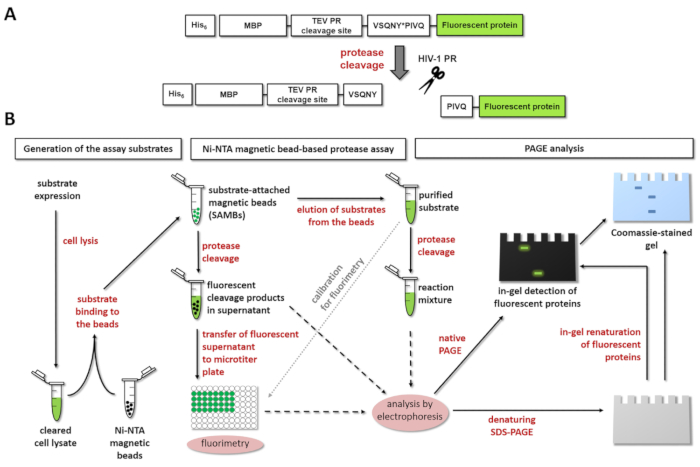
Figura 1: Princípio do ensaio fluorescente protease. Protease (A), a representação esquemática de um substrato fluorescente e sua clivagem pelo vírus da imunodeficiência humana tipo 1 (HIV-1) é mostrado. A seta indica a posição de clivagem dentro da sequência de sítio de clivagem de matriz/capsídeo de protease do HIV-1 (VSQNY * PIVQ). Substratos (B) o fluorescente podem ser usados para analisar reacções enzimáticas por ensaio baseado em grânulo magnético Ni-NTA e por eletroforese em gel de poliacrilamida, bem, como é mostrado no diagrama de fluxo de trabalho. Clique aqui para ver uma versão maior desta figura.
Embora proteolíticas ensaios usando a proteína recombinante semelhante substratos contendo uma tag de afinidade, um local de clivagem proteolítica e um proteína fluorescente-ter já sido descrito8,9,10, o sistema apresentado aqui tem a intenção de integrar e melhorar as vantagens destes métodos. Uma diferença importante é que os substratos de proteína de fusão nesta plataforma de ensaio estão equipados com MBP para aumentar a solubilidade de proteínas13 e conter um local de clivagem de controle para TEV PRs. Além disso, os substratos contêm proteínas fluorescentes de nova geração, que são altamente estáveis e têm uma forma monomérica para prevenir a agregação de substrato. Além do aplicativo publicado anteriormente de mTurquoise2 e mApple-fundido formas14, aqui nós também mostramos resultados dada pelo uso de um substrato recombinante contendo um monomérico reforçada etiqueta fluorescente proteína fluorescente amarela (mEYFP). Pelo presente, podemos demonstrar a compatibilidade do sistema com outras proteínas fluorescentes e representam alguns tipos gerais de resultados que podem ser adquiridos por meio de ensaio o protease.
As proteínas recombinantes de fusão são expressos em células de Escherichia coli BL21(DE3) e são utilizadas como substratos para o ensaio em um ácido nitrilotriacético-níquel (Ni-NTA)-magnético-agarose-grânulo-Unido forma revestida. Os produtos de clivagem do C-terminal são liberados da superfície do grânulo no sobrenadante após clivagem pela protease de interesse. Após a separação do sobrenadante (contendo a enzima e os produtos de clivagem) das esferas magnéticas, a fluorescência pode ser medida para determinar as propriedades de clivagem da enzima. Em contraste com os métodos anteriormente descritos, no sistema apresentado aqui, a quantidade de substrato e produtos de clivagem do C-terminal é excepcionalmente quantificada com base em um procedimento de calibração de substrato detalhadas. O sistema de ensaio pode ser suportado por uma análise de SDS-PAGE das amostras de ensaio; uma visualização de em-gel fluorescente subsequente pode ser aplicada imediatamente após a electroforese ou após a renaturalização em-gel dos nondenatured e desnaturados componentes fluorescentes, respectivamente14.
A flexibilidade e a estrutura da fita' clonagem' permitir uma inserção e custo-eficiente tempo de uma grande variedade de sequências para a construção e, assim, promove a geração de bibliotecas de substrato. Uma vez que todas as etapas do ensaio são automação e HTS-compatível, o sistema pode ser especialmente atraente para, por exemplo, medições de especificidade de protease e de mutagénese ou também pode ser efetivamente utilizado para rastreio de inibidor de protease industrial e/ou desenvolvimento de drogas antivirais, também.
Parâmetros de cinética enzimática (kgato, Km) podem ser determinados pelo teste desenvolvido com base em separação; Portanto, pode ser apropriado realizar medições cinética enzimática individuais, tais como os estudos de tempo-curso, dependente do substrato e inibição. Isto prova que os substratos de proteínas recombinantes de fusão fornecem boas alternativas para os substratos de oligopeptídeos sintética frequentemente utilizada, e devido a sua alta similaridade com os substratos poliproteína, eles representam a ocorrência natural interações enzima-substrato com mais precisão.
Protocolo
1. geração de plasmídeo de expressão o substrato-codificação
- Linearizar o plasmídeo de expressão pDest-His6- MBP-FP pela PacI e NheI endonucleases de restrição. Para a geração de sua pDest6- MBP-FP, ver Bozóki et al.14.
- Adicione 1.500-2.000 µ g do plasmídeo de expressão pDest-His6- MBP-FP, 2 μL de cada de PacI e NheI endonucleases de restrição, 10 µ l de tampão de x 10 (ver Tabela de materiais) e água livre de nuclease (NFW) de 100 µ l em um tubo de microcentrifugadora.
- Incube a mistura a 37 ° C por 1h.
- Adicionar 20 µ l de 6 x roxo de ADN carregando o corante para a mistura de reação e separar os produtos de clivagem por eletroforese, utilizando gel de agarose 1%. Aplica uma escada de DNA kB 1 como padrão.
- Lave o gel durante 15 min em 20 mL de TAE buffer (40 mM Tris, ácido acético de 20 mM, 1 mM EDTA, pH 8,5) contendo 20 µ l de solução SYBR green e impostos especiais de consumo da banda do plasmídeo linear fora o gel de agarose, usando uma ferramenta afiada.
Nota: Enquanto iluminando o gel por um transiluminador azul escuro-leitura (DRBT), o plasmídeo de - MBP-FP pDest-His linear6aparece como uma banda discreta e luminosa em cerca de 7-8 kB. - Purifica o plasmídeo de expressão linear da fatia gel usando um kit de extração do gel de acordo com as instruções do fabricante.
- Inserir a sequência de substrato o plasmídeo de expressão linear, sua pDest6- MBP-FP.
- Recoze a frente (FWD) e as inversa (REV) Escherichia coli códon otimizado primeiras demão do oligonucleotide codificação para a sequência de substrato de interesse.
Nota: Os primers recozidos vão ser ladeados pelas extremidades coesas correspondente a PacI e NheI sítios de clivagem de endonuclease de restrição (Figura 2).- Mix 150 ng do plasmídeo de expressão linear com 200 ng de FWD e 200 ng do oligonucleotide REV primers em 0,2 mL uma reação em cadeia do polymerase (PCR) tubo e ajuste o volume para µ l 17 adicionando NFW.
- Incube a mistura a 65 ° C por 2 min e, em seguida, a 4 ° C, durante pelo menos 2 min.
- Execute a inserção dos primers recozidos para o plasmídeo linear pela ligadura.
- Adicione 2 µ l de tampão de T4 ligase (10x) e 1 µ l de T4 ligase à mistura contendo o plasmídeo linear e as primeiras demão recozidas.
- Incube a mistura de ligadura a 16 ° C, durante 16 h.
- Recoze a frente (FWD) e as inversa (REV) Escherichia coli códon otimizado primeiras demão do oligonucleotide codificação para a sequência de substrato de interesse.

Figura 2 : Primeiras demão do oligonucleotide que codifica para uma clivagem proteolítica site sequência. Cartilhas para diante e reversos codificam o VSQNY * sequência de sítio de clivagem PIVQ de HIV-1 PR. Após as primeiras demão do oligonucleotide complementares de recozimento, o DNA de cadeia dupla curto contém extremidades pegajosas, correspondente do PacI e NheI endonucleases de restrição. Clique aqui para ver uma versão maior desta figura.
- Transformar 100 µ l de células competentes de BL21(DE3) por 5 µ l de mistura de ligadura e espalhar as células em placas de ágar caldo (LB) lisogenia contendo ampicilina.
Nota: As proteínas fluorescentes será no mesmo quadro de leitura aberto com as tags de fusão do N-terminal somente após uma bem sucedida da ligadura. Alguns dias depois da transformação, as colônias (contendo o plasmídeo de expressão que codifica para o local de clivagem inserido de interesse) mostrará fluorescência visível com ou mesmo sem usar um DRBT. - Prepare o estoque de glicerol das colônias retratadas.
- Lave uma colônia discreta em um tubo de centrífuga de 50 mL contendo 5 µ l de meio LB com ampicilina (em uma concentração final de 100 µ g/mL).
- -Incubar a 37 ° C para 8 h agitando continuamente a 220 rpm; em seguida, recolher as células por centrifugação a 1.000 x g durante 5 min à temperatura ambiente.
- Delicadamente, suspender as células em 1 mL de solução de glicerol de 80% (diluída com água destilada) e adicionar 500 µ l de solução de MgCl2 a 10 mm para a suspensão.
- Transfira a suspensão para um tubo de congelação e armazenar os estoques a-70 ° C.
- Verifique se a sequência do plasmídeo gerado pelo sequenciamento de DNA.
- Adicione 10 µ l do estoque de glicerol (preparado na etapa 1.7) para 5 mL de meio LB contendo 100 ampicilina µ g/mL em um tubo de centrífuga de 50 mL.
- Incube a suspensão a 37 ° C durante 16 h agitando continuamente a 220 rpm; em seguida, recolher as células por centrifugação a 2.000 x g durante 10 minutos a 4 ° C.
- Isolar o plasmídeo de expressão do centrifugado por miniprep um plasmídeo kit (veja a Tabela de materiais) de acordo com as instruções do fabricante e use o plasmídeo purificado para o sequenciamento de DNA.
Nota: para o sequenciamento, 5'-GATGAAGCCCTGAAAGACGCGCAG-3' (em frente) e 5'-GCAAGGCGATTAAGTTGGGTAACGC-3' primeiras demão do oligonucleotide (reverso) podem ser usadas.
2. a expressão de substratos fluorescentes
- Prepare a cultura starter.
- Adicione 10 µ l do estoque de glicerol (preparado na etapa 1.7) para 5 mL de meio LB contendo 100 ampicilina µ g/mL em um tubo de centrífuga de 50 mL.
- Incube a suspensão a 37 ° C por 15 h agitando continuamente a 220 rpm.
- Transferi a cultura bacteriana (5 mL) a 50 mL de meio LB fresco contendo 100 ampicilina µ g/mL em um Erlenmeyer de 500 mL estéril.
- Crescem as células a 37 ° C para uma absorvância de 0.6-0.8 no comprimento de onda de nm 600, agitando continuamente a 220 rpm.
Nota: Se um tratamento de tetraciclina é para ser aplicado na etapa 2.5, não se recomenda a crescer as células para uma absorvância de mais de 0,6 a 600 nm. - Adicione isopropílico β-D-1-thiogalactopyranoside (IPTG) para a concentração final de 1 mM para induzir a expressão da proteína.
- Se não é aplicado um tratamento de tetraciclina, incubar a cultura por 3 h a 37 ° C, agitando continuamente a 220 rpm e continuar o protocolo com passo 2.6. Se for aplicado um tratamento de tetraciclina, continue o protocolo com passos 2.5.1-2.5.3.
Nota: Alguns FPs produzido pelas células de e. coli pode ter um tempo de maturação (ver trabalhos anteriores)16,,17; nesses casos, a tradução de proteínas pode ser opcionalmente preso pelo tratamento a tetraciclina, a fim de aumentar o rendimento fluorescente da solução de substrato.- Incube a suspensão de eritrócitos durante 2 h a 37 ° C, agitando continuamente a 220 rpm; em seguida, adicione uma solução de tetraciclina (em uma concentração final de 200 µ g/mL).
- Incube a cultura de células de acordo com o tempo de maturação da proteína fluorescente de escolha a 37 ° C, agitando continuamente a 220 rpm.
- 2 x 25 ml da cultura para limpar tubos de centrífuga de 50 mL e colher as células por centrifugação a 4.000 x g durante 15 min a 4 ° C.
- Desprezar o sobrenadante e armazenar as pelotas de célula bacteriana a-70 ° C durante pelo menos 1 h.
Nota: As células contendo os substratos fluorescentes expressados apresentam fluorescência visível com ou mesmo sem usar um DRBT.
3. rompimento da pilha
- Coloque o centrifugado congelado no gelo e deixe descongelar por 15 min.
- Adicionar 2 mL de tampão de lise (50mm NaH2PO4, 300 mM de NaCl, imidazol 10 mM, 0,05% de Tween 20, pH 8) para a pelota e suspender as células.
- Adicione 10 µ l de solução de inibidor de protease recentemente preparada phenylmethanesulfonyl-fluoreto (PMSF) (8,7 mg/mL, dissolvido em etanol) à suspensão.
- Adicionar 2 mg de lisozima e 20 unidades de DNase à suspensão e suspendê-lo.
- Incube a suspensão no gelo por 15 min e vórtice que ocasionalmente.
- 2 x 1 mL da suspensão de transferência para tubos de 1,5 mL microcentrifuga e proceda à sonicação as suspensões por 3 min, nas rodadas de 10 s de sonication e 5 s de descansar.
- Centrifugar os tubos a 10.000 x g por 20 min à temperatura ambiente; em seguida, remover o sobrenadante fluorescente (apurado bacteriano lisado celular) cuidadosamente cada tubo e transferi-lo para novos tubos de microcentrifuga.
Nota: Lysates apuradas contendo o substrato fluorescente mostrar fluorescência visível com ou mesmo sem usar um DRBT e podem ser armazenadas a 4 ° C por até 2 semanas. Não congele. Lisados limpos podem ser utilizados diretamente para a preparação de amostra no ensaio de protease (ver seção 4.1) ou também pode ser usado para a purificação de substrato (ver passo 4.5.1).
4. ensaio de Ni-NTA magnética do grânulo-baseado protease
Nota: Devido à flexibilidade da plataforma de ensaio, ele pode ser otimizado para muitos tipos diferentes de estudos. Por este motivo e devido à diferença na taxa de atividade das enzimas de escolha, alguns dos parâmetros do ensaio (onde ele é denotado) não podem ser descrito explicitamente, mas precisam ser otimizado para os objectivos individuais e delineamento experimental. Como uma orientação, parâmetros de alguns tipos de estudos são indicados nas etapas a seguir particular.
- Preparação da amostra
- Geração de grânulos magnéticos substrato-inscritos
- Coloca um fechado 2ml baixa proteína-ligação (consulte a Tabela de materiais) microcentrifuga tubo contendo novos ou reciclados (ver secção 4.7) grânulos de agarose magnética Ni-NTA em um concentrador de partículas magnéticas (MPC).
Nota: A quantidade de suspensão aplicada do grânulo é definida com base no desenho experimental. Usamos 1 mL de solução magnética do grânulo (5%, v/v) em cada experimento. - Grânulos podem aderir à parede e/ou na tampa do tubo microcentrifuga; Portanto, transformar o MPC de ponta-cabeça em todas as direções para certificar-se de que todos os grânulos são coletados.
- Remover o sobrenadante e descartá-lo.
- Lave os grânulos por Lise.
- Adicionar 1,8 mL de tampão de Lise para os grânulos e retire o tubo fechado o MPC.
- Suspenda os grânulos no tubo de agitação e/ou transformando os tubos de cabeça para baixo até que a amostra é completamente homogênea.
- Coloque o tubo de volta para o MPC e transformá-lo de ponta-cabeça para coletar os grânulos.
- Abra o tubo e descartar o sobrenadante.
- Adicionar 1,0-1,8 ml de solução a limpo lisado (na etapa 3.7) para as contas e retire o tubo do MPC.
- Gire o tubo fechado de ponta-cabeça até que os grânulos são completamente homogêneos e giram lentamente o tubo por um rotador em temperatura ambiente por 30 min.
- Coloque-o no MPC e remover o lisado celular total apurado de grânulos e da tampa.
Nota: O lisado celular total apurado pode ser descartado ou salvos de continuar a utilizar (consulte a observação após etapa 3.7). - Adicionar 1% Tween 20 (pH 7) para os grânulos magnéticos conectado à carcaça (SAMBs).
Nota: SAMBs mostrar fluorescência visível com ou mesmo sem usar um DRBT.
- Coloca um fechado 2ml baixa proteína-ligação (consulte a Tabela de materiais) microcentrifuga tubo contendo novos ou reciclados (ver secção 4.7) grânulos de agarose magnética Ni-NTA em um concentrador de partículas magnéticas (MPC).
- Lavagem dos SAMBs
- Colocar o tubo com a suspensão SAMB o MPC e descartar o sobrenadante.
- Lavar os SAMBs 3 x com cada buffer: i) 1,8 mL de 1% Tween 20 (pH 7); II) 1,8 mL de tampão de lavagem (50mm NaH2PO4, 300 mM de NaCl, imidazol 5mm, 0,05% de Tween 20, pH 7); III) 1,8 mL de tampão de clivagem (50mm NaH2PO4, 300 mM de NaCl, 0,05% de Tween 20, pH 7).
Nota: Para o procedimento de lavagem, consulte a etapa 4.1.1.4. O buffer de clivagem pode ser alterado de acordo com as necessidades de experimentais, mas recomenda-se verificar o manual dos grânulos Ni-NTA magnéticos para determinar a compatibilidade.
- Preparação da solução-mãe SAMB
- Adicione um buffer de clivagem para os lavado SAMBs para criar uma solução stock de SAMB.
Nota: Após a adição do buffer, não agitar ou girar o tubo de ponta-cabeça. O volume da reserva clivagem varia de acordo com o desenho experimental individual e deve ser calculado com base no número de grânulos magnéticos (consulte a etapa 4.1.1.1) e sobre os volumes a ser usado na etapa 4.1.4.2. Para tubos de 2 mL, o volume aplicado é até 1.900 µ l (ver tabela 1). A densidade recomendada do grânulo magnético da solução SAMB é 2% - 10% (v/v).Tipo de estudo Volume de tampão de clivagem (µ l) Medições de S-dependente (Fig. 4) 1600 Medições de tempo-curso (Fig. 5A) 1600 Estudo de inibição (Fig. 5B) 1900 estudo de dependência do pH (Fig. 6) 1400
Tabela 1: Volume do decote buffer usado para preparar a solução de estoque SAMB nos diferentes tipos de medições. - Remova o tubo fechado o MPC. Use a solução de estoque SAMB imediatamente ou armazená-lo em 4 ° C por até 24 h.
- Adicione um buffer de clivagem para os lavado SAMBs para criar uma solução stock de SAMB.
- Geração das amostras de ensaio, utilizando a solução de estoque SAMB
Nota: Os detalhes desta parte do ensaio são fortemente dependentes do delineamento experimental individual (amostra tipos são mostrados na tabela 2).Tipo de amostra Notas Amostra de reação (R) -usado para avaliar Propriedades de clivagem
-contém a enzima e o substrato no buffer de clivagemAmostra em branco do substrato (B) -usado para avaliar a dissociação espontânea do substrato (ver passo 4.6.2)
-contém apenas o substrato no buffer de clivagemAmostra de controle de substrato (C) -para a concentração de substrato detemining (consulte a etapa 4.6.3)
-contém apenas o substrato em tampão de eluição
Tabela 2: Tipos de amostras do ensaio Ni-NTA magnética do grânulo-baseado protease.- Prepare-se para as amostras de ensaio 2 mL de tubos microcentrifuga de baixa proteína-ligação.
Nota: Outra baixa proteína de ligação de utensílios de plástico também pode ser usada. Use tubos ou plano-fundo redondo para garantir a livre circulação de SAMBs. Consulte o número recomendado de tubos na tabela 3.Tipo de estudo R B C Medições de S-dependente (Fig. 4) 5 5 2 Medições de tempo-curso (Fig. 5A) 6 6 2 Estudo de inibição (Fig. 5B) 7 7 1 estudo de dependência do pH (Fig. 6) 5 5 1
Tabela 3: Número de tubos de microcentrifuga 2ml necessários para cada tipo de amostra em estudos de demonstrada. - Suspender a solução estoque SAMB até homogeneidade e transferir a quantidade de substrato a ser analisado nas reações imediatamente em frascos de amostra. O volume recomendado é de 25-300 µ l, mas é a ser definido de acordo com o projeto experimental individual (tabela 4).
Nota: Verifique se todos os SAMBs foram medidos no fundo dos tubos. SAMBs pode aderir à parede do tubo, que pode distorcer os resultados do ensaio. Se diferentes volumes devem ser medidos sequencialmente, começar alíquotas com maior volume e tentar minimizar a alteração da pipeta e/ou pontas de pipetas.Tipo de estudo R B C Medições de S-dependente (Fig. 4) 25 – 50 – 100 – 150 – 250 25 – 50 – 100 – 150 – 250 25 Medições de tempo-curso (Fig. 5A) 25 25 25 Estudo de inibição (Fig. 5B) 120 120 120 estudo de dependência do pH (Fig. 6) 100 100 100
Tabela 4: Volume da solução SAMB medido em frascos de amostra de cada tipo de amostra em estudos de demonstrada. - Coloque os tubos de amostra contendo a suspensão SAMB aliquotada para o MPC e deslocar ligeiramente o MPC e para trás.
- Cuidadosamente remova os SAMBs o sobrenadante e descartá-lo.
- Retirar os tubos do MPC e adicionar o volume calculado de amortecedor da reação (clivagem ou eluição tampão [100 mM EDTA, 0,05% de Tween 20, pH 7]) com cuidado para os SAMBs.
Nota: Calcule o volume de reserva de acordo com o projeto experimental individual (tabela 5). Para tubos de 2 mL, volume final da mistura de reação (o volume do buffer a ser adicionado nesta etapa de reação + o volume da solução a ser adicionado na etapa 4.2.3) recomendado é 50-150 µ l. Certifique-se de que todos os SAMBs são lavados no buffer adicionado. Tampão de eluição é usado em vez de buffer de clivagem nos casos de amostras de controlo (C) substrato. Para um estudo de inibição, o inibidor da escolha é recomendado para ser adicionado a este passo.Tipo de estudo Volume de tampão de reação (µ l) Medições de S-dependente (Fig. 4) Buffer de clivagem µ l 68 Medições de tempo-curso (Fig. 5A) Buffer de clivagem µ l 68 Estudo de inibição (Fig. 5B) 67,3 buffer de clivagem µ l + inibidor de 0,7 µ l estoque solução * estudo de dependência do pH (Fig. 6) µ L 69.5 clivagem reserva * *
Tabela 5: Volume de tampão de reação nos estudos demonstraram. * Amprenavir foi resolvido em dimetil-sulfóxido; soluções estoque amprenavir (variando de 1 nanômetro a concentrações de 1 µM) foram aplicados para estudo inibitório (ver Figura 5B). * * O pH do buffer clivagem aplicada variou entre pH 6.0-8.5. - Feche as tampas dos tubos. Agora as amostras estão prontas para o ensaio.
Nota: As amostras podem ser armazenadas a 4 ° C por até 24 h, mas o armazenamento só é aplicável se a solução estoque SAMB foi usada imediatamente após o preparo (ver passo 4.1.3.2).
- Prepare-se para as amostras de ensaio 2 mL de tubos microcentrifuga de baixa proteína-ligação.
- Geração de grânulos magnéticos substrato-inscritos
- Iniciação das reações proteolíticas
- Prepare a solução de enzima proteolítica de acordo com as necessidades de experimentais.
Nota: É recomendável usar um buffer de clivagem para dissolver ou diluir a enzima. Protocolos para a purificação do HIV-114 e TEV PRs18 foram publicados anteriormente. - Defina do thermoshaker taxa de agitação (600 rpm) e temperatura de incubação (quadro 6).
Tipo de estudo Temperatura de incubação (° C) Medições de S-dependente (Fig. 4) 37 Medições de tempo-curso (Fig. 5A) 37 Estudo de inibição (Fig. 5B) 37 estudo de dependência do pH (Fig. 6) 30
Tabela 6: temperaturas de incubação aplicadas no estudo de diferentes tipos. Para HIV-1 PR, 37 ° C é recomendado, enquanto 30 ° C é recomendado para TEV PR. - Adicione a solução de enzima para as amostras de reação para inicializar reações proteolíticas.
Nota: No caso do branco do substrato (B) e amostras C, adicione clivagem buffer (buffer de enzima) e tampão de eluição, respectivamente. O volume é calculado de acordo com as necessidades individuais de experimentais (tabela 7). Para tubos de 2 mL, recomendado volume final da mistura reacional (o volume da reação reserva adicionado na etapa 4.1.4.5 + o volume da solução a ser adicionado nesta etapa) é de 50-150 µ l.Tipo de estudo Volume de tampão de eluição/tampão de solução/enzima a enzima (µ l) Medições de S-dependente (Fig. 4) 2 Medições de tempo-curso (Fig. 5A) 2 Estudo de inibição (Fig. 5B) 2 estudo de dependência do pH (Fig. 6) 0,5
Tabela 7: Volume da enzima enzima/solução tampão/eluição reserva adicionado durante a inicialização das amostras de ensaio, no caso os estudos demonstraram. - Agitar as contas cuidadosamente movendo suavemente os tubos e colocar imediatamente os tubos da thermoshaker já tremendo.
Nota: Terminação Manual da amostra (ver secção 4.3) leva mais tempo do que a iniciação; Portanto, recomenda-se um atraso registrado de pelo menos 2 min entre iniciações das reações. - Incube as amostras de acordo com o projeto experimental (tabela 8).
Tipo de estudo Tempos de incubação (min) Medições de S-dependente (Fig. 4A) 7 Medições de S-dependente (Fig. 4B) 120 Medições de tempo-curso (Fig. 5A) 0 – 2,5 – 5 – 10 – 15 – 20 Estudo de inibição (Fig. 5B) 10 estudo de dependência do pH (Fig. 6) 60
Tabela 8: Os tempos de incubação aplicados para as amostras de ensaio nas diferentes medições.
- Prepare a solução de enzima proteolítica de acordo com as necessidades de experimentais.
- Estaçãation das reações proteolíticas
- Tire o agitador, 30 s antes do final da incubação, a amostra e girá-lo prontamente.
- Coloque o tubo sobre o MPC, deixe 15 s, e deslocar ligeiramente o MPC e para trás.
- Abra a tampa e transferir o sobrenadante cuidadosamente para um prato ou um tubo novo.
Nota: Não toque os grânulos concentrados com a ponta da pipeta. O sobrenadante coletado de amostras C e R amostras com um alto grau de segmentação pode mostrar fluorescência visível com ou mesmo sem o uso de um DRBT.
- Deteção fluorescente
- Transferi 2 x 30 µ l dos sobrenadantes de amostra separada para uma microplaca de metade-área preta.
- Medir a fluorescência usando os filtros apropriados de excitação e emissão.
Nota: Medir a fluorescência básica do buffer de clivagem e tampão de eluição, também. Combinações de filtro precisa ser escolhido com base na proteína fluorescente medida (tabela 9).Proteína fluorescente Filtros de excitação (nm) Filtros de emissão (nm) mTurqiouse2 355/40 460/25 mEYFP 544/15 590/10 mApple 544/15 590/10
Tabela 9: Excitação e emissão de filtros usados para detectar diferentes proteínas fluorescentes.
- Calibração
Nota: Para gerar as curvas de calibração na etapa 4.6.1, valores de intensidade de fluorescência de clivagem - ou eluição-tampão-resolvido substratos purificados em diferentes concentrações precisam ser medido.- Purifica os substratos fluorescentes.
Nota: Para a purificação, SAMBs de substrato em branco (B) amostras após o ensaio de protease pode ser recolhido ou um SAMB nova suspensão pode também ser preparado (ver seções 4.1.1 e 4.1.2).- Coloque um tubo com SAMBs suspendidos em 1 mL de tampão de clivagem (2% - 10%; v/v) para o MPC e coletar os grânulos magnéticos girando o MPC de ponta-cabeça em todas as direções.
- Abra o tubo e retire o buffer de clivagem, tanto do tubo e a tampa.
- Retire o tubo do MPC e adicionar 400-600 µ l de tampão de eluição para as SAMBs.
- Gire lentamente o tubo fechado com um rotador em temperatura ambiente por 5 min.
- Coloque o tubo sobre o MPC e coletar os grânulos girando o MPC de ponta-cabeça.
- Remover o sobrenadante contendo o substrato fluorescente intacto purificado (eluído) e transferi-lo para um novo tubo de baixa proteína-vinculação microcentrifuga.
Nota: O eluato mostra fluorescência claramente visível com ou mesmo sem usar um DRBT.
- Realizar a troca de amortecedor paralela usando dois dispositivos de ultrafiltração de 0,5 mL 10K.
- Medir a metade do volume do eluato preparado (200-300 µ l) para cada dispositivo de ultrafiltração.
- Depois em cada etapa de centrifugação, diluir o eluído concentrado no primeiro e os segundo dispositivos de ultrafiltração por buffers de eluição e clivagem, respectivamente.
- Após a recuperação, ajuste as amostras concentradas resolvidas nos buffers diferentes para o mesmo volume, entre 120-200 µ l.
Nota: Agora o teor de proteínas do substrato reserva-resolvido clivagem é idêntico ao substrato de resolvido-tampão de eluição; Portanto, não é necessário determinar o conteúdo de um último passo 4.5.3, a proteína se o método usado para medir a concentração de proteína interfere com EDTA.
- Determinar o teor de proteínas dos substratos dissolvido em tampão de eluição ou clivagem medindo a absorbância em 280 nm.
Nota: Outros métodos (por exemplo, Bradford ou bicinchoninic ácido (BCA) ensaios) também podem ser usados para medir a concentração de proteína, mas a possível interferência com EDTA (presente no buffer de eluição) ou com a absorção do substrato fluorescente precisa ser considera-se. Recomenda-se o teor de proteínas inicial da solução de substrato a ser aplicado na etapa 4.5.4 estar entre 0,4-2,0 mg/mL, a fim de gerar uma calibração curva em uma gama adequada. Ver tabela 10 para coeficientes de extinção.Substrato Peso molecular
(Da)Coeficiente de extinção
(Cm M-1 -1, em 280 nm medido na água)Seu6- MBP-VSQNY * PIVQ-mTurquoise2 72101.7 96845 Seu6- MBP-KARVL * macacos-mTurquoise2 72042.7 95355 Seu6- MBP-VSQNY * PIVQ-mEYFP 72367.1 94325 Seu6- MBP-VSQNY * PIVQ-mApple 72145.9 105200
Quadro 10: pesos moleculares e os coeficientes de extinção de substratos de proteína da fusão fluorescente recombinante diferentes. - Prepare uma diluição serial duplo pelo menos oito passos, tanto da eluição - e a partir das soluções de substrato de clivagem-tampão-resolvido, usando o buffer de clivagem ou eluição para a diluição, respectivamente.
- Transferi 30 µ l de cada diluição ponto para uma microplaca de metade-área preta.
- Medir a fluorescência com um fluorímetro, usando a configuração aplicada na etapa 4.4.2.
Nota: Medir a fluorescência básica de ambos o decote e o tampão de eluição.
- Purifica os substratos fluorescentes.
- Avaliação do ensaio
- Traça as curvas de calibração.
- Calcule a concentração (em mM) das soluções de substrato purificado (usado na etapa 4.5.4), baseada-se o teor de proteínas, determinado na etapa 4.5.3.
- Corrigi os valores de intensidade de fluorescência relativo (RFU) de pontos de diluição serial pelos valores RFU básicos do buffer (buffer de clivagem ou o tampão de eluição) diluição aplicada.
- Plotar os valores RFU corrigidos contra a concentração molar de substratos purificados clivagem - ou eluição-tampão-resolvido e realizar uma regressão linear (força a interceptar a zero).
Nota: Um valor elevado de2 R (˃0.97) indica uma boa correlação linear entre a fluorescência e a concentração da proteína fluorescente. Neste caso, o declive da linha de regressão pode ser usado para avaliar as concentrações dos componentes do ensaio na faixa analisada em etapas 4.6.2 e 4.6.3. Distribuição de ponto de dados e erros experimental pode afetar a confiabilidade da calibração; assim, uma avaliação gráfica pode ser realizada com a ajuda de zoom em gráficos (como mostrado na Figura 3), a fim de verificar se R2 e os valores de inclinação são influenciados pelos dados.
- Calcule a quantidade de produto de clivagem fluorescente C-terminal nas amostras de reação.
- Corrigi os valores RFU de cada amostra R com os valores RFU da amostra B correspondente.
- Calcular a concentração do produto de clivagem (em mM) nas amostras de reação dividindo o corrigido valores RFU pela encosta da calibração clivagem reserva-com base em curva (consulte a etapa 4.6.1.3).
- Calcule a concentração de substrato aplicado nas amostras de reação.
- Corrigi os valores RFU da amostra C com o valor RFU do buffer de base de eluição.
- Calcular a concentração do substrato (em mM) eluted nos sobrenadantes da amostra C dividindo sua corrigido valores RFU pela encosta da calibração eluição reserva-com base em curva (consulte a etapa 4.6.1.3).
- Determine a concentração de substrato (em mM) da solução-mãe SAMB usada para criar as amostras de ensaio na etapa 4.1.4.2, com base na seguinte equação:
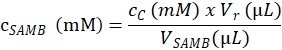
Aqui, cSAMB é a concentração molar da solução-mãe SAMB preparada na seção 4.1.3; cC é a concentração molar do substrato eluted na amostra C calculado no passo 4.6.3.3; Vr é o volume da mistura de reação, criado pela adição de buffer reação na etapa 4.1.4.5 e o buffer de enzima na etapa 4.2.3.; e VSAMB é o volume da solução da amostra C (etapa 4.1.4.2) SAMB. - Calcule a concentração molar de substratos em cada amostra R baseada a concentração molar da SAMB solução-mãe (em mM) de acordo com o volume (em µ l) medida em cada tubo de amostra de reação na etapa 4.1.4.2.
- Execute o processamento de dados.
Nota: A análise dos dados depende do objetivo do experimento. O vídeo mostra um exemplo de processamento de dados de um estudo cinético do substrato dependentes em HIV-1 PR usando seu6- MBP-VSQNY * PIVQ-mTurquoise2 substrato. Os valores de velocidade inicial são calculados a partir do número de fragmentos de clivagem do C-terminal e são plotados contra a concentração de substrato aplicado. Os parâmetros cinéticos são determinados por uma análise de regressão não-linear de Michaelis-Menten.
- Traça as curvas de calibração.
- Reciclagem dos grânulos magnéticos
Nota: Após a realização de um ensaio, os grânulos de agarose magnético podem ser recolhidos e reciclados.- Recolher os grânulos magnéticos usados com o MPC e descartar o sobrenadante.
- Lave os grânulos com 1,8 mL de buffers de seguir, na ordem dada: tampão de regeneração A (0,05% Tween 20, 0.5 M de NaOH), buffer de regeneração B (0,05% Tween 20), buffer de regeneração C (0,05% Tween 20, 100 mM EDTA, pH 8), buffer de regeneração B, regeneração tampão D ( 0,05% Tween 20, NiSO4a 100 mM, pH 8), buffers de regeneração B e regeneração E (0,5% de Tween 20, 30% de etanol, pH 7).
Nota: Para o procedimento de lavagem, consulte a etapa 4.1.1.4.
- Lave os grânulos com 1,8 mL de buffers de seguir, na ordem dada: tampão de regeneração A (0,05% Tween 20, 0.5 M de NaOH), buffer de regeneração B (0,05% Tween 20), buffer de regeneração C (0,05% Tween 20, 100 mM EDTA, pH 8), buffer de regeneração B, regeneração tampão D ( 0,05% Tween 20, NiSO4a 100 mM, pH 8), buffers de regeneração B e regeneração E (0,5% de Tween 20, 30% de etanol, pH 7).
- Armazenar os grânulos reciclados em buffer de regeneração E a 4 ° C.
- Recolher os grânulos magnéticos usados com o MPC e descartar o sobrenadante.
5. análise de página
-
Preparação da amostra
Nota: Depois de executar o ensaio de Ni-NTA magnética do grânulo-baseado, os sobrenadantes de ensaio podem ser analisados por página. Nesse caso, ignore os passos 5.1.1 e 5.1.2. No entanto, também é possível analisar a solução purificada substratos fluorescentes e/ou seus fragmentos de clivagem após digestão na solução com a protease de interesse. Neste caso, continue o protocolo com passo 5.1.1.- Prepare a solução de substrato fluorescente purificado conforme item 4.5.1.
- Execute em solução de digestão.
- Troca o tampão de eluição com buffer de clivagem no dispositivo de ultrafiltração de 0,5 mL 10K e alíquota as amostras para ser digerido em tubos de microcentrifuga de 1,5 mL.
Nota: para a análise de página, nós aliquotadas 68 µ l de cada substrato, mas o número de tubos de amostra e o volume de solução de substrato para ser aliquotadas podem ser otimizadas de acordo com o projeto experimental individual. - Adicione a solução de enzima para as amostras.
Nota: Para a análise de página, aplicamos 2 µ l de HIV-1 PR, preparado como descrito por Bozóki et al.14, mas o volume pode ser otimizado de acordo com o projeto experimental individual. É recomendável usar o buffer de clivagem para dissolver ou diluir a enzima. - Incube as amostras de acordo com o projeto experimental.
Nota: Para a análise de página, estamos incubados a mistura de reação por 45 min a 37 ° C, mas o tempo de incubação e temperatura precisam ser definidas de acordo com o projeto experimental. - Terminar a reação executando passo 5.1.3.
- Troca o tampão de eluição com buffer de clivagem no dispositivo de ultrafiltração de 0,5 mL 10K e alíquota as amostras para ser digerido em tubos de microcentrifuga de 1,5 mL.
- Prepare a amostra para a página.
Nota: As amostras fluorescentes-substrato-contendo podem estar preparadas para a página por um nondenaturing ou por um método de desnaturação. Para o uso de condições nondenaturing ou desnaturação, siga o passo 5.1.3.1 ou 5.1.3.2, respectivamente.- Preparar uma amostra de nondenatured: misturar 30 µ l da amostra com 6 µ l de 6 x nondenaturing tampão de amostra-carregamento (300 mM Tris, glicerol 20%, 0,05% de bromofenol, pH 6,8).
- Preparar uma amostra de desnaturado: misturar 30 µ l da amostra com 6 µ l de 6 x tampão de amostra-carregamento desnaturação (300 mM Tris, 20% de glicerol, 100mm de bromofenol, SDS de 12%, 0,05% β-Mercaptoetanol, pH 6,8) e aquecer as amostras a 95 ° C por 10 min.
-
Análise de SDS-PAGE
Nota: Opcionalmente, se apenas amostras nondenatured (preparado no passo 5.1.3.1) estão a ser analisado, uma página nativa também pode ser realizada. Nesse caso, ignore a secção 5.3.- Preparar um gel de SDS-poliacrilamida (uso 14% separando e 4% gel de empilhamento) e encha o depósito com tampão de eletroforese (2,5 mM Tris, glicina 19,2 mM, 0,01% SDS).
- Adicionar as amostras (preparadas na etapa 5.1.3.1 ou 5.1.3.2) nos poços de um gel de poliacrilamida e executar eletroforese na tensão de 120 V.
- Remova o cartucho de gel do módulo de execução e colocar o gel em um tanque de lavar roupa.
Nota: Amostras de Nondenatured já são visíveis no gel, até mesmo para o olho nu ou com um DRBT.
-
Renaturalização em-gel e detecção de proteínas fluorescentes
Nota: Para detectar as proteínas fluorescentes em amostras desnaturadas (preparadas na etapa 5.1.3.2) sobre o DRBT, SDS precisa ser lavada do gel, a renaturalização parcialmente as proteínas.- Adicionar ~ 100 mL de água destilada para o gel e lave o gel pelo menos durante 30 min.
Nota: Para melhorar a remoção de SDS, substituir a água a cada 10 minutos, ou lave até 60 min. - Visualize as proteínas fluorescentes, usando um DRBT, ou imagem de UV.
- Adicionar ~ 100 mL de água destilada para o gel e lave o gel pelo menos durante 30 min.
-
Coloração de Coomassie convencional do gel
- Manche o gel com corante azul brilhante de Coomassie Visualizar nonfluorescent proteínas.
Resultados
A figura 1A mostra a estrutura esquemática de um substrato de proteína recombinante fluorescente representativos que pode ser processado pelo PR de HIV-1 em sua sequência de sítio de clivagem específica. Figura 1B representa a produção de substrato e suas possíveis aplicações em ensaios de protease, incluindo ensaio de Ni-NTA magnética do grânulo-baseada e/ou página.
Para obter dados fiáveis por fluorimetria, um procedimento de calibração é necessário, a fim de determinar as quantidades de substratos fluorescentes e produtos de clivagem. Para isso, os valores de intensidade de fluorescência de substratos diferentes das condições de reserva diferentes precisam ser medidos e precisam ser correlacionada com as respectivas concentrações na faixa de concentração analisado (Figura 3). Os valores de inclinação das curvas de calibração podem ser aplicados para determinar as quantidades de substratos e produtos de clivagem nas amostras de ensaio. As inclinações das curvas de calibração são independentes das sequências clivagem local inseridas os substratos (tabela 11) e potencialmente podem ser usadas para uma série de substratos fundida-se para o mesmo tipo de proteína fluorescente. Zoom em gráficos são mostrados para todas as regressões lineares, para ampliar os intervalos de concentração mais baixos também (Figura 3). É importante notar que a calibração precisa ser executada com cuidado porque uma adequada distribuição de pontos de dados é necessária para uma calibração confiável. Por esta razão, dupla diluição serial é aplicada para preparar as amostras para a calibração, porque o valor de2 R indica uma boa correlação entre a concentração de proteína fluorescente e fluorescência, somente se um número suficiente de pontos de dados têm sido utilizados para cobrir o intervalo de concentração toda. Além disso, os erros experimentais altamente podem afetar a exatidão da calibração; assim, uma avaliação gráfica das linhas de regressão pode ser também necessária.
Uma variedade de medições enzimáticas pode ser executada pelo ensaio de protease, incluindo uma análise do efeito da concentração de substrato na velocidade da reação (Figura 4A). Por regressão não-linear, os dados podem ser usados para determinar os parâmetros cinéticos de enzima (por exemplo, vmax e Km). Uma suspensão de grânulo insuficiente e dispersão e uma finalização de reação inadequada podem causar resultados suboptimal (Figura 4B), que não são adequados para calcular valores de cinética enzimática confiável.
Uma dependência da formação do produto no tempo pode ser determinada por ensaio (Figura 5A) (por exemplo, durante a otimização dos parâmetros de reação de clivagem). Atividade enzimática na presença de um inibidor também pode ser investigado (Figura 5B) para a determinação da concentração de enzima activa e constante inibitória. Usando a mesma metodologia, efeitos de outros inibidores também podem ser rastreados por ensaio.
O ensaio de protease é útil quando investigando os efeitos do pH na atividade enzimática, também. Figura 6A representa a dependência da atividade enzimática no pH pelo exemplo de TEV PR, que tem uma faixa de largura ideal de pH (pH 6-9). Se a dependência do pH da atividade da enzima é estudada (ou enzimas, tendo uma ótima de pH ácido precisam ser medido), é necessário considerar que a ligação de afinidade de substratos recombinantes para os grânulos pode ser restrita em pH ligeiramente ácido. Uma dissociação elevada de substratos de grânulos (Figura 6B) pode causar uma distorção dos resultados do ensaio. Para considerar a dissociação espontânea do substrato de grânulos, os valores medidos para amostras de reação precisam ser corrigido das amostras de B.
A Figura 7 mostra que a nondenatured proteínas fluorescentes podem ser diferenciadas no gel com base em suas cores, usando transiluminação de luz azul (Figura 7A). Se a determinação dos pesos moleculares dos fragmentos de substratos/clivagem é necessária, desnaturação condições pode também ser utilizado para a preparação da amostra, porque proteínas fluorescentes podem ser parcialmente renatured no gel e podem ser detectadas por iluminação UV (Figura 7B) ou por Coomassie coloração (Figura 7). Se as amostras de R são analisadas, apenas os produtos de clivagem do C-terminal são visíveis (Figura 7), enquanto os fragmentos de clivagem do N-terminal e os substratos uncleaved permanecem ligados aos talões. Ocasionalmente, as proteínas podem desnaturar parcialmente apesar de usar condições nondenaturing (Figura 7), e enquanto as proteínas nondenatured são mais abundantes, desnaturadas formas também são detectáveis na amostra. Este fenômeno não influencia a detecção de clivagem proteolítica, mas precisa ser considerado no caso de densitometria quantitativa de amostras nondenatured.
Embora a descrição detalhada é mostrada apenas para um ensaio 2 mL tubo-baseados, o ensaio pode ser adaptado para um 96 poços baseados na placa de sistema (Figura 8), que já foi testado com sucesso em nosso laboratório (não mostrado). O formato de placa-adaptado de 96 poços é totalmente compatível com o fluorimétrica eletroforética análises e, também, e os dados obtidos também podem ser avaliados com base em métodos descritos neste artigo.
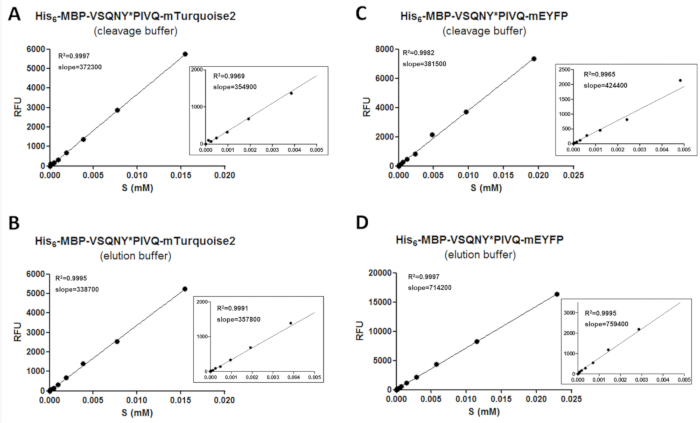
Figura 3 : As curvas de calibração. As curvas de calibração do substrato representativos são demonstradas com o exemplo de dois substratos recombinantes fundidos para diferentes etiquetas fluorescentes do C-terminal: (A e B) seu6- MBP-VSQNY * PIVQ-mTurquoise2 e (C e D de ) Seu6- MBP-VSQNY * PIVQ-mEYFP. Zoom em figuras também são mostradas para representar a regressão linear de pontos de dados na 0,005-0 intervalo de concentração de substrato de milímetros. Clique aqui para ver uma versão maior desta figura.
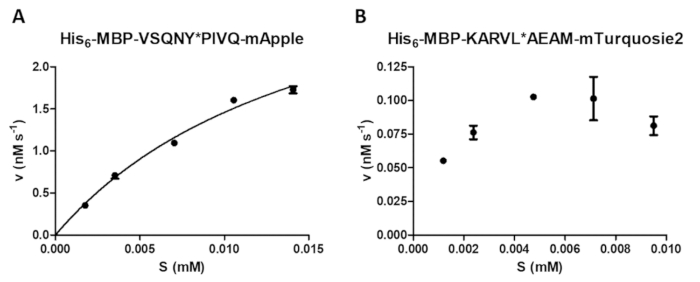
Figura 4 : Determinação de parâmetros cinéticos de enzima. Foram realizadas medidas cinéticas de substrato dependentes por HIV-1 PR (em uma concentração final de ativa de 41,2 nM). Os valores de velocidade inicial foram plotados contra a concentração de substrato e realizou-se uma análise de regressão não-linear de Michaelis-Menten. As barras de erro representam SD (n = 2). (A), A representante ideal resultado é mostrado no exemplo de seu6- MBP-VSQNY * PIVQ-mApple substrato de proteína de fusão. (B), um representante suboptimal resultado também é indicado para a sua6- MBP-KARVL * substrato de macacos-mTurquoise2, onde o cenário das concentrações de substrato adequado era problemático devido a uma homogeneização insuficiente da solução SAMB , enquanto relativamente elevados erros foram causados por rescisão de reação imprópria. Clique aqui para ver uma versão maior desta figura.
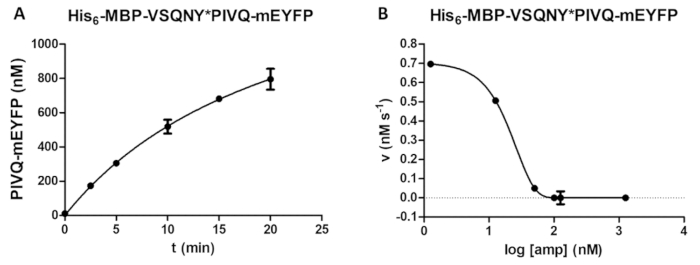
Figura 5 : Estudo do tempo-curso e inibitório. () Seu 6- MBP-VSQNY * PIVQ-mEYFP substrato de proteínas recombinantes de fusão (em uma concentração final de 0,00326 mM) foi clivado por HIV-1 PR (em uma concentração final de ativa de 41,2 nM), e o lançamento de fragmentos de proteolíticas fluorescentes PIVQ-mEYFP foi medido para realizar uma análise de tempo-curso. As medições foram realizadas em cinco pontos diferentes do tempo. As barras de erro representam SD (n = 2). (B) seu6- MBP-VSQNY * PIVQ-mEYFP foi usado como substrato (a 0,0015 mM) para determinar o efeito inibitório sobre a atividade do HIV-1 PR de amprenavir (numa concentração total de 163,8 nM). Por plotar os dados, o meia concentração inibitória máxima (IC50) podem ser avaliados e a concentração da enzima ativa (uma concentração final de ativa de 41,2 nM) do HIV-1 PR aplicada pode também ser calculada com base na curva de inibição. As barras de erro representam SD (n = 3). Clique aqui para ver uma versão maior desta figura.
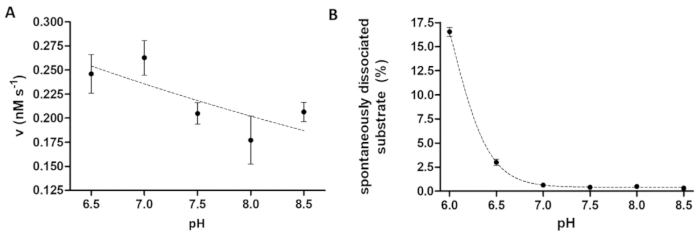
Figura 6 : Estudando a dependência da atividade enzimática e dissociação espontânea do substrato em pH (A) o seu MBP -6-VSQNY * PIVQ-mTurquoise2 substrato (em 0,033 mM) foi usado para medir a atividade de enzima de TEV PR (numa concentração total final de 91.42 nM) no buffer de clivagem definido para um pH diferente, entre o intervalo de 6.5-8.5. As barras de erro representam SD (n = 2). Os dados plotados tem sido publicadas anteriormente14. (B) com base nos valores a intensidade fluorescente relativo as amostras em branco do substrato, a dissociação espontânea de seus6- MBP-VSQNY * PIVQ-mTurquoise2 substrato (0,033 mM) de grânulos magnéticos foi estudado usando buffer de clivagem com um pH diferente, entre 6.0-8.5. Os dados plotados tem sido publicadas anteriormente14. Clique aqui para ver uma versão maior desta figura.
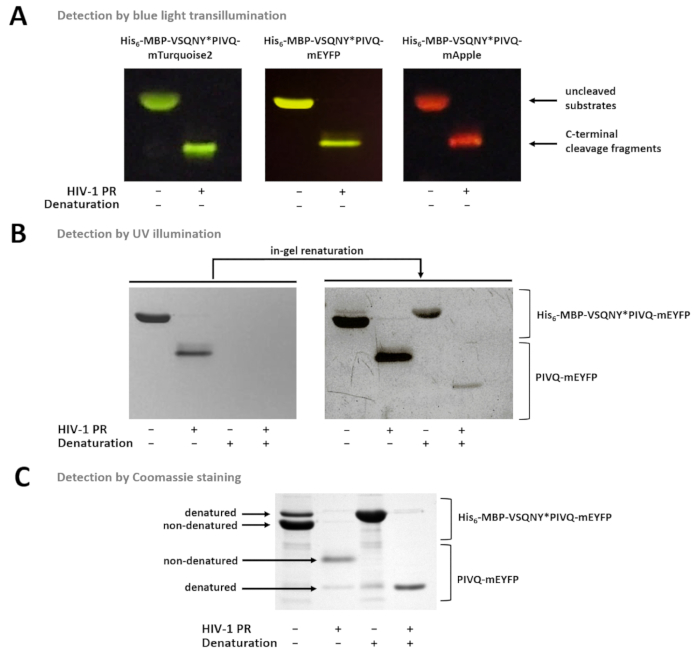
Figura 7 : Detecção de proteínas no gel por diferentes métodos. (A) Uncleaved e substratos de proteína de fusão do HIV-1 PR-digerido após a preparação da amostra nondenaturing foram visualizados por transiluminação luz azul depois de SDS-PAGE. A reação de clivagem foi realizada por digestão na solução. (B) imediatamente após a página, só nondenatured proteínas pode ser detectada no gel por iluminação UV, enquanto após a remoção do SDS, as proteínas fluorescentes previamente desnaturadas tornou-se parcialmente renatured e detectável. As amostras foram preparadas a partir os sobrenadantes do ensaio Ni-NTA magnética do grânulo-baseados. (C) coloração Coomassie também pode ser usado para detecção de proteínas, após a renaturalização em-gel. O SDS-presente em gel-maio causam a desnaturação parcial da proteína nativa, mas em amostras nativas, as formas de nondenatured são mais abundantes. As amostras foram preparadas a partir os sobrenadantes do ensaio Ni-NTA magnética do grânulo-baseados. Clique aqui para ver uma versão maior desta figura.

Figura 8 : adaptação baseada em placa de 96 poços da plataforma de ensaio. (A), o ensaio pode ser realizada não só em tubos de 2 mL, mas nos poços de uma placa de 96 poços, também. Aqui nós mostramos a representação esquemática para a aplicação do ensaio estudar a especificidade de uma protease fictícia usando uma série de substratos fluorescentes, que podem conter o selvagem-tipo (wt) ou mutante (mut-1 a mut-4) sequências de sítio de clivagem. Para lidar com os grânulos magnéticos, um concentrador de partícula magnética compatível com 96 poços (MPC) é para ser usado nos experimentos. Todos os volumes indicados estão relacionados com um único poço. Para comparar a eficiência de clivagem de diferentes substratos, conversão de substrato pode ser avaliado da percentagem de substrato-em branco-corrigido valores RFU das amostras de reação, considerando os valores RFU substrato-em branco-corrigida das amostras de controlo de substrato como 100. (B) depois de fluorimetria, os sobrenadantes separados do ensaio as amostras também podem ser analisadas por página e os componentes de proteína fluorescente pode ser analisado diretamente ou após a renaturalização em-gel no caso de amostra nondenaturing e desnaturação preparação, respectivamente. Os três tipos de amostra de ensaio diferentes também são ilustrados em cada figura: C = controle de substrato, B = branco do substrato e R = reação. Amostras de controlo de substrato são em tampão de eluição, enquanto o branco do substrato e as amostras de reação estão no buffer de clivagem. Clique aqui para ver uma versão maior desta figura.
| Buffer de | Proteína fluorescente | CV % das pistas (%) |
| Eluição | mTurquoise2 | 6.04 |
| Clivagem | 9.11 | |
| Eluição | mApple | 10.92 |
| Clivagem | 12,68 |
Quadro 11: coeficiente de valores de variação (% CV) das encostas das curvas de calibração do substrato. Para testar se a fluorescência de substratos a proteína recombinante é dependente o local de clivagem inserido, calibrações foram realizadas por uma série de substratos de mApple - e mTurquoise2-fundido (seis variantes para cada um, contendo o local de clivagem diferente sequências de protease do HIV-1), ambos nos buffers de eluição e clivagem. Nós achamos que valores de CV % das encostas estão abaixo dos 15% em todos os casos, o que implica que uma calibração de substrato único pode ser utilizada para a avaliação das diferentes medições realizadas por variantes de substrato contendo a mesma marca de fluorescente.
Discussão
Devido as investigações industriais e acadêmicas intensivas de enzimas proteolíticas e a constante procura de célere e acessíveis plataformas de ensaio de protease HTS-compatível nesse sentido, temos desenvolvido uma protease fluorescente magnético do grânulo-baseados do ensaio. O ensaio baseia-se na utilização de proteínas recombinantes de fusão que podem ser novas alternativas para os substratos de peptídeo sintético amplamente utilizada.
No formato de ensaio desenvolvidos, os substratos de proteína de fusão são imobilizados nas superfícies dos grânulos de agarose magnética Ni-quelato-revestido. O acessório de substrato é fornecido pelo N-terminal sua marca6 afinidade da proteína de fusão, que é fundida diretamente a um tag MBP para facilitar a dobradura e aumentar a solubilidade em água do substrato13. O MBP é seguido por sítios de clivagem de TEV PR e uma protease de interesse. O primeiro pode servir como um local de clivagem de controle no ensaio, enquanto o último pode ser processado pela protease a ser investigada. O local de clivagem é intercambiável; uma sequência curta dsDNA codificação para o local de clivagem de interesse pode ser inserida dentro do flexível' clonagem' do plasmídeo de expressão pela ligadura. As proteínas recombinantes de fusão contêm uma marca altamente estável, monomérica proteína fluorescente no C-terminal, que permite a detecção de ponto de extremidade de enzima-liberado, fluorescentes C-terminal clivagem produtos divulgada mediante clivagem proteolítica ( Figura 1A). Os substratos intactos fluorescentes purificados resolvidos nos buffers diferentes também são usados para calibração para avaliar as concentrações molares de substratos e produtos de clivagem. Além disso, após fluorimetria, os componentes do ensaio podem ser analisados por SDS-PAGE, também. Ambos os nativos (nondenatured) e proteínas desnaturadas fluorescentes podem ser visualizadas no gel, imediatamente após a electroforese ou após subsequente em-gel renaturalização, respectivamente. Este adicional procedimento-em combinação com um convencional azul de Coomassie brilhante coloração-maio ser usada eficientemente para a verificação dos resultados do ensaio (figura 1B).
O procedimento de ensaio consiste em passos simples, fácil-para-executar em um formato de baixo volume que pode ser totalmente adaptado a um ambiente de alta produtividade automático. No entanto, independentemente da realização do ensaio, manualmente ou com um sistema de automação, as seguintes partes do ensaio são consideradas ser crucial e precisam de atenção especial durante a realização do procedimento. i) homogeneidade da solução magnética do grânulo. Uma solução homogênea do grânulo magnético deve ser usada durante todo o ensaio, tanto na purificação e etapas de lavagem. Particularmente, a confiabilidade dos ensaios de protease depende fortemente corretamente alíquotas das soluções estoque conectado à carcaça magnética do grânulo (Claudecir). Para aumentar a eficácia da suspensão e dispersão, recomenda-se definir a concentração do grânulo entre 2% e 10% (v/v). Durante a preparação da amostra, o uso de buffers suplementados com detergente não iônico (tais como o Triton X-100 ou Tween 20) até 2% também pode diminuir a aderência dos grânulos magnéticos para superfícies de plástico. A aderência dos grânulos nas paredes dos frascos de amostra pode ser evitada se as suspensões do grânulo são aplicadas com cuidado para o fundo dos frascos em vez de nas paredes dos tubos de amostra. A homogeneidade dos grânulos magnéticos durante a reação enzimática é também crítica e pode ser assegurada agitando continuamente as amostras a 600 rpm durante a incubação. Grânulos são devidamente dispersos em utensílios de plástico arredondados ou achatados, enquanto não se recomenda o uso de frascos de V-fundo. Um resultado de qualidade inferior causado por homogeneização inadequada do grânulo é representado na Figura 4B. II) término de amostras de reação. Outra vantagem do método é que a reação enzimática pode ser encerrada sem a utilização de tratamento de calor desnaturação ou qualquer de agentes químicos potencialmente interferentes15. A rescisão pode ser realizada simplesmente separando as esferas magnéticas da mistura de reação, usando um concentrador de partícula magnética convencional. Enquanto o amortecedor da reação removido contém a enzima activa e os produtos de clivagem fluorescentes gerados C-terminal, os substratos uncleaved permanecem ligados aos talões. Devido à presença da enzima activa no buffer de reação, o procedimento de separação precisa ser realizada com cuidado para a deteção de confiança do ponto de extremidade. Antes de colocar os frascos de amostra para o concentrador, é aconselhável aplicar uma centrifugação de curta rotação. Depois de colocar os tubos para o concentrador, fornecer pelo menos 15 s para as contas a cobrar. Ligeiro movimento do separador e para trás pode facilitar a recolha dos grânulos. Por favor, considere que, durante uma separação realizada manualmente, a rescisão geralmente leva mais tempo do que o início das reações. Portanto, um atraso de cerca de 2 min registrado recomenda-se, entre as iniciações, se o mesmo tempo de incubação deve ser aplicado para todas as amostras.
O princípio do ensaio descrito proteolítica é relativamente simples; no entanto, a versatilidade do sistema é garantida pela estrutura de substrato flexível e estável. A otimização individual do ensaio pode ser limitada apenas pela compatibilidade dos grânulos afinidade com as condições aplicadas, reagentes e aditivos. De acordo com o protocolo do fabricante, nós também achamos que a ligação de afinidade de substratos para as superfícies de grânulo de Ni-NTA enfraquece substancialmente a pH ≤ 6,515. Portanto, é recomendável aplicar amostras em branco do substrato paralelas para as amostras de reação, e a taxa de dissociação espontânea do substrato precisa ser considerado durante a avaliação dos resultados.
Nesses casos, onde magnética do grânulo-ensaios baseados não podem ser executados devido ao uso de componentes incompatíveis do grânulo ou um baixo pH, em solução de digestão de substratos recombinantes purificados pode igualmente ser aplicado. Nestes casos, as misturas de reação podem ser analisadas por eletroforese, e as proteínas podem ser visualizadas no gel baseado no protocolo descrito. Para investigar a atividade proteolítica, digestão em solução e em gel detecção de proteínas também podem ser ferramentas alternativas de fluorimetria. Uma novidade do sistema projetado substrato é a aplicação de uma etapa de renaturalização de em-gel após desnaturação de SDS-PAGE. Enquanto o nativas proteínas fluorescentes (nondenatured) retêm sua fluorescência durante a electroforese, o Propriedade fluorescente é abolido após desnaturação (Figura 7B). No entanto, a fluorescência de proteínas desnaturadas pode ser parcialmente recuperada pela remoção do gel de SDS. Assim, uma separação dos componentes de reação utilizando condições desnaturação possibilita não só a base de fluorescência mas a identificação molecular baseados em peso. Outra vantagem da detecção fluorescente em-gel em comparação com a análise de um gel manchado de Coomassie é que as proteínas fluorescentes (nativo ou renatured) podem ser facilmente identificadas no gel com base em sua fluorescência (ver Figura 7). Isto pode ser importante se as reações de clivagem são realizadas em amostras contendo contaminantes nonfluorescent ou proteínas altamente, assemelhando-se os pesos moleculares do outro.
Ensaios de protease similarmente usando substratos projetados já foram publicadas anteriormente8,9,10, e embora o local de clivagem de interesse nesses casos também estava localizado entre uma tag de afinidade e um proteína fluorescente, o sistema de ensaio apresentado aqui não apenas repete as ideias descritas mas combina as vantagens diferentes das plataformas anteriores e também conclui-los com novas melhorias: i) a utilização de um parceiro de fusão de MBP, ii) a presença de um local de clivagem do controle do PEV PR, iii) o uso de FPs monomérico recentemente projetado e iv) a aplicação de um procedimento de calibração do substrato original. O ensaio em si foi projetado especialmente para ser útil para a especificidade da enzima e estudos cinéticos de forma segura, tempo e custo-eficiente forma, sem a necessidade de instrumentação cara. O método é destinado a ser uma ferramenta acessível e adequada para ambos os fins de pesquisa acadêmica e industrial. Devido à flexibilidade da fita' clonagem' do plasmídeo de expressão, o sistema pode ser adequado para a geração rápida e barata de bibliotecas de substrato recombinante. O ensaio descrito neste documento é uma ferramenta viável para a implementação de especificidade de substrato, mutagênese de enzima, e inibição estudos e, também, fornecer uma ferramenta alternativa para executar cinética enzimática. A plataforma de ensaio (a partir de rompimento da pilha bacteriana para a determinação dos parâmetros cinéticos) pode ser adaptada a um ambiente de HTS e automação baseada e, potencialmente, pode ser aplicada em triagem de inibidor de protease industrial e/ou drogas antivirais desenvolvimento. Além disso, a adaptação do ensaio para proteólise competitiva também é no futuro escopo de nosso laboratório. Em um ensaio tão competitivo, dois diferentes substratos, cada contendo um sítio de clivagem diferente fundido a um diferente C-terminal fluorescente marca-são destinado a ser utilizados simultaneamente na mesma reação de clivagem para investigar a preferência dos estudados enzima para as sequências de determinado destino. Além disso, o uso de um formulário de ensaio adaptado de placa de 96 poços (Figura 8) também está sendo otimizado para rastreio de mutação usando uma série de substratos com sequências de sítio de clivagem modificada em caso de proteases de cisteína.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi parcialmente suportado pelo projeto "Unindo PHARMPROT" GINOP-2.3.2-15-2016-00044 e, também, financiado pelo programa de excelência institucional de ensino superior de Ministério das capacidades humanas na Hungria, no âmbito da Programa temático de biotecnologia da Universidade de Debrecen. Os autores são gratos por membros do laboratório de bioquímica Retroviral pela ajuda durante o desenvolvimento do ensaio científica e também pela sua paciência durante as filmagens do ensaio (especialmente para Norbert Kassay, Krisztina Joóné Matúz e Vanda Toldi, que estão aparecendo no fundo do vídeo). Os autores também queria dizer especial graças a Gedeon Richter Plc., especialmente ao Dr. Zoltán Urbányi para permitir que o trabalho de Beáta Bozóki no departamento de Bioquímica e Biologia Molecular, como pesquisador convidado. Os autores também gostaria de estender sua gratidão ao György Zsadányi, Balázs Tőgyi, Balázs Pöstényi e Zoltán Király do centro técnico de E-learning da Universidade de Debrecen e multimídia para a assistência profissional em áudio e vídeo produção.
Materiais
| Name | Company | Catalog Number | Comments |
| 10K Amicon tubes | Merck-Millipore | UFC501096 | |
| 2-Mercaptoethanol (β-ME) | Sigma-Aldrich (St Louis, MO, USA) | M6250 | |
| 40% Acrylamide/Bis solution 37.5:1 | Bio-Rad | 1610148 | |
| Acetic acid | Merck | 100063 | |
| Agarose | SERVA | 11404.04 | |
| Alpha Imager HP gel documentation system | ProteinSimple | ||
| Ammonium persulfate (APS) | Sigma-Aldrich (St Louis, MO, USA) | A3678 | |
| Ampicillin sodium salt | Sigma-Aldrich (St Louis, MO, USA) | A9518 | |
| Beckman Coulter Allegra X-22 centrifuge | Beckman Coulter | 392185 | |
| Black half-area plates | Greiner bio-One | 675086 | |
| Bromophenol blue | Sigma-Aldrich (St Louis, MO, USA) | B0126 | |
| CutSmart buffer (10x) | New England Biolabs | B7204S | For plasmid linearization (step 1.1.1) |
| Dark Reader transilluminator | Clare Chemical Research | DR-45M | |
| DNase I | New England Biolabs | M0303L | |
| Dynamag-2 magnetic particle concentrator | Thermo Fischer Scientific | 12321D | |
| Escherichia coli BL21(DE3) competent cells | Thermo Fischer Scientific (Invitrogen) | C600003 | |
| Ethanol | Merc-Millipore | 100983 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich (St Louis, MO, USA) | 798681 | |
| Gel Loading Dye, Purple (6X) | New England Biolabs | B7024S | |
| Glycerol | Merck | 356350 | |
| Glycine | Sigma-Aldrich (St Louis, MO, USA) | G7126 | |
| High-Speed Plasmid Mini Kit | GeneAid | PD300 | |
| Imidazole | Sigma-Aldrich (St Louis, MO, USA) | 56750 | |
| Isopropyl β-D-1-thiogalactopyranoside (IPTG) | Thermo Fischer Scientific (Invitrogen) | AM9464 | |
| Jouan CR 412 centrifuge | Jouan | CR412 | |
| Labinco LD-76 Rotator | Labinco | 7600 | |
| Luria-Bertani (LB) broth | Sigma-Aldrich (St Louis, MO, USA) | L3022 | |
| Lysozyme | Sigma-Aldrich (St Louis, MO, USA) | L6876 | |
| Magnesium chloride | Scharlau | MA0036 | |
| MERCK eurolab ultrasonic bath | MERCK | USR54H | |
| Millifuge Eppendorf spin centrifuge | Millipore | CT10 | |
| Mini-PROTEAN 3 Electrophoresis Cell | Bio-Rad | ||
| N,N,N′,N′-Tetramethylethylenediamine (TEMED) | Sigma-Aldrich (St Louis, MO, USA) | T9281 | |
| NanoDrop 2000 | Thermo Fischer Scientific | ||
| NheI-HF restriction endonuclease | New England Biolabs | R3131L | |
| Nickel(II) sulfate (NiSO4) | Sigma-Aldrich (St Louis, MO, USA) | 656895 | |
| Ni-NTA magnetic agarose beads | Qiagen | 36113 | |
| Orbital shaker | Biosan | OS-20 | |
| PacI restriction endonuclease | New England Biolabs | R0547L | |
| PageBlue Protein Staining Solution | Thermo Fischer Scientific | 24620 | |
| Phenylmethanesulfonyl-fluoride (PMSF) | Sigma-Aldrich (St Louis, MO, USA) | P7626 | |
| Protein Lobind Micro-centrifuge tubes | Eppendorf | 22431102 | |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| Snijders Press-to-Mix shaker | Gemini | 34524 | |
| Sodium-acetate trihydrate | Sigma-Aldrich (St Louis, MO, USA) | S7670 | |
| Sodium-chloride | Sigma-Aldrich (St Louis, MO, USA) | S9888 | |
| Sodium-hydroxide | Sigma-Aldrich (St Louis, MO, USA) | S5881 | |
| SYBR Green I Nucleic Acid Gel Stain | Thermo Fischer Scientific | S7563 | |
| T4 DNA ligase | Promega | M180A | |
| Thermo shaker | Biosan | TS-100 | with SC-24 accessory block |
| Tris | Sigma-Aldrich (St Louis, MO, USA) | T1503 | |
| Tween 20 | Sigma-Aldrich (St Louis, MO, USA) | P2287 | |
| WALLAC VICTOR2 1420 multilabel counter | Wallac Oy, Turku, Finland |
Referências
- Meldal, M. Smart Combinatorial Assays for the Determination of Protease Activity and Inhibition. Molecular Informatics. 24 (10), 1141-1148 (2005).
- Rao, M. B., Tanksale, A. M., Ghatge, M. S., Deshpande, V. V. Molecular and biotechnological aspects of microbial proteases. Microbiology and Molecular Biology Reviews. 62 (3), 597-635 (1998).
- Zhang, G., Sittampalam, G. S., et al. Protease assays. The Assay Guidance Manual. , (2012).
- Woelcke, J., Hassiepen, U., Chen, T. Fluorescence-based biochemical assay formats. A Practical Guide to Assay Development and High-Throughput Screening in Drug Discovery. , (2010).
- Richardson, P. L. The determination and use of optimized protease substrates in drug discovery and development. Current Pharmaceutical Design. 8 (28), 2559-2581 (2002).
- Diamond, S. L. Methods for mapping protease specificity. Current Opinion in Chemical Biology. 11 (1), 46-51 (2007).
- Schilling, O., Overall, C. M. Proteome-derived, database-searchable peptide libraries for identifying protease cleavage sites. Nature Biotechnology. 26 (6), 685-694 (2008).
- Askin, S. P., Morin, I., Schaeffer, P. M. Development of a protease activity assay using heat-sensitive Tus-GFP fusion protein substrates. Analytical Biochemistry. 415 (2), 126-133 (2011).
- Chaparro-Riggers, J. F., Breves, R., Michels, A., Maurer, K. H., Bornscheuer, U. A GFP based assay for the determination of hydrolytic activity and substrate specificity of subtilisins under washing conditions. Journal of Molecular Catalysis B: Enzymatic. 35, 74-77 (2005).
- Patel, D., Frelinger, J., Goudsmit, J., Kim, B. In vitro assay for site-specific proteases using bead-attached GFP substrate. Biotechniques. 31 (5), 1194-1198 (2001).
- Branchini, B. R., et al. Sequential bioluminescence resonance energy transfer-fluorescence resonance energy transfer-based ratiometric protease assays with fusion proteins of firefly luciferase and red fluorescent protein. Analytical Biochemistry. 414 (2), 239-245 (2011).
- Zhou, C., Yan, Y., Fang, J., Cheng, B., Fan, J. A new fusion protein platform for quantitatively measuring activity of multiple proteases. Microbial Cell Factories. 13 (1), 44 (2014).
- Fox, J. D., Waugh, D. S. Maltose-binding protein as a solubility enhancer. Methods in Molecular Biology. 205, 99-117 (2003).
- Bozóki, B., et al. A recombinant fusion protein-based, fluorescent protease assay for high throughput-compatible substrate screening. Analytical Biochemistry. 540-541, 52-63 (2018).
- Mótyán, J. A., Miczi, M., Bozóki, B., Tözsér, J. Data supporting Ni-NTA magnetic bead-based fluorescent protease assay using recombinant fusion protein substrates. Data in Brief. 18, 203-208 (2018).
- Balleza, E., Kim, J. M., Cluzel, P. Systematic characterization of maturation time of fluorescent proteins in living cells. Nature Methods. 15, 47-51 (2018).
- Hebisch, E., Knebel, J., Landsberg, J., Frey, E., Leisner, M. High variation of fluorescence protein maturation times in closely related Escherichia coli strains. PLoS One. 8, e75991 (2013).
- Kapust, R. B., et al. Tobacco etch virus protease: mechanism of autolysis and rational design of stable mutants with wild-type catalytic proficiency. Protein Engineering, Design and Selection. 14, 993-1000 (2001).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados