É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Vetor de Lentivirus de plataforma para a entrega eficiente de ferramentas de edição de Epigenoma em humanos induzida por modelos de doenças derivadas de células-tronco pluripotentes
Neste Artigo
Resumo
Alvo de DNA Epigenoma edição representa uma poderosa abordagem terapêutica. Este protocolo descreve a produção, purificação e concentração de vetores Lentivirus all-in-one, abrigando o transgene CRISPR-dCas9-DNMT3A para aplicativos de edição de Epigenoma em células-tronco pluripotentes induzidas humana (hiPSC)-derivado de neurônios.
Resumo
O uso de derivados de hiPSC células representa uma abordagem valiosa para estudar doenças neurodegenerativas humanas. Aqui, descrevemos um protocolo otimizado para a diferenciação de hiPSCs derivado de um paciente com a triplicação do locus de gene (SNCA) alfa-synuclein para a doença de Parkinson (PD)-populações neuronal dopaminergic relevantes. Acumular provas demonstrou-se que altos níveis de SNCA são causais para o desenvolvimento de PD reconhecer o insatisfeitas precisam estabelecer novas abordagens terapêuticas para a polícia, especialmente os alvos a regulação da expressão SNCA , nós recentemente desenvolveu um sistema CRISPR/dCas9-metilação-baseado no ADN para modular epigenetically transcrição SNCA enriquecendo os níveis de metilação na região reguladora SNCA intron 1. Para entregar o sistema, consistindo de um morto (desativado) versão do Cas9 (dCas9) fundiu-se com o domínio catalítico do DNA metiltransferase enzima 3A (DNMT3A), um vetor de Lentivirus é usado. Este sistema é aplicado às células com a triplicação do locus a SNCA e reduz os níveis de proteína e SNCA-mRNA por cerca de 30% através da metilação do DNA alvo da SNCA intron 1. A aperfeiçoá-lo downregulation dos níveis SNCA resgata doença relacionada com fenótipos celulares. O protocolo atual, nosso objetivo é descrever um procedimento passo a passo para diferenciar hiPSCs em células progenitoras neurais (NPCs) e a criação e a validação dos ensaios pyrosequencing para a avaliação do perfil de metilação na SNCA intron 1. Para descrever mais detalhadamente o sistema de lentivirus-CRISPR/dCas9 usado nesses experimentos, este protocolo descreve como produzir, purificar e concentrar Lentivirus vetores e para destacar a sua adequação para Epigenoma e genoma-edição de aplicações usando hiPSCs e NPCs. O protocolo é facilmente adaptável e pode ser usado para produzir lentivírus de alta concentração para aplicações in vitro e in vivo.
Introdução
Múltiplas plataformas Epigenoma-edição foram recentemente desenvolvidas para direcionar as sequências de DNA nas regiões que controlam a expressão de gene1,2. As ferramentas de edição Epigenoma criadas destinam-se a (i) regulam a transcrição, (ii) alterar modificações do histone posttranslational, (iii) modificar a metilação do DNA e (iv) modular interações elemento regulamentar. A abordagem para ancorar os modificadores de transcrição/cromatina para um Cas9 (morto) desativado (dCas9) ressuscitou anteriormente desenvolvidas Epigenoma-edição plataformas, tais como zinco dedo proteínas (ZFPs) e efetores, como ativador de transcrição (contos), abrigando um domínio efetor transcriptional potente (ED) fundido ao domínio de ligação a DNA projetado (DBD)3. Os resultados do fenótipo desejado como ativação ou repressão é definida pela molécula efetora ancorada ao Locus endógenos (Figura 1). Para criar ativadores transcricionais programáveis, dCas9/gRNA módulos estão ligados à VP164,5,6 (figura 1A), um domínio de ativação viral que recrutas Pol II e a maquinaria de transcrição geral. A modificação deste sistema incluiu VP64, um tetrâmero de domínios VP16, fornecendo um ainda mais robusta5,taxa de ativação6. O sistema tem sido empregado com sucesso para ativar regiões não-codificadoras e codificação, orientando os promotores e dos elementos reguladores. Importante, mesmo que VP64 moléculas diretamente não modifique a estrutura da cromatina na região de destino, ele recruta modificadores de cromatina, que se ligam os resultados no depoimento das marcas ativo (eucromatina), incluindo como acetilação H3/H4 e H3-K4 di / Tri-metilação5,6. Além de VP64, a subunidade p65 do NF-κB humano complexo tem sido amarrada ao dCas9/gRNA módulo7. Curiosamente, o tethering desses efetores às regiões montante dos locais de início da transcrição (TSSs) e dentro de promotores resulta em uma indução do gene forte. No entanto, VP64 e p65 efetores também podem exercer os efeitos activatory enquanto ser linkadas para as regiões situadas a jusante de TSSs e no distal potenciadores de7,8. Para eliciar uma resposta transcricional mais robusta, várias fusões de dCas9-VP64 ou dCas9-p65 deveriam ser recrutados para um destino único locus9,10. Como tal, o desenvolvimento recente de ativadores de próxima geração, que recrutar vários domínios efetoras por um complexo único dCas9-gRNA, tais como SunTag, resultou em uma capacidade de ativação mais forte comparando com dCas9-VP64 fusão homólogos11 , 12. uma ativação transcricional melhorada foi obtida através da fusão de VP64, p65 e Rta (VPR), um domínio do transactivation do gama-herpesviruses, para o C-terminal da dCas913 (figura 1A). Desenvolveram-se sistemas semelhantes de CRISPR/dCas9 para a repressão de destino específico (figura 1B).
Repressão do gene endógeno pode ser conseguido com fusões repressor projetadas através de uma variedade de mecanismos (figura 1B). Foi demonstrado que sistemas CRISPR/dCas9, ligados ao repressor DBD (mesmo sem um domínio efetor/s), com eficiência podem silenciar a expressão de gene, enquanto amarrados a um promotor ou montante/jusante-TSS regiões3,6 ,14. Os efeitos na transcrição é causado por interferência estérica de ligação do fator de transcrição e processamento de RNA polimerase. No entanto, são necessárias abordagens mais abrangentes, como repressão do gene por estérico sozinho muitas vezes não é suficiente para silenciar robusto. O recente desenvolvimento da próxima geração de silenciadores baseado em sistemas CRISPR/dCas9 carregando domínios transcricional repressor (TRDs), modificadores de histona (H3-K9 di-/ tri-metilação, H3-K27 di-/ tri-metilação; H3-K36 di-/ tri-metilação, deacetilação de histona H3/H4) e metilação de DNA (CpG), levado à construção de ferramentas epigenéticas, permitindo mais robusto silenciar efeitos4,5,15,16, 17,18,19,20. Foi demonstrado que o recrutamento desses modificadores epigenéticas ao DNA pode levar à formação de cromatina mais condensada e fechada, que normalmente geram um mais potente silencioso resultado21,22. O domínio de silenciar mais comumente usado com DBDs é o caixa Krüppel-associado (CASCUDO)4,5. O recrutamento do fator foi demonstrado para corresponder com as alterações da cromatina; no entanto, os mecanismos destas modificações são ainda ser elucidado16,17,18. Recentemente, tem sido demonstrado que a localização do Siri CASCUDO para DNA pode promover a montagem do methyltransferase do histone SETDB1 e os complexos de NuRD histona deacetilação (HDAC), sugerindo a possibilidade de que estas interações mediam a formação de condensação da cromatina e transcriptional silencioso3,13. Como uma abordagem alternativa, domínios effector podem ser fundidos a DBDs para criar uma proteína de silenciamento epigenética personalizada. Este sistema diretamente catalisa repressivas marcas de DNA ou modificações do histone.
Recentemente, a utilização de sistemas CRISPR/dCas9 sintéticos amarrados à enzima DNMT3A tem foi realocada para desativação transcricional. Dnmt3a catalisa a metilação do DNA que exerce repressão transcriptional em toda a formação de heterocromatina em promotores de genes endógenos e outras regiões reguladoras (figura 1B)18,20. McDonald et al.18 e Vojta et al20 foram os primeiros autores para relatar que a metilação do DNA pode ser usada para Epigenoma-silenciamento ou repressão, demonstrando que o sistema de fusão de dCas9-DNMT3A entregue em plasmídeo potente pode melhorar metilação de citosina em torno do TSS18,20. McDonald ' s e colegas demonstraram que o emprego da estratégia pode resultar em uma redução significativa (cerca de 40%) em um gene supressor de tumor, os níveis de mRNA de CDKN2A 18. Da mesma forma, visando a região unmethylated promotor dos genes BACH ou IL6ST mostra aumentada metilação de CpG que tem sido correlacionada com a expressão de gene20redução dupla. Nosso laboratório tem recentemente realocado o uso de metilação do DNA para atenuar os resultados patológicos de SNCA superexpressão (Figura 2)23. A estratégia baseia-se na seletiva do realce na metilação do DNA dentro da região de intron 1 SNCA , como anteriormente foi relatado para ser hypomethylated em PD e demência com Lewy (DLB) de corpos cérebros24,25, 26. Esta hypomethylation tem sido associada a SNCA superexpressão, oferecendo assim um alvo atraente para a intervenção terapêutica de27,24,28. Recentemente mostramos um baixo nível de metilação do DNA na SNCA intron 1 na região de hiPSC-derivado dopaminérgico NPCs obtidos de um paciente de PD com o SNCA triplicação23. A vantagem deste modelo experimental é que os NPCs podem ser robustamente propagados na cultura ou ainda mais diferenciados em neurônios maduros, permitindo uma triagem eficiente identificar fatores genéticos que medeiam fenótipos celulares, incluindo oxidativo stress e apoptose29. Além disso, este modelo sistema permite aos cientistas recapitular os eventos do desenvolvimento que ocorreram antes do aparecimento de sintomas em pacientes. Além disso, hiPSC-derivado de NPCs representam uma ótima ferramenta para testar as vias celulares e moleculares associadas a expressão gênica. Importante, NPCs derivado de hiPSC, combinados com estado-da-arte tecnologia CRISPR/Cas9-Epigenoma podem facilitar muito o desenvolvimento de "drogas de última geração" para muitas doenças neurodegenerativas.
Para reduzir níveis patológicos de expressão SNCA, recentemente desenvolvemos um sistema baseado em lentivirus carregando uma proteína de fusão de dCas9-DNMT3A e gRNA para especificamente alvo CpG do methylation dentro o SNCA intron 1 (Figura 2A)23. Este protocolo irá descrever design vetor de Lentivirus (LV) e produção em detalhe. VLS representam um meio eficaz de fornecer componentes CRISPR/dCas9 por várias razões, nomeadamente (i) sua capacidade de transportar DNA volumoso insere, (ii) uma alta eficiência de transducing uma ampla variedade de células, incluindo células de divisão e nondividing30 e (iii) sua habilidade de induzir respostas imunogênicas e citotóxicas mínimas. Recentemente, aplicamos o sistema LV para os neurônios dopaminérgicos hiPSC-derivado de um paciente com a triplicação do locus a SNCA e demonstrou o potencial terapêutico de LVs para a entrega da edição de Epigenoma metilação ferramentas23 ( Figura 2B). Com efeito, um sistema de LV-gRNA/dCas9-DNMT3A ocasiona um aumento significativo na metilação do DNA na região do intron 1 SNCA . Este aumento corresponde com a redução dos níveis de SNCA mRNA e proteína23. Além disso, a SNCA downregulation resgata fenótipos relacionados com PD na SNCA triplicação/hiPSC-derivado dopaminérgico neurônios (por exemplo, mitocondrial ROS produção e célula viabilidade)23. Importante, temos demonstrado que a redução na expressão SNCA pelo sistema LV-gRNA-dCas9-DMNT3A é capaz de reverter os fenótipos que são característicos para os neurônios dopaminérgicos hiPSC-derivado de um paciente de PD que carregava a SNCA triplicação, tais como mitocondrial ROS produção e célula viabilidade23. O objetivo do presente protocolo é 1) para delinear o protocolo de produção e concentração de uma plataforma otimizada de LV para gerar alta-tittered virais preparações e 2) para descrever a diferenciação de hiPSCs em NPCs padronizada para tornar-se maduro dopaminergic os neurônios31,32 e a caracterização dos níveis de metilação da região alvo dentro SNCA intron 1.
Particulas de Lentivirus plataformas tem uma grande vantagem sobre a plataforma mais popular do vetor, ou seja adeno-associado vetores (AAVs), que é capacidade a ex de acomodar maior inserções genética33,34. AAVs podem ser gerados em rendimentos significativamente mais elevados, mas possuem uma capacidade baixa de empacotamento (< 4,8 kb), comprometer a sua utilização para fornecimento de sistemas de CRISPR/Cas9 all-in-one. Assim, parece que o LVs seria a plataforma de escolha nas aplicações envolvidas no fornecimento de ferramentas CRISPR/dCas9. Portanto, o protocolo descrito aqui vai ser uma ferramenta valiosa para pesquisadores desejando entregar efetivamente Epigenoma-editando componentes para as células e órgãos. O protocolo mais descreve a estratégia para aumentar as capacidades de produção e expressão dos vetores através de uma modificação na CEI dos elementos dentro do vetor expressão gaveta30,35. A estratégia é baseada no romance o sistema desenvolvido e estudado em nosso laboratório e destaca sua capacidade de produzir partículas virais no intervalo de 1010 unidades virais (VU) /mL30,35.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. sistema Design e produção de vírus
- Construção e design de plasmídeo
Nota: A construção de um vetor de LV-gRNA-dCas9-DNMT3A all-in-one é realizada por meio de uma produção- e otimizados para expressão expressão cassette, publicado por Ortinski et al.30. A gaveta de vetor carrega uma repetição do site reconhecimento do fator de transcrição Sp1 e uma estado-da-arte exclusão dentro o untranslated (U3') região de um terminal 3'-longa repetição (LTR) (Figura 2A)30,36. A espinha dorsal de vetor foi encontrada para ser eficaz na entrega e expressando CRISPR/Cas930,35.- Obter a versão deactivate (mortos) da SpCas9 (dCas9) através do mutagenesis local-dirigido (dados não mostrados). Substitua o clone abrigando mutações D10A e H840A em HNH e RuvC domínios catalíticos da enzima, respectivamente, com o ativo Cas9 pBK30129 através do intercâmbio entre AgeI-BamHI fragmentos (Figura 3).
- Derivam do domínio catalítico DNMT3A pdCas9-DNMT3A-eGFP (ver Tabela de materiais) amplificando a DNMT3A parcela BamHI-429/R 5 '- GAGCGGATCCCCCTCCCG - 3' BamHI-429/L 5' - CTCTCCACTGCCGGATCCGG - 3' (Figura 3). Para amplificar a região que contém DNMT3A, use as seguintes condições: (1) 95 ° C por 60 s, (2) 95 ° C, durante 10 s, (3) 60 ° C durante 20 s, (4) 68 ° C por 60 s. repetir condições 2 a 4 x 30. Para a extensão final, use a 68 ° C por 3 min e manter a 4 ° C.
- Clone o fragmento DNMT3A, digerido por uma enzima de restrição de BamHI, no local de BamHI do vetor pBK301 modificado carregando dCas9. Verifique se a clonagem por Sanger direto sequenciamento. Observe que o plasmídeo resultou abriga dCas9-DNMT3A-p2a-puromicina transgene. O plasmídeo expressa gRNA andaimes do humano U6 promotor (Figura 3).
- Substitua o gene repórter puromicina com proteína verde fluorescente (GFP) para criar a dCas9-DNMT3A-p2a-GFP. Digeri o plasmídeo dCas9-DNMT3A-p2a-Puro com FseI. Purifica o fragmento de vetor usando um método de purificação do gel. Preparar a inserção por digestão de pBK201a (pLenti-GFP) com FseI. Clone o fragmento de FseI para o vetor. O resultado do plasmídeo pBK539 abriga dCas9-DNMT3A-p2a-GFP transgene (Figura 3).
- Cultivo de células HEK-293T e chapear pilhas para transfeccao
Nota: Rim embrionário humano 293T (HEK-293T) as células são cultivadas em alta-glicose completo Dulbecco modificada do meio da águia (DMEM; 10% de soro de bovino bezerro, 1 x antibiótico antimicótico, piruvato de sódio 1 x, 1 x não essenciais aminoácidos, 2 mM L-glutamina) a 37 ° C com 5% CO 2. para a reprodutibilidade do protocolo, é recomendável testar o soro de vitelo, quando se muda para um novo lote/lote. Até seis 15 cm placas são necessários para a produção de Lentivirus.- Use células de baixo-passagem para iniciar uma nova cultura (inferior a passagem 20). Uma vez que as células alcançar 90% – 95% confluência, aspirar a mídia e lavá-lo suavemente com estéril 1x tampão fosfato salino (PBS).
- Adicionar 2 mL de tripsina-EDTA (0.05%) e -incubar a 37 ° C por 3 – 5 min. Para desativar o reagente de dissociação, adicione 8 mL de DMEM completa de alta-glicose e pipeta 10 x x-15 com uma pipeta sorológica 10ml para criar uma célula única suspensão de 4 x 106 células/mL.
- Para os transfections, revestimento placas de 15cm com 0,2% de gelatina. Adicionar 22,5 mL de meio de alto-glicose e as células da semente adicionando 2,5 mL da suspensão de células (total ~ 1 x 107 células/placa). Incube as placas a 37 ° C com 5% CO2 até 70%-80% confluência é alcançada.
- Transfecção de células HEK-293T
- Prepare 2 x solução tampão BES BBS e 1 M CaCl2, de acordo com Tiago e Kantor35. Filtrar as soluções, passando-os através de um filtro de 0,22 µm e armazená-los em 4 ° C. A mistura de transfeccao tem de ser limpo antes de sua adição para as células. Se a mistura torna-se nublado durante a incubação, prepare-se fresco 2 x BBS (pH = 6,95).
- Para preparar a mistura do plasmídeo, use os quatro Plasmideos conforme listado (a seguinte mistura é suficiente para uma placa de 15 cm): 37,5 µ g do vetor CRISPR/dCas9-transferência (pBK492 [DNMT3A-Puro-n-gRNA] ou pBK539 [DNMT3A-GFP-n-gRNA]); 25 µ g de pBK240 (psPAX2); 12,5 µ g de pMD2.G; 6,25 µ g de pRSV-rev (Figura 4A). Calcular o volume dos Plasmideos baseada as concentrações e adicionar as quantidades exigidas em um tubo cônico de 15 mL. Adicione 312,5 µ l de 1 M CaCl2 e trazer o volume final de 1,25 mL, usando estéril dd-H2O. delicadamente adicione 1,25 mL de solução de x BBS 2 enquanto num Vortex mistura. Incube durante 30 min à temperatura ambiente. As células estão prontas para transfeccao, uma vez que eles são 70%-80% de Confluencia.
- Aspirar a mídia e substituí-lo com 22,5 mL de acabadas alta-glicose DMEM sem soro. Adicione 2,5 mL da mistura de transfeccao gota a gota para cada placa de 15 cm. Agite as placas e incubar a 37 ° C com 5% de CO2 para 2-3 h.
- Depois de 3 h, adicionar 2,5 mL (10%) de soro por placa e incubar durante uma noite a 37 ° C com 5% de CO2.
- No dia 1 após o transfeccao, observar as células para garantir que não há nenhum ou morte celular mínima e que as células formavam uma cultura confluente (100%).
- Altere a mídia adicionando 25 mL de soro DMEM e 10% alta-glicose recentemente preparado para cada placa.
- Incube a 37 ° C com 5% de CO2 por 48 h.
- Colheita do vírus
- Recolher o sobrenadante de todas as células transfectadas e piscina-los em tubos de Erlenmeyer de 50 mL. Centrifugar a 400-450 x g por 10 min. filtrar o sobrenadante através de uma unidade de filtro de vácuo 0,45 µm. Após filtração, o sobrenadante pode ser mantido a 4 ° C para armazenamento de curto prazo (até 4 dias). Para armazenamento a longo prazo, preparar alíquotas e armazená-los a-80 ° C.
Nota: As preparações nonconcentrated virais são esperadas ser ~ 2 x 107 3 x 107 vu/ml (consulte a seção 1.5 para determinação do título). É altamente recomendável para preparar alíquotas de uso único, uma vez que vários ciclos de gelo-degelo irão resultar em uma perda de 10%-20% em títulos funcionais.
- Recolher o sobrenadante de todas as células transfectadas e piscina-los em tubos de Erlenmeyer de 50 mL. Centrifugar a 400-450 x g por 10 min. filtrar o sobrenadante através de uma unidade de filtro de vácuo 0,45 µm. Após filtração, o sobrenadante pode ser mantido a 4 ° C para armazenamento de curto prazo (até 4 dias). Para armazenamento a longo prazo, preparar alíquotas e armazená-los a-80 ° C.
- Concentração de partículas virais
Nota: Para a purificação, um método duplo-sacarose em duas fases envolvendo uma etapa de gradiente de sacarose e um passo de almofada de sacarose é realizada (Figura 4B).- Para criar um gradiente de sacarose, preparar os tubos de ultracentrifugação cónico na seguinte ordem: 0,5 mL de 70% de sacarose em 1X PBS, 0,5 mL de 60% de sacarose em DMEM, 1 mL de 30% de sacarose em DMEM e 2 mL de 20% de sacarose em 1X PBS.
- Cuidadosamente, adicione o sobrenadante, recolhido de acordo com a seção 1.4, o gradiente. Desde que o volume total coletado de quatro placas de 15cm é 100 mL, use seis tubos de ultracentrifugação para processar o sobrenadante viral.
- Distribua igualmente o sobrenadante viral entre cada tubo de ultracentrifugação. Para evitar a ruptura do tubo durante a centrifugação, encha os tubos de ultracentrifugação a pelo menos três quarto da sua capacidade de volume total. Equilibre os tubos com PBS 1x. Centrifugar as amostras a 70.000 x g durante 2 h a 17 ° C.
Nota: Para manter a camada de sacarose durante as etapas de aceleração e desaceleração, permitir que o se lentamente acelerar e desacelerar o rotor de 0 a 200 x g e 200-0 x g durante o primeiro e o último 3 min do spin, respectivamente. - Delicadamente colete frações de 30 – 60% de sacarose em tubos limpos (Figura 4B). Adicione 1 x acima (fria) PBS para 100 mL de volume total. Misture pipetando várias vezes.
- Cuidadosamente, estratificar a preparação viral em uma almofada de sacarose adicionando 4 mL de sacarose 20% (em 1X PBS) para o tubo. Continue por pipetagem ~ 20-25 mL da solução de viral por cada tubo. Encha os tubos com PBS 1x se o volume do seu conteúdo é menos de três quartos por tubo. Cuidadosamente, equilibre os tubos. Centrifugar a 70.000 x g por 2 h a 17 ° C. Esvazie o sobrenadante e inverter os tubos em papel toalha para permitir que o restante líquido escorrer.
- Retire todo o líquido cautelosamente aspirando o líquido restante. Observe que, neste passo, Pelotas, que contêm o vírus são pouco visíveis como pequenos pontos translúcidos. Adicione 70 µ l de 1X PBS para o primeiro tubo para Ressuspender o precipitado. Cuidadosamente, pipetar a suspensão e transferi-lo para o próximo tubo até todas as pelotas são resuspended.
- Lave os tubos com um adicional 50 µ l de 1X PBS e mistura como antes. Observe que, nesta etapa, o volume da suspensão final ~ 120 µ l e aparece ligeiramente leitoso. Para obter uma suspensão clara, prosseguir-se com uma centrifugação s 60 a 10.000 x g. Transferir o sobrenadante para um tubo novo, fazer 5 alíquotas µ l e armazená-los a-80 ° C.
Nota: Preparações de vetor de Lentivirus são sensíveis os ciclos repetidos de congelamento e degelo. Além disso, sugere-se que as etapas restantes são feitas em cultura de tecidos de contenção ou em designadas áreas qualificadas em termos de ser em níveis adequados de normas de biossegurança (Figura 4B).
- Quantificação da concentração viral
Nota: A estimativa da concentração viral é executada usando o método de ensaio (ELISA) de enzima-lig da imunoabsorção p24 (p24amordaçar ELISA) e de acordo com o National Institutes of Health (NIH) de vacina contra AIDS de protocolo para o HIV-1 p24 Antigen capturar ensaio com pequenas modificações.- Use 200 µ l de 0,05% Tween 20 em frio 1X PBS (PBS-T) para lavar os poços de uma placa de 96 poços 3 x.
- Para revestir a placa, use 100 µ l de anticorpo monoclonal anti-p24 diluído na 1:1,500 em 1X PBS. Incubar a placa durante a noite a 4 ° C.
- Preparar o reagente de bloqueio (1% albumina de soro bovino [BSA] em 1X PBS) e adicionar 200 µ l de cada poço para evitar a ligação inespecífica. Use 200 µ l de PBS-T para lavar o bem 3x pelo menos 1 h à temperatura ambiente.
- Prosseguir com a preparação da amostra. Ao trabalhar com preparações de vetor concentrado, dilua o vetor em 1: 100 usando 1 µ l da amostra, 89 µ l de dd-H20 e 10 µ l de Triton X-100 (em uma concentração final de 10%). Para preparações nonconcentrated, dilua as amostras às 01:10.
- Obter padrões de HIV-1 usando uma diluição serial dupla (a concentração inicial é de 5 ng/mL).
- Diluir as amostras de concentrado (preparadas na etapa 1.6.4) em RPMI 1640 suplementado com 0,2% Tween 20 e 1% de BSA para obter 01:10, 000, 01:50, 000 e diluições de 1:250,000. Da mesma forma, diluir as amostras nonconcentrated (preparadas na etapa 1.6.4) em RPMI 1640 suplementado com 0,2% Tween 20 e 1% de BSA para estabelecer a 1: 500, 1:2, 500 e diluições de 1:12,500.
- Adicione as amostras e as normas sobre a placa em triplica. Incubar durante uma noite a 4 ° C.
- No dia seguinte, lavar os poços 6 x.
- Adicione 100 µ l de anticorpo anti-p24 de policlonal de coelho, diluído em 1:1,000 em RPMI 1640, 10% de soro fetal bovino (FBS), 0,25% de BSA e soro normal do rato de 2% (NMS) e incubar a 37 ° C por 4 h.
- Lavar os poços 6 x. Adicionar a peroxidase de rábano de anticabra coelho IgG diluído na 1:10,000 em RPMI 1640 suplementado com 5% de soro de cabra normal, 2% NMS, BSA de 0,25% e 0,01% Tween 20. Incube a 37 ° C, durante 1 h.
- Lavar os poços 6 x. Adicione o substrato de peroxidase TMB e incubar a temperatura ambiente por 15 min.
- Para parar a reação, adicione 100 µ l de 1 N HCl. Em um leitor de microplacas, medir a absorvância a 450 nm.
- Medição da intensidade de repórter fluorescentes
- Use a suspensão viral para obter uma diluição serial 10 vezes (de 10-1 a 10-5) em PBS 1x.
- Placa de 5 x 105 HEK-293T células em cada poço de uma placa de 6. Aplicar 10 µ l de cada diluição viral para as células e incubar a 37 ° C com 5% de CO2 por 48 h.
- Prossiga para a célula de fluorescência-ativado classificação análise (FACS) da seguinte forma: separar as células, adicionando 200 µ l de solução de tripsina-EDTA 0,05%. Incubar as células a 37 ° C por 5 min e Resuspenda-los em 2 mL do meio DMEM (com soro). Coletar as amostras em um tubo cônico de 15 mL e centrifugar a 400 x g a 4 ° C. Resuspenda o pellet em 500 µ l de PBS 1x frio.
- Corrigir as células adicionando 500 µ l de paraformaldeído 4% (PFA) e incubar durante 10 minutos à temperatura ambiente.
- Centrifugar a 400 x g a 4 ° C e resuspenda o pellet em 1 mL de 1X PBS. Analise a expressão de GFP, usando um instrumento de FACS, conforme descrito em Ortinski et al.30. Para determinar a concentração de vírus funcional, use a seguinte fórmula.

Aqui,
TG = número de células GFP-positivo;
TN = número total de células;
N = número total de células transduzidas;
V = volume utilizado para transdução (em microlitros).
- Contagem de células GFP-positivo
Nota: Determine a multiplicidade de infecção (MOI) que é utilizada por transdução. Uma ampla gama de MOIs de teste (de MOI = 1 para MOI = 10).- Sementes de 3 x 105 4 x 105 HEK-293T células por cada poço de uma placa de 6.
- Quando atingem as células > 80% confluência, transduce-los com o vetor para o MOI de interesse.
- Incubar a 37 ° C com 5% de CO2e monitorar o sinal GFP nas células de 1 a 7 dias.
- Conte o número de células GFP-positivo. Empregar um microscópio fluorescente (plano objetiva de 4x, 0.1 N.A., ampliação de 40 x) usando um filtro GFP (comprimento de onda de excitação = 470 nm, comprimento de onda de emissão = 525 nm). Use untransduced células para definir a população de controle de células GFP-negativo.
- Emprega a seguinte fórmula para determinar o título funcional do vírus.
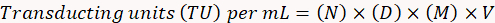
Aqui,
N = número de células GFP-positivo;
D = fator de diluição;
M = fator de ampliação;
V = volume de vírus usada por transdução.
Nota: Calcular os resultados seguindo este exemplo: para 10 células GFP-positivo (N) contadas a uma diluição (D) de 10-4 (01:10, 000) em ampliação de 20 x (M) em uma amostra de 10 µ l (V), o TU por mililitro será (10 x 104) x (20) x (10) x (100) = 2 x 108 vu/mL.
2. diferenciação de células progenitoras neurais de dopaminergic
- Cultivo de hiPSCs
Nota: hiPSCs de um paciente com a triplicação do locus SNCA, ND34391, foram obtidos do Instituto Nacional de Disorders Neurological e catálogo de Stroke (NINDS) (ver tabela de materiais).- Cultura hiPSCs sob condições de alimentador independente em meio de cultura de ESC-iPSC alimentador-livre (veja a Tabela de materiais) na membrana hESC qualificado matriz básica (BMM)-revestido de placas (ver Tabela de materiais). Lave as colônias confluentes com 1 mL de DMEM/F12, adicionar 1 mL do reagente de dissociação (ver Tabela de materiais) e incube por 3 min à temperatura ambiente.
- Aspirar o reagente de dissociação e adicionar 1 mL de meio de cultura de ESC-iPSC alimentador-livre.
- Raspar a placa usando um levantador de célula e ressuspender as colônias em 11 mL de meio de cultura de ESC-iPSC alimentador-livre por pipetagem x-5 4 x usando pipetas de borosilicato.
- 2 mL de suspensão de colônia em placas BMM-revestido da placa e coloque a placa a 37 ° C com 5% de CO2. Realizar uma mudança diária média e dividir as células em 5-7 dias.
- Diferenciação em células progenitoras neurais de dopaminergic
Nota: A diferenciação de hiPSCs em células progenitoras neurais de dopaminérgico (MD NPCs) é realizada utilizando um protocolo de indução neural disponíveis comercialmente médio segundo as instruções dos fabricantes, com pequenas modificações (de32 31, consulte Tabela de materiais). O primeiro dia da diferenciação é considerado como dia 0. HiPSCs de alta qualidade são necessários para a diferenciação neural eficiente. A indução de NPCs MD é performedusing um corpo do embryoid (EB)-com base em protocolo.- Antes de começar a diferenciação de hiPSCs, preparar uma placa de cultura de microplacas (ver Tabela de materiais) de acordo com as instruções do seu fabricante.
- Depois de preparar a placa de cultura de microplacas, adicionar 1 mL de meio de indução neural (NIM; ver Tabela de materiais) suplementado com 10 µM Y-27632.
- Reserve a placa até estar pronto para uso.
- Lavar hiPSCs com DMEM/F12, adicionar 1 mL de solução de desprendimento de célula (ver Tabela de materiais) e incube por 5 min a 37 ° C com 5% de CO2.
- Ressuspender as células únicas em DMEM/F12 e centrifugá-los a 300 x g durante 5 min.
- Cuidadosamente, aspirar o sobrenadante e ressuspender as células em NIM + 10 µM Y-27632 para obter uma concentração final de 3 x 106 células/mL.
- Adicione 1 mL de suspensão de célula única para um único poço da placa de cultura de microplacas e centrifugar a placa a 100 x g durante 3 min.
- Examine a placa sob o microscópio para garantir uma distribuição uniforme das células entre os micropoços e incubar a 37 ° C com 5% CO2células.
- Nos dias 1 – 4, realizar uma alteração de média diária parcial.
- Utilizando uma micropipeta de 1 mL, 1,5 mL do meio de remover e descartar. Lentamente, adicione 1,5 mL de NIM fresca sem Y-27632.
- Repita a etapa 2.2.10 até dia 4.
- No dia 5, revestir um poço de uma placa de 6 com BMM.
- Coloque um filtro reversível 37 µm (ver Tabela de materiais) em cima de um tubo cónico de 50 mL (resíduos). Aponte a seta do filtro reversível para cima.
- Remova o meio da placa de cultura de microplacas sem perturbar o EBs formado.
- Adicionar 1 mL de DMEM/F12 e prontamente coletar o EBs com a pipeta de borosilicato e filtrá-los através do filtro.
- Repita a etapa 2.2.15 até todos os EBs são removidos da placa de cultura de microplacas.
- Inverta a peneira sobre um novo tubo cónico de 50 mL e adicionar 2 mL de NIM para coletar todo o EBs.
- Placa de 2 mL de suspensão de EB num único poço da placa BMM-revestido com uma pipeta de borosilicato. Incube a EBs a 37 ° C com 5% de CO2.
- No dia 6, prepare-se 2 mL de NIM + 200 ng/mL SHH (ver Tabela de materiais) e realizar uma mudança de média diária.
- No dia 8, examine a porcentagem de indução neuronal.
- Contar todo ligado EBs e, especificamente, determinar o número de cada individual EB que é preenchido com rosetas neurais. Quantificar a indução de Roseta neural usando a seguinte fórmula.
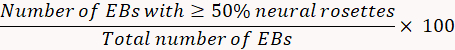
Nota: Se a indução neural é < 75%, seleção de Roseta neural pode ser ineficiente. - No dia 12, preparar 250 mL do meio de N2B27 com 119 mL de meio neurobasal, 119 mL de DMEM/F12 médio, 2,5 mL de GlutaMAX, 2,5 mL de timina, 2,5 mL de suplemento de N2, 5 mL de B27 sem vitamina A, 250 μL de gentamicina (50 mg/mL) e 19. 66 μL de BSA (7 mg/mL).
- Para preparar 50 mL de meio de N2B27 completo, adicione 3 μM CHIR99021, 2 μM SB431542, 20 ng/mL bFGF, 20 ng/mL EGF e 200 ng/mL SHH.
Nota: É importante preparar o concluído médio bem antes do uso. - Aspire o médio dos poços que contêm rosetas neurais e lavar com 1 mL de DMEM/F12.
- Anúncio 1 mL de reagente de selecção de Roseta neural (ver Tabela de materiais) e incubar a 37 ° C com 5% de CO2 por 1h.
- Remover o reagente de selecção e, usando uma pipeta de 1 mL, visam diretamente os clusters de Roseta.
- Adicionar a suspensão a um tubo cônico de 15 mL e repita as etapas 2.2.25 e 2.2.26 até a maioria dos clusters de Roseta neural foram recolhidas.
Nota: Para evitar a contaminação com tipos de células nonneuronal, não overselect. - Centrifugar a suspensão de Roseta 350 x g durante 5 min. Aspire o sobrenadante e ressuspender as rosetas neurais em N2B27 + 200 ng/mL SHH. Adicionar a suspensão de Roseta neural para um poço de BMM-revestido e incubar a placa a 37 ° C com 5% de CO2.
- Nos dias 13-17, execute uma mudança diária média usando concluído N2B27 médio. Passagem das células quando as culturas são confluente 80%-90%.
- Para dividir as células, prepare um prato de BMM-revestido.
- Lavar as células com 1 mL de DMEM/F12, Aspire o meio e adicione 1 mL do reagente de dissociação (ver Tabela de materiais).
- Incubar durante 5 min a 37 ° C, adicionar 1 mL de DMEM/F12 e desalojar anexadas células pipetando para cima e para baixo. Recolha a suspensão NPC em um tubo cônico de 15 mL. Centrifugar x g por 5 min.
- Aspirar o sobrenadante e ressuspender as células em 1 mL de N2B27 completa + 200 ng/mL SHH.
- Contar as células da placa-los em uma densidade de 1,25 x 105 células/cm2e incubar a 37 ° C com 5% CO2células.
- Mude o meio de todos os outros dias, usando N2B27 completa + 200 ng/mL SHH.
Nota: A esta passagem, NPCs são considerados como passagem (P) 0. SHH pode ser retirado do meio N2B27 em P2. - Passagem das células quando atingem a confluência de 80%-90%.
- Nesta fase, confirmam que as células expressam marcadores Nestin e FoxA2 usando imunocitoquímica e qPCR. Este protocolo leva à geração de células de duplo-positivo de 85% para os marcadores Nestin e FoxA2.
- Para as células de passagem, repita os passos 2.2.31–2.2.36.
- Congele as células, a partir de passagem P2. Para o congelamento das células, repita as etapas 2.2.31–2.2.36 e resuspenda o pellet de células em 2 x 106 a 4 x 106 células/mL usando fria progenitoras neurais congelamento médio (ver Tabela de materiais).
- Transferir 1 mL da suspensão de célula para cada cryovial e congelar as células usando um padrão sistema de arrefecimento lento-taxa-controlada. Para armazenamento a longo prazo, manter as células em nitrogênio líquido.
- Descongelamento de NPCs MD
- Preparar um prato de BMM-revestido e quentes N2B27 completa. Adicione 10 mL de DMEM/F12 quente para um tubo cônico de 15 mL. Coloque um cryovial em um bloco de calor de 37 ° C por 2 min.
- Transferi as células do cryovial para o tubo contendo DMEM/F12. Centrifugar x g por 5 min.
- Aspire o sobrenadante e ressuspender as células em 2 mL de N2B27 adicionar a suspensão de células para um poço da placa BMM-revestido. Incube a 37 ° C com 5% CO2células.
- Preparar um prato de BMM-revestido e quentes N2B27 completa. Adicione 10 mL de DMEM/F12 quente para um tubo cônico de 15 mL. Coloque um cryovial em um bloco de calor de 37 ° C por 2 min.
3. transdução da MD NPCs e a análise das mudanças de metilação
-
Transdução de NPCs MD
- Transduce MD NPCs na confluência de 70% com vetores de LV-gRNA/dCas9-DNMT3A em MOI = 2. Substitua o posttransduction médio 16 h de N2B27.
- Adicione N2B27 com 5 puromicina µ g/mL, 48 h após a transdução. Cultura das células por 3 semanas em N2B27 além de puromicina para obter as linhas MD NPC estáveis. As células estão prontas para aplicações a jusante (DNA, RNA, análises de proteínas, Caracterização fenotípica23, congelamento e passagem).
-
Caracterização do perfil de metilação de intrão SNCA 1
- Extrair o DNA de cada célula estàvel transduzida kit de linha usando uma extração de DNA (ver Tabela de materiais).
- Uso 800 ng de DNA para executar uma conversão de bissulfito, usando um comercialmente disponível kit (veja a Tabela de materiais). Após a conversão de bissulfito, Eluir o DNA de bissulfito-convertido a 20 ng / µ l.
-
PCR para a análise de pyrosequencing
- Prepare a mistura de mestre do PCR em um tubo de nuclease-livre. Para cada reação, use 0,4 µ l de primer reverso (10 µM), 0,4 µ l de primer para a frente (10 µM), 1,6 µ l de MgCl2 (25 mM), 2 µ l de 10 x CoralLoad concentrado, 10 µ l de mistura de mestre 2 x PCR, 4 µ l de 5 x Q-solução, 1 µ l de DNA e 0,6 µ l de água livre de nuclease.
- Transferir a placa de reação para um thermocycler e realizar PCR usando as seguintes condições: 95 ° C por 15 min, 50 ciclos de 94 ° C por 30 s, 56 ° C durante 30 s e 72 ° C por 30 s, com uma etapa de extensão de 10min final a 72 ° C. Primers utilizados para a pyrosequencing de intrão SNCA 1 estão listados na tabela 1, Figura 7Ae complementar a Figura 1.
- Após a amplificação, visualize os amplicons usando 2 µ l de produto PCR com brometo de etídio coloração, seguir eletroforese em gel de agarose.
- Pyrosequencing ensaios são validados usando misturas de unmethylated (U) e misturado (M) DNAs de bissulfito-convertido nas relações seguintes, nomeadamente 100U:0M, 75U:25M, 50U:50M, 25U:75M e 0U:100M (ver Tabela de materiais).
- Conduta pyrosequencing usando reagentes pyrosequencing (ver Tabela de materiais) e calcular os valores de metilação para cada site de CpG usando pyrosequencing software. Para um protocolo de pyrosequencing detalhada, consulte Bassil et al.37.
Access restricted. Please log in or start a trial to view this content.
Resultados
Validação dos títulos de produção dos vetores LV-dCas9-DNMT3A-GFP/Puro em comparação com a contrapartida GFP ingênuo
Realizamos p24amordaçar ELISA para comparar entre uma concentração física de LV-dCas9-DNMT3A-GFP/Puro com as contrapartes GFP/Puro ingênuo. Resultados representativos, apresentados na Figura 5A, demonstram que o rendimento físico dos vetores, ...
Access restricted. Please log in or start a trial to view this content.
Discussão
VLS começaram a emergir como o veículo de escolha para edição Epigenoma, especialmente no contexto de doenças genéticas, principalmente devido à sua capacidade de (i) acomodar grandes cargas de DNA e (ii) transduce eficientemente uma grande variedade de dividir e células nondividing. A eficácia de grandes embalagens dos VLS é especialmente benéfica para as aplicações que envolvem embalagens dos sistemas CRISPR/dCas9 que são de grandes dimensões. Nesta perspectiva, LVs representam a plataforma de escolha pa...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Duke University apresentou um pedido provisório de patente relacionado com este estudo.
Agradecimentos
Este trabalho foi financiado em parte pelo prêmio de desenvolvimento de Neurotecnologia Kahn (de The O.C.) e o nacional institutos de saúde nacionais Instituto de doenças neurológicas e Stroke (NINDS/NIH) (R01 NS085011 de O.C.).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| 0.22 μM filter unit, 1 L | Corning | 430513 | |
| 0.45-μm filter unit, 500 mL | Corning | 430773 | |
| 100 mm TC-Treated Culture Dish | Corning | 430167 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 50 mL conical centrifuge tubes | Corning | 430291 | |
| 6-well plates | Corning | 3516 | |
| Aggrewell 800 | StemCell Technologies | 34811 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| BD FACS | Becton Dickinson | 338960 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| Eppendorf Cell Imaging Slides | Eppendorf | 30742060 | |
| High-binding 96-well plates | Corning | 3366 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Reversible Strainer | StemCell Technologies | 27215 | |
| SW32Ti rotor | Beckman Coulter | 369650 | |
| VWR® Disposable Serological Pipets, Glass, Nonpyrogenic | VWR | 93000-694 | |
| VWR® Vacuum Filtration Systems | VWR | 89220-694 | |
| xMark™ Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| Accutase | StemCell Technologies | 7920 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| Anti-FOXA2 Antibody | Abcam | Ab60721 | |
| Anti-Nestin Antibody | Abcam | Ab18102 | |
| Antibiotic-antimycotic solution, 100x | Sigma Aldrich | A5955-100ML | |
| B-27 Supplement (50x), minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| BES | Sigma Aldrich | B9879 - BES | |
| Bovine Albumin Fraction V (7.5% solution) | Thermo Fisher Scientific | 15260037 | |
| CHIR99021 | StemCell Technologies | 72052 | |
| Corning Matrigel hESC-Qualified Matrix | Corning | 08-774-552 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| DMEM-F12 | Lonza | 12-719 | |
| DMEM, high glucose media | Gibco | 11965 | |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | |
| EpiTect PCR Control DNA Set | Qiagen | 596945 | |
| EZ DNA Methylation Kit | Zymo Research | D5001 | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Gentamicin | Thermo Fisher Scientific | 15750078 | |
| Gentle Cell Dissociation Reagent | stemCell Technologies | 7174 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Human Recombinant bFGF | StemCell Technologies | 78003 | |
| Human Recombinant EGF | StemCell Technologies | 78006 | |
| Human Recombinant Shh (C24II) | StemCell Technologies | 78065 | |
| MEM Non-Essential Amino Acids Solution (100x) | Thermo Fisher Scientific | 11140050 | |
| mTeSR1 | StemCell Technologies | 85850 | |
| N-2 Supplement (100x) | Thermo Fisher Scientific | 17502001 | |
| Neurobasal Medium | Thermo Fisher Scientific | 21103049 | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| PyroMark PCR Kit | Qiagen | 978703 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| SB431542 | StemCell Technologies | 72232 | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| STEMdiff Neural Induction Medium | StemCell Technologies | 5835 | |
| STEMdiff Neural Progenitor Freezing Medium | StemCell Technologies | 5838 | |
| TaqMan Assay FOXA2 | Thermo Fisher Scientific | Hs00232764 | |
| TaqMan Assay GAPDH | Thermo Fisher Scientific | Hs99999905 | |
| TaqMan Assay Nestin | Thermo Fisher Scientific | Hs04187831 | |
| TaqMan Assay OCT4 | Thermo Fisher Scientific | Hs04260367 | |
| TaqMan Assay PPIA | Thermo Fisher Scientific | Hs99999904 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| Y27632 | StemCell Technologies | 72302 | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Goat serum, Sterile, 10 mL | Sigma | G9023 | Working concentration 1:1000 |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Normal mouse serum, Sterile, 500 mL | Equitech-Bio | SM30-0500 | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| pMD2.G | Addgene | 12253 | |
| pRSV-Rev | Addgene | 52961 | |
| psPAX2 | Addgene | 12259 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsmBI | New England Biolabs | R0580S | |
| BsrGI | New England Biolabs | R0575S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
Referências
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Gaj, T., Gersbach, C. A., Barbas, C. F. 3rd ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology. 31 (7), 397-405 (2013).
- Thakore, P. I., Black, J. B., Hilton, I. B., Gersbach, C. A. Editing the epigenome: technologies for programmable transcription and epigenetic modulation. Nature Methods. 13 (2), 127-137 (2016).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Gilbert, L. A., et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159 (3), 647-661 (2014).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Chavez, A., et al. Highly efficient Cas9-mediated transcriptional programming. Nature Methods. 12 (4), 326-328 (2015).
- Horlbeck, M. A., et al. Nucleosomes impede Cas9 access to DNA in vivo and in vitro. eLife. 5, (2016).
- Chavez, A., et al. Comparison of Cas9 activators in multiple species. Nature Methods. 13 (7), 563-567 (2016).
- Zhou, H., et al. In vivo simultaneous transcriptional activation of multiple genes in the brain using CRISPR-dCas9-activator transgenic mice. Nature Neuroscience. 21 (3), 440-446 (2018).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 159 (3), 635-646 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2015).
- Holtzman, L., Gersbach, C. A. Editing the Epigenome: Reshaping the Genomic Landscape. Annual Review of Genomics and Human Genetics. , (2018).
- Perez-Pinera, P., et al. Synergistic and tunable human gene activation by combinations of synthetic transcription factors. Nature Methods. 10 (3), 239-242 (2013).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nature Methods. 12 (12), 1143-1149 (2015).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- McDonald, J. I., et al. Reprogrammable CRISPR/Cas9-based system for inducing site-specific DNA methylation. Biology Open. 5 (6), 866-874 (2016).
- Huang, Y. H., et al. DNA epigenome editing using CRISPR-Cas SunTag-directed DNMT3A. Genome Biology. 18 (1), 176(2017).
- Vojta, A., et al. Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic Acids Research. 44 (12), 5615-5628 (2016).
- Razin, A., Kantor, B. DNA methylation in epigenetic control of gene expression. Progress in Molecular and Subcellular Biology. 38, 151-167 (2005).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18786-18791 (2009).
- Kantor, B., et al. Downregulation of SNCA Expression by Targeted Editing of DNA Methylation: A Potential Strategy for Precision Therapy in PD. Molecular Therapy. , (2018).
- Jowaed, A., Schmitt, I., Kaut, O., Wullner, U. Methylation regulates alpha-synuclein expression and is decreased in Parkinson's disease patients' brains. Journal of Neuroscience. 30 (18), 6355-6359 (2010).
- Wang, Y., et al. A DNA methyltransferase inhibitor, 5-aza-2'-deoxycytidine, exacerbates neurotoxicity and upregulates Parkinson's disease-related genes in dopaminergic neurons. CNS Neuroscience & Therapeutics. 19 (3), 183-190 (2013).
- Matsumoto, L., et al. CpG demethylation enhances alpha-synuclein expression and affects the pathogenesis of Parkinson's disease. PLOS One. 5 (11), e15522(2010).
- Desplats, P., et al. Alpha-synuclein sequesters Dnmt1 from the nucleus: a novel mechanism for epigenetic alterations in Lewy body diseases. Journal of Biological Chemistry. 286 (11), 9031-9037 (2011).
- Ai, S. X., et al. Hypomethylation of SNCA in blood of patients with sporadic Parkinson's disease. Journal of the Neurological Sciences. 337 (1-2), 123-128 (2014).
- Tagliafierro, L., Chiba-Falek, O. Up-regulation of SNCA gene expression: implications to synucleinopathies. Neurogenetics. 17 (3), 145-157 (2016).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Molecular Therapy - Methods & Clinical Development. 5, 153-164 (2017).
- Tagliafierro, L., et al. Genetic analysis of alpha-synuclein 3' untranslated region and its corresponding microRNAs in relation to Parkinson's disease compared to dementia with Lewy bodies. Alzheimer’s & Dementia. 13 (11), 1237-1250 (2017).
- Tagliafierro, L., Zamora, M. E., Chiba-Falek, O. Multiplication of the SNCA locus exacerbates neuronal nuclear aging. Human Molecular Genetics. , (2018).
- Kantor, B., McCown, T., Leone, P., Gray, S. J. Clinical applications involving CNS gene transfer. Advances in Genetics. 87, 71-124 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Advances in Genetics. 87, 125-197 (2014).
- Vijayraghavan, S., Kantor, B. A Protocol for the Production of Integrase-deficient Lentiviral Vectors for CRISPR/Cas9-mediated Gene Knockout in Dividing Cells. Journal of Visualized Experiments. (130), e56915(2017).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Molecular Therapy. 16 (12), 1968-1976 (2008).
- Bassil, C. F., Huang, Z., Murphy, S. K. Bisulfite pyrosequencing. Methods in Molecular Biology. 1049, 95-107 (2013).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Research. 43 (13), 6450-6458 (2015).
- Nishimasu, H., et al. Crystal Structure of Staphylococcus aureus Cas9. Cell. 162 (5), 1113-1126 (2015).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. Journal of Virology. 68 (4), 2632-2648 (1994).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. Journal of Virology. 71 (8), 6113-6127 (1997).
- Goffin, V., et al. Transcription factor binding sites in the pol gene intragenic regulatory region of HIV-1 are important for virus infectivity. Nucleic Acids Research. 33 (13), 4285-4310 (2005).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. Journal of Biological Chemistry. 280 (22), 21545-21552 (2005).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Molecular Therapy. 19 (3), 547-556 (2011).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados