É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um pipeline de bioinformática para analisar com precisão e eficiência os transcriptomas de MicroRNA em plantas
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Um pipeline de bioinformática, ou seja, miRDeep-P2 (miRDP2 para breve), com critérios de miRNA de plantas atualizados e um algoritmo revisado, poderia analisar com precisão e eficiência transcriptomas microRNA em plantas, especialmente para espécies com genomas complexos e grandes.
Resumo
MicroRNAs (miRNAs) são 20- a 24 nucleotídeos (nt) pequenos RNAs endógenos (sRNAs) extensivamente existentes em plantas e animais que desempenham papéis potentes na regulação da expressão gênica no nível pós-transcricional. Sequenciar bibliotecas de sRNA por métodos de sequenciamento de próxima geração (NGS) tem sido amplamente empregado para identificar e analisar transcriptomas miRNA na última década, resultando em um rápido aumento da descoberta de miRNA. No entanto, dois grandes desafios surgem na anotação de miRNA de plantas devido ao aumento da profundidade das bibliotecas sRNA sequenciadas, bem como o tamanho e a complexidade dos genomas das plantas. Em primeiro lugar, muitos outros tipos de sRNAs, em particular, RNAs interfantes interferindo (siRNAs) de bibliotecas sRNA, são erroneamente anotados como miRNAs por muitas ferramentas computacionais. Em segundo lugar, torna-se um processo extremamente demorado para analisar transcriptomas miRNA em espécies vegetais com genomas grandes e complexos. Para superar esses desafios, recentemente atualizamos o miRDeep-P (uma ferramenta popular para análises de transcriptoma miRNA) para miRDeep-P2 (miRDP2 para abreviação) empregando uma nova estratégia de filtragem, revisando o algoritmo de pontuação e incorporando miRNA de plantas recém-atualizado critérios de anotação. Testamos miRDP2 contra populações sequenciadas de sRNA em cinco plantas representativas com crescente complexidade genômica, incluindo arabidopse, arroz, tomate, milho e trigo. Os resultados indicam que o miRDP2 processou essas tarefas com eficiência muito alta. Além disso, miRDP2 superou outras ferramentas de previsão sobre sensibilidade e precisão. Em conjunto, nossos resultados demonstram miRDP2 como uma ferramenta rápida e precisa para analisar transcriptomas de miRNA de plantas, portanto, uma ferramenta útil para ajudar a comunidade a anotar melhor miRNAs em plantas.
Introdução
Uma das descobertas mais emocionantes nas últimas duas décadas em biologia é o papel proliferante das espécies de sRNA na regulação de diversas funções do genoma1. Em particular, os miRNAs constituem uma classe importante de 20 a 24-nt sRNAs em eucariontes, e principalmente funcionam em nível pós-transcricional como reguladores genéticos proeminentes ao longo dos estágios de desenvolvimento do ciclo de vida, bem como em respostas de estímulo e estresse2,3. Nas plantas, miRNAs surgem de transcrições primárias chamadas pri-miRNAs, que geralmente são transcritas pela RNA polymerase II como unidades de transcrição individuais4,5. Processado sigativamente conservado máquinas celulares (Drosha RNase III em animais, DICER-like em plantas), pri-miRNAs são extirbolsados para os precursores miRNA imediatos, pré-miRNAs, que contêm seqüências que formam estruturas intra-moleculares de laço-tronco6,7. Pré-miRNAs são então processados em intermediários duplos, ou seja, duplex miRNA, consistindo da vertente funcional, miRNA maduro, e o parceiro menos freqüentemente funcional, miRNA *2,8. Depois de carregados no complexo de silenciamento induzido pelo RNA (RISC), os miRNAs maduros puderam reconhecer seus alvos mRNA com base na complementaridade da sequência, resultando em uma função regulatória negativa2,8. miRNAs poderia desestabilizar suas transcrições alvo ou impedir a tradução alvo, mas a maneira anterior é dominada em plantas8,9.
Desde a descoberta fortuita do primeiro miRNA no nematóide Caenorhabditis elegans10,11, muita pesquisa tem sido comprometida com a identificação miRNA e sua análise funcional, especialmente após a disponibilidade do método NGS. A ampla aplicação do método NGS promoveu muito a utilização de ferramentas computacionais que foram projetadas para capturar a característica única dos miRNAs, como a estrutura de precursores de stem-loop e seu acúmulo preferencial de leituras de sequências em miRNA e miRNA maduros*. Como resultado, os pesquisadores alcançaram um sucesso notável na identificação de miRNAs em diversas espécies. Com base em um modelo de probabilidade descrito anteriormente12,desenvolvemos o miRDeep-P13,que foi a primeira ferramenta computacional para a descoberta de miRNAs de plantas a partir de dados NGS. o miRDeep-P visava especificamente conquistar os desafios da decodificação de miRNAs de plantas com maior comprimento de precursor e grandes famílias paralogous13,14,15. Após seu lançamento, este programa foi baixado milhares de vezes e usado para anotar transcriptomas miRNA em mais de 40 espécies de plantas16. Impulsionado por ferramentas baseadas em NGS como miRDeep-P, tem havido um aumento dramático no número de miRNAs registrados no miRBase repositório miRBASE17público , onde mais de 38.000 itens miRNA estão atualmente hospedados (lançamento 22,1) em comparação com apenas ~ 500 itens miRNA (lançamento 2.0) em 200818.
No entanto, dois novos desafios surgiram a partir da anotação de miRNA de plantas. Em primeiro lugar, as elevadas proporções de falsos positivos têm impactado fortemente a qualidade das anotações de miRNA de plantas16,19 pelas seguintes razões: 1) um dilúvio de RNAs endógenos de interferência curta (siRNAs) de bibliotecas NGS sRNA foram erroneamente anotados como miRNAs devido à falta de uma rigorosa critérios de annotação miRNA; 2) para espécies sem informações priori miRNA, os falsos positivos previstos com base em dados ngs são difíceis de eliminar. Usando miRBase como exemplo, Taylor et al.20 encontraram um terço das entradas de miRNA de plantas no repositório público21 (lançamento 21) não tinham provas convincentes de apoio e até três quartos das famílias de miRNA de plantas eram questionáveis. Em segundo lugar, torna-se um processo extremamente demorado para prever miRNAs vegetais com genomas grandes e complexos16. Para superar esses desafios, atualizamos o miRDeep-P adicionando uma nova estratégia de filtragem, revisando o algoritmo de pontuação e integrando novos critérios para a anotação de miRNA de plantas e lançou a nova versão miRDP2. Além disso, testamos miRDP2 usando conjuntos de dados NGS sRNA com tamanhos genômicos aumentando gradualmente: Arabidopsis, arroz, tomate, milho e trigo. Em comparação com outras cinco ferramentas amplamente utilizadas e sua versão antiga, o miRDP2 analisou esses dados de sRNA e analisou transcriomas miRNA mais rapidamente com maior precisão e sensibilidade.
Conteúdo do pacote miRDP2
O pacote miRDP2 consiste em seis scripts Perl documentados que devem ser executados sequencialmente pelo script bash preparado. Dos seis scripts, três(convert_bowtie_to_blast.pl, filter_alignments.ple excise_candidate.pl)são herdados do miRDeep-P. Os outros scripts são modificados a partir da versão original. As funções dos seis scripts são descritas no seguinte:
preprocess_reads.pl filtros de entrada lê, incluindo leituras que são muito longas ou muito curtas (<19 nt ou >25 nt), e lê correlacionado com seqüências de Rfam ncRNA, bem como lê com RPM (Lê Per Million) menos de 5. O script, em seguida, recupera lê correlacionado s sequências conhecidas miRNA maduro. Os arquivos de entrada são leituras originais em formato FASTA/FASTQ e saída bowtie2 de leituras de mapeamento para sequências de miRNA e ncRNA.
A fórmula para calcular o RPM é como a seguinte:
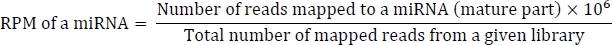
convert_bowtie_to_blast.pl muda o formato de gravata borboleta em formato BLAST-parsed. O formato blast-analisado é um formato separado tabular personalizado derivado do formato padrão NCBI BLASToutput.
filter_alignments.pl filtra os alinhamentos de seqüenciamento profundo lê a um genoma. Ele filtra alinhamentos parciais, bem como leituras multi-alinhadas (corte de frequência especificado pelo usuário). A entrada básica é um arquivo em formato BLAST-analisados.
excise_candidate.pl corta sequências precursoras potenciais de uma sequência de referência usando leituras alinhadas como diretrizes. A entrada básica é um arquivo em formato BLAST-analisados e um arquivo FASTA. A saída é todas as sequências precursoras potenciais no formato FASTA.
mod-miRDP.pl precisa de dois arquivos de entrada, arquivo de assinatura e arquivo de estrutura, que é modificado a partir do algoritmo miRDeep-P principal, alterando o sistema de pontuação com parâmetros específicos da planta. Os arquivos de entrada são arquivo de estrutura precursora do suporte de pontos e lê em arquivo de assinatura de distribuição.
mod-rm_redundant_meet_plant.pl precisa de três arquivos de entrada: chromosome_length, precursores e original_prediction gerados por mod-miRDP.pl. Ele gera dois arquivos de saída, arquivo previsto não redundante e arquivo previsto filtrado por critérios de miRNA de plantas recém-atualizados. Detalhes sobre o formato do arquivo de saída são descritos na seção 1.4.
Protocolo
1. Instalação e testes
- Baixe as dependências necessárias: Bowtie222 e RNAfold23. Pacotes compilados são recomendados.
- Baixe Bowtie2, uma ferramenta de mapeamento de leitura, a partir de seu site de origem(http://bowtie-bio.sourceforge.net/bowtie2/index.shtml).
- Baixe RNAfold, uma ferramenta do pacote de Viena usado para prever a estrutura secundária de RNA, a partir de http://www.tbi.univie.ac.at/~ivo/RNA/.
- Antes de instalar miRDP2, certifique-se de que essas duas dependências sejam corretamente instaladas e personalize o arquivo do ambiente bash (por exemplo, .bashrc) para definir um CAMINHO correto para essas duas dependências.
NOTA: Outras ferramentas de mapeamento, como Bowtie24 também são adequados para miRDP2; Bowtie ou Bowtie2 podem ser usados após a versão 1.1.3.
- Para baixar o pacote miRDP2, vá para https://sourceforge.net/projects/mirdp2/files/latest_version/ e buscar os arquivos tarball.
- Antes de instalar miRDP2, certifique-se de que Perl está no PATH. Para instalar miRDP2, extraia todo o conteúdo do arquivo de tarball baixado em uma pasta (linhas de comando como em 1,4,2), e, em seguida, definir o caminho da pasta para o PATH.
NOTA: Um computador ou nó de computação com pelo menos 8 GB ram e 100 GB de armazenamento são recomendados para executar miRDP2. - Teste o oleoduto MiRDP2.
- Para testar se o miRDP2 foi instalado corretamente, use os dados de teste e a saída esperada encontrada em https://sourceforge.net/projects/mirdp2/files/TestData/. Os dados de teste contêm um arquivo de sequenciamento gsm formatado e um arquivo do genoma de thaliana de Arabidopsis.
- Mova todos os arquivos baixados para o diretório de trabalho atual:
mv miRDP2-v*.tar.gz TestData.tar.gz ncRNA_rfam.tar.gz
cd - Extraia os arquivos de tarball comprimidos:
alcatrão -xvzf miRDP2-v*.tar.gz
alcatrão -xvzf TestData.tar.gz
alcatrão -xvzf ncRNA_rfam.tar.gz - Construa o índice de referência do genoma da Arabidopsis:
bowtie2-build -f ./TestData/TAIR10_genome.fa ./TestData/TAIR10_genome - Construa o índice de referência ncRNA:
bowtie2-build -f ./ncRNA_rfam.fa ./1.1.3/script/index/rfam_index - Executar o oleoduto miRDP2:
bash ./1.1.3/miRDP2-v1.1.3_pipeline.bash -g ./TestData/TAIR10_genome.fa -i ./ TestData/TAIR 10_genome -f ./TestData/GSM2094927.fa -o .
NOTA: Os comandos linux usados estão em fontes arrojadas e itálicas, com opções de linha de comando em itálico. *indica a versão do miRDP2 (a versão atual é 1.1.3). O comando bowtie2-build deve levar cerca de 10 minutos, eo gasoduto miRDP2 deve terminar dentro de vários minutos
- Verifique as saídas de teste.
- Note-se que uma pasta chamada 'GSM2094927-15-0-10' é gerada automaticamente em , contendo todos os arquivos intermediários e resultados.
- Verifique se o arquivo de saída delimitada de guiaGSM2094927-15-0-10_filter_P_prediction, a saída final dos miRNAs previstos, contém colunas que indicam cromossomo id, direção de vertente, representante lê id, id precursor, localização miRNA madura, precursor localização, seqüência madura e sequência precursora. Observe o arquivo adicional da cama derivado deste arquivo para facilitar uma análise mais aprofundada.
- Verifique o arquivo "progress_log", que fornece informações sobre as etapas terminadas, e os arquivos "script_log" e "script_err", que contêm saída do programa e avisos.
NOTA: Atualmente, testamos miRDP2 em duas plataformas Linux, incluindo a liberação CentOS 6.5 em um servidor de cluster, e Cygwin 2.6.0 no sistema PC Windows, e miRDP2 deve funcionar em sistemas semelhantes que suportam Perl.
2. Identificação de miRNAs romance
- Antes de executar o pipeline, certifique-se de que as leituras de entrada sejam pré-processadas em formato adequado.
NOTA: A nova versão 1.1.3 do miRDP2 pode aceitar arquivos originais do formato FASTQ como entradas, embora o processo de leituras de formatação seja realizado como nas versões anteriores.- Primeiro, retire os adaptadores das extremidades de 5 e 3' das leituras de sequenciamento profundo (se presente).
- Em segundo lugar, analise as leituras de sequenciamento profundo no formato FASTA.
- Em terceiro lugar, remover a redundância, de tal forma que lê com seqüência idêntica são representados com uma entrada FASTA única e única.
- Finalmente, certifique-se de que todos os identificadores fasta são únicos. Cada identificador de sequência deve terminar com um '_x' e um inteiro, indicando o número de cópia da sequência exata que foi recuperada nos conjuntos de dados de sequenciamento profundo. Uma maneira de garantir que o identificador FASTA exclusivo é incluir um número de execução no ID. Para referência, consulte o arquivo GSM2094927.fa nos dados do teste (https://sourceforge.net/projects/mirdp2/files/TestData/).
- Veja o seguinte para exemplos de leituras corretamente formatado:
>read0_x29909
TTTGGATTGAAGGGAGCTCTA TTTGGATTGAAGGGAGCTCTA
>read1_x36974
TTCCACAGCTTTCTTGAACTG TTCCACAGCTTTCTTGAACTG
>read2_x32635
TTCCACAGCTTTCTTGAACTT TTCACAGCAGCTTTCTTGAACTT
- Construir índices de referência.
- Para a referência do genoma, para economizar tempo, baixe arquivos de índice Bowtie2 do site iGenomes(https://support.illumina.com/sequencing/sequencing_software/igenome.html)se as sequências do genoma das espécies de interesse tiverem sido indexadas. Caso contrário, os usuários indexam sequências de referência e mantêm o arquivo do índice por um tempo até que o projeto seja concluído, já que a sequência do genoma pode precisar ser reindexada. Detalhes sobre como indexar uma referência do genoma estão incluídos no manual bowtie2(http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml).
- Outro índice ncRNA não-miRNA também é necessário para filtrar sequências ruidosas de outros fragmentos de RNA não codificadores. O arquivo é uma coleção de seqüências principais ncRNA de Rfam, incluindo rRNA, tRNA, snRNA e snoRNA. Para construir este índice, consulte a parte 1.4, pois o índice deve ser colocado e nomeado corretamente, ou seja, & miRDP2_version>/script/index/rfam_index.
- Executar miRDP2.
- Para usar miRDP2 para detectar novos miRNAs a partir de dados de sequenciamento profundo, executar o script bash no pacote para iniciar o pipeline de análise (Um exemplo pode ser encontrado na etapa 1.4):
/miRDP2-v*.*_pipeline.bash -g -i -f -o
onde * indica a versão do script bash pipeline. Há três parâmetros que podem ser modificados: 1) o número de locais diferentes para os os dois locais para os qual uma leitura pode ser mapeada, 2) o número de incompatibilidade para a execução de bowtie2 e 3) o limiar do RPM (Lê Por Milhão). Modifique-os usando as opções -L, -M e -R, respectivamente. Uma explicação detalhada está na seção 3.1.
- Para usar miRDP2 para detectar novos miRNAs a partir de dados de sequenciamento profundo, executar o script bash no pacote para iniciar o pipeline de análise (Um exemplo pode ser encontrado na etapa 1.4):
- Verifique as saídas miRDP2.
- Note-se que a pasta de saída será gerada automaticamente , e nomeada '-15-0-10'; os últimos 3 números indicam os valores (padrão neste caso) para parâmetros 1, 2 e 3, respectivamente. O arquivo _filter_P_prediction contém informações dos miRNAs previstos finais satisfazendo os critérios de anotação de miRNA de plantas recém-atualizados. Detalhes sobre o formato do arquivo de saída são descritos na parte 1.4.
3. Modificações e cautela usando miRDP2
- Parâmetros que podem ser modificados
- Use a opção '-L' para definir o limite de quantos locais uma leitura poderia ser mapeada (parâmetro 1). Leia o mapeamento de muitos sites são possivelmente associados com seqüências de repetição, e não são susceptíveis de miRNAs. A configuração padrão é 15. Para espécies específicas, se houver famílias miRNA com muitos membros, o primeiro parâmetro pode ser aumentado manualmente para se adaptar à paisagem do genoma.
- Use a opção '-M' para definir as incompatibilidades permitidas para gravata borboleta (parâmetro 2). A configuração padrão é 0.
- Use a opção '-R' para definir o limite para leituras potencialmente correspondentes a miRNAs maduros (parâmetro 3). Para reduzir o consumo de tempo e falsos positivos, o filtro lê por RPM. Apenas leituras excedendo um certo limite de RPM podem representar sequências maduras de miRNAs em vez de ruído de fundo, e seriam mantidas para análise mais aprofundada. A configuração padrão é 10 RPM.
- Observe que a alteração desses parâmetros pode afetar potencialmente o desempenho e o consumo de tempo. Em geral, um aumento do parâmetro 1 e 2 e uma diminuição do parâmetro 3 geraria um resultado menos rigoroso e maior tempo de execução e vice-versa.
- Redundância e miRNA*
- Observe que os miRNAs de saída do miRDP2 podem diferir dos miRNAs conhecidos. Descobrimos que isso se deve principalmente a uma das duas razões: heterogeneidade dos miRNAs maduros ou a abundância relativa de miRNA e miRNA*. Descobrimos que isso não afeta a seleção de comprimento ideal de precursores e o perfil de genes miRNA conhecidos.
Resultados
O pipeline de anotação miRNA, miRDP2, descrito aqui é aplicado a 10 bibliotecas públicas sRNA-seq de 5 espécies de plantas com comprimento gradualmente maior do genoma, incluindo Arabidopsis thaliana, Oryza sativa (arroz), Solanum lycopersicum (tomate), Zea mays (milho) e Triticum aestivum (trigo) (Figura 1A). No geral, para cada espécie, 2 bibliotecas representativas de sRNA de diferentes tecidos (colapsadas em leituras ún...
Discussão
Com o advento do NGS, um grande número de miRNA loci foram identificados a partir de uma quantidade cada vez maior de dados de sequenciamento de sRNA em diversas espécies29,30. No banco de dados da comunidade centralizada miRBase21, os itens de miRNA depositados aumentaram quase 100 vezes na última década. No entanto, em comparação com miRNAs em animais, os miRNAs vegetais têm muitas características únicas que tornam a identifica?...
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Este trabalho foi apoiado pela Academia de Agricultura e Ciências Florestais de Pequim (KJCX201917, KJCX20180425 e KJCX20180204) para a XY e a National Natural Science Foundation of China (31621001) para ll.
Materiais
| Name | Company | Catalog Number | Comments |
| Computer/computing node | N/A | N/A | Perl is required; at least 8 GB RAM and 100 GB storage are recommended |
Referências
- Ghildiyal, M., Zamore, P. D. Small silencing RNAs: an expanding universe. Nature Reviews Genetics. 10 (2), 94-108 (2009).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Moran, Y., Agron, M., Praher, D., Technau, U. The evolutionary origin of plant and animal microRNAs. Nature Ecology Evolution. 1 (3), 27 (2017).
- Xie, Z., et al. Expression of Arabidopsis MIRNA genes. Plant Physiology. 138 (4), 2145-2154 (2005).
- Zhao, X., Zhang, H., Li, L. Identification and analysis of the proximal promoters of microRNA genes in Arabidopsis. Genomics. 101 (3), 187-194 (2013).
- Bologna, N. G., Mateos, J. L., Bresso, E. G., Palatnik, J. F. A loop-to-base processing mechanism underlies the biogenesis of plant microRNAs miR319 and miR159. EMBO JOURNAL. 28 (23), 3646-3656 (2009).
- Rogers, K., Chen, X. Biogenesis, turnover, and mode of action of plant microRNAs. Plant Cell. 25 (7), 2383-2399 (2013).
- Voinnet, O. Origin, biogenesis, and activity of plant microRNAs. Cell. 136 (4), 669-687 (2009).
- Iwakawa, H. O., Tomari, Y. The Functions of MicroRNAs: mRNA Decay and Translational Repression. Trends in Cell Biology. 25 (11), 651-665 (2015).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75 (5), 855-862 (1993).
- Friedlander, M. R., et al. Discovering microRNAs from deep sequencing data using miRDeep. Nature Biotechnology. 26 (4), 407-415 (2008).
- Yang, X., Li, L. miRDeep-P: a computational tool for analyzing the microRNA transcriptome in plants. Bioinformatics. 27 (18), 2614-2615 (2011).
- Meyers, B. C., et al. Criteria for annotation of plant MicroRNAs. Plant Cell. 20 (12), 3186-3190 (2008).
- Yang, X., Zhang, H., Li, L. Global analysis of gene-level microRNA expression in Arabidopsis using deep sequencing data. Genomics. 98 (1), 40-46 (2011).
- Kuang, Z., Wang, Y., Li, L., Yang, X. miRDeep-P2: accurate and fast analysis of the microRNA transcriptome in plants. Bioinformatics. , (2018).
- Kozomara, A., Birgaoanu, M., Griffiths-Jones, S. miRBase: from microRNA sequences to function. Nucleic Acids Research. 47 (1), 155-162 (2019).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucleic Acids Research. 36, 154-158 (2008).
- Axtell, M. J., Meyers, B. C. Revisiting Criteria for Plant MicroRNA Annotation in the Era of Big Data. Plant Cell. 30 (2), 272-284 (2018).
- Taylor, R. S., Tarver, J. E., Hiscock, S. J., Donoghue, P. C. Evolutionary history of plant microRNAs. Trends in Plant Science. 19 (3), 175-182 (2014).
- Kozomara, A., Griffiths-Jones, S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research. 42, 68-73 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Lorenz, R., et al. ViennaRNA Package 2.0. Algorithms for Molecular Biology. 6, 26 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), 25 (2009).
- An, J., Lai, J., Sajjanhar, A., Lehman, M. L., Nelson, C. C. miRPlant: an integrated tool for identification of plant miRNA from RNA sequencing data. BMC Bioinformatics. 15, 275 (2014).
- Lei, J., Sun, Y. miR-PREFeR: an accurate, fast and easy-to-use plant miRNA prediction tool using small RNA-Seq data. Bioinformatics. 30 (19), 2837-2839 (2014).
- Evers, M., Huttner, M., Dueck, A., Meister, G., Engelmann, J. C. miRA: adaptable novel miRNA identification in plants using small RNA sequencing data. BMC Bioinformatics. 16, 370 (2015).
- Mathelier, A., Carbone, A. MIReNA: finding microRNAs with high accuracy and no learning at genome scale and from deep sequencing data. Bioinformatics. 26 (18), 2226-2234 (2010).
- Zhu, Q. H., et al. A diverse set of microRNAs and microRNA-like small RNAs in developing rice grains. Genome Research. 18 (9), 1456-1465 (2008).
- Fahlgren, N., et al. MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana. Plant Cell. 22 (4), 1074-1089 (2010).
- Fromm, B., et al. A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome. Annual Review of Genetics. 49, 213-242 (2015).
- Blevins, T., et al. Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guiding de novo DNA methylation in Arabidopsis. Elife. 4, 09591 (2015).
- Zhai, J., et al. A One Precursor One siRNA Model for Pol IV-Dependent siRNA Biogenesis. Cell. 163 (2), 445-455 (2015).
- Werner, S., Wollmann, H., Schneeberger, K., Weigel, D. Structure determinants for accurate processing of miR172a in Arabidopsis thaliana. Current Biology. 20 (1), 42-48 (2010).
- Mateos, J. L., Bologna, N. G., Chorostecki, U., Palatnik, J. F. Identification of microRNA processing determinants by random mutagenesis of Arabidopsis MIR172a precursor. Current Biology. 20 (1), 49-54 (2010).
- Vitsios, D. M., et al. Mirnovo: genome-free prediction of microRNAs from small RNA sequencing data and single-cells using decision forests. Nucleic Acids Research. 45 (21), 177 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados