É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Uma plataforma de espectrometria de massa de troca de hidrogênio (HDX-MS) para investigar enzimas biossintéticas de peptídeos
Neste Artigo
Resumo
As sintetizações lanthipeptide catalisam reações multistep durante a biossíntese de produtos naturais de peptídeos. Aqui, descrevemos um fluxo de trabalho contínuo, de baixo para cima, de espectrometria de massa de troca de hidrogênio -deutério (HDX-MS) que pode ser empregado para estudar a dinâmica conformacional das sintetizações de lanthipeptídeo, bem como outras enzimas semelhantes envolvidas na biossíntese de produtos naturais peptídeos.
Resumo
Espectrometria de massa de troca de hidrogênio-deutério (HDX-MS) é um método poderoso para a caracterização biofísica de alterações conformacionais enzimáticos e interações entre enzimas enzimistas. Entre seus muitos benefícios, o HDX-MS consome apenas pequenas quantidades de material, pode ser realizado em condições próximas de nativos sem a necessidade de rotulagem enzimada/substrato, e pode fornecer informações espacialmente resolvidas sobre a dinâmica conformacional enzimático– mesmo para grandes enzimas e complexos multiproteínas. O método é iniciado pela diluição da enzima de interesse em tampão preparado em D2O. Isso desencadeia a troca de protium em peptídeos (N-H) com deutério (N-D). Nos pontos de tempo de troca desejados, as alíquotas de reação são saciadas, a enzima é proteolisada em peptídeos, os peptídeos são separados por cromatografia líquida de ultra-desempenho (UPLC), e a mudança na massa de cada peptídeo (devido à troca de hidrogênio por deutério) é registrada pela MS. A quantidade de absorção de deutério por cada peptídeo depende fortemente do ambiente local de ligação de hidrogênio desse peptídeo. Peptídeos presentes em regiões muito dinâmicas do deutério de troca de enzimas muito rapidamente, enquanto peptídeos derivados de regiões bem ordenadas passam por troca muito mais lentamente. Desta forma, a taxa HDX relata a dinâmica conformacional da enzima local. Perturbações aos níveis de absorção de deutério na presença de diferentes ligantes podem então ser usadas para mapear sites de ligação de ligantes, identificar redes alusicais e entender o papel da dinâmica conformacional na função enzimática. Aqui, ilustramos como usamos o HDX-MS para entender melhor a biossíntese de um tipo de peptídeos naturais chamados lanthipeptides. Lanthipeptídeos são peptídeos geneticamente codificados que são modificados pós-translacionalmente por enzimas grandes, multifuncionais e conformacionalmente dinâmicas que são difíceis de estudar com abordagens tradicionais de biologia estrutural. O HDX-MS fornece uma plataforma ideal e adaptável para investigar as propriedades mecanicistas desses tipos de enzimas.
Introdução
Proteínas são moléculas estruturalmente dinâmicas que amostram diferentes conformações em escalas de tempo que vão desde vibração de ligação em escala femtosegundo até rearranjos de domínios proteicos inteiros que podem ocorrer ao longo de muitos segundos1. Estas flutuações conformais são frequentemente aspectos críticos da função enzima/proteína. Por exemplo, alterações conformais induzidas pela ligação de ligasão são muitas vezes criticamente importantes para a modulação da função enzimática, seja organizando resíduos ativos do local necessários para a catálise, definindo locais de ligação de substrato em mecanismos cinéticos sequenciais, protegendo intermediários reativos do ambiente ou modulando a função enzimática através de redes alostêmicas. Estudos recentes também mostraram que a dinâmica conformacional pode ser conservada ao longo da evolução e que perturbações a movimentos moleculares conservados podem ser correlacionadas com mudanças na especificidade do substrato e no surgimento de novas funções enzimárias2,3.
Nos últimos anos, a espectrometria de massa de troca de hidrogênio-deutério (HDX-MS) tem emergido rapidamente como uma técnica poderosa para sondar como as paisagens conformais da proteína respondem a perturbações como ligadura ou mutagênese4,5,6,7. Em um experimento típico hdx-ms(Figura 1), uma proteína de interesse é colocada em tampão preparado em D2O, que desencadeia a substituição de prótons trocaveis de solventes por deuteria. A taxa de troca da moiety amida dos títulos de peptídeo depende fortemente do pH, da sequência local de aminoácidos, e do ambiente estrutural local da amida8. Amidas que estão engajadas em interações de ligação de hidrogênio (como as presentes em α-helices e β-folhas) trocam mais lentamente do que em meios em regiões não estruturadas da proteína que são expostas a solventes a granel. Assim, a extensão da captação do deutério é um reflexo da estrutura da enzima. Enzimas que são conformamente dinâmicas, ou que passam por transições estruturais sobre ligação ligante, seriam esperadas para produzir uma resposta HDX mensurável.
A base mecanicista para a taxa de câmbio lenta de uma amida estruturada é mostrada na Figura 25,8,9. Para se submeter ao HDX, a região estruturada deve primeiro amostrar transitoriamente uma conformação desdobrada, de tal forma que as moléculas de solvente que catalisam a troca de HDX através de um mecanismo químico ácido/base específico, tenham acesso ao amide tromável. Em última análise, as magnitudes relativas da taxa de câmbio química (kchem) e as taxas de dobra e reagem(kopen e kclose) determinam a taxa HDX medida no experimento5,8. A partir deste modelo cinético simples, é claro que a extensão da absorção do deutério refletirá a dinâmica conformacional subjacente (definida por kopen e kclose). A maioria dos experimentos HDX-MS são realizados em um fluxo de trabalho de baixo para cima onde, após a reação de troca, a proteína de interesse é digerida em peptídeos e a absorção do deutério por cada peptídeo é medida como um aumento na massa7. Dessa forma, o HDX-MS permite que perturbações para a dinâmica conformacional enzimária sejam mapeadas na escala espacial local dos peptídeos, permitindo que o pesquisador avalie como a perturbação altera a dinâmica em diferentes regiões da enzima de interesse.
As vantagens da abordagem HDX-MS para a dinâmica estrutural proteica elucidação são inúmeras. Em primeiro lugar, o método pode ser realizado com pequenas quantidades de proteína nativa ou em complexos proteicos em sistemas com estrutura quaternária10. Não é mesmo necessário que a preparação da enzima usada no ensaio seja altamente purificada11,12, desde que o fluxo de trabalho HDX-MS de baixo para cima forneça um número suficiente de peptídeos confiantemente identificados que cobrem a sequência proteica de interesse. Além disso, o HDX-MS pode fornecer informações sobre a dinâmica conformacional em condições próximas de nativos sem a necessidade de rotulagem de proteínas específicas do local, como seria usado em estudos de fluorescência de moléculas únicas13, e não há limite de tamanho para o complexo proteico ou proteico que possa ser investigado (o que torna desafiadoras abordagens como ressonância magnética nuclear [NMR] espectroscopia)7,14. Finalmente, métodos HDX-MS resolvidos pelo tempo podem ser empregados para estudar proteínas intrinsecamente desordenadas, que são difíceis de estudar com cristalografia de raios-X15,16,17,18. A principal limitação do HDX-MS é que os dados são de baixa resolução estrutural. Os dados HDX-MS são úteis para apontar para onde a dinâmica conformacional está mudando e para revelar alterações conformais acopláveis, mas muitas vezes não fornecem muita visão do mecanismo molecular preciso que conduz a mudança observada. Avanços recentes na combinação de métodos de dissociação de captura de elétrons com dados de proteína HDX-MS mostraram-se promissores para mapear locais de troca para resíduos de aminoácidosúnicos 19,mas estudos bioquímicos e estruturais de acompanhamento ainda são frequentemente necessários para fornecer clareza aos modelos estruturais encaminhados pelos dados hdx-MS.
Abaixo, um protocolo detalhado para o desenvolvimento de um ensaio HDX-MS é apresentado20. Os protocolos de preparação da amostra apresentados abaixo devem ser geralmente aplicáveis a qualquer proteína que exaque boa solubilidade em tampões aquosos. Métodos de preparação de amostras mais especializados e fluxos de trabalho HDX-MS estão disponíveis para proteínas do que precisam ser avaliados na presença de detergentes ou fosfolipídios21,22,23,24. As configurações instrumentais para a coleta de dados HDX-MS são descritas para um espectrômetro de massa quadrúpole de alta resolução acoplado ao sistema de cromatografia líquida. Dados de complexidade e resolução semelhantes poderiam ser coletados em qualquer um dos vários sistemas de espectrometria de massa líquida (LC-MS) disponíveis comercialmente. Aspectos-chave do processamento de dados usando um pacote de software comercialmente disponível também são fornecidos. Também apresentamos diretrizes para coleta e análise de dados que são consistentes com as recomendações feitas pela comunidade HDX-MS mais ampla12. O protocolo descrito é usado para estudar as propriedades estruturais dinâmicas do HalM2, uma sintetização lanthipeptide que catalisa a maturação multistep de um produto natural de peptídeo antimicrobiano20. Ilustramos como o HDX-MS pode ser usado para revelar sites de ligação de substratos e propriedades alotésicas que iludiram a caracterização anterior. Vários outros protocolos sobre proteína HDX-MS foram publicados nos últimos anos25,26. Juntamente com o presente trabalho, essas contribuições anteriores devem proporcionar ao leitor alguma flexibilidade no design experimental.
Protocolo
1. Preparação de reagentes deuterados e soluções de estoque de enzimas
- Prepare os reagentes necessários para as reações HDX (incluindo quaisquer buffers, sais, substratos, ligantes, etc.) como soluções de estoque concentradas de 100-200x em D2O (99,9% fração de átomo D). Prepare pelo menos 50 mL de solução de estoque tampão.
NOTA: Para caracterização do HalM2, foram preparadas as seguintes soluções: 500 mM MgCl2, 100 mM tris (2 carboxyethyl)fospfina (TCEP), ATP de 750 mM (em tampão HEPES), 800 mM HEPES pD 7.1, 500 μM HalA2 e 500 mM AMPP. - Congele e liofilize as soluções de estoque para o ressecamento.
- Ressulimação em D2O, e ciclo de liofilização repetido pelo menos um tempo adicional para substituir o maior número possível de prótons trocaveis por deuterons.
- Ajuste o pD do estoque tampão HEPES desuterado ao valor desejado com NaOD/DCl concentrado, tendo em mente a seguinte relação27:
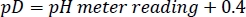
NOTA: A taxa de AMIDE HDX é fortemente dependente do pL da solução (pL = pH ou pD)5. Diferentes lotes de soluções de estoque tampão precisam ser preparados, armazenados e usados de forma idêntica para evitar uma leve deriva de pL entre os experimentos. - Calcule a quantidade de cada reagente necessário para um ensaio HDX de 300 μL e armazene como alíquotas de uso único a -80 °C.
- Prepare uma solução concentrada de estoque de enzimas (~100-200 μM) em um tampão de armazenamento de enzimas protiado usando um filtro centrífuga(Tabela de Materiais) ou dispositivo equivalente.
NOTA: O tampão exato e o corte de peso molecular do filtro centrífuga dependerão da proteína/enzima de interesse. O HalM2 é armazenado em HEPES de 50 mM, pH 7,5, 100 mM KCl e 10% glicerol. Foram utilizados filtros de 10 kDa para preparar a enzima concentrada. - Aliquotar a enzima em porções de uso único e armazenar a -80 °C.
NOTA: Este pode ser um ponto de parada. Todas as soluções de estoque descritas na seção 1 podem ser preparadas antes das reações HDX. Se armazenado a -80 °C, a maioria das enzimas/soluções de estoque desuterados ficará estável por muitos meses.
2. Calibração do volume de saciecia HDX
- Prepare uma reação HDX de 300 μL em D2O usando os reagentes deuterados e estoques concentrados de enzimas preparados na seção 1.
- Use uma concentração enzimática final de 1-5 μM.
- Use uma concentração final de buffer HEPES desuterada de pelo menos 50-100 mM.
- Certifique-se de que as concentrações de outros componentes são suficientes para manter a atividade/função da enzima desejada.
- Prepare 1 L de solução de sátdia HDX (fosfato de 100 mM, 0,8 M guanidine-HCl, pH 1.9). Congele e armazene em ambas as porções de 50 mL (para estoque de longo prazo) e 1 mL (para alíquotas de uso único).
NOTA: A composição exata do tampão de saciamento dependerá da enzima que é usada na etapa de proteólise do fluxo de trabalho HDX-MS de baixo para cima (etapa 3.3.3). O tampão de sumosa dado aqui é compatível com pepsina, o protease mais usado para HDX-MS. Se for usada uma protease diferente, verifique com o fornecedor protease para garantir a compatibilidade com o buffer. - Calibrar o volume de tampão de sutura necessário para ajustar o pL final da mistura de reação HDX saciada a um valor de leitura do medidor de pH de 2,3.
NOTA: A taxa de câmbio H/D solvente do vínculo peptídeo amide N-H é um processo dependente de pH que está sujeito tanto à catalise ácido quanto à base-catálise. A taxa de câmbio mínima ocorre a um valor de pH 2,5 (leitura do medidor de pH = 2,3 para uma mistura de 50:50 H2O:D2O). Assim, um valor de pL final perto de 2,5 minimizará a troca de recuo de hidrogênio que ocorre durante a análise de LC-MS de baixo para cima, preservando assim o rótulo de deutério nos peptídeos.- Misture 50 μL da mistura de reação HDX da etapa 2.1 com 50 μL de tampão de saciar e meça o pL da mistura saciada com um eletrodo de micropéia.
- Aumente o volume da solução de saciar conforme necessário para ajustar a leitura final do medidor de pH a um valor de 2,3.
- Uma vez determinado o volume de saciamento apropriado, repita o processo de saciamento várias vezes usando alíquotas frescas de 50 μL da reação HDX (etapa 2.1) para garantir que um pL final consistente seja alcançado após a adição de uma quantidade fixa de tampão de saciamento.
3. Elaboração de amostras de referência e otimização do fluxo de trabalho LC-MS de baixo para cima
- Prepare amostras de referência não deuteradas para a proteína de interesse em triplicado em tubos de 0,5 mL. Certifique-se de que as condições finais da mistura de reação são idênticas às usadas nas autênticas reações HDX (etapa 2.1), exceto que as reações são preparadas em H2O usando soluções de estoque de reagente também preparadas em H2O.
- Sacie as amostras como na etapa 2.3 adicionando o volume apropriado de tampão de saciar para ajustar o pH final para 2,5. O flash congele as amostras em nitrogênio líquido e armazene a -80 °C até ficar pronto para análise.
- Analise as amostras de referência de enzimas protiadas usando um fluxo de trabalho LC-MS de baixo para cima.
NOTA: Antes de executar estas etapas, o sistema LC-MS a ser utilizado para aquisição de dados deve ser devidamente calibrado e pronto para uso. O tempo e a temperatura de todas as etapas do fluxo de trabalho LC-MS inferior para cima devem ser rigorosamente controlados, a fim de minimizar as diferenças na troca de volta entre as amostras. Com a instrumentação MS utilizada neste protocolo (Tabela de Materiais e Informações de suporte), a maioria das etapas pode ser controlada através do software de instrumentos. Para garantir a coleta de réplicas precisas, recomenda-se automatizar o maior número possível de etapas no fluxo de trabalho.- Remova uma amostra de referência de enzima individual (preparada como na etapa 3.2) do congelador e descongele a 37 °C por 1 min em banho-maria.
- Precisamente 2 minutos após a remoção da amostra do congelador e do descongelamento, injete uma porção de 40 μL da amostra de referência saciada em uma coluna de cromatografia líquida de ultra-desempenho (UPLC) (2,1 x 30 mm, 300 Å, 5 μM) contendo uma fase estacionária funcionalizada com pepsina (protease ácido-estável).
- Digerir a amostra a uma taxa de fluxo de 100 μL/min por 3 min a 15 °C usando ácido fórmico de 0,1% em H2O (pH = 2,5) como solvente.
- Colete os peptídeos peptic enquanto se eluem da coluna pepsina em uma coluna de armadilha C18 mantida a 0,4 °C para minimizar a troca de costas.
- Passe os peptídeos péticos dessatados da coluna armadilha para uma coluna analítica C18 (1 mm x 100 mm, 1,7 μM, 130 Å) realizada e operada a 0,4 °C para separação dos peptídeos pepéticos.
NOTA: As etapas 3.3.3-3.3.5 podem ser automatizadas por certos sistemas LC-MS usados para aquisição de dados HDX-MS. Alternativamente, essas etapas podem ser executadas de forma independente, tendo em mente que o tempo e a temperatura de cada etapa precisam ser cuidadosamente controlados para alcançar uma troca traseira consistentemente baixa. - Elute a coluna C18 com um sistema de solvente de ácido fórmico/acetonitrilo/água/0,1% paramórico. Otimize o gradiente LC para a proteína de interesse, a fim de maximizar a separação e preservar o rótulo de deutério nos peptídeos pepticos.
NOTA: Os detalhes de elução de gradiente são fornecidos nas informações de suporte. - Sujeito o digestor peptic à espectrometria de massa de eletroprai (ESI).
NOTA: As condições de origem fornecidas nas Informações de Suporte fornecerão ionização suficiente para a maioria dos peptídeos pepticos.- Uma vez ionizado no instrumento MS, realize uma separação de mobilidade de íons de fase gasosa usando nitrogênio como gás tampão para melhorar a capacidade máxima do método.
- Após a separação da mobilidade de íons, sujeitos aos íons precursores do peptídeos a um fluxo de trabalho MSE envolvendo ciclos alternados de baixa energia de colisão (4 V) e alta energia de colisão (21-40 V).
NOTA: Os regimes alternados de energia de baixa e alta colisão permitem a coleta de dados de MS (baixa energia de colisão) simultaneamente com dados MSMS (alta energia de colisão). Isso, por sua vez, permite a correlação temporal dos íons precursores com seus respectivos íons fragmentado. Esta correlação é essencial para a identificação confiante de peptídeos descritas na seção 4. - Detecte o precursor do peptídeo e íons fragmentado usando um analisador de massa com um poder de resolução de pelo menos 20.000.
- Simultaneamente à aquisição de dados, adquira dados MS para um padrão externo [Glu-1]-fibrinopeptide B (GluFib).
NOTA: O fluxo de trabalho MS descrito na etapa 3.3.7 é referido como um protocolo MSE. Configurações instrumentais completas para um protocolo MSE adequado para HDX-MS de baixo para cima são fornecidas nas Informações de Suporte.
- Avalie a qualidade dos dados LC-MS.
NOTA: Utilizando o protocolo descrito acima e as configurações instrumentais fornecidas nas Informações de Suporte,as amostras de referência devem produzir um cromatógrafo de íon total com uma intensidade máxima de sinal de aproximadamente 1 x 108. Deve haver muitos peptídeos pepticos eluindo entre 3-9 min (Figura 3A−C). - Injete 40 μL de amostras em branco (0,1% de ácido fórmico na água) para limpar a pepsina e as colunas C18 analíticas.
NOTA: Em geral, 2-3 espaços em branco devem ser suficientes. - Repetir passos 3.3.1-3.3.9 para cada uma das amostras de proteína de referência triplicada.
4. Processar os dados de referência e definir uma lista de peptídeos
- Analisar os dados mse brutos (etapa 3.3.7) utilizando software de proteômica(Tabela de Materiais). Usando o software de proteômica, navegue até bibliotecas | Protein Sequence Databanks para definir o banco de dados de proteínas importando a sequência de aminoácidos da proteína de interesse.
NOTA: O objetivo desta etapa é pesquisar os dados de MS de referência para peptídeos peptídeos derivados da proteína de interesse, e utilizar os dados MSMS (adquiridos simultaneamente com os dados de MS) para validar quaisquer identificações de peptídeos putativos. - Dê um nome à sequência de proteínas de interesse. Importe a sequência proteica (em formato FASTA). O software realizará uma digestão em silico da proteína do banco de dados para gerar uma lista de peptídeos que serão usados para pesquisar os dados do LC-MS.
- Defina os parâmetros de processamento (localizados no menu Biblioteca). Selecione Electrospray MSE como o tipo de aquisição de dados. No campo Lock Mass for Charge 2, digite 785.8426 para o m/z para o íon 2+ de [Glu-1]-fibrinopeptídeo B (GluFib) e clique no acabamento.
- Defina os parâmetros de fluxo de trabalho (localizados no menu Biblioteca).
- Selecione Electrospray MSE para o tipo de pesquisa. Sob o | de fluxo de trabalho Título de Consulta de Pesquisa de banco de dados, selecione a proteína de banco de dados criada na etapa 4.2 no campo do Databank.
- Altere o reagente de digestão primário para inespecífico, e limpe o campo de reagente modificador fixo segurando o botão Ctrl enquanto clica em Carbamidomethyl C.
- Especifique o diretório de saída navegando para opções | configuração de automação | Identidade E. Verifique as caixas para Apex 3D e Peptide 3D Output e Ion Accounting Output e e especifique o diretório desejado.
- Processe os dados da amostra de referência.
- Na barra de ferramentas esquerda do espaço de trabalho da plataforma proteômica, crie uma nova placa clicando à direita na Placa Microtiter. Destaque três poços na placa de microtítmetro (um para cada amostra de referência coletada na seção 3). Clique esquerdo em um poço, segure e arraste para três poços.
- Clique com o botão direito do mouse e selecione adicionar dados brutos. Na janela que aparece, navegue até o diretório contendo os três arquivos de referência da seção 3 e selecione-os ao mesmo tempo.
- Clique no próximo e escolha os parâmetros de processamento definidos na etapa 4.3. Clique no próximo e selecione os parâmetros de fluxo de trabalho definidos na etapa 4.4. Em seguida, clique no acabamento.
- Uma vez que os dados brutos, parâmetros de processamento e parâmetros de fluxo de trabalho tenham sido atribuídos a cada poço na placa, os poços parecerão azuis. Selecione os poços, clique com o botão direito do mouse e selecione processar os dados brutos mais recentes. Clique no canto inferior direito da janela para acompanhar o processamento dos dados. Uma vez que a mensagem Nenhum trabalho a ser executado aparece, o processamento é totalmente feito.
- Depois que o processamento dos dados estiver concluído, os poços na placa ficarão verdes. Clique com o botão direito do mouse nos poços e selecione exibir resultados do fluxo de trabalho. Uma janela separada será aberta para cada arquivo de dados de referência.
- Inspecione os dados para garantir que a maioria dos sinais de MS nos dados da amostra de referência foram mapeados com sucesso para peptídeos previstos a partir da digestão in-silico da proteína de interesse. Os peptídeos combinados serão coloridos de azul no espectro de saída(Figura 4). Clique duas vezes no filtro OK e verifique se a cobertura percentual é superior a 99%.
NOTA: Após o processamento, a saída de dados será salva automaticamente com a extensão do arquivo (raw_data_file_name_IA_final_peptide) no diretório especificado na etapa 4.5. - Importe a saída do software de proteômica para o software de processamento HDX(Tabela de Materiais)para limiares adicionais.
- Clique em Data no canto esquerdo da janela de software de processamento HDX. Clique em importar os resultados do PLGS e clique no ícone adicionar. Escolha os arquivos de dados processados a partir da etapa 4.9 navegando até o diretório apropriado.
- Clique em Next e especifique os seguintes parâmetros: íons consecutivos mínimos ≥ 2, erro de massa = 5 ppm e limiar de arquivo = 3. Clique no acabamento.
- Uma vez satisfeito com os parâmetros de limiar, salve o projeto HDX. Todos os dados HDX serão importados para este projeto para análise e exibição.
NOTA: As amostras trocadas por deutério descritas na próxima seção precisarão ser processadas com um fluxo de trabalho LC-MS idêntico. Portanto, antes de prosseguir com os ensaios HDX (seção 5), certifique-se de que a preparação da amostra (seção 2), o fluxo de trabalho LC-MS de baixo para cima (seção 3) e os fluxos de trabalho de processamento de dados (seção 4) estejam fornecendo a reprodutibilidade desejada e a cobertura sequencial da proteína alvo. Se algum desses processos precisar ser alterado para melhorar a cobertura, é aconselhável retornar à etapa 2.1, preparar amostras de referência frescas em triplicado e repetir as seções 2-4 (enquanto faz os ajustes necessários ao protocolo) para garantir que cada peptídeo possa ser reproduzido e detectado.
5. Realização de reações HDX
- Prepare o espaço de trabalho para reações HDX.
- O tampão de saciar a linha de fundo pré-alíquota em tubos de 0,5 mL devidamente rotulados. Prepare um tubo diferente para cada ponto de tempo, cada réplica, e cada estado bioquímico a ser analisado. Use o volume apropriado de tampão de saciar a partir da etapa 2.2 necessária para ajustar a leitura final do medidor de pH de uma porção de 50 μL da reação HDX a um valor de 2,3.
- Centrifufique brevemente as banheiras de 0,5 mL para transferir todo o tampão de saciar para o fundo do tubo. Coloque os tubos no gelo.
- Encha um pequeno Dewar com nitrogênio líquido e mantenha-se adjacente ao espaço de trabalho.
- Prepare as reações do HDX. Certifique-se de que há volume de reação suficiente para coletar o número desejado de pontos de tempo de troca (uma alíquota de 50 μL para cada ponto de tempo desejado). Colete pelo menos 4-5 pontos de tempo acima de 3-4 ordens de magnitude em escala de tempo (por exemplo, saciar tempos de 15 s, 60 s, 300 s [5 min], 1.800 s [30 min], e 14.400 s [4h] fornecem cobertura adequada da dinâmica de troca para a maioria das enzimas).
- Pré-misture todos os componentes deutados (menos enzima) da etapa 1.1 em D2O.
- Para que cada estado bioquímico seja examinado (enzima livre, enzima + ligante, enzima + inibidor, etc.), prepare reações HDX em pelo menos triplicado.
- Incubar as misturas de reação em um banho de água controlado pela temperatura a 25 °C por 10 minutos antes da adição da enzima.
NOTA: A enzima deve ser preparada como uma solução concentrada de estoque (~100-200 μM, passo 1.6) de modo a minimizar a adição de prótium no ensaio HDX. - Ao adicionar a enzima a uma concentração final de 1-5 μM, inicie o temporizador. Misture cuidadosamente e rapidamente a solução usando uma pipeta de 200 μL para garantir que a enzima seja distribuída uniformemente na amostra.
- Nos pontos de tempo de troca desejados, remova 50 alíquotas de μL da reação HDX e misture de forma rápida e uniforme com o tampão de sada frio pré-aliquoted em um tubo de 0,5 mL.
NOTA: Os volumes de mistura e o procedimento de mistura devem ser o mais precisos e reprodutíveis possíveis para garantir que a saciação final desejada pL de 2.3 seja alcançada rapidamente em todas as amostras. Manter o gelo de tampão de saciar ajudará a minimizar a troca de volta após a desnaturação da enzima. - Imediatamente após a saciar a amostra HDX, cubra o tubo e o flash congele em nitrogênio líquido.
- Continue coletando pontos de tempo até que todos os ensaios estejam concluídos e, em seguida, transfira amostras para o congelador -80 °C para armazenamento.
NOTA: Este pode ser um ponto de parada. Depois de coletar todos os pontos de tempo HDX, as amostras podem ser armazenadas a -80 °C até estarem prontas para análise de LC-MS. O ideal é que as reações do HDX triplicado sejam realizadas para todos os estados bioquímicos de interesse no mesmo dia. No mínimo, todas as reações de HDX para um determinado estado bioquímico devem ser executadas em paralelo no mesmo dia.
- Depois de coletar todos os pontos de tempo HDX saciados, submá-los ao fluxo de trabalho otimizado de baixo para cima LC-MS desenvolvido conforme descrito na etapa 3.3. Injete amostras de HDX em uma ordem aleatória com um número apropriado de espaços em branco entre as amostras para garantir que qualquer peptídeo seja mínimo.
NOTA: Os dados HDX não precisam ser coletados no modo MSE. Assim, o segmento de energia de alta colisão (etapa 3.3.7.2) deve ser removido do ciclo de plantão de MS. Esta deve ser a única alteração feita no fluxo de trabalho descrito na etapa 3.3. - Avalie a qualidade dos dados HDX como eles estão sendo coletados.
- Certifique-se de que os picos cromatográficos presentes no cromatograma de íon total das amostras de referência não deuteradas aparecem no mesmo tempo de retenção nas amostras desuteradas (como na Figura 3D−F).
- Certifique-se de que os espectros de massa somados em intervalos de tempo específicos de amostras de referência e deuterados mostram evidências de deuteração (ou seja, uma mudança no envelope isotópico de peptídeos individuais para valores mais elevados de m/z nas amostras desuteradas [Figura 6]).
6. Processamento de dados HDX
- Importe os dados HDX para o projeto HDX criado na etapa 4.11 clicando em Data | MS Arquivos na barra de ferramentas superior.
- Clique em Novo Estado e Nova Exposição conforme necessário para definir os estados bioquímicos (por exemplo, enzima livre, enzima + ligante, etc.) e tempos de exposição ao deutério, respectivamente, pertinentes à análise.
- Clique em New Raw para selecionar os arquivos de dados HDX a serem analisados. Atribua os tempos de troca apropriados e o estado bioquímico a cada arquivo de dados brutos importados.
NOTA: Os dados podem ser importados e processados em lotes, ou todos de uma só vez. Adicionar dados ao projeto não desfazerá qualquer análise que tenha sido realizada anteriormente dentro desse projeto.
- Uma vez que os arquivos de dados tenham sido adicionados, clique em terminar para iniciar o processamento de dados. Após um curto atraso, o software perguntará se o usuário deseja salvar os dados antes de continuar. Clique em sim.
NOTA: O processamento inicial pode levar até várias horas, dependendo de quantas amostras estão sendo analisadas, quantos peptídeos estão na lista final de peptídeos (etapa 4.10), do tamanho da janela cromatográfica e da frequência de aquisição espectral. - Se desejar, altere os parâmetros de processamento no menu Configuração para alterar os parâmetros de pesquisa de íons. Certifique-se de empregar os mesmos parâmetros de pesquisa de íons para todos os dados em um determinado projeto.
7. Análise e visualização dos dados HDX
NOTA: Uma vez concluído o processamento inicial dos dados brutos (etapa 6.2), o software de processamento HDX terá peptídeos localizados da lista de peptídeos (gerados na etapa 4.10) em cada um dos arquivos de dados brutos analisados. Uma vez que a distribuição de isótopos para um peptídeo na lista está localizada em um arquivo de dados bruto, o software de processamento HDX representa cada isótopo com um "stick" (como na Figura 6C−E). As intensidades relativas das varas para um determinado peptídeo são então usadas para calcular a absorção do deutério em relação ao espectro de referência. Embora o software de processamento HDX faça um trabalho admirável de atribuir corretamente "sticks" à maioria dos peptídeos, uma curadoria manual significativa dos valores de absorção do deutério ainda será necessária.
- Analisando valores de absorção de peptídeos de deutério
- Selecione o primeiro peptídeo na lista de peptídeos e abra o enredo espectral empilhado no menu Vistas. Role para cima e para baixo na janela do enredo de espectro empilhado para ver o espectro de massa para o peptídeo selecionado em função do tempo de troca de deutério(Figura 7D,E).
- Atribuir e não assinar varas conforme necessário usando cliques do mouse para garantir que a distribuição adequada do isótopo tenha sido localizada nos dados e que cada pico de isótopos tenha sido atribuído (varas atribuídas aparecerão azuis). Clicar em qualquer um dos espectros na trama espectral empilhada(Figura 7D,E) permitirá que o usuário atribua/não se insignirá varas na janela do visualizador de dados ativos(Figura 7C).
- Verifique as atribuições de cada estado de carga, toggling o estado de carga no topo da janela de parcela espectral empilhada.
- Repetir as etapas 7.1.1-7.1.3 para cada estado bioquímico de interesse. O estado bioquímico também pode ser alternado no topo da janela do enredo espectral empilhado. Para as medidas de diferença de absorção de deutério mais precisas, certifique-se de que as varas sejam atribuídas para o mesmo conjunto de estados de carga para cada estado bioquímico.
- Repetir as etapas 7.1.1−7.1.4 para cada peptídeo na lista de peptídeos.
- Verifique o desvio padrão dos valores de absorção de deutério de peptídeos usando o mapa de cobertura.
- Acesse o mapa de cobertura do menu Views, que exibe cada peptídeo na lista de peptídeos mapeados ao longo da sequência de aminoácidos da proteína de interesse(Figura 8C). Colorir os peptídeos de acordo com o desvio padrão relativo (unidades de Da).
- Procure visualmente o mapa por peptídeos outlier com desvio de alto padrão relativo. Clique nos peptídeos outlier no mapa de cobertura para preencher as parcelas espectrais empilhadas (Figura 8B) e janela de visualização de dados(Figura 8A) com o peptídeo de destino.
- Usando o enredo espectral empilhado, verifique cuidadosamente se todos os estados de carga e todos os pontos de tempo do peptídeo outlier têm varas apropriadamente atribuídas.
NOTA: Na maioria das vezes, peptídeos com grandes desvios padrão (>0.3 Da) têm picos isotópicos que não foram atribuídos adequadamente pelo software (como indicado na Figura 8B). A atribuição de quaisquer varas faltantes geralmente permitirá que o desvio padrão relativo de um peptídeo seja reduzido para <0.3 Da. - Esconda o peptídeo da lista se o desvio padrão relativo não puder ser reduzido a menos de 0,3 Da.
- Exportar dados de diferença HDX entre dois estados bioquímicos para mapeamento em um modelo estrutural da proteína de interesse.
- Mostre a diferença de interesse no mapa de cobertura. Clique com o botão direito do mouse no mapa de cobertura para exportar os dados de diferença para um arquivo .csv. Exporte os dados estaduais (em formato .csv) navegando para Data | Exportar Dados estaduais na barra principal de ferramentas.
NOTA: A formatação adequada dos dados de diferença e arquivos de dados estaduais é fornecida nas Informações de Suporte. - Importar os dados de diferença, os dados estaduais e o arquivo pdb da proteína de interesse emDeuteronômes 28. Escolha o intervalo de confiança de 99%, selecione ativar a somae processe os dados.
NOTA: O MATLAB deve ser instalado no PC para executar o Deuteronômes. O Deuteros usará as medidas de replicação no conjunto de dados para calcular o desvio padrão dos dados de absorção de cada peptídeo. Este desvio padrão será usado para definir o intervalo de confiança para troca significativa, que será exibido nas parcelas. - Em Opções PyMOL, selecione a captação de exportação | exportar para gerar um script Pymol para mapear regiões de diferença cambial significativa na estrutura pdb da proteína de interesse usando o software PyMOL.
NOTA: Usando o fluxo de trabalho descrito neste protocolo, o intervalo de confiança de 99% para uma diferença significativa de absorção de deutério para um determinado peptídeo em um único ponto de tempo é tipicamente 0,3-0,5 Da. O intervalo de confiança de 99% para a diferença somada em todos os pontos de tempo de câmbio é tipicamente de 0,7-1,0 Da.
- Mostre a diferença de interesse no mapa de cobertura. Clique com o botão direito do mouse no mapa de cobertura para exportar os dados de diferença para um arquivo .csv. Exporte os dados estaduais (em formato .csv) navegando para Data | Exportar Dados estaduais na barra principal de ferramentas.
Resultados
É necessário avaliar a qualidade da digestão proteolítica e a reprodutibilidade do fluxo de trabalho para cada conjunto de injeções amostrais. Assim, antes de realizar ensaios HDX-MS, é essencial estabelecer condições efetivas para a proteólise da proteína de interesse, para a separação de peptídeos utilizando cromatografia líquida de fase inversa e mobilidade de íons de fase de gás, e para a detecção de peptídeos utilizando EM. Para isso, as amostras de referência para a proteína de interesse (cole...
Discussão
O fluxo de trabalho HDX-MS apresentado neste protocolo fornece uma plataforma notavelmente robusta para mapear a distribuição espacial de elementos estruturalmente dinâmicos em proteínas e para investigar como essas dinâmicas mudam em resposta à perturbação (ligação de ligante, mutagênese enzimática, etc.). O HDX-MS possui várias vantagens distintas sobre outras abordagens de biologia estrutural que são comumente usadas para investigar a dinâmica conformacional. Mais notavelmente, apenas pequenas quantidad...
Divulgações
Não temos nada para revelar.
Agradecimentos
Este trabalho foi apoiado pelo Conselho de Pesquisa em Ciências Naturais e Engenharia do Canadá, pela Fonds de Recherche du Quebec Nature et Technologie, pela Canadian Foundation for Innovation e pela McGill University.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| [glu-1]-fibrinopeptide B (Glu-Fib) | BioBasic | NA | |
| 0.5 mL Amicon Ultracel 10k centrifugal filtration device (Millipore) | Milipore Sigma | UFC501096 | |
| acetonitrile | Fisher | A955-1 | |
| AMP-PNP | SIGMA | A2647-25MG | |
| ATP | SIGMA | a2383-5G | |
| D2O | ALDRICH | 435767-100G | |
| formic acid | Thermo Fisher | 28905 | |
| guanidine-HCl | VWR | 97063-764 | |
| HEPES | Fisher | BP310-1 | |
| Magnesium chloride | SiGMA-Aldrich | 63068-250G | |
| Potassium chloride | BioBasic | PB0440 | |
| potassium phosphate | BioBasic | PB0445 | |
| TCEP Hydrochloride | TRC Canada | T012500 | peptide was synthesized upon request |
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| software | |||
| Deuteros | Andy M C Lau, et al | version 1.08 | |
| DynamX | Waters | version 3.0 | |
| MassLynx | Waters | version 4.1 | |
| Protein Lynx Global Server (PLGS) | Waters | version 3.0.3 | |
| PyMOL | Schrödinger | version 2.2.2 | |
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| Instrument and equipment | |||
| ACQUITY UPLC BEH C18 analytical Column | Waters | 186002346 | |
| ACQUITY UPLC BEH C8 VanGuard Pre-column | Waters | 186003978 | |
| ACQUITY UPLC M-Class HDX System | Waters | ||
| HDX Manager | Waters | ||
| microtip pH electrode | Thermo Fisher | 13-620-291 | |
| Waters Enzymate BEH column or Pepsin solumn | Waters | 186007233 | |
| Waters Synapt G2-Si | Waters |
Referências
- Karplus, M., McCammon, J. A. Molecular dynamics simulations of biomolecules. Nature Structural & Molecular Biology. 9 (9), 646-652 (2002).
- Campbell, E., et al. The role of protein dynamics in the evolution of new enzyme function. Nature Chemical Biology. 12 (11), 944-950 (2016).
- Tokuriki, N., Tawfik, D. S. Protein dynamism and evolvability. Science. 324 (5924), 203-207 (2009).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chemical Society Reviews. 40 (3), 1224 (2011).
- Katta, V., Chait, B. T. Conformational changes in proteins probed by hydrogen-exchange electrospray-ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 5 (4), 214 (1991).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Science. 2 (4), 522 (1993).
- Smith, D. L., Deng, Y., Zhang, Z. Probing the non-covalent structure of proteins by amide hydrogen exchange and mass spectrometry. Journal of Mass Spectrometry. 32 (2), 135-146 (1997).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Xiao, K., et al. Revealing the architecture of protein complexes by an orthogonal approach combining HDXMS, CXMS, and disulfide trapping. Nature Protocols. 13 (6), 1403-1428 (2018).
- Smith, D. L., Deng, Y., Zhang, Z. Probing the non-covalent structure of proteins by amide hydrogen exchange and mass spectrometry. Journal of Mass Spectrometry. 32 (2), 135-146 (1997).
- Masson, G. R., et al. Recommendations for performing, interpreting and reporting hydrogen deuterium exchange mass spectrometry (HDX-MS) experiments. Nature Methods. 16 (7), 595-602 (2019).
- Liu, J., et al. An Efficient Site-Specific Method for Irreversible Covalent Labeling of Proteins with a Fluorophore. Scientific Reports. 5, 16883 (2015).
- Marion, D., et al. Overcoming the overlap problem in the assignment of proton NMR spectra of larger proteins by use of three-dimensional heteronuclear proton-nitrogen-15 Hartmann-Hahn-multiple quantum coherence and nuclear Overhauser-multiple quantum coherence spectroscopy: application to interleukin 1.beta. Biochemistry. 28 (15), 6150-6156 (1989).
- Balasubramaniam, D., Komives, E. A. Hydrogen-exchange mass spectrometry for the study of intrinsic disorder in proteins. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1834 (6), 1202-1209 (2013).
- Goswami, D., et al. Time window expansion for HDX analysis of an intrinsically disordered protein. Journal of the American Society for Mass Spectrometry. 24 (10), 1584-1592 (2013).
- Keppel, T. R., Howard, B. A., Weis, D. D. Mapping Unstructured Regions and Synergistic Folding in Intrinsically Disordered Proteins with Amide H/D Exchange Mass Spectrometry. Biochemistry. 50 (40), 8722-8732 (2011).
- Mitchell, J. L., et al. Functional Characterization and Conformational Analysis of the Herpesvirus saimiri Tip-C484 Protein. Journal of Molecular Biology. 366 (4), 1282-1293 (2007).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Hydrogen/deuterium exchange mass spectrometry with top-down electron capture dissociation for characterizing structural transitions of a 17 kDa protein. Journal of the American Chemical Society. 131 (35), 12801-12808 (2009).
- Habibi, Y., Uggowitzer, K. A., Issak, H., Thibodeaux, C. J. Insights into the Dynamic Structural Properties of a Lanthipeptide Synthetase using Hydrogen-Deuterium Exchange Mass Spectrometry. Journal of the American Chemical Society. 141 (37), 14661-14672 (2019).
- Hebling, C. M., et al. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Analytical Chemistry. 82 (13), 5415-5419 (2010).
- Duc, N. M., et al. Effective application of bicelles for conformational analysis of G protein-coupled receptors by hydrogen/deuterium exchange mass spectrometry. Journal of the American Society for Mass Spectrometry. 26 (5), 808-817 (2015).
- Canul-Tec, J. C., et al. Structure and allosteric inhibition of excitatory amino acid transporter 1. Nature. 544 (7651), 446-451 (2017).
- Reading, E., et al. Interrogating Membrane Protein Conformational Dynamics within Native Lipid Compositions. Angewandte Chemie International Edition. 56 (49), 15654-15657 (2017).
- Hentze, N., Mayer, M. P. Analyzing protein dynamics using hydrogen exchange mass spectrometry. Journal of Visualized Experiments. (81), e50839 (2013).
- Lento, C., et al. Time-resolved ElectroSpray Ionization Hydrogen-deuterium Exchange Mass Spectrometry for Studying Protein Structure and Dynamics. Journal of Visualized Experiments. (122), e55464 (2017).
- Schowen, K. B., Schowen, R. L. Solvent isotope effects on enzyme systems. Methods in Enzymology. 87, 551-606 (1982).
- Lau, A. M. C., Ahdash, Z., Martens, C., Politis, A. Deuteros: software for rapid analysis and visualization of data from differential hydrogen deuterium exchange-mass spectrometry. Bioinformatics. 35 (7), 3171-3173 (2019).
- Houde, D., Berkowitz, S. A., Engen, J. R. The utility of hydrogen/deuterium exchange mass spectrometry in biopharmaceutical comparability studies. Journal of Pharmaceutical Sciences. 100 (6), 2071 (2011).
- Burkitt, W., O'Connor, G. Assessment of the repeatability and reproducibility of hydrogen/deuterium exchange mass spectrometry measurements. Rapid Communications in Mass Spectrometry. 22 (23), 3893-3901 (2008).
- Rob, T., Gill, P. K., Golemi-Kotra, D., Wilson, D. J. An electrospray ms-coupled microfluidic device for sub-second hydrogen/deuterium exchange pulse-labelling reveals allosteric effects in enzyme inhibition. Lab on a Chip. 13, 2528-2532 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados