Method Article
Multiplexação automatizada da amostra usando rotulagem isotópica precursora combinada e marcação isobárica (cPILOT)
Neste Artigo
Resumo
A rotulagem isotópica precursora combinada e a marcação isobárica (cPILOT) é uma estratégia de multiplexação de amostra aprimorada que é capaz de aumentar o número de amostras que podem ser analisadas simultaneamente com tags isobáricas disponíveis. A incorporação de uma plataforma robótica aumentou consideravelmente o rendimento experimental, a reprodutibilidade e a precisão quantitativa.
Resumo
Introduzimos um fluxo de trabalho de proteômica quantitativa de alto rendimento, rotulagem isotópica precursora combinada e marcação isobárica (cPILOT) capaz de multiplexar até 22 ou 24 amostras com etiquetas de massa tandem ou etiquetas isobáricas N,N-dimethyl leucina isobaric, respectivamente, em um único experimento. Este multiplexagem de amostra aprimorado reduz consideravelmente os tempos de aquisição de espectrometria de massa e aumenta a utilidade dos reagentes isobáicos comerciais caros. No entanto, o processo manual de manuseio de amostras e etapas de pipetação na estratégia pode ser trabalhoso, demorado e introduzir perda de amostra e erro quantitativo. Essas limitações podem ser superadas através da incorporação da automação. Aqui transferimos o protocolo manual cPILOT para um dispositivo de manuseio líquido automatizado que pode preparar grandes números de amostra (ou seja, 96 amostras) em paralelo. No geral, a automação aumenta a viabilidade e a reprodutibilidade do cPILOT e permite um amplo uso por outros pesquisadores com dispositivos de automação comparáveis.
Introdução
A espectrometria de massa (mS) é uma ferramenta de pesquisa indispensável na identificação de biomarcadores específicos da doença, na compreensão da progressão da doença e na criação de leads para o desenvolvimento terapêutico. Isso pode ser obtido a partir de uma série de amostras clínicas relacionadas à doença, como soro/plasma sanguíneo, fluidos proximais e tecidos1,2. A descoberta e validação de biomarcadores de proteômica ganharam recentemente significativa consideração devido ao poder das estratégias de multiplexação da amostra3,4. Multiplexação amostral é uma técnica que permite a comparação simultânea e quantificação de duas ou mais condições amostrais dentro de uma única injeção de MS5,6. O multiplexing amostral é obtido através de peptídeos de barcodificação ou proteínas de múltiplas amostras com marcas químicas, enzimáticas ou metabólicas e obtenção de informações de MS de todas as amostras em um único experimento em MS ou MS/MS. Entre as etiquetas isobáricas disponíveis estão reagentes isobáicos de marcação (iTRAQ), tags de massa tandem comercial (TMT) e em casa sintetizados reagentes isobáricos N,N-dimethyl leucina (DiLeu) com capacidades de até 16-plex7 e 21-plex8, respectivamente.
A rotulagem isotópica precursora combinada e a marcação isobárica (cPILOT) é uma tecnologia de multiplexação de amostra aprimorada. cPILOT combina rotulagem isotópica de peptídeo N-termini com leve [-(CH3)2] e pesado [−(13C2H3)2] isótopos em pH baixo (∼2,5), que mantém o resíduo de liseina disponível para rotulagem isobárica de pH (8,5) DiLeu, ou iTRAQ marcando3,9,10,11,12,13,14. O esquema de rotulagem dupla da estratégia cPILOT é retratado na Figura Suplementar 1 com duas amostras usando um peptídeo de exemplo. A precisão e precisão da quantificação baseada em TMT no nível MS2 pode ser comprometida devido à presença de íons co-isolados e co-fragmentados considerados como efeito de interferência15. Essa limitação em proporções de íons de repórter imprecisas pode ser superada com a ajuda de espectrômetros de massa tribrid Orbitrap. Por exemplo, o efeito de interferência pode ser superado isolando um pico em um par dimetilado no nível MS1 no espectrômetro de massa, submetendo o pico leve ou pesado à fragmentação de MS2 na armadilha de íons lineares e, em seguida, submetendo o fragmento mais intenso de MS2 para HCD-MS3 para obter informações quantitativas. A fim de aumentar as chances de seleção dos peptídeos sem aminas de liseina disponíveis para geração de íons repórteres, uma aquisição seletiva de MS3 com base no fragmento y-1 também pode ser usada e é uma abordagem que pode resultar em uma maior porcentagem de peptídeos quantificáveis com cPILOT9. A combinação de rotulagem leve e pesada aumenta as capacidades de multiplexação da amostra em um fator de 2x ao alcançado com tags isobáricas individuais. Recentemente, usamos o cPILOT para combinar até 24 amostras em um único experimento com reagentes DiLeu16. Além disso, o cPILOT tem sido usado para estudar modificações oxidativas pós-translacionais14, incluindo nitração proteica17, outros proteomes globais9, e demonstrou aplicações em várias amostras de tecido em um modelo de camundongo da doença de Alzheimer11.
A preparação robusta da amostra é um passo crítico em um experimento cPILOT e pode ser demorado, trabalhoso e extenso. Multiplexing de amostra aprimorado requer tubulação extensiva e pessoal de laboratório altamente qualificado, e há vários fatores que podem influenciar fortemente a reprodutibilidade do experimento. Por exemplo, o manuseio cuidadoso das amostras é necessário para garantir tempos de reação semelhantes para todas as amostras e manter o pH tampão adequado para amostras fracastiladas leves e pesadas. Além disso, a preparação manual de dezenas a centenas de amostras pode introduzir um alto erro experimental. Portanto, para reduzir a variabilidade de preparação da amostra, melhorar a precisão quantitativa e aumentar o throughput experimental, desenvolvemos um fluxo de trabalho automatizado cPILOT. A automação é alcançada utilizando um dispositivo de manuseio líquido robótico que pode completar muitos aspectos do fluxo de trabalho (Figura 1). A preparação da amostra da quantificação proteica para a rotulagem de peptídeos foi realizada em um manipulador líquido automatizado. O manipulador líquido automatizado é integrado a um aparelho de pressão positivo (PPA) para trocas de buffer entre as placas de extração em fase sólida (SPE), shaker orbital e um dispositivo de aquecimento/resfriamento. A plataforma robótica contém 28 locais de deck para acomodar placas e buffers. Existem dois pods com um gripper para transferir as placas dentro dos locais do convés: uma cabeça de tubulação de volume fixo de 96 canais (5-1100 μL) e sondas de volume variável de 8 canais (1-1000 μL). A plataforma robótica é controlada usando um software. O usuário precisa ser treinado profissionalmente antes de usar o manipulador líquido robótico. O presente estudo se concentra na automatização do fluxo de trabalho manual cPILOT, que pode ser intensivo em mão-de-obra para o processamento de mais de 12 amostras em um único lote. A fim de aumentar o rendimento da abordagem cPILOT11,transferimos o protocolo cPILOT para um manipulador líquido robótico para processar mais de 10 amostras em paralelo. A automação também permite reações semelhantes para cada amostra em paralelo durante várias etapas do processo de preparação da amostra, o que exigiu que usuários altamente treinados alcançassem durante o CPILOT manual. Este protocolo se concentra na implementação do dispositivo de manuseio líquido automatizado para realizar o cPILOT. O presente estudo descreve o protocolo para o uso deste sistema automatizado e demonstra seu desempenho usando uma análise de 22 plex "prova de conceito" de homogeneizadores hepáticos de camundongos.
Protocolo
Todos os protocolos animais foram aprovados pelo Comitê Institucional de Cuidados e Uso de Animais da Universidade de Pittsburgh. Um rato de controle masculino (C57/BLJ) foi comprado comercialmente e alojado na Divisão de Recursos Animais laboratoriais da Universidade de Pittsburgh. Os camundongos foram alimentados com laboratório de roedores padrão chow ad libitum e mantidos em um ciclo claro/escuro de 12 horas. O tecido hepático foi colhido e armazenado a -80 °C.
1. Extração de proteína
NOTA: Estas etapas são executadas manualmente.
- Lave o fígado do rato (100 mg) com soro fisiológico e homogeneizado com 500 μL de ureia de 8 M utilizando um homogeneizador mecânico utilizando contas de matriz de lise A para 4 m/s para 20 s.
NOTA: Neste estudo, os inibidores de protease ou fosfatase não foram adicionados, mas podem ser adicionados ao buffer, se necessário, com base no experimento. Além disso, as etapas de extração de proteínas podem ser ajustadas de acordo para vários tipos de amostra. - Transfira o tecido homogeneizar para um novo tubo de microcentrífuga, enxágüe e combine os tubos de lise com 100-500 μL de PBS com ureia de 8 M.
- Centrifugar os tecidos homogeneizados (12.800 x g, 4 °C e 15 min) e coletar o sobrenante.
NOTA: O restante das etapas são realizadas no manipulador líquido robótico disponível no laboratório do usuário. O manipulador líquido deve ser capaz de aspirar e distribuir buffers de e para os tipos de placa especificada da ANCI, com participação mínima do operador. - Determine a concentração proteica utilizando um ensaio de ácido bicinchonínico (BCA) de acordo com as instruções do fabricante.
- Ligue o PPA, o dispositivo de aquecimento/resfriamento e as bombas de vácuo e conecte todos os acessórios com o manipulador líquido. O manipulador líquido mostra uma luz azul uma vez que está conectada ao computador e pronta para operar.
- Para a placa de reagente 1 (96 placas de bem), adicione 30 μL de 25 mM 1,4-dithiothiothreitol (DTT), 25 mM 30 μL de cisteína e 25 mM 30 μL de iodoacetamida (IAA) para as linhas 1, 2 e 3, respectivamente.
- Adicione 8 M de ureia e 20 mM Tris com 10 mM CaCl2 (pH 8.2) a uma placa de reservatório. Adicione 500 μL de ácido fórmico de 5% a 2 mL de placa de poço profundo, 20 μL de trippsina para a linha 1 da placa de trippsina e coloque no local do deck conforme especificado pelo método.
NOTA: IAA e trippsina foram adicionados à placa de 96 poços antes de adicionar à amostra. - Aliquot 300 μL do fígado homogeneizar em um tubo de 500 μL e colocar em uma placa de poço de profundidade de 2 mL e colocar a 4 °C até o início do protocolo. Para este experimento, foram geradas 22 alíquotas a partir do monogênato hepático.
NOTA: Adicione um padrão de controle de qualidade interna (por exemplo, caseína alfa bovina ou outra proteína exógena) com a razão 1 μg padrão: amostra de proteína de 100 μg. - Defina o volume de buffers a serem adicionados às amostras por nomes variáveis e valor conforme tabela 1. Com base no BCA, digite o volume de amostra e buffer de normalização (8 M de ureia) em uma planilha e conecte-se ao software(Tabela 2).
NOTA: Uma diluição adicional com tampão pode ser necessária se a concentração de proteína for muito alta. As concentrações resultantes para amostra do tecido hepático foram de 10 μg/μL. O volume de tampões foi otimizado para 100 μg de proteína. - Remova os tubos de amostra de 4 °C, coloque na localização do convés especificado do manipulador líquido automatizado e deixe aquecer por 10 minutos.
- Abra o método e siga as instruções para colocar as dicas necessárias (1070, 90 e 230 μL), e labware nas posições desejadas. Uma vez que todos os labware e dicas estejam no lugar, cruze o layout final do deck e clique em Seguir para continuar o protocolo.
NOTA: A configuração guiada informa o usuário a adicionar um volume necessário de buffer ao labware específico, dependendo do protocolo e do local do deck a ser mantido.
2. Redução da amostra, alquilação e digestão
- Carregue 230 pontas μL e aspire 90 μL de 8 M de ureia do reservatório e dispense para a linha 1 da placa de poço de profundidade de 2 mL preta. Descarregue as pontas. Repita esta etapa até que a ureia de 8 M seja adicionada a todos os poços correspondentes às 22 amostras.
NOTA: O tampão de desnaturação desenrola a estrutura tridimensional da proteína para produzir a estrutura primária para que a trippsina possa agir e quebrar a proteína efetivamente. A pipeta de 8 canais pode aspirar diferentes volumes para os 8 canais e pode dispensar para diferentes locais no labware. Nesta etapa todos os canais aspiraram o mesmo volume definido no software. - Depois de adicionar o tampão de desnaturação, carregue as pontas de 90 μL e aspire 10 μL (corresponde a 100 μg) de homogeneizar o fígado do rato usando a pipeta de 8 canais na placa de poço de profundidade preta de 2 mL. Descarregue as pontas. Repita esta etapa até que todas as 22 amostras sejam transferidas.
- Após cada transferência, realize uma etapa de mistura para aspirar e dispensar 50 μL do conteúdo do poço três vezes para garantir a mistura da proteína com o tampão para exibir uma razão de 1:1.
NOTA: Remova a amostra homogeneizante do estoque e coloque em -80 °C. - Para reduzir as proteínas desnaturadas, carregue uma linha de 90 pontas de μL e aspire 3 μL de DTT da placa de reagente 1. Dispense dtt para as linhas 1 e 2 da placa preta de 2 mL de profundidade e descarregue as pontas.
NOTA: A relação de molar DTT é mantida em 1:40 para redução de ligações de dissulfeto. O cálculo para a razão molar é baseado na massa proteica da albumina de soro bovino (BSA), que é 66,5 x 103 g/mol. - Sele a placa de amostra com papel alumínio e incubar a 37 °C por 600 s a 300 rpm.
- Adicione 30 μL de 0,25 M IAM para remar 3 da placa de reagente 1 pouco antes do uso e coloque no convés. Desemsarar a placa de amostra e retornar ao convés.
NOTA: O IAM é sensível à luz. - Carregue uma linha de 90 pontas μL, aspire 6 μL de IAM da placa de reagente 1 na linha 3 e dispense a linha 1 da placa de amostra. Descarregue as pontas.
NOTA: A relação de molar IAM é mantida em 1:80 para cada amostra. Esta reação deve ser feita no escuro. - Sele a placa de amostra e incubar a 4 °C por 30 min a 300 rpm no dispositivo de aquecimento/resfriamento.
NOTA: A vedação da placa é realizada para evitar que a amostra leve, evaporação e derramamento do poço. - Dessempegue a placa de amostra e carregue uma linha de 90 pontas μL e aspire 5 μL de cisteína da placa de reagente 1 na linha 2. Dispense as linhas 1 e 2 da placa de amostra e descarregue as pontas. Incubar em temperatura ambiente por 30 minutos.
NOTA: A proteína: A relação molar L-cisteína é mantida em 1:40. - Coloque a placa de amostra no agitador orbital para realizar um shake cronometrado de 1800 rpm por 30 minutos.
- Adicione 800 μL de tampão Tris de 20 mM com 10 mM CaCl2 (pH 8.2) a cada poço da placa de amostra para diluir a concentração de ureia para 2 M. Descarregue as pontas.
NOTA: A atividade de trippsina é dificultada em concentrações de ureia mais altas e, portanto, deve ser reduzida para menos de 2 M. Este estudo foi realizado com uma relação molar de proteína para trippsina de 50: 1 para 14 h. - Adicione 20 μL de trippsina à linha 1, coluna 1-12 de uma placa de 96 poços e coloque em um local especificado no convés.
- Carregue uma linha de 90 pontas μL, aspire 2 μL de trippsina da linha de placa de trypsin 1 e dispense para as linhas 1 e 2 da placa de amostra. Descarregue as pontas.
- Sele a placa e incubar por 14 h a 37 °C a 600 rpm no dispositivo de aquecimento/resfriamento.
- Após a incubação, dessemça a placa de amostra e adicione 5% de ácido fórmico à linha 3, coluna 1-12 de uma placa de coleta de poços profundos e coloque-a no convés especificado.
- Pare a digestão adicionando 150 μL de ácido fórmico de 5% da linha 3 da placa de ácido fórmico e dispense a placa de amostra nas linhas 1 e 2. Descarregue as pontas.
3. Desalting passo 1
- Desalque os peptídeos com placa SPE contendo 20 mg de material Targa C-18. Corrija o volume para cada troca de buffer como 600 μL e fixe a pressão em 100 mbar.
- Carregar 1070 pontas μL e aspirar 600 μL de acetonitrilo e dispensar as linhas 1 e 2 de placa SPE. Coloque a placa SPE no PPA e aplique pressão.
- Usando as mesmas pontas, aspire 600 μL de acetonitrilo e dispense as linhas 1 e 2 da placa SPE. Coloque a placa SPE no PPA e aplique pressão.
NOTA: O fluxo pode ser drenado para resíduos usando uma bomba de sucção. - Carregar 1070 pontas μL e aspirar 600 μL de Buffer A (100 % de água em 0,1 % de ácido fórmico) e dispensar as linhas 1 e 2 da placa SPE. Coloque a placa SPE no PPA e aplique pressão.
NOTA: Se o volume do buffer não reduzir significativamente tente aumentar a pressão ou o tempo. - Carregar 2 linhas de 1070 pontas μL, aspirar 534 μL de amostras digeridas e dispensar as linhas 1 e 2 de placa SPE. Coloque a placa SPE no PPA e aplique pressão. Repita esta etapa até que todas as amostras estejam carregadas.
NOTA: Uma vez que o volume total após a adição de ácido fórmico será de 1068 μL e as amostras foram adicionadas em duas passagens. - Carregue 2 linhas de pontas usadas de 1070 μL, aspire 600 μL de Buffer A e dispense para as linhas 1 e 2 de placa SPE. Coloque a placa SPE no PPA e aplique pressão.
- Repita o passo acima uma vez para limpar a amostra.
- Carregar 1 linha de 1070 pontas μL, aspirar 600 μL de ACN: Água (60:40) e dispensar as linhas 1 e 2 de placa SPE. Coloque a placa SPE em cima de uma placa de coleta para elutar os peptídeos usando PPA e aplique pressão.
- Repita o passo acima para eluto os peptídeos na placa de coleta. Seque a placa de coleta para secar e armazene a -80° C até que seja mais processamento.
4. Rotulagem de dimetilação (peptídeo N-termini)
- Coloque as dicas necessárias (1070, 90 e 230 μL), e labware nas posições desejadas no software. Uma vez que todos os labware e dicas estejam no lugar, cruze o layout final do deck e clique em Seguir para continuar o protocolo.
- À placa de reagente 2, adicione 450 μL de ácido acético de 1%, 50 μL de formaldeído leve (CH2O), 50 μL de formaldeído pesado(13CD2O) e 150 μL de ácido fórmico para as linhas 1, 2, 3 e 4, respectivamente.
- À placa de reagente 3 adicione 50 μL de CB leve, 50 μL de CB pesado, para as linhas 1 e 2 e 50 μL de Amônia para a linha 3 e 4.
- Carregue 2 linhas de 230 pontas μL e aspire 100 μL de ácido acético de 1% da linha 1 da placa de reagente 2 e dispense aos peptídeos secos na placa de amostra 2 (placa de coleta da etapa de desalamento) linhas 1 e 2 e realize um tremor cronometrado por 5 min a 1800 rpm.
- Carregar 90 pontas μL e aspirar 16 μL de 60 mM (4%) CH2O (37% wt/v) da linha 2 da placa de reagente 2 e dispense a linha 1 da placa de amostra. Descarregue as pontas.
- Carregar 1 linha de 90 μL pontas e aspirar 16 μL de 60 mM (4%) 13 CD2O (20% wt/v) da linha 3 da placa de reagente 2 e dispense para a linha 2 da placa de amostra. Descarregue as pontas.
NOTA: Neste estudo, a linha 1 corresponde à luz e a linha 2 corresponde a amostras de dimetilados pesados (Ver Figura 1). - Carregar 2 linhas de 90 μL pontas e aspirar 16 μL de 24 mM NaBH3CN e 24 mM NaBD3CN da linha 1 e 2 da placa de reagente 3 e dispensar as linhas 1 e 2, respectivamente da placa de amostra.
- Descarregue as pontas e realize um shake cronometrado por 15 min a 1800 rpm usando o agitador orbital.
NOTA: A adição de cianoboroidride de sódio inicia a reação, portanto, para reduzir a variabilidade entre os experimentos esta etapa é realizada uma vez por lote. - Carregar 2 linhas de 90 μL pontas e aspirar 32 μL de amônia de 1% (~28-30% v/v) das linhas 3 e 4 da placa de reagente 3 e dispensar as linhas 1 e 2, respectivamente, da placa de amostra 2. Descarregue as pontas.
NOTA: A adição de amônia interrompe a reação, portanto, para reduzir a variabilidade entre os experimentos esta etapa é realizada uma vez por lote. - Combine volumes iguais de peptídeos dimetilados leves e pesados (1:1) a uma nova placa de poço de 2 mL para desalar.
NOTA: Neste estudo, 90 μL do conteúdo do poço da linha 1 foi combinado com 90 μL de linha 2 da placa de amostra 2 para uma nova placa de coleta de 2 mL (Placa de Amostra 3). A razão da leve: a mistura pesada depende do protocolo experimental, neste estudo foi utilizada uma razão de 1:1. - Carregue 2 linhas de 230 pontas μL e aspire 32 μL de 5% de ácido fórmico às amostras combinadas e realize um tremor cronometrado para 30 s a 1800 rpm.
NOTA: A eficiência da dimetilação depende do pH da mistura de reação e qualquer alteração no pH tampão resultará na rotulagem incompleta do peptídeo N-termini. A eficiência da dimetilação deve ser superior a 97% quando pesquisada como modificação dinâmica no peptídeo N-termini. Neste estudo, a eficiência de rotulagem de peptídeos leves e pesados foi de 99,7% e 99,5%, respectivamente.
5. Desalting passo 2
- Realize a desaltação de amostra semelhante ao desalto passo 1 para as amostras combinadas.
- Seque as amostras na placa de amostra usando um vac de velocidade e armazene a -80 °C até um processamento adicional.
6. Marcação isobárica (resíduos de Lys)
- Siga a configuração guiada do labware e coloque as dicas necessárias (1070, 90 e 230 μL), com buffers apropriados. Uma vez que todos os labware e dicas estejam no lugar, cruze o layout final do deck e clique em Seguir para continuar o protocolo.
- Adicione 250 μL de 100 mM triethyl amônio bicarbonato (TEAB), 30 μL de hidroxilamina (10% w/v) às linhas 1 e 2 de placa de reagente 4. Coloque os tubos TMT na placa de poço profundo de 2 mL, conforme a planilha na Tabela 3.
- Mantenha um tubo vazio de 1,5 mL em um suporte de rack de tubo para agrupar os peptídeos marcados. Coloque a placa de amostra seca na placa de processamento P9 e TMT em P14.
- Para reconstituir os peptídeos, carregue 230 pontas μL e aspire 100 μL de 100 mM triethyl amônio bicarbonato (TEAB) tampão (pH ~8,5) da linha 1 na placa TEAB e dispense aos peptídeos secos na linha 3 da placa de amostra 3. Coloque a placa no agitador orbital por 30 s a 1800 rpm.
NOTA: Reconstituir 100 μg de peptídeos dimetilados a uma concentração de 1 μg/μL. - Carregue 90 pontas μL e aspire 10 μL de acetonitrilo anidro de H12 da placa TMT e dispense em cada um dos tubos TMT secos.
- Remova os tubos TMT, vórtice e gire rapidamente os tubos e retorne ao convés em placa profunda.
- Carregar 1 linha de 90 pontas μL e aspirar 12,5 μL dos peptídeos dimetilados combinados e dispensar para a linha 1 da placa de processamento TMT. Descarregue as pontas.
NOTA: A razão TMT: peptídeo foi mantida em 1:8 para este experimento. - Carregue 1 linha de pontas de 90 μL e aspire 10 μL de TMT e dispense para a linha 1 da placa de processamento TMT. Descarregue as pontas, realize um shake cronometrado por 1 hora a 1800 rpm.
- Carregar 1 linha de 90 μL pontas e aspirar 8 μL de hidroxilamina (10% w/v) da linha 2 na placa TEAB e dispensar para a linha 1 da placa de processamento TMT. Descarregue as pontas, realize um tremor cronometrado por 15 min a 1800 rpm.
- Combine 30,5 μL dos peptídeos rotulados TMT da placa de processamento TMT ao tubo de processamento de 1,5 mL. Descarregue as dicas após cada transferência.
- Remova o tubo com as amostras agrupadas e seque para evaporar a acetonitrila. Reconstituir peptídeos em 0,2 mL de água em ácido fórmico de 0,1 % e retornar ao convés.
NOTA: A eficiência de rotulagem da marcação isobárica também é específica do pH e a eficiência de rotulagem deve estar acima de 98% por instruções do fabricante. Neste estudo, a eficiência de rotulagem TMT dos peptídeos leves e pesados foi de 99,4% e 99,5%, respectivamente.
7. Passo desalhente
- Realize a desaltação da amostra semelhante ao desalto 1 para uma amostra.
- Seque as amostras e armazene em -80 °C até a análise.
8. Cromatografia Líquida -Espectrometria de Massa Tandem (LC-MS/MS) e MS3
- Reconstitua os peptídeos na água de grau MS com FA de 0,1% para obter ~1 μg/μL de concentração. Filtrar amostras com tubos de microcentrifuuge contendo um filtro de 0,65 μm. Peptídeos centrífugas a 12.000 x g por 3 min e coloque o fluxo através de um frasco de amostrador automático.
NOTA: A concentração de peptídeos pode ser confirmada nesta fase, se desejar. Os peptídeos precisariam ser reconstituídos em água de grau LC-MS e sujeitos a um ensaio de peptídeo bca. Neste estudo, o ensaio de peptídeo BCA não foi realizado e todas as quantidades de peptídeo foram baseadas no ensaio inicial da proteína BCA. - Prepare os buffers de fase móvel da seguinte forma: 100% (v/v) água com FA 0,1% (A) e 100% ACN com FA (B) de 0,1%.
- Injete 1 μL de amostra em uma coluna de armadilha embalada com 2 cm de material C18 (3 μm, 100 Å tamanho de poros).
NOTA: A limpeza da amostra na armadilha é a seguinte: 10 min, 100% A; 2 μL/min utilizando um sistema de cromatografia líquida 2D. - Execute o método de separação analítica. Use uma coluna capilar de sílica fundida de 100 μm x 26 cm de ponta puxada com laser embalada com material C18 (2,5 μm, 100 Å). O gradiente é: 0-10 min, 10% B; 10-30 min, 10-15% B; 30-75 min, 15-30% B; 75-88 min, 30-60% B; 88-92 min, 60-90% B; 92-99 min, 90% B; 99-100 min, 90-10% B, 100-120 min, 10% B; 300 nL/min, 120 min.
- Execute a aquisição de dados para o espectrômetro de massa enquanto o método de separação analítica estiver em execução.
- Use os seguintes parâmetros para a pesquisa em MS: 375-1.500 m/z,resolução de 120.000, tempo de ciclo 3 s (velocidade máxima), controle automático de ganho (AGC) alvo 4.0e5, tempo máximo de injeção de 50 ms.
- Use os seguintes parâmetros para CID-MS/MS com armadilha de íon: varreduras de aquisição dependente de dados (DDA) por resultado, largura de isolamento de 2 m/z, energia de colisão normalizada de 35%, 0,25 de ativação q, tempo de ativação de 10 ms, 1,0e4 AGC, tempo máximo de injeção de 100 ms.
- Use os seguintes parâmetros para HCD-MS3 SPS 10: Intervalo de varredura 100-400 m/z, número de varreduras dependentes 10, AGC 5.0e4, tempo máximo de injeção 118 ms, energia de colisão HCD 55%, janela de isolamento MS2.
9. Análise de dados
- Pesquisa gerou arquivos RAW para a lista de proteínas e peptídeos usando um software de análise de proteínas contra um banco de dados apropriado.
NOTA: Os arquivos RAW gerados neste estudo foram pesquisados contra um banco de dados do Mouse Uniprot. - Uma vez que há rotulagem leve e pesada em um arquivo RAW, pesquise cada arquivo com dois fluxos de trabalho para peptídeos dimetilados leves e pesados.
- Pesquise os arquivos RAW contra o banco de dados do Mouse Uniprot (13/07/2019) com sequência 53035 com os seguintes parâmetros: decote trypsin com máximo dois decotes perdidos, peptídeo com comprimento mínimo de 6 aminoácidos, tolerância à massa dos pais de 15 ppm, 1 Tolerância à fragmentação da Modificações estáticas: dimetilação leve (+28.031, peptídeo N-terminus) ou dimetilação pesada (+36.076 Da, peptídeo N-terminus), carbamidomtila (+57.021 Da, C), Modificações dinâmicas: oxidação (+15.995 Da, M), tag isobárica 11-plex (229.163 Da, K), 1% FDR, quantificação de íons repórteres com tolerância de integração de pico de 30 ppm, centroid mais confiante para método de integração.
NOTA: O pico de dimetilado pesado também inclui outra modificação que é ~7 Da (+35.069 Da, peptídeo N-terminus) do pico dimetilado leve e, portanto, outro nó de pesquisa deve ser incorporado para incluir esta modificação também. - Realizar quantificação de íon repórter com base na intensidade, 65% de correspondência de massa SPS, proporção média S/N 10, correção isotópica, normalização e dimensionamento não foram realizadas.
NOTA: A normalização e a escala podem ser realizadas com base na quantidade total de peptídeo ou uma proteína específica adicionada à amostra. As amostras de QC também podem ser incluídas nos canais para normalização entre lotes ou intra-lotes com base em um dimensionamento de referência interna de duas caudas18. As contaminações isotópicas de diferentes canais TMT não foram fornecidas à pesquisa, os usuários são aconselhados a adicionar a contaminação de isótopos de diferentes íons repórteres.
Resultados
A Figura 2 mostra dados representativos de MS de um peptídeo identificado em todos os 22 canais de íons de repórter de um experimento de 22-plex cPILOT, incluindo réplicas de fluxo de trabalho. A Figura 2 (topo) retrata um par de pico duplamente carregado separado por 4 espaçamentos de m/z indicando um único grupo de dimetila incorporado ao peptídeo. Os pares de pico dimetilado leves e pesados foram isolados e fragmentados independentemente para produzir a sequência do peptídeo. A sequência do peptídeo é G(dimethyl)AAELMQQK(TMT-11plex) e corresponde à proteína Betaine-homocysteine S-metil transferase. Os íons fragmentos mais intensos para os picos leves e pesados dimetilados(não mostrados) foram ainda mais isolados para a fragmentação de MS3 e os íons repórteres(m/z 126-131) são mostrados na Figura 2 (inferior). As intensidades de íons repórteres são diretamente proporcionais à abundância de peptídeos na amostra. A abundância de peptídeos das amostras implica que a capacidade de pipetação da plataforma robótica é bastante uniforme nas 22 amostras. No geral, este experimento de 22 plex cPILOT resultou em 1326 (1209-light/1181-heavy) identificações de proteínas resultantes de 3098 (6137-light/5872-heavy) peptídeos(Tabela 4). A Figura 3 mostra o enredo da caixa de abundância log10 versus intensidades totais de íons de repórter em todos os 22 canais mostrando menor variabilidade entre bem/inter-amostra. A avaliação da automação total foi feita examinando o erro na abundância de íons repórteres em cada proteína nas 22 amostras. A Figura 4 mostra que o processamento de amostras com a plataforma robótica resultou em valores cv muito baixos. Especificamente, nos 3098 peptídeos identificados, o CV médio em abundância de íons repórteres foi de 12,36 % e 15,03 % para peptídeos leves e pesados dimetilados, respectivamente. Entre esses peptídeos 2032 desses peptídeos tinham sinal de íon repórter acima do limiar mínimo e foram considerados quantificáveis.
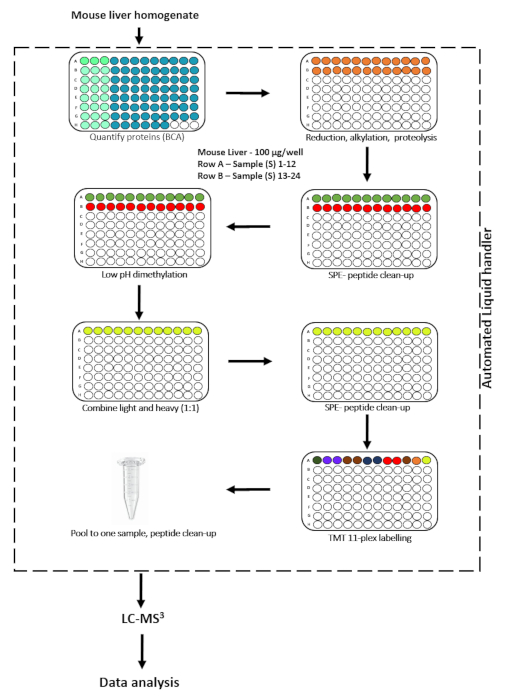
Figura 1. Fluxo de trabalho experimental para processar 22 amostras em paralelo com um protocolo cPILOT automatizado. Clique aqui para ver uma versão maior desta figura.
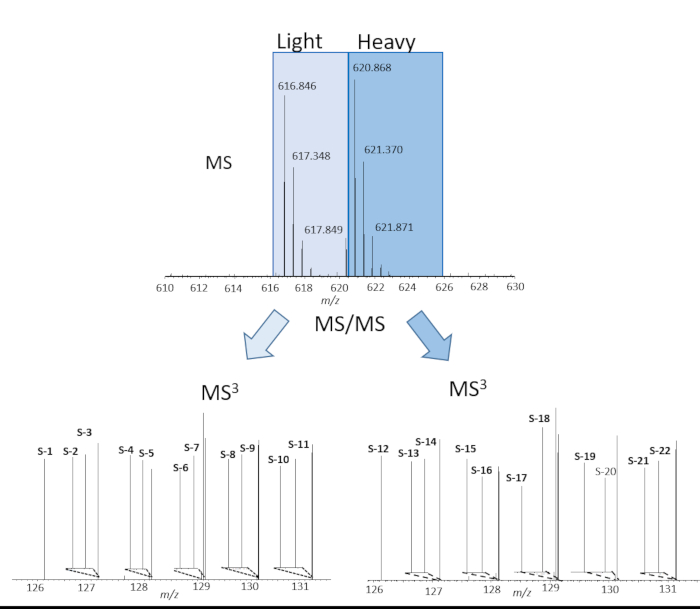
Figura 2. Quantificação de peptídeos em 22 amostras. Exemplo MS (superior) e MS3 (inferior) espectros do peptídeo G (dimethyl)AAELMQQK(TMT-11plex) quantificados em 22-plex experimento cPILOT automatizado para picos dimetilados leves (inferior esquerdo) e dimetilados pesados (inferior direito). Clique aqui para ver uma versão maior desta figura.
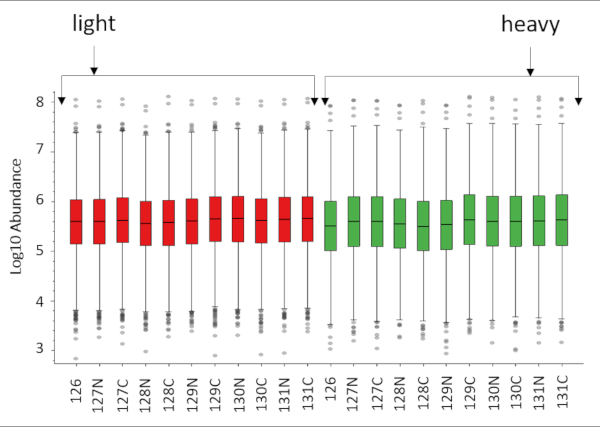
Figura 3. Gráfico de caixa de intensidades totais de íons de repórter versus log10 abundância de 22 amostras usando o descobridor proteome 2.3. O arquivo RAW foi pesquisado duas vezes por peptídeos leves e pesados, IDs de proteínas separadamente com TMT como modificação dinâmica, leve (+28.031 Da) e pesada (+36.076 & +35.069 Da) dimetilação no peptídeo N-termini como modificação estática. Uma pesquisa combinada com todas as modificações acima foi executada usando Proteome Discover 2.3 para obter o Log 10 Abundância de intensidades de peptídeos em todos os canais. Clique aqui para ver uma versão maior desta figura.
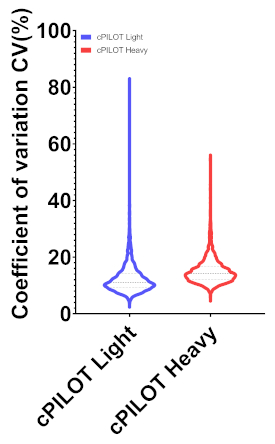
Figura 4. Gráficos de violino de co-eficiente de variação da abundância de peptídeos a partir de intensidades de íons repórteres somadas através dos canais 126-131 m/z. O peptídeo foi quantificado com um valor médio cv de 12,36 e 15,03 para peptídeos leves (2373) e pesados (2533). Clique aqui para ver uma versão maior desta figura.
Figura Suplementar 1. Ilustração do cPILOT com um único peptídeo. Mostrando a rotulagem isotópica de duas amostras diferentes e a marcação isobárica com TMT126,a mistura resultante foi injetada em MS para LC-MS3. Clique aqui para baixar este arquivo.
| Nome variável | Valor | Descrição |
| DesaltSamp1 | 1065 | Volume a ser usado para desalto passo 1 |
| DesaltSamp2 | 392 | Volume a ser usado para desalto passo 2 |
| DesaltSamp3 | 100 | Volume a ser usado para desalto passo 3 |
| Devmode | False | Falso reduzirá os tempos de incubação para 30seg- True seguirá o tempo de incubação no protocolo. |
| DTTVol | 3 | Volume de DTT |
| Placa de filtro | Targa | Placa usada para desalvar |
| FilterPlateVol | 600 | Volume para desalting |
| HAWaterWashes | False | Número de lavagens de água na placa SPE |
| IAMVol | 2 | Volume de iodoacetamida |
| PeptideTMTVol | 12.5 | Volume de peptídeo para rotulagem TMT |
| Pressão | 100 | pressão mbar no PPA |
| TempOffSet | 1 | Mudança de temperatura |
| TMTVol | 10 | Volume de tags isobárica a ser adicionado |
| TrisVol | 800 | Volume para diluir amostra antes da digestão |
| TrypsinVol | 2 | Volume de trippsina |
| UsePopTimer | Verdade | True exibe as opções para aplicar pressão na placa, se necessário |
Mesa 1. Lista de variáveis utilizadas no protocolo cPILOT automatizado.
| Fonte dil | DilWell | Dest | DestWell | DilVolume | StockSource | Stockwell | SampleVol | Amostraid |
| 8M_Urea | 1 | Amostras | A1 | 90 | Stock_Samples | A1 | 10 | 1 |
| 8M_Urea | 1 | Amostras | A2 | 90 | Stock_Samples | A1 | 10 | 2 |
| 8M_Urea | 1 | Amostras | A3 | 90 | Stock_Samples | A1 | 10 | 3 |
| 8M_Urea | 1 | Amostras | A4 | 90 | Stock_Samples | A1 | 10 | 4 |
| 8M_Urea | 1 | Amostras | A5 | 90 | Stock_Samples | A1 | 10 | 5 |
| 8M_Urea | 1 | Amostras | A6 | 90 | Stock_Samples | A1 | 10 | 6 |
| 8M_Urea | 1 | Amostras | A7 | 90 | Stock_Samples | A1 | 10 | 7 |
| 8M_Urea | 1 | Amostras | A8 | 90 | Stock_Samples | A1 | 10 | 8 |
| 8M_Urea | 1 | Amostras | A9 | 90 | Stock_Samples | A1 | 10 | 9 |
| 8M_Urea | 1 | Amostras | A10 | 90 | Stock_Samples | A1 | 10 | 10 |
| 8M_Urea | 1 | Amostras | A11 | 90 | Stock_Samples | A1 | 10 | 11 |
| 8M_Urea | 1 | Amostras | A12 | 90 | Stock_Samples | A1 | 10 | 12 |
| 8M_Urea | 1 | Amostras | B1 | 90 | Stock_Samples | A1 | 10 | 13 |
| 8M_Urea | 1 | Amostras | B2 | 90 | Stock_Samples | A1 | 10 | 14 |
| 8M_Urea | 1 | Amostras | B3 | 90 | Stock_Samples | A1 | 10 | 15 |
| 8M_Urea | 1 | Amostras | B4 | 90 | Stock_Samples | A1 | 10 | 16 |
| 8M_Urea | 1 | Amostras | B5 | 90 | Stock_Samples | A1 | 10 | 17 |
| 8M_Urea | 1 | Amostras | B6 | 90 | Stock_Samples | A1 | 10 | 18 |
| 8M_Urea | 1 | Amostras | B7 | 90 | Stock_Samples | A1 | 10 | 19 |
| 8M_Urea | 1 | Amostras | B8 | 90 | Stock_Samples | A1 | 10 | 20 |
| 8M_Urea | 1 | Amostras | B9 | 90 | Stock_Samples | A1 | 10 | 21 |
| 8M_Urea | 1 | Amostras | B10 | 90 | Stock_Samples | A1 | 10 | 22 |
| 8M_Urea | 1 | Amostras | B11 | 90 | Stock_Samples | A1 | 10 | 23 |
| 8M_Urea | 1 | Amostras | B12 | 90 | Stock_Samples | A1 | 10 | 24 |
Mesa 2. Volume de fígado de rato homogeneado e 8 M de ureia.
| FonteWell | FonteWell2 | Repórter Ion | DestWell1 | DestWell2 | Volume | Amostraid |
| A1 | C1 | 126 | A1 | E1 | 10 | 1 |
| A3 | C3 | 127N | A2 | E2 | 10 | 2 |
| A5 | C5 | 127C | A3 | E3 | 10 | 3 |
| A7 | C7 | 128N | A4 | E4 | 10 | 4 |
| A9 | C9 | 128C | A5 | E5 | 10 | 5 |
| A11 | C11 | 129N | A6 | E6 | 10 | 6 |
| B2 | D2 | 129C | A7 | E7 | 10 | 7 |
| B4 | D4 | 130N | A8 | E8 | 10 | 8 |
| B6 | D6 | 130C | A9 | E9 | 10 | 9 |
| B8 | D8 | 131N | A10 | E10 | 10 | 10 |
| B10 | D10 | 131C | A11 | E11 | 10 | 11 |
Mesa 3. Número total de peptídeos, proteínas e fósforos espectrais de peptídeos (PSMs).
| CPILOT automatizado | ||
| Luz | Pesado | |
| Proteínas | 1209 | 1181 |
| Peptídeos | 6137 | 5872 |
| PSMs | 14948 | 16762 |
Mesa 4. Barcoding as etiquetas isobáricas com as amostras leves e pesadas rotuladas.
Discussão
cPILOT é uma estratégia de multiplexação aprimorada que pode analisar até 24 amostras em um único experimento. A capacidade de multiplexing depende do número de combinações isotópicas e isobáricas disponíveis. A introdução do TMTpro7, que é capaz de marcar 16 amostras em um único experimento, pode empurrar os limites do cPILOT para 32-plex. cPILOT consiste em múltiplas etapas de pipetação e requer cuidados extensivos e habilidades do usuário para realizar a preparação da amostra. Mesmo com um usuário especialista, erros manuais são inevitáveis, o que convida o uso de plataformas robóticas para processar amostras na estratégia cPILOT. Uma vez que o cPILOT utiliza a marcação dependente de pH dos peptídeos, o pH precisa ser mantido para a luz e o conjunto de amostras estilizadas pesadas. PH levemente ácido-básico pode resultar em dimetilação tanto de resíduos de N-termini quanto de lisina. Uma vantagem do cPILOT é que ele requer apenas metade das tags isobáricas, uma vez que peptídeo N-termini são ocupados com os grupos de dimetila. Isso permite que um número maior de amostras seja rotulada pela metade do custo. O manuseio de números amostrais maiores requer que os tempos de exposição ao reagente sejam semelhantes para a primeira e a última amostra em um lote. Um distribuidor de pipetas que pode acomodar até 32 amostras em paralelo pode ser melhor alcançado com o uso de dispositivos robóticos de manuseio líquido.
Para processar várias amostras por cPILOT, o fluxo de trabalho manual foi alterado para incorporar a automação. O manipulador líquido robótico usado neste estudo possui dois pods com habilidades de pipetação de 96 canais e 8 canais, com uma pinça para colocar as placas nos 28 locais disponíveis. O manipulador líquido é integrado com um aparelho de pressão positivo, shaker orbital e um dispositivo para aquecer/resfriar amostras na placa de 96 poços. O aparelho de pressão positiva auxilia na realização de trocas tampão nas placas de SPE durante a limpeza, enquanto o agitador orbital ajuda a vórtice/misturar as amostras. A plataforma robótica foi programada para aspirar e distribuir tampões e amostras para placas de 96 poços, incubar, amostras de vórtice e placas de transferência. Líquidos com diferentes viscosidades, como acetonitrilo e água, requerem considerações específicas de pipetização que também podem ser programadas no método.
O fluxo de trabalho cPILOT, desde a quantificação proteica pelo BCA até a rotulagem dos peptídeos com tags isobáricas (ou seja, TMT), foi realizado no sistema de manipuladores líquidos. O protocolo completo foi dimensionado para usar 96 placas de poços profundos que podem conter 2 mL por poço. Os buffers foram preparados antes do início do experimento e adicionados à placa do poço 96, de modo a permitir o processamento paralelo da amostra. No presente estudo, 22 réplicas do fluxo de trabalho do homogeneato do fígado do camundongo foram adicionadas às placas profundas do poço e levadas através do protocolo cPILOT. Finalmente, uma única amostra constituída pelo fígado de rato equimolar de 22 plex foi injetada no espectrômetro de massa. Intensidades de íons repórteres correspondentes à abundância de peptídeos nas amostras demonstraram que as amostras processadas com o manipulador líquido têm CVs menores do que o protocolo manual(dados não mostrados). A plataforma robótica também melhorou muito a reprodutibilidade do processamento de amostras. A reprodutibilidade e robustez são fatores muito importantes ao processar um grande número de amostras. Erros de pipetação podem levar à interpretação errada completa dos dados e aqui a plataforma robótica forneceu baixa variação entre amostras. Também o uso da plataforma robótica para cPILOT reduziu o tempo necessário para preparar amostras. Por exemplo, depois de desenvolver o método automatizado, foi necessário 2,5 h para processar 22 amostras em comparação com 7,5 h para cPILOT manual. Experimentos estão em andamento em nosso laboratório para avaliar ainda mais as comparações dos fluxos de trabalho cPILOT manuais e automatizados. Com base em relatórios anteriores do nosso laboratório, os CV%'s de intensidades de íons de repórter de proteína no cPILOT manual foram, em média, 20% com alguns outliers excedendo esse valor12.
cPILOT é uma estratégia de derivação química no nível do peptídeo, que pode ser usada para qualquer tipo de amostra, como células, tecidos e fluidos corporais. o cPILOT oferece multiplexing de amostra aprimorado e com a incorporação da automação pode facilitar o multiplexing de amostra de alto rendimento em proteômica. Esse rendimento é necessário para avançar ainda mais a compreensão biológica e a descoberta de biomarcadores.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores reconhecem os Fundos de Start-up da Universidade Vanderbilt e o prêmio NIH (R01GM117191) ao RASR.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.6 mL eppendorf tubes, 500 pk | Fisher Scientific | 05-408-120 | Any brand of 0.6 mL eppendorf tubes are sufficient |
| 0.65 µm Ultrafree MC DV centrifugal filter units | EMD Millipore | UFC30DV00 | |
| 1.5 mL eppendorf tubes, 500 pk | Fisher Scientific | 05-408-129 | Any brand of 1.5 mL eppendorf tubes are sufficient |
| 2 ml black deep well plate | Analytical Sales and Services, Inc. | 59623-23BKGC | Any brand of black 96-well plate is sufficient |
| 2 ml clear deep well plate | VWR | 75870-796 | |
| Acetic Acid | J.T. Baker | 9508-01 | |
| Acetonitrile - MS Grade | Fisher Scientific | A955-4 | 4 L quantity is not necessary |
| Agilent 500µL plate | Agilent | 203942-100 | Reagent plate for adding buffers |
| Ammonium formate | Acros Organics | 208-753-9 | |
| Ammonium hydroxide solution (28 - 30%) | Sigma Aldrich | 320145-500ML | |
| Analytical balance | Mettler Toledo | AL54 | |
| BCA protein assay kit | Pierce Thermo Fisher Scientific | 23227 | |
| Biomek i7 hybrid | Beckmann | Any liquid handling device with ability to use positive pressure, heating/cooling and Vortex the samples. | |
| C18 packing material (2.5 µm, 100 Å) | Bruker | This item is no longer available from Bruker. Alternative packing material with listed specifications will be sufficient | |
| Centrifuge with plate rotor | Thermo Scientific | 69720 | |
| Micro 21R Centrifuge | Sorval | 5437 | |
| Dionex 3000 UHPLC | Thermo Scientific | This model is no longer available. Any nano LC with an autosampler is sufficient. | |
| Dithiothreiotol (DTT) | Fisher Scientific | BP172-5 | |
| Formaldehyde (13CD2O) solution; 20 wt % in D2O, 98 atom % D, 99 atom % 13C | Sigma Aldrich, Chemistry | 596388-1G | |
| Formaldehyde (CH2O) solution; 36.5 - 38% in H2O | Sigma Aldrich, Life Science | F8775-25ML | |
| Formic Acid | Fluka Analytical | 94318-250ML-F | |
| Fusion Lumos Mass Spectrometer | Thermo Scientific | This model is no longer available. Other high resolution instruments (e.g. Orbitrap Elite, Orbitrap Fusion, or Orbitrap Fusion Lumos) can be used. | |
| Hydroxylamine hydrochloride | Sigma Aldrich, Chemistry | 255580-100G | |
| Iodoacetamide (IAM) | Acros Organics | 144-48-9 | |
| Isobaric Tagging Kit (TMT 11-plex) | Thermo Fisher Scientific | 90061 | |
| L-1-tosylamido-2 phenylethyl cholormethyl ketone (TPCK)-treated Trypsin from bovine pancreas | Sigma Aldrich, Life Science | T1426-100MG | |
| L-Cysteine | Sigma Aldrich, Chemistry | 168149-25G | |
| Mechanical Homogenizer (i.e. FastPrep-24 5G) | MP Biomedicals | 116005500 | |
| pH 10 buffer | Fisher Scientific | 06-664-261 | Any brand of pH buffer 10 is sufficient |
| pH 7 buffer | Fisher Scientific | 06-664-260 | Any brand pH buffer 7 is sufficient |
| pH meter (Tris compatiable) | Fisher Scientific (Accumet) | 13-620-183 | Any brand of a pH meter is sufficient |
| Protein software (e.g. Proteome Discoverer) | Thermo Scientific | ||
| Reservior plate 200ml | Agilent | 204017-100 | |
| Sodium Cyanoborodeuteride; 96 atom % D, 98% CP | Sigma Aldrich, Chemistry | 190020-1G | |
| Sodium Cyanoborohydride; reagent grade, 95% | Sigma Aldrich | 156159-10G | |
| Speed-vac | Thermo Scientific | SPD1010 | any brand of speed vac that can accommodate a deep well plate is sufficient |
| Stir plate | VWR | 12365-382 | Any brand of stir plates are sufficient |
| Targa 20 mg SPE plates | Nest Group, Inc. | HNS S18V | These are C18 cartridges |
| Triethyl ammonium bicarbonate (TEAB) buffer | Sigma Aldrich, Life Science | T7408-100ML | |
| Tris | Biorad | 161-0716 | |
| Biomek 24-Place Tube Rack Holder | Beckmann | 373661 | |
| Urea | Biorad | 161-0731 | |
| Water - MS Grade | Fisher Scientific | W6-4 | 4 L quantity is not necessary |
Referências
- Diamandis, E. P. Mass spectrometry as a diagnostic and a cancer biomarker discovery tool: opportunities and potential limitations. Molecular and Cell Proteomics. 3 (4), 367-378 (2004).
- Qian, W. -J., Jacobs, J. M., Liu, T., Camp, D. G., Smith, R. D. Advances and challenges in liquid chromatography-mass spectrometry-based proteomics profiling for clinical applications. Molecular and Cellular Proteomics. 5 (10), 1727-1744 (2006).
- Arul, A. B., Robinson, R. A. S. Sample Multiplexing Strategies in Quantitative Proteomics. Analytical Chemistry. 91 (1), 178-189 (2019).
- Everley, R. A., Kunz, R. C., McAllister, F. E., Gygi, S. P. Increasing Throughput in Targeted Proteomics Assays: 54-Plex Quantitation in a Single Mass Spectrometry Run. Analytical Chemistry. 85 (11), 5340-5346 (2013).
- Shiio, Y., Aebersold, R. Quantitative proteome analysis using isotope-coded affinity tags and mass spectrometry. Nature Protocol. 1 (1), 139-145 (2006).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. TMTpro: Design, Synthesis, and Initial Evaluation of a Proline-Based Isobaric 16-Plex Tandem Mass Tag Reagent Set. Analytical Chemistry. 91 (24), 15941-15950 (2019).
- Frost, D. C., Feng, Y., Li, L. 21-plex DiLue Isobaric Tags for High-Throughput Quantitative Proteomics. Anal. Chem. 92 (12), 8228-8234 (2020).
- Evans, A. R., Robinson, R. A. Global combined precursor isotopic labeling and isobaric tagging (cPILOT) approach with selective MS(3) acquisition. Proteomics. 13 (22), 3267-3272 (2013).
- Robinson, R. A., Evans, A. R. Enhanced sample multiplexing for nitrotyrosine-modified proteins using combined precursor isotopic labeling and isobaric tagging. Analytical Chemistry. 84 (11), 4677-4686 (2012).
- King, C. D., Dudenhoeffer, J. D., Gu, L., Evans, A. R., Robinson, R. A. S. Enhanced Sample Multiplexing of Tissues Using Combined Precursor Isotopic Labeling and Isobaric Tagging (cPILOT). Journal of Visual Experiments. (123), e55406(2017).
- King, C. D., Robinson, R. A. S. Evaluating Combined Precursor Isotopic Labeling and Isobaric Tagging Performance on Orbitraps To Study the Peripheral Proteome of Alzheimer's Disease. Analytical Chemistry. , (2020).
- Evans, A. R., Gu, L., Guerrero, R., Robinson, R. A. Global cPILOT analysis of the APP/PS-1 mouse liver proteome. Proteomics & Clinical Applications. 9 (9-10), 872-884 (2015).
- Gu, L., Evans, A. R., Robinson, R. A. Sample multiplexing with cysteine-selective approaches: cysDML and cPILOT. Journal of American Society of Mass Spectrometer. 26 (4), 615-630 (2015).
- Ting, L., Rad, R., Gygi, S. P., Haas, W. MS3 eliminates ratio distortion in isobaric multiplexed quantitative proteomics. Nature Methods. 8 (11), 937-940 (2011).
- Frost, D. C., Rust, C. J., Robinson, R. A. S., Li, L. Increased N,N-Dimethyl Leucine Isobaric Tag Multiplexing by a Combined Precursor Isotopic Labeling and Isobaric Tagging Approach. Analytical Chemistry. 90 (18), 10664-10669 (2018).
- Gu, L., Robinson, R. A. High-throughput endogenous measurement of S-nitrosylation in Alzheimer's disease using oxidized cysteine-selective cPILOT. Analyst. 141 (12), 3904-3915 (2016).
- Amin, B., Ford, K. I., Robinson, R. A. S. Quantitative proteomics to study aging in rabbit liver. Mechanisms of Ageing and Development. 187, 111227(2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados