É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Permitindo compensação em tempo real em oxidações fotoquímicas rápidas de proteínas para a determinação de mudanças de topografia de proteínas
Neste Artigo
Resumo
A oxidação fotoquímica rápida das proteínas é uma técnica emergente para a caracterização estrutural das proteínas. Diferentes aditivos e ligantes de solventes têm variadas propriedades de limpeza radical hidroxyl. Para comparar a estrutura proteica em diferentes condições, a compensação em tempo real dos radicais hidroxis gerados na reação é necessária para normalizar as condições de reação.
Resumo
A oxidação fotoquímica rápida de proteínas (FPOP) é uma técnica de biologia estrutural baseada em espectrometria de massa que sonda a área de superfície acessível de solventes das proteínas. Esta técnica se baseia na reação de cadeias laterais de aminoácidos com radicais hidroxil livremente difundindo em solução. FPOP gera esses radicais in situ por fotolise laser de peróxido de hidrogênio, criando uma explosão de radicais hidroxil que está esgotado na ordem de um microssegundo. Quando esses radicais hidroxis reagem com uma cadeia lateral de aminoácidos acessível a solventes, os produtos de reação exibem uma mudança de massa que pode ser medida e quantificada pela espectrometria de massa. Uma vez que a taxa de reação de um aminoácido depende, em parte, da superfície acessível de solvente médio desse aminoácido, mudanças medidas na quantidade de oxidação de uma determinada região de uma proteína podem estar diretamente correlacionadas com mudanças na acessibilidade solvente daquela região entre diferentes conformações (por exemplo, ligadura versus sem ligante, monômero vs. agregado, etc.) O FPOP tem sido aplicado em uma série de problemas na biologia, incluindo interações proteína-proteína, alterações conformacionais proteicas e ligação proteína-ligante. Como a concentração disponível de radicais hidroxilas varia de acordo com muitas condições experimentais no experimento FPOP, é importante monitorar a dose radical eficaz à qual o analito proteico é exposto. Este monitoramento é eficientemente alcançado incorporando um dosímetro inline para medir o sinal da reação FPOP, com fluência laser ajustada em tempo real para alcançar a quantidade desejada de oxidação. Com essa compensação, mudanças na topografia proteica refletindo alterações conformais, superfícies de ligação de ligantes e interfaces de interação proteína-proteína podem ser determinadas em amostras heterogêneas usando quantidades amostrais relativamente baixas.
Introdução
A oxidação fotoquímica rápida de proteínas (FPOP) é uma técnica emergente para a determinação de alterações topográficas proteicas por modificação covalente ultrarrápida da área de superfície exposta ao solvente de proteínas seguida pela detecção por LC-MS1. FPOP gera uma alta concentração de radicais hidroxílicos in situ por fotolise flash laser UV de peróxido de hidrogênio. Esses radicais hidroxis são muito reativos e de curta duração, consumidos em aproximadamente uma escala de tempo microssegundos sob as condições FPOP2. Esses radicais hidroxis se difundem através da água e oxidam vários componentes orgânicos em solução a taxas cinéticas geralmente variando de rápido (~106 M-1 s-1) a3controlados por difusão . Quando o radical hidroxyl encontra uma superfície proteica, o radical oxidará as cadeias laterais de aminoácidos na superfície da proteína, resultando em uma mudança em massa desse aminoácido (mais comumente a adição líquida de um átomo de oxigênio)4. A taxa de reação de oxidação em qualquer aminoácido depende de dois fatores: a reatividade inerente desse aminoácido (que depende da cadeia lateral e do contexto sequencial)4,5 e a acessibilidade dessa cadeia lateral ao radical hidroxílico difusor, que se correlaciona intimamente com a área média de superfície acessívelsolvente 6,,7. Todos os aminoácidos padrão, exceto a glicina, foram observados como rotulados por esses radicais hidroxil altamente reativos em experimentos FPOP, embora em rendimentos amplamente diferentes; na prática, Ser, Thr, Asn e Ala raramente são vistos como oxidados na maioria das amostras, exceto sob altas doses radicais e identificados pela cuidadosa e sensível fragmentação de ETD8,,9. Após a oxidação, as amostras são saciadas para remover peróxido de hidrogênio e oxidantes secundários (superóxido, oxigênio singlet, hidroperóxidos de peptidyl, etc.) As amostras saciadas são então digeridas proteolíticos para gerar misturas de peptídeos oxidados, onde as informações estruturais são congeladas como um "instantâneo" químico nos padrões dos produtos de oxidação dos vários peptídeos(Figura 1). A cromatografia líquida acoplada à espectrometria de massa (LC-MS) é usada para medir a quantidade de oxidação de aminoácidos em um determinado peptídeo proteolítico baseado nas intensidades relativas das versões oxidadas e nãoxidizadas desse peptídeo. Comparando esta pegada oxidativa da mesma proteína obtida em diferentes condições conformais (por exemplo, ligadura versus sem ligante), as diferenças na quantidade de oxidação de uma determinada região da proteína podem estar diretamente correlacionadas com diferenças na superfície acessível ao solvente daquela região6,,7. A capacidade de fornecer informações topográficas proteicas torna o FPOP uma tecnologia atraente para a determinação da estrutura de alta ordem de proteínas, inclusive na descoberta e desenvolvimento terapêutico de proteínas10,11.

Figura 1: Visão geral do FPOP. A superfície da proteína é covalentemente modificada por radicais hidroxil altamente reativos. Os radicais hidroxis reagirão com cadeias laterais de aminoácidos da proteína a uma taxa fortemente influenciada pela acessibilidade solvente da cadeia lateral. As alterações topográficas (por exemplo, devido à ligação de um ligante como mostrado acima) protegerão os aminoácidos na região de interação de reagir com radicais hidroxílicos, resultando em uma diminuição na intensidade do peptídeo modificado no sinal LC-MS. Clique aqui para ver uma versão maior desta figura.
Diferentes constituintes presentes na solução FPOP (por exemplo, ligantes, excipientes, tampões) têm diferentes atividades de limpeza em direção aos radicais hidroxílicos gerados sobre a fotolise laser do peróxido de hidrogênio3. Da mesma forma, uma pequena mudança na concentração de peróxido, fluência laser e composição de buffer pode alterar a dose radical eficaz, tornando a reprodução de dados FPOP desafiadora entre as amostras e entre diferentes laboratórios. Portanto, é importante poder comparar a dose radical hidroxyl disponível para reagir com proteína em cada amostra usando um dosímetros radicais hidroxílicos disponíveis12,,13,,14,,15,,16. Os dosímetros radicais hidroxilas atuam competindo com o analito (e com todos os catadores em solução) para a piscina de radicais hidroxila; a dose efetiva de radicais hidroxis é medida medindo a quantidade de oxidação do dosímetro. Note que a "dose radical hidroxíl" eficaz é uma função tanto da concentração inicial de radical hidroxílico gerada quanto da meia-vida do radical. Esses dois parâmetros são parcialmente dependentes um do outro, tornando a modelagem cinética teórica um pouco complexa(Figura 2). Duas amostras poderiam ter meias-vidas radicais iniciais extremamente diferentes enquanto ainda mantinham a mesma dose radical eficaz alterando a concentração inicial de radicais hidroxis formados; eles ainda vão gerar pegadas idênticas17. Adenina13 e Tris12 são dosímetros radicais hidroxílicos convenientes porque seu nível de oxidação pode ser medido pela espectroscopia UV em tempo real, permitindo que os pesquisadores identifiquem rapidamente quando há um problema com a dose radical hidroxílida eficaz e para solucionar seus problemas. Para resolver esse problema, um dosímetro inline localizado no sistema de fluxo logo após o local da irradiação que pode monitorar o sinal a partir de mudanças de absorção de adenina em tempo real é importante. Isso ajuda na realização de experimentos FPOP em buffers ou qualquer outro excipiente com níveis amplamente diferentes de capacidade de limpeza radical hidroxis17. Essa compensação de dosagem radical pode ser realizada em tempo real, produzindo resultados estatisticamente indistinguíveis para o mesmo conformador, ajustando a dose radical eficaz.
Neste protocolo, temos procedimentos detalhados para a realização de um experimento típico de FPOP com compensação de dosagem radical usando adenina como um dosímetro radical óptico interno. Este método permite que os pesquisadores comparem pegadas em condições FPOP que têm capacidade de limpeza diferente, realizando compensação em tempo real.
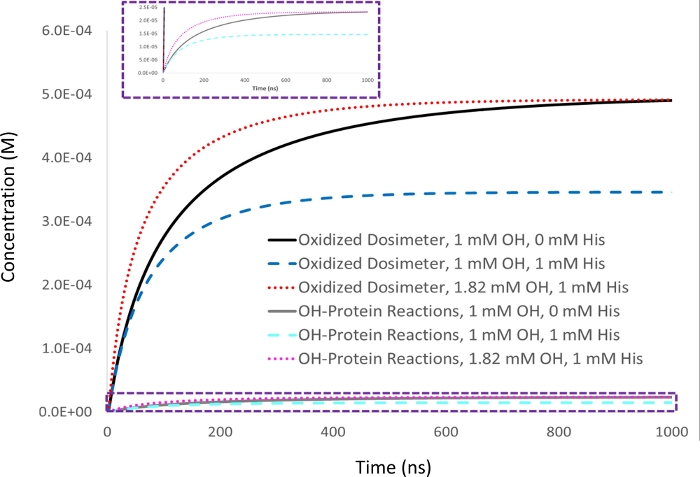
Figura 2: Simulação cinética de compensação baseada em dosimetria. A resposta dosímetro de adenina de 1 mM é medida em 5 μM de analito de lymo com uma concentração radical hidráxil inicial de 1 mM (▪OH t1/2=53 ns), e definida como resposta dosímetro alvo (preto). Após a adição de 1 mM da histidina excipiente catraca, a resposta dosímetro (azul) diminui junto com a quantidade de oxidação proteica de forma proporcional (ciano). A meia-vida do radical hidroxíll também diminui (▪OH t1/2=39 ns). Quando a quantidade de radical hidroxílico gerada é aumentada para dar um rendimento equivalente de dosímetro oxidado na amostra com 1 mM de histidina como alcançado com 1 mM radical hidroxyl na ausência de sca verador (vermelho), a quantidade de oxidação proteica que ocorre de forma semelhante torna-se idêntica (magenta), enquanto a meia-vida radical hidroxis diminui ainda mais (▪OH t1/2=29 ns). Adaptado com permissão da Sharp J.S., Am Pharmaceut Rev 22, 50-55, 2019. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Prepare o Banco Óptico e o Capilar para FPOP
ATENÇÃO: Os lasers excimer krf são perigos extremos dos olhos, e a luz direta ou refletida pode causar danos permanentes nos olhos. Use sempre proteção ocular adequada, evite a presença de quaisquer objetos reflexivos perto do caminho do feixe quando possível, e use controles de engenharia para impedir o acesso não autorizado a um laser ativo e conter quaisquer reflexos perdidos.
- Prepare o banco óptico FPOP.
- Ligue o laser para aquecer. Coloque o laser no gatilho externo, energia constante, sem substituição de gás. Defina a energia do laser por pulso (tipicamente entre 80-120 mJ/pulso).
- Configure o banco óptico com a lente plano-convexa (30 mm Dia. x 120 mm FL sem revestimento) diretamente no caminho do raio laser e um backstop não reflexivo para absorver a luz como mostrado na Figura 3A.

Figura 3: Banco óptico para o experimento FPOP. (A) A amostra é misturada com H2O2, dosímetro radical de adenina e catador de glutamina e carregada na seringa. A amostra é empurrada através da capilar de sílica fundida através do caminho de feixe focalado de um laser UV excimer KrF. A luz UV folliposa H2O2 em radicais hidroxil, que oxida a proteína e os dosímetros de adenina. O fluxo de seringa empurra a amostra iluminada para fora do caminho do laser antes do próximo pulso de laser, com um volume de exclusão nãoilluinado entre regiões iluminadas. Imediatamente após a oxidação, a amostra é passada através de um espectrofotômetro UV inline, que mede a absorvância UV da adenina a 265 nm. A amostra é então depositada em um tampão de saciar para eliminar os oxidantes restantes H2O2 e secundários. (B) O tamanho da mancha é medido após irradiar uma nota adesiva colorida afixada atrás do capilar com o laser a 248 nm. A largura do local é usada para calcular a taxa de fluxo amostral, e a silhueta do capilar no centro da mancha é usada para alinhar o banco óptico. Clique aqui para ver uma versão maior desta figura.
- Corte um comprimento apropriado da capilaridade de sílica fundida (360 μm de diâmetro externo e 100 μm de diâmetro interno) e usando uma manga, conecte-se à seringa apertada a gás usando um conector de baixo volume morto.
- Queime suavemente o revestimento de poliimida do capilar com uma tocha de butano no local onde o dosímetro inline lê o sinal de absorvência a 265 nm após a exposição a laser das amostras. Limpe os detritos do capilar gentilmente usando metanol em um lenço sem fiapos. O revestimento de poliimida no local da incidência de laser pode ser igualmente queimado com a tocha de butano ou queimado com o laser excimer disparando em baixa potência.
NOTA: Aguarde que o capilar esfrie, pois é um risco de incêndio usar o metanol no capilar quente. - Coloque este capilar através do caminho do feixe do laser e no dosímetro inline.
- Pressione a alavanca na parte superior do dosímetro da linha para abrir a dobradiça. Remova os suportes magnéticos. Coloque o capilar no sulco usinado do dosímetro inline, utilizando os suportes magnéticos para manter o capilar no lugar. Feche a dobradiça dosímetro sobre o capilar, pressionando-a até que a alavanca se eixe no lugar.
- Usando o software de dosimetria, clique no botão Iniciar Flash para começar a disparar o laser excimer. Defina a potência laser predefinida entre 50-100 mJ/pulse no próprio software de controle a laser e defina a taxa de repetição predefinida entre 10-20 Hz na guia Configurações do software de dosimetria.
- Concentre o raio laser usando uma lente convexa plano montada em um estágio motorizado linear. Meça a largura e a altura do ponto laser na posição do capilar em uma nota pegajosa precisamente usando uma pinça para calcular fluência incidente (mJ/mm2) como mostrado na Figura 3B.
- Coloque uma abertura opaca perto do capilar para garantir uma largura iluminada consistente do capilar, independentemente de alterações no tamanho do feixe devido ao movimento da lente ou alteração da energia por pulso do laser18.
- Com o disparo a laser, mova o estágio motorizado através de seu alcance de movimento. Certifique-se de que o feixe permanece centrado na abertura e a silhueta do capilar pode ser observada por toda parte. O diâmetro da abertura deve ser menor do que a largura do feixe focalizado em cada ponto da faixa do estágio motorizado.
- Passe a água pelo capilar a 20 μL/min por pelo menos um minuto para lavar o capilar.
- Clique no botão Iniciar Dados + AutoZero no software dosímetro para zerar o dosímetro na água e iniciar a coleta de dados.
NOTA: Se o sistema tampão para FPOP tiver absorvância UV significativa a 265 nm, o sistema FPOP deve ser zerado no buffer, não na água.
- Clique no botão Iniciar Dados + AutoZero no software dosímetro para zerar o dosímetro na água e iniciar a coleta de dados.
- Defina a taxa de fluxo calculada na bomba de seringa.
- A taxa de fluxo da amostra de proteína depende do volume irradiado por tiro(VIrr),do número de tiros a laser por segundo(R),e da desejada fração de volume de exclusão unirradiated(FEx)para corrigir os efeitos de fluxo laminar e a difusão amostral (0,15-0,30 recomendado)2,,19,,20. Calcule o VIrr (em μL) com base no diâmetro interno do capilar em mm (d) e na largura do ponto laser que impinge sobre o capilar (ou seja, a largura da abertura) em mm(w) usando a seguinte equação:
VIrr = π(d/2)2w - Calcule a taxa de fluxo desejada (em μL/min) com base na seguinte equação:
Fluxo = 60R[VIrr (1 + FEx)]
- A taxa de fluxo da amostra de proteína depende do volume irradiado por tiro(VIrr),do número de tiros a laser por segundo(R),e da desejada fração de volume de exclusão unirradiated(FEx)para corrigir os efeitos de fluxo laminar e a difusão amostral (0,15-0,30 recomendado)2,,19,,20. Calcule o VIrr (em μL) com base no diâmetro interno do capilar em mm (d) e na largura do ponto laser que impinge sobre o capilar (ou seja, a largura da abertura) em mm(w) usando a seguinte equação:
2. Preparação da solução proteica para FPOP
- Prepare a proteína nas duas ou mais condições diferentes a serem comparadas (por exemplo, ligadas a ligas e sem ligante; agregada e monômera; sozinha e com um parceiro de ligação proteína-proteína; etc.) para detectar as alterações de conformação.
- Defina o volume total utilizado para fpop para atender às necessidades do experimento. O limite mínimo geralmente depende do volume da capilarização de irradiação e do material necessário para detecção robusta e quantificação relativa, e variará dependendo em grande parte do sistema LC-MS/MS utilizado e do método de processamento da amostra pós-rotulagem. O volume total de soluções FPOP comumente utilizadas em nosso grupo é de 20 μL após a adição de peróxido de hidrogênio. A concentração final da proteína é comumente 1-10 μM, com glutamina de 17 mM (para limitar a vida útil do radical hidroxila), 1 mM de adenina (para agir como um dosímetro radical)13,,17 e 10 mM tampão fosfato (um tampão que é um pobre catador de radicais hidroxil). As amostras são geralmente preparadas com múltiplas réplicas para permitir a modelagem estatística dos resultados.
- Para fins mais gerais, prepare amostras em triplicado em ambos os estados, além de pelo menos uma amostra para usar como controle sem laser para medir a oxidação de fundo. Prepare 18 μL deste mix de soluções FPOP.
NOTA: Muitos tampões e aditivos comumente usados na bioquímica são catadores radicais hidroxis. Estes aditivos e buffers podem ser usados; no entanto, podem ocorrer reduções na oxidação devido à limpeza radical hidroxílida do tampão. Em geral, mantenha todos os aditivos no mínimo exigido pelo sistema biológico para maximizar o rendimento da oxidação proteica. Sulfóxido de dimetila deve ser evitado devido à propensão de gerar radicais secundários; dimetilformamida tem sido uma alternativa útil em nossas mãos. Ao usar tampões que são fortes catadores radicais hidroxis, a glutamina pode muitas vezes ser excluída do mix de soluções FPOP.
- Para fins mais gerais, prepare amostras em triplicado em ambos os estados, além de pelo menos uma amostra para usar como controle sem laser para medir a oxidação de fundo. Prepare 18 μL deste mix de soluções FPOP.
- Prepare 1 M de peróxido de hidrogênio imediatamente antes do experimento FPOP.
NOTA: 30% de peróxido de hidrogênio como comumente vendido pelos fornecedores inclui um estabilizador, o que aumenta a vida útil. Uma vez diluído, o peróxido de hidrogênio deve ser usado rapidamente, definitivamente no mesmo dia. Peróxido de hidrogênio também deve ser regularmente testado para decomposição por FPOP usando um dosímetro radical hidroxílico. - Prepare tubos de microcentrifuuge contendo 25 μL de solução de saciamento de 0,5 μg/μL de amida de methionina e 0,5 μg/μL catalase. Se um volume amostral superior a 20 μL for usado para FPOP, aumente proporcionalmente o volume da solução de saciamento.
3. Realize o experimento FPOP
- Adicione 2 μL de peróxido de hidrogênio nos 18 μL da mistura de solução FPOP. Misture o conteúdo suavemente com uma pipeta e gire rapidamente a solução para a parte inferior dos tubos de microcentrifuuge. Colete imediatamente com uma seringa gastight e carregue na bomba de seringa.
- Inicie o fluxo na bomba de seringa com a taxa de fluxo determinada na etapa 1.8.1 (tipicamente entre 8-16 μL/min) clicando no botão Iniciar bomba no software dosímetro.
- Monitore a leitura de adenina em tempo real utilizando o dosímetro inline (ver Tabela de Materiais) e colete a amostra em resíduos. Espere o sinal do Abs265 estabilizar.
- Clique no botão Iniciar Flash no software dosímetro para começar a disparar o laser na taxa de repetição e energia predefinidas.
- Monitore a leitura de adenina em tempo real usando um dosímetro inline (ver Tabela de Materiais); a diferença no Abs265 com o laser desligado e o laser ligado é a leitura ΔAbs265.
NOTA: O aparecimento de leituras altamente instáveis do Abs265 ao disparar o laser na presença de peróxido de hidrogênio deve-se à geração de bolhas em solução. Reduza a fluência do laser e/ou a concentração de peróxido de hidrogênio para eliminar as bolhas.
4. Realizar compensação
NOTA: Diferentes ligantes, tampões, etc. podem ter capacidade de limpeza diferente para radicais hidroxil. É importante garantir que doses radicais hidroxil eficazes comparáveis estejam disponíveis para reagir com proteína em diferentes amostras. Isso é feito garantindo uma resposta radical dosímetro hidroxílico igual entre as amostras. Utilizando dosimetria de adenina, a mudança na absorção uv a 265 nm (ΔAbs265) reflete a dose radical hidroxila eficaz; quanto maior o ΔAbs265,maior a dose radical de hidroxyl eficaz.
- Compare a leituraΔAbs 265 obtida com o dosímetro inline com a leitura desejada ΔAbs265 obtida por experimentos ou controles anteriores. Uma leituraΔAbs 265 inferior à leitura desejada indica uma dose eficaz insuficiente de radicais hidroxil; uma leitura ΔAbs265 indica uma dose radical eficaz que é muito alta. Se a leituraΔAbs 265 estiver no nível desejado, colete a amostra imediatamente após a irradiação a laser no tampão de sásia17.
- Compense a dose radical eficaz para equalizar os ΔAbs265. Essa compensação pode ser realizada de três maneiras: alterar a concentração de peróxido de hidrogênio, aumentar a fluência do laser alterando a energia do laser por pulso, ou aumentar a fluência do laser alterando o plano focal da lente focal.
- Para fazer uma grande mudança (>10 mAU) na leituraΔAbs 265, refaça a amostra com mais ou menos peróxido de hidrogênio e reprise a amostra conforme a Seção 3.
- Para fazer uma pequena alteração na leituraΔAbs 265 em tempo real, ajuste o plano focal do feixe incidente ajustando a posição da lente focal usando o Estágio Motorizado de 50 mm. Aproximar o plano focal da posição do capilar aumentará a leituraΔAbs 265; trazer o plano focal mais longe da posição do capilar diminuirá a leitura ΔAbs265.
- Monitore a adenina ΔAbs265 para medir a quantidade efetiva de radical hidroxyl presente na amostra após a irradiação a laser13. O monitoramento em tempo real com um detector capilar UV inline permite compensação em tempo real, conforme descrito em 4.2.2; ajustar a posição da lente usando o estágio motorizado até que a leituraΔAbs 265 seja igual à leitura desejada. As medidas de absorção pós-experimental com um espectrofotômetro UV também são precisas, mas exigem que novas amostras sejam usadas para cada dose radical eficaz.
5. Digerir as amostras de proteína
NOTA: A trippsina é mais comumente usada para digerir amostras de proteínas para FPOP, e é a protease usada neste protocolo. É um protease confiável que gera peptídeos com locais básicos tanto no N- quanto no C-terminus, promovendo íons de peptídeos carregados multiplicados em MS. Além disso, ele se apóia após lisina e arginina, dois aminoácidos que são apenas moderadamente reativos aos radicais hidroxil; portanto, alterações no padrão de digestão devido à oxidação de analito é rara. Outras proteases têm sido utilizadas com sucesso com fpop21,mas deve-se tomar cuidado para garantir que os padrões de digestão sejam comparáveis entre amostras nãoxidizadas e oxidadas.
- Meça o volume final da amostra FPOP saciada. Adicione 500 mM Tris, pH 8.0 com 10 mM CaCl2 contendo 50 mM dithiothiothreitol (DTT) à solução proteica após saciar a uma concentração final de 50 mM Tris, 1 mM CaCl2 e 5 mM DTT.
- Aqueça a amostra de proteína a 95 °C por 15 minutos.
- Esfrie imediatamente a amostra no gelo por 2 minutos.
- Adicione a razão de peso de trippsina/proteína de 1:20 às amostras.
- Digerir a proteína durante a noite a 37 °C com a mistura.
- Pare a reação de digestão pela adição de ácido fórmico de 0,1% e/ou aquecimento da amostra a 95 °C por 10 minutos.
- Adicione 2 mM DTT às amostras e aqueça a 60 °C por 15 minutos imediatamente antes de LC-MS/MS.
NOTA: Embora outros grupos tenham relatado alquilação de thiols em experimentos FPOP, em nossas mãos temos notado produtos laterais sobre alquilação de proteínas oxidadas (possivelmente devido à reação com carbonisoes nucleofílicos formados como um produto de oxidação menor). Por isso, optamos por evitar a alquilação de thiols quando possível.
6. Realize espectrometria de massa cromatografia líquida tandem (LC-MS/MS)
- Prepare a fase móvel A consistindo em água contendo 0,1% de ácido fórmico e fase B móvel consistindo de acetonitrilo com ácido fórmico de 0,1%.
- Carregue a amostra primeiro em uma coluna de armadilha C18 (300 μm I.D. x 5 mm 100 Å tamanho de poros, 5 μm tamanho de partícula) trapping cartucho e lavagem com 2% solvente B por 3 minutos a uma taxa de fluxo de 5,0 μL/min para remover sais e moléculas pequenas hidrofílicas.
- Em seguida, separe os peptídeos na nanocolumn C18 (0,75 mm x 150 mm, tamanho de partícula de 2 μm, 100 Å poro) a uma taxa de fluxo de 300 nL/min. O gradiente consiste em um aumento linear de 2 a 35% de solvente B ao longo de 22 min, rampado para 95% solvente B acima de 5 min e mantido por 3 min para lavar a coluna, e depois retornou a 2% B sobre 3 min e segurou por 9 min para ree equilibrar a coluna.
NOTA: Este gradiente é suficiente para LC-MS/MS da maioria das misturas FPOP de uma e duas proteínas que buscam fazer quantificação no nível de peptídeo. A porcentagem de solvente B pode precisar ser alterada para aumentar a resolução de peptídeos em casos raros em que peptídeos interferem uns com os outros devido a tempos de retenção semelhantes e valores m/z. FPOP22 em escala proteome ou projetos experimentais que buscam separar isômeros de produto de oxidação de peptídeos1,,23,,24,25 podem exigir gradientes de LC mais longos e estão fora do escopo deste relatório. - Elute os peptídeos diretamente na fonte de nanospray de um espectrômetro de massa de alta resolução usando um emissor de nanospray condutivo.
- Adquira os dados no modo íon positivo. Coloque a tensão do spray em 2400 V, e a temperatura do tubo de transferência de íons para 300 °C.
- Adquira os exames de MS completos de m/z 250 a 2000 em uma resolução nominal em m/z 200 de 60.000 seguidos por oito subsequentes varreduras de íons lineares dependentes de dados MS/MS nos oito íons peptídeos mais abundantes usando dissociação induzida por colisão a 35% de energia normalizada para identificar os peptídeos. Fragmente os peptídeos até cinco vezes dentro dos 30 s e, em seguida, transfira para uma lista de exclusão para 60 s.
7. Processamento de dados e cálculo da oxidação média dos peptídeos
- Determine a cobertura sequencial dos valores de proteína, m/z e tempos de retenção de peptídeos não oxidados usando o mecanismo de busca de proteômica MS/MS.
- Defina a tolerância de massa precursora a 10 ppm e permita até dois locais de decote perdidos para as amostras digeridas de trippsina, usando a especificidade padrão do decote de trippsina.
- Defina a tolerância em massa do fragmento de peptídeo para 0,4 Daltons.
- Com base na razão m/z dos peptídeos não modificados detectados e nas mudanças de massa conhecidas dos principais produtos de oxidação, calcule o m/z dos vários produtos teóricos de oxidação de cada peptídeo4,,26,,27,,28,,29.
- Identifique o cromatograma de íon extraído desses valores m/z usando software para visualizar a corrida espectrométrica de massa(Figura 4). Identifique os produtos de oxidação de peptídeos com base em seu m/z,seu estado de carga e a semelhança no tempo de eluição com o peptídeo não modificado. Em nossas mãos, os produtos de oxidação de peptídeos eluto entre 240 segundos antes de 180 segundos após o peptídeo não modificado usando o gradiente LC acima. Como a oxidação muitas vezes resultará em múltiplos produtos de oxidação isomeric, é comum observar múltiplos picos parcialmente resolvidos nos cromatagramas de íons extraídos de produtos de oxidação de peptídeos, como mostrado na Figura 4. Os produtos de oxidação de peptídeos são quantificados com base na área do pico nos cromatgramas de íons extraídos.
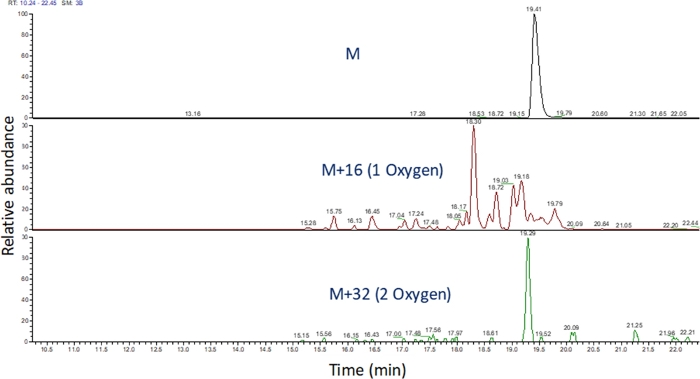
Figura 4: Cromatograma de íon extraído de um peptídeo e seus produtos de oxidação após fpop. O m/z dos produtos de oxidação de peptídeos é calculado com base no m/z do peptídeo não oxidado e nos produtos de oxidação conhecidos; e as áreas desses produtos peptídeos são determinadas. A área dos produtos peptídeos é então utilizada para o cálculo dos eventos médios de oxidação por peptídeo. Clique aqui para ver uma versão maior desta figura.
- Calcule a oxidação média dos peptídeos usando a seguinte equação.
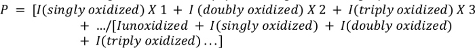
onde P denota o número médio de eventos de oxidação por molécula de peptídeo, e eu represento a área de pico do peptídeo nãooxidado (Iunoxidizado) e o peptídeo com n eventos de oxidação. Note que eu (singly oxidado) incluiria não apenas adições de um único átomo de oxigênio, mas também outros eventos de oxidação única menos comuns que o pesquisador pode optar por medir (por exemplo, descarboxingação oxidativa, formação de carbonyl, etc.) 4,26,27,28,29.
Resultados
Comparação da pegada de peptídeo de corrente pesada do biossimilar adalimumabe em tampão fosfato e quando aquecido a 55 °C por 1 h mostram resultados interessantes. O teste t do aluno é utilizado para a identificação de peptídeos que são significativamente alterados nessas duas condições (p ≤ 0,05). Os peptídeos 20-38, 99-125, 215-222, 223-252, 260-278, 376-413 e 414-420 mostram proteção significativa do solvente quando a proteína é aquecida para formar agregados(Figura 5)...
Discussão
Técnicas estruturais baseadas em espectrometria de massa, incluindo troca de deutério de hidrogênio, ligação química, rotulagem covalente e espectrometria de massa de spray nativo e mobilidade de íons têm crescido rapidamente em popularidade devido à sua flexibilidade, sensibilidade e capacidade de lidar com misturas complexas. O FPOP possui várias vantagens que aumentaram sua popularidade na área de técnicas estruturais baseadas em espectrometria de massa. Como a maioria das estratégias de rotulagem covalen...
Divulgações
Joshua S. Sharp revela um interesse financeiro significativo na GenNext Technologies, Inc., uma pequena empresa que busca comercializar tecnologias para análise de estrutura de ordem superior de proteínas, incluindo a pegada de proteína radical hidroxíl.
Agradecimentos
Reconhecemos que o financiamento de pesquisa do Instituto Nacional de Ciências Médicas Gerais concede R43GM125420-01 para apoiar o desenvolvimento comercial de um dispositivo FPOP benchtop e R01GM127267 para o desenvolvimento de protocolos de padronização e dosimetria para FPOP de alta energia.
Materiais
| Name | Company | Catalog Number | Comments |
| Adenine | Acros Organics | 147440250 | Soluble in water upto 3.5 mM |
| Aperture | Edmund Optics | 39-905 | 1000 μm Aperture Diameter, Gold-Plated Copper Aperture |
| Aperture holder | Edmund Optics | 53-287 | 25.8mm Outer Diameter, Precision Pinhole Mount |
| Catalse | Sigma Aldrich | C-40 | Catalase from bovine liver, lyophilized powder, ≥10,000 units/mg protein |
| COMPex Pro laser | Coherent | 1113836 | COMPexPRO 102, F-Vversion, KrF laser, No XeCl |
| Dithiotheitol (DTT) | Promega | V3151 | DTT, Molecular Grade (DL-Dithiothreitol) |
| Fraction collector | GenNext Technologies, Inc. | N/A | Automated fraction collector |
| Fused silica capillay | Molex | 1068150023 | Polymicro Flexible Fused Silica Capillary Tubing, Inner Diameter 100 µm, Outer Diameter 375 µm, TSP100375 |
| Glutamine | Acros Organics | 119951000 | L(+)-Glutamine, 99% |
| Holder for lens | Edmund Optics | 03-668 | 53 mm Outer Diameter, Three-Screw Adjustable Ring Mount |
| Hydrogen peroxide | Fisher Scientific | H325-100 | Hydrogen Peroxide, 30% (Certified ACS), Fisher Chemical |
| LC-MS/MS system | Thermo Scientific | IQLAAEGAAPFADBMBCX | Dionex Ultimate 3000 coupled to Orbitap Fusion Tribrid mass spectrometer |
| Mas spec grade Acetonitrile | Fisher Scientific | A955-1 | Acetonitrile, Optima LC/MS Grade, Fisher Chemical |
| Mass spec grade formic acid | Fisher Scientific | A117-50 | Formic Acid, 99.0+%, Optima™ LC/MS Grade, Fisher Chemical |
| Mass spec grade water | Fisher Scientific | W6-4 | Water, Optima LC/MS Grade, Fisher Chemical |
| MES buffer | Sigma Aldrich | M0164 | MES hemisodium salt |
| Methionine amide | Bachem | 4000594.0005 | H-met-NH2.HCl |
| Micro V clamp | Thor Labs | VK250 | Micro V-clamp with stainless steel blades |
| Motorized stage | Edmund Optics | 68-638 | 50mm Travel Motorized Stage System with Manual Control |
| Nano C18 colum | Thermo Scientific | 164534 | Acclaim PepMap 100 C18 HPLC Columns |
| Optical bench | Edmund Optics | 56-935 | 18" x 18" breadboard |
| Pioneer FPOP Module System | GenNext Technologies, Inc. | N/A | Inline FPOP Radical Dosimetry System |
| Post holder | Edmund Optics | 58-979 | 3" Length, ¼-20 Thread, Post Holder |
| Sodium phosphate dibasic | Fisher Scientific | BP331-500 | Sodium Phosphate Dibasic Heptahydrate (Colorless-to-White Crystals), Fisher BioReagents |
| Sodium phosphate monobasic | Fisher Scientific | BP330-500 | Sodium Phosphate Monobasic Monohydrate (Colorless-to-white Crystals), Fisher BioReagents |
| Syringe | Hamilton | 81065 | 100 µL, Model 1710 RN SYR, Small Removable NDL, 22s ga, 2 in, point style 3 |
| Syringe pump | KD Scientific | 788101 | Legato 101 syringe pump |
| Trap C18 column | Thermo Scientific | 160454 | Thermo Scientific Acclaim PepMap 100 C18 HPLC Columns |
| Tris | Sigma Aldrich | 252859 | Tris(hydroxymethyl)aminomethane |
| Trypsin | Promega | V5111 | Sequencing Grade Modified Trypsin |
| UV plano convex lens | Edmund Optics | 84-285 | 30 mm Dia. x 120 mm FL Uncoated, UV Plano-Convex Lens |
Referências
- Kaur, P., Kiselar, J., Yang, S., Chance, M. R. Quantitative protein topography analysis and high-resolution structure prediction using hydroxyl radical labeling and tandem-ion mass spectrometry (MS). Molecular & Cellular Proteomics. 14 (4), 1159-1168 (2015).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of the American Society for Mass Spectrometry. 16 (12), 2057-2063 (2005).
- Buxton, G. V., Greenstock, C. L., Helman, W. P., Ross, A. B. Critical review of rate constants for reactions of hydrated electrons, hydrogen atoms and hydroxyl radicals (·OH/·O- in Aqueous Solution. Journal of Physical and Chemical Reference Data. 17 (2), 513 (1988).
- Xu, G., Chance, M. R. Radiolytic modification and reactivity of amino acid residues serving as structural probes for protein footprinting. Analytical Chemistry. 77 (14), 4549-4555 (2005).
- Sharp, J. S., Tomer, K. B. Effects of anion proximity in peptide primary sequence on the rate and mechanism of leucine oxidation. Analytical Chemistry. 78 (14), 4885-4893 (2006).
- Huang, W., Ravikumar, K. M., Chance, M. R., Yang, S. Quantitative mapping of protein structure by hydroxyl radical footprinting-mediated structural mass spectrometry: a protection factor analysis. Biophysical Journal. 108 (1), 107-115 (2015).
- Xie, B., Sood, A., Woods, R. J., Sharp, J. S. Quantitative protein topography measurements by high resolution hydroxyl radical protein footprinting enable accurate molecular model selection. Scientific Reports. 7 (1), 4552 (2017).
- Li, Z., et al. High structural resolution hydroxyl radical protein footprinting reveals an extended Robo1-heparin binding interface. Journal of Biological Chemistry. 290 (17), 10729-10740 (2015).
- Li, X., et al. Structural analysis of the glycosylated intact HIV-1 gp120-b12 antibody complex using hydroxyl radical protein footprinting. Biochemistry. 56 (7), 957-970 (2017).
- Li, K. S., Shi, L., Gross, M. L. Mass spectrometry-based fast photochemical oxidation of proteins (FPOP) for higher order structure characterization. Accounts of Chemical Research. 51 (3), 736-744 (2018).
- Li, J., Chen, G. The use of fast photochemical oxidation of proteins coupled with mass spectrometry in protein therapeutics discovery and development. Drug Discovery Today. 24 (3), 829-834 (2019).
- Roush, A. E., Riaz, M., Misra, S. K., Weinberger, S. R., Sharp, J. S. Intrinsic buffer hydroxyl radical dosimetry using Tris(hydroxymethyl)aminomethane. Journal of the American Society for Mass Spectrometry. 31 (2), 169-172 (2020).
- Xie, B., Sharp, J. S. Hydroxyl radical dosimetry for high flux hydroxyl radical protein footprinting applications using a simple optical detection method. Analytical Chemistry. 87 (21), 10719-10723 (2015).
- Niu, B., Zhang, H., Giblin, D., Rempel, D. L., Gross, M. L. Dosimetry determines the initial OH radical concentration in fast photochemical oxidation of proteins (FPOP). Journal of the American Society for Mass Spectrometry. 26 (5), 843-846 (2015).
- Niu, B., et al. Incorporation of a reporter peptide in FPOP compensates for adventitious scavengers and permits time-dependent measurements. Journal of the American Society for Mass Spectrometry. 28 (2), 389-392 (2017).
- Garcia, N. K., Sreedhara, A., Deperalta, G., Wecksler, A. T. Optimizing hydroxyl radical footprinting analysis of biotherapeutics using internal standard dosimetry. Journal of the American Society for Mass Spectrometry. 31 (7), 1563-1571 (2020).
- Sharp, J. S., Misra, S. K., Persoff, J. J., Egan, R. W., Weinberger, S. R. Real time normalization of fast photochemical oxidation of proteins experiments by inline adenine radical dosimetry. Analytical Chemistry. 90 (21), 12625-12630 (2018).
- Zhang, B., Cheng, M., Rempel, D., Gross, M. L. Implementing fast photochemical oxidation of proteins (FPOP) as a footprinting approach to solve diverse problems in structural biology. Methods. 144, 94-103 (2018).
- Konermann, L., Stocks, B. B., Czarny, T. Laminar flow effects during laser-induced oxidative labeling for protein structural studies by mass spectrometry. Analytical Chemistry. 82 (15), 6667-6674 (2010).
- Gau, B. C., Sharp, J. S., Rempel, D. L., Gross, M. L. Fast photochemical oxidation of protein footprints faster than protein unfolding. Analytical Chemistry. 81 (16), 6563-6571 (2009).
- Li, K. S., et al. Hydrogen-Deuterium exchange and hydroxyl radical footprinting for mapping hydrophobic interactions of human bromodomain with a small molecule Inhibitor. Journal of the American Society for Mass Spectrometry. 30 (12), 2795-2804 (2019).
- Espino, J. A., Jones, L. M. Illuminating biological interactions with in vivo protein footprinting. Analytical Chemistry. 91 (10), 6577-6584 (2019).
- Charvatova, O., et al. Quantifying protein interface footprinting by hydroxyl radical oxidation and molecular dynamics simulation: application to galectin-1. Journal of the American Society for Mass Spectrometry. 19 (11), 1692-1705 (2008).
- Gau, B., Garai, K., Frieden, C., Gross, M. L. Mass spectrometry-based protein footprinting characterizes the structures of oligomeric apolipoprotein E2, E3, and E4. Biochemistry. 50 (38), 8117-8126 (2011).
- Gau, B. C., Chen, J., Gross, M. L. Fast photochemical oxidation of proteins for comparing solvent-accessibility changes accompanying protein folding: Data processing and application to barstar. Biochimica et Biophysica Acta. 1834 (6), 1230-1238 (2013).
- Garrison, W. M. Reaction mechanisms in the radiolysis of peptides, polypeptides, and proteins. Chemical Reviews. 87 (2), 381-398 (1987).
- Xu, G., Chance, M. R. Radiolytic modification of sulfur-containing amino acid residues in model peptides: fundamental studies for protein footprinting. Analytical Chemistry. 77 (8), 2437-2449 (2005).
- Xu, G., Chance, M. R. Radiolytic modification of acidic amino acid residues in peptides: probes for examining protein-protein interactions. Analytical Chemistry. 76 (5), 1213-1221 (2004).
- Xu, G., Takamoto, K., Chance, M. R. Radiolytic modification of basic amino acid residues in peptides: probes for examining protein-protein interactions. Analytical Chemistry. 75 (24), 6995-7007 (2003).
- Misra, S. K., Orlando, R., Weinberger, S. R., Sharp, J. S. Compensated hydroxyl radical protein footprinting measures buffer and excipient effects on conformation and aggregation in an adalimumab biosimilar. AAPS Journal. 21 (5), 87 (2019).
- Simmons, D. A., Konermann, L. Characterization of transient protein folding intermediates during myoglobin reconstitution by time-resolved electrospray mass spectrometry with on-line isotopic pulse labeling. Biochemistry. 41 (6), 1906-1914 (2002).
- Vahidi, S., Konermann, L. Probing the time scale of FPOP (fast photochemical oxidation of proteins): radical reactions extend over tens of milliseconds. Journal of the American Society for Mass Spectrometry. 27 (7), 1156-1164 (2016).
- Chance, M. R. Unfolding of apomyoglobin examined by synchrotron footprinting. Biochemical and Biophysical Research Communications. 287 (3), 614-621 (2001).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chemical Reviews. 107 (8), 3514-3543 (2007).
- Zhang, Y., Rempel, D. L., Zhang, H., Gross, M. L. An improved fast photochemical oxidation of proteins (FPOP) platform for protein therapeutics. Journal of the American Society for Mass Spectrometry. 26 (3), 526-529 (2015).
- Cornwell, O., Radford, S. E., Ashcroft, A. E., Ault, J. R. Comparing hydrogen deuterium exchange and fast photochemical oxidation of proteins: a structural characterisation of wild-type and ΔN6 β(2)-microglobulin. Journal of the American Society for Mass Spectrometry. 29 (2), 2413-2426 (2018).
- Xie, B., Sharp, J. S. Relative Quantification of sites of peptide and protein modification using size exclusion chromatography coupled with electron transfer dissociation. Journal of the American Society for Mass Spectrometry. 27 (8), 1322-1327 (2016).
- Srikanth, R., Wilson, J., Vachet, R. W. Correct identification of oxidized histidine residues using electron-transfer dissociation. Journal of Mass Spectrometry. 44 (5), 755-762 (2009).
- Li, X., Li, Z., Xie, B., Sharp, J. S. Improved identification and relative quantification of sites of peptide and protein oxidation for hydroxyl radical footprinting. Journal of the American Society for Mass Spectrometry. 24 (11), 1767-1776 (2013).
- Li, X., Li, Z., Xie, B., Sharp, J. S. Supercharging by m-NBA Improves ETD-Based Quantification of Hydroxyl Radical Protein Footprinting. Journal of the American Society for Mass Spectrometry. 26 (8), 1424-1427 (2015).
- Khaje, N. A., Sharp, J. S. Rapid quantification of peptide oxidation isomers from complex mixtures. Analytical Chemistry. 92 (5), 3834-3843 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados