É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Câncer hipoxico impresso em células 3D para recapitulando progressão patológica do câncer sólido
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
A hipóxia é uma marca registrada do microambiente tumoral e desempenha um papel crucial na progressão do câncer. Este artigo descreve o processo de fabricação de um câncer hipóxico baseado na tecnologia de impressão celular 3D para recapitular uma patologia relacionada à hipóxia do câncer.
Resumo
O microambiente do câncer tem um impacto significativo na progressão da doença. Em particular, a hipóxia é o principal motor da sobrevivência do câncer, invasão e chemoresistance. Embora vários modelos in vitro tenham sido desenvolvidos para estudar a patologia do câncer relacionada à hipóxia, a complexa interação do microambiente cancerígeno observado in vivo ainda não foi reproduzida devido à falta de controle espacial preciso. Em vez disso, foram propostas abordagens de biofabricação 3D para criar sistemas microfisiológicos para melhor emulação da ecologia do câncer e avaliação precisa do tratamento anticâncer. Aqui, propomos uma abordagem de impressão celular 3D para fabricar um câncer hipóxico em um chip. Os componentes indutores de hipóxia no chip foram determinados com base em uma simulação computacional da distribuição de oxigênio. Anéis concêntricos de câncer foram impressos usando bioinks contendo células de glioblastoma e células endoteliais para recapitular um tipo de câncer sólido. O chip resultante percebeu a hipóxia central e a malignidade agravada no câncer com a formação de marcadores fisiodicosiológicos representativos. No geral, espera-se que a abordagem proposta para a criação de um sistema microfisiológico mímético de câncer sólido e mímica seja a ponte entre modelos in vivo e in vitro para pesquisa de câncer.
Introdução
O microambiente do câncer é um fator crítico que impulsiona a progressão do câncer. Múltiplos componentes, incluindo sinais bioquímicos, biofísicos e celulares, determinam as características patológicas do câncer. Entre elas, a hipóxia está fortemente associada à sobrevivência do câncer, à proliferação e à invasão1. Devido ao crescimento ilimitado e divisão de células cancerosas, nutrientes e oxigênio são continuamente esgotados, e um gradiente hipóxico é gerado. Em condições de baixo oxigênio, as células ativam o fator de transcrição indutível de hipóxia (HIF) em cascata molecular associada ao HIF. Esse processo induz um núcleo necrosado, desencadeia alterações metabólicas e inicia hiperplasia e metástase do vaso sanguíneo2,3. Posteriormente, a hipóxia nas células cancerosas causa a destruição de tecidos normais vizinhos. Além disso, a hipóxia está fortemente associada à resistência terapêutica de tumores sólidos de modos multifatoriais. A hipóxia pode impedir severamente a radioterapia, uma vez que a radiosensibilidade é limitada devido à espécie de oxigênio reativo1,4. Além disso, diminui os níveis de pH de microambientes de câncer, o que diminui o acúmulo de medicamentos1. Portanto, a reprodução de características patológicas relacionadas à hipóxia in vitro é uma estratégia promissora para achados científicos e pré-clínicos.
A modelagem de um microambiente específico do câncer é essencial para entender o desenvolvimento do câncer e explorar tratamentos adequados. Embora os modelos animais tenham sido amplamente utilizados devido à sua forte relevância fisiológica, existem questões relacionadas às diferenças de espécies e problemas éticos5. Além disso, embora os modelos convencionais 2D e 3D permitam a manipulação e a imagem em tempo real das células cancerosas para uma análise aprofundada, sua complexidade arquitetônica e celular não pode ser totalmente recapitulada. Por exemplo, modelos de spheroid de câncer têm sido amplamente utilizados, já que a agregação de células cancerosas em um esferoide pode naturalmente gerar hipóxia no núcleo. Além disso, um grande número de esferoides celulares de tamanho uniforme foram produzidos utilizando sistemas multi-poços à base de plástico ou silicone6,7. No entanto, a menor flexibilidade no que diz respeito à captura da estrutura heterogênea exata dos tecidos cancerígenos com plataformas convencionais exigiu o estabelecimento de uma tecnologia avançada de biofabização para construir uma plataforma altamente biomimética para melhorar a pesquisa sobre o câncer8.
Os sistemas microfisiológicos 3D (MPSs) são ferramentas úteis para recapitular a geometria complexa e a progressão patológica das célulascancerosas 9. À medida que as células cancerígenas sentem o gradiente bioquímico de fatores de crescimento e quimiocinas e a heterogeneidade mecânica reproduzida no sistema, características importantes do desenvolvimento do câncer podem ser investigadas in vitro. Por exemplo, viabilidade do câncer, malignidade metastática e resistência a medicamentos, dependendo das variadas concentrações de oxigênio tem sido estudada usando MPSs10,11. Apesar dos avanços recentes, a geração de condições hipóxiis de modelos in vitro depende de procedimentos complexos de fabricação, incluindo conexão com bombas de gás físico. Portanto, são necessários métodos simples e flexíveis para construir microambientes específicos para o câncer.
A tecnologia de impressão celular 3D ganhou considerável atenção devido ao seu controle preciso do arranjo espacial de biomateriais para recapitular arquiteturas biológicas nativas12. Em particular, essa tecnologia supera as limitações existentes dos modelos de hipóxia 3D devido à sua alta controlabilidade e viabilidade para a construção das características espaciais do microambiente do câncer. A impressão 3D também facilita a fabricação auxiliada por computador através de um processo camada por camada, fornecendo assim uma construção rápida, precisa e reprodutível de geometrias complexas para imitar arquiteturas de tecidos reais. Além das vantagens das estratégias de fabricação existentes para MPSs 3D, as características fisiofológicas da progressão do câncer podem ser reproduzidas pela padronização dos componentes bioquímicos, celulares e biofísicos13,14.
Aqui, apresentamos uma estratégia de impressão celular 3D para um câncer hipóxico em um chip para recapitular a heterogeneidade de um câncer sólido (Figura 1)15. Os parâmetros de fabricação foram determinados através de uma simulação computacional da formação central de hipóxia no sistema. Anéis concêntricos de câncer foram impressos usando bioinks de colágeno contendo células de glioblastoma e células endoteliais para emular a fisiopatologia do glioblastoma, um tipo de câncer sólido. A formação de um gradiente de oxigênio radial agravou a malignidade do câncer, indicando uma agressividade reforçada. Além disso, indicamos perspectivas futuras para as aplicações do chip a modelos pré-clínicos específicos do paciente. Espera-se que a abordagem proposta para a criação de um sistema microfisiológico mímético de câncer sólido e que altere a distância entre modelos in vivo e in vitro de câncer.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Simulação computacional da formação de gradiente de oxigênio
-
Geração de um modelo de geometria 3D para impressão hipoxica de câncer em um chip
- Execute um software CAD 3D.
- Esboce o modelo de geometria do câncer hipóxico em um chip. Clique em Sketch e selecione o plano desejado para desenhar a geometria. Consulte o desenho (Figura 2A) para a escala de detalhes de cada peça.
- Defina a espessura da geometria clicando em Feature-Protrusion Boss/Base. Digite a espessura desejada (consulte a Figura 2A) na caixa vazia e selecione o ícone de verificação verde para formar a geometria 3D.
NOTA: A dimensão do câncer-on-a-chip é definida com base nos volumes desejados de mídia e hidrogel. No presente experimento, os volumes desejados de mídia e hidrogel foram de aproximadamente 1.500 μL e 500 μL, respectivamente, com base nas experiências práticas anteriores para resolução de bioimpressora baseada em extrusão. - Salve o arquivo de geometria como um formato de arquivo CAD 3D (.prt ou .stl).
-
Determinação da densidade celular para indução do núcleo hipóxico
- Execute um programa de simulação de difusão física.
- Clique no LiveLink e selecione o programa CAD usado. Clique em Sincronizar para importar a geometria do câncer hipóxico no programa de simulação. Como o espaço interno da câmara será preenchido com um meio cultural em um ambiente experimental real, o oxigênio se difundirá através do espaço interno da câmara e da construção celular, que será composta de hidrogéis carregados de células.
NOTA: Consulte o estudo anterior para obter detalhes sobre os parâmetros físicos15. - Defina a geometria 3D importada como um volume de controle do espaço em que o oxigênio se difunde, e as células consomem oxigênio(Figura 2B).
- Execute uma análise de computador para análise de difusão de gás seguindo um guia do usuário e métodos previamente estabelecidos16,17.
- A partir dos resultados da análise do computador, exporte os dados estimados de concentração de oxigênio sobre a seção transversal A-A' em cada ponto de tempo seguindo o guia do usuário. A equação governante é baseada na primeira lei de Fick, expressa em Eq. (1) (Figura 2C).
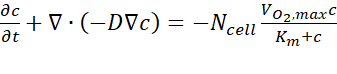
onde c é a concentração, D é o coeficiente de difusão de oxigênio, acélula N é a densidade das células, é a taxa máxima de aumento de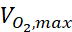 oxigênio, e Km é a constante Michaelis-Menten. As constantes foram aplicadas conforme descrito em publicação anterior15.
oxigênio, e Km é a constante Michaelis-Menten. As constantes foram aplicadas conforme descrito em publicação anterior15.
NOTA: Cada ponto de tempo significa um ponto de passo para observar a variação de difusão de oxigênio ao longo do tempo. - Avalie se o nível mínimo de oxigênio atinge um limiar de hipóxia e repita o processo de análise do computador com um incremento ou decréscito da densidade celular.
NOTA: Defina que o gradiente de hipóxia é formado no construto se o nível de oxigênio de 80% na área do hidrogel for inferior a 0,02 mM após 24 h. - Confirme o número de células necessárias para gerar a hipóxia indutora de oxigênio na região central da primeira lei de Fick na etapa 1.2.5 e a simulação resulta da etapa 1.2.6.
NOTA: Neste protocolo, o número de celular foi de 2 × 106 células/cada construção.
2. Cultura celular de células cancerosas e células estromicas
-
Preparação de meios de cultura celular para evitar estresse fisiológico
- Para células U-87 MG (linha celular glioblastoma humana imortalizada), coloque 12 mL de médio águia modificado de alta glicose Dulbecco contendo 10% de soro bovino fetal, Penicilina de 100 U/mL, e 100 μg/mL estreptomicina em um frasco de cultura celular T-75 em uma incubadora umidificada de 37 °C, 5% CO2 por 30 minutos para minimizar os efeitos térmicos e alcalinos do meio nas células.
NOTA: O glioblastoma foi escolhido como um tipo de câncer sólido por ter características agressivas em um ambiente hipóxico. Outros vários tipos de cânceres podem ser aplicados a este modelo. - Para células endoteliais de veia umbilical humana (HUVECs), coloque 12 mL de meio de crescimento celular endotelial em um frasco de cultura celular T-75 em uma incubadora umidificada co2 de 5% a 37 °C por 30 min.
NOTA: Os HUVECs foram escolhidos por ser uma das linhas celulares endoteliais mais representativas. Vários tipos de células estromas também podem ser aplicadas a este modelo.
- Para células U-87 MG (linha celular glioblastoma humana imortalizada), coloque 12 mL de médio águia modificado de alta glicose Dulbecco contendo 10% de soro bovino fetal, Penicilina de 100 U/mL, e 100 μg/mL estreptomicina em um frasco de cultura celular T-75 em uma incubadora umidificada de 37 °C, 5% CO2 por 30 minutos para minimizar os efeitos térmicos e alcalinos do meio nas células.
-
Degelo rápido de células cancerígenas criopreservadas e células estrogonais e sua manutenção
- Mova criovias contendo células 5 x 105 U-87 MG e HUVECs do recipiente de nitrogênio líquido para um gabinete de fluxo laminar. Solte imediatamente e aperte a tampa para liberar a pressão interna.
- Coloque delicadamente as células criopreservadas em um banho de água a 37 °C por 2 minutos, mantendo a tampa fora da água. Enxágüe os frascos com 70% de etanol sob fluxo laminar para evitar contaminação.
- Transfira as células descongeladas para os frascos contendo os meios de cultura celular preparados descritos na etapa 2.1 e coloque os frascos contendo células em uma incubadora umidificada de 5% de CO2 a 37 °C para recuperação celular.
- Refrescar a mídia de cultura celular a cada 2 dias e manter o crescimento celular.
- Após 24 horas de descongelamento, substitua a mídia de cultura celular para evitar a citotoxicidade do sulfóxido de dimetila (DMSO), que foi usado para congelamento celular. Use HUVECs, que já passaram por menos de 6 passagens.
3. Preparação da solução pré-gel de colágeno
-
Solubilização da esponja de colágeno com ácido clorídrico de 0,1 N (HCl)
- Prepare uma solução de 0,1 N HCl e filtre-a com um filtro de seringa de 0,2 μm.
- Para 3 mL de uma solução de pré-gel de colágeno neutralizado de 1% (w/v), prepare esponjas de colágeno cortadas em 5 x 5 mm2 peças e pesando 30 mgs.
- Transfira as peças de colágeno cortadas para um frasco de vidro estéril de 10 mL.
NOTA: Prepare o volume de 1,5 vezes o hidrogel de colágeno necessário, considerando a perda do hidrogel devido à característica pegajosa da solução de colágeno. - Adicione 2,4 mL de 0,1 N HCl no frasco de vidro contendo colágeno e incuba-o no roqueiro a 15 rpm e 4 °C por 3 dias.
NOTA: O volume da solução 0.1 N HCl foi de quatro quintos do volume final do hidrogel de colágeno necessário. Neste caso, 3 mL de colágeno foi preparado. - Após a digestão, peneirar as partículas de colágeno não digeridas usando um coador de células de 40 μm. Armazene a solução de colágeno ácido a 4 °C e use dentro de 7 dias.
-
ajuste de pH para solução de pré-gel de colágeno neutralizado de 1%
- Centrifugar a solução de colágeno ácido a 1224 x g por 5 min a 4 °C.
- Adicione 30 μL de solução vermelha de fenol como indicador de pH a uma concentração final de 1% (v/v) e 300 μL de tampão salino tampão tampão de fosfato de 10x (PBS) a uma concentração final de 10% (v/v) na solução pré-gel de colágeno.
- Neutralize o pH para 7 com hidróxido de sódio de 1 N (NaOH), verificando a mudança de cor.
NOTA: Com base na fórmula, mols H+ = molaridade H+ x volume H+ = mols OH-= molaridade OH- x volume OH-, adicione 240 μL de NaOH. - Adicione água destilada para obter um volume total de 3 mL.
- Após o ajuste do pH, armazene a solução de pré-gel neutralizada de colágeno de 1% (p/v) a 4 °C e use dentro de 3 dias.
NOTA: Para verificar previamente a gelação da solução de pré-gel de colágeno neutralizado, faça gotículas de colágeno de 50 μL em um prato pequeno usando uma pipeta de deslocamento positiva e incuba-as em uma incubadora de 37 °C por 1h. Consulte os seguintes três métodos para verificar a ligação cruzada das gotículas de colágeno. - Verifique se a cor do colágeno se transformou em branco opaco de cor transparente.
- Incline o recipiente e verifique se o colágeno está aderido ao fundo do recipiente.
- Despeje 1x PBS nas gotículas e verifique se a construção do colágeno não está quebrada na solução.
Impressão .3D 4 de barreira permeável a gás
-
Impressão 3D de um molde de poli sacrificial (acetato de etileno-vinil) (PEVA)
- Gere a geometria 3D do molde PEVA sacrificial definido na etapa 1 utilizando um software CAD 3D(Figura 3A).
NOTA: A geometria 3D e a escala detalhada do modelo, incluindo dimensão, unidades e tipos de linha foram mostrados na Figura 2A. - Converta o arquivo CAD 3D em um formato de arquivo STL clicando em Arquivo | Tipo de arquivo de salvamento como STL. Além disso, clique em Opção | Formulário de saída como ASCII para geração de código G.
- Clique em | de Arquivo Abra o arquivo STL e selecione o arquivo STL salvo para importar o arquivo STL gerado. Clique no modelo Slice do trocador STL-CAD para gerar automaticamente o código G do molde PEVA sacrificial (Figura 3B, C).
NOTA: O caminho de impressão é gerado com a conexão de pontos intersetoriados entre a figura fundamental do arquivo STL e o plano de corte (ou seja, camada). Basicamente, a figura fundamental de um fragmento em um arquivo STL é um triângulo que contém as coordenadas 3D. Depois que os pontos cruzados entre o triângulo e a camada são obtidos, um código G para impressão é gerado conectando cada ponto sem um caminho sobreposto em uma camada18. Qualquer algoritmo de geração de código G a bordo pode ser usado para gerar caminhos de impressão para a fabricação do chip. - Prepare um slide de histologia adesiva e hidrofílica e estéril.
NOTA: O vidro de lâmina hidrofílica é fundamental para a ligação permanente de polidimtilsiloxano (PDMS) no vidro e a adesão do colágeno constrói células cancerígenas e células estromáticas. - Imprima o molde peva sacrificial no slide com um bocal de precisão de 50 G a uma pressão pneumática de 500 kPa a 110 °C.
NOTA: A largura da linha é afetada pela taxa de alimentação, medidor de bocal e temperatura do material. O bocal de 50 G foi usado e uma taxa de alimentação de 400 foi aplicada para gerar 500 μm de largura de linha para a parede sacrificial. O medidor de bocal, a pressão pneumática e a taxa de alimentação são definidos com resultados práticos19. A parede de sacrifício precisa ser suficientemente grossa para segurar a solução PDMS, que é o próximo passo de fabricação.
- Gere a geometria 3D do molde PEVA sacrificial definido na etapa 1 utilizando um software CAD 3D(Figura 3A).
-
Fundição de barreira de polidimometiletilaxano (PDMS)
- Misture 6 mL PDMS base elastomer e 0,6 mL agente de cura homogêneamente mais de 5 min em um reservatório plástico. Isso pode fabricar 6 câncer hipóxicos em chips, considerando a perda devido à característica pegajosa do PDMS.
- Carregue a solução PDMS combinada em uma seringa descartável de 10 mL e encaixe a cabeça da seringa com uma ponta de dispensa afilada de plástico de 20 G.
- Encha o molde peva sacrificial com a solução PDMS combinada na seringa. O PDMS misturado encherá o molde de PEVA sacrificial com uma superfície convexa. A altura da barreira PDMS será maior que a do molde PEVA.
- Cure a barreira PDMS em um forno a 40 °C por mais de 36 h para evitar o derretimento do PEVA. Não aumente a temperatura para mais de 88 °C, que é a temperatura de fusão do PEVA.
- Retire o molde de PEVA sacrificial com um par de pinças de precisão e esterilize a barreira permeável a gás a 120 °C em uma autoclave.
5. Preparação de bio-tintas de colágeno encapsuladas por células
-
Desprendimento das células cancerígenas preparadas e células estromas
NOTA: Considerando a viabilidade celular, todo o processo de impressão deve ser concluído o mais rápido possível após a descolagem das células.- Lave o câncer e as células estromicanas com 10 mL de 1x PBS usando uma pipeta sorológica; tratar com 2 mL de 0,25% de trippsina-etilenodiaminetetraactic ácido (EDTA) usando uma pipeta e incuba-los por 3 min a 37 °C.
- Neutralizar as células experimentpsinizadas com 3 mL de mídia de cultura celular; coletar as suspensões das células em tubos cônicos de 15 mL e centrífuga a 516 x g por 5 min a 20 °C.
- Aspirar o supernante lentamente; resuspenda as pelotas de células em mídia de cultura celular de 5 mL e conte o número de células usando um hemótmetro.
- Transfira 5 x 106 células de cada tipo de célula em novos tubos cônicos de 15 mL e centrifuá-los a 516 x g por 5 min a 20 °C.
- Aspire o supernatante e coloque-o em gelo molhado.
-
Mistura de cada tipo de célula com a solução de pré-gel de colágeno neutralizado de 1%
NOTA: Para evitar a solidificação térmica da solução de pré-gel de colágeno neutralizado de 1%, este processo deve ser realizado em gelo molhado.- Resuspenda cada tipo de pelota celular coletada na etapa 5.1.4 com 20 μL de mídia de cultura celular cada.
- Adicione 1 mL da solução de pré-gel de colágeno neutralizado de 1% em cada uma das suspensões celulares resuspended e misture-as homogêneamente usando uma pipeta de deslocamento positiva. A concentração final de cada tipo de célula será de 5 x 106 células/mL.
- Transfira as bioinks de colágeno encapsuladas por células em seringas descartáveis de 3 mL usando uma pipeta descartável positiva e armazene as seringas a 4 °C até a impressão celular 3D.
6.3D impressão celular de anéis concêntricos cancero-stroma
-
Impressão celular 3D de bioinks de colágeno encapsulando células cancerosas e células estromicas
- Gere a geometria 3D dos anéis concêntricos de câncer-stroma definidos na etapa 1.2 usando um software CAD 3D.
NOTA: As dimensões dos anéis concêntricos de estroma do câncer são definidas através de parâmetros simulados. As dimensões do parâmetro de dimensão final são mostradas na Figura 3A. - Converta o arquivo CAD 3D em um formato de arquivo STL e gere um código G dos anéis concêntricos de câncer-stroma usando um trocador STL-CAD.
NOTA: Consulte a nota na etapa 4.1.2 para o algoritmo de geração de código G. - Carregue os bioinks de colágeno encapsulados por células contidos em seringas descartáveis de 3 mL na cabeça da impressora 3D e coloque a temperatura da cabeça e da placa em 15 °C.
NOTA: Se a temperatura da cabeça e da placa da impressora atingir mais de 37 °C, o bioink fica cruzado e não é mais impresso. - Carregue o caminho de impressão gerado no software de controle da impressora 3D.
- Clicando no botão Iniciar, imprima os bioinks de colágeno encapsulando células cancerosas e células estromais na barreira permeável a gás seguindo o código G carregado com uma agulha de plástico de 18 G à pressão pneumática de aproximadamente 20 kPa a 15 °C.
- No final de cada operação de impressão, coloque manualmente uma tampa de vidro esterilizada de 22 mm x 50 mm em cima da barreira permeável a gás para gerar o gradiente hipoxico.
NOTA: Compare dois grupos dependendo da presença de tampa de vidro (GR+) e ausência (GR-) disso para verificar a geração do gradiente hipóxico. - Depois de gerar três câncer hypoxic em chips, transfira os chips para uma incubadora a 37 °C por 1 h para cruzar os bioinks de colágeno.
- Gere a geometria 3D dos anéis concêntricos de câncer-stroma definidos na etapa 1.2 usando um software CAD 3D.
-
Conclusão do processo de fabricação e manutenção do câncer hipóxico em um chip
- Após a conclusão de todos os processos de impressão celular 3D do câncer hipóxico em um chip, esfregue suavemente os óculos de cobertura em cima das barreiras permeáveis a gás com o raspador de células para ligação apertada(Figura 4A, B).
NOTA: O vidro de cobertura e a barreira gaso-permeável são montados através de ligação hidrofóbica sem colas químicas, simplesmente raspando a parte colada entre o vidro de cobertura e a barreira PDMS. - Introduza 1,5 mL de meio de crescimento de células endoteliais a cada chip. Para evitar o descolamento da construção do câncer, introduza o meio de cultura celular de um lado do chip. Incline o chip para permitir que a mídia de cultura celular flua usando uma pipeta.
- Refrescar a mídia da cultura celular todos os dias durante uma semana. Use uma pipeta para aspirar o meio de cultura celular; não use uma bomba de pressão.
- Após a conclusão de todos os processos de impressão celular 3D do câncer hipóxico em um chip, esfregue suavemente os óculos de cobertura em cima das barreiras permeáveis a gás com o raspador de células para ligação apertada(Figura 4A, B).
7. Avaliação da viabilidade celular pós-impressão
-
Preparação de amostras e tratamento com solução calcein AM e EthD-1
- Aqueça 1x PBS em um banho de água a 37 °C.
- Prepare a solução de ensaio adicionando 0,75 μL de acetoximilo de calceína (calceina AM) e 3 μL de homodimer de ethidium (EthD-1) a 1,5 mL PBS pré-aquecido.
- Aspire cuidadosamente todas as mídias do chip usando uma pipeta.
- Lave a construção do câncer com PBS pré-armado. Encha 1,5 mL PBS no chip usando uma pipeta e deixe-o ficar por 10 minutos à temperatura ambiente. Para evitar a deformação da construção do câncer, introduza 1x PBS de um lado dos chips e incline os chips para permitir que o PBS 1x flua.
- Aspirar o PBS do chip; trate a solução de ensaio de 1,5 mL e incubar o chip a 37 °C por 20 min usando uma folha para proteger da luz. Use uma pipeta para aspirar 1x PBS; não use uma bomba de sucção.
-
Imagem da viabilidade celular usando um microscópio de fluorescência
- Visualize e capture as células rotuladas usando um microscópio de fluorescência(Figura 4C).
NOTA: Calcein AM marca células vivas com fluorescência verde (comprimento de onda ~488 nm). O EthD-1 representa o sinal de células mortas com fluorescência vermelha (comprimento de onda ~594 nm). - Conte o número de células vivas e mortas usando software de imagem, um programa de processamento de imagem de código aberto e calcule a viabilidade com os números .
- Visualize e capture as células rotuladas usando um microscópio de fluorescência(Figura 4C).
8. Imunofluorescência para validar a formação de hipóxia central e seu efeito sobre a malignidade do câncer
-
Fixação, permeabilização e bloqueio da construção do câncer
- Prepare 1x PBS, 4% paraformaldeído (PFA), 0,1% (v/v) Triton X-100 e 2% (w/v) albumina de soro bovino (BSA) à temperatura ambiente.
- Aspire cuidadosamente toda a mídia do chip usando uma pipeta e enxágue o chip três vezes com 1x PBS. Para evitar a deformação da construção do câncer, introduza 1x PBS de um lado dos chips e incline os chips para permitir que o PBS 1x flua. Entre cada etapa de lavagem, deixe o chip ficar com 1x PBS por 5 minutos para remover soluções residuais.
NOTA: 1x PBS foi aspirado usando uma pipeta, não uma bomba de pressão. - Adicionar 500 μL de 4% PFA à construção de câncer no chip usando uma pipeta; Deixe por 15 minutos e lave três vezes com 1x PBS para corrigir as células na construção do câncer.
- Trate a construção do câncer com 500 μL de 0,1% Triton X-100 usando uma pipeta à temperatura ambiente por 5 minutos e lave três vezes com 1x PBS para solubilizar e permeabilizar a membrana celular.
- Tratar a construção do câncer com 500 μL de 2% de BSA usando uma pipeta à temperatura ambiente por 1h para bloquear epítopos reativos.
NOTA: Cubra o chip com filme de parafina para evitar a evaporação. - Depois de 1h, lave o chip três vezes com 1x PBS.
-
Tratamento com anticorpo primário, anticorpo secundário e DAPI e imagem da estrutura usando um microscópio confocal.
- Prepare anticorpos de controle de isótipo e o coquetel de anticorpos primários diluindo os anticorpos em 1x PBS para cada concentração de trabalho desejada.
NOTA: Os detalhes específicos dos anticorpos estão listados na Tabela de Materiais. As mesmas concentrações de trabalho dos anticorpos de controle do isótipo que os anticorpos primários devem ser utilizadas. - Aspire cuidadosamente todos os 1x PBS do chip usando uma pipeta e trate o chip com solução de anticorpos primários de 200 μL a 4 °C durante a noite. Cubra os chips com filme de parafina para evitar a evaporação.
- Aspire a solução de anticorpos primária e lave o chip três vezes com 1x PBS.
- Diluir anticorpos secundários e DAPI em 1x PBS à concentração de trabalho desejada.
NOTA: Um anticorpo secundário conjugado com fluorescência verde é usado neste caso a uma razão de 1:200. O DAPI foi utilizado em uma proporção de 1:1000. - Aspire cuidadosamente todos os PBS 1x do chip usando uma pipeta e trate o chip com solução de anticorpos secundários-DAPI de 200 μL a 4 °C por 3h. Cubra o chip com filme de parafina para evitar a evaporação e, em seguida, enrole-o com papel alumínio para evitar fotobleaching.
- Aspire a solução secundária de anticorpos-DAPI e lave o chip três vezes com 1x PBS.
- Depois de terminar a etapa de coloração, transfira a construção do câncer para um prato confocal, agarrando suavemente com fórceps.
- Visualize e capture as células rotuladas usando um microscópio confocal(Figura 5).
NOTA: O comprimento de onda do microscópio confocal foi ajustado, dependendo do tipo dos marcadores fluorescentes. Os detalhes específicos dos anticorpos estão listados na Tabela de Materiais. Para detectar eficientemente a posição celular, seria melhor observar os núcleos manchados de DAPI da construção em um primeiro momento. Os comprimentos de onda de detecção/emissão dos sinais fluorescentes foram de 358/461 nm (DAPI, Azul), 494/517 nm (Verde) e 590/617 nm (Vermelho). As ampliações foram 4x, 10x e 20x, ajustadas das mais baixas para as mais altas.
- Prepare anticorpos de controle de isótipo e o coquetel de anticorpos primários diluindo os anticorpos em 1x PBS para cada concentração de trabalho desejada.
9. Análise estatística
-
Contagem de células com programa de processamento de imagens
- Execute um programa de processamento de imagens para contar o número de células vivas e mortas.
- Abra os arquivos de imagem fluorescentes. Clique em | de Arquivo Abra e importe as imagens TIFF.
- Converta as imagens em imagens em escala de cinza de 16 bits. Clique em Imagem | Tipo | Escala de cinza de 16 bits.
- Ajuste o limiar clicando em Imagem | Ajuste | Limiar e, em seguida, selecione a cor das células para ser preto.
- Corte as células mescladas clicando em Process | | binário Divisor de águas para contagem precisa de células.
- Conte o número de células clicando em Analisar e, em seguida, em Analisar partículas três vezes; calcular a média e apresentar os dados como erro médio ± padrão.
NOTA: Os marcadores de imunofluorescência foram analisados comparando a intensidade da fluorescência.
Access restricted. Please log in or start a trial to view this content.
Resultados
O câncer hipóxico foi desenvolvido usando tecnologia de impressão celular 3D auxiliada por computador para recapitular a hipóxia e a patologia relacionada ao câncer(Figura 1). O transporte e o consumo de oxigênio foram simulados utilizando o modelo de geometria 3D. O chip foi projetado na forma de anéis concêntricos para imitar a difusão e esgotamento do oxigênio radial, em tecidos cancerígenos(Figura 2A). Após a defi...
Access restricted. Please log in or start a trial to view this content.
Discussão
Neste estudo, descrevemos o processo de fabricação de um câncer hipóxico baseado na tecnologia de impressão celular 3D. A formação do gradiente hipóxico no chip projetado foi prevista através de simulações de computador. O ambiente que pode induzir um gradiente hipóxico heterogêneo foi reproduzido através de uma estratégia simples combinando a barreira permeável a gás impresso em 3D e a tampa de vidro. As características patológicas relacionadas à hipóxia do glioblastoma, incluindo pseudopalisade e u...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm revelações.
Agradecimentos
Esta pesquisa foi apoiada pela Fundação Nacional de Pesquisa da Coreia (NRF) financiada pelo Ministério da Educação (nº 2020R1A6A1A1A03047902 e NRF-2018H1A2A1062091) e pelo governo da Coreia (MSIT) (N. No. NRF-2019R1C1C1009606 e NRF-2019R1A3A3005437).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Cells | |||
| Human umbilical vein endothelial cells | Promocell | C-12200 | |
| U-87 MG cells | ATCC | ATCC HTB-14 | |
| Disposable | |||
| 0.2 μm syringe filter | Sartorius | 16534-K | |
| 10 mL disposable syringe | Jung Rim | 10ml 21G32 | |
| 10 mL glass vial | Hubena | A0039 | |
| 10 mL Serological pipette tip | SPL lifescience | 91010 | |
| 15 mL conical tube | SPL lifescience | 50015 | |
| 18G plastic needle | Musashi engineering | PN-18G-B | |
| 20G plastic tapered dispense tip | Musashi engineering | TPND-20G-U | |
| 22x50 glass cover | MARIENFIELD | 0101142 | |
| 25 mL Serological pipette tip | SPL lifescience | 90125 | |
| 3 mL disposable syringes | HENKE-JET | 4020-X00V0 | |
| 40 µm cell strainer | Falcon | 352360 | |
| 5 mL Serological pipette tip | SPL lifescience | 91005 | |
| 50 mL conical tube | SPL lifescience | 50050 | |
| 50 mL Serological pipette tip | SPL lifescience | 90150 | |
| 50N precision nozzle | Musashi engineering | HN-0.5ND | |
| Aluminum foil | SINKWANG | ||
| Capillary tips | Gilson | CP1000 | |
| Cell-scrapper | SPL lifescience | 90030 | |
| Confocal dish | SPL lifescience | 200350 | |
| Parafilm | Bemis | PM996 | |
| Pre-coated histology slide | MATSUNAMI | MAS-11 | |
| Reservoir | SPL lifescience | 23050 | |
| T-75 cell culture flask | SPL lifescience | 70075 | |
| Equipment | |||
| 3DX printer | T&R Biofab | ||
| Autoclave | JEIOTECH | AC-12 | |
| Centrifuger | Cyrozen | 1580MGR | |
| Confocal laser microscopy | Olympus Life Science | FV 1000 | |
| Fluorescence microscope | FISHER SCEINTIFIC | O221S366 | |
| Forcep | Korea Ace Scientific | HC.203-30 | |
| Hand tally counter | KTRIO | ||
| Hemocytometer | MARIENFIELD | 0650030 | |
| Incubator | Panasonic | MCO-170AIC | |
| Laminar flow cabinet | DAECHUNG SCIENCE | CB-BMMS C-001 | |
| Metal syringe | IWASHITA engineering | SUS BARREL 10CC | |
| Operating Scissors | Hirose | HC.13-122 | |
| Oven | JEIOTECH | OF-12, H070023 | |
| Positive displacement pipette | GILSON | NJ05652 | |
| Refrigerator | SAMSUNG | CRFD-1141 | |
| Voltex Mixer | DAIHAN scientific | VM-10 | |
| Water bath | DAIHAN SCIENTIFIC | WB-11 | |
| Water purifier | WASSER LAB | DI-GR | |
| Materials | |||
| 0.25 % Trypsin-EDTA | Gibco | 25200-072 | |
| 10x PBS | Intron | IBS-BP007a | |
| 4% Paraformaldehyde | Biosesang | ||
| 70% Ethanol | Daejung | 4018-4410 | |
| Anti-CD31 antibody | Abcam | ab28364 | |
| Anti-HIF-1 alpha antibody | Abcam | ab16066 | |
| Anti-SHMT2/SHMT antibody | Abcam | ab88664 | |
| Anti-SOX2 antibody | Abcam | ab75485 | |
| Bovine Serum Albumin | Thermo scientific | J10857-22 | |
| Collagen from porcine skin | Dalim tissen | PC-001-1g | |
| DAPI (4',6-Diamidino-2-Phenylindole, Dihydrochloride) | Thermofisher | D1306 | |
| Endothelial Cell Growth Medium-2 | Promocell | C22011 | |
| Fetal bovine serum | Gibco | 12483-020 | |
| Goat anti-Mouse IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Theromofisher | A-11001 | |
| Goat anti-Rabbit IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 594 | Theromofisher | A-11012 | |
| High-glucose Dulbecco’s Modified Eagle Medium(DMEM) | Hyclone | SH30243-0 | |
| Hydrochloric acid | Sigma-Aldrich | 311413-100ML | |
| Live/dead assay kit | Invitrogen | L3224 | |
| Mouse IgG1, kappa monoclonal [15-6E10A7] - Isotype Control | Abcam | ab170190 | |
| Penicillin/streptomycin | Gibco | 15140-122 | |
| Phenol red solution | Sigma-Aldrich | P0290-100ML | |
| Poly(ethylene-vinyl acetate) | Poly science | 06108-500 | |
| Polydimethylsiloxane | Dowhitech | sylgard 184 | |
| Rabbit IgG, polyclonal - Isotype Control | Abcam | ab37415 | |
| Sodium hydroxide solution | Samchun | S0610 | |
| Triton X-100 | Biosesang | TRI020-500-50 | |
| Trypan Blue | Sigma-Aldrich | T8154 | |
| Software | |||
| COMSOL Multiphysics 3.5a | COMSOL AB | ||
| IMS beamer | in-house software | ||
| SolidWorks Package | Dassault Systems SolidWorks Corporation |
Referências
- Jing, X., et al. Role of hypoxia in cancer therapy by regulating the tumor microenvironment. Molecular Cancer. 18 (1), 157(2019).
- Al Tameemi, W., Dale, T. P., Al-Jumaily, R. M. K., Forsyth, N. R. Hypoxia-modified cancer cell metabolism. Frontiers in Cell and Developmental Biology. 7, 4(2019).
- Petrova, V., Annicchiarico-Petruzzelli, M., Melino, G., Amelio, I. The hypoxic tumour microenvironment. Oncogenesis. 7 (1), 1-13 (2018).
- Hockel, M., Vaupel, P. Tumor hypoxia: definitions and current clinical, biologic, and molecular aspects. Journal of the National Cancer Institute. 93 (4), 266-276 (2001).
- Kim, H., Lin, Q., Glazer, P. M., Yun, Z. The hypoxic tumor microenvironment in vivo selects the cancer stem cell fate of breast cancer cells. Breast Cancer Research. 20 (1), 16(2018).
- Jeong, G. S., Lee, J., Yoon, J., Chung, S., Lee, S. -H. Viscoelastic lithography for fabricating self-organizing soft micro-honeycomb structures with ultra-high aspect ratios. Nature Communications. 7 (1), 1-9 (2016).
- Razian, G., Yu, Y., Ungrin, M. Production of large numbers of size-controlled tumor spheroids using microwell plates. Journal of Visualized Experiments:JoVE. (81), e50665(2013).
- Nunes, A. S., Barros, A. S., Costa, E. C., Moreira, A. F., Correia, I. J. 3D tumor spheroids as in vitro models to mimic in vivo human solid tumors resistance to therapeutic drugs. Biotechnology and Bioengineering. 116 (1), 206-226 (2019).
- Wan, L., Neumann, C., LeDuc, P. Tumor-on-a-chip for integrating a 3D tumor microenvironment: chemical and mechanical factors. Lab on a Chip. 20 (5), 873-888 (2020).
- Nam, H., Funamoto, K., Jeon, J. S. Cancer cell migration and cancer drug screening in oxygen tension gradient chip. Biomicrofluidics. 14 (4), 044107(2020).
- Palacio-Castañeda, V., Kooijman, L., Venzac, B., Verdurmen, W. P., Le Gac, S. Metabolic switching of tumor cells under hypoxic conditions in a tumor-on-a-chip model. Micromachines. 11 (4), 382(2020).
- Ronaldson-Bouchard, K., Vunjak-Novakovic, G. Organs-on-a-chip: a fast track for engineered human tissues in drug development. Cell Stem Cell. 22 (3), 310-324 (2018).
- Mi, S., Du, Z., Xu, Y., Sun, W. The crossing and integration between microfluidic technology and 3D printing for organ-on-chips. Journal of Materials Chemistry B. 6 (39), 6191-6206 (2018).
- Yi, H. -G., Lee, H., Cho, D. -W. 3D printing of organs-on-chips. Bioengineering. 4 (1), 10(2017).
- Yi, H. -G., et al. A bioprinted human-glioblastoma-on-a-chip for the identification of patient-specific responses to chemoradiotherapy. Nature Biomedical Engineering. 3 (7), 509-519 (2019).
- Kang, T. -Y., Hong, J. M., Jung, J. W., Yoo, J. J., Cho, D. -W. Design and assessment of a microfluidic network system for oxygen transport in engineered tissue. Langmuir. 29 (2), 701-709 (2013).
- Woo Jung, J., et al. Evaluation of the effective diffusivity of a freeform fabricated scaffold using computational simulation. Journal of Biomechanical Engineering. 135 (8), (2013).
- Brown, A. C., De Beer, D. Development of a stereolithography (STL) slicing and G-code generation algorithm for an entry level 3-D printer. 2013 Africon (IEEE). , 1-5 (2013).
- Shim, J. -H., Lee, J. -S., Kim, J. Y., Cho, D. -W. Bioprinting of a mechanically enhanced three-dimensional dual cell-laden construct for osteochondral tissue engineering using a multi-head tissue/organ building system. Journal of Micromechanics and Microengineering. 22 (8), 085014(2012).
- Gillispie, G., et al. Assessment methodologies for extrusion-based bioink printability. Biofabrication. 12 (2), 022003(2020).
- Kim, B. S., Das, S., Jang, J., Cho, D. -W. Decellularized extracellular matrix-based bioinks for engineering tissue-and organ-specific microenvironments. Chemical Reviews. 120 (19), 10608-10661 (2020).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados