Method Article
Cristalização e Determinação Estrutural de uma Enzima:Complexo de Substrato por Cristalografia Serial em um Chip Microfluido Versátil
Neste Artigo
Resumo
Um dispositivo microfluido versátil é descrito que permite a cristalização de uma enzima usando o método de contra-difusão, a introdução de um substrato nos cristais por imersão, e a determinação da estrutura 3D da enzima:complexo substrato por uma análise serial de cristais dentro do chip à temperatura ambiente.
Resumo
A preparação de cristais bem difundidos e seu manuseio antes de sua análise de raios-X são duas etapas críticas de estudos biocristagráficos. Descrevemos um versátil chip microfluido que permite a produção de cristais pelo método eficiente de contra-difusão. O ambiente livre de convecção fornecido pelos canais microfluidos é ideal para o crescimento cristalino e útil para difundir um substrato no local ativo da enzima cristalina. Aqui aplicamos essa abordagem à enzima de adição de CCA da bactéria psicofilílica Planococcus halocryophilus no exemplo apresentado. Após cristalização e difusão/imersão de substratos, a estrutura cristalina do complexo enzimágio:substrato foi determinada à temperatura ambiente por cristalografia serial e a análise de múltiplos cristais diretamente dentro do chip. Todo o procedimento preserva as propriedades genuínas de difração das amostras porque não requer manuseio de cristais.
Introdução
Cristalografia é um método para decifrar a arquitetura 3D de macromoléculas biológicas. Este último é importante para entender como uma enzima seleciona e processa seus substratos. A determinação de uma estrutura cristalina requer a cristalização da macromolécula alvo e o condicionamento dos cristais para sua análise por difração de raios-X1. Tanto a preparação e o manuseio de cristais são etapas cruciais, mas delicadas, que podem afetar a qualidade do cristal e as propriedades de difração, e, assim, a resolução (ou seja, a precisão) da estrutura 3D resultante. Para facilitar a preparação de cristais de alta qualidade e eliminar o manuseio desnecessário para preservar suas propriedades de difração, projetamos um dispositivo microfluido fácil de usar e versátil chamado ChipX2,3,4.
Neste artigo, demonstraremos como carregar a solução proteica nos canais ChipX usando material de laboratório convencional para preparar cristais por contra-difusão. Este método de cristalização fornece uma triagem eficiente da supersaturação e das condições potenciais de nucleação ao longo dos canais microfluidos contendo a solução enzimática devido ao gradiente de concentração gerado pela difusão do agente cristalizador5,6.
A configuração do chip é simples, usa apenas tubos de laboratório padrão e não requer nenhum equipamento caro. Quando os cristais cresceram em ChipX, ligantes da enzima podem ser introduzidos por difusão. Os dados de difração são então coletados à temperatura ambiente em uma série de cristais contidos nos canais do chip usando uma fonte de raio-X síncrotron. O estudo estrutural aqui descrito levou à determinação de estruturas de uma enzima de maturação de tRNA em sua forma apo e em complexo com um análogo de seu substrato CTP introduzido pela imersão. Esta proteína chamada enzima de adição de CCA polimeriza a cauda trinucleotídea CCA no final de 3' de tRNAs. A comparação das duas imagens 3D obtidas pela cristalografia serial revela as alterações conformais locais relacionadas à ligação do ligante em condições mais fisiológicas do que as utilizadas na crio-cristalografia. O protocolo descrito neste vídeo é geralmente aplicável a qualquer biomolécula, seja uma proteína, um ácido nucleico ou um complexo multicomponiário.
Protocolo
1. Configuração de ensaios de cristalização no ChipX
NOTA: O dispositivo microfluido ChipX pode ser obtido dos autores. Uma descrição do chip é dada na Figura 1. As soluções que contenham o cristalante (ou agente cristalizador) utilizadas para desencadear a cristalização podem ser de origem comercial ou preparadas pelo experimentador.
- Carregando a amostra de biomolécula
NOTA: O volume de amostra realmente necessário para realizar um ensaio individual de contrafusão na seção reta de cada canal do ChipX é de 300 nL. No entanto, por conveniência, sugerimos carregar 5 μL para preencher completamente os oito canais levando em conta o comprimento variável de sua seção curvada e volumes mortos de entrada.- Pipet 5 μL de soluções enzimástas usando tubulação e ponta padrão de 10 μL.
- Introduza a ponta verticalmente na entrada da amostra e injete a solução até que os oito canais sejam preenchidos até a extremidade oposta (entrada do reservatório cristalino).
- Injete 1 μL de óleo de parafina na entrada da amostra, a fim de desconectar os canais uns dos outros.
- Recupere a solução extra no reservatório cristalino na extremidade de cada canal usando uma tubulação padrão de 10 μL.
- Sele a entrada da amostra com um pedaço de fita de 1 cm x 1 cm.
- Carregando as soluções de cristalização
- Pipet 5 μL de solução de cristalização utilizando tubulação e ponta padrão de 10 μL. O volume do reservatório é de 10 μL, mas carregar apenas metade dele evita o transbordamento ao selar com fita adesiva e facilita a adição adicional de ligante para experimentos de imersão. Se as condições iniciais de cristalização forem obtidas por difusão de vapor, aumente a concentração cristalina em um fator de 1,5 a 2. As soluções podem ser diferentes em cada reservatório (no caso apresentado, 1 M de fosfato de hidrogênio diamônio, acetato de sódio de 100 mM, pH 4.5 foi usado em todo o país).
- Oriente a ponta da tubulação em direção à entrada do canal na parte moldada do funil do reservatório para evitar a formação de uma bolha de ar na deposição da solução. Impediria o contato entre as duas soluções e a difusão cristalina no canal.
- Injete a solução cristalina no reservatório.
- Sele os reservatórios com uma fita de fita de 2,5 cm x 1 cm.
- Incubar o chip a 20 °C (a temperatura pode ser ajustada dependendo do alvo, tipicamente entre 4 e 37 °C 4).
2. Rotulagem de proteína com carboxyrhodamina para detecção de fluorescência
NOTA: Esta etapa é opcional. Deve ser realizado antes do carregamento da amostra para facilitar a detecção de cristais no chip usando fluorescência. O método detalhado de rotulagem fluorescente de traços foi descrito por Pusey e colegas de trabalho7. Todas as etapas são realizadas em temperatura ambiente.
- Dissolver 5 mg de pó de éster carboxyrhodamine em 1 mL de dimetil-formamida anidro, dividir a solução em alíquotas de 0,6 μL a serem armazenadas a -20 °C.
- Prepare uma solução de estoque de 1 M Na-borate pH 8.75.
- Diluir o estoque para preparar o buffer de reação em 0,05 M Na-borate pH 8.75.
- Enxágüe uma coluna de desalamento (7 kDa MWCO, 0,5 mL) com 800 μL de tampão de reação.
- Centrifugar a coluna por 1 min a 1400 x g, remova o filtrado.
- Repita esta operação duas vezes (etapas 2.4-2.5) para lavar a coluna.
- Deposite 80 μL de proteína em seu tampão de armazenamento na coluna (a proteína pode ser diluída até 1 mg/mL para aumentar o volume, se necessário).
- Centrifugar a coluna por 1 min a 1400 x g. Esta etapa destina-se a transferir a proteína do seu buffer de armazenamento para o buffer de reação.
- Recupere o fluxo -through (contendo a proteína no tampão de reação) e misture-o com 0,6 μL de solução de carboxyrhodamina.
- Incubar 5 min em temperatura ambiente.
- Enquanto isso, enxágue a coluna 3 x com o tampão de armazenamento, centrifufique a coluna por 1 min a 1400 x g e descarte o filtrado.
- Deposite a solução de reação na coluna.
- Centrifugar a coluna por 1 min a 1400 x g e recuperar o fluxo -through (ou seja, solução de proteína rotulada em seu buffer de armazenamento).
- Suplemente a solução de proteína de estoque com 0,1-1 % (w/w) de proteína rotulada.
- Configure os ensaios de cristalização chipX descritos na seção 1.
- Verifique a presença de cristais proteicos nos ensaios, excitando a sonda fluorescente com uma fonte de luz de comprimento de onda de 520 nm.
3. Observação de cristal
NOTA: O dispositivo ChipX pode ser manuseado sem cuidados especiais, mesmo com cristais dentro, exceto se a temperatura precisar ser controlada.
- Use qualquer estereómico para verificar o resultado dos ensaios de cristalização no ChipX. Sua pegada tem as dimensões padrão dos slides do microscópio e é compatível com qualquer sistema e suporte de slides.
- Verifique o conteúdo dos canais microfluidos a partir do reservatório onde a concentração cristalina é a mais alta para a entrada da amostra onde a concentração cristalina é a mais baixa. O material ChipX é transparente à luz visível, compatível com o uso de polarizadores, bem como com iluminação UV para identificação de cristal proteico por fluorescência tripptophane intrínseca8.
- Registo de cristal usando as etiquetas estampadas ao longo dos canais ou marque locais de cristal com um marcador permanente desenhando pontos de cor ao lado deles na superfície do chip.
4. Imersão de cristal com ligantes
NOTA: Este procedimento é opcional. É usado para introduzir ligantes, substratos enzimados ou átomos pesados nos cristais e deve ser realizado pelo menos 24-48 h antes da análise de raios-X para permitir a difusão composta ao longo dos canais e para os cristais.
- Remova suavemente a fita de vedação dos reservatórios.
- Adicione até 5 μL de solução de ligante em um ou vários reservatórios usando uma solução de micropipet de 10 μL (no exemplo, 3 μL de 10 mM cytidine-5'-[α,β)-metileno] triphosfato (CMPcPP) foi adicionado para alcançar uma concentração final de 3,75 mM). CMPcPP é um análogo não hidrollizável da CTP, um substrato natural da enzima.
- Sele os reservatórios com uma fita de fita de 2,5 cm x 1 cm.
- Incubar o chip sob temperatura controlada por 24-48 h para permitir a difusão de ligantes ao longo dos canais do chip.
5. Análise de cristal por cristalografia serial
NOTA: Esta parte do protocolo precisa ser adaptada dependendo da configuração da linha de luz e das propriedades de difração dos cristais. Apenas indicações gerais são dadas para a análise cristalográfica baseada em experimentos realizados na linha de feixe X06DA (SLS, Villigen, Suíça).
- Montagem chipX no goniômetro de linha de feixe
NOTA: O arquivo para impressão 3D do titular ChipX é fornecido no ref4.- Desligue o crio-jato da linha de vigas. A análise aqui é realizada à temperatura ambiente.
- Monte o ChipX em um suporte dedicado com o canal contendo os cristais a serem analisados posicionados no centro do suporte. O porta-chaves ChipX4 não requer nenhum parafuso ou peça adicional, pois foi projetado para fornecer um ajuste perfeito para ChipX.
- Coloque o suporte no goniômetro.
- Coleta de dados
- Oriente a camada mais grossa (camada superior, Figura 1) do ChipX (nesta orientação as etiquetas ao longo dos canais são diretamente legíveis usando a câmera centralizante da linha de feixe), em direção ao feixe direto e ao rosto mais fino atrás do cristal para minimizar a atenuação do sinal ditracado conforme descrito no ref3.
- Para evitar colisão do ChipX com o material circundante (beamstop, collimator), restringir os movimentos do goniômetro na faixa ±30° (0° correspondente aos canais perpendiculares ao feixe de raios-X).
- Encontre a posição dos cristais com a ajuda dos rótulos gravados ao longo dos canais.
- Selecione uma posição de cristal.
- Centralizar o cristal por grade de dose baixa padrão/rastreamento de raster ou procedimento de 1 clique (o vídeo mostra um exemplo de triagem de grade).
- Coletar dados de difração dentro da faixa -30° / +30°.
- Reinicie o procedimento nas etapas 5.2.4-5.2.6 em outro cristal no mesmo canal após a tradução do chip.
- Realinhar manualmente outro canal ChipX no centro do suporte e realizar a coleta de dados em cristais presentes neste canal.
- Use pacotes e procedimentos cristalográficos padrão para processar e fundir os dados, em seguida, para resolver e refinar a estrutura.
Resultados
O chip microfluido descrito aqui foi projetado para permitir a fácil configuração de ensaios de cristalização e análise de cristal à temperatura ambiente. O procedimento descrito acima e no vídeo foi aplicado no quadro da caracterização estrutural da enzima de adição de CCA da bactéria adaptada a frio Planococcus halocryophilus. Esta enzima pertence a uma família essencial de polimerase que catalisa a adição sequencial da sequência cca de 3' em tRNAs usando CTP e ATP9,10.
O chip foi usado pela primeira vez para preparar cristais da enzima para análise estrutural pelo método de contra-difusão. Para isso, a solução enzimária foi carregada nos oito canais microfluidos (câmaras de cristalização) por uma única injeção na entrada amostral do chip (ver Figura 1). A enzima foi usada a 5,5 mg/mL em seu buffer de armazenamento contendo 20 mM Tris/HCl pH 7.5, 200 mM NaCl e 5 mM MgCl2. Esta etapa foi realizada manualmente com um micropipet padrão de 10 μL. As soluções de cristalização (acetato de sódio de 100 mM pH 4,5,1 M de fosfato de hidrogênio de diamônio) foram então depositadas nos reservatórios nas outras extremidades dos canais.
O procedimento de carregamento é simples e não demora mais do que cinco minutos(Figura 2). O cristalino então difunde para os canais, cria um gradiente de concentração que desencadeia nucleação e crescimento cristalinos. Este gradiente evolui dinamicamente e explora um contínuo dos estados de supersaturação5,6 até atingir um equilíbrio de concentração cristalina entre os canais e o reservatório. Os ensaios de cristalização são tipicamente verificados sob o micoscópio durante um período de 2 a 4 semanas para acompanhar o crescimento de cristais. Cristais bipyramidais de enzima de adição de CCA apareceram em todos os canais após alguns dias de incubação a 20 °C(Figura 3). A rotulagem fluorescente opcional7 da proteína facilita muito a identificação de cristais proteicos e sua discriminação por cristais de sal(Figura 4).
Exploramos o ambiente difuso nos canais de chips para entregar um substrato à enzima que constrói os cristais. No presente caso, o CMPcPP, um analógico CTP, foi adicionado às soluções do reservatório em uma concentração final de 3,75 mM (Figura 5). Essa adição foi realizada dois dias antes da análise cristalográfica para permitir que o CMPcPP alcançasse e ocupasse o local catalítico da enzima, como confirmado posteriormente pela estrutura cristalina (veja abaixo).
Fabricamos um suporte de chip(Figura 6) em ácido polilático usando uma impressora 3D. O suporte permite a montagem do chip em goniômetros usando cabeças magnéticas padrão. Assim, o chip pode ser facilmente posicionado e traduzido no feixe de raios-X para trazer os cristais em posição de difração. A estratégia de coleta de dados precisa ser adaptada dependendo das características do feixe e das propriedades de cristal. No caso da enzima de adição de CCA, os dados foram coletados nas linhas de luz X06DA e X10SA, Swiss Light Source (SLS), com um comprimento de onda de raios-X de 1,0 Å e pilatus 2M-F e detectores de pixels de 6M, respectivamente. Foram coletados 30-60° de rotação em cada cristal à temperatura ambiente com imagens de exposição de 0,1° ou 0,2° e 0,1 s (ver Tabela 1). Os conjuntos de dados parciais foram processados individualmente e cortados quando a resolução dos padrões de difração começou a decair devido a danos causados pela radiação (detectado pela diminuição da relação sinal-ruído 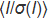 e CC1/2, e um aumento demeas Rna concha de alta resolução). Conjuntos de dados completos foram reconstituídos pela fusão de dados de 5 cristais(Tabela 1). As estruturas cristalinas foram derivadas pela substituição molecular utilizando embalagens e procedimentos cristalográficos padrão para processamento de dados11 e refinamento12. A comparação das estruturas da enzima e do seu complexo com o CMPcPP revela a adaptação conformacional local que acompanha a ligação do substrato no local ativo da enzima de adição de CCA(Figura 7).
e CC1/2, e um aumento demeas Rna concha de alta resolução). Conjuntos de dados completos foram reconstituídos pela fusão de dados de 5 cristais(Tabela 1). As estruturas cristalinas foram derivadas pela substituição molecular utilizando embalagens e procedimentos cristalográficos padrão para processamento de dados11 e refinamento12. A comparação das estruturas da enzima e do seu complexo com o CMPcPP revela a adaptação conformacional local que acompanha a ligação do substrato no local ativo da enzima de adição de CCA(Figura 7).
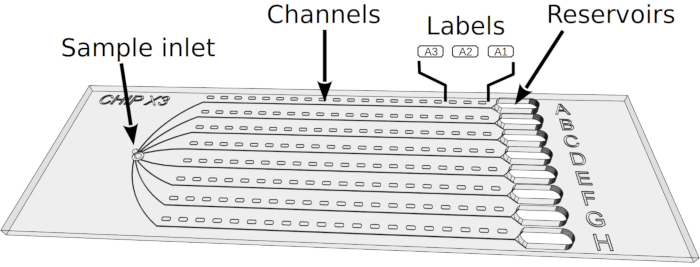
Figura 1: Design ChipX. O chip consiste em uma camada superior feita de COC (espessura: 1 mm) na qual oito canais microfluidos e reservatórios são impressos. O chip inteiro é selado com uma camada de COC (espessura: 0,1 mm). Todos os canais estão conectados a uma única entrada no lado esquerdo para injeção simultânea de amostras e a reservatórios individuais no lado direito em que as soluções de cristalização são depositadas. Os canais, que constituem as câmaras de cristalização reais do chip, têm 4 cm de comprimento e possuem uma seção transversal de 80 μm x 80 μm. Rótulos (A1, A2, A3, etc.) gravados ao longo dos canais facilitam o posicionamento cristalino sob o microscópio e a elaboração de uma lista de amostras para coleta de dados. ChipX tem o tamanho de um slide de microscópio padrão (7,5 cm x 2,5 cm). Clique aqui para ver uma versão maior desta figura.
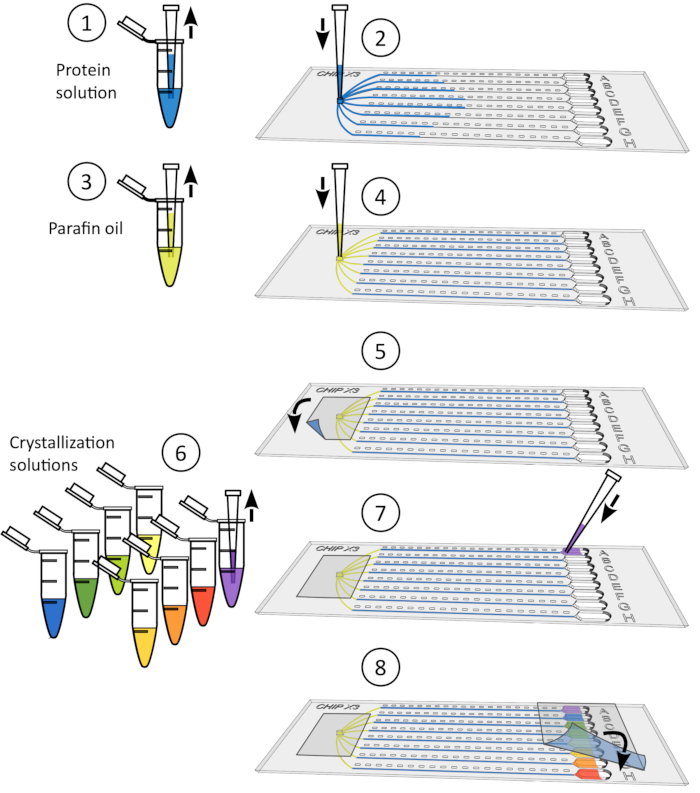
Figura 2: Configuração de ensaios de cristalização em ChipX. 1) Depositar 5-6 μL de soluções enzimáticos usando tubulação e ponta padrão de 10 μL. 2) Introduza a ponta verticalmente na entrada da amostra e injete a solução nos oito canais. 3) Pipet 1 μL de óleo de parafina. 4) Introduza a ponta verticalmente na entrada da amostra e injete o óleo para desconectar os canais uns dos outros. 5) Sele a entrada com um pedaço de fita. 6) Pipet 5 μL de solução de cristalização utilizando tubulação e ponta padrão de 10 μL. As soluções podem ser diferentes em cada reservatório (por exemplo, a partir de um kit de triagem). 7) Oriente a ponta da tubulação para a entrada do canal na parte moldada do funil do reservatório (para evitar a formação de uma bolha de ar na deposição da solução) e injete a solução cristalina no reservatório. 8) Selar os reservatórios com um pedaço de fita e incubar o chip a temperatura controlada. Clique aqui para ver uma versão maior desta figura.

Figura 3: Cristais de enzima de adição de CCA cultivadas por contra-difusão nos canais microfluídicos do ChipX. A barra de escala é de 0,1 mm. Clique aqui para ver uma versão maior desta figura.
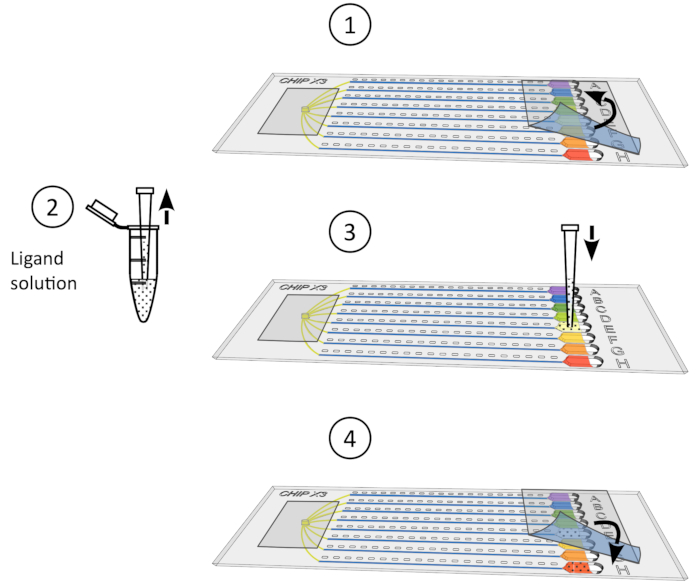
Figura 4: Procedimento de imersão de cristal. 1) Remova suavemente a fita dos reservatórios. 2) Deposite até 5 μL de solução de ligante utilizando um micropipet de 10 μL. 3) Adicione o ligante a um ou vários reservatórios. 4) Selar novamente os reservatórios com um pedaço de fita e incubar o chip sob temperatura controlada por 24-48 h antes da coleta de dados. Clique aqui para ver uma versão maior desta figura.

Figura 5: A rotulagem fluorescente de traço discrimina a proteína (esquerda) dos cristais de sal (direita). A solução enzimática de adição de CCA continha 0,4 % (w/w) de proteína rotulada com carboxyrhodamina. À direita, os cristais são iluminados com uma fonte de luz de comprimento de onda de 520 nm e a imagem é tirada com um filtro de passagem baixa a 550 nm (LP550); (inset) estrutura de carboxyrhodamine-succinimidyl éster. Clique aqui para ver uma versão maior desta figura.

Figura 6: (Esquerda) Desenho do suporte ChipX e (à direita) ChipX montado no goniômetro da linha de feixe X06DA no SLS (Villigen, Suíça) para análise de cristal serial. Clique aqui para ver uma versão maior desta figura.
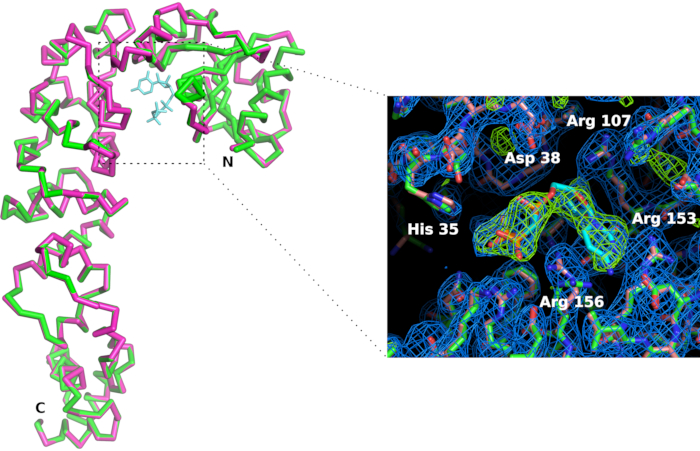
Figura 7: Comparação do sítio ativo da enzima de adição de CCA na forma apo (em rosa) e no complexo com um analógico CTP (em verde). Embora a conformação geral da enzima não seja afetada, a vinculação do ligante CMPcPP é acompanhada por uma leve reorganização das cadeias laterais no local ativo. O mapa de densidade de elétrons 2Fo-Fc (em azul) é contornado a 1,2 sigma. O mapa de densidade de elétrons de diferença contornado em 4 sigma (em verde) confirma a presença do ligante no local ativo. Clique aqui para ver uma versão maior desta figura.
| Amostra cristalizada | Enzima de adição de CCA | Enzima de adição de CCA + CMPcPP |
| Análise de cristal | ||
| Linha de raio-X | SLS – X06DA | SLS – X10SA |
| Comprimento de onda (Å) | 1.000 | 1.000 |
| Temperatura (K) | 293 | 293 |
| Detector | Pilatus 2M-F | Pilatus 6M |
| Distância do detector de cristal (mm) | 300 | 400 |
| Cristais coletados | 6 | 14 |
| Cristais selecionados | 5 | 5 |
| Faixa de rotação por imagem (°) | 0.1 | 0.2 |
| Tempo de exposição por imagem (s) | 0.1 | 0.1 |
| Não. de imagens selecionadas | 1000 | 540 |
| Faixa de rotação total (°) | 100 | 108 |
| Grupo espacial | P43212 | P43212 |
| a, c (Å) | 71.5, 293.8 | 71.4, 293.6 |
| Mosaico médio (°) | 0.04 | 0.04 |
| Faixa de resolução (Å) | 46 – 2.54 (2.6 – 2.54) | 48 – 2.3 (2.4 – 2.3) |
| Total Nº. de reflexões | 176105 (9374) | 232642 (32937) |
| Não. de reflexões únicas | 23922 (1598) | 34862 (4066) |
| Completude (%) | 90.6 (84.6) | 99.5 (100.0) |
| Redundância | 7.5 (6.0) | 6.7 (8.1) |
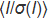 | 8.1 (1.3) | 6.9 (0.7) |
| Rmeas (%) § | 18.6 (126.0) | 18.0 (231.2) |
| CC1/2 (%) £ | 98.7 (55.0) | 98.7 (46.9) |
| Fator B geral da trama de Wilson (Å2) | 57.4 | 60.6 |
| Refinamento cristalográfico | ||
| Não. de reflexões, conjunto de trabalho / conjunto de testes | 23583 / 1180 | 34840 / 3405 |
| Rcryst final (%) / Rgrátis (%) | 18.8 / 21.4 | 20.0 / 22.9 |
| Não. de átomos não-H: geral / proteína / ligante / solvente | 2998 / 2989 / 0 / 9 | 3057 / 2989 / 29 / 10 |
| R.m.s. desvios para ligações (Å) / ângulos (°) | 0.009 / 1.23 | 0.010 / 1.22 |
| Fatores B médios (Å2): geral / proteína / ligante / solvente | 60.1 / 60.1 / 0 / 52.7 | 62.5 / 62.6 / 60.1 / 55.5 |
| Trama de Ramachandran: mais favorecida (%) / permitida (%) | 98.1 / 1.9 | 97.2 / 2.8 |
| ID PDB | 6IBP | 6T52 |
Tabela 1: Estatísticas de coleta e refinamento de dados
§ Rmeas independentes de redundância = Σhkl(N/N-1)1/2Σi | Ii(hkl)- (hkl)>| / ΣhklΣi i i(hkl),onde N é a multiplicidade de dados 17.
£ Os dados com baixo em shell externo (<2.0) foram incluídos com base no critério CC1/2 (correlação entre duas metades aleatórias do conjunto de dados > 50%) como proposto por Karplus & Diederichs 18.
Discussão
Os protocolos atuais na biocristalografia envolvem a preparação de cristais usando métodos como difusão de vapor ou lote13,14, e sua transferência para um microloop para crio-resfriamento15,16 antes de realizar a análise de difração em um jato de nitrogênio em condições criogênicas. Em contraste, o crio-resfriamento direto de cristal não é possível no ChipX3 e os cristais não podem ser extraídos de seu canal microfluido, o que pode ser visto como limitações dessa configuração. No entanto, o protocolo descrito no artigo fornece um pipeline totalmente integrado para a determinação de estruturas cristalinas à temperatura ambiente (ou seja, em condições mais fisiológicas). Embora a coleta de dados à temperatura ambiente cause um aumento de danos à radiação19,esse efeito é contrabalançado pelo rápido tempo de aquisição de dados (um máximo de rotação de 60° é coletado em cada cristal) e pela fusão de vários conjuntos de dados parciais. Tanto o design chipX quanto o material foram otimizados para reduzir a dispersão de fundo e a atenuação do sinal de difração3, e a coleta de dados pode ser realizada em cristais com dimensões equivalentes à metade do tamanho dos canais (40 μm)4.
Resumindo, as principais vantagens do protocolo são as seguintes. Os cristais são produzidos em um ambiente livre de convecção (canais microfluidos), o que é muito favorável ao crescimento de cristais de alta qualidade. O método de contrafusão implementado no ChipX é muito eficiente na triagem do cenário de supersaturação; a difusão de cristallantes no canal do chip cria uma onda de concentração e supersaturação que ajuda a determinar as condições adequadas de nucleação e crescimento5. Os cristais nunca são tratados diretamente, mas são analisados in situ, dentro do chip, que preserva suas propriedades genuínas de difração (ou seja, não altera a mosaico cristalina por interação física ou criocooling)20. A análise de difração é realizada em uma série de cristais distribuídos ao longo dos canais de chip com baixa exposição de dose para minimizar danos causados pela radiação, e um conjunto de dados completo é montado pela fusão de dados parciais da série. A pegada padrão e o design simples do ChipX permitirão no futuro uma automação completa da coleta de dados in situ usando instalações síncrotrons ou XFEL. Todas as etapas do protocolo são realizadas no ChipX. Do ponto de vista do experimentador, a configuração do chip é simples e fácil de executar com tubos padrão e não requer nenhum equipamento extra. A conexão do canal semelhante a uma árvore na entrada da amostra minimiza os volumes mortos no sistema, o que é importante quando se trabalha com amostras difíceis de purificar ou que só estão disponíveis em quantidade limitada.
Em conclusão, a abordagem lab-on-a-chip implementada no ChipX simplifica e miniaturiza eficientemente o processo de cristalização por contra-difusão e determinação da estrutura cristalina, permitindo passar da amostra para sua estrutura 3D em um único dispositivo. É amplamente aplicável e oferece uma solução fácil de usar e econômica para investigações de biocristalografia serial de rotina à temperatura ambiente.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores reconhecem a Fonte de Luz Suíça (Villigen, Suíça) pela alocação de feixes para o projeto nas linhas de luz X10SA (PXII) e X06DA (PXIII), Alexandra Bluhm por sua contribuição para o refinamento de estrutura, Clarissa Worsdale para a gravação da narração e François Schnell (Université de Strasbourg) por sua ajuda na edição de vídeo e SFX. Este trabalho foi apoiado pelo Centro Nacional de la Recherche Scientifique (CNRS), a Universidade de Estrasburgo, o consórcio LabEx "NetRNA" (ANR-10-LABX-0036_NETRNA), um financiamento de doutorado para R.dW da iniciativa excelência (IdEx) da Universidade de Estrasburgo no quadro do Programa Nacional Francês "Investissements d'Avenir", um financiamento de doutorado para k.R. da Universidade Franco-Alemã (UFA-DFH, não conceder. CT-30-19), o Deutsche Forschungsgemeinschaft (concessão nº. Mo 634/10-1). Os autores se beneficiaram do programa de cooperação PROCOPE Hubert Curien (Ministério francês das Relações Exteriores e Deutscher Akademischer Austauschdienst).
Materiais
| Name | Company | Catalog Number | Comments |
| Axioscope A1 stereomicroscope | Zeiss | Crystal observation (step 3) | |
| Carboxyrhodamine succinimidyl ester | Invitrogen | C-6157 | Protein labeling (step 2) |
| CMPcPP | Jena Bioscience | NU-438 | Crystal soaking (step 4) |
| Crystal clear sealing tape | Hampton research | HR3-511 | ChipX sealing (step 1) |
| Parafin oil | Hampton research | HR3-411 | ChipX loading (step 1) |
| Ultimaker 2 extended+ | Ultimaker | 3D printer - Representative results | |
| UV light source | Xtal Concepts Gmbh | XtalLight100c | Crystal observation (step 3) |
| Zeba spin desalting column 7K MWCO | ThermoFisher Scientific | 89882 | Protein labeling (step 2) |
Referências
- Giegé, R., Sauter, C. Biocrystallography: past, present, future. HFSP Journal. 4 (3-4), 109-121 (2010).
- Dhouib, K., et al. Microfluidic chips for the crystallization of biomacromolecules by counter-diffusion and on-chip crystal X-ray analysis. Lab on a Chip. 9 (10), 1412-1421 (2009).
- Pinker, F., et al. ChipX: A Novel Microfluidic Chip for Counter-Diffusion Crystallization of Biomolecules and in Situ Crystal Analysis at Room Temperature. Crystal Growth & Design. 13 (8), 3333-3340 (2013).
- de Wijn, R., et al. A simple and versatile microfluidic device for efficient biomacromolecule crystallization and structural analysis by serial crystallography. IUCrJ. 6 (3), 454-464 (2019).
- García-Ruiz, J. M. A supersaturation wave of protein crystallization. Journal of Crystal Growth. 232 (1-4), 149-155 (2001).
- Otálora, F., Gavira, J. A., Ng, J. D., García-Ruiz, J. M. Counterdiffusion methods applied to protein crystallization. Progress in Biophysics and Molecular Biology. 101 (1-3), 26-37 (2009).
- Pusey, M., Barcena, J., Morris, M., Singhal, A., Yuan, Q., Ng, J. Trace fluorescent labeling for protein crystallization. Acta Crystallographica Section F Structural Biology Communications. 71 (7), 806-814 (2015).
- Meyer, A., Betzel, C., Pusey, M. Latest methods of fluorescence-based protein crystal identification. Acta Crystallographica Section F Structural Biology Communications. 71 (2), 121-131 (2015).
- Betat, H., Rammelt, C., Mörl, M. tRNA nucleotidyltransferases: ancient catalysts with an unusual mechanism of polymerization. Cellular and Molecular Life Sciences. 67 (9), 1447-1463 (2010).
- Ernst, F. G. M., Erber, L., Sammler, J., Jühling, F., Betat, H., Mörl, M. Cold adaptation of tRNA nucleotidyltransferases: A tradeoff in activity, stability and fidelity. RNA Biology. 15 (1), 144-155 (2018).
- Kabsch, W. XDS. Acta Crystallographica. Section D, Biological Crystallography. 66 (2), 125-132 (2010).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallographica. Section D, Biological Crystallography. 66 (2), 213-221 (2010).
- Dessau, M. A., Modis, Y. Protein Crystallization for X-ray Crystallography. Journal of Visualized Experiments. (47), e2285 (2011).
- Sauter, C., Lorber, B., McPherson, A., Giegé, R., Arnold, E., Himmel, D. M., Rossmann, M. G. Crystallization - General Methods. International Tables of Crystallography, Vol. F, Crystallography of Biological Macromolecules. , 99-120 (2012).
- Garman, E. "Cool" crystals: macromolecular cryocrystallography and radiation damage. Current Opinion in Structural Biology. 13 (5), 545-551 (2003).
- Li, D., Boland, C., Aragao, D., Walsh, K., Caffrey, M. Harvesting and Cryo-cooling Crystals of Membrane Proteins Grown in Lipidic Mesophases for Structure Determination by Macromolecular Crystallography. Journal of Visualized Experiments. (67), e4001 (2012).
- Diederichs, K., Karplus, P. A. Improved R-factors for diffraction data analysis in macromolecular crystallography. Nature Structural Biology. 4 (4), 269-275 (1997).
- Karplus, P. A., Diederichs, K. Linking Crystallographic Model and Data Quality. Science. 336 (6084), 1030-1033 (2012).
- de la Mora, E., Coquelle, N., Bury, C. S., Rosenthal, M., Holton, J. M., Carmichael, I., Garman, E. F., Burghammer, M., Colletier, J. -. P., Weik, M. Radiation damage and dose limits in serial synchrotron crystallography at cryo- and room temperatures. Proceedings of the National Academy of Sciences of the United States of America. 117 (8), 4142-4151 (2020).
- Nave, C. A. Description of Imperfections in Protein Crystals. Acta Crystallographica. Section D, Biological Crystallography. 54 (5), 848-853 (1998).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados