Method Article
Explorando epitransomos m6A e m5C sobre infecção viral: um exemplo com HIV
Neste Artigo
Resumo
O papel das modificações do RNA em infecções virais está apenas começando a ser explorado e pode destacar novos mecanismos de interação viral-host. Neste trabalho, fornecemos um pipeline para investigar modificações de RNA m6A e m5C no contexto de infecções virais.
Resumo
O papel das modificações do RNA nos processos biológicos tem sido o foco de um número crescente de estudos nos últimos anos e é conhecido hoje em dia como epitranscriptomia. Entre outros, as modificações de RNA N6-metiladenosine (m6A) e 5-metilcytosina (m5C) foram descritas em moléculas de mRNA e podem ter um papel na modulação dos processos celulares. A epitranscriptomia é, portanto, uma nova camada de regulação que deve ser considerada além das análises transcriômicas, pois também pode ser alterada ou modulada pela exposição a qualquer agente químico ou biológico, incluindo infecções virais.
Aqui, apresentamos um fluxo de trabalho que permite a análise da paisagem epitransomica celular e viral das marcas m6A e m5C simultaneamente, em células infectadas ou não com o vírus da imunodeficiência humana (HIV). Após o isolamento e fragmentação do mRNA a partir de células infectadas pelo HIV e não infectadas, utilizamos dois procedimentos diferentes: MeRIP-Seq, uma técnica baseada em imunoprecipitação de RNA, para enriquecer para fragmentos de RNA contendo a marca m6A e BS-Seq, uma técnica baseada em conversão de bisullfita, para identificar a marca m5C em uma única resolução nucleotídea. Após a captura específica da metilação, as bibliotecas RNA são preparadas para sequenciamento de alto rendimento. Também desenvolvemos um pipeline de bioinformática dedicado para identificar transcrições diferencialmente metiladas (DM) independentemente de seu perfil de expressão basal.
No geral, a metodologia permite a exploração de múltiplas marcas epitranscriptômicas simultaneamente e fornece um atlas de transcrições de DM sobre infecção viral ou qualquer outra perturbação celular. Essa abordagem oferece novas oportunidades para identificar novos atores e novos mecanismos de resposta celular, como fatores celulares que promovem ou restringem a replicação viral.
Introdução
Sabe-se há muito tempo que as moléculas de RNA podem ser modificadas, e mais de 150 modificações pós-transcrições foram descritas até o momento1. Eles consistem na adição de grupos químicos, principalmente grupos de metila, a praticamente qualquer posição dos anéis pirimidina e purina das moléculas de RNA2. Tais modificações pós-transcrição já foram demonstradas como altamente enriquecidas na transferência de RNA (tRNA) e RNA ribossômico (rRNA) e foram recentemente descritas em moléculas de mRNA também.
O surgimento de novas tecnologias, como o Sequenciamento de Próxima Geração (NGS), e a produção de anticorpos específicos que reconhecem modificações químicas definitivas permitiram, pela primeira vez, a investigação do local e a frequência de modificações químicas específicas em nível de transcriptome-em todo o nível. Esses avanços levaram a uma melhor compreensão das modificações do RNA e ao mapeamento de várias modificações nas moléculas de mRNA3,4.
Enquanto a epigenética investiga o papel das modificações de DNA e histona na regulação do transcriptome, a epitranscriptomia de forma semelhante se concentra nas modificações do RNA e seu papel. A investigação de modificações epitransomicas oferece novas oportunidades para destacar novos mecanismos de regulação que podem sintonizar uma variedade de processos celulares (ou seja, emendas de RNA, exportação, estabilidade e tradução)5. Não foi, portanto, grande surpresa que estudos recentes descobriram muitas modificações epitranscriômicas sobre infecção viral tanto no celular quanto no viral RNAs6. Os vírus investigados até agora incluem tanto os vírus DNA quanto o RNA; entre eles, o HIV pode ser considerado como um exemplo pioneiro. Ao todo, a descoberta da metilação de RNA no contexto de infecções virais pode permitir a investigação de mecanismos ainda não atribuídos de expressão ou replicação viral, fornecendo assim novas ferramentas e metas para controlá-los7.
No campo da epitranscriptomia do HIV, modificações de transcrições virais têm sido amplamente investigadas e mostraram que a presença dessa modificação foi benéfica para a replicação viral8,9,10,11,12,13. Até o momento, várias técnicas podem ser usadas para detectar marcas epitranscriômicas no nível de transcriptome em todo o mundo. As técnicas mais utilizadas para identificação m6A dependem de técnicas de precipitação imunológica, como MeRIP-Seq e miCLIP. Enquanto o MeRIP-Seq depende da fragmentação do RNA para capturar fragmentos contendo resíduos metilados, o miCLIP baseia-se na geração de mutações específicas de assinatura de anticorpos α-m6A no crosslinking UV RNA-anticorpos, permitindo assim um mapeamento mais preciso.
A detecção da modificação m5C pode ser alcançada por tecnologias baseadas em anticorpos semelhantes à detecção de m6A (rip m5C), ou por conversão de bisullfita ou por AZA-IP ou por miCLIP. Tanto o Aza-IP quanto o m5C miCLIP usam um metiltransferase específico como isca para atingir o RNA enquanto passa pela metilação de RNA. Em Aza-IP, as células-alvo são expostas a 5-azacytidina, resultando na introdução aleatória de sítios analógicos de 5 azacytidina de citidina em RNA nascente. No miCLIP, o NSun2 metiltransferase é geneticamente modificado para abrigar a mutação C271A14,15.
Neste trabalho, focamos na caracterização dupla das modificações m6A e m5C em células infectadas, utilizando o HIV como modelo. Após a otimização metodológica, desenvolvemos um fluxo de trabalho que combina imunoprecipitação de RNA metilado (MeRIP) e conversão de bisullfita de RNA (BS), permitindo a exploração simultânea de marcas epitransomicas m6A e m5C em nível transcriptome-wide, tanto em contextos celulares quanto virais. Este fluxo de trabalho pode ser implementado em extratos de RNA celular, bem como em RNA isolado de partículas virais.
A abordagem metilada RNA ImmunoPrecipitation (MeRIP)16 permitindo a investigação do m6A no nível de transcriptome-em todo o nível está bem estabelecida e uma matriz de anticorpos específicos m6A estão disponíveis comercialmente até o momento17. Este método consiste na captura seletiva de peças de RNA contendo m6A usando um anticorpo específico m6A. As duas principais desvantagens dessa técnica são (i) a resolução limitada, que é altamente dependente do tamanho dos fragmentos de RNA e, portanto, fornece uma localização e região aproximadas contendo o resíduo metilado, e (ii) a grande quantidade de material necessária para realizar a análise. No protocolo otimizado a seguir, padronizamos o tamanho do fragmento para cerca de 150 nt e reduzimos a quantidade de material inicial de 10 μg de RNA poli-A selecionado, que atualmente é a quantidade aconselhada de material inicial, para apenas 1 μg de RNA poli-A selecionado. Também maximizamos a eficiência de recuperação de fragmentos de RNA m6A ligados a anticorpos específicos usando uma eluição por uma abordagem de competição com um peptídeo m6A em vez de métodos de elução mais convencionais e menos específicos usando técnicas à base de fenol ou proteinase K. A principal limitação deste ensaio baseado em RIP, no entanto, continua sendo a resolução subótima que não permite a identificação do nucleotídeo A modificado preciso.
A análise da marca m5C pode ser realizada atualmente usando duas abordagens diferentes: um método baseado em RIP com anticorpos específicos m5C e conversão de bisullfita de RNA. Como o RIP oferece apenas resolução limitada sobre a identificação do resíduo metilado, utilizamos conversão de bisulfito que pode oferecer resolução de nucleotídeo único. A exposição do RNA ao bisulfita (BS) leva à deaminação da citosina, convertendo assim o resíduo de citosina em uracil. Assim, durante a reação de conversão de bisulfita de RNA, toda citosina não metilada é desaminada e convertida em uracil, enquanto a presença de um grupo de metila na posição 5 da citosina tem um efeito protetor, impedindo a desaminação induzida pela BS e preservando o resíduo de citosina. A abordagem baseada em BS permite a detecção de um nucleotídeo modificado por m5C em resolução de base única e para avaliação da frequência de metilação de cada transcrição, fornecendo insights sobre a dinâmica de modificação m5C18. A principal limitação dessa técnica, no entanto, baseia-se na taxa falsa positiva de resíduos metilados. De fato, a conversão de BS é eficaz em RNA de fio único com resíduos C acessíveis. No entanto, a presença de uma estrutura secundária de RNA apertada poderia mascarar a posição N5C e dificultar a conversão de BS, resultando em resíduos C não metilados que não são convertidos em resíduos de U e, portanto, falsos positivos. Para contornar essa questão e minimizar a taxa falsa positiva, aplicamos 3 rodadas de ciclos de conversão de desnaturação e bisulfato19. Também introduzimos 2 controles nas amostras para permitir a estimativa da eficiência de conversão de bisulfato: nós, controles de sequenciamento de ERCC (sequências padronizadas e comercialmente disponíveis não metiladas)20, bem como RNAs poli-A-esgotadas para avaliar a taxa de conversão de bisulfatos por um lado, e verificar por RT-PCR a presença de um local metilado conhecido e bem conservado, C4447, em 28S ribossômico RNA, por outro lado,21.
No campo da virologia, o acoplamento desses dois métodos de investigação epitransomica com sequenciamento de próxima geração e análise bioinformática precisa permite o estudo aprofundado da dinâmica m6A e m5C (ou seja, alterações temporais de modificação de RNA que poderiam ocorrer após a infecção viral e poderiam descobrir uma série de novos alvos terapeuticamente relevantes para uso clínico).
Protocolo
1. Preparação celular
NOTA: Dependendo do tipo de célula e do seu conteúdo de RNA, o número inicial de células pode variar.
- Tenha células suficientes para obter entre 200-500 μg de RNA total ou 5-7 μg de RNA poli-A selecionado. Por exemplo, as células 50 x 106 SupT1 devem produzir cerca de 500 μg de RNA total após a extração com reagentes à base de fenol, e, portanto, é necessário para cada condição individual testada.
- Prepare o número necessário de células de acordo com o desenho experimental e, portanto, de acordo com o número de condições testadas (infecção, pontos de tempo, tratamento). Se o experimento visa obter células não infectadas e células infectadas pelo HIV a 24 horas após a infecção, é necessário um total de 100 x 106 células, metade para condição não infectada e metade para condição infectada.
2. Extração de RNA
- De Células: Extração de RNA com Fenol-Cloroform
- Para cada condição, colete células (por exemplo, 50 x 106) por centrifugação e descarte o sobrenante.
- Adicione 5 mL de reagente à base de fenol a cada pelota de 50 x 106 células e misture por pipetar para cima e para baixo várias vezes.
- Incubar por 5 minutos à temperatura ambiente para permitir a lise completa. As células lysed podem ser armazenadas a -80 °C ou processadas diretamente.
NOTA: Se necessário, as células também podem ser divididas em alíquotas de 10 x 106 células por tubo em tubos de 1,5 mL e diluídas em 1 mL de reagente à base de fenol para armazenamento mais conveniente. - Adicione 1 mL de clorofórmio e misture por inversão.
- Incubar por 3 minutos à temperatura ambiente.
- Centrifugar por 15 min a 2.000 x g e 4 °C.
- Pipeta para fora da fase aquosa (fase superior) e transferir para um novo tubo. Finalize a transferência da fase aquosa angling do tubo a 45° e tubos cuidadosamente a solução para fora.
NOTA: A quantidade de fase aquosa pode variar entre as amostras, mas deve estar próxima da quantidade de clorofórmio adicionada à amostra (ou seja, 1 mL). Não transfira nenhuma camada interfase ou orgânica! O uso de tubos de bloqueio de fase ou de phase-maker pode facilitar esse processo. - Adicione 0,5 mL de isopropanol de grau molecular 100% à fase aquosa.
- Incubar por 1h a -80 °C para permitir a precipitação do RNA.
- Centrifugar por 10 min a 12.000 x g e 4 °C para pelotar o RNA precipitado.
- Descarte o supernasal e resuspenque a pelota de RNA em 1 mL de 75% de etanol de grau de biologia molecular. Vórtice brevemente.
- Centrifugar por 5 min a 7.500 x g e 4 °C e descartar o supernaspe.
- Seque a pelota por 15 minutos.
- Resuspenque a pelota em 20 μL de água sem RNase e transfira para um novo tubo.
- Lave o tubo vazio com mais 20 μL de água para maximizar a recuperação do RNA e acumule com o primeiro volume de 20 μL.
- Quantifique o RNA total com um espectotômetro e avalie a qualidade do RNA com um analisador de fragmentos.
- De Partículas Virais: Extração de RNA com kit de extração de RNA viral baseado em coluna
NOTA: A extração de RNA de partículas virais com reagente à base de fenol resulta em RNA viral de baixa qualidade e em bibliotecas de baixa qualidade. Assim, uma extração de RNA baseada em coluna deve ser favorecida. Os kits de extração de RNA utilizando RNA portador para elução e recuperação de RNA não são apropriados para este procedimento e devem ser evitados. Uma vez que o RNA do HIV é poli-adenilado, a extração direta de RNA sem mais isolamento mRNA é suficiente para entrar nos gasodutos MeRIP-Seq e BS-Seq. Normalmente, 1-2 mL de supernanato viral de células universalmente infectadas deve fornecer RNA suficiente para realizar todo o fluxo de trabalho.- Prepare o buffer adicionando 150 μL de beta-mercaptoetanol a 30 mL de tampão de lise. Reconstitua o Tampão de Lavagem Viral adicionando 96 mL de 100% de etanol.
- Coletar supernacantas contendo vírus e centrífugas em detritos celulares de pelotas para minimizar a contaminação celular do RNA.
- Transfira 1 mL de supernanato viral para um tubo de 15 mL.
- Adicione 3 mL de Tampão de RNA Viral a 1 mL de amostra viral e misture por vórtice.
- Transfira 700 μL de amostra em uma coluna, inserida em um tubo de coleta.
- Centrifugar por 2 min a 13.000 x g em temperatura ambiente.
- Descarte o fluxo através do fluxo.
- Repita as 3 etapas anteriores até que toda a amostra tenha sido processada e, portanto, todo o RNA foi capturado na coluna matriz baseada em sílica.
- Adicione 500 μL de tampão de lavagem viral à coluna.
- Centrifugar por 1 min a 10.000 x g em temperatura ambiente. Descarte o fluxo através do fluxo.
- Adicione 200 μL de Tampão de Lavagem Viral à coluna.
- Centrifugar por 1 min a 10.000 x g em temperatura ambiente. Descarte o fluxo através do fluxo.
- Coloque a coluna em um tubo de coleta vazio.
- Centrifugar por 1 min a 10.000 x g à temperatura ambiente para descartar ainda mais qualquer contaminante tampão de lavagem restante.
- Transfira cuidadosamente a coluna para um tubo de 1,5 mL.
- Adicione 20 μL de água livre de DNase/RNase diretamente ao centro da matriz da coluna e centrífuga a 10.000 x g para 30 s à temperatura ambiente.
- Adicione um adicional de 10 μL de água livre de DNase/RNase diretamente ao centro da matriz da coluna e centrífuga novamente para 30 s.
- Quantifique o RNA total com um espectotômetro e avalie a qualidade do RNA com um analisador de fragmentos.
NOTA: A extração de RNA pode ser realizada com qualquer método, se a qualidade do RNA recuperado for alta, com um número de integridade/qualidade de RNA > 9. O RNA total pode ser armazenado a -80 °C até um processamento adicional.
3. mRNA Isolamento por seleção poli-A com Oligo(dT)25
NOTA: Devido à presença de RNA ribossômico altamente metilado em extratos celulares, é altamente recomendável isolar o RNA poli-A, seja por esgotamento de rRNA ou preferencialmente por seleção positiva poli-A. Esta etapa é opcional e deve ser realizada apenas para amostras de RNA celular, para obter resultados de sequenciamento em maior resolução. Se analisar a metilação de RNAs virais não poli-adenylated, favoreça o esgotamento do rRNA em vez da seleção poli-A ou eventualmente realize a análise no RNA total.
-
Preparação de contas para captura poli-A
- Resuspend o Oligo(dT)25 contas magnéticas estoque de frasco de vórtice para >30 s.
- Transfira 200 μL de contas magnéticas para um tubo de 1,5 mL. Prepare o número de tubos com contas magnéticas de acordo com a quantidade total de amostras de RNA a serem processadas.
NOTA: Um tubo com 200 μL de solução de estoque Dynabead corresponde a 1 mg de contas e pode acomodar uma amostra de 75 μg de RNA total. - Coloque os tubos em um ímã por 1 min e descarte o supernatante. Remova os tubos do ímã.
- Adicione 1 mL de Buffer de Ligação (20 mM Tris-HCl, pH 7.5, 1.0 M LiCl, 2 mM EDTA) e resuspend por vórtice. Coloque os tubos no ímã por 1 min e descarte o sobrenatante. Remova os tubos do ímã. Repetir.
- Resuspenda as contas magnéticas lavadas em 100 μL de Buffer de Ligação.
-
Preparação total do RNA
- Diluir o RNA total em uma concentração final de 0,75 μg/μL com água sem RNase, que corresponde a 75 μg/100 μL.
NOTA: Se o RNA estiver em uma concentração menor, proceda como descrito abaixo sem modificar os volumes. - Aliquot o RNA total em vários tubos, dispensando 100 μL de amostra de RNA por tubo.
- Adicione 100 μL de buffer de ligação a cada amostra de RNA.
- Aqueça o RNA total a 65 °C por 2 min para interromper estruturas secundárias.
- Coloque imediatamente no gelo até que esteja pronto para dar o próximo passo.
NOTA: O tempo de incubação pode variar de acordo com o número de amostras a serem processadas, mas não deve exceder 1h para evitar qualquer degradação do RNA.
- Diluir o RNA total em uma concentração final de 0,75 μg/μL com água sem RNase, que corresponde a 75 μg/100 μL.
-
Seleção Poli-A
- A cada tubo de RNA (a partir da etapa 3.2), adicione 100 μL de contas magnéticas lavadas (a partir do passo 3.1).
- Misture bem por tubulação para cima e para baixo e deixe a ligação em uma roda giratória à temperatura ambiente por 15 minutos.
- Abra todos os tubos, coloque-os no ímã por 1 min, e remova cuidadosamente todo o supernatante.
- Recupere o supernante em um novo tubo e mantenha-se de lado para uma segunda rodada de captura de RNA (passo 3.3.14), a fim de melhorar a recuperação final poli-A.
- Remova o tubo do ímã e adicione 200 μL de Tampão de Lavagem (10 mM Tris-HCl, pH 7.5, 0,15 M LiCl, 1 mM EDTA). Misture por pipetar cuidadosamente 4 a 5 vezes.
- Coloque o tubo no ímã por 1 min e descarte o supernasce.
- Repita a etapa de lavagem uma vez (repetia as etapas 3.3.5 e 3.3.6).
- Adicione 20 μL de gelo frio de 10 mM Tris-HCl ao elute poly-A RNA das contas.
- Incubar a 80 °C por 2 min.
- Coloque o tubo sobre o ímã e transfira rapidamente o supernasce contendo o RNA poli-A para um novo tubo sem RNase. Coloque o tubo no gelo.
- Repita a etapa de elução (passos 3.3.8 a 3.3.10) para aumentar o rendimento.
- Lave as mesmas contas uma vez com 200 μL de tampão de lavagem. Misture por pipetar cuidadosamente 4 a 5 vezes.
- Coloque no ímã por 1 min e descarte o tampão de lavagem.
- Adicione o fluxo da etapa 3.3.4 às contas e repita o procedimento da vinculação à eluição (etapas 3.3.2 a 3.3.10). Mantenha os eluatos de RNA em tubos separados por enquanto.
NOTA: Opcionalmente, mantenha novamente o equivalente supernante ao passo 3.3.4 em um novo tubo, pois ele pode ser usado como controle. Ao final do procedimento, purificar e concentrar o RNA por precipitação de etanol ou com um método de escolha baseado em coluna (ou seja, RNA clean e concentrador). Esta amostra corresponde a uma amostra de RNA poli-A esgotada e pode ser usada como controle para conversão de bisulfato (etapa 8.2.2). - Quantifique o RNA elucido com um espectotômetro e mantenha uma alíquota de 2 μL para avaliar melhor a qualidade do RNA com um analisador de fragmentos.
NOTA: O RNA poly-A pode ser armazenado a -80 °C até que seja necessário.
4. Fluxo de trabalho de RNA
- Divida as amostras de RNA poli-A celular (mRNA) e RNA viral em 2 alíquotas, dedicadas ao respectivo pipeline de análise epitransomica:
i 5 μg de mRNA celular ou 1 μg de RNA viral para MeRIP-Seq e controles de entrada (vá para as etapas 5 a 7, e passo 9).
(ii) 1 μg de mRNA celular ou 500 ng de RNA viral para BS-Seq (vá para as etapas 8 e 9).
5. Fragmentação do RNA
NOTA: A fragmentação do RNA é realizada com o reagente de fragmentação de RNA e destina-se a MeRIP-Seq e ao controle de amostras de RNA. Este é um passo muito importante que requer uma otimização cuidadosa para obter fragmentos que variam entre 100-200 nt.
- Divida o volume total de mRNA em tubos PCR de 0,2 mL com 18 μL de mRNA/tubo.
NOTA: Trabalhe rapidamente. Não trabalhe com mais de 8 amostras por vez para ter resultados reprodutíveis. O dimensionamento do volume não garantirá uma fragmentação reprodutível e uniforme. - Aqueça um termociclador a 70 °C.
- Adicione 2 μL de reagente de fragmentação na borda de cada tubo PCR.
- Feche o tubo e gire para baixo (para que o reagente entre em contato com o RNA ao mesmo tempo para os 8 tubos).
- Incubar as amostras 15 min a 70 °C no termociclizado pré-aquecido.
- Assim que a incubação acabar, adicione rapidamente 2 μL de solução Stop em cada tubo.
- Gire para baixo e deixe sentar-se no gelo até pronto para prosseguir para o próximo passo.
NOTA: O tempo de incubação pode variar de acordo com o número de amostras a serem processadas, mas não deve exceder 1h para evitar qualquer degradação do RNA. - Repita o procedimento para todas as amostras (se houver mais de 8 alíquotas).
- Misture os tubos e prossiga para a purificação do RNA com um kit RNA limpo e concentrador (etapa 6) ou qualquer kit personalizado baseado em coluna para se livrar dos buffers e recuperar RNA fragmentado limpo na água.
6. Purificação de RNA
NOTA: Esta etapa pode ser realizada por precipitação de etanol ou com qualquer tipo de método de purificação e concentração de RNA baseado em coluna (ou seja, RNA Clean e Concentrador).
- Elute ou resuspense o RNA purificado em um volume total de 50-75 μL de água livre de DNase/RNase.
NOTA: Se um método baseado em coluna for usado, duas rodadas de eluição são fortemente recomendadas para garantir a recuperação máxima. - Quantifique o mRNA fragmentado purificado com um espectrofotômetro e avalie a qualidade do RNA com um analisador de fragmentos.
- Mantenha 100 ng de mRNA fragmentado como controle de entrada para preparação e sequenciamento da biblioteca (vá para o passo 9). O mRNA fragmentado restante (mínimo 2,5 μg) pode ser usado para MeRIP (ir para a etapa 7.2).
7. MeRIP
NOTA: É necessário um mínimo de 2,5 μg de mRNA fragmentado para cada imunoprecipitação (IP), seja usando um anticorpo anti-m6A específico (condição de teste) ou usando um anticorpo anti-IgG (controle negativo).
- Preparação de contas magnéticas para imunoprecipitação
- Para cada amostra, prepare 4 mL de tampão IP 1x em um novo tubo cônico, diluindo 800 μL de mRNA IP buffer 5x (50 mM Tris-HCl pH 7.4, 750 mM NaCl, 0,5% Igepal CA-630, e água sem nuclease) com 3,2 mL de água sem nuclease.
NOTA: São necessárias pelo menos 2 reações (um teste e um controle IgG). - Coloque o tubo no gelo.
- Rotule o número apropriado de tubos de microcentrifuus de 1,5 mL para o número de reações IP desejadas:
n tubos (teste) para anticorpo anti-m6A.
n tubos (controle negativo) para IgG do Mouse Normal. - Resuspend as contas magnéticas (por exemplo, Magna ChIP Protein A/G ) invertendo e vórtice. Nenhum monte de contas deve ser visível.
- Para cada reação planejada, transfira 25 μL de contas magnéticas para um tubo de microcentrifutura.
- Adicione dez vezes mais tampão IP 1x (da etapa 7.1.1) em relação ao volume original de contas usadas (ou seja, 250 μL de 1x tampão IP por 25 μL de contas magnéticas).
- Misture as contas ao empinar suavemente várias vezes para a ressuspensão completa.
- Coloque o tubo no separador magnético por 1 min.
- Remova e descarte o supernatante, certificando-se de não aspirar quaisquer contas magnéticas. Remova o tubo do ímã.
- Repita a etapa de lavagem (passos 7.1.6 a 7.1.9).
- Resuspenda as contas em 100 μL de 1x TAMPão IP por 25 μL de volume original de contas magnéticas.
- Adicione 5 μL de anticorpo (1 μg/μL) por 25 μL de volume original de contas magnéticas.
n tubos (teste) com anticorpo anti-m6A (clone 17-3-4-1) [1 μg/μL].
n tubos (controle negativo) com IgG do Mouse Normal (1 μg/μL). - Incubar na roda giratória por 30 minutos à temperatura ambiente para permitir a conjugação dos anticorpos com as contas magnéticas.
- Coloque o tubo no separador magnético por 1 min. Descarte o supernatante. Remova o tubo do ímã e resuspense a mistura de anticorpos em 100 μL de 1x de buffer IP.
- Para cada amostra, prepare 4 mL de tampão IP 1x em um novo tubo cônico, diluindo 800 μL de mRNA IP buffer 5x (50 mM Tris-HCl pH 7.4, 750 mM NaCl, 0,5% Igepal CA-630, e água sem nuclease) com 3,2 mL de água sem nuclease.
- Imunoprecipitação de RNA (RIP)
- Prepare 500 μL de mistura de reação RIP para cada amostra de 2,5 μg mRNA da seguinte forma: 2,5 μg em 100 μL de RNA fragmentado (a partir da etapa 6.12); 295 μL de água sem nuclease; 5 μL de 40 Inibidores de RNase U/μL; e 100 μL de buffer IP de 5x.
- Adicione 500 μL de mistura de reação RIP a cada mistura de anticorpos (~100 μL da etapa 7.1.14). Misture suavemente a pipetação várias vezes para resuspensar completamente as contas. Coloque no gelo.
- Incubar todos os tubos RIP em uma roda giratória por 2 horas a 4 °C.
- Centrifugar as reações de MeRIP brevemente para girar gotículas líquidas da tampa e dos lados do tubo. Coloque os tubos em um separador magnético por 1 min.
- Transfira o supernatante em um novo tubo de centrífuga, tomando cuidado para não perturbar as contas magnéticas.
NOTA: O fluxo pode ser mantido como controle para verificar a eficiência do RIP (vá para a etapa 7.3.9). - Remova os tubos do ímã. Lave as contas adicionando 500 μL de tampão IP frio de 1x. Misture as contas ao empinar suavemente várias vezes para resuspensar completamente as contas.
- Coloque os tubos em um separador magnético por 1 min e descarte o supernaspeso.
- Repita o procedimento de lavagem (etapas 7.2.6-7.2.7) duas vezes para um total de 3 lavagens.
- Coloque os tubos no gelo e proceda imediatamente para a eluição.
- Eluição
- Prepare a solução m6A de 20 mM dissolvendo 10 mg de N6-Metiladenosina, 5′-monofosfato de sal de sódio (m6A) em 1,3 mL de água sem nuclease. Prepare 150 alíquotas μL e armazene a -20 °C.
- Para cada amostra (teste e controles): Prepare 225 μL de tampão de eluição misturando os seguintes componentes: 45 μL de 5x IP Buffer, 75 μL de 20 mM m6A, 3,5 μL de 40U/μL RNase Inibidor e 101,5 μL de água sem nuclease.
- Adicione 100 μL de tampão de eluição (da etapa 7.3.2) às contas (a partir do passo 7.2.9). Misture suavemente a pipetação várias vezes para resuspensar completamente as contas.
- Incubar todos os tubos por 1h com agitação contínua em um roqueiro a 4 °C.
- Centrifugar brevemente as reações RIP para girar gotículas líquidas da tampa e dos lados do tubo. Coloque os tubos em um separador magnético por 1 min.
- Transfira o supernascimento contendo fragmentos de RNA elucidos para um novo tubo de microcentrífuga de 1,5 mL. Tenha cuidado para não aspirar as contas, pois aumentará o ruído de fundo.
- Repetição de etapas de elução (7.3.3 a 7.3.6) adicionando novamente 100 μL de tampão de eluição, incubando 1h a 4 °C e coletando o elunato após a separação magnética.
- Combine todos os eluatos da mesma amostra (o volume total de eluição deve ser de 200 μL).
- Purificar o RNA e o fluxo (opcional, a partir da etapa 7.2.5) por precipitação de etanol ou por um método de escolha baseado em coluna (ou seja, RNA Clean e Concentrador).
- Avalie a quantidade de RNA e a qualidade do fluxo através e eluvado amostras com um analisador de fragmentos usando um kit de detecção de alta sensibilidade. Se a qualidade do RNA for satisfatória, proceda à preparação da biblioteca e sequenciamento de alto rendimento (etapa 9).
NOTA: A quantidade de RNA recuperada no MeRIP é muito baixa e requer, imperativamente, kits de detecção de alta sensibilidade para garantir a quantificação. Se não houver bioanalítômetro disponível, é possível prosseguir cegamente para a preparação da biblioteca.
8. Conversão de Bisullfita de RNA
- Preparação de Controle e Reagente
- Controle de pico de mistura ERCC: Adicione o mix ERCC seguindo as instruções do fabricante, que recomendam a adição de 0,5 μL de mistura ERCC não diluída a 500 ng de mRNA. Este controle pode ajudar a avaliar a eficiência da conversão de bisulfato.
- RNA despovado de Pico Poly-A (da etapa 3.3.14) a uma razão de 1/1000 (ou seja, 500 pg de RNA poli-A esgotada para 500 ng de mRNA). Esta amostra é enriquecida em RNA ribossômico e deve conter assim o rRNA 28S, um controle positivo para conversão de bisulfato.
NOTA: O RNA total também pode ser usado como controle positivo em vez de RNA poli-A esgotado. - Realize a conversão de bisulfita com um kit de metilação de RNA (por exemplo, Zymo EZ).
- RNA Wash Buffer: Adicione 48 mL de 100% de etanol (ou 52 mL de 95% de etanol) a 12 mL de concentrado RNA Wash Buffer antes do uso.
- Conversão de Bisullfita
NOTA: A conversão de bisulfito foi realizada com um kit de conversão de bisulfito de RNA comercialmente disponível, seguindo o procedimento do fabricante, conforme indicado abaixo.- Em tubos PCR de 0,2 mL, adicione 1000 ng de mRNA (ou entre 300 e 1000 ng). Adicione controles de espeto: 1 μL de mistura ERCC (etapa 8.1.1) e 1000 pg de RNA poli-A esgotado (passo 8.1.2). Volume completo de até 20 μL com água livre de DNase/RNase.
- Adicione 130 μL de reagente de conversão de RNA a cada amostra de RNA de 20 μL.
- Misture a amostra por pipetting para cima e para baixo.
- Gire brevemente para garantir que não haja gotículas na tampa ou nas laterais do tubo.
- Coloque os tubos PCR em um cicloviário térmico e execute as seguintes etapas: desnaturação a 70 °C por 5 min; conversão a 54 °C para 45 min; repetir etapas de desnaturação e conversão para um total de 3 ciclos; e, em seguida, manter a 4 °C indefinidamente.
NOTA: Três ciclos de desnaturação e conversão de bisullfita garantem a conversão completa de bisulfito da amostra. As amostras podem ser armazenadas a -80 °C ou processadas diretamente. - Proceda com dessulphonação na coluna. Coloque uma coluna em um tubo de coleta vazio e adicione 250 μL de tampão de ligação de RNA à coluna.
- Carregue a amostra (~150 μL a partir da etapa 8.2.5) na coluna que contém o buffer de ligação do RNA e misture por tubulação para cima e para baixo.
- Adicione 400 μL de 95-100% de etanol à mistura de buffer de ligação amostra-RNA na coluna. Feche a tampa e misture imediatamente invertendo a coluna várias vezes.
- Centrifugar a toda velocidade (≥ 10.000 x g) para 30 s. Descarte o fluxo através do fluxo.
- Adicione 200 μL de RNA Wash Buffer à coluna e centrífuga a toda velocidade por 30 s.
- Adicione 200 μL de tampão de dessefonação de RNA à coluna e incubar em temperatura ambiente por 30 minutos. Após a incubação, centrífuga a toda velocidade por 30 s. Descarte o fluxo através do fluxo.
- Adicione 400 μL de RNA Wash Buffer à coluna e centrífuga a toda velocidade por 30 s. Repita a etapa de lavagem com um adicional de 400 μL de RNA Wash Buffer. Descarte o fluxo através do fluxo.
- Centrifugar a coluna no tubo de coleta esvaziado a toda velocidade por 2 minutos. Transfira a coluna para um tubo sem RNase.
- Adicione ≥ 10 μL de água livre de DNase/RNase diretamente à matriz da coluna e incubar por 1 min a temperatura ambiente. Centrífuga a toda velocidade por 30 s.
NOTA: Normalmente eluto em um volume de 20 μL. O RNA elucido pode ser usado imediatamente ou armazenado a -20 °C por até 3 meses. Para armazenamento a longo prazo, mantenha-se a -80 °C. - Tire 2,5 μL para avaliação do analisador de fragmentos da qualidade e quantidade do RNA e prossiga para a preparação da biblioteca e sequenciamento de alto rendimento (etapa 9).
- Tome 4 μL de RNA convertido para controle de conversão de bisullfita de eficiência (etapa 8.3).
- Controle de conversão de bisulfato por RT-PCR
NOTA: Esta etapa garante que a conversão de bisulfato tenha sido bem sucedida antes de prosseguir para o sequenciamento . 28S ribossômico RNA do Homo sapiens será usado como controle positivo para análise de metilação de RNA, como o resíduo C na posição 4447 (adesão do GenBank # NR_003287) foi descrito como sendo 100% metilado.
Sequências de primer:
H 28SF primer: 5'-GGGGTTTTAYGATTTTTTTGATTTTTTTTGGG-3'
H 28SR primer: 5'-CCAACTCACRTTCCCTATAAAAC-3'- Prepare a mistura de reação de transcrição reversa (RT) usando um kit de transcrição reversa cDNA de alta capacidade. Descongele os componentes do kit no gelo e prepare a mistura mestre RT no gelo da seguinte forma:
4 μL de RNA convertido bisulfo (da etapa 8.2.14):
2 μL de buffer 10xRT
0,8 μL de 25x dNTP Mix [100 mM]
2 μL de primers aleatórios RT de 10x
1 μL de Transcriptase reversa multiscrito
1 μL de Inibidor de RNase
9,2 μL de H2O sem nuclease
NOTA: Cada reação rt deve conter um volume final de 20 μL em tubos PCR de 0,2 mL. - Coloque os tubos no cicloviário térmico com o seguinte programa RT: 25 °C por 10 min; 37 °C por 120 min; 85 °C por 5 min; em seguida, a 4 °C indefinidamente.
- Prepare a reação do PCR para amplificar especificamente o rRNA 28S com uma enzima de revisão pcr. Descongele os componentes do kit no gelo, suavemente vórtice e centrífugue brevemente. Prepare a mistura mestre pcr no gelo ou em um suporte de placa de metal gelado da seguinte forma:
0,6 μL de 10 μM H 28SF primer
0,6 μL de 10 μM H 28SF primer
6,5 μL de modelo cDNA
22,5 μL de DNA Polymerase master mix
NOTA: Cada reação do PCR deve conter um volume final de 20 μL em tubos PCR de 0,2 mL. - Coloque os tubos no cicloviário térmico com o seguinte programa PCR: denaturação inicial a 95 °C por 5 min; 45 ciclos de desnaturação (95 °C para 15 s), ressarem (57 °C para 30 s) e alongamento (72 °C para 15 s), alongamento final a 72°C por 10 min e, em seguida, mantendo-se a 4 °C indefinidamente.
- Execute 10 μL da reação em um gel de 2% de agarose. O tamanho esperado da banda é de 130 a 200 bp.
- Prepare a mistura de reação de transcrição reversa (RT) usando um kit de transcrição reversa cDNA de alta capacidade. Descongele os componentes do kit no gelo e prepare a mistura mestre RT no gelo da seguinte forma:
- Sequenciamento de produtos PCR
- Purifique os produtos PCR com um método de escolha baseado em coluna para remover enzimas e resíduos dNTP e elute o DNA amplificado em pelo menos 20 μL de água livre de DNase/RNase.
- Quantifique o DNA purificado com espectotômetro.
- Reação de sequenciamento
- Use 40 ng de reação de produto/sequenciamento PCR.
- Sequência em ambas as direções com as primers H 28SF e H 28SR.
- Alinhe as sequências com a sequência não convertida conhecida (28S ribossmal N5 (RNA28SN5). Verifique a presença de um resíduo C na posição C4447, e para resíduos T em vez de C em outros lugares.
9. Preparação da biblioteca e Sequenciamento de Alto Rendimento
- Prepare bibliotecas para sequenciamento usando kits de mRNA (por exemplo, Illumina TruSeq Stranded), iniciando o protocolo na etapa Elute-Prime-Fragment e seguindo as instruções do fabricante.
- No entanto, para as amostras de entrada RNA-Seq e MeRIP-Seq, incubar as amostras a 80 °C por 2 min para apenas prime, mas não fragmentá-las mais.
- Realize o sequenciamento usando plataformas Illumina. Reações de sequenciamento podem ser realizadas de acordo com preferências e design experimental, extremidades individuais ou emparelhadas, com um mínimo de 100 nt de comprimento.
10. Análises bioinformáticas
- processamento de dados m6A
- Execute o FASTQC24 para avaliar a qualidade de leitura em m6A e insira arquivos FASTQ a partir de sequenciamento.
- Execute o Atropos25 para aparar sequências de extremidade e adaptador de baixa qualidade das leituras. Defina os seguintes parâmetros na execução de Atropos.
- Remova as seguintes sequências de adaptador: AGATCGGAAGAG, CTCTTCCGATCT, AACACTCTTTCCCT, AGATCGGAAGAGCG, AGGGAAAGAGTGTT, CGCTCTTCCGATCT.
- Use o seguinte corte de qualidade Phred: 5, para aparar extremidades de baixa qualidade conforme especificado pelo fabricante (https://support.illumina.com/downloads/illumina-adapter-sequences-document-1000000002694.html).
- Use o seguinte comprimento mínimo de leitura após o corte: 25 pares de base.
- Mesclar a referência GRh38 do genoma humano e do HIV [linear integrado pNL4-3Env-GFP] no formato FASTA.
- Indexe a referência mesclada com HISAT226.
- Execute HISAT2 em leituras aparadas para alinhar à referência indexada. Use parâmetros HISAT padrão.
- Classificar e indexar as leituras alinhadas com SAMtools27.
- Execute a estatística SAMtools e qualimap 228, para verificação de qualidade pós-alinhamento das bibliotecas sequenciadas.
- Opcionalmente, colete e resumir medidas de qualidade da etapa anterior com multiQC29.
- O genoma do HIV tem sequências homólogas de 634 bps na LTR de 5' e 3' LTR: Realinhar leituras multimapping de 5''LTR para a região correspondente de 3' LTR com SAMtools.
- Para identificar os picos m6A, execute o software de chamada de pico MACS230 (v 2.1.2). Selecione cuidadosamente os parâmetros de execução do MACS2, a fim de garantir o funcionamento correto dos dados do RNA-Seq, pois a chamada de pico pode ser afetada pelo nível de expressão genética, e exons curtos podem ser mal chamados como picos. Assim, o sinal de entrada deve ser subtraído do sinal m6A, sem a suavização rotineiramente aplicada pelo MACS2 aos dados baseados em DNA. Aplique os seguintes parâmetros ao subcomandamento 'callpeak' do MACS2:
-keep-dup auto (controla o comportamento MACS2 para leituras duplicadas, 'auto' permite que macs calcule o número máximo de leituras no mesmo local com base na distribuição binomial usando 1e-5 como corte de valor p)
-g 2.7e9 (tamanho do genoma humano em bp)
-q 0,01 (corte mínimo de FDR para chamar picos significativos)
-nomodel (para contornar a construção do modelo de mudança, que é adaptado para experimentos ChIP-Seq)
-slocal 0
-llocal 0 (definir este e o parâmetro anterior para 0 permite que macs2 subtraia diretamente, sem suavização, a entrada lê a partir das leituras m6A)
-extsize 100 (comprimento médio de fragmentos em bp)
-B - Execute o sub-comando diferencial de chamada de pico de MACS2, 'bdgdiff' para comparar amostras infectadas vs não infectadas. 'bdgdiff' toma como entradas os arquivos bedGraph gerados por 'callpeak' na etapa anterior. Para cada ponto de tempo, execute a comparação de amostras infectadas versus não infectadas com 'bdgdiff', subtraindo o respectivo sinal de entrada do sinal m6A e fornecendo os parâmetros adicionais: -g 60 -l 120.
- processamento de dados m5C
- Execute Cutadapt31 para aparar sequências de adaptador das leituras brutas, com os seguintes parâmetros:
adaptador "AGATCGGAAGAGCACACGTCTGAAC"
-comprimento mínimo=25. - Complemente inversa as leituras aparadas usando seqkit32, à medida que o protocolo de sequenciamento produz leituras da vertente inversa.
- Execute o FastQC para examinar a qualidade da leitura.
- Mesclar a referência de genoma humano GRh38 e HIV [linear integrado pNL4-3Env-GFP] no formato FASTA.
- Indexe a referência mesclada com o aplicativo meRanGh do pacote meRanTK33.
- Alinhe-se com o meRanGh com os seguintes parâmetros:
-ONU permitindo que leituras não mapeadas sejam escritas para arquivos de saída
-MM permitindo que leituras multi mapeadas sejam escritas para arquivo de saída
-bg para saída no bedGraph
-mbgc 10 filtro relatado região por cobertura (pelo menos 10 leituras de cobertura) - O genoma do HIV tem sequências homologous de 634 bps nas LTR de 5' e 3'LTR: realinhar leituras multimapping de 5''LTR para a região correspondente de 3' LTR com SAMtools.
- Execute a chamada de metilação através da ferramenta meRanCall, fornecida pelo meRanTK, com os seguintes parâmetros:
-rl = 126, comprimento de leitura
-ei = 0,1, intervalo de erro para o cálculo da taxa de metilação p-valor
-cr = 0,99, conversão esperada - Execute o utilitário do MeRanTK estimateSizeFactors.pl para estimar fatores de tamanho de cada amostra. Os fatores de tamanho serão usados como parâmetros na próxima etapa.
- Execute o MeRanCompare para análise diferencial de metilação de não infectados versus infectados através dos pontos de tempo 12, 24 e 36h. São aplicados os seguintes parâmetros: um valor de significância de 0,01 como o limiar mínimo para relatórios e fatores de tamanho da etapa anterior.
- Execute Cutadapt31 para aparar sequências de adaptador das leituras brutas, com os seguintes parâmetros:
Resultados
Este fluxo de trabalho tem se mostrado útil para investigar o papel da metilação m6A e m5C no contexto da infecção pelo HIV. Para isso, usamos um modelo de linha celular CD4+ T (SupT1) que ou infectamos com HIV ou não tratados. Iniciamos o fluxo de trabalho com 50 milhões de células por condição e obtivemos uma média de 500 μg de RNA total com um número de qualidade de RNA de 10 (Figura 1A-B). Após a seleção poli-A recuperamos entre 10 e 12 μg de mRNA por condição (representando cerca de 2% do RNA total) (Figura 1B). Neste ponto, usamos 5 μg de RNA poli-A selecionado para o gasoduto MeRIP-Seq e 1 μg para o gasoduto BS-Seq. Uma vez que o RNA do HIV é poli-adenilado, nenhuma ação adicional é necessária e os procedimentos MeRIP-Seq e BS-Seq podem ser aplicados diretamente.
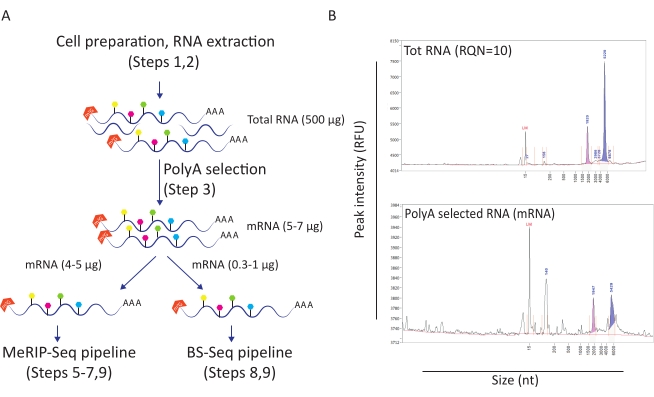
Figura 1: Preparação de RNA para aplicações a jusante. A) Fluxo de trabalho que retrata a preparação e distribuição do RNA para os gasodutos MeRIP-Seq e BS-Seq simultâneos. Cada forma hexagonal preenchida representa um tipo de modificação de RNA, como m6A (verde) ou m5C (rosa). São indicadas quantidades de material de RNA necessários para a realização do experimento. B) Resultados representativos que retratam os perfis de distribuição de RNA esperados (tamanho e quantidade) na extração total de RNA (painel superior) e seleção poli-A (painel inferior). As amostras foram carregadas no analisador de fragmentos com kit de sensibilidade padrão, a fim de avaliar a qualidade do RNA antes de inserir procedimentos específicos de MeRIP-Seq e BS-Seq. RQN: número de qualidade de RNA; nt: nucleotídeos. Clique aqui para ver uma versão maior desta figura.
O gasoduto MeRIP-Seq é uma técnica baseada em imunoprecipitação de RNA que permite a investigação da modificação m6A ao longo das moléculas de RNA. Para isso, o RNA é primeiro fragmentado e depois incubado com anticorpos específicos m6A acoplados a contas magnéticas para imunoprecipitação e captura. Fragmentos de RNA enriquecidos por MeRIP e a fração intocada (entrada) são então sequenciados e comparados para identificar regiões de RNA modificadas por m6A e, portanto, transcrições m6A-metiladas (Figura 2A). A resolução da técnica depende da eficiência da fragmentação do RNA. De fato, fragmentos mais curtos permitem uma localização mais precisa do resíduo m6A. Aqui, rnas poli-A selecionadas celulares e RNAs virais foram submetidos à fragmentação baseada em íons com tampão de fragmentação de RNA durante 15 min em um volume final de 20 μL para obter fragmentos de RNA de 100-150 nt. A partir de 5 μg de mRNA, recuperamos 4,5 μg de RNA fragmentado, correspondendo a uma taxa de recuperação de 90% (Figura 2B). Usamos 100 ng de RNA fragmentado e purificado como controle de entrada, submetido diretamente à preparação e sequenciamento da biblioteca. O RNA restante (~4,4 μg) foi processado de acordo com o gasoduto MeRIP-Seq, que começa com a incubação de RNA fragmentado com contas ligadas a anticorpos específicos anti-m6A ou a anticorpos anti-IgG como controle. RIP (MeRIP) específico para m6A de 2,5 μg de RNA fragmentado permitiu recuperar cerca de 15 ng de material enriquecido com m6A que passou por preparação e sequenciamento da biblioteca (Figura 2B). RIP com controle anti-IgG, como esperado, não rendeu RNA suficiente para permitir uma análise mais aprofundada (Figura 2B).
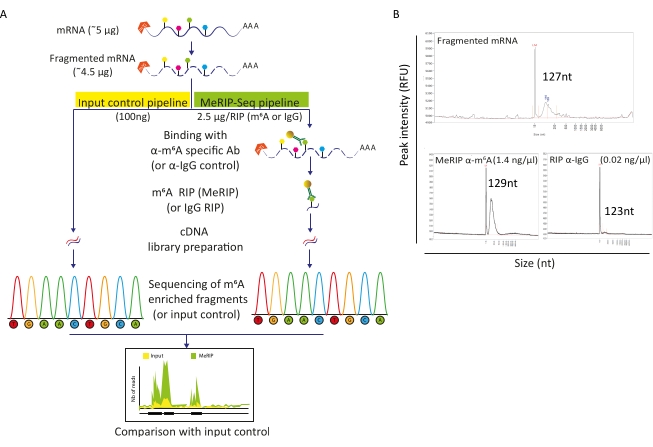
Figura 2: Pipeline MeRIP-Seq. A) Representação esquemática do fluxo de trabalho MeRIP-Seq e controle de entrada. Após a seleção poli-A, as amostras foram fragmentadas em peças de 120-150 nt e, diretamente submetidas a sequenciamento (100 ng, controle de entrada), ou utilizadas para imunoprecipitação de RNA (2,5 μg, RIP) com anticorpo específico anti-m6A ou anticorpo anti-IgG como controle negativo antes do sequenciamento. B) Resultados representativos mostrando os perfis de distribuição de RNA esperados (tamanho e quantidade) após a fragmentação (painel superior) e RIP (painéis inferiores, MeRIP: esquerda, controle IgG: à direita). As amostras foram carregadas no analisador de fragmentos para avaliar a qualidade e concentração do RNA antes de posterior processamento para preparação e sequenciamento da biblioteca. A análise fragmentada do RNA foi realizada utilizando-se o kit de sensibilidade padrão RNA, enquanto o RNA imunoprecipitado utilizou o kit de alta sensibilidade. Clique aqui para ver uma versão maior desta figura.
O gasoduto BS-Seq permite a exploração da modificação de RNA m5C na resolução de nucleotídeos e leva à identificação de transcrições m5C-metiladas. Após a conversão de bisulfita, citosinas não metiladas são convertidas em uracil, enquanto as citosinas metiladas permanecem inalteradas (Figura 3A). Devido às condições adversas do procedimento de conversão de bisullfita (ou seja, alta temperatura e pH baixo), os mRNAs convertidos são altamente degradados (Figura 3B), porém isso não interfere na preparação e sequenciamento da biblioteca. A conversão de bisulfita é eficiente apenas no RNA de uma única-encalhada e, portanto, pode ser potencialmente prejudicada por estruturas secundárias de RNA de dupla-encalhadas. Para avaliar a eficiência da conversão C-U introduzimos dois controles. Como controle positivo, aproveitamos a presença previamente descrita de uma citosina altamente metilada na posição C4447 do 28S rRNA23. Após a amplificação e sequenciamento do RT-PCR de um fragmento de 200 bps em torno do local metilado pudemos observar que todas as citosinas foram convertidas com sucesso em uracils, aparecendo assim como timmidinas na sequência de DNA, exceto a citosina na posição 4447 que permaneceu inalterada. Como um controle para taxa de conversão de bisulfita, usamos sequências RNA sintéticas ercc ercc comercialmente disponíveis. Esta mistura consiste em uma piscina de sequências de RNA conhecidas, não metiladas e poli-adenylated, com uma variedade de estruturas secundárias e comprimentos. Após a preparação e sequenciamento da biblioteca, focamos nessas sequências de ERCC para calcular a taxa de conversão, que pode ser realizada contando o número de C convertidos entre os resíduos C totais em todas as sequências de ERCC e em cada amostra. Obtivemos uma taxa de conversão de 99,5%, confirmando a eficiência e o sucesso da reação de conversão de bisulfato (Figura 3D).
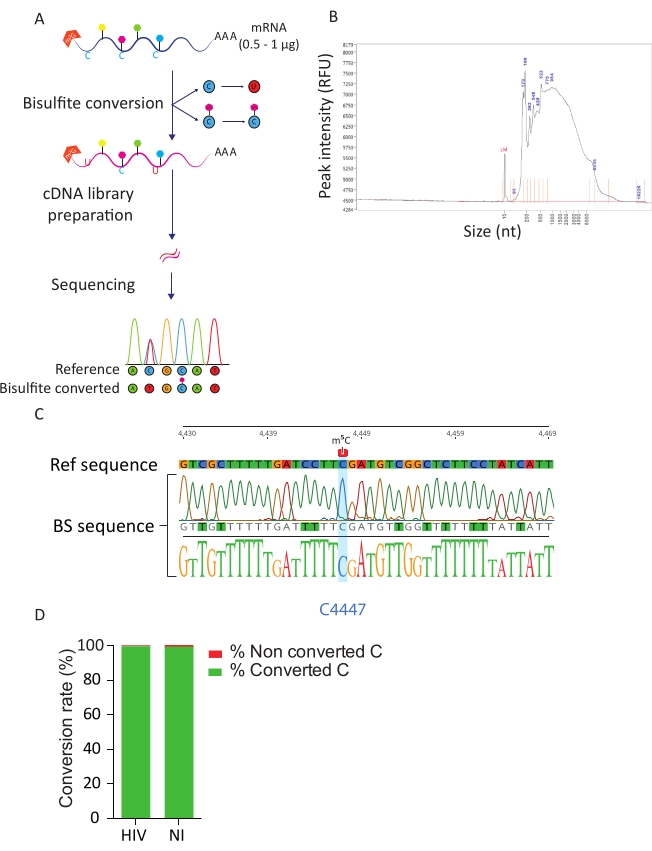
Figura 3: Gasoduto BS-Seq. A) Representação esquemática do fluxo de trabalho BS-Seq. Após a seleção poli-A, as amostras são expostas ao bisulfato, resultando na conversão de C para U (devido à desaminação) para resíduos C não metilados. Em contraste, os resíduos de C metilados (m5C) não são afetados pelo tratamento de bisulfato e permanecem inalterados. B) Resultado representativo do perfil de distribuição de RNA convertido de bisulfato (tamanho e quantidade) após análise no analisador de fragmentos com um kit de sensibilidade padrão. C) Eletroferógrama mostrando resultado representativo de sequenciamento de amplicon RT-PCR da região em torno do C 100% metilado na posição 4447 em 28S rRNA (destacado em azul). Em contraste, os resíduos C da sequência de referência foram identificados como resíduos T na sequência de amplicon devido ao sucesso de conversão de bisullfita. D) Avaliação da taxa de conversão de C-U por análise de sequências de pico do ERCC em células infectadas pelo HIV e não infectadas. A taxa média de conversão é de 99,5%. Clique aqui para ver uma versão maior desta figura.
Amostras enriquecidas com M6A, amostras convertidas por bisulfato e controles de entrada são processados para preparação de bibliotecas, sequenciamento e análise bioinformática (Figura 4). De acordo com o desenho experimental e as perguntas biológicas abordadas, podem ser aplicadas múltiplas análises bioinformáticas. Como prova de princípio aqui, mostramos resultados representativos de uma aplicação potencial (ou seja, análise diferencial de metilação), que se concentra na identificação de transcrições diferencialmente metiladas induzidas pela infecção pelo HIV. Brevemente, investigamos o nível de metilação m6A ou metilação m5C, independentemente de seu nível de expressão genética, tanto em células não infectadas quanto infectadas pelo HIV, a fim de entender melhor o papel das metilações de RNA durante o ciclo de vida viral. Após a normalização da expressão genética, identificamos que a transcrição ZNF469 foi diferencialmente m6A-metilada de acordo com o estado de infecção, na verdade esta transcrição não foi metilada em células não infectadas enquanto exibia vários picos metilados sobre a infecção pelo HIV (Figura 5A). Uma análise de metilação diferencial semelhante no m5C revelou que a transcrição phlpp1 continha vários resíduos metilados, que tendem a ser mais frequentemente metilados na condição do HIV (Figura 5B). Nesse contexto, ambas as análises sugerem que a infecção pelo HIV impacta o epitranscripto celular.
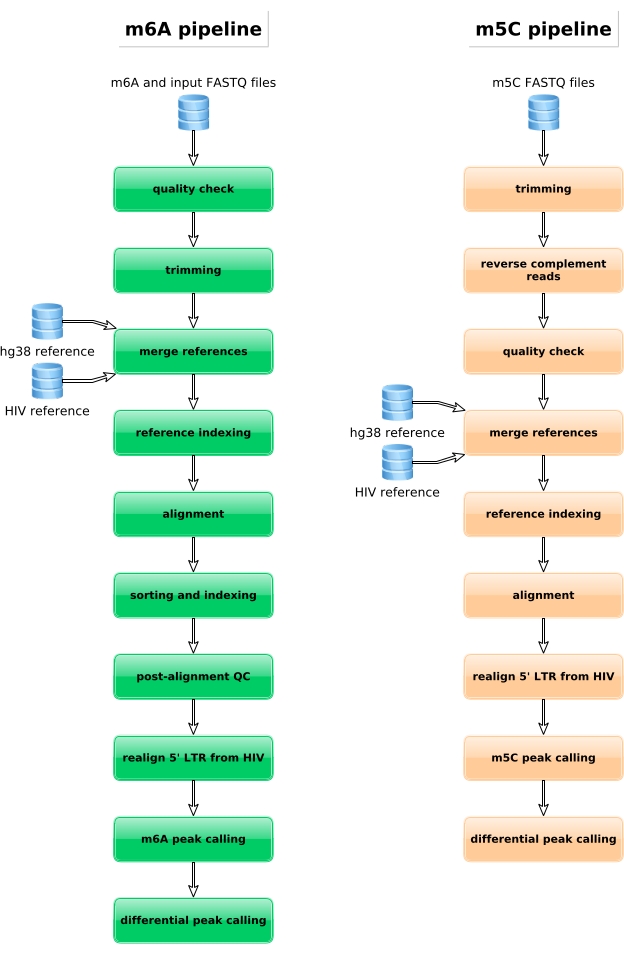
Figura 4: Representação esquemática do fluxo de trabalho bioinforático para a análise dos dados m6A e m5C. Clique aqui para ver uma versão maior desta figura.
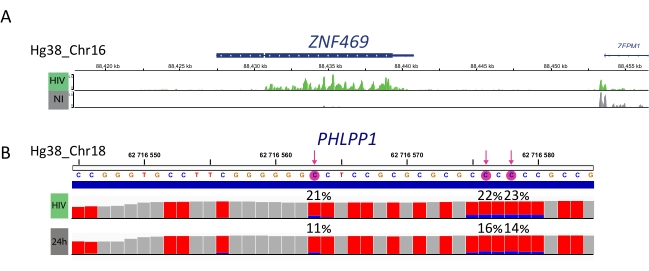
Figura 5: Exemplo de transcrições diferencialmente metiladas após a infecção. A) Resultado representativo mostrando metilação m6A de transcrição ZNF459 em células infectadas pelo HIV (verde) e não infectadas (cinza). A intensidade máxima (após subtração de expressão de entrada) é mostrada no eixo y e posição no cromossomo ao longo do eixo x. A análise diferencial da metilação revela que a transcrição ZFN469 é hipermetilada após a infecção pelo HIV. B) Resultado representativo de células metiladas m5C em células infectadas pelo HIV (pista superior) e não infectadas (pista inferior). A altura de cada barra representa o número de leituras por nucleotídeo e permite a avaliação da cobertura. Cada resíduo C representado em vermelho, e a proporção de C metilado é representada em azul. A taxa exata de metilação (%) é relatada acima de cada resíduo C. Setas destacam diferencialmente significante diferencialmente metilado C. As amostras foram visualizadas utilizando-se do visualizador IGV. Clique aqui para ver uma versão maior desta figura.
Discussão
O papel das modificações do RNA na infecção viral ainda é amplamente desconhecido. Uma melhor compreensão do papel das modificações epitranscriptômicas no contexto da infecção viral poderia contribuir para a busca de novas metas de tratamento antiviral.
Neste trabalho, fornecemos um fluxo de trabalho completo que permite a investigação dos epitranscriptos m6A e m5C de células infectadas. Dependendo da questão biológica, aconselhamos usar o RNA poli-A selecionado como material inicial. Embora opcional, como o gasoduto poderia ser usado com RNA total, é importante ter em mente que as rRNAs, bem como os pequenos RNAs são altamente modificados e contêm um importante número de resíduos metilados. Isso poderia resultar em uma diminuição da qualidade e quantidade de dados significativos de sequenciamento.
No entanto, se o foco do estudo for RNA não poli-adenylated, a etapa de extração de RNA deve ser adaptada para evitar descartar o pequeno RNA (em caso de extração de RNA baseada em coluna) e privilegiar técnicas de esgotamento ribossomo em vez de seleção poli-A para entrar no pipeline.
A fim de garantir RNA de alta qualidade, fragmentação correta e qualidade de RNA enriquecida com m6A e BS adequada para preparação da biblioteca, aconselhamos fortemente usar um analisador de fragmentos ou um bioanalíter. No entanto, este equipamento nem sempre está disponível. Como alternativa, a qualidade do RNA, mRNA e tamanho do RNA fragmentado também poderia ser avaliada pela visualização em gel de agarose. Alternativamente, a preparação da biblioteca pode ser realizada sem avaliação prévia da quantidade de RNA.
Usamos a técnica MeRIP-Seq16 baseada em anticorpos para explorar a paisagem epitranscriptômica m6A. Esta técnica é baseada na imunoprecipitação de RNA e é bem sucedida; no entanto, algumas etapas precisam de uma otimização cuidadosa e podem ser críticas. Embora a metilação m6A tenha sido descrita para ocorrer principalmente dentro da sequência de consenso RRA*CH, este motivo é altamente frequente ao longo de moléculas de mRNA e não permite a identificação precisa do local metilado. Assim, é fundamental alcançar uma fragmentação de RNA reprodutível e consistente, gerando pequenos fragmentos de RNA, para melhorar a resolução baseada em RIP. Neste protocolo, recomendamos um procedimento otimizado, proporcionando resultados reprodutíveis e consistentes em nosso cenário experimental; no entanto, essa etapa de fragmentação pode precisar de maior otimização de acordo com características específicas da amostra.
Recentemente, uma nova técnica que permite o sequenciamento direto m6A foi descrita. Baseia-se no uso de variantes específicas de transcriptase reversa que exibem assinaturas rt exclusivas como resposta à alteração de RNA m6A24. Essa tecnologia, após uma otimização cuidadosa, poderia contornar a maior limitação enfrentada pelo MeRIP-Seq (diminuindo a quantidade de material inicial e permitindo uma resolução maior). Para explorar a modificação m5C decidimos usar a técnica de conversão de bisulfato para detectar na resolução nucleotídea os resíduos C modificados. A fim de reduzir a taxa de falso positivo devido à presença de estruturas secundárias de RNA, realizamos 3 ciclos de conversão de denaturação/bisulfita e maior desempenho da taxa de conversão de bisulfita de controle, graças ao uso de controles de pico de ERCC. Uma das limitações ligadas a esta técnica é que a conversão de bisulfito é muito dura e três ciclos de conversão de desnaturação/bisulfato poderiam degradar algum RNA e, consequentemente, reduzir a resolução. No entanto, em nosso cenário, optamos por se contentar com uma resolução potencialmente ligeiramente menor, a fim de aumentar a qualidade do conjunto de dados.
Graças a essas otimizações e controles, conseguimos fornecer um fluxo de trabalho confiável e sólido que pode ser explorado para investigar a paisagem epitransomica e sua alteração no contexto de infecções virais, interações hospedeiro-patógeno ou qualquer exposição a tratamentos específicos.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado pela Fundação Nacional de Ciência da Suíça (bolsas 31003A_166412 e 314730_188877).
Materiais
| Name | Company | Catalog Number | Comments |
| AccuPrime Pfx SuperMix | Invitrogen | 12344-040 | |
| anti-m6A antibody _Clone 17-3-4-1 | Millipore | MABE1006 | |
| Chloroform | Merck | 67-66-3 | |
| ERCC | Invitrogen | 4456740 | |
| EZ RNA Methylation Kit | Zymo Research | EZR5001 | |
| Fragment analyzer RNA Kit - HS RNA Kit | Agilent | DNF-472-0500 | |
| Fragment analyzer RNA Kit - RNA Kit | Agilent | DNF-471-0500 | |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystem | 4368814 | |
| Illumina TruSeq Stranded mRNA | Illumina | 20020594 | |
| Magnetic Beads A/G Blend | Merck | 16-663 | |
| N6-Methyladenosine, 5′-monophosphate sodium salt (m6A) | Sigma Aldrich | M2780-10MG | |
| Normal Mouse IgG | Merk | 12371 | |
| Oligo(dT)25 | Life Technologies | 61005, | |
| PCRapace | Stratec | 1020220300 | |
| Quick RNA Viral Kit | Zymo Research | 1034 | |
| RNA Clean & Concentrator | Zymo Research | R1015 | |
| RNA Fragmentation Reagent | Ambion | AM8740 | |
| RNase Inhibitor | Ambion | AM2684 | |
| Trizol | TRIzol Reagent | 15596026 |
Referências
- Machnicka, M. A., et al. MODOMICS: a database of RNA modification pathways--2013 update. Nucleic Acids Research. 41, 262-267 (2013).
- Zaccara, S., Ries, R. J., Jaffrey, S. R. Reading, writing and erasing mRNA methylation. Nature Reviews Molecular Cell Biology. 20 (10), 608-624 (2019).
- Davalos, V., Blanco, S., Esteller, M. SnapShot: Messenger RNA Modifications. Cell. 174 (2), 498 (2018).
- Saletore, Y., et al. The birth of the Epitranscriptome: deciphering the function of RNA modifications. Genome Biology. 13 (10), 175 (2012).
- Zhao, B. S., Roundtree, I. A., He, C. Post-transcriptional gene regulation by mRNA modifications. Nature Reviews Molecular Cell Biology. 18 (1), 31-42 (2017).
- Netzband, R., Pager, C. T. Epitranscriptomic marks: Emerging modulators of RNA virus gene expression. Wiley Interdisciplinary Reviews: RNA. 11 (3), 1576 (2020).
- Pereira-Montecinos, C., Valiente-Echeverria, F., Soto-Rifo, R. Epitranscriptomic regulation of viral replication. Biochimica et Biophysica Acta. 1860 (4), 460-471 (2017).
- Lichinchi, G., et al. Dynamics of the human and viral m(6)A RNA methylomes during HIV-1 infection of T cells. Nature Microbiology. 1, 16011 (2016).
- Courtney, D. G., et al. Epitranscriptomic Addition of m(5)C to HIV-1 Transcripts Regulates Viral Gene Expression. Cell Host & Microbe. 26 (2), 217-227 (2019).
- Kennedy, E. M., et al. Posttranscriptional m(6)A Editing of HIV-1 mRNAs Enhances Viral Gene Expression. Cell Host & Microbe. 19 (5), 675-685 (2016).
- Tirumuru, N., Wu, L. HIV-1 envelope proteins up-regulate N (6)-methyladenosine levels of cellular RNA independently of viral replication. Journal of Biological Chemistry. 294 (9), 3249-3260 (2019).
- Tirumuru, N., et al. N(6)-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression. Elife. 5, (2016).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. Frontiers in Virology. 1 (11), (2021).
- Khoddami, V., Cairns, B. R. Transcriptome-wide target profiling of RNA cytosine methyltransferases using the mechanism-based enrichment procedure Aza-IP. Nature Protocols. 9 (2), 337-361 (2014).
- Hussain, S., Aleksic, J., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (11), 215 (2013).
- Dominissini, D., Moshitch-Moshkovitz, S., Salmon-Divon, M., Amariglio, N., Rechavi, G. Transcriptome-wide mapping of N6-methyladenosine by m6A-seq based on immunocapturing and massively parallel sequencing. Nature Protocols. 8 (1), 176-189 (2013).
- Dominissini, D., et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature. 485 (7397), 201-206 (2012).
- Shobbir Hussain, J. A., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (215), (2013).
- Amort, T., et al. Distinct 5-methylcytosine profiles in poly(A) RNA from mouse embryonic stem cells and brain. Genome Biology. 18 (1), 1 (2017).
- Endrullat, C., Glökler, J., Franke, P., Frohme, M. Standardization and quality management in next-generation sequencing. Applied & Translational Genomics. 10, 2-9 (2016).
- Schaefer, M., Pollex, T., Hanna, K., Lyko, F. RNA cytosine methylation analysis by bisulfite sequencing. Nucleic Acids Research. 37 (2), 12 (2009).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. biorxiv. 1 (11), (2021).
- Squires, J. E., et al. Widespread occurrence of 5-methylcytosine in human coding and noncoding RNA. Nucleic Acids Research. 40 (11), 5023-5033 (2012).
- Aschenbrenner, J., et al. Engineering of a DNA Polymerase for Direct m(6) A Sequencing. Angewandte Chemie (International ed. in English). 57 (2), 417-421 (2018).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Kim, D., Langmead, B., Salzberg, S. L. HISAT: a fast spliced aligner with low memory requirements. Nature Methods. 12 (4), 357-360 (2015).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Okonechnikov, K., Conesa, A., García-Alcalde, F. Qualimap 2: advanced multi-sample quality control for high-throughput sequencing data. Bioinformatics. 32 (2), 292-294 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Zhang, Y., et al. Model-based Analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), 137 (2008).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet Journal. 17 (1), (2011).
- Shen, W., Le, S., Li, Y., Hu, F. SeqKit: A Cross-Platform and Ultrafast Toolkit for FASTA/Q File Manipulation. PLOS ONE. 11 (10), 0163962 (2016).
- Rieder, D., Amort, T., Kugler, E., Lusser, A., Trajanoski, Z. meRanTK: methylated RNA analysis ToolKit. Bioinformatics. 32 (5), 782-785 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados