Method Article
Estabelecendo Culturas Binárias Estáveis de Saccharibacterias simbióticas da Cavidade Oral
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Demonstramos um método para isolar membros difíceis de crescer do novo filo bacteriano, Saccharibacteria, filtrando placas dentárias e co-culminando com bactérias hospedeiras.
Resumo
Muitas espécies bacterianas não podem ser cultivadas em laboratório usando métodos padrão, representando uma barreira significativa para estudar a maioria da diversidade microbiana na Terra. Novas abordagens são necessárias para cultivar essas bactérias não cultivadas para que os pesquisadores possam estudar efetivamente sua fisiologia e estilo de vida usando as poderosas ferramentas disponíveis em laboratório. A Radiação Phyla Candidata (RCP) é um dos maiores grupos de bactérias não cultivadas, compreendendo ~15% da diversidade viva na Terra. O primeiro isolado deste grupo foi um membro do filo saccharibacteria, 'Nanosynbacter lyticus' cepa TM7x. TM7x é uma bactéria extraordinariamente pequena que vive como um simbionte em contato direto com um hospedeiro bacteriano, Schaalia odontolytica, cepa XH001. Aproveitando o tamanho inusitadamente pequeno das células e seu estilo de vida como organismo simbiótico, desenvolvemos um protocolo para cultivar rapidamente saccharibacterias a partir de placas dentárias. Este protocolo mostrará como filtrar uma suspensão da placa dentária através de um filtro de 0,2 μm, em seguida, concentrar as células saccharibacteria coletadas e infectar uma cultura de organismos hospedeiros. A cocultura resultante pode ser aprovada como qualquer cultura bacteriana normal e a infecção é confirmada pela PCR. A cultura binária resultante pode ser mantida em laboratório e usada para experimentos futuros. Embora a contaminação seja uma possibilidade, a cultura binária pode ser purificada por filtrar e reinfeccionar mais o hospedeiro, ou por chapeamento da cultura binária e triagem para colônias infectadas. Esperamos que este protocolo possa ser expandido para outros tipos de amostras e ambientes, levando ao cultivo de muitas outras espécies na RCP.
Introdução
A colheita de novas espécies de bactérias e trazê-las para o laboratório permite que experimentos poderosos compreendam melhor sua fisiologia e interações mais amplas dentro de sua comunidade microbiana. Embora existam métodos livres de cultura para interrogar essas questões (por exemplo, "meta-omics"), as interações complexas de diversas populações microbianas dificultam a provocação de variáveis únicas e a chegar a conclusões significativas. Embora a cultura de bactérias tenha muitos benefícios, existem muitas barreiras potenciais para isolar uma bactéria e cultuá-la em cultura pura. Os potenciais requisitos específicos de crescimento incluem pH, tensão de oxigênio, vitaminas, fatores de crescimento, moléculas de sinalização ou até mesmo contato celular direto para obter crescimento1. No entanto, acredita-se que auxotrophies específicos são o principal impedimento para a cultura de novas espécies de bactérias. Formulações de mídia padrão carecem de muitos nutrientes exigidos por bactérias não cultivadas, como vitaminas específicas ou fontes de carbono. Essas moléculas ausentes podem ser a chave para a fisiologia das bactérias não culturadas e geralmente são fornecidas por outro organismo na comunidade microbiana ou por um organismo hospedeiro. Por exemplo, carboidratos complexos como mucinas podem ser fornecidos por hospedeiros animais. Somando-as à mídia, permitiu o cultivo de várias bactérias das tripas animais, incluindo a Akkermansia muciniphila e os mucinivoranos hirudinis2,3,4. Muitas bactérias patogênicas desenvolveram a capacidade de usar ferro ligado à hemin em células animais, incluindo o patógeno oral Porphyromonas gingivalis5. Em laboratório, o crescimento de Porphyromonas e outros organismos, pode ser estimulado pela adição de hemin6.
Recentemente, muitos avanços na cultura de novos isolados de bactérias vieram através da co-cultivo, usando um organismo "alimentador" para fornecer fatores específicos para bactérias não culturadas necessárias para seu crescimento. Um estudo elegante de Vartoukian e colegas mostrou que siderophores, moléculas de ligação de ferro produzidas por bactérias, estimularam o crescimento de vários novos isolados orais. Pyoverdines, um tipo de siderophore produzido por espécies pseudomonad, mostrou-se para facilitar significativamente o crescimento de uma nova espécie Prevotella 7. No mesmo estudo, foi cultivado o primeiro isolado oral para o filo Chloroflexi, utilizando-se também f. nucleatum como auxiliar para fornecer algum composto ainda desconhecido7. Mais recentemente, uma bactéria do gênero Ruminococcaceae foi isolada usando bacteroides fragilis como organismo auxiliar8. Mais tarde foi demonstrado que o ácido gama aminobutírico (GABA), um neurotransmissor inibidor, era necessário para o crescimento em mídia laboratorial. O uso de organismos alimentador provou ser uma estratégia fundamental para imitar microambientes específicos onde crescem bactérias não culturadas, sendo mais eficientes do que reformulando continuamente as mídias de crescimento com diferentes aditivos em concentrações variadas.
Um dos maiores grupos de bactérias não culturadas está na "Radiação Phyla Candidata" (RCP), um grupo monofilético de vários filos bacterianos candidatos9,10. A partir deste escrito, apenas membros da sacaribactéria filo dentro da RCP foram cultivados com sucesso em laboratório. O primeiro isolado,'Nanosynbacter lyticus', foi isolado usando o antibiótico estreptomicina, que havia sido previsto para enriquecer para o TM711,12. Uma descoberta fundamental deste trabalho foi que o novo isolado cresceu como um parasita crescendo em contato direto com um hospedeiro bacteriano, Schaalia odontolytica, e a microscopia mostrou que esses parasitas eram bactérias ultrasmall.
Usando essas pistas, criamos um método para estabelecer rapidamente coculturas binárias de Saccharibacteria com seus parceiros filtrando placas dentárias e outras amostras orais através de um filtro de 0,2 μm, coletando células no filtrado por centrifugação e usando-as para infectar culturas de bactérias hospedeiras candidatas. Este método tem a vantagem de evitar culturas de enriquecimento, que podem ser sobrecarregadas com organismos de rápido crescimento. Também evita o uso de antibióticos, o que poderia parar o crescimento das espécies de Sacaribacteria direcionadas ou de seus hospedeiros. Usando o método demonstrado aqui, conseguimos cultivar 32 isolados do filo saccharibacteria.
Protocolo
Ao desenvolver esse protocolo, foi solicitada e aprovada a aprovação do IRB (#14-10) e o consentimento informado foi obtido de todos os sujeitos.
1. Preparação
- Ao trabalhar com seres humanos, obtenha aprovação necessária do IRB e consentimento informado.
- Inicie culturas de bactérias hospedeiras com tempo suficiente para crescer até a fase estacionária inicial. Por exemplo, inocular 2-5 mL de caldo de soja tripptic com extrato de levedura de 0,1% adicionado (TSBY) com propionica de aracnia e incubar a 37 °C por 24 h.
- Monte os suportes do filtro com a membrana do filtro de 0,2 μm gravada. Enrole o conjunto em papel alumínio e esterilize autoclaving.
- Esterilizar tubos de centrífuga e conjuntos de tampas.
2. Obter amostra de bactérias orais
NOTA: Enquanto muitas amostras orais contêm Sacaribacterias (por exemplo, saliva, cotonetes de amígdalas, raspas de língua) a placa dentária rotineiramente é a mais bem sucedida.
- Pegue uma placa raspando usando um ponto de papel estéril, curette gracey, ou, se auto-amostragem, use um palito de dente estéril ou ponta de pipeta. Transferir placa para um buffer adequado, como diluente de recuperação máxima (MRD, 0,85% NaCl, 0,1% peptone) ou PBS. Se não for processado imediatamente, mantenha a amostra no gelo até que esteja pronta para prosseguir.
- Resuspenque vigorosamente a placa dentária no tampão MRD usando uma combinação de vórtice e pipetação com uma pequena ponta de tubulação.
- Adicione a amostra de placa resuspended a um adicional de 9 mL de buffer MRD.
3. Prepare o filtrado da amostra oral
- Usando técnica asséptica, desembrulhe um conjunto de filtro estéril. Desembrulhar uma curva de quarto e retighten o suporte do filtro para ter certeza de que os fios estão devidamente ligados e o aparelho está fechado corretamente. Usando uma seringa, lave a membrana passando 10 mL de tampão mrd através do aparelho. A montagem inadequada é revelada nesta etapa pelo fluido que vaza do suporte do filtro. Se ocorrer vazamento, obtenha outro conjunto de filtro estéril e repita a etapa de lavagem.
- Aplique a amostra no filtro lavado. Remova o êmbolo de uma seringa e fixe-o no aparelho do filtro. Despeje a amostra dentária dispersa, agora em 10 mL de tampão mrd, em uma seringa e carregue-a no filtro.
- Coloque um tubo de centrífuga estéril sob o aparelho do filtro para capturar o filtrado e, em seguida, aplique pressão leve no êmbolo para empurrar a amostra através do filtro.
- Repita o procedimento com mais 10 mL de tampão mrd para lavar a membrana, coletando o fluxo através no mesmo tubo da amostra filtrada. Asepticamente tampa o tubo.
4. Concentrar células de sacaribacteria por centrifugação
- Faça uma marca de orientação no tubo e na tampa e coloque o tubo em uma centrífuga de alta velocidade com a marca na parte superior. A pelota formada a partir da centrifugação é geralmente invisível. A marcação ajudará a determinar onde a pelota das células saccharibacteria está localizada quando a centrifugação é feita e o tubo removido da centrífuga.
- Centrifugar as amostras a 60.000 x g por 1 h a 4 °C.
NOTA: Esta força e tempo são suficientes para doar todas as células de Saccharibacteria. No entanto, as células saccharibacteria podem ser pelo menos parcialmente pelotas por centrifugação por apenas 20 min a 20.000 x g. - Remova cuidadosamente os tubos da centrífuga. Despeje o líquido do tubo, mantendo a pelota na parte superior do tubo.
- Resuspend a pelota geralmente invisível em 1-2 mL de buffer MRD por vórtice vigoroso.
5. Infectar culturas de hospedagem com filtrado enriquecido com sacaribacteria
- Prepare tubos de cultura aliquoting 2 mL de mídia de crescimento apropriada (por exemplo, TSBY, BHI, etc.) em tubos.
- Adicione 200 μL de cultura durante a noite de organismos hospedeiros a cada tubo. Adicione 100-200 μL de amostra resuspended, filtrada a cada tubo.
- Incubar amostras combinadas conforme apropriado para o organismo hospedeiro (por exemplo, 37 °C, em atmosfera aeróbica para A. propionica).
- Transfira as células a cada dois ou três dias transferindo 200 μL de cultura binária para 2 mL de meio de crescimento fresco em um novo tubo. Se parece que as culturas passagens não mostram crescimento (ou seja, a turbidez/densidade óptica da cultura não aumenta após a passagem para a mídia fresca) as Saccharibacterias podem estar sobrecarregando ou matando todo o organismo hospedeiro. Para remediar isso, adicione 200 μL de cultura de host não infectada ao passar as células. Repita por pelo menos 5 passagens.
6. Confirmar infecção por PCR
- Após 5 passagens, confirme a infecção com PCR. Cinco passagens garantirão que quaisquer células saccharibacteria não em crescimento tenham sido esgotadas além do limite de detecção usando 25 ciclos de PCR.
- Prepare um mastermix pcr. Uma receita sugerida usando é a seguinte, para cada tubo necessário, prepare 12,5 μL 2x pcr tampão, 0,75 μL 10 μM Primer para frente (580F12 - AYT GGG CGT AAA GAG TTG C), 0,75 μL 10 primer reverso μM (1177R13- GAC CTG ACA TCA TCC CCT CCT CCT TCC TCC), 1 μL 25 mM MgCl2 e 9 μL de água. Misture por vórtice.
- Aliquot 24 μL de mastermix em um tubo PCR de 0,2 mL. Adicione 1 μL de cultura infectada por sacaribacteria à reação do PCR. Coloque o tubo em um termociclador e realize o seguinte protocolo: Denaturação inicial 95 °C para 5 min, 25 ciclos de 95 °C para 30 s, 60 °C para 30 s, 72 °C para 30 s, alongamento final a 72 °C por 2 min, depois espera final em 4 °C.
- Carregue e execute os produtos PCR em um gel de 1% de agarose. Uma banda de ~600 bases indicará a presença de Saccharibacteria.
7. Verifique se há pureza e remova organismos contaminantes
- Confirme a pureza das culturas positivas ao emplacar a cocultura de Saccharibacteria em ágar nutrientes suficientes para o crescimento do organismo hospedeiro. Realize uma diluição serial de 10 vezes da cultura em tampão estéril (por exemplo, MRD ou PBS) e espalhe 100 μL em uma placa de ágar. Realizar diluições de tal forma que haverá aproximadamente 20-200 colônias crescendo na placa de ágar.
- Incubar a cultura em condições adequadas de crescimento e observar para contaminar organismos.
NOTA: Colônias de sacaribacterias axenic nunca foram observadas, por isso apenas o organismo hospedeiro é observado crescendo nessas placas. Mais de um tipo de colônia geralmente indica contaminação. Caso ocorra contaminação, a cultura deve ser refil filtrada para remover os contaminantes indesejados e re-inoculada em hospedeiro fresco. - Ocasionalmente, ocorre uma dupla infecção, onde duas espécies de Saccharibacteria infectam o mesmo hospedeiro. Para separar as espécies de Saccharibacteria, resuspenque colônias individuais em PBS estéreis de 20 μL e use 1 μL de suspensão em uma reação pcr para detectar colônias com infecções por Sacaribacteria.
- Transfira as suspensões que deram um resultado positivo ao meio de crescimento para iniciar uma cultura binária. A taxa de sucesso dependerá do título de cada espécie de Saccharibacteria na cultura. Pode ser necessário selecionar mais de 50 colônias para encontrar uma infectada para propagação.
8. Armazenamento de culturas
- Cresça uma cultura binária de volume suficiente (10-50 mL) durante a noite.
- Células de pelotização por centrifugação (4.000 x g por 10 min).
NOTA: A centrifugação de alta velocidade não é necessária aqui, pois as células saccharibacterias serão anexadas às células maiores e mais pesadas do hospedeiro e serão pelotas com elas. - Células resuspend em crioprotetor. A mídia de crescimento complementada com 5% de DMSO ou 20% de glicerol é geralmente suficiente. Certifique-se de que o organismo hospedeiro é compatível com mídia crioprotetor (por exemplo, o crescimento da propionica A. é inibido por glicerol e os estoques congelados podem não reviver). Teste a viabilidade de uma cultura hospedeira não infectada com crioprotetor para garantir que os estoques possam sobreviver ao congelamento.
- Resuspend pelotas de célula no mesmo volume de cultura (10-50 mL). Alíquota de 0,5 mL em criovias rotuladas. Congele as culturas celulares a -80 °C.
Resultados
PCR para detecção de Sacaribacteria pode parecer negativo (ou seja, nenhum produto visto) em culturas iniciais de infecção devido a um baixo número de symbionts saccharibacteria. No entanto, após algumas passagens, um produto PCR forte deve aparecer mostrando que ocorreu uma infecção estável(Figura 1A). Por outro lado, algumas infecções inicialmente parecerão positivas pelo PCR, mas diminuirão para indetectáveis após 1-4 passagens (não mostradas). Isso indica que uma grande quantidade de células saccharibacteria estavam presentes no filtrado inicial, mas foram diluídas por passagem e nenhuma foi capaz de entrar em uma simbiose estável com a cultura hospedeira. Testar várias espécies hospedeiras com o mesmo filtrado saccharibacteria da cavidade oral geralmente terá uma baixa taxa de sucesso(Figura 1B), pois a interação simbionte-hospedeiro é muito específica. Um pesquisador também pode testar o mesmo hospedeiro com filtrados de vários assuntos diferentes. Se um bom organismo hospedeiro (como a aracnia propionica ou Schaalia odontolytica) é usado, uma taxa de sucesso de 50% pode ser esperada.
O teste por PCR é crucial. Pesquisadores experientes tentaram confirmar a infecção por microscopia, apenas para relatar falsos positivos. As células saccharibacteria são pequenas e difíceis de distinguir entre vesículas ou protuberâncias irregulares de envelopes celulares. Um sinal PCR estável através de várias passagens é a chave para confirmar uma infecção bem sucedida.
À medida que as culturas infectadas crescem, o crescimento normal do turvo deve aparecer. À medida que a simbiose se estabelece, as culturas infectadas aparecerão menos turvas do que as culturas de organismos hospedeiros não infectados(Figura 2A). Em alguns casos, as culturas infectadas podem parecer parar de crescer completamente e não se tornar turva. Isso pode ser devido a uma infecção onde as células saccharibacteria estão sobrecarregando o organismo hospedeiro. Adicionar hospedeiro "fresco" (não infectado) a essas culturas deve fornecer uma população suficiente de hospedeiros para apoiar o crescimento contínuo dos parasitas saccharibacteria.
O crescimento excessivo, ou culturas excessivamente turvas, pode indicar contaminação por um contaminante de laboratório ou por outra pequena bactéria oral que foi capaz de passar pelo filtro de 0,2 μm. Campylobacter e Capnocytophaga spp. são contaminantes orais típicos desses experimentos. Isso pode ser confirmado emplacando a cultura e procurando colônias atípicas do organismo hospedeiro, seguidas pelo sequenciamento de rRNA 16S. Se a contaminação for vista, filtrar essas culturas através de um filtro de 0,2 μm geralmente é suficiente para remover os contaminantes. As células sacaribacterias no filtrado podem ser concentradas por centrifugação e usadas para rein infectar uma cultura hospedeira pura.
Outra maneira de purificar uma cultura contaminada é escolhendo colônias infectadas de uma cultura infectada. Colônias de hospedeiros infectados podem às vezes ser identificadas pela forma irregular de colônias em comparação com colônias não infectadas(Figura 2B-D). Essas colônias irregulares dependem do título de Saccharibacterias na cultura binária e uma proporção menor de colônias parecerá irregular se o titer for baixo. Isso pode facilitar a identificação de colônias infectadas, que podem ser colhidas e usadas para iniciar uma cultura binária pura. Se nenhuma colônia irregular for vista em revestimento de uma cultura infectada por Saccharibacteria, é possível que o simbionte esteja em um nível baixo ou não cause colônias de forma irregular. Nesse caso, colônias de triagem de PCR com aparência normal podem apresentar infecção por Sacaribacterias, mas a uma taxa baixa (2-10% de todas as colônias).
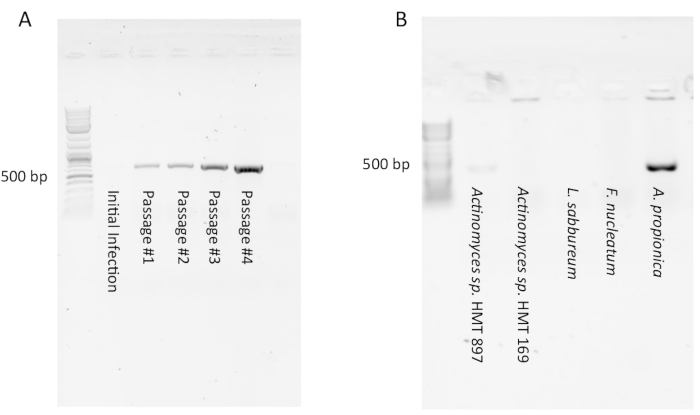
Figura 1: Resultados típicos de PCR de infecções de Saccharibacteria de culturas hospedeiras. (A) Um produto PCR indicando presença de Saccharibacteria pode não aparecer com a infecção inicial, mas pode aparecer e se tornar mais forte em passagens subsequentes à medida que a cocultura se estabelece. (B) A maioria dos hospedeiros infectados com filtrado enriquecido com sacaribacteria não suportará seu crescimento devido à especificidade da simbiose. Neste exemplo, apenas A. propionica foi infectada com sucesso. Clique aqui para ver uma versão maior desta figura.
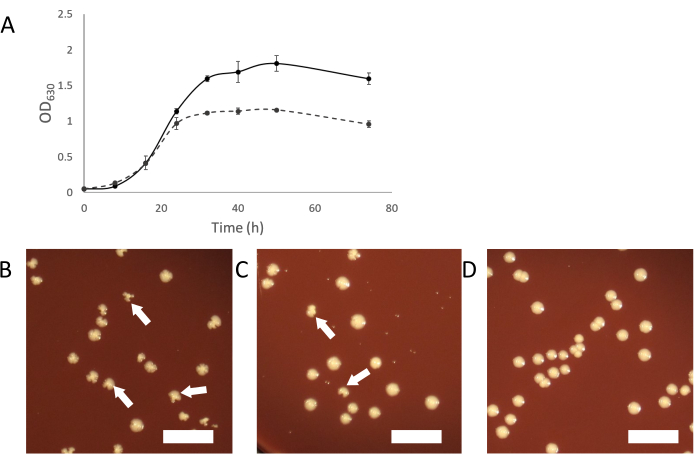
Figura 2: Características de crescimento das culturas infectadas por Sacaribacteria. (A) Curvas de crescimento de culturas infectadas e não infectadas de A. propionica mostrando cultura infectada não crescerá na mesma densidade que o controle não infectado e parecerá menos turva. (B-D) O revestimento de coculturas pode produzir colônias irregulares, causadas por infecções de Saccharibateria. Colônias irregulares diminuirão em proporção em relação ao título de Saccharibacteria na cocultura. (B) Hospedeiro com alto nível de Sacaribacteria. (C) 10 vezes a cultura de saccharibacteria diluída(D) Cultura hospedeira não infectada. Setas brancas indicam exemplos de colônias irregulares. Barra de escala= 1 cm. Clique aqui para ver uma versão maior desta figura.
Discussão
Nosso método de filtrar placas e aplicá-la a culturas puras de organismos hospedeiros é em grande parte baseado em observações anteriores sobre a primeira saccharibacteria cultivada, 'Nanosynbacter lyticus' cepa TM7x11,14,15. Dado o tamanho das células pequenas, deduzimos que elas poderiam ser separadas da placa dentária usando um filtro e concentradas com centrifugação. Em segundo lugar, como esses organismos vivem como parasitas, fornecer a essas células culturas puras de hospedeiros permitiria que eles entrassem em uma simbiose e crescessem como culturas binárias.
Uma vantagem deste método é que ele não requer uma cultura de enriquecimento ou pressão seletiva. 'Nanosynbacter lyticus' cepa TM7x foi cultivado a partir de uma cultura de enriquecimento usando estreptomicina como um agente seletivo, que sequenciamento tinha sugerido seria eficaz no enriquecimento para sacaribacteria. Fortuitamente, o hospedeiro de 'Nanosynbacter lyticus' Schaalia odontolytica, é conhecido por ser resistente à estreptomicina16. O uso de antibióticos como agente seletivo também poderia impedir que o organismo hospedeiro crescesse, o que, por sua vez, impediria o crescimento de Sacaribacterias.
Uma questão maior de usar culturas de enriquecimento é que organismos de rápido crescimento irão rapidamente deslocar organismos de interesse. Na cavidade oral, por exemplo, as espécies de Estreptococos podem crescer rapidamente e, se o açúcar estiver presente no meio de crescimento, produzem ácido suficiente para acidificar o meio, selecionando ainda mais contra organismos de interesse. Ao evitar uma cultura de enriquecimento e antibióticos seletivos, nosso método fornece uma abordagem geral que poderia ser aplicada a uma gama mais ampla de Saccharibacterias e potenciais hospedeiros sem as complicações desses outros métodos.
Há alguns obstáculos ao método aqui apresentado. Primeiro, este método pressupõe que saccharibacterias vivem em uma cultura binária. Não testamos combinações de culturas trinárias ou ternárias para medir sua eficácia, mas há provavelmente saccharibacterias que requerem fatores de crescimento que um único organismo hospedeiro não pode fornecer. Testar as vastas combinações de bactérias orais que poderiam suportar o crescimento de Sacaribacterias seria uma tarefa assustadora. Em segundo lugar, o método pressupõe que todas as Saccharibacterias são pequenas o suficiente para passar por um filtro de 0,2 μm. Pode ser que outras Sacaribacterias sejam maiores do que se acreditava e o filtro esteja selecionando contra esses organismos. Um filtro com um tamanho maior de poros poderia ser usado, mas isso corre o risco de permitir mais bactérias orais indesejadas na cocultura infectada. Por fim, é muito difícil encontrar espécies hospedeiras fora daquelas que já foram publicadas. Até agora, os únicos hospedeiros bem sucedidos são espécies dos gêneros Actinomyces, Schaalia, Aracnia e Celulosimicrobium, todos membros da actinobactériasphylum 15,17,18. No entanto, esses hosts só suportam o crescimento de saccharibacterias específicas. Para cultivar mais espécies de Saccharibacteria, muitos mais hospedeiros devem ser explorados.
Esperamos que o método aqui apresentado ajude futuras pesquisas das sacariactérias e outros organismos de RCP. O sequenciamento metagenômico sugere que esses organismos também possuem genomas pequenos e são suspeitos de serem simmbiontos ou dependem da comunidade microbiana local para fornecer metabólitos e outros fatores críticos à sua sobrevivência19. Uma estratégia de filtragem semelhante poderia ser usada para isolar esses organismos, desde que sejam pequenos o suficiente e que seus organismos hospedeiros possam ser cultivados. Os métodos descritos aqui são um primeiro passo para trazer as poderosas ferramentas da cultura laboratorial para este grande e diversificado grupo de bactérias.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores desejam agradecer a Anne Tanner, Bruce Paster, Heike Boisvert, Xuesong He e Batbileg Bor por discussões úteis e por fornecer cepas bacterianas. Agradecemos a Susan Yost e Jessica Woods pela assistência técnica microbiana. A pesquisa relatada nesta publicação contou com o apoio do Instituto Nacional de Pesquisas Odontológicas e Craniofaciais dos Institutos Nacionais de Saúde sob os números de premiação R37 DE016937 (FED), R01 DE024468 (FED) e T32 DE007327 (AJC). O conteúdo é de responsabilidade exclusiva dos autores e não representa necessariamente as opiniões oficiais dos Institutos Nacionais de Saúde.
Materiais
| Name | Company | Catalog Number | Comments |
| Agarose | Fisher Scientific | BP160-100 | |
| Alphaimager | Cell Biosciences | FluorChem HD2 | Or equivalent UV gel imaging system |
| Aluminum foil | Fisher Scientific | 01-213-101 | |
| Brain Heart Infusion Broth (dehydrated powder) | Becton-Dickinson | 211059 | Or other growth media suitable for target organisms |
| Centrifuge Rotor 70-Ti | Beckman Coulter | 337922 | |
| Cryovials | Fisher Scientific | 12-567-500 | |
| DMSO | Fisher Scientific | BP231-100 | |
| Electrophoresis Power Supply | Bio-Rad | 1645052 | |
| Electrophoresis Rig | Bio-Rad | 1704467 | |
| Filter Forceps | Millipore Sigma | XX6200006P | Not essential, helps ensure filters are not punctured during handling |
| Glycerol | Fisher Scientific | G33-500 | |
| GoTaq Green Mastermix | Promega | M7122 | |
| Mastercycler Pro Thermocycler | Eppendorf | 950040025 | Or equivalent thermocycler for PCR |
| MgCl2 solution 25mM | Promega | A3513 | |
| Molecular Biology grade water | Fisher Scientific | BP2819100 | |
| O2 Control InVitro Glove Box | Coy Laoratories | 031615 | If needed for microaerobic organisms |
| Optima L-100 XP High Speed Centrifuge | Beckman Coulter | 8043-30-1124 | |
| P-10 micro pipette | Gilson | F144802 | |
| P-1000 micro pipette | Gilson | F123601G | |
| P-2 micro pipette | Gilson | F144801 | |
| P-20 micro pipette | Gilson | F123600 | |
| P-200 micro pipette | Gilson | F123602G | |
| PBS | Fisher Scientific | BP399500 | |
| PCR tubes 0.2 mL | Fisher Scientific | 14-230-205 | |
| Peptone | Fisher Scientific | BP1420-500 | |
| Pipette tips - 10 μL | Fisher Scientific | 02-717-157 | |
| Pipette tips - 1000 μL | Fisher Scientific | 02-717-166 | |
| Pipette tips - 20 μL | Fisher Scientific | 02-717-161 | |
| Pipette tips - 200 μL | Fisher Scientific | 02-717-165 | |
| Polycarbonate filters - 47mm, 0.2 μm pore size | Millipore | GTTP04700 | |
| Screw-cap conical centrifuge tubes 15 mL | Falcon | 352096 | Or other tube suitable for bacterial culture |
| Sodium chloride | Fisher Scientific | BP358-1 | |
| Swin-Lok Filter - 47mm | Whatman | 4200400 | |
| SYBR Safe DNA Gel stain | ThermoFisher Scientific | S33102 | |
| Syringes - 20 mL | Fisher Scientific | 14-955-460 | |
| TAE Buffer (50x) concentrate | Fisher Scientific | P1332500 | |
| Thickwall Polycarbonate 25 x 89 mm (26.3mL capacity) centrifuge tubes with caps | Beckman Coulter | 355618 | |
| Tryptic Soy Blood Agar Plates | Northeast Laboratory Services | P1100 | Or other agar plate sufficient for growth of host organisms |
| Tryptic Soy Broth (dehydrated powder) | Becton-Dickinson | 211825 | Or other growth media suitable for target organisms |
| Vinyl Anaerobic Chamber | Coy Laboratories | 032714 | If needed for anaerobic organisms |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Yeast Extract | Fisher Scientific | BP1422-500 |
Referências
- Stewart, E. J. Growing unculturable bacteria. Journal of Bacteriology. 194 (16), 4151-4160 (2012).
- Bomar, L., Maltz, M., Colston, S., Graf, J. Directed culturing of microorganisms using metatranscriptomics. mBio. 2 (2), 00012 (2011).
- Derrien, M., Vaughan, E. E., Plugge, C. M., de Vos, W. M. Akkermansia muciniphila gen. nov., sp. nov., a human intestinal mucin-degrading bacterium. International Journal of Systematic and Evolutionary Microbiology. 54, 1469-1476 (2004).
- Nelson, M. C., Bomar, L., Maltz, M., Graf, J. Mucinivorans hirudinis gen. nov., sp. nov., an anaerobic, mucin-degrading bacterium isolated from the digestive tract of the medicinal leech Hirudo verbana. International Journal of Systematic and Evolutionary Microbiology. 65, 990-995 (2015).
- Scott, J. C., Klein, B. A., Duran-Pinedo, A., Hu, L., Duncan, M. J. A two-component system regulates hemin acquisition in porphyromonas gingivalis. PLoS One. 8 (9), 0073351 (2013).
- Martin, B., et al. New growth media for oral bacteria. Journal of Microbiological Methods. 153 (22), 10-13 (2018).
- Vartoukian, S. R., et al. In vitro cultivation of 'unculturable' oral bacteria, facilitated by community culture and media supplementation with siderophores. Plos One. 11 (1), 0146926 (2016).
- Strandwitz, P., et al. GABA-modulating bacteria of the human gut microbiota. Nature Microbiology. 4 (3), 396-403 (2019).
- Hug, L. A., et al. A new view of the tree of life. Nature Microbiology. 1, 16048 (2016).
- Castelle, C. J., Banfield, J. F. Major new microbial groups expand diversity and alter our understanding of the tree of life. Cell. 172 (6), 1181-1197 (2018).
- He, X., et al. Cultivation of a human-associated TM7 phylotype reveals a reduced genome and epibiotic parasitic lifestyle. Proceedings of the National Academy of Sciences of the United States of America. 112 (1), 244-249 (2015).
- Hugenholtz, P., Tyson, G., Webb, R. I., Wagner, A. M., Blackall, L. L. Investigation of candidate division TM7, a recently recognized major lineage of the domain Bacteria with no known pure-culture representatives. Applied and Environmental Microbiology. 67 (1), 411-419 (2001).
- Brinig, M., Lepp, P. Prevalence of bacteria of division TM7 in human subgingival plaque and their association with disease. Applied and Environmental Microbiology. 69 (3), 1687-1694 (2003).
- Bor, B., et al. Insights obtained by culturing saccharibacteria with their bacterial hosts. Journal of Dental Research. , 689-694 (2020).
- Bor, B., et al. Phenotypic and physiological characterization of the epibiotic interaction between TM7x and its basibiont actinomyces. Microbial Ecology. 71 (1), 243-255 (2016).
- Skopek, R. J., Liljemark, W. F., Bloomquist, C. G., Rudney, J. D. Dental plaque development on defined streptococcal surfaces. Oral Microbiology and Immunology. 8 (1), 16-23 (1993).
- Murugkar, P. P., Collins, A. J., Chen, T., Dewhirst, F. E. Isolation and cultivation of candidate phyla radiation Saccharibacteria (TM7) bacteria in coculture with bacterial hosts. Journal of Oral Microbiology. 12 (1), 1814666 (2020).
- Cross, K. L., et al. Targeted isolation and cultivation of uncultivated bacteria by reverse genomics. Nature Biotechnology. 37 (11), 1314-1321 (2019).
- Kantor, R., et al. Small genomes and sparse metabolisms of sediment-associated bacteria from four candidate phyla. MBio. 4 (5), 00708-00713 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados