É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Sondagem do metabolismo e viscosidade de células cancerígenas usando microscopia de imagem de fluorescência ao longo da vida
Neste Artigo
Resumo
Aqui, demonstramos o uso da microscopia de imagem de fluorescência (FLIM) para obter imagens sequenciais do metabolismo celular e da viscosidade da membrana plasmática em cultura de células cancerígenas vivas. As avaliações metabólicas são realizadas pela detecção de fluorescência endógena. A viscosidade é medida usando um rotor molecular fluorescente.
Resumo
A viscosidade é uma propriedade física importante de uma membrana biológica, pois é um dos parâmetros-chave para a regulação do estado morfológico e fisiológico das células vivas. Sabe-se que as membranas plasmáticas das células tumorais apresentam alterações significativas em sua composição, estrutura e características funcionais. Juntamente com o metabolismo desregulado da glicose e dos lipídios, essas propriedades específicas da membrana ajudam as células tumorais a se adaptarem ao microambiente hostil e a desenvolverem resistência às terapias medicamentosas. Aqui, demonstramos o uso da microscopia de imagem de fluorescência (FLIM) para obter imagens sequenciais do metabolismo celular e da viscosidade da membrana plasmática em cultura de células cancerígenas vivas. As avaliações metabólicas são realizadas pela detecção de fluorescência de cofatores metabólicos endógenos, como redução do dinucleotídeo de nicotinamida adenina NAD(P)H e flavinas oxidadas. A viscosidade é medida usando um rotor molecular fluorescente, um corante sintético sensível à viscosidade, com uma forte dependência do tempo de vida de fluorescência da viscosidade do ambiente imediato. Em combinação, essas técnicas nos permitem entender melhor as ligações entre o estado da membrana e o perfil metabólico das células cancerígenas e visualizar as alterações induzidas pela quimioterapia.
Introdução
A transformação maligna das células é acompanhada por múltiplas alterações em seu estado morfológico e fisiológico. O crescimento rápido e descontrolado das células cancerígenas requer uma reorganização fundamental das vias bioquímicas responsáveis pela produção de energia e biossíntese. As características do metabolismo do câncer são o aumento da taxa de glicólise, mesmo sob as concentrações normais de oxigênio (efeito Warburg), o uso de aminoácidos, ácidos graxos e lactato como combustíveis alternativos, alta produção de ROS na presença de altos níveis de antioxidantes e aumento da biossíntese de ácidos graxos 1,2. Agora é reconhecido que o metabolismo das células cancerígenas é altamente flexível, o que lhes permite adaptar-se ao ambiente desfavorável e heterogêneo e fornece uma vantagem adicional de sobrevivência3.
O metabolismo alterado suporta a organização e composição específicas das membranas das células tumorais. O perfil lipídico da membrana plasmática nas células cancerígenas difere quantitativamente das células não cancerosas. As principais alterações no lipidoma são o aumento do nível de fosfolipídios, incluindo fosfatidilinositol, fosfatidilserina, fosfatidiletanolamina e fosfatidilcolina, a diminuição do nível de esfingemielina, aumento da quantidade de colesterol e menor grau de insaturação de ácidos graxos, para citar alguns 4,5,6. Portanto, as propriedades físicas da membrana, como a viscosidade da membrana, o inverso da fluidez, inevitavelmente mudam. Como a viscosidade determina a permeabilidade das membranas biológicas e controla a atividade das proteínas associadas à membrana (enzimas, transportadores, receptores), sua regulação homeostática é vital para o funcionamento celular. Ao mesmo tempo, a modificação da viscosidade através do ajuste do perfil lipídico da membrana é importante para a migração/invasão celular e sobrevivência após alterações condicionais.
A microscopia de imagem de fluorescência ao longo da vida (FLIM) surgiu como uma abordagem poderosa para a avaliação não invasiva de múltiplos parâmetros em células vivas, usando fluorescência endógena ou sondas exógenas7. O FLIM é comumente realizado em um microscópio de varredura a laser multifotônico, que fornece resolução (sub)celular. Sendo equipado com o módulo de contagem de fótons únicos correlacionados ao tempo (TCSPC), ele permite medições de fluorescência resolvidas no tempo com alta precisão8.
A sondagem do metabolismo celular por FLIM baseia-se na medição de fluorescência de cofatores endógenos de desidrogenase, o dinucleotídeo (fosfato) de nicotinamida adenina reduzido NAD(P)H e flavinas oxidadas - dinucleotídeo de flavina adenina FAD e mononucleotídeo de flavina FMN, que atuam como carreadores de elétrons em várias reações bioquímicas 7,9,10. A fluorescência detectada de NAD(P)H é de NADH e sua forma fosforilada, NADPH, pois são espectralmente quase idênticas. Normalmente, os decaimentos de fluorescência de NAD (P) H e flavinas se ajustam a uma função bi-exponencial. No caso do NAD (P) H, o primeiro componente (~ 0,3-0,5 ns, ~ 70% -80%) é atribuído ao seu estado livre, associado à glicólise, e o segundo componente (~ 1,2-2,5 ns, ~ 20% -30%) ao seu estado ligado à proteína, associado à respiração mitocondrial. No caso das flavinas, o componente curto (~ 0,3-0,4 ns, ~ 75% -85%) pode ser atribuído ao estado temperado de FAD e o componente longo (~ 2,5-2,8 ns, ~ 15% -25%) a FAD não extinto, FMN e riboflavina. Alterações nos níveis relativos de glicólise, glutaminólise, fosforilação oxidativa e síntese de ácidos graxos resultam em alterações nas frações de vida curta e longa dos cofatores. Além disso, a razão de intensidade de fluorescência desses fluoróforos (a razão redox) reflete o status redox celular e também é usada como um indicador metabólico. Embora a razão redox apresente uma métrica mais simples, em comparação com o tempo de vida da fluorescência, em termos de aquisição de dados, o FLIM é vantajoso para estimar NAD(P)H e FAD, pois o tempo de vida da fluorescência é uma característica intrínseca do fluoróforo e quase não é influenciado por fatores como poder de excitação, fotobranqueamento, focalização, espalhamento e absorção de luz, especialmente em tecidos, ao contrário da intensidade de emissão.
Uma das maneiras convenientes de mapear a viscosidade em células e tecidos vivos no nível microscópico é baseada no uso de rotores moleculares fluorescentes, pequenos corantes sintéticos sensíveis à viscosidade, nos quais os parâmetros de fluorescência dependem fortemente da viscosidade local11,12. Em um meio viscoso, a vida útil da fluorescência do rotor aumenta devido à desaceleração da torção ou rotação intramolecular. Entre os rotores moleculares, os derivados do dipirrometeno de boro (BODIPY) são adequados para detectar a viscosidade em sistemas biológicos, pois possuem uma boa faixa dinâmica de tempos de vida de fluorescência na faixa fisiológica de viscosidades, independência de temperatura, decaimentos de fluorescência monoexponenciais que permitem interpretação direta dos dados, solubilidade em água suficiente e baixa citotoxicidade13,14. Avaliações quantitativas de microviscosidade usando rotores baseados em BODIPY e FLIM foram demonstradas anteriormente em células cancerígenas in vitro, esferóides tumorais multicelulares e tumor de camundongo in vivo15,16.
Aqui, apresentamos uma descrição detalhada das metodologias de sondagem sequencial para estudar o metabolismo celular e a viscosidade da membrana plasmática em células cancerígenas in vitro por FLIM. Para evitar a contaminação da fluorescência endógena relativamente fraca com a fluorescência do rotor baseado em BODIPY, a imagem da mesma camada de células é realizada sequencialmente com a fluorescência de NAD (P) H e FAD fotografada primeiro. O tempo de vida de fluorescência dos cofatores é medido no citoplasma, e o tempo de vida de fluorescência do rotor é medido nas membranas plasmáticas das células pela seleção manual das zonas correspondentes como regiões de interesse. O protocolo foi aplicado para correlacionar o estado metabólico e a viscosidade para diferentes linhagens de células cancerígenas e avaliar as alterações após a quimioterapia.
O protocolo para preparação de amostras FLIM não difere daquele para microscopia de fluorescência confocal. Uma vez que os dados tenham sido adquiridos, a principal tarefa é extrair o tempo de vida da fluorescência dos dados brutos. O desempenho do protocolo é demonstrado usando células HCT116 (carcinoma colorretal humano), CT26 (carcinoma de cólon murino), HeLa (carcinoma cervical humano) e huFB (fibroblastos da pele humana).
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Descrição da configuração mínima para executar o FLIM
- Para realizar este experimento, certifique-se de que a configuração necessária esteja disponível: um microscópio confocal invertido, um laser pulsado, normalmente um ps ou fs, com o sinal de sincronização, um detector de contagem rápida de fótons (resposta de tempo 150 ps) e eletrônica de contagem de fótons, portas de saída e entrada disponíveis para o detector e o laser, respectivamente, no microscópio, os pulsos do relógio de varredura do controlador de varredura do microscópio, a cabeça de varredura do microscópio com os combinadores de feixe de laser e os divisores de feixe dicróico principais adequados ao comprimento de onda do laser usado para FLIM.
- Se a excitação de dois fótons for usada para FLIM, certifique-se de que o microscópio contenha a porta NDD.
- Para estudos com células de mamíferos, especialmente para experimentos de longo prazo, certifique-se de ter uma incubadora de CO2 mantida na temperatura desejada.
NOTA: Para o sistema usado neste experimento, consulte a Tabela de Materiais.
2. Preparação de células para microscopia
- Cultive as células rotineiramente em uma incubadora a 37 ° C com 5% de CO2 e uma atmosfera úmida.
- Para microscopia, preparar a suspensão celular num meio de cultura completo na concentração de 1 x 106 células/ml.
NOTA: As concentrações celulares e as condições do meio dependem da célula. O número de células utilizadas para a semeadura e o tempo de incubação devem ser adaptados para obter 70%-80% de confluência na placa microscópica. - Semear as células em placas de cultura de células de 35 mm com fundo de vidro (1 x 105 células em 100 μL por placa) usando uma pipeta automática de 200 μL.
NOTA: Use placas com fundo de vidro quadriculado para a semeadura de células para monitorar células nos mesmos campos microscópicos de view em dinâmica.- Durante a semeadura manual, certifique-se de que a ponta da pipeta não risque o fundo ou as laterais do prato para evitar danificar o fundo.
- Colocar a placa na incubadora de CO2 (37 °C, 5% de CO2, atmosfera húmida) e incubar as células durante 24 h.
- Após 24 h, retirar a placa da incubadora e verificar a morfologia e a confluência das células ao microscópio óptico. Se as células não atingirem cerca de 80% de confluência, incubar por mais 24 horas.
- Remova suavemente o meio antigo do prato usando uma pipeta automática de 1.000 μL e adicione 2 mL de meio DMEM sem vermelho de fenol (por exemplo, DMEM Life ou FluoroBrite).
NOTA: Diferentes meios de cultura podem ser usados para geração de imagens. Evite o vermelho de fenol no meio ao usar células para microscopia. - Coloque o prato na incubadora por 60-120 min para permitir a adaptação das células.
3. FLIM de cofatores metabólicos
- Coloque o prato com fundo de vidro com as células (da etapa 2.7) no microscópio stage.
- Clique na guia Localizar no software do microscópio de varredura a laser (por exemplo, ZEN - ZEISS Efficient Navigation) e, em seguida, clique em Luz de transmissão (TL) para acender a luz.
- Encontre o plano focal da amostra visualizando através da ocular no nível da fatia central das células, onde um quadrado é ocupado ao máximo por células (com ampliação de 40x).
- Clique no botão OFF para desligar a luz.
- Abra a guia Aquisição . Para obter imagens de intensidade de transmissão e autofluorescência de NAD(P)H endógeno, insira as seguintes configurações: Comprimento de onda de excitação: modo de dois fótons 750 nm, Faixa de registro: 450-490 nm, Potência do laser: 5% (~6 mW), Tamanho da imagem: 1024 x 1024 pixels.
NOTA: A escolha do comprimento de onda de excitação e da faixa de registro é baseada nas características espectrais (excitação máxima e emissão máxima) do NAD(P)H17.- Use uma lente objetiva de imersão em óleo С Plan-Apochromat 40x/1.3 NA para a aquisição da imagem.
- Pressione o botão Snap e salve a imagem no formato ZEN.
- Para obter as imagens de intensidade de transmissão e autofluorescência do FAD, altere o comprimento de onda de excitação para 900 nm. Defina o intervalo de registro: 500-550 nm, potência do laser: 9% (~6 mW) e tamanho da imagem: 1024 x 1024 pixels.
NOTA: A escolha do comprimento de onda de excitação e da faixa de registro é baseada nas características espectrais (excitação máxima e emissão máxima) do FAD18.- Use uma lente objetiva de imersão em óleo С Plan-Apochromat 40x/1.3 NA para aquisição de imagem.
- Pressione o botão Snap e salve a imagem no formato ZEN.
- Para NAD(P)H, defina os parâmetros conforme descrito na etapa 3.5 no software do microscópio de varredura a laser. Altere o tamanho da imagem para 256 x 256 pixels.
- Insira os seguintes parâmetros no menu do SPCM (Single Photon Counting Modules) Software operacional do módulo FLIM: Tempo de coleta: 60 s; Faixa TAC: 5.00E-8; Limite de CFD Baixo: -29,41; Resolução ADC: 256, Tamanho da imagem: 256 x 256 pixels.
- Digitalize a amostra por 60 s, pare a varredura e salve a imagem FLIM obtida de NAD(P)H.
- Verifique os dados FLIM obtidos. Para isso, Abra os dados brutos no software de imagem, selecione um pixel no citoplasma da célula colocando o cursor sobre ele e analise o decaimento da fluorescência neste pixel. As intensidades de pixel devem ser ≥ 3.000 fótons por curva de decaimento no compartimento 1.
NOTA: Se o número de fótons estiver abaixo de 3.000, aumente a potência do laser ou o tempo de coleta de imagens, enquanto controla a morfologia das células e a taxa de contagem de fótons. Normalmente, se a queda na taxa de contagem exceder 10% do valor inicial, ocorre o fotobranqueamento. - Para FAD, defina os parâmetros conforme descrito na etapa 3.7 no software do microscópio de varredura a laser. Altere o tamanho da imagem para 256 x 256 pixels.
- Insira os seguintes parâmetros no menu do SPCM (Single Photon Counting Modules) Software operacional do módulo FLIM: Tempo de coleta: 60 s; Faixa TAC: 5.00E-8; Limite de CFD Baixo: -29,41; Resolução ADC: 256, Tamanho da imagem: 256 x 256 pixels.
- Escaneie a amostra por 60 s. Pare a digitalização e salve a imagem FLIM obtida do FAD.
NOTA: Os parâmetros indicados nas etapas 3.10 e 3.15 são específicos para a eletrônica e o detector usado. - Verifique os dados obtidos conforme descrito na etapa 3.12.
- Repita as etapas 3.5 a 3.16 para gravar imagens FLIM de diferentes campos de view.
4. Coloração de células com o rotor molecular fluorescente
NOTA: As células são visualizadas na solução fluorescente do rotor molecular sem lavagem à temperatura ambiente (~ 20 ° C) para retardar a internalização do rotor. A viscosidade da membrana depende da temperatura, como demonstrado em nossos trabalhos anteriores 19,20. O estágio de temperatura controlada do microscópio deve ser desligado com antecedência, ou seja, antes que o rotor seja adicionado às células. Para nossa configuração, o resfriamento do palco leva cerca de 10 minutos.
- Preparar uma solução geral do rotor molecular fluorescente BODIPY 2 (Stock 1, 25,7 mM).
- Abra o BODIPY 2 em um ambiente estéril e pese aproximadamente 2 mg, usando uma balança precisa. Coloque-o cuidadosamente em um tubo de microcentrífuga.
- Use uma pipeta automática de 20 μL para adicionar 3 μL de um solvente adequado (por exemplo, DMSO).
- Assim que a amostra se dissolver completamente em DMSO, adicione 297 μL de PBS estéril e misture bem usando uma pipeta automática de 200 μL.
NOTA: Armazene a solução estoque na geladeira a +4 °C em embalagem escura. Uma vez ressuspenso, pode ser armazenado na geladeira por vários meses.
- Prepare um estoque 2 (8,9 mM) adicionando 25 μL da solução estoque geral (estoque 1) a um tubo de microcentrífuga, seguido por 48 μL de PBS estéril. Misturar delicadamente utilizando uma pipeta automática de 200 μL.
NOTA: Use o estoque 2 para preparar o estoque de coloração final, que é aplicado para coloração celular, pois a concentração micromolar é necessária. - Substitua suavemente os meios de cultura na placa (a partir do passo 3.1) por uma solução de Hank gelada sem Ca2+/Mg2+ e incube as células a +4 °C durante 3 min.
NOTA: O uso de solução gelada e incubação a +4 °C retarda a internalização do rotor molecular e a coloração local da membrana persiste por 20-30 min. - Prepare a solução de coloração final contendo 4,5 μM de BODIPY 2 adicionando 1 μL da solução de Hank gelada Stock 2 a 999 μL gelada ou PBS.
NOTA: A concentração de BODIPY 2 na solução de coloração final pode ser aumentada para ~ 10 μM sem nenhum efeito tóxico nas células, o que resulta em uma coloração mais eficiente e maior número de fótons coletados. Em concentrações mais altas, pode ocorrer sobrecarga do detector FLIM. - Aspirar a solução de Hank da placa de cultura de células e substituí-la por uma solução gelada de 4,5 μM de BODIPY 2. As células são visualizadas em solução BODIPY 2 sem lavagem.
5. FLIM do rotor molecular fluorescente nas células
NOTA: Sempre execute FLIM do rotor molecular fluorescente após FLIM de cofatores metabólicos porque o espectro de fluorescência de BODIPY 2 é sobreposto à emissão de cofatores endógenos NAD(Р)H e FAD 12,17,18.
- Transfira o prato com as células coradas para o estágio de microscópio (~ 20 ° C) para obter imagens.
- Defina os seguintes parâmetros para o modo de um fóton no software do microscópio de varredura a laser: Excitação no comprimento de onda de 488 nm com um laser de íons de argônio, potência do laser 1% -2%, faixa de registro de comprimento de onda de 500-550 nm.
- Use uma lente objetiva de imersão em óleo С Plan-Apochromat 40x/1.3 NA para aquisição de imagem.
- Pressione o botão Ao vivo . Comece a digitalizar e usar o posicionamento XY e Z por um estágio motorizado integrado, ajuste o foco e obtenha imagens de transmissão e intensidade de fluorescência das células em uma janela de visualização. Salve as imagens obtidas, se necessário.
- Verifique a transmissão sobreposta e a imagem de fluorescência para ver se a fluorescência do rotor está vindo do local esperado (membrana plasmática da célula).
- Insira os seguintes parâmetros no menu do software SPCM do módulo FLIM: Tempo de coleta: 60 s; Faixa TAC: 5.00E-8; Limite de CFD Baixo: -29,41; Resolução ADC: 256, Tamanho da imagem: 256 x 256 pixels.
NOTA: Dependendo da configuração do sistema e dos detectores usados para FLIM, os parâmetros de aquisição de imagem podem variar. - Ajuste o laser Ti:Sapphire do microscópio para um comprimento de onda de 850 nm e a potência do laser para 1%-2% para FLIM de dois fótons.
- Selecione a guia Contínuo no software do microscópio de varredura a laser e pressione Iniciar no software SPCM. Digitalize a amostra por 60 s, pare a varredura e salve a imagem FLIM obtida.
- Verifique os dados FLIM obtidos. Para isso, carregue os dados brutos no software SPCImage de análise de dados FLIM, selecione um pixel na membrana da célula colocando o cursor sobre ele e analise o decaimento de fluorescência neste pixel. As intensidades de pixel devem ser de ≥5.000 por decaimento (possivelmente incluindo binning) em um tempo de coleta razoável (60-120 s).
- Repita as etapas 5.4 a 5.8 para gravar imagens FLIM de células de diferentes campos de visão.
NOTA: As medições FLIM de células vivas coradas com BODIPY 2 devem ser limitadas a ~ 30 min após a adição de BODIPY 2.
6. Análise dos dados
- Análise da intensidade de fluorescência: razão redox
- Imagens abertas da intensidade de fluorescência de NAD(P)H e FAD usando ImageJ.
- Realce uma área livre de células na imagem NAD(P)H usando uma opção de círculo ou quadrado. Clique em Medir e, em seguida, clique em Subtrair (selecione Processar no painel principal e, em seguida, Matemática e Subtrair) para subtrair o valor obtido do sinal de fundo.
- Repita a etapa 6.1.2 para a imagem FAD.
- Obter a imagem do rácio redox dividindo a intensidade de fluorescência do DCP pela intensidade de fluorescência do NAD(P)H. Faça isso selecionando Processar no painel principal e, em seguida, selecione Calculadora de imagens e dividir; marque a caixa Criar nova janela e pressione OK.
- Salve a imagem no formato TIFF.
- Para calcular a razão redox, selecione a região do citoplasma na célula específica na imagem TIFF e pressione a tecla M . Repita para todas as células de interesse.
- Importe a medida para um documento de planilha.
NOTA: Alternativamente, as intensidades de fluorescência de NAD(P)H e FAD nas células podem ser medidas usando o software padrão do microscópio e a razão redox pode ser obtida dividindo esses valores no software da planilha.
- Análise de dados FLIM: metabolismo
- Importe a imagem FLIM do NAD(P)H para o software SPCImage.
- Aplique um ajuste de decaimento bi-exponencial à imagem colocando 2 na seção Componentes .
- Corrija o parâmetro Offset marcando a caixa correspondente no software SPCImage.
- Vá para Opções e selecione Modelo. Use o modelo de ajuste multiexponencial incompleto e o método de ajuste MLE.
- Ajuste o binning para atingir intensidades de pixel de ≥5000 fótons por curva de decaimento.
- Verifique o valor χ2. O χ2≤ 1,20 indica que o modelo usado fornece um ajuste razoável.
- Calcule o histograma da vida útil da fluorescência em cada imagem clicando no menu superior Calcular e, em seguida, em Matriz de decaimento.
- Selecione a área no citoplasma da célula específica como uma região de interesse.
- Analise os componentes de vida curta e longa (τ1 e τ2, respectivamente) e as amplitudes relativas dos componentes de vida útil (a1 e a2, onde a1 + a2 = 100%) usando a opção Cor .
- Exporte as medições para um software de planilha.
- Repita as etapas 6.2.8 a 6.2.10 para cada célula de interesse.
- Repita as etapas 6.2.1-6.2.11 para a imagem FAD.
- Análise de dados FLIM: viscosidade
- Importe a imagem FLIM para o software SPCImage de análise de dados FLIM.
- Remova a marca na caixa Dispersão .
- Coloque 1 na seção Componentes , pois o decaimento da fluorescência do rotor deve se ajustar a um modelo monoexponencial.
- Ajuste o binning para atingir uma intensidade de pixel de ≥5000 fótons por curva de decaimento.
- Verifique o valor χ2 na membrana plasmática. Um valor de χ2≤ 1,20 indica que o modelo usado fornece um ajuste razoável. No caso de χ2≥ 1,20, a aproximação monoexponencial não é aplicável, tais dados podem indicar a agregação do corante e devem ser descartados. A agregação impossibilita o uso das curvas de calibração e leva a estimativas incorretas de viscosidade.
NOTA: Decaimentos bi-exponenciais podem ser indicativos de agregação. Em um microscópio com módulo FLIM com filtros variáveis, isso pode ser detectado testando as faixas de comprimento de onda de emissão específicas de monômeros e agregados, 500-550 nm e 580-650 nm, conforme descrito na referência21. - Gere o histograma do tempo de vida da fluorescência τ para cada imagem clicando no menu superior Calcular e, em seguida, em Matriz de decaimento.
- Selecione a região da membrana plasmática da célula individual com decaimento monoexponencial, χ2≤ 1,20, usando a opção ROI.
- Exporte o valor da vida útil da fluorescência para um software de planilha.
- Repita as etapas 6.3.7-6.3.8 para cada célula de interesse.
- Converter o tempo de vida de BODIPY 2 medido experimentalmente (em ns) em valores de viscosidade (em cP) utilizando a seguinte equação (previamente obtida com base no gráfico de calibração de BODIPY em misturas de metanol/glicerol):
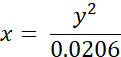
onde x - viscosidade (em cP), y - tempo de vida de fluorescência τ (em ns).
NOTA: IRF (Instrument Response Function) é uma parte importante do ajuste FLIM. No SPCImage, o IRF é calculado automaticamente a partir da borda ascendente das curvas de decaimento de fluorescência. Enquanto isso, o IRF pode ser registrado usando amostras não fluorescentes, por exemplo, cerâmicas ou uma amostra que produz sinal SHG (Second Harmonic Generation), por exemplo, colágeno, cristais de uréia ou açúcar. O uso dos IRFs gravados não é recomendado se houver uma opção para calculá-lo no software.
Access restricted. Please log in or start a trial to view this content.
Resultados
Usando o protocolo descrito aqui, visualizamos os cofatores metabólicos e a viscosidade da membrana microscópica em células vivas cultivadas usando FLIM. As medições foram feitas em diferentes linhagens de células cancerígenas - carcinoma colorretal humano HCT116, carcinoma de cólon murino CT26, câncer cervical humano HeLa Kyoto e fibroblastos de pele humana huFB.
A razão redox baseada...
Access restricted. Please log in or start a trial to view this content.
Discussão
Este protocolo ilustra as possibilidades do FLIM para análise multiparamétrica, funcional e biofísica de células cancerígenas. A combinação da imagem metabólica óptica baseada em fluorescência endógena e as medidas de viscosidade da membrana plasmática usando marcação exógena com rotor molecular fluorescente nos permite caracterizar as interconexões entre esses dois parâmetros em células cancerígenas vivas em uma cultura de células e acompanhar as mudanças em respost...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
O desenvolvimento do protocolo de imagem metabólica foi apoiado pelo Ministério da Saúde da Federação Russa (Atribuição do Governo, registro nº АААА-А20-120022590098-0). O estudo da viscosidade foi apoiado pela Fundação Russa de Ciência (Projeto nº 20-14-00111). Os autores agradecem a Anton Plekhanov (PRMU) por sua ajuda na produção do vídeo.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Item/Device | |||
| Cell culture incubator | Sanyo | 37°C, 5% CO2, humidified atmosphere | |
| Centrifuge 5702 R | Eppendorf | 5703000010 | |
| imageJ 1.53c | Wayne Rasband (NIH) | ||
| FLIM module Simple Tau 152 TCSPC (in LSM 880) | Becker & Hickl GmbH | ||
| Laminar flow hood | ThermoFisher Scientific | ||
| Leica microscope DFC290 | Leica Microsystems | ||
| LSM 880 confocal microscope | Carl Zeiss | ||
| Ti:Sapphire femtosecond laser Mai Tai | Spectra Physics | ||
| Microscope incubator XLmulti S DARK LS | PeCon GmbH | 273-800 050 | |
| Mechanical pipettor | Sartorius mLINE | volume 0.5-10 μL; 20-200 μL; 100-1000 μL | |
| Oil-immersion objective C-Apochromat 40×/1.2 NA W Korr (in LSM 880) | Carl Zeiss | 421767-9971-790 | |
| Power-Tau 152 module with the detector HPM-100-40 | Becker&Hickl GmbH | ||
| SPCImage software | Becker & Hickl GmbH | SPC 9.8; SPCImage 8.3 | |
| ZEN software | Carl Zeiss | ZEN 2.1 SP3 (black), Version 14.0.0.201 | |
| Reagent/Material | |||
| 5-fluorouracil | Medac GmbH | 3728044 | |
| DMEM | Gibco, Life Technologies | 31885023 | |
| DMSO | PanEco | F135 | |
| FBS | Hyclone | A3160801 | |
| FluoroBright DMEM | Gibco, Life Technologies | A1896701 | |
| Hank’s solution without Ca2+/Mg2+ | Gibco, Life Technologies | 14175 | |
| l-Glutamine | PanEco | F032 | |
| Mammalian cells | HCT116, CT26, HeLa Kyoto, huFB | ||
| Molecular rotor BODIPY 2 | Synthesized and Supplied by Marina Kuimova Group, Imperial College London | ||
| Penicillin/streptomycin | PanEco | A065 | |
| Tissue culture dish with cover glass-bottom FluoroDishes | World Precision Instruments, Inc | ||
| Trypsin- EDTA 0.25% | PanEco | P034 | |
| Versen buffer | PanEco | R080p |
Referências
- Vazquez, A., et al. Cancer metabolism at a glance. Journal of Cell Science. 129 (18), 3367-3373 (2016).
- Li, Z., Zhang, H. Reprogramming of glucose, fatty acid, and amino acid metabolism for cancer progression. Cellular and Molecular Life Sciences. 73 (2), 377-392 (2015).
- Strickaert, A., et al. Cancer heterogeneity is not compatible with one unique cancer cell metabolic map. Oncogene. 36 (19), 2637-2642 (2016).
- Szlasa, W., Zendran, I., Zalesińska, A., Tarek, M., Kulbacka, J. Lipid composition of the cancer cell membrane. Journal of Bioenergetics and Biomembranes. 52, 321-342 (2020).
- Preta, G. New insights into targeting membrane lipids for cancer therapy. Frontiers in Cell and Developmental Biology. 8, 571237(2020).
- Kojima, K. Molecular aspects of the plasma membrane in tumor cells. Nagoya Journal of Medical Science. 56, 1-18 (1993).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 071203(2020).
- Becker, W. Advanced time-correlated single photon counting applications. Springer Series in Chemical Physics. , Springer. (2015).
- Shirmanova, M. V., Shcheslavskiy, V. I., Lukina, M. M., Becker, W., Zagaynova, E. V. Exploring tumor metabolism with time-resolved fluorescence methods: From single cells to a whole tumor. Multimodal Optical Diagnostics of Cancer. 3, 133-155 (2020).
- Kalinina, S., Rück, A. FLIM and PLIM in biomedical research. An innovative way to combine autofluorescence and oxygen measurements. Photonics & Lasers in Medicine. 5 (4), 257-266 (2016).
- Kuimova, M. K. Molecular rotors image intracellular viscosity. Chimia. 66 (4), 159-165 (2012).
- Kuimova, M. K. Mapping viscosity in cells using molecular rotors. Physical Chemistry Chemical Physics. 14 (37), 12671(2012).
- Haidekker, M. A., Theodorakis, E. A. Molecular rotors-fluorescent biosensors for viscosity and flow. Organic & Biomolecular Chemistry. 5, 1669-1678 (2007).
- Liu, X., et al. Molecular mechanism of viscosity sensitivity in BODIPY rotors and application to motion-based fluorescent sensors. ACS Sensors. 5 (3), 731-739 (2020).
- Shirmanova, M. V., Shimolina, L. E., Lukina, M. M., Zagaynova, E. V., Kuimova, M. K. Live cell imaging of viscosity in 3D tumour cell models. Multi-Parametric Live Cell Microscopy of 3D Tissue Models. Advances in Experimental Medicine and Biology. Dmitriev, R. 1035, 143-153 (2017).
- Shimolina, L. E., et al. Imaging tumor microscopic viscosity in vivo using molecular rotors. Scientific Reports. 7, 41097(2017).
- Scott, T. G., Spencer, R. D., Leonard, N. J., Weber, G. Synthetic spectroscopic models related to coenzymes and base pairs. V. Emission properties of NADH. Studies of fluorescence lifetimes and quantum efficiencies of NADH, AcPyADH, [reduced acetylpyridineadenine dinucleotide] and simplified synthetic models. Journal of the American Chemical Society. 92 (3), 687-695 (1970).
- Harvey, R. A., Damle, S. A fluorescent modification of flavin adenine dinucleotide. FEBS Letters. 26 (1-2), 341-343 (1972).
- Kubánková, M., Summers, P., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Microscopic viscosity of neuronal plasma membranes measured using fluorescent molecular rotors: Effects of oxidative stress and neuroprotection. ACS Applied Materials and Interfaces. 11, 36307-36315 (2019).
- Kubánková, M., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Molecular rotors report on changes in live cell plasma membrane microviscosity upon interaction with beta-amyloid aggregates. Soft Matter. 14, 9466-9474 (2018).
- Wu, Y., et al. Molecular rheometry: Direct determination of viscosity in Lo and Ld lipid phases via fluorescence lifetime imaging. Physical Chemistry Chemical Physics. 15 (36), 14986(2013).
- Shimolina, L. E., et al. Mapping cisplatin-induced viscosity alterations in cancer cells using molecular rotor and fluorescence lifetime imaging microscopy. Journal of Biomedical Optics. 25 (12), 126004(2020).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014(2007).
- Rück, A., Hauser, C., Mosch, S., Kalinina, S. Spectrally resolved fluorescence lifetime imaging to investigate cell metabolism in malignant and nonmalignant oral mucosa cells. Journal of Biomedical Optics. 19 (9), 096005(2014).
- Lee, D. -H., Li, X., Ma, N., Digman, M. A., Lee, A. P. Rapid and label-free identification of single leukemia cells from blood in a high-density microfluidic trapping array by fluorescence lifetime imaging microscopy. Lab on a Chip. 18 (9), 1349-1358 (2018).
- Lukina, M. M., et al. Interrogation of tumor metabolism in tissue samples ex vivo using fluorescence lifetime imaging of NAD(P)H. Methods and Applications in Fluorescence. 8 (1), 014002(2019).
- Alam, S. R., et al. Investigation of mitochondrial metabolic response to doxorubicin in prostate cancer cells: An NADH, FAD and Tryptophan FLIM assay. Scientific Reports. 7 (1), 10451(2017).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Lukina, M. M., et al. In vivo metabolic and SHG imaging for monitoring of tumor response to chemotherapy. Cytometry Part A. 95 (1), 47-55 (2019).
- Shah, A. T., et al. Optical metabolic imaging of treatment response in human head and neck squamous cell carcinoma. PLoS One. 9 (3), 90746(2014).
- Sergeeva, T. F., et al. Relationship between intracellular pH, metabolic co-factors, and caspase-3 activation in cancer cells during apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1864 (3), 604-611 (2017).
- Shirmanova, M. V., et al. Chemotherapy with cisplatin: insights into intracellular pH and metabolic landscape of cancer cells in vitro and in vivo. Scientific Reports. 7, 8911(2017).
- Lukina, M. M., et al. Metabolic cofactors NAD(P)H and FAD as potential indicators of cancer cell response to chemotherapy with paclitaxel. Biochimica et Biophysica Acta (BBA) - General Subjects. 1862 (8), 1693-1700 (2018).
- Druzhkova, I. N., et al. The metabolic interaction of cancer cells and fibroblasts - coupling between NAD(P)H and FAD, intracellular pH and hydrogen peroxide. Cell Cycle. 15 (9), 1257-1266 (2016).
- Lakowicz, J., Szmacinski, H., Nowaczyk, K., Johnson, M. Fluorescence lifetime imaging of free and protein bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Vanden Berg, P. A. W., Feenstra, K. A., Mark, A. E., Berendsen, H. J. C., Visser, A. J. W. G. Dynamic conformations of flavin adenine dinucleotide: Simulated molecular dynamics of the flavin cofactor related to the time-resolved fluorescence characteristics. The Journal of Physical Chemistry B. 106 (34), 8858-8869 (2002).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5, 3936(2014).
- Lu, W., Wang, L., Chen, L., Hui, S., Rabinowitz, J. D. Extraction and quantitation of nicotinamide adenine dinucleotide redox cofactors. Antioxidants and Redox Signaling. 28 (3), 167-179 (2018).
- Ranjit, S., Malacrida, L., Jameson, D. M., Gratton, E. Fit-free analysis of fluorescence lifetime imaging data using the phasor approach. Nature Protocols. 13 (9), 1979-2004 (2018).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116 (48), 24019-24030 (2019).
- Wang, S., Chacko, J. V., Sagar, A. K., Eliceiri, K. W., Yuan, M. Nonparametric empirical Bayesian framework for fluorescence-lifetime imaging microscopy. Biomedical Optics Express. 10 (11), 5497-5517 (2019).
- Quinn, K. P., et al. Quantitative metabolic imaging using endogenous fluorescence to detect stem cell differentiation. Scientific Reports. 3 (1), 3432(2013).
- Hou, J., et al. Correlating two-photon excited fluorescence imaging of breast cancer cellular redox state with seahorse flux analysis of normalized cellular oxygen consumption. Journal of Biomedical Optics. 21 (6), 060503(2016).
- Wang, H., et al. Red-emitting fluorescence probe for sensing viscosity in living cells. Chemical Papers. 74, 1071-1078 (2020).
- Chen, B., et al. Sensing and imaging of mitochondrial viscosity in living cells by a red photoluminescent probe with long lifetime. Chemical Communications. 55, 7410(2019).
- Shen, B., Wang, L. F., Zhi, X., Qian, Y. Construction of a red emission BODIPY-based probe for tracing lysosomal viscosity changes in culture cells. Sensors and Actuators B: Chemical. 304, 127271(2019).
- Chen, T., Chen, Z., Liu, R., Zheng, S. NIR fluorescent probe for detection of viscosity and lysosome imaging in live cells. Organic and Biomolecular Chemistry. 17, 6398(2019).
- Angelucci, C., et al. Epithelial-stromal interactions in human breast cancer: effects on adhesion, plasma membrane fluidity and migration speed and directness. PLoS One. 7 (12), 50804(2012).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados