É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Estudo do Desenvolvimento de Células Dendríticas por Short Hairpin RNA-Mediado Gene Knockdown em uma linha de célula hematopoiética e progenitora in vitro
Neste Artigo
Resumo
Aqui fornecemos um protocolo para triagem de potenciais fatores de transcrição envolvidos no desenvolvimento de células dendríticas (DC) usando transdução lentiviral de shRNA para obter linhas celulares de knockdown estáveis para diferenciação in vitro DC.
Resumo
As células dendríticas (DCs) são células importantes que apresentam antígenos que conectam respostas imunes inatas e adaptáveis. Os DCs são heterogêneos e podem ser divididos em DCs convencionais (cDCs) e DCs plasmacitóides (pDCs). cDCs é especializado em apresentar antígenos e ativar células T ingênuas. Por outro lado, os pDCs podem produzir grandes quantidades de interferons tipo I (IFN-I) durante a infecção viral. A especificação dos DCs ocorre em um estágio inicial de progenitores de DC na medula óssea (MM) e é definida por uma rede de fatores de transcrição (TFs). Por exemplo, cDCs altamente expressam ID2, enquanto pDCs altamente expressos E2-2. Uma vez que cada vez mais subconjuntos de DCs estão sendo identificados, há um crescente interesse em entender TFs específicos controlando o desenvolvimento de DC. Aqui, estabelecemos um método para tela de TFs críticos para a diferenciação de DCs in vitro , fornecendo lentivírus carregando RNA de grampo de cabelo curto (shRNA) em uma linha de células hematopoiéticas e progenitoras imortalizadas (iHSPCs). Após a seleção e diferenciação in vitro , o potencial de CDC e pDC das linhas celulares de knockdown estáveis são analisados por citometria de fluxo. Essa abordagem fornece uma plataforma para identificar genes potencialmente regendo os destinos dc de progenitores in vitro.
Introdução
Os DCs são os principais reguladores da imunidade inata e adaptativa1. Os DCs são classificados principalmente em duas populações funcionalmente distintas, ou seja, pDCs e cDCs. Além disso, os cDCs compreendem dois subconjuntos, ou seja, cDCs tipo I e tipo II ou cDC1s e cDC2s, respectivamente2. pDCs, expressando BST2, Siglec-H, e níveis intermediários de CD11c em camundongos3,4, são as células que podem segregar grandes quantidades de IFN-I durante inflamação e infecção viral5. Devido à sua robusta capacidade de produção ifn-I, eles também são suspeitos de desempenhar um papel fundamental na progressão de doenças autoimunes, incluindo lúpus eritematoso sistêmico (SLE)6. cDC1s, definidos pela expressão superficial de XCR1, CD8a, CLEC9A e CD103 em camundongos7, são especializados na ativação e polarização de células CD8+ (CTLs) citotóxicas através da apresentação cruzada de antígenos, iniciando assim a imunidade tipo I em resposta a patógenos intracelulares e câncer8,9. Por outro lado, cDC2s, expressando CD11b e CD172α (também conhecido como Sirpα) em humanos e camundongos, podem ativar células T CD4+ e promover a resposta imune tipo II contra alérgenos e parasitas10, bem como modular a imunidade tipo III após o reconhecimento extracelular de bactérias e microbiota11,12.
A diversificação de DCs é determinada por um grupo de TFs de células-tronco hematopoiéticas e progenitoras (HSPCs) no BM. E2-2 (codificado por Tcf4) é um regulador mestre para diferenciação e função dos pDCs13,14. Em contraste, o inibidor da ligação de DNA 2 (ID2) impulsiona a especificação do CDC e inibe o desenvolvimento do PDC através do bloqueio da atividade proteica E15. Além disso, o desenvolvimento de cDC1s requer IRF8 e BATF3, enquanto a diferenciação de cDC2s depende muito do IRF416. Trabalhos recentes têm explorado a heterogeneidade dos pDCs17 e cDCs e sua regulamentação transcricional18. Devido à complexidade da rede DC, há uma necessidade não atendida de estabelecer uma plataforma para identificar outros TFs que controlam o desenvolvimento e funcionalidade dos DCs.
Aqui, utilizamos um iHSPC que foi gerado expressando translocação nuclear regulamentada por estrogênio de Hoxb8 em células BM (também conhecidas como células Hoxb8-FL)19. os iHSPCs podem proliferar e permanecer em um estágio indiferenciado na presença de ligantes β-estradiol e Flt3 (FL), enquanto eles começam a se diferenciar em diferentes tipos de DC na presença de FL após a retirada de β-estradiol19. Com base nesse recurso, podemos derrubar genes de interesse no estágio progenitor, seguido por examinar o efeito sobre a diferenciação in vitro de pDCs e cDCs. Portanto, este método é uma ferramenta poderosa para descobrir os genes que regulam o desenvolvimento e a função dos DCs.
Protocolo
O manuseio do lentivírus é realizado de acordo com a regulamentação do Departamento de Saúde e Segurança Ambiental da Faculdade de Medicina da Universidade Nacional de Taiwan.
1. Preparação de linhas de células hematopoiéticas e progenitoras imortalizadas (iHSPCs)
- Mantenha a linha celular iHSPC em meio RPMI completo 1640 contendo FL de 100 ng/mL e 1 μM β-estradiol.
- Passar as células a uma proporção de 1:10 a cada 3 dias.
NOTA: Faça o meio RPMI 1640 completo suplementando com 10% de soro bovino fetal (FBS), 5 x 10-5 M β-mercaptoethanol e 10 μg/mL gentamicin. Recombinante murine FL também está disponível comercialmente.
2. Transdução lentiviral
- Placa iHSPCs a uma densidade de 1 x 105 células/bem em placas de 12 poços em 1 mL de meio completo contendo 100 ng/mL FL, 1 μM β-estradiol e 8 μg/mL polibrene.
NOTA: A concentração de polibrene depende dos tipos celulares e geralmente está na faixa de 4-8 μg/mL. - Adicione o lentivírus portador de shRNA em cada poço em uma multiplicidade de infecção (MOI) de 100.
NOTA: O vetor lentiviral é pLKO.1-Puro com um marcador de seleção de puramicina (Figura 1). As sequências de alvos de shRNAs contra LacZ, Tcf4 e Id2, respectivamente, estão listadas na Tabela de Materiais. O MOI é definido pelo número de virions que são adicionados por célula durante a infecção.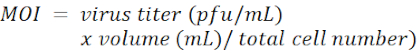
- Gire as placas a 1.100 x g por 90 min a 37 °C.
NOTA: A infecção por giro é realizada usando um rotor de balde de balanço. - Incubar a placa contendo células infectadas durante a noite a 37 °C em uma incubadora.
NOTA: Se as células forem sensíveis à polibrene, refresque as células com o meio completo sem polibrene após a infecção por giro. - Células de atualização com meio completo contendo FL de 100 ng/mL e 1 μM β-estradiol 24 h após a infecção.
- Adicione 6 μg/mL de puramicina ao meio para selecionar as células infectadas após mais 24 h.
NOTA: O vetor lentiviral transduzido geralmente leva 48 h para expressar genes, incluindo gene resistente à puramicina. Certifique-se de que a concentração de puromicina seja otimizada para cada linha celular. - Atualize o meio de seleção contendo FL de 100 ng/mL, 1 μM β-estradiol e 6 μg/mL puromicina a cada 3 dias e mantenha as células por pelo menos uma semana para expandir as células iHSPC transduzidas com facadas.
NOTA: A seleção de puramicina geralmente tem efeito 48h depois, e o período de seleção depende de tipos de células.
3. Medição da eficiência do knockdown por transcrição reversa e PCR em tempo real (RT-PCR)
- Extrair RNA total de 1 x 107 shLacZ, shTcf4 e células shId2 estáveis de knockdown iHSPC usando reagente de extração de RNA comercial e precipitar RNA da camada aquosa com isopropanol, seguido pela lavagem do precipitado RNA com 75% de etanol.
- Dissolva o RNA (~ 5 μg) com 5 μL de H2O tratado com DEPC e ajuste a concentração para 1 μg/μL.
- Pegue 1-3 μg de RNA, misture com H2O tratado com DEPC a um volume final de 17,4 μL, adicione 1 μL de 1 unidade/μL RNase-free DNase I e incubar por 20 min a 37 °C.
NOTA: Este passo é digerir o DNA genômico nas amostras de RNA. - Adicione 1 μL de 20 mM EDTA às amostras de RNA, incubar a 65 °C por 10 minutos para inativar DNase I e colocar as amostras de RNA imediatamente a 4 °C.
- Adicione 11,6 μL da mistura de reação contendo 1 μL de primers oligo (dT) (45 μM), 6 μL de tampão de 5x 1º fio, 3 μL de dNTP (2 mM), 0,6 μL de inibidor de RNase (50 Unit/μL) e 1 μL de transcriptase reversa (200 Unit/μL) para as amostras de RNA e incubar a 40 °C por 1 h.
- Pare a reação aquecendo a 70 °C por 10 min e dilua a mistura de reação com 30 μL de H2O.
- Tome 2 μL da mistura de reação RT diluída como um modelo de DNA e PCR amplificá-lo usando primers contra Tcf4 ou Id2 (ver Tabela 1 para condições termociclistas).
NOTA: As sequências de primer estão incluídas na Tabela de Materiais.
4. Diferenciação in vitro das linhas de células de knockdown estável iHSPC
- Mantenha o knockdown único estável de LacZ (shLacZ), Tcf4 (shTcf4) ou Id2 (shId2) em iHSPCs em meio completo contendo 100 ng/mL de FL e 1 μM β-estradiol.
- Colete iHSPCs shLacZ, shTcf4 e shId2 não diferenciados em um tubo de 15 mL e centrífuga por 5 min a 500 x g para pelotizar as células.
- Descarte o supernatante e adicione 10 mL de PBS para lavar as células. Repita este passo duas vezes.
- Resuspend e semente as células iHSPC a uma densidade de 2 x 105 células/mL em uma placa de 12 poços em 1 mL de meio completo contendo apenas 100 ng/mL FL.
- Adicione 1 mL de meio completo fresco contendo 100 ng/mL FL três dias depois.
- Analise as células diferenciadas (shLacZ, shTcf4 e shId2 iHSPCs) por citometria de fluxo dois dias depois.
5. Análise citométrica de fluxo dos DCs diferenciados
- Colete as células em tubos de 1,5 mL, encaixe as células para cima e para baixo 2-3 vezes na placa, e centrífuga por 5 min a 500 x g à temperatura ambiente (RT).
NOTA: Os DCs diferenciados in vitro se prenderão ligeiramente às placas. A tubulação suave ajudará a recuperar os DCs das placas. - Descarte o supernatante e resuspenque as células em 50 μL de tampão FACS. Em seguida, adicione 50 μL de anti-CD16/32 hybridoma supernante e incubar por 5-10 min no gelo.
NOTA: O anticorpo de bloqueio fc previne a ligação inespecífica de anticorpos aos receptores Fc expressos em algumas células mielóides e células B e está comercialmente disponível através de vários fornecedores. - Adicione anticorpos fluorescentes conjugados por corante (0,04 μg para cada anticorpo) diretamente às células e incubar por 15 minutos no gelo no escuro. Os anticorpos utilizados são APC/cy7 anti-mouse CD11c, FITC anti-mouse CD11b e PE anti-mouse B220.
NOTA: os pDCs são definidos como CD11c+CD11b-B220+, e os cDCs são definidos como CD11c+CD11b+B220-. - Lave as células com 1 mL de tampão FACS e centrífuga por 5 min a 500 x g em RT.
- Resuspense as células em 100 μL de tampão FACS e analise por citometria de fluxo.
Resultados
O mapa do vetor lentiviral pLKO.1-Puro é mostrado (Figura 1). Após a entrega do lentivírus que expressa shRNA contra LacZ (um controle não direcionado), Tcf4 e Id2 em iHSPCs, a eficiência de knockdown confirmada pelo RT-qPCR revelou que a expressão de Tcf4 foi reduzida em shTcf4 iHSPCs, em comparação com shLacZ iHSPCs (Figura 2A). Por outro lado, a diminuição da expressão do Id2 também fo...
Discussão
Vetores de shRNA baseados em lentivírus são frequentemente usados para silenciamento genético por transdução viral em células e permitem integração estável no genoma hospedeiro. No entanto, várias eficiências de transdução em diferentes tipos de células precisam ser consideradas, e uma série de abordagens foram tomadas para superar esse problema.
Polybrene é um polímero policalítico que pode neutralizar as cargas na membrana celular, aumentando assim a ligação da virion à...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos o apoio técnico do Dr. Tz-Ling Chen. Agradecemos ao National RNAi Core Facility (Academia Sinica, Taiwan) por fornecer shRNA lentivirus (http://rnai.genmed.sinica.edu.tw). Este trabalho foi apoiado pelo Ministério da Ciência e Tecnologia de Taiwan (MOST 108-2320-B-002-037-MY3 e MOST 109-2320-B-002-054-MY3).
Materiais
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| APC/Cy7 anti-mouse CD11c Antibody | Biolegend | 117324 | (Clone: N418) |
| FITC anti-mouse/human CD11b Antibody | Biolegend | 101206 | (Clone: M1/70) |
| PE anti-mouse/human B220 Antibody | Biolegend | 103208 | (Clone: RA3-6B2) |
| Cell culture | |||
| 1.5 mL Micro tube | ExtraGene | TUBE-170-C | |
| 12-well tissue culture-treated plate | Falcon | 353043 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| RPMI 1640 medium | gibco | 11875-085 | |
| Reagent | |||
| β-estradiol | Sigma-Aldrich | E2758-250MG | |
| β-mercaptoethanol (β-ME) | Sigma-Aldrich | M6250 | |
| FACS buffer | home-made | Formula: 1xPBS+0.5 %FBS+0.1%NaN3 | |
| Flt3 ligand (FL) | home-made | ||
| Polybrene | Sigma-Aldrich | TR-1003-G | |
| Puromycin | Invivogen | ant-pr-1 | |
| TRIsure | BIOLINE | BIO-38032 | |
| shRNA (Taregt sequence/clone ID) | Company | ||
| shId2 (GCTTATGTCGAATGATAGCAA/TRCN0000054390) | The RNAi Consortium (TRC) | ||
| shLacZ (CGCGATCGTAATCACCCGAGT/TRCN0000072224) | The RNAi Consortium (TRC) | ||
| shTcf4 (GCTGAGTGATTTACTGGATTT/TRCN0000012094) | The RNAi Consortium (TRC) |
Referências
- Steinman, R. M., Witmer, M. D. Lymphoid dendritic cells are potent stimulators of the primary mixed leukocyte reaction in mice. Proceedings of the National Academy of Sciences. 75 (10), 5132-5136 (1978).
- Guilliams, M., et al. Dendritic cells, monocytes and macrophages: a unified nomenclature based on ontogeny. Nature Reviews Immunology. 14 (8), 571-578 (2014).
- Blasius, A. L., Cella, M., Maldonado, J., Takai, T., Colonna, M. Siglec-H is an IPC-specific receptor that modulates type I IFN secretion through DAP12. Blood. 107 (6), 2474-2476 (2006).
- Swiecki, M., Colonna, M. Unraveling the functions of plasmacytoid dendritic cells during viral infections, autoimmunity, and tolerance. Immunology Reviews. 234 (1), 142-162 (2010).
- Liu, Y. -. J. IPC: Professional Type 1 interferon-producing cells and plasmacytoid dendritic cell precursors. Annual Review of Immunology. 23 (1), 275-306 (2005).
- Panda, S. K., Kolbeck, R., Sanjuan, M. A. Plasmacytoid dendritic cells in autoimmunity. Current Opinion in Immunology. 44, 20-25 (2017).
- Durai, V., Murphy, K. M. Functions of murine dendritic cells. Immunity. 45 (4), 719-736 (2016).
- Mashayekhi, M., et al. CD8α(+) dendritic cells are the critical source of interleukin-12 that controls acute infection by Toxoplasma gondii tachyzoites. Immunity. 35 (2), 249-259 (2011).
- Anderson, D. A., Dutertre, C. -. A., Ginhoux, F., Murphy, K. M. Genetic models of human and mouse dendritic cell development and function. Nature Reviews Immunology. 21 (2), 101-115 (2021).
- Plantinga, M., et al. Conventional and monocyte-derived CD11b(+) dendritic cells initiate and maintain T helper 2 cell-mediated immunity to house dust mite allergen. Immunity. 38 (2), 322-335 (2013).
- Persson, E. K., et al. IRF4 transcription-factor-dependent CD103(+)CD11b(+) dendritic cells drive mucosal T helper 17 cell differentiation. Immunity. 38 (5), 958-969 (2013).
- Kumamoto, Y., et al. CD301b+ dermal dendritic cells drive T helper 2 cell-mediated immunity. Immunity. 39 (4), 733-743 (2013).
- Cisse, B., et al. Transcription factor E2-2 is an essential and specific regulator of plasmacytoid dendritic cell development. Cell. 135 (1), 37-48 (2008).
- Grajkowska, L. T., et al. Isoform-specific expression and feedback regulation of E protein TCF4 control dendritic cell lineage specification. Immunity. 46 (1), 65-77 (2017).
- Jackson, J. T., et al. Id2 expression delineates differential checkpoints in the genetic program of CD8α+ and CD103+ dendritic cell lineages. The EMBO Journal. 30 (13), 2690-2704 (2011).
- Suzuki, S., et al. Critical roles of interferon regulatory factor 4 in CD11bhighCD8alpha- dendritic cell development. Proceedings of the National Academy of Science U. S. A. 101 (24), 8981-8986 (2004).
- Rodrigues, P. F., et al. Distinct progenitor lineages contribute to the heterogeneity of plasmacytoid dendritic cells. Nature Immunology. 19 (7), 711-722 (2018).
- Brown, C. C., et al. Transcriptional basis of mouse and human dendritic cell heterogeneity. Cell. 179 (4), 846-863 (2019).
- Redecke, V., et al. Hematopoietic progenitor cell lines with myeloid and lymphoid potential. Nature Methods. 10 (8), 795-803 (2013).
- Abe, A., Miyanohara, A., Friedmann, T. Polybrene increases the efficiency of gene transfer by lipofection. Gene Therapy. 5 (5), 708-711 (1998).
- Sorgi, F. L., Bhattacharya, S., Huang, L. Protamine sulfate enhances lipid-mediated gene transfer. Gene Therapy. 4 (9), 961-968 (1997).
- Kirkling, M. E., et al. Notch signaling facilitates in vitro generation of cross-presenting classical dendritic cells. Cell Reports. 23 (12), 3658-3672 (2018).
- Pearce, E. J., Everts, B. Dendritic cell metabolism. Nature Reviews. Immunology. 15 (1), 18-29 (2015).
- Wculek, S. K., Khouili, S. C., Priego, E., Heras-Murillo, I., Sancho, D. Metabolic Control of Dendritic Cell Functions: Digesting Information. Frontiers in Immunology. 10 (775), (2019).
- Saxton, R. A., Sabatini, D. M. mTOR signaling in growth, metabolism, and disease. Cell. 168 (6), 960-976 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados