É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Usando o analisador de fluxo metabólico de células em tempo real para monitorar bioenergetics osteoblast
Neste Artigo
Resumo
O ensaio de fluxo metabólico celular em tempo real mede a taxa de consumo de oxigênio e a taxa de acidificação extracelular, que corresponde à produção de triptosfato de adenosina mitocondrial e glicolítico, usando sensores de pH e oxigênio. O manuscrito explica um método para compreender o estado energético dos osteoblastos e a caracterização e interpretação do estado bioenergéstico celular.
Resumo
A formação óssea por osteoblastos é um processo essencial para a aquisição óssea adequada e a rotatividade óssea para manter a homeostase esquelética e, finalmente, prevenir a fratura. No interesse de otimizar o pico de massa óssea e combater várias doenças musculoesquelletas (ou seja, osteoporose pós-menopausa, anorexia nervosa, diabetes mellitus tipo 1 e 2), esforços incríveis têm sido feitos no campo da biologia óssea para caracterizar totalmente os osteoblastos durante seu processo de diferenciação. Dado o papel primário dos osteoblastos maduros para segregar proteínas matriciais e vesículas de mineralização, observou-se que esses processos tomam uma quantidade incrível de energia celular, ou triptosfato de adenosina (ATP). O status geral de energia celular é frequentemente referido como bioenergéstico celular, e inclui uma série de reações metabólicas que sentem a disponibilidade de substrato para derivar ATP para atender às necessidades celulares. Portanto, o método atual detalha o processo de isolação das células estrômicas primárias de medula óssea murina (BMSCs) e o monitoramento de seu status bioenergénico utilizando o analisador de fluxo metabólico de células reais em vários estágios na diferenciação do osteoblasto. É importante ressaltar que esses dados demonstraram que o perfil metabólico muda drasticamente ao longo da diferenciação do osteoblasto. Assim, o uso desse tipo de célula fisiologicamente relevante é necessário para apreciar plenamente como o status bioenergértico de uma célula pode regular a função geral.
Introdução
A formação do osso pelo osteoblasto é acompanhada de destruição coordenada ou resorção dos ossos por osteoclastos. O equilíbrio entre a formação óssea osteoblástica e a resorção do osteoclato é um processo acoplado que descreve a rotatividade óssea ou a remodelação, essencial para a homeostase esquelética. A disfunção do osteoblasto leva à formação óssea prejudicada e resulta em várias doenças, incluindo a osteoporose 1,2,3. A diferenciação ex vivo/in vitro de células-tronco estrômicas de medula óssea (BMSCs) aos precursores do osteoblasto e osteoblastos maduros resulta na formação e deposição da matriz óssea mineralizada no vaso cultural ao longo do tempo 4,5,6. Esta formação óssea pelo osteoblasto requer uma quantidade significativa de energia celular. Especificamente, a síntese e secreção de colágeno têm se mostrado fortemente dependentes de ATP celular: as proporções de ADP e, presumivelmente, o tráfico e a secreção mineralizados requerem ATP 7,8,9,10,11 adicionais. Muitos pesquisadores demonstraram que o processo de osteoblastogênese e função osteoblastogista requer um fornecimento adequado de energia para atender à demanda metabólica de formaçãoóssea 12,13,14,15,16. Portanto, o objetivo deste método é caracterizar o estado bioenergéstico das células primárias e estrômicas murinas ao longo da diferenciação do osteoblasto utilizando o analisador de fluxo metabólico celular em tempo real. Essas técnicas auxiliam no desenvolvimento de uma melhor compreensão da homeostase esquelética, o que pode, em última análise, levar ao desenvolvimento de novas opções terapêuticas capazes de melhorar os transtornos esqueléticos.
O analisador de fluxo metabólico celular em tempo real pode ser usado para medir a taxa de consumo de oxigênio (OCR) e a taxa de acidificação extracelular (ECAR) de osteoblastos vivos, o que corresponde à produção de ATP mitocondrial e glicolítico, respectivamente. Fundamental para essa metodologia é o fato de que um íon H+ por lactato é lançado durante a glicolise na conversão de glicose em lactato, o que altera o pH da mídia refletido nos valores ECAR. Por outro lado, durante o ciclo TCA (ácido tricarboxílico), a fosforilação oxidativa através das mitocôndrias produz CO2 utilizando ou consumindo oxigênio, e, portanto, o monitoramento de OCR é reflexo desse processo metabólico. O analisador mede tanto o OCR quanto o ECAR no microambiente extracelular simultaneamente e em tempo real, o que permite um enorme potencial ao estudar bioenergeticscelulares 6,17. Além disso, a realização desses ensaios é relativamente simples e facilmente personalizável dependendo do objetivo experimental. Técnicas semelhantes têm sido empregadas para entender melhor a regulação metabólica t-célula do sistema imunológico18,19, iniciação do câncer e progressão20, juntamente com vários outros tipos de células contribuindo para síndromes metabólicas21,22.
As vantagens do analisador de fluxo metabólico em tempo real sobre técnicas alternativas incluem (1) a capacidade de medir bioenergésicos celulares de células vivas em tempo real, (2) capacidade de realizar ensaios com um número relativamente pequeno de células (requer até 5.000 células), (3) portas de injeção para manipular paralelamente múltiplos tratamentos em um sistema de alta produtividade de 96 poços, (4) uso de imagens celulares automatizadas sem rótulo radioativo para normalização18, 23,24. Os seguintes métodos visam fornecer uma descrição generalizada, mas detalhada, do monitoramento de bioenergésticos celulares em BMSCs murinas durante a diferenciação do osteoblasto usando o analisador. Incluirá ensaios realizados rotineiramente; no entanto, como acontece com muitas técnicas e métodos, é altamente encorajado que laboratórios individuais determinem detalhes específicos para seus experimentos.
Seleção de ensaios e diferentes tipos de ensaios disponíveis: Uma grande variedade de kits de ensaio e reagentes estão disponíveis para estudar os bioenergésticos das células, garantindo a confiabilidade e consistência dos resultados experimentais. Além disso, o software de desktop também oferece modelos de ensaio que podem ser facilmente personalizados. O ensaio pode ser definido com base nas necessidades do usuário de medir diferentes parâmetros metabólicos. Esses ensaios podem ser modificados de várias maneiras com base no objetivo experimental e/ou na questão científica. Por exemplo, com quatro portas de injeção, vários compostos podem ser injetados na mídia de ensaio para analisar a resposta celular específica a cada via metabólica.
Teste de fenótipo de energia celular: Este ensaio mede o fenótipo metabólico das células vivas e o potencial metabólico. Este ensaio também é recomendado como o primeiro passo para obter uma ideia generalizada de metabolismo específico da via. Uma mistura de oligomicina A-an inibidora de atp synthase e cianeto carbonil 4-(trifluorometoxy) fenilhydrazone (FCCP)-um agente mitocondrial de desacoplamento é injetada para entender o potencial energético celular. A injeção de oligomicina A inibe a síntese de ATP, resultando em um aumento na taxa de glicolise (ECAR) para permitir que as células atendam às suas demandas energéticas; por outro lado, a injeção de FCCP resulta em maior OCR devido à despolarização da membrana mitocondrial. Essencialmente, este ensaio retrata a respiração metabólica basal, e seguindo as injeções duplas, empurra ou estressa, a resposta metabólica. Com base nesses parâmetros, o software então traça OCR e ECAR das células classificando as células como estado aeróbico, quiescente, glicolítico ou energético ao longo do tempo25,26.
Ensaio da taxa de produção em tempo real da ATP: Isso mede a produção de ATP celular simultaneamente a partir da glicolise e da respiração mitocondrial. Este ensaio mede quantitativamente as mudanças metabólicas das duas vias energéticas e fornece dados sobre as taxas de produção atpicondrial e glicolítico atp ao longo do tempo. O ensaio obtém dados basais de OCR e ECAR seguidos pelo cálculo da taxa de produção de ATP mitocondrial através da injeção de oligomicina A e taxa de produção de ATP glicolítico através da injeção de rotenona + mistura de antimicina A (inibição total da função mitocondrial), resultando na acidificação mitocondrial17,27.
Teste de estresse das mitocôndrias celulares (ou teste de estresse mito celular): Isso mede a função mitocondrial através da respiração ligada à ATP, quantifica a bioenergetic celular, identifica a disfunção mitocondrial e mede a resposta das células ao estresse. Vários parâmetros, incluindo capacidade respiratória basal e sobressal, respiração ligada a ATP, respiração máxima e consumo de oxigênio não mitocondrial, podem ser obtidos em um ensaio. Este ensaio envolve injeções sequenciais de oligomicina A, FCCP (agente de desacoplamento mitocondrial), uma mistura de inibidores de rotenona/antimicina A para analisar eficientemente o efeito destes na função mitocondrial28.
Teste de flexibilidade mito combustível flex: Isso mede a taxa de respiração mitocondrial pela oxidação dos três combustíveis mitocondriais primários pela presença e ausência de seus inibidores. A inibição sequencial de glicose, glutamina e ácidos graxos auxilia na medição da dependência, capacidade e flexibilidade das células e da dependência das células em várias vias celulares para atender à demanda energética. Quando as mitocôndrias não conseguem atender às demandas do caminho bloqueado de interesse oxidando outros combustíveis, as células entram em um estado de dependência. A capacidade das células é calculada pela inibição das outras duas vias alternativas seguidas pela inibição da via de interesse. A flexibilidade das células ajuda a entender a capacidade das mitocôndrias de compensar e atender às necessidades de combustível da via inibida. É calculado subtraindo a dependência das células da capacidade das células. Três inibidores diferentes são usados independentemente ou como uma mistura de dois para calcular efetivamente os parâmetros de ensaio. 2-ciano-3-(1-fenil-1H-indol-3-yl)-2-ácido propenóico (UK5099) inibe a oxidação da glicose bloqueando o portador de piruvato em glicólise. Bis-2-(5-fenillacetamido-1,3,4-thiadiazol-2-yl) (BPTES) sulfeto etílico inibe a via de oxidação da glutamina, e o etomoxir inibe a oxidação de ácidos graxos de cadeia longa29.
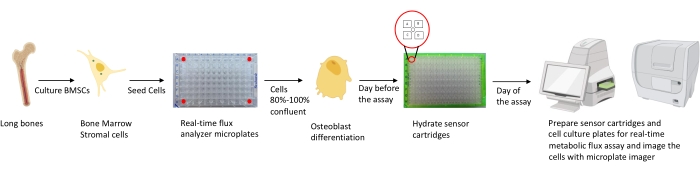
Figura 1: Representação esquemática da metodologia de cultivo e preparação de osteoblastos para análise. Os BMSCs murinos são isolados de ossos longos, cultivados e semeados em placas de 96 poços a 25.000 células/densidade de poços. A culização dessas células em mídia específica do Osteoblast é iniciada quando elas atingem 80%-100% de confluência para iniciar sua diferenciação. Os ensaios são realizados em diferentes estágios de diferenciação. As placas do cartucho estão hidratadas um dia antes do ensaio. No dia do ensaio, diferentes inibidores são injetados nas portas dos cartuchos do sensor com base nos requisitos do ensaio, e um buffer de calibração é adicionado à placa de calibração de 96 poços. Após a calibração, é realizado o ensaio de fluxo metabólico de células em tempo real, seguido por imagens da microplacão da cultura celular usando o imager microplalá para normalizar os dados do analisador de fluxo metabólico celular em tempo real com a contagem celular. Clique aqui para ver uma versão maior desta figura.
Protocolo
Todos os procedimentos foram baseados nas diretrizes e aprovação do Comitê Institucional de Cuidados e Uso de Animais no Centro Médico da Universidade Vanderbilt.
1. Preparação de reagentes e configuração de ensaio
- Isolamento e cultivo de células estrômicas de medula óssea (veja também o artigo anterior30).
- Prepare a mídia essencial mínima completa (αMEM) da cultura celular, suplementando o mínimo de mídia essencial com modificação alfa com 10% de FBS (soro bovino fetal), 100 U/mL de penicilina e 100 μg/mL de estreptomicina.
- Prepare o tubo de coleta de medula óssea aparando a extremidade de um tubo de microcentrifuuge de 0,6 mL para que as células possam passar e inseri-lo em um tubo de microcentrifuuge de 1,5 mL contendo 100 μL de αMEM completo.
- Eutanize os camundongos usando tratamento de CO2 da seguinte forma. Coloque o animal na câmara de CO2 por 2-3 minutos ou até que a respiração cesse. Espere pelo menos 1 minuto depois que o animal ficar inconsciente para remover os ratos da câmara e deslocar cervicalmente.
- Usando fórceps estéreis e um par de tesouras, abra o abdômen inferior dos ratos eutanizados para fazer uma pequena incisão. Isole os ossos longos (fêmur, tíbia e crista ilíaca) dos camundongos.
- Corte os ossos longos para remover todo o tecido mole. Uma vez que o osso é limpo, corte ~1-2 mm das extremidades distal e proximal para criar uma abertura para a medula para lavar.
NOTA: Esta abertura deve ser conservadora para evitar perder a medula, permitindo que ela saia. - Coloque os ossos em um tubo de coleta contendo 100 μL de PBS estéril de 1x (soro fisiológico tamponado para fosfato) para isolar a medula óssea total.
- Lave a medula por centrifugação a 10.000 x g para 15-20 s em temperatura ambiente. Células de medula pelota na parte inferior do tubo.
- Reincluada centrifugação até que a cavidade óssea pareça branca e desprovida da maioria dos elementos da medula. Resuspend a população mista de medula óssea, gentilmente pipetting para cima e para baixo.
- Cultume as células de um animal (tanto fêmur quanto tíbia) em um frasco de cultura celular de 75 cm2 em 10 mL de mídia de cultura celular e incubar a 37 °C em uma incubadora de cultura celular com 5% de CO2. Se agrupar células de 2-3 animais, use um frasco de cultura celular de 150 cm2 (recomendado).
- Após 24-48 h de incubação da população mista, aspirar a população de células hematopoiéticas não aderentes contidas na mídia cultural e lavar as células aderentes com 1x PBS.
- Semeadura celular de BMSCs e diferenciação de osteoblasto
- Tentesinizar as células aderentes adicionando 0,25% de trippsina-EDTA (aproximadamente 3-4 mL) para cobrir ligeiramente a superfície do frasco, seguido por uma incubação de 3 min a 37 °C.
- Adicione 6-7 mL de αMEM completo ao frasco/trypsin para resuspenque os BMSCs aderentes, tubos cuidadosamente para cima e para baixo. Transfira a suspensão do BMSC para um tubo de centrífuga cônica.
- Remova uma alíquota de 50 μL da suspensão BMSC e adicione 50 μL de azul trypan (diluição 1:1) a ele. Conte o número total de células viáveis que excluem o corante por tubos de 10 μL desta mistura em um hemótmetro e observando-o sob o microscópio. Não conte nenhuma célula morta ou insalubre que pareça de cor azul (<10% células).
- Com base na contagem de células, calcule o volume de suspensão celular em αMEM completo necessário para uma concentração final de 2,4 x 106 células/mL para um volume total de pelo menos 10 mL por placa.

Figura 2: Destacam-se a microplacão da cultura celular, especificamente projetada para o analisador. (A) Os quatro poços de correção de fundo, A1, A12, H1, H12, são destacados. Estes poços só contêm mídia de ensaio sem células. (B) O código de barras na lateral da placa para digitalizar a placa usando o leitor de imagens e o analisador. Clique aqui para ver uma versão maior desta figura.
- Centrifugar as células no tubo cônico a 1.000 x g por 5 min e resuspensar as células para a concentração final desejada de 2,4 x 106 células/mL.
- Transfira a suspensão celular para um reservatório e, usando um tubo multicanal, resuspente cuidadosamente as células para garantir uma mistura homogênea de células.
- Sementes 2,5 x 104 células por poço na microplacão de cultura celular de 96 poços com 80 μL de αMEM completo. Não faça células de sementes nos poços de correção de fundo (A1, A12, H1, H12); em vez disso, basta adicionar o meio nestes quatro poços.
NOTA: Os BMSCs para os ensaios são emplacados em microplacas de cultura celular de 96 poços projetadas para o analisador em conjunto com os cartuchos do sensor. A área de superfície dessas placas é diferente de uma placa normal de 96 poços. A área de superfície de cada poço na placa é de 0,106 cm2, que é aproximadamente 40% da área típica da placa de 96 poços. A densidade ideal de semeadura celular é escolhida com base no tipo celular. Normalmente, o analisador pode detectar entre 0,5-4 x 104 células por poço. Os osteoblastos precisam estar em contato para se diferenciar efetivamente; para este fim, foi selecionado o revestimento entre 2,0 x 10 4-3,0 x 104 BMSCs/well em 80 μL de αMEM completo. - Agitar suavemente a placa para garantir a distribuição uniforme das células nos poços e incubar a 37 °C, 5% de CO2. Verifique o crescimento das células e da confluência celular sob o microscópio após 48 h. Mude a mídia de cultura celular, se necessário.
- Dependendo da meta do ensaio, quando os BMSCs são 60%-80% confluentes (tipicamente 48-72 h), iniciam a diferenciação do osteoblasto alterando a mídia de cultura celular para mídia de diferenciação de osteoblasto (αMEM completo suplementado com fosfato de 5 mM β-glicerol e 50 μg/mL de ácido L-asbico).
- Se forem analisadas células estromais indiferenciadas (Dia 0), mantenham as células sob αMEM completos.
- Mude a mídia de diferenciação do osteoblasto a cada dois dias e visualize as células sob o microscópio para garantir que elas estejam saudáveis até o dia do ensaio. Preferencialmente, 24h antes do ensaio programado, altere a mídia e mantenha um cronograma de mudança média consistente (recomendado).
NOTA: Altere cuidadosamente a mídia inclinando ligeiramente as placas em um ângulo; isso evita o contato acidental de pontas de pipeta para as placas de cultura celular e interrupção da monocamada das células.
2. Preparação do cartucho do sensor para calibração de fluxo extracelular
- Hidrate os cartuchos do sensor do kit de ensaio extracelular antes do dia do ensaio. Remova os cartuchos do sensor (placa verde) e coloque os sensores de cabeça para baixo.
- Usando um tubo multicanal, adicione 200 μL de H2O a cada poço da placa de utilidade. Coloque cuidadosamente os cartuchos do sensor de volta na placa de utilidade e incubar a placa durante a noite em temperatura ambiente.
NOTA: O fabricante recomenda a incubação dos cartuchos do sensor em uma incubadora não CO2 37 °C durante a noite. No entanto, pode ocorrer uma evaporação significativa dos cartuchos do sensor. Se isso acontecer, os cartuchos de sensores podem ser incubados à temperatura ambiente. Estas placas devem ser incubadas por um mínimo de 4h e no máximo 72 h. - No dia do ensaio, descarte o H2O da placa do utilitário e adicione 200 μL de calibrante. Incubar a placa de utilidade por pelo menos 1h antes do ensaio.
3. Preparação de mídia do analisador de fluxo metabólico de células em tempo real
- Use a mídia DMEM livre de fenol vermelho com um pH pré-ajustado de 7,4 (recomendado) para executar o ensaio com BMSCs.
- Prepare 80 mL de mídia de ensaio suplementando mídia DMEM com piruvato de sódio de 1 mM, glutamina de 2 mM, 10 mM de glicose, insulina 200 nM, 50-200 μM ácido oleico BSA.
- Incubar a mídia completa de ensaio a 37 °C em um banho de água.
4. Preparação de compostos para os cartuchos de sensores
- Descongele oligomicina A, rotenona e antimicina A no gelo. Pipeta para cima e para baixo para solubilizar os compostos antes de usar.
- Adicione 3 mL de ensaio preparado médio a cada tubo, seguido da adição do respectivo tubo composto A: 26,4 μL de 2,5 mM oligomicina A; tubo B: 3,1 μL de 12,67 mM rotenona + 4,1 μL de 9,4 mM antimicina A + 30 μL Mancha hoechst.
- Carregue uma concentração de 10x desses inibidores na porta correspondente. A concentração final das soluções de injeção necessárias é 2 μM de oligomicina A, 1 μM de rotenona e 4,1 μM de antimicina A.
NOTA: Hoechst é adicionado à porta de injeção final para colorir fluorescentemente os núcleos para fins de imagem e normalização. Essas concentrações podem ser otimizadas com base no tipo celular. - Carregue 20 μL desses compostos nas células em 180 μL de mídia de ensaio.
5. Prepare microplacares de cultura celular para ensaio
- Remova a microplaca da cultura celular da incubadora de 37 °C e observe as células sob o microscópio.
- Remova o meio de ensaio do banho de água.
- Lave suavemente as células com 200 μL de média de ensaio duas vezes e adicione 200 μL de mídia de ensaio por poço.
NOTA: Uma vez que a mídia de ensaio final é adicionada às células, o tempo até as placas entrarem no analisador é crucial. Portanto, não comece a substituir a mídia até que as seguintes etapas sejam realizadas dentro de 1h. - Verifique as células sob o microscópio para garantir que as células permaneçam aderidas aos poços.
- Certifique-se de que as células em D5 e E8 são aderidas com uma monocamada consistente e não foram lavadas durante a etapa de lavagem. O software de imagem celular usa esses dois poços para definir o foco automático e a autoexposição.
NOTA: O fabricante recomenda a incubação da placa em uma incubadora não CO2 37 °C por 1 h; esta etapa pode ser ignorada se a imagem automatizada for preferida. Por exemplo, o imager de microplacão mantém as mesmas condições em uma câmara fechada, e as células podem ser imagens sob um campo brilhante.
6. Configuração do ensaio e imagem
Figura 3: O software do controlador. O software verifica se o equipamento está conectado e está definido para 37 °C. Os arquivos de modelo para diferentes ensaios que podem ser realizados com o analisador de fluxo extracelular podem ser selecionados para personalizar o ensaio ainda mais com base nos objetivos experimentais. Clique aqui para ver uma versão maior desta figura.
- Abra o software de desktop no computador ao lado do equipamento.
- Verifique o status da conexão no canto inferior esquerdo do software do controlador.
- Vá para Modelos e selecione o arquivo de modelo XF ATP Rate Assay ou o modelo de ensaio apropriado.
- Selecione Definições de grupo na parte superior da tela e defina os grupos.
- Selecione o Layout do Mapa da placa e atribua os poços dependendo dos grupos definidos.
- Verifique o protocolo de instrumentos, certifique-se de que os compostos adicionados estejam corretamente listados e inclua as informações do projeto para referências futuras.
- Clique em Executar ensaio; isso solicitará a seleção do local de armazenamento do arquivo de resultado.
- Selecione o local para salvar o arquivo de resultado.
- Salve o arquivo com a data do ensaio e clique em Iniciar Execução.
- Coloque o cartucho do sensor e a placa de utilidade na bandeja e clique em Estou pronto para iniciar a calibração.
- Antes de iniciar a calibração, certifique-se de que a tampa do cartucho seja removida e o cartucho do sensor seja colocado na orientação correta na placa do utilitário. Esta etapa levará de 10 a 20 minutos e, uma vez concluída, o software exibirá a caixa de diálogo Load Cell Plate .
7. Obtenha imagens de brightfield
NOTA: Esta etapa é opcional. Se não houver equipamento de imagem disponível, pule para a etapa 8.

Figura 4: O software de imagem celular comunica-se ao leitor de imagens através do computador. As células da microplacão podem ser imagens antes e depois do ensaio, e a contagem de células/bem é obtida após o ensaio para normalizar os dados. Clique aqui para ver uma versão maior desta figura.
- Abra o software de imagem celular no computador.
- Certifique-se de que o imager de microplacar está ligado e as portas estão conectadas ao computador.
- Verifique a barra de status no canto inferior esquerdo da tela para garantir que a temperatura esteja definida para 37 °C e que o status da conexão deve ser destacado em verde como pronto.
- Escaneie o código de barras da placa para iniciar o processo de imagem.
- Forneça um nome para a placa de celular e aperte Save (Este é o nome onde tanto o campo brilhante quanto as imagens fluorescentes serão salvos). Clique em Executar brightfield scan.
- A próxima tela, prato e menu de varredura, mostram as opções de imagem. Antes do ensaio, selecione Iniciar varredura brightfield.
- Coloque a microplacão de cultura celular junto com a tampa/tampa da placa no suporte da bandeja e alinhe bem a A1 com a marca A1. Clique em Close Tray.
- A próxima tela, aquisição de imagem brightfield, com um mapa de placas aparece. Clique em Scan All Wells, que inicia o processo de inicialização do sistema seguido de 30 a 35 minutos de uma varredura.
- Após a varredura de campo brilhante, remova a microplacão de cultura celular e coloque-a no analisador para realizar o ensaio.
8. Executando o ensaio
- Uma vez concluída a calibração, o software exibe a caixa de diálogo Placa de célula de carga .
- Clique em Abrir bandeja para substituir a bandeja de utilidade por uma microplaca de cultura celular. Certifique-se de que a tampa está removida e que o A1 da placa se encaixe na orientação correta.
- Em seguida, clique em Carregar a placa celular para iniciar o ensaio. O cartucho do sensor permanecerá dentro do analisador para as injeções de ensaio.
- Aguarde até que o ensaio comece e exiba o tempo estimado de conclusão.
- Após a conclusão do ensaio, o software exibe a caixa de diálogo Do cartucho de descarregamento . Clique em Ejetar e remova a microplaca de cultura celular do analisador.
- Remova cuidadosamente o cartucho do sensor e substitua a tampa da placa da célula. As células estão prontas para imagens fluorescentes e contagem de células.
- Depois de remover a placa celular e o cartucho do sensor, a caixa de diálogo Assay Complete aparece.
- Clique em Exibir resultados para abrir o arquivo de resultado do ensaio e normalizar os dados imediatamente ou clique em Home.
9. Obtenha imagens de fluorescência e normalize
NOTA: Este passo é um método opcional, mas preferido para a normalização de BMSCs e osteoblastos. Se não houver equipamento de imagem disponível, outro método de normalização precisa ser realizado, como isolamento de proteínas ou DNA e quantificação.

Figura 5: Imagens representativas do software de imagem utilizado para a normalização dos dados do ensaio. (A) Imagem de campo brilhante costurada mostrando a confluência celular em todo o poço. (B) Imagem de fluorescência costurada mostrando núcleos manchados de hoechst de osteoblastos usados para contar números de células para normalizar os resultados do ensaio. São osteoblastos após 7 dias de diferenciação. Clique aqui para ver uma versão maior desta figura.
- Após a conclusão do ensaio, escaneie o código de barras da placa com o leitor de código de barras portátil. Se a placa já foi imageda, não exigirá um novo nome.
- Selecione Fluorescência & Contagem de células, coloque a placa de célula no suporte da bandeja e clique em Close Tray.
- Na janela de aquisição de imagens, selecione Digitalizar Todos os Poços para iniciar a imagem. A imagem da fluorescência leva cerca de 15-20 minutos para escanear toda a placa. Observe a marca do carrapato verde denotando que a varredura foi concluída.
- Revise as imagens fluorescentes e a contagem de células no aplicativo de imagem e imagem celular clicando aleatoriamente em alguns dos poços.
NOTA: Há uma opção para visualizar células contadas no canto inferior direito da tela. Esta opção mostra uma imagem mascarada, destacando os objetos que foram contados. - Uma vez que a imagem de fluorescência esteja completa, exporte as imagens para referências adicionais.
- Uma vez que a contagem de imagens e células esteja completa, abra o arquivo Resultados e clique em Normalizar. A tela de normalização dará ao layout da placa e uma opção para importar a contagem de células.
- Clique em Importar e selecione Solicitar o software de desktop para normalizar o ensaio com contagem de células automaticamente.
Resultados
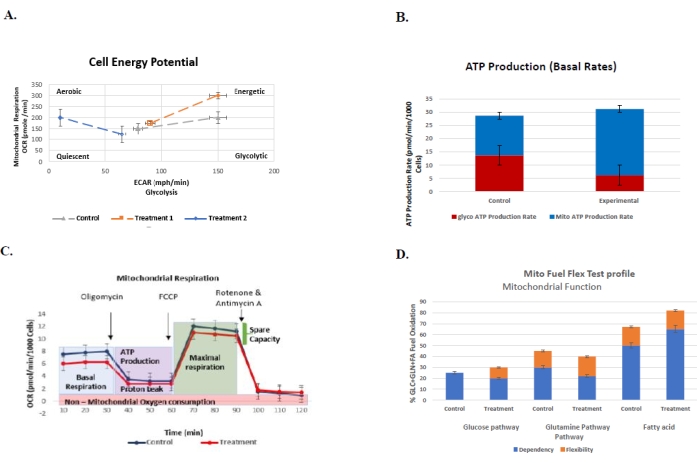
Figura 6: Gráficos representativos para ensaios realizados rotineiramente para compreender o perfil bioenergéstico celular do grupo de controle versus tratamento com seus respectivos erros padrão. (A) O teste do fenótipo de energia celular. O enredo representa a glicolise (ECAR) vs. respiração mitocondrial (OCR) do cont...
Discussão
O analisador de fluxo metabólico celular em tempo real pode ser usado para explorar energias celulares em diferentes condições. O protocolo ilustra o isolamento eficiente dos BMSCs, a cultura de células em placas de cultura celular apropriadas e sua diferenciação com os osteoblastos maduros, que podem ser usados para vários ensaios usando o analisador de fluxo extracelular. Além disso, as etapas críticas do ensaio de fluxo metabólico celular em tempo real, incluindo hidratação de cartuchos de sensores, carreg...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado pelo Instituto Nacional de Saúde (NIH) Instituto Nacional de Artrite e Doenças Musculoesqueléticos e de Pele (NIAMS) Grant AR072123 e Instituto Nacional de Envelhecimento (NIA) Grant AG069795 (para ERR).
Materiais
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin EDTA | Sigma-Aldrich | T4049 | |
| 2-cyano-3-(1-phenyl-1H-indol-3-yl)-2-propenoic acid | Sigma - Aldrich | PZ0160 | UK5099 |
| Antimycin A | Sigma - Aldrich | A8674 | |
| Ascorbic acid | Sigma-Aldrich | A4544-100G | |
| Bis-2-(5-phenylacetamido-1,3,4-thiadiazol-2-yl)ethyl sulfide | Sigma - Aldrich | SML0601 | BPTES |
| Carbonyl cyanide 4-(trifluoromethoxy)phenylhydrazone | Sigma - Aldrich | C2920 | FCCP |
| Cytation 5 imaging reader | BioTek | N/A | Microplate imager |
| Etomoxir sodium salt hydrate | Sigma - Aldrich | E1905 | |
| Hoechst 33342 Solution (20 mM) | Thermo Scientific | 62249 | |
| Insulin | Sigma - Aldrich | I6634 | |
| Oleic Acid-Albumin from bovine serum | Sigma - Aldrich | O3008 | |
| Oligomycin A - 5 mg | Sigma - Aldrich | 75351 | |
| Rotenone | Sigma - Aldrich | R8875-1G | |
| Seahorse XF 1.0 M Glucose Solution | Agilent Technologies | 103577-100 | |
| Seahorse XF 100mM Pyruvate Solution | Agilent Technologies | 103578-100 | |
| Seahorse XF 200mM Glutamine solution | Agilent Technologies | 103579-100 | |
| Seahorse XF DMEM media | Agilent Technologies | 103575-100 | DMEM assay media eith 5mM HEPES, pH 7.4, without phenol red, sodium bicarbonate, glucose, pyruvate, and L-glutamine |
| Seahorse XFe96 Analyzer | Agilent Technologies | S7800B | Real- Time Metabolic flux analyzer |
| Seahorse XFe96 FluxPak | Agilent Technologies | 102416-100 | Includes XFe96 Sensor cartridges, Cell culture microplates, and Seahorse XF Calibrant solution |
| The Cell imaging 1.1.0.11 software | Agilent Technologies - BioTek | ||
| Wave software 2.6.1 | Agilent Technologies | ||
| β-glycerol phosphate | Sigma-Aldrich | G9422-50G |
Referências
- Rodan, G. A. Bone homeostasis. Proceedings of the National Academy of Sciences of the United States of America. 95 (23), 13361-13362 (1998).
- Nakahama, K. I. Cellular communications in bone homeostasis and repair. Cellular and Molecular Life Sciences. 67 (23), 4001-4009 (2010).
- Kim, J. M., Lin, C., Stavre, Z., Greenblatt, M. B., Shim, J. H. Osteoblast-osteoclast communication and bone homeostasis. Cells. 9 (9), 2073 (2020).
- Gao, J., et al. SIRT3/SOD2 maintains osteoblast differentiation and bone formation by regulating mitochondrial stress. Cell Death and Differentiation. 25 (2), 229-240 (2018).
- Baron, R. Molecular mechanisms of bone resorption by the osteoclast. The Anatomical Record. 224 (2), 317-324 (1989).
- Tian, L., Rosen, C. J., Guntur, A. R. Mitochondrial Function and Metabolism of Cultured Skeletal Cells. Methods in Molecular Biology. 2230, 437-447 (2021).
- Zanotelli, M. R., et al. Regulation of ATP utilization during metastatic cell migration by collagen architecture. Molecular Biology of the Cell. 29 (1), 1-9 (2018).
- Gonzales, S., Wang, C., Levene, H., Cheung, H. S., Huang, C. Y. C. ATP promotes extracellular matrix biosynthesis of intervertebral disc cells. Cell and Tissue Research. 359 (2), 635-642 (2015).
- Kruse, N. J., Bornstein, P. The metabolic requirements for transcellular movement and secretion of collagen. Journal of Biological Chemistry. 250 (13), 4841-4847 (1975).
- Rendina-Ruedy, E., Guntur, A. R., Rosen, C. J. Intracellular lipid droplets support osteoblast function. Adipocyte. 6 (3), 250-258 (2017).
- Sinnott-Armstrong, N., et al. A regulatory variant at 3q21.1 confers an increased pleiotropic risk for hyperglycemia and altered bone mineral density. Cell Metabolism. 33 (3), 615-628 (2021).
- Esen, E., Lee, S. Y., Wice, B. M., Long, F. PTH promotes bone anabolism by stimulating aerobic glycolysis via IGF signaling. Journal of Bone and Mineral Research. 30 (11), 1959-1968 (2015).
- Borle, A. B., Nichols, N., Nichols, G. Metabolic studies of bone in vitro: I. Normal bone. Journal of Biological Chemistry. 235, 1206-1210 (1960).
- Borle, A. B., Nichols, N., Nichols, G. Metabolic studies of bone in vitro: II. The metabolic patterns of accretion and resorption. Journal of Biological Chemistry. 235, 1211-1214 (1960).
- Adamek, G., Felix, R., Guenther, H. L., Fleisch, H. Fatty acid oxidation in bone tissue and bone cells in culture. Characterization and hormonal influences. The Biochemical Journal. 248 (1), 129-137 (1987).
- Frey, J. L., et al. Wnt-Lrp5 signaling regulates fatty acid metabolism in the osteoblast. Molecular and Cellular Biology. 35 (11), 1979-1991 (2015).
- Romero, N., Rogers, G., Neilson, A., Dranka, B. P. . Quantifying cellular ATP production rate using agilent seahorse XF technology. , (2018).
- vander Windt, G., Chang, C., Pearce, E. Measuring bioenergetics in T cells using a Seahorse Extracellular Flux Analyzer. Current Protocols in Immunology. 113, 1 (2016).
- Traba, J., Miozzo, P., Akkaya, B., Pierce, S. K., Akkaya, M. An optimized protocol to analyze glycolysis and mitochondrial respiration in lymphocytes. Journal of Visualized Experiments:JoVE. (117), e54918 (2016).
- Noel, P., et al. Preparation and metabolic assay of 3-dimensional spheroid co-cultures of pancreatic cancer cells and fibroblasts. Journal of Visualized Experiments:JoVE. (126), e56081 (2017).
- Nicholls, D., et al. Bioenergetic profile experiment using C2C12 myoblast cells. Journal of Visualized Experiments: JoVE. (46), e2511 (2010).
- Sakamuri, S. S. V. P., et al. Measurement of respiratory function in isolated cardiac mitochondria using Seahorse XFe24 Analyzer: applications for aging research. GeroScience. 40 (3), 347-356 (2018).
- . What are the advantages of using Seahorse XF technology Available from: https://wwwagilent.com/en/support/cell-analysis/advantages-of-using-xf-tech (2018)
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. Journals of Gerontology - Series A Biological Sciences and Medical Sciences. 67 (10), 1022-1035 (2012).
- . XF cell energy phenotype test Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740884 (2021)
- Leung, D. T. H., Chu, S. Measurement of oxidative stress: Mitochondrial function using the seahorse system. Methods in Molecular Biology. 1710, 285-293 (2018).
- . XF ATP rate assay Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740889 (2021)
- . XF cell mito stress test Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740885 (2021)
- . XF cell mito fuel flex test Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740888 (2021)
- Maridas, D. E., Rendina-Ruedy, E., Le, P. T., Rosen, C. J. Isolation, culture, and differentiation of bone marrow stromal cells and osteoclast progenitors from mice. Journal of Visualized Experiments: JoVE. (131), e56750 (2018).
- Wei, J., et al. Glucose uptake and Runx2 synergize to orchestrate osteoblast differentiation and bone formation. Cell. 161 (7), 1576-1591 (2015).
- Zoch, M. L., Abou, D. S., Clemens, T. L., Thorek, D. L. J., Riddle, R. C. In vivo radiometric analysis of glucose uptake and distribution in mouse bone. Bone Research. 4, 16004 (2016).
- Divakaruni, A. S., Paradyse, A., Ferrick, D. A., Murphy, A. N., Jastroch, M. Analysis and interpretation of microplate-based oxygen consumption and pH data. Methods in Enzymology. 547, 309-354 (2014).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. . Normalization of agilent seahorse XF data by in-situ cell counting using a BioTek cytation 5 application note. , (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados