Method Article
Protocolos Otimizados de Amostragem Óssea para a Recuperação de DNA Antigo de Vestígios Arqueológicos
Neste Artigo
Resumo
O protocolo apresenta uma série de protocolos de melhores práticas para a coleta de pó ósseo de oito locais de amostragem anatômica recomendados (locais específicos em um determinado elemento esquelético) em cinco elementos esqueléticos diferentes de indivíduos medievais (radiocarbono datado de um período de cerca de 1040-1400 CE, faixa calibrada de 2-sigma).
Resumo
Os métodos aqui apresentados procuram maximizar as chances de recuperação do DNA humano de antigos vestígios arqueológicos, limitando a entrada de material de amostra. Isso foi feito visando locais de amostragem anatômica previamente determinados para produzir as maiores quantidades de DNA antigo (aDNA) em uma análise comparativa da recuperação de DNA em todo o esqueleto. Pesquisas anteriores sugeriram que esses protocolos maximizam as chances de recuperação bem-sucedida do DNA humano e patogênico antigo de vestígios arqueológicos. Os rendimentos de DNA foram previamente avaliados por Parker et al. 2020 em uma ampla pesquisa de preservação de aDNA em vários elementos esqueléticos de 11 indivíduos recuperados do cemitério medieval (radiocarbono datado de um período de cerca de (ca.) 1040-1400 CE, calibrado 2-sigma range) cemitério em Krakauer Berg, um assentamento medieval abandonado perto de Peißen Alemanha. Esses oito pontos de amostragem, que abrangem cinco elementos esqueléticos (pars petrosa, molares permanentes, vértebra torácica, falange distal e tálus) produziram com sucesso DNA humano antigo de alta qualidade, onde os rendimentos foram significativamente maiores do que a média geral em todos os elementos e indivíduos. Os rendimentos foram adequados para uso na maioria das análises genéticas populacionais a jusante mais comuns. Nossos resultados apoiam o uso preferencial desses locais de amostragem anatômica para a maioria dos estudos envolvendo as análises de DNA humano antigo de vestígios arqueológicos. A implementação desses métodos ajudará a minimizar a destruição de preciosos espécimes arqueológicos.
Introdução
A amostragem de restos humanos antigos para fins de recuperação e análise de DNA é inerentemente destrutiva 1,2,3,4. As amostras em si são espécimes preciosos e a preservação morfológica deve ser preservada sempre que possível. Como tal, é imperativo que as práticas de amostragem sejam otimizadas para evitar a destruição desnecessária de material insubstituível e para maximizar a probabilidade de sucesso. As técnicas de melhores práticas atuais baseiam-se numa pequena coorte de estudos limitados a inquéritos forenses5,6, estudos de espécimes antigos em que o desenvolvimento de amostragem óptima não é o objectivo directo do estudo7, ou estudos dedicados que utilizam restos não humanos8 ou que visam uma selecção muito pequena de locais de amostragem anatómica (utilizados aqui para denotar uma área específica de um elemento esquelético a partir do qual o pó ósseo, para uso em análises de DNA a jusante, foi gerado)9,10. Os protocolos de amostragem aqui apresentados foram otimizados no primeiro estudo sistemático em larga escala da preservação do DNA em múltiplos elementos esqueléticos de múltiplos indivíduos11. Todas as amostras resultaram de elementos esqueléticos recuperados de 11 indivíduos escavados do cemitério da igreja do assentamento medieval abandonado de Krakauer Berg, perto de Peißen, Saxônia-Anhalt, Alemanha (ver Tabela 1 para amostras demográficas detalhadas) e, como tal, podem precisar de modificação para uso com amostras fora dessa faixa geográfica/temporal.
| Individual | Sexo | Idade estimada no momento da morte | 14 anos Datas C (CE, Cal 2-sigma) |
| KRA001 | Macho | 25-35 | 1058-1219 |
| KRA002 | Fêmea | 20-22 | 1227-1283 |
| KRA003 | Macho | 25 | 1059-1223 |
| KRA004 | Macho | 15 | 1284-1392 |
| KRA005 | Macho | 10-12 | 1170-1258 |
| KRA006 | Fêmea | 30-40 | 1218-1266 |
| KRA007 | Fêmea | 25-30 | 1167-1251 |
| KRA008 | Macho | 20 | 1301-1402 |
| KRA009 | Macho | Desconhecido | 1158-1254 |
| KRA010 | Macho | 25 | 1276-1383 |
| KRA011 | Fêmea | 30-45 | 1040-1159 |
Tabela 1: Sexo geneticamente determinado, idade estimada arqueologicamente determinada no momento da morte e datação por radiocarbono (14C Cal 2-sigma) para todos os 11 indivíduos amostrados. Esta tabela foi adaptada de Parker, C. et al. 202011.
Esses protocolos permitem uma geração relativamente simples e eficiente de pó ósseo a partir de oito locais de amostragem anatômica em cinco elementos esqueléticos (incluindo a pars petrosa) com contaminação limitada de DNA induzida por laboratório. Desses cinco elementos esqueléticos, sete locais de amostragem anatômica encontrados em quatro elementos esqueléticos foram determinados como alternativas viáveis à amostragem destrutiva da pirâmide petrosa11,12. Estes incluem o cemento, a dentina e a câmara pulpar dos molares permanentes; osso cortical coletado da incisura vertebral superior, bem como do corpo das vértebras torácicas; osso cortical decorrente da superfície inferior do tufo apical e da diáfise das falanges distais; e o osso cortical denso ao longo da porção externa do tali. Embora existam vários métodos amplamente aplicados para a amostragem da pars petrosa 4,12,13,14, da dentina e da câmara da polpa dentária 1,2,15, métodos publicados descrevendo a geração bem-sucedida de pó ósseo a partir do cemento 16 , corpo vertebral, entalhe vertebral inferior e tálus podem ser difíceis de obter. Como tal, aqui demonstramos protocolos de amostragem otimizados para a pirâmide petrosa (etapa 3.1); cemento (etapa 3.2.1), dentina (etapa 3.2.2) e polpa dentária (etapa 3.2.3) de molares adultos; osso cortical do corpo vertebral (passo 3.3.1) e arco vertebral superior (passo 3.3.2); a falange distal (etapa 3.4); e o tálus (etapa 3.5), a fim de tornar o uso efetivo desses elementos esqueléticos para aDNA e pesquisa forense mais amplamente acessível.
Protocolo
Todas as pesquisas aqui apresentadas foram realizadas em conformidade com as diretrizes estabelecidas pelo Instituto Max Planck para a Ciência da História Humana, Jena, Alemanha, para trabalhar com restos humanos antigos. Antes de executar qualquer etapa deste protocolo, certifique-se de aderir a todos os requisitos éticos locais / estaduais / federais relativos à obtenção de permissão para o estudo científico e ao uso de restos humanos para amostragem destrutiva em sua área. Todos os procedimentos/armazenamento de produtos químicos devem ser realizados de acordo com as diretrizes de segurança institucionais individuais.
1. Considerações antes do processamento da amostra
- Trate as amostras com cuidado, pois os restos antigos são um recurso irreplicável e finito (por exemplo, a amostragem deve ser o menos desperdiçadora possível, e todos os restos devolvidos aos seus respectivos fornecedores legais, se possível).
- Execute todas as etapas em um ambiente de sala limpa, de preferência em uma instalação dedicada ao DNA antigo17,18,19. Use equipamentos de proteção individual (EPIs) que consistam em macacões microporosos estéreis com capuz, luvas estéreis (dois pares), máscara cirúrgica, óculos de proteção e botas estéreis ou sapatos antiderrapantes com capas estéreis (consulte Tabela de Materiais). Troque as luvas com frequência, especialmente entre as amostras.
- Limpe e desinfete completamente todos os equipamentos e superfícies com solução de descontaminação de água sanitária/DNA/etanol e irradiação UV (comprimento de onda: 254 nm) sempre que possível (por exemplo, brocas, brocas, vises/grampos, etc.). Finalmente, é altamente recomendável fazer pausas ergonômicas regulares (a cada 2-3 h, se possível) para evitar o excesso de exaustão devido ao ambiente de sala limpa.
NOTA: Todos os restos esqueléticos devem ser adequadamente documentados (por exemplo, fotografados, pesados e, se possível, micro-TC digitalizados, imagens 3D, etc.) antes da amostragem (protocolos para documentação apropriada não são cobertos neste manuscrito). Todos os protocolos de amostragem podem ser pausados entre as iterações de amostragem e as amostras podem ser armazenadas indefinidamente em um ambiente seco, com temperatura controlada (25 °C), estéril.
2. Pré-tratamento
- Descontaminar todos os locais de amostragem anatômica antes da geração do pó ósseo para minimizar o risco de contaminação18.
NOTA: A eficácia da lixívia e/ou remoção de superfície (ver NOTA na etapa 3.3.2 para as etapas de remoção de superfície) para a descontaminação de amostras ainda é uma questão de debate entre os pesquisadores de aDNA 8,19,20,21,22,23,24,25, pois ambas podem influenciar o rendimento geral de DNA, especialmente em amostras altamente degradadas. Dessa forma, as etapas a seguir são consideradas opcionais e estão incluídas aqui, pois foram utilizadas em todas as amostras para gerar os resultados representativos apresentados neste trabalho. Recomenda-se que o uso desses protocolos de pré-tratamento seja determinado caso a caso com base na aplicação molecular, idade, raridade e nível de degradação morfológica de cada conjunto de amostras.- Realize toda a amostragem em uma sala limpa dedicada sob um exaustor de reação em cadeia da polimerase (PCR) equipado com luz UV ou gabinete de biossegurança com o fluxo de ar desligado. Espalhe a folha de alumínio estéril pela bancada para capturar qualquer pó/fragmento de osso perdido.
- Certifique-se de que todos os fragmentos ósseos sejam recuperados (para repatriação) antes de descartar a folha. Troque a folha entre o tratamento de cada elemento esquelético. Eliminar a folha usada num saco/recipiente de risco biológico autoclavável.
- Remova o máximo possível de sujeira/detritos soltos dos locais de amostragem anatômica limpando suavemente a área com uma limpeza estéril seca e sem fiapos (consulte Tabela de Materiais). Eliminar os toalhetes em sacos ou recipientes de risco biológico autoclaváveis.
- Descontaminar a superfície limpa limpando com um lenço umedecido estéril humedecido com lixívia comercial diluída (~0,01% v/v, diluído com água ultrapura isenta de DNase/RNase) e deixar incubar durante 5 min. Eliminar os toalhetes em sacos ou recipientes de risco biológico autoclaváveis.
CUIDADO: A água sanitária é um produto químico altamente corrosivo e reativo; por conseguinte, devem existir precauções de segurança adequadas antes da sua utilização. - Remova o máximo de água sanitária residual possível do local de amostragem anatômica com um lenço umedecido estéril com água ultrapura isenta de DNase/RNase. Eliminar os toalhetes em sacos ou recipientes de risco biológico autoclaváveis.
- Exponha todos os locais de amostragem anatômica limpos à radiação UV por 30 minutos (comprimento de onda: 254 nm) e, em seguida, deixe secar totalmente à temperatura ambiente. Certifique-se de que os locais de amostragem anatômica estejam completamente secos antes de prosseguir com a amostragem ou retornar ao armazenamento para não apenas facilitar a geração de pó ósseo, mas também para evitar uma maior degradação da amostra (por exemplo, mofo).
CUIDADO: A exposição à radiação UV pode ser prejudicial para os olhos. - Deslocar-se imediatamente para a amostragem ou armazenar elementos esqueléticos num ambiente seco e estéril com temperatura controlada (25 °C).
3. Geração de pó ósseo
NOTA: Os protocolos a seguir destinam-se ao uso na extração de DNA seguindo o protocolo26 de Dabney et al. 2019.
- Amostragem de pars petrosa
NOTA: Este protocolo é adaptado dos procedimentos descritos em Pinhasi et al. 20194 e é apresentado aqui para facilitar o uso. Este protocolo não representa o método atual, menos destrutivo, para a amostragem de pars petrosa. Como tal, recomenda-se a utilização do protocolo descrito por Sirak et al. 201713 ou Orfanou et al. 202014 para amostras cuja preservação morfológica é de máxima importância.- Realize toda a amostragem em uma sala limpa dedicada sob um exaustor de PCR equipado com luz UV ou gabinete de biossegurança (comprimento de onda: 254 nm) com o fluxo de ar desligado. Espalhe a folha de alumínio estéril pela bancada para capturar qualquer pó/fragmento de osso perdido.
- Certifique-se de que todos os fragmentos ósseos e o máximo de pó possível sejam recuperados (para repatriação) antes de descartar a folha. Troque a folha entre cada amostragem. Eliminar a folha utilizada num saco/recipiente de risco biológico autoclavável.
- Prenda o elemento seco e descontaminado usando um grampo ou vise esterilizado.
- Cortar a pars petrosa ao meio ao longo do sulco petrosus superior (ver figura 1) utilizando uma serra de joalharia padrão equipada com uma lâmina de 0,6 mm (ver Tabela de Materiais) a uma velocidade média para evitar o sobreaquecimento (ver NOTA abaixo passo 3.1.6).
CUIDADO: A pars petrosa é muito densa e, como tal, pode ser difícil de cortar. Tome cuidado para manter o elemento firmemente preso para evitar lesões. Descarte todas as lâminas de serra quebradas no recipiente apropriado dos perfurocortantes. - Remova as porções petrosas da braçadeira. Recupere e salve qualquer material solto/excedente.
- Coloque o papel de pesagem em um barco de pesagem estéril
- Segure a porção petrosa sobre o papel de pesagem, cortado lateralmente inclinado em direção à bandeja de pesagem. Perfure o osso cortical denso entre o canal facial e o antro mastoideo (parece mais brilhante do que o material circundante, ver Figura 1) usando uma broca dentária equipada com um bit de calibre pequeno (ver Tabela de Materiais) e ajustada para velocidade média, torque médio para produzir pó ósseo.
NOTA: A perfuração/corte deve ser feita em rajadas curtas a velocidades baixas a médias para evitar o superaquecimento do osso e potencialmente destruir/danificar o DNA. Curiosamente, quando a porção densa do petroso começa a superaquecer, um cheiro descrito como bacon de cozimento pode ser observado. Pare de perfurar/serrar imediatamente e deixe o osso descansar até arrefecer suficientemente antes de retomar. - Repetir a perfuração até que aproximadamente 50-100 mg de pó sejam coletados no papel de pesagem, conforme medido usando uma balança fechada com precisão de pelo menos 0,01 mg (ver Tabela de Materiais).
NOTA: Sempre que possível, sugere-se reunir 100 mg de pó ósseo para permitir duas repetições de extração de DNA de 50 mg cada. No entanto, isso nem sempre pode ser possível com base na limitação dos próprios locais de amostragem anatômica (por exemplo, a falange distal, a câmara da polpa dentária) ou na necessidade de preservação morfológica. Para outros locais, como o cemento, podem estar disponíveis consideravelmente menos de 50 mg do material. No entanto, o cemento, a câmara da polpa dentária e a falange distal demonstraram produzir DNA endógeno significativo 11,27,28, apesar da menor entrada inicial de pó ósseo do processo de extração. - Transfira o pó do papel de pesagem para um tubo de 2 mL rotulado com baixa ligação e trava segura para extração ou armazenamento. Conservar as amostras a -20 °C, por tempo indeterminado.
- Armazenar o osso remanescente/excesso de pó em um ambiente seco e estéril com temperatura controlada (25 °C) até que o retorno/repatriamento possa ser concluído.
- Eliminar todos os resíduos em sacos ou recipientes de risco biológico autoclaváveis. Esterilizar/descontaminar todos os equipamentos reutilizáveis (por exemplo, grampos, brocas, brocas, serras, etc.) usando água sanitária/solução de descontaminação de DNA/etanol e exposição UV (comprimento de onda: 254 nm), conforme aplicável, entre cada amostragem.
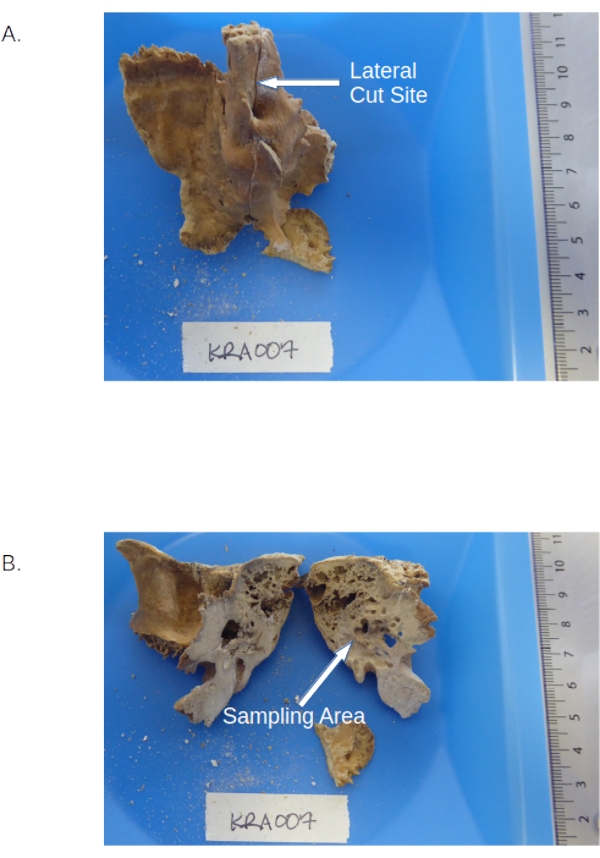
Figura 1: Osso temporal incluindo a pars petrosa. (A) Pré-corte de amostra mostrando as localizações da pirâmide petrosa e do sulco petrosa. (B) Porção petrosa pós-corte destacando as áreas densas a serem perfuradas. Por favor, clique aqui para ver uma versão maior desta figura.
- Amostragem de molares permanentes
NOTA: Para a amostragem de molares permanentes, pré-selecione in situ molares com raízes fundidas e, idealmente, vazios de cáries, rachaduras no esmalte ou desgaste excessivo para melhores resultados. Remova qualquer amostragem de cálculo dentário e armazene a -20 °C para possíveis análises futuras do microbioma oral (procedimento não abordado aqui).- Amostragem do cemento
- Realize toda a amostragem em uma sala limpa dedicada sob um exaustor de PCR equipado com luz UV ou gabinete de biossegurança (comprimento de onda: 254 nm) com o fluxo de ar desligado. Espalhe a folha de alumínio estéril pela bancada para capturar qualquer pó/fragmento de osso perdido.
- Certifique-se de que todos os fragmentos ósseos e o máximo de pó possível sejam recuperados (para repatriação) antes de descartar a folha. Troque a folha entre cada amostragem. Eliminar a folha usada num saco/recipiente de risco biológico autoclavável.
- Coloque uma folha de papel de pesagem em uma bandeja de pesagem estéril.
- Segure/prenda o molar descontaminado pelo esmalte, raiz para baixo, sobre uma bandeja de pesagem usando uma braçadeira de mão, como uma chave ajustável (consulte Tabela de materiais).
- Equipe uma broca dentária com uma roda de corte circular com bordas de diamante. Com a broca ajustada para uma configuração de velocidade/torque média, toque levemente a borda do bit na raiz em um ângulo de aproximadamente -20°.
- Raspe para baixo na bandeja para remover / coletar o material amarelo e mais externo da raiz (cemento). Pare a coleta quando o material mais claro (branco) da dentina se tornar visível.
NOTA: É importante combinar a direção de rotação do bit de corte em relação à bandeja de coleta para evitar que o pó se torne aerossolizado e potencialmente desperdice a amostra por perder a bandeja inteiramente. O cemento é particularmente rico em DNA; no entanto, os rendimentos típicos de material são muito menores do que outros locais de amostragem anatômica (~7-20 mg)11,27,28. - Registar a massa de pó recolhida em papel de pesagem utilizando uma balança fechada com uma precisão de, pelo menos, 0,01 mg (ver Tabela de Materiais).
- Transfira o pó do papel de pesagem para um tubo de bloqueio seguro e de baixa ligação de 2 mL para extração. Conservar a -20 °C, por tempo indeterminado.
- Amostragem da câmara pulpar
- Após a coleta do cemento (se desejado), corte o molar ao longo da junção cemento-esmalte usando uma serra de joalheiro para remover a coroa (ver Figura 2).
- Coloque uma nova folha de papel de pesagem em uma nova bandeja de pesagem.
- Prenda a seção da coroa em uma braçadeira ou viseira portátil, sobre a bandeja de pesagem. Segure o lado cortado inclinado para baixo e o material de perfuração/raspagem como a primeira passagem com uma broca dentária equipada com uma pequena broca de calibre (consulte Tabela de Materiais) ao longo das bordas da câmara de celulose dentro da porção da coroa (ver Figura 2).
NOTA: Apenas a primeira passagem do interior da câmara pulpar deve ser coletada e rotulada como material pulpar (rendimento típico de 5-15 mg), qualquer coisa mais profunda no dente é considerada dentina. - Vire o dente com a porção inferior voltada para baixo, bata no grampo com um martelo e colete o pó liberado no papel de pesagem.
- Registar o peso do pó recolhido no papel de pesagem utilizando uma balança fechada com uma precisão de, pelo menos, 0,01 mg (ver Tabela de Materiais).
- Transfira o pó do papel de pesagem para um tubo de 2 mL de baixa ligação e bloqueio seguro para extração. Conservar a -20 °C, por tempo indeterminado.
- Amostragem da dentina
- Coloque uma nova folha de papel de pesagem em uma nova bandeja de pesagem.
- Manter a secção da coroa sobre o tabuleiro de pesagem (de acordo com o passo 3.2.2.3), perfurar e recolher mais 50-100 mg de dentina, medidos utilizando uma balança fechada com precisão de 0,01 mg (ver Tabela de Materiais) do interior da câmara pulpar da mesma forma para posterior amostragem da dentina (ver figura 2).
- Transfira o pó ósseo do papel de pesagem para um tubo de 2 mL de baixa ligação e trava segura para extração. Conservar a -20 °C, por tempo indeterminado.
- Armazenar as peças dentárias restantes / excesso de pó em um ambiente seco, com temperatura controlada (25 °C) estéril até que o retorno/repatriação possa ser concluído.
- Eliminar todos os resíduos em sacos ou recipientes de risco biológico autoclaváveis. Esterilizar/descontaminar todos os equipamentos reutilizáveis (por exemplo, grampos, brocas, brocas, serras, etc.) usando as exposições de lixívia/solução de descontaminação de DNA/etanol e UV (comprimento de onda: 254 nm), conforme aplicável, entre cada amostragem.
- Amostragem do cemento
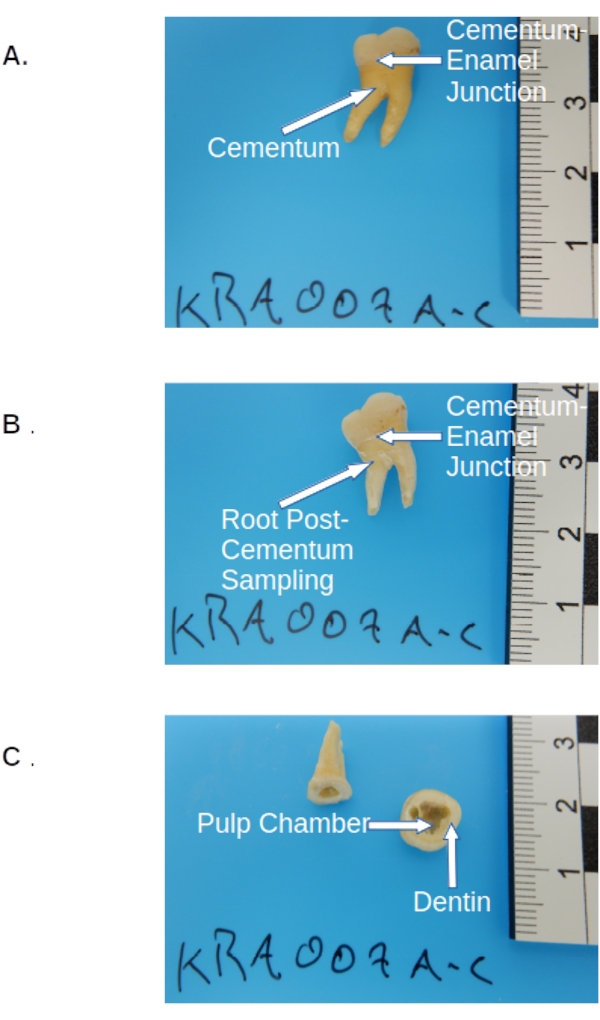
Figura 2: Pré-amostragem molar permanente . (A) Molar pré-tratado antes da amostragem, mostrando coroa, cemento (camada amarelada da raiz) e o local de corte na junção cemento-esmalte. (B) A mesma coleção molar pós-cemento, mostrando o local de corte na junção cemento-esmalte. (C) Pós-corte molar e amostragem mostrando locais de amostragem anatômica para a câmara da polpa dentária e dentina dentro da coroa. Por favor, clique aqui para ver uma versão maior desta figura.
- Amostragem das vértebras torácicas
- Amostragem do corpo vertebral
- Realize toda a amostragem em uma sala limpa dedicada sob um exaustor de PCR equipado com luz UV ou gabinete de biossegurança (comprimento de onda: 254 nm) com o fluxo de ar desligado. Espalhe a folha de alumínio estéril pela bancada para capturar qualquer pó/fragmento de osso perdido.
- Certifique-se de que todos os fragmentos ósseos e o máximo de pó possível sejam recuperados (para repatriação) antes de descartar a folha. Troque a folha entre cada amostragem. Eliminar a folha usada num saco/recipiente de risco biológico autoclavável.
- Coloque uma pequena folha de papel de pesagem em uma bandeja de pesagem padrão.
- Prenda as vértebras com um grampo ou viseira de mão, com o corpo vertebral para fora.
- Segure as vértebras sobre a bandeja de pesagem com o corpo vertebral inclinado para baixo. Usando uma broca dentária equipada com uma broca de perfuração de calibre pequeno (ver Tabela de Materiais) ajustada para torque alto torque de baixa velocidade, perfure ao longo da borda mais externa (inferior e superior) do osso cortical ao redor do tecido interno esponjoso do corpo vertebral (ver Figura 3).
- Raspe o bit contra a camada cortical sobre uma bandeja de pesagem padrão até que 50-100 mg de material sejam coletados, conforme medido usando uma balança fechada com precisão de 0,01 mg (ver Tabela de Materiais).
- Transfira o pó ósseo do papel pesado para um tubo de bloqueio seguro e de baixa ligação de 2 mL para extração. Conservar a -20 °C, por tempo indeterminado.
- Amostragem do arco vertebral superior
Observação : esta etapa é opcional. Remova e descarte a camada mais externa do osso cortical do arco vertebral superior usando uma broca dentária equipada com uma broca de perfuração de calibre pequeno (ver Tabela de Materiais), raspando-a ao longo da superfície19. Isto não é sugerido para amostragem do corpo vertebral, uma vez que a camada de osso cortical é geralmente muito fina e é provável que seja totalmente esgotada por este processo (ver NOTA na secção 2).- Coloque uma pequena folha de papel de pesagem em uma bandeja de pesagem padrão.
- Prenda as vértebras em uma braçadeira / vise de mão com o processo vertebral para fora, aspecto superior para baixo.
- Enquanto segura as vértebras, aspecto superior para baixo, sobre uma bandeja de pesagem, perfure para cima no centro do entalhe em forma de V formado pela fusão do processo espinhoso com as lamelas (ver Figura 3) usando uma broca dentária com um pequeno bit de calibre (ver Tabela de Materiais) ajustado para baixa velocidade e alto torque.
- Pare de perfurar quando houver uma queda perceptível na resistência. Altere ligeiramente a posição de perfuração e repita até que 50-100 mg de pó ósseo sejam coletados, conforme medido usando uma balança fechada com precisão de 0,01 mg (ver Tabela de Materiais).
- Transfira o pó ósseo do papel pesado para um tubo de baixa ligação de 2 mL para extração. Conservar a -20 °C, por tempo indeterminado.
- Armazenar o osso remanescente/excesso de pó num ambiente seco e estéril com temperatura controlada (25 °C) até à devolução/repatriamento.
- Eliminar todos os resíduos em sacos ou recipientes de risco biológico autoclaváveis. Esterilizar/descontaminar todos os equipamentos reutilizáveis (por exemplo, grampos, brocas, brocas, serras, etc.) utilizando água sanitária/solução de descontaminação de DNA/etanol e exposição UV (comprimento de onda: 254 nm), conforme aplicável, entre cada amostragem.
- Amostragem do corpo vertebral

Figura 3: Locais de amostragem anatômica do corpo vertebral e do osso cortical superior do arco vertebral superior da vértebra torácica. Clique aqui para ver uma versão maior desta figura.
- Amostragem da falange distal
Observação : esta etapa é opcional. Remover e descartar a camada mais externa do osso cortical do eixo e/ou tufo apical usando uma broca dentária equipada com uma pequena broca de perfuração, raspando-a ao longo da superfície19. Isto pode não ser possível para amostras com osso cortical excessivamente fino ou restos juvenis (ver NOTA na secção 2).- Realize toda a amostragem em uma sala limpa dedicada, sob um exaustor de PCR equipado com luz UV ou gabinete de biossegurança (comprimento de onda UV: 254 nm) com o fluxo de ar desligado. Espalhe a folha de alumínio estéril pela bancada para capturar qualquer pó/fragmento de osso perdido.
- Certifique-se de que todos os fragmentos ósseos e o máximo de pó possível sejam recuperados (para repatriação) antes de descartar a folha. Troque a folha entre cada amostragem. Eliminar a folha usada num saco/recipiente de risco biológico autoclavável.
- Coloque uma pequena folha de papel de pesagem em uma bandeja de pesagem padrão.
- Prenda a amostra em braçadeira/viseira portátil, lado superior para cima.
- Segure a amostra sobre a bandeja de pesagem, recolha o pó ósseo do osso cortical do lado inferior do tufo apical e do eixo perfurando as camadas densas mais exteriores (ver Figura 4) utilizando uma broca dentária equipada com uma broca de perfuração de calibre pequeno (ver Tabela de Materiais).
- Pare de perfurar quando houver uma diminuição acentuada na resistência, pois isso significa material mais leve e esponjoso. Repita este processo, irradiando para fora da perfuração inicial até que pelo menos 50-100 mg de pó ósseo sejam coletados, conforme medido usando uma balança fechada com precisão de 0,01 mg (ver Tabela de Materiais).
- Transfira o pó ósseo do papel de pesagem para um tubo de 2 mL de baixa ligação e trava segura para extração. Conservar a -20 °C, por tempo indeterminado.
- Conservar o osso restante/excesso de pó num ambiente seco e estéril com temperatura controlada (25 °C) até à devolução/repatriamento.
- Eliminar todos os resíduos em sacos ou recipientes de risco biológico autoclaváveis. Esterilizar/descontaminar todos os equipamentos reutilizáveis (por exemplo, grampos, brocas, brocas, serras, etc.) usando água sanitária/solução de descontaminação de DNA/etanol e exposição UV, conforme aplicável, entre cada amostragem.
NOTA: Para amostras menores (por exemplo, amostras juvenis) pode haver consideravelmente menos do que os 50-100 mg sugeridos de osso cortical disponíveis para amostra. No entanto, mesmo em baixas quantidades, esse local de amostragem anatômica tem se mostrado particularmente rico em DNA11.

Figura 4: Falange distal mostrando a localização do osso cortical denso ao longo da diáfise e do lado inferior do tufo apical. Por favor, clique aqui para ver uma versão maior desta figura.
- Amostragem do Talus
- Realize toda a amostragem em uma sala limpa dedicada sob um exaustor de PCR equipado com luz UV ou gabinete de biossegurança (comprimento de onda: 254 nm) com o fluxo de ar desligado. Espalhe a folha de alumínio estéril pela bancada para capturar qualquer pó/fragmento de osso perdido.
- Certifique-se de que todos os fragmentos ósseos e o máximo de pó possível sejam recuperados (para repatriação) antes de descartar a folha. Troque a folha entre cada amostragem. Eliminar a folha usada num saco/recipiente de risco biológico autoclavável.
- Coloque uma pequena folha de papel de pesagem em uma bandeja de pesagem padrão.
- Prenda a amostra em braçadeira/viseira portátil, cúpula para cima.
- Segure o tálus, a cúpula para cima e a superfície medial em direção ao coletor, sobre a bandeja de pesagem. Raspe o osso cortical do colo do tálus a uma profundidade de ~1 mm (ver Figura 5) usando uma broca dentária com um bit de calibre baixo (ver Tabela de Materiais) ajustado para baixa velocidade e alto torque.
- Altere ligeiramente a posição de perfuração e repita até que aproximadamente 50-100 mg de pó ósseo sejam coletados, conforme medido usando uma balança fechada com precisão de 0,01 mg (ver Tabela de Materiais).
- Transfira o pó ósseo do papel pesado para um tubo de baixa ligação de 2 mL para extração. Conservar a -20 °C, por tempo indeterminado.
- Armazenar o osso restante/excesso de pó num ambiente seco e estéril com temperatura controlada (25 °C) até que o retorno/repatriamento possa ser concluído.
- Eliminar todos os resíduos em sacos ou recipientes de risco biológico autoclaváveis. Esterilizar/descontaminar todos os equipamentos reutilizáveis (por exemplo, grampos, brocas, brocas, serras, etc.) usando água sanitária/solução de descontaminação de DNA/etanol e exposição UV (comprimento de onda: 254 nm), conforme aplicável, entre cada amostragem.
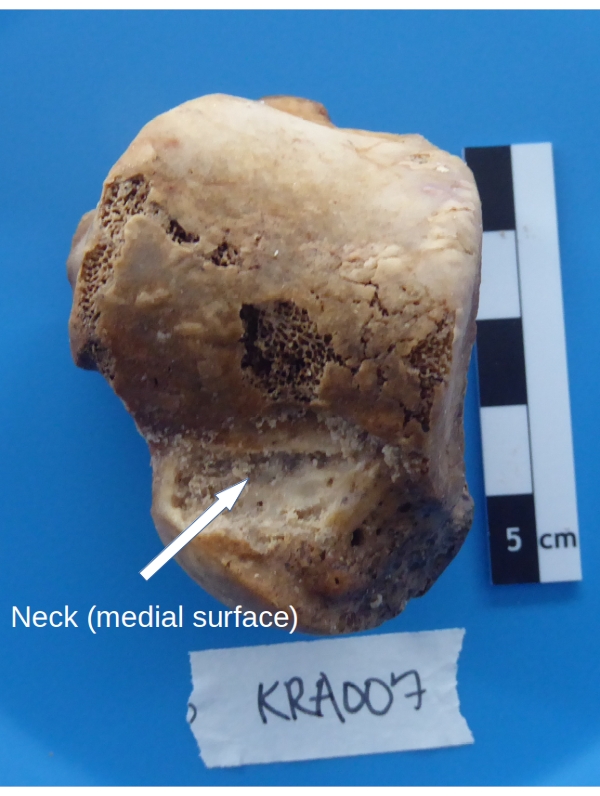
Figura 5: Área de amostragem do tálus para recuperação óssea cortical. Por favor, clique aqui para ver uma versão maior desta figura.
NOTA: O tálus tem muito pouco osso cortical (uma fina camada externa). O material não deve ser apenas coletado da superfície, mas também da camada densa subjacente de osso esponjoso.
Resultados
Em um estudo separado11, o DNA foi extraído do pó ósseo gerado de cada local de amostragem anatômica em 11 indivíduos, utilizando um protocolo padrão de extração de DNA otimizado para fragmentos curtos de tecido calcificado2. Bibliotecas de fita simples foram então produzidas28 e sequenciadas em um HiSeq 4000 (75 pb emparelhado-end) a uma profundidade de ~20.000.000 leituras por amostra. Os dados da sequência resultante foram então avaliados quanto ao conteúdo endógeno de DNA humano usando o pipeline EAGER29 (configurações BWA: comprimento da semente de 32, penalidade de incompatibilidade de 0,1, filtro de qualidade de mapeamento de 37). Todos os resultados representativos são relatados usando as mesmas métricas de Parker et al. 202011 para consistência. As bibliotecas das porções em pó da pars petrosa produziram, em média, DNA endógeno mais alto do que qualquer um dos outros 23 locais de amostragem anatômica pesquisados (Figura 6A-B). Os sete locais adicionais de amostragem anatômica apresentados neste protocolo (cemento, primeira passagem da câmara da polpa dentária e dentina dos molares permanentes; osso cortical do corpo vertebral e arco vertebral superior da vértebra torácica; osso cortical do tufo apical da falange distal; e osso cortical do colo do tálus) produziram os próximos maiores rendimentos (sem significância estatística entre esses locais de amostragem anatômica; Figura 6A-B; Arquivo Suplementar 1: EndogenousDNAPreCap). Todos esses locais alternativos produziram consistentemente rendimentos de DNA adequados para análises genéticas populacionais padrão, como análises mitocondriais e análises de polimorfismo de nucleotídeo único (SNP). As taxas de duplicação em bibliotecas decorrentes de todos os locais de amostragem anatômica foram baixas (fatores de conglomerados < 1,2 em média, calculados como a razão entre todas as leituras de mapeamento e leituras únicas de mapeamento, Tabela 2; Arquivo Suplementar 1: ClusterFactor), indicando que todas as bibliotecas examinadas eram de altíssima complexidade. Da mesma forma, as estimativas médias de contaminação exógena do DNA humano foram baixas, com média < 2% (contaminação do cromossomo X em machos, n = 7, conforme relatado pelo pipeline ANGSD30) em todos os locais de amostragem anatômica, exceto no arco vertebral superior (contaminação média estimada: 2,11%, com uma amostra removida como outlier; KRA005: 19,52%, ver Tabela 2; Arquivo Suplementar 1: Xcontaminação). O comprimento médio do fragmento (após filtragem para remoção de todas as leituras < 30 pb) foi menor no material coletado da câmara da polpa dentária e dentina, sem variação significativa entre os demais locais de amostragem anatômica (55,14 pb e 60,22 pb, respectivamente, em comparação com uma mediana média de 62,87, valores de p pareados, < 0,019, Tabela 2; Arquivo suplementar 1: AvgFragLength). Além disso, os dentes e as vértebras torácicas contêm múltiplos locais de amostragem anatômica onde foi observada alta recuperação endógena de DNA, tornando-os particularmente adequados como alternativas à pars petrosa.
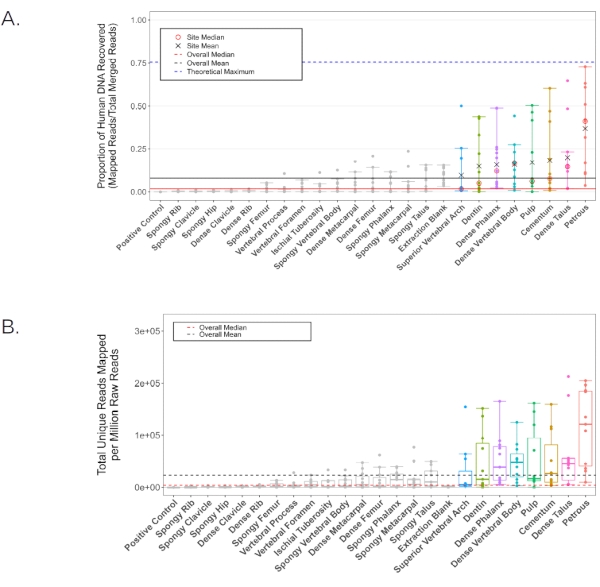
Figura 6: Conteúdo de ADN humano para todas as amostras triadas. As linhas pretas representam a média geral, enquanto as linhas vermelhas representam a mediana (sólido: proporção do DNA humano, tracejado: leituras humanas mapeadas por milhão de leituras geradas). Locais de amostragem anatômica individuais com uma proporção média de DNA humano maior do que a média geral (8,16%) são coloridos em todas as análises. (A) A proporção de leituras mapeadas para o genoma de referência hg19. A linha tracejada azul representa o máximo teórico dado os parâmetros de mapeamento da tubulação (gerados usando Gargammel31 para simular uma distribuição aleatória de 5.000.000 de leituras do genoma de referência hg19 com danos simulados). Médias individuais (X preto) e medianas (círculo vermelho) são relatadas para as amostras com uma proporção média de DNA humano maior do que a média geral. Os intervalos de confiança indicam limites superiores e inferiores, excluindo valores atípicos estatísticos. (B) O número de leituras únicas mapeadas para o genoma de referência hg19 por milhão de leituras de esforço de sequenciamento (extremidade emparelhada de 75 pb). Os intervalos de confiança indicam limites superiores e inferiores, excluindo valores atípicos estatísticos. Este número foi adaptado de Parker, C. et al. 202011. Por favor, clique aqui para ver uma versão maior desta figura.
Tabela 2: Níveis médios de duplicação (leituras de mapeamento/leituras únicas), comprimentos médios e medianos de fragmentos e estimativas de contaminação do cromossomo X para todos os locais de amostragem anatômica. Erro relatado como o erro padrão da média. Esta tabela foi adaptada de Parker, C. et al. 202011.
| Local de amostragem | Fator de duplicação médio (# leituras mapeadas /# leituras mapeadas exclusivas) | Comprimento médio do fragmento em pb | Proporção média estimada de contaminação do cromossomo X |
| Pirâmide petrosa | 1,188 ± 0,006 | 65,40 ± 1,36 | 0,000 ± 0,003 |
| Cimento | 1,197 ± 0,028 | 67,28 ± 1,76 | 0,011 ± 0,003 |
| Dentina | 1,188 ± 0,061 | 60,22 ± 2,37 | 0,002 ± 0,007 |
| Polpa | 1,179 ± 0,024 | 55,14 ± 2,90 | 0,013 ± 0,006 |
| Falange distal | 1,191 ± 0,049 | 65,95 ± 1,08 | 0,013 ± 0,005 |
| Corpo vertebral | 1,194 ± 0,037 | 66,14 ± 1,03 | 0,008 ± 0,003 |
| Arco vertebral superior | 1,19 ± 0,017 | 63,02 ± 1,23 | 0,021 ± 0,009* |
| Tálus | 1,198 ± 0,010 | 68,20 ± 1,24 | 0,011 ± 0,003 |
| *Amostra KRA005 removida como um outlier em 0,1952 |
Disponibilidade de código
Todos os programas de análise e módulos R utilizados nas análises deste manuscrito estão disponíveis gratuitamente junto aos seus respectivos autores. Todo o código R personalizado está disponível mediante solicitação.
Disponibilidade dos dados
Todos os dados brutos utilizados no cálculo dos resultados representativos estão disponíveis gratuitamente no repositório de dados ENA do Arquivo Europeu de Nucleotídeos (número de adesão PRJ-EB36983) ou materiais suplementares de Parker, C. et al.11.
Arquivo Suplementar 1. Clique aqui para baixar este arquivo.
Discussão
A prática atual na genética de populações humanas antigas é coletar amostras preferenciais da pars petrosa (etapa 2.1) sempre que possível. No entanto, a pars petrosa pode ser uma amostra de difícil obtenção, pois é altamente valorizada para uma miríade de avaliações esqueléticas (por exemplo, história populacional32, estimativa da idade fetal na morte33 e determinação do sexo34) e, historicamente, a amostragem da pars petrosa para análise de DNA pode ser altamente destrutiva 3,4 (incluindo o protocolo aqui apresentado, embora novos protocolos minimamente invasivos13,14 tenham sido amplamente adotados para aliviar essa preocupação). Isso é agravado pelo fato de que, até muito recentemente, um estudo sistemático em larga escala da recuperação do DNA humano em todo o esqueleto não havia sido tentado11, tornando desafiadora encontrar uma estratégia de amostragem apropriada quando a pirâmide petrosa não está disponível.
Os protocolos aqui apresentados ajudam a aliviar esse desafio, fornecendo um conjunto de procedimentos otimizados para amostragem de DNA de restos esqueléticos arqueológicos / forenses, incluindo a pars petrosa, bem como sete locais alternativos de amostragem anatômica em quatro elementos esqueléticos adicionais. As etapas críticas incluídas destinam-se a minimizar a possibilidade de perda/dano ao DNA devido a amostragem ineficiente (etapas 2.1.6 e 3.2.1.3) ou superaquecimento das amostras durante a perfuração/corte (etapa 3.1.6). Além disso, observou-se ao longo do protocolo que pode ser necessário modificar/omitir as etapas de pré-tratamento para garantir o melhor desempenho em amostras altamente degradadas. Deve-se notar também que, mesmo entre os elementos selecionados aqui apresentados, ainda existem várias técnicas alternativas de amostragem possíveis (particularmente para a pars petrosa13,14), bem como amplo espaço para otimização adicional dos locais de amostragem anatômica subexplorados aqui apresentados (ou seja, o tálus: etapa 2.5 e as vértebras: etapa 2.3).
Também é importante ter em mente que esses protocolos foram projetados e testados usando restos antigos juvenis-adultos de alta qualidade (boa preservação morfológica) para fins de análises endógenas de DNA humano. Os resultados apresentados podem não se estender a materiais mais altamente degradados, outros contextos de preservação, restos de lactentes, restos não humanos ou estudos de patógenos ou do microbioma, pois ainda é necessária uma maior exploração do uso desses protocolos em contextos adicionais. Além disso, os elementos esqueléticos alternativos aqui apresentados (dentes, vértebras, falange distal e tali) podem ser difíceis de atribuir a um único indivíduo entre restos misturados, necessitando de amostragem de vários elementos para garantir uma única origem. Apesar dessas limitações, tornar esses protocolos amplamente disponíveis pode ajudar a aliviar parte da heterogeneidade em torno da seleção e processamento de amostras, fornecendo uma estrutura generalizada e quantitativamente otimizada para uso em uma ampla gama de futuros estudos de aDNA/forenses em restos humanos.
Divulgações
Os autores não têm conflitos de interesse para relatar.
Agradecimentos
Os autores gostariam de agradecer à equipe de laboratório do Instituto Max Planck para a Ciência da História Humana por sua ajuda no desenvolvimento e implementação desses protocolos. Este trabalho não teria sido possível sem a contribuição e o trabalho árduo do Dr. Guido Brandt, da Dra. Elizabeth Nelson, de Antje Wissegot e de Franziska Aron. Este estudo foi financiado pela Sociedade Max Planck, pelo Conselho Europeu de Investigação (CEI) no âmbito do programa de investigação e inovação Horizonte 2020 da União Europeia ao abrigo dos contratos de subvenção n.º 771234 - PALEoRIDER (WH, ABR) e da Subvenção de Início n.º 805268 CoDisEASe (para o KIB).
Materiais
| Name | Company | Catalog Number | Comments |
| #16 Dental Drill Bit | NTI | H1-016-HP | example drilling bit |
| 0.6 mm scroll saw blade | Fisher Scientific | 50-949-097 | blade for Jewellers Saw |
| 22mm diamond cutting wheel | Kahla | SKU 806 104 358 514 220 | Dremel cutting attachment |
| Commercial Bleach | Fisher Scientific | NC1818018 | |
| Control Company Ultra-Clean Supreme Aluminum Foil | Fisher Scientific | 15-078-29X | |
| DNA LoBind Tubes (2 mL) | Eppendorf | 22431048 | |
| Dremel 225-01 Flex Shaft Attachment | Dremel | 225-01 | Dremel flexible extension |
| Dremel 4300 Rotary Tool | Dremel | 4300 | Example drill |
| Dremel collet and nut kit | Dremel | 4485 | Adapters for various Dremel tool attachments/bits |
| Eagle 33 Gallon Red Biohazard Waste Bag | Fisher Scientific | 17-988-501 | |
| Eppendorf DNA LoBind 2 mL microcentrifuge tube | Fisher Scientific | 13-698-792 | |
| Ethanol (Molecular Biology Grade) | Millipore Sigma | 1.08543 | |
| FDA approved level 2 Surgical Mask | Fisher Scientific | 50-206-0397 | PPE |
| Fisherbrand Comfort Nitrile Gloves | Fisher Scientific | 19-041-171X | PPE |
| Fisherbrand Safety Glasses | Fisher Scientific | 19-130-208X | PPE |
| Granger Stationary Vise | Fisher Scientific | NC1336173 | benchtop vise |
| Invitrogen UltraPure DNase/Rnase free distilled water | Fisher Scientific | 10-977-023 | |
| Jewellers Saw | Fisher Scientific | 50-949-231 | |
| Kimwipes | Sigma-Aldritch | Z188956 | |
| Labconco Purifier Logic Biosafety cabinet | Fisher Scientific | 30-368-1101 | |
| LookOut DNA Erase | Millipore Sigma | L9042-1L | |
| Medium weighing boat | Heathrow Scientific | HS120223 | |
| MSC 10pc plier/clamp set | Fisher Scientific | 50-129-5352 | Miscellaneous clamps/vise grips for securely holding samples while drilling/cutting |
| Sartorius Quintix Semi-Micro Balance | Fisher Scientific | 14-560-019 | enclosed balance |
| Tyvek coveralls with hood | Fisher Scientific | 01-361-7X | PPE |
| Weigh paper | Heathrow Scientific | HS120116 |
Referências
- Adler, C. J., Haak, W., Donlon, D., Cooper, A. Survival and recovery of DNA from ancient teeth and bones. Journal of Archaeological Science. 38 (5), 956-964 (2011).
- Dabney, J., Meyer, M. Extraction of highly degraded DNA from ancient bones and teeth. Ancient DNA: Methods and Protocols. , 25-29 (2019).
- Palsdottir, A. H., Bläuer, A., Rannamäe, E., Boessenkool, S., Hallsson, J. Not a limitless resource: ethics and guidelines for destructive sampling of archaeofaunal remains. Royal Society Open Science. 6 (10), 191059 (2019).
- Pinhasi, R., Fernandes, D. M., Sirak, K., Cheronet, O. Isolating the human cochlea to generate bone powder for ancient DNA analysis. Nature Protocols. 14 (4), 1194-1205 (2019).
- Latham, K. E., Miller, J. J. DNA recovery and analysis from skeletal material in modern forensic contexts. Forensic Sciences Research. 4 (1), 51-59 (2019).
- Mundorff, A. Z., Bartelink, E. J., Mar-Cash, E. DNA preservation in skeletal elements from the World Trade Center disaster: Recommendations for mass fatality management. Journal of Forensic Sciences. 54 (4), 739-745 (2009).
- Gamba, C., et al. Genome flux and stasis in a five millennium transect of European prehistory. Nature Communications. 5 (1), 1-9 (2014).
- Alberti, F., et al. Optimized DNA sampling of ancient bones using Computed Tomography scans. Molecular Ecology Resources. 18 (6), 1196-1208 (2018).
- Hansen, H. B., et al. Comparing ancient DNA preservation in petrous bone and tooth cementum. PLoS ONE. 12 (1), 0170940 (2017).
- Sirak, K., et al. Human auditory ossicles as an alternative optimal source of ancient DNA. Genome Research. 30 (3), 427-436 (2020).
- Parker, C., et al. A systematic investigation of human DNA preservation in medieval skeletons. Scientific Reports. 10 (1), 18225 (2020).
- Pinhasi, R., et al. Optimal ancient DNA yields from the inner ear part of the human petrous bone. PLoS ONE. 10 (6), 0129102 (2015).
- Sirak, K. A., et al. A minimally-invasive method for sampling human petrous bones from the cranial base for ancient DNA analysis. BioTechniques. 62 (6), 283-289 (2017).
- . Minimally-invasive sampling of pars petrosa (os temporale) for ancient DNA extraction. protocols.io Available from: https://www.protocols.io/view/minimally-invasive-sampling-of-pars-petrosa-os-tem-bqd8ms9w (2020)
- Damgaard, P. B., et al. Improving access to endogenous DNA in ancient bones and teeth. Scientific Reports. 5 (1), 1-12 (2015).
- Harney, &. #. 2. 0. 1. ;., et al. A minimally destructive protocol for DNA extraction from ancient teeth. Genome Research. 31 (3), 472-483 (2021).
- Cooper, A., Poinar, H. N. Ancient DNA: Do it right or not at all. Science. 289 (5482), 1139 (2000).
- Llamas, B., et al. From the field to the laboratory: Controlling DNA contamination in human ancient DNA research in the high-throughput sequencing era. STAR: Science & Technology of Archaeological Research. 3 (1), 1-14 (2017).
- Llamas, B., et al. From the field to the laboratory: Controlling DNA contamination in human ancient DNA research in the high-throughput sequencing era. STAR: Science & Technology of Archaeological Research. 3 (1), 1-14 (2017).
- Boessenkool, S., et al. Combining bleach and mild predigestion improves ancient DNA recovery from bones. Molecular Ecology Resources. 17 (4), 742-751 (2017).
- García-Garcerà, M., et al. Fragmentation of contaminant and endogenous DNA in ancient samples determined by shotgun sequencing; Prospects for human palaeogenomics. PLoS ONE. 6 (8), 24161 (2011).
- Malmström, H., et al. More on contamination: The use of asymmetric molecular behavior to identify authentic ancient human DNA. Molecular Biology and Evolution. 24 (4), 998-1004 (2007).
- Basler, N., et al. Reduction of the contaminant fraction of DNA obtained from an ancient giant panda bone. BMC Research Notes. 10, 754 (2017).
- Kemp, B. M., Smith, D. G. Use of bleach to eliminate contaminating DNA from the surface of bones and teeth. Forensic Science International. 154 (1), 53-61 (2005).
- Korlević, P., et al. Reducing microbial and human contamination in DNA extractions from ancient bones and teeth. BioTechniques. 59 (2), 87-93 (2015).
- Dabney, J., Meyer, M. Extraction of highly degraded DNA from ancient bones and teeth. Methods in Molecular Biology. 1963, 25-29 (2019).
- Hansen, H. B., et al. Comparing ancient DNA preservation in petrous bone and tooth cementum. PLoS ONE. 12 (1), 0170940 (2017).
- Gansauge, M. -. T., et al. Single-stranded DNA library preparation from highly degraded DNA using T4 DNA ligase. Nucleic Acids Research. 45 (10), 79 (2017).
- Peltzer, A., et al. EAGER: efficient ancient genome reconstruction. Genome Biology. 17 (1), 60 (2016).
- Korneliussen, T. S., Albrechtsen, A., Nielsen, R. ANGSD: analysis of next generation sequencing data. BMC Bioinformatics. 15, 356 (2014).
- Renaud, G., Hanghøj, K., Willerslev, E., Orlando, L. Gargammel: A sequence simulator for ancient DNA. Bioinformatics. 33 (4), 577-579 (2017).
- Ponce de León, M. S., et al. Human bony labyrinth is an indicator of population history and dispersal from Africa. Proceedings of the National Academy of Sciences of the United States of America. 115 (16), 4128-4133 (2018).
- Nagaoka, T., Kawakubo, Y. Using the petrous part of the temporal bone to estimate fetal age at death. Forensic Science International. 248, 188 (2015).
- Norén, A., Lynnerup, N., Czarnetzki, A., Graw, M. Lateral angle: A method for sexing using the petrous bone. American Journal of Physical Anthropology. 128 (2), 318-323 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados