Method Article
Monitorando Cinética de Agregação de Proteínas In Vivo usando Contagem Automatizada de Inclusão em Elegans Caenorhabditis
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, é apresentado um método para a análise da cinética de agregação de proteínas no nematode Caenorhabditis elegans. Animais de uma população sincronizada por idade são imagens em diferentes momentos, seguidos pela contagem de inclusão semiatomada no CellProfiler e adaptação a um modelo matemático em Amilafit.
Resumo
A agregação de proteínas em inclusões insolúveis é uma marca registrada de uma variedade de doenças humanas, muitas das quais estão relacionadas à idade. O nematode Caenorhabditis elegans é um organismo modelo bem estabelecido que tem sido amplamente utilizado no campo para estudar a agregação e toxicidade proteica. Sua transparência óptica permite a visualização direta da agregação de proteínas por microscopia de fluorescência. Além disso, o ciclo reprodutivo rápido e a vida útil curta fazem do nematode um modelo adequado para a triagem de genes e moléculas que modulam esse processo.
No entanto, a quantificação da carga agregada em animais vivos é pouco padronizada, normalmente realizada por inclusão manual contando sob um microscópio de dissecção de fluorescência em um único ponto de tempo. Essa abordagem pode resultar em alta variabilidade entre observadores e limita a compreensão do processo de agregação. Em contraste, a agregação de proteínas semelhantes a amiloide in vitro é rotineiramente monitorada pela fluorescência thioflavvin T de forma altamente quantitativa e resolvida pelo tempo.
Aqui, um método análogo é apresentado para a análise imparcial da cinética de agregação em C. elegans vivos, utilizando um microscópio confocal de alta produtividade combinado com análise de imagem personalizada e encaixe de dados. A aplicabilidade deste método é demonstrada pelo monitoramento da formação de inclusão de uma proteína de poliglutamina (polyQ) fluorescentemente rotulada nas células musculares da parede corporal. O fluxo de trabalho de análise de imagem permite a determinação do número de inclusões em diferentes momentos, que são ajustados a um modelo matemático baseado em eventos de nucleação independentes em células musculares individuais. O método aqui descrito pode ser útil para avaliar os efeitos dos fatores de proteostase e potenciais terapêuticas para doenças de agregação de proteínas em um animal vivo de forma robusta e quantitativa.
Introdução
O acúmulo de proteínas desdobradas em depósitos insolúveis ocorre em uma ampla gama de doenças. Exemplos bem conhecidos são a agregação de amilóide-β e tau na doença de Alzheimer, α-sinucleína na doença de Parkinson, e a caça com polq expandido na doença de Huntington 1,2. O descompactamento desses polipeptídeos em fibrilas amiloides está associado à toxicidade e morte celular por mecanismos que ainda não estão claros. Elucidar os mecanismos de formação amiloide será crucial para o desenvolvimento de terapias eficazes, que atualmente não estão disponíveis.
Investigações detalhadas da formação amiloide têm sido realizadas in vitro com base em medidas de fluorescência thioflavina T, levando a uma compreensão mecanicista do processo de agregação e do efeito das moléculas inibitórias 3,4,5. No entanto, não está claro se os mesmos mecanismos de agregação se mantêm verdadeiros no complexo ambiente de células vivas e organismos. O verme nematode Caenorhabditis elegans é um organismo modelo adequado para estudar a agregação de proteínas in vivo. Tem uma anatomia relativamente simples, mas consiste em múltiplos tecidos, incluindo músculo, intestino e um sistema nervoso. É geneticamente bem caracterizado, e ferramentas para modificação genética estão prontamente disponíveis. Além disso, tem um curto tempo de geração de ~3 dias e uma vida útil total de 2-3 semanas. Como tal, a agregação de proteínas pode ser examinada ao longo da vida útil do animal em uma escala de tempo experimentalmente conveniente. Finalmente, o nematoide é opticamente transparente, permitindo o rastreamento da agregação de proteínas fluorescentes rotuladas em animais vivos.
Essas características de C. elegans foram previamente exploradas para investigar a agregação de proteínas de polq como modelo para doenças de expansão de Huntington e outras doenças de expansão do POLQ. Acima do limiar patogênico de 35-40 resíduos de glutamina, as proteínas de polq rotuladas com proteína fluorescente amarela (YFP) podem ser observadas para formar inclusões insolúveis no tecido muscular 6,7, neurônios8 e intestino 9,10. Essas características têm sido amplamente utilizadas para a triagem de genes 11,12,13 e modificadores de pequenas moléculas14 de agregação e toxicidade de proteínas.
C. elegans tem o potencial de desempenhar um papel importante na ponte entre estudos in vitro de agregação de proteínas e modelos de doenças mais complexas, como camundongos15. C. elegans é favorável ao rastreamento de drogas16 , mas também pode ser explorado para obter uma compreensão fundamental dos mecanismos moleculares de agregação de proteínas in vivo, como demonstrado recentemente17. No entanto, para ambas as aplicações, é de grande importância extrair uma medida quantitativa e reprodutível de agregação de proteínas. Aqui, isso é conseguido com o uso de um microscópio confocal de alta produtividade combinado com um pipeline de análise de imagem dedicado (Figura 1).
Protocolo
1. Crescimento de uma população sincronizada por idade de C. elegans
- Manter as cepas de C. elegans em placas de médio de crescimento de nematoide (NGM) semeadas com Escherichia coli OP50 a 20 °C de acordo com os procedimentos padrão18.
- Realize um ovo-lay sincronizado colocando 10 nematoides adultos em uma placa de NGM semeada de 6 cm com uma picareta de verme de platina. Deixe os adultos colocarem ovos por ~2 h a 20 °C antes de removê-los. Prepare 1-4 placas por cepa, dependendo da fertilidade da cepa e do número de pontos de tempo a serem tomados.
- Coloque as placas com ovos na incubadora a 20 °C. Monitore o desenvolvimento dos animais até chegarem à idade adulta.
NOTA: O dia em que os animais atingiram a idade adulta é definido aqui como o primeiro dia. Normalmente, isso é três dias depois do ovo-lay. - A partir do primeiro dia, transfira os animais para novas placas de GNL semeadas diariamente para separá-los de seus filhotes. Para compensar os animais que morrem ou são perdidos durante a transferência, transfira ~40 animais por cepa vezes o número de pontos a serem retratados (ver passo 2). Prossiga até que os animais deixem de colocar ovos fertilizados (~dia 6 da idade adulta).
NOTA: Exclua animais com ensacamento ou outros fenótipos de desenvolvimento. O ensacamento é comumente observado em cepas que expressam proteínas propensas à agregação.
2. Preparação amostral de C. elegans em uma placa multiwell
NOTA: Como o procedimento de imagem requer anestésicos que eventualmente matarão os animais, os mesmos animais não podem ser reutilizados para pontos de tempo subsequentes. Em vez disso, animais diferentes do mesmo lote sincronizado por idade são imagens em dias diferentes. Embora a maioria das cepas tenha poucas inclusões no primeiro dia, recomenda-se incluir este ponto de tempo como linha de base.
- Prepare a placa de 384 poços preenchendo o número necessário de poços com 100 μL de tampão M9 complementado com 25 mM NaN3 como anestésico. Encha um poço por cepa a ser imageado.
NOTA: O azida de sódio (NaN3) é tóxico e deve ser tratado com cuidado. - Para cada cepa, transfira 20 animais para um poço usando uma picareta de verme de platina.
NOTA: Os vermes devem ser colocados fora do gramado bacteriano antes de colocá-los no poço. Bactérias fazem com que os animais aderam à picareta de vermes, o que pode impedir sua liberação e irá nublar o conteúdo do poço. Geralmente, 20 é o número ideal de animais por poço para evitar a sobreposição entre os vermes, limitando imagens desnecessárias de espaço vazio. - Cubra a placa com a tampa para evitar a evaporação e imagem a placa dentro de 1h após a preparação.
- Repetir passos 2.1-2.3 diariamente até que um patamar constante nos números de inclusão seja atingido ou até que a maioria dos animais tenha morrido. Realize a preparação e a imagem da amostra ao mesmo tempo todos os dias para garantir intervalos de 24h.
3. Aquisição de imagem no microscópio confocal de alto rendimento
NOTA: Este experimento também pode ser configurado em um microscópio confocal de disco giratório regular com um suporte de placa multiwell. Uma câmera com um grande campo de visão é benéfica para limitar o número de telhas necessárias para serem imagens para cobrir todo o poço. Consulte a Tabela de Materiais para obter detalhes sobre o microscópio e o software utilizados neste protocolo.
- Ligue o instrumento e abra o software.
- Inicie um novo protocolo indo para Configurações de Medição | É novo. Selecione o tipo de placa multiwell correta e clique em Criar uma nova configuração de medição.
- Configure o canal para fluorescência indo para Ch 1. Estabeleça o objetivo em 10x. Selecione 488 nm como fonte de luz e BP525/25 como o filtro de emissão para imagem YFP. Defina binning para 2x2 para reduzir o tamanho do arquivo.
- Clique em Adicionar canal e selecione Brightfield como o método.
- Para adicionar uma imagem de fluorescência confocal z-stack à medição, escolha a aquisição de fluorescência 3D em Action List. Vá para Selecionar e escolha Ch 1. Para minimizar o tamanho do arquivo, defina o processamento de imagem ao máximo para que a imagem máxima de projeção seja salva em vez da pilha z completa.
- Clique em BF/Ph Acquisition | Selecione | Ch 2 para o canal Brightfield.
- Clique no botão de reprodução (procure o símbolo do triângulo de ponta direita) ao lado de Placas de Poço de Descarga e coloque a placa de 384 poços no microscópio.
- Em Aquisição de Fluorescência 3D, clique em Testar e selecione um poço contendo worms para determinar a distância de deslocamento ideal na qual os worms estão centrados corretamente. Defina distância ascendente a 50 μm, distância descendente para -50 μm e intervalo de corte para 2 μm para capturar toda a espessura dos animais na pilha z.
- Otimize o tempo de exposição para obter uma boa intensidade de sinal para todas as quatro cepas, evitando a saturação. Use o mesmo tempo de exposição para todas as cepas e pontos de tempo.
- Selecione os poços a serem imagens na configuração de varredura de placas de poço. Selecione azulejo e adquira bem.
- Salve a configuração de medição e inicie o experimento clicando em Iniciar a Medição. Para os pontos de tempo subsequentes, abra a mesma Configuração de Medição e ajuste a distância de deslocamento e os poços a serem imagens.
4. Costurando imagens de ladrilhos no ImageJ
NOTA: Esta etapa só é necessária ao usar um objetivo maior que 4x, para o qual a imagem de cada poço é adquirida como múltiplas telhas. Nesta análise, a costura das telhas é realizada utilizando-se o software livre FIJI/ImageJ19 (Figura 2). Dependendo do instrumento utilizado na etapa 3, também pode ser possível realizar a costura diretamente no software de acompanhamento.
- Baixe FIJI20 e abra-o.
- Vá para Plugins | | de costura Grade/Coleta Costura21.
- Na janela pop-up, Grade/Collection Stitching, selecione o tipo e a ordem pela qual as telhas foram coletadas. Escolha Grade: linha por linha e Right & Down.
- Na próxima janela, Costura de grade: Grade: linha por linha, Direita & Para Baixo, insira o número de telhas nas direções x e y. Para o objetivo de 10x usado aqui, escolha 2 como o tamanho da grade x, 3 como o tamanho da grade y e 0 como a sobreposição de ladrilhos.
- Clique em Procurar e selecione a pasta que contém as imagens TIFF a serem costuradas.
- Insira o nome do arquivo comum em nomes de arquivo para telhas, usando {i} na posição do número de ladrilho em cada nome do arquivo.
- Desnaça todas as caixas abaixo.
- Ligue para o plugin.
- Salve as imagens resultantes como arquivos TIFF para análise na próxima etapa.
5. Contagem automatizada de inclusão usando CellProfiler22
- Baixe e instale o software de análise de imagem de código aberto, CellProfiler23. Baixe o pipeline InclusionCounting.cpproj de github.com/sinnigelab/aggregate-quantification.
- Abra o CellProfiler e arraste o pipeline para a janela de soltar um arquivo de pipeline aqui. Clique em Sim para carregar o projeto.
- Clique no módulo de entrada Imagens e arraste as imagens costuradas para a janela Soltar arquivos e pasta aqui.
- Clique no módulo de entrada Metadata . Ajuste a expressão regular para extrair do nome do arquivo de acordo com os nomes das imagens costuradas.
- Clique no módulo de entrada NamesandTypes e ajuste Selecione os critérios de regra para combinar com os canais nos nomes dos arquivos.
NOTA: Nas configurações padrão do pipeline, os nomes dos arquivos contendo BF são reconhecidos como imagens de campo brilhante e são chamados de Worms. Os nomes dos arquivos contendo YFP são reconhecidos como imagens de fluorescência e são chamados de Fluorescência. - Clique em Exibir configurações de saída para selecionar uma pasta padrão para salvar a saída do CellProfiler.
- Clique no Modo de Teste iniciar para verificar as configurações do pipeline usando o primeiro conjunto de dados de imagem. Clique em Executar para executar todos os módulos do pipeline ou Passo para executar através do pipeline um módulo de cada vez. Para ajustar os contornos do worm no módulo EditObjectsManually , clique em Ajudar para ver as instruções e clique em Feito para continuar executando o pipeline.
NOTA: As medidas extraídas não serão exportadas enquanto estiver em modo de teste. Os parâmetros de limiar para detectar vermes e inclusões podem precisar ser ajustados com base nas cepas e ampliação utilizadas. - Clique no modo de teste de saída e analise imagens.
- Abra a pasta de saída para visualizar os arquivos de saída. Abra as imagens com o nome do arquivo original seguido de contornos para verificar se os worms e inclusões foram corretamente sobrepostos.
NOTA: O número de inclusões por worm pode ser encontrado no arquivo chamado ExpandedWormObjects. Mais informações sobre as imagens de entrada podem ser encontradas no arquivo chamado Imagem. A saída adicional pode ser selecionada no módulo ExportToSpreadsheet no pipeline.
6. Ajuste global de dados de contagem de inclusão usando AmyloFit5
NOTA: Esta etapa só pode ser realizada quando os dados para múltiplas concentrações proteicas estiverem disponíveis. Para o Q40-YFP, um conjunto de quatro cepas com diferentes níveis de superexpressão nas células musculares da parede corporal foi criado anteriormente17. Em outros casos, novas cepas devem ser geradas usando microinjeção plasmida e integração genômica24.
- Vá para a plataforma de ajuste on-line gratuita para agregação de cinética AmyloFit25. Faça login ou faça login com uma conta existente.
NOTA: Um extenso manual sobre como usar o AmyloFit pode ser acessado para obter ajuda adicional. Consulte o link no canto superior esquerdo da página web (após o login) para obter mais informações. - Para começar a usar o AmyloFit, nomeie o projeto e clique em Criar projeto. Abra o projeto clicando em Abrir e criar uma sessão dando-lhe um nome e clicando na sessão Criar & carregar.
- Clique em Adicionar dados e carregue o arquivo contendo o número médio de inclusões por animal, seguindo os requisitos de formato de dados mostrados no painel esquerdo. Clique em Carregar novos dados.
- Pule as etapas de pré-processamento, que não são necessárias para dados de contagem de inclusão, definindo número de pontos para média sobre para deslocamento de ponto zero e número de pontos para a média sobre para planalto para 0. Clique em Enviar. Repita esta etapa para cada concentração de proteína (ou seja, cada coluna no arquivo carregado).
- Selecione Personalizado no painel de modelos, digite Ncells*(1-exp(-Kcell*m**(n)*(t-1)) na caixa de equação e clique no modelo Carregar.
NOTA: Como o AmyloFit foi originalmente projetado para a análise de dados cinéticos de ensaios in vitro, um modelo personalizado deve ser carregado para analisar os dados de contagem de inclusão de C. elegans. Na equação utilizada aqui, as células N é o número de células em que a formação de inclusão ocorre, a célula K é a constante da taxa de nucleação, m a concentração de proteína intracelular, e n a ordem de reação da nucleação. - Defina os tipos de parâmetros para Global Const para células N, global fit para célula K e n, e Const para m. Definir valor das células N para 95 para células musculares da parede corporal e palpite inicial para célulaS K e n a 1. Digite os valores de m para as diferentes cepas no painel esquerdo.
NOTA: Os palpites iniciais não são relevantes para o modelo relativamente simples usado aqui. Para modelos mais complexos, é benéfico inserir uma estimativa dos valores esperados para encurtar o tempo de cálculo. - Deixe o número de lúpulos da bacia inalterado e clique em Encaixar no painel de encaixe.
- Extrair o ajuste clicando em Baixar dados e encaixar.
NOTA: Os parâmetros extraídos pelo ajuste global do modelo serão listados no canto inferior direito. Um gráfico dos dados e ajuste será gerado automaticamente no painel superior direito. Este plot pode ser extraído clicando em Download pdf e personalizado indo para Exibir opções de plot. Acélula K tem unidades de moléculas de concentração-n tempo-1 célula-1. Para comparar valores com diferentes n, a célula K pode ser convertida à taxa de nucleação em uma dada concentração de proteína, multiplicando-a com mn.
Resultados
O método descrito aqui (Figura 1) foi utilizado para analisar a cinética de agregação de uma construção composta por 40 glutaminas fundidas a YFP (Q40-YFP). A proteína é expressa sob o controle do promotor unc-54 , conduzindo expressão nas células musculares da parede corporal. Como estes são relativamente grandes e fáceis de visualizar, o uso de um objetivo de 10x é suficiente para resolver as inclusões formadas pelo Q40-YFP neste tecido. Quatro cepas (linhas A-D) foram previamente desenvolvidas expressando a proteína em diferentes extensões para avaliar a concentração-dependência da agregação de polq in vivo17.
As populações sincronizadas por idade das linhas A-D foram geradas por um ovo-lei de 2 h, seguido pela transferência diária assim que a prole atingiu a idade adulta. Do 1º ao 10º dia da idade adulta, 20 animais de cada uma das quatro cepas foram retratados em uma placa de 384 poços, usando um microscópio confocal de alta produtividade. As imagens dos poços foram adquiridas como 6 telhas, que foram costuradas juntas utilizando um plugin na ImageJ21 (Figura 2). As imagens costuradas foram posteriormente analisadas usando um pipeline personalizado cellprofiler22 (Figura 3) para quantificar o número médio de inclusão por animal para cada cepa e ponto de tempo.
Os dados foram então ajustados a um modelo matemático em AmyloFit5 (Figura 4). O modelo baseia-se no pressuposto de que cada uma das 95 células musculares da parede corporal adquire independentemente uma inclusão por um evento de nucleação que limita a taxa, seguido pelo rápido crescimento agregado17. O ajuste rendeu uma taxa de nucleação constante de 9,9 × 105 moléculas M-2.1 d-1 cell-1 e uma ordem de reação de 2,1, correspondendo a uma taxa de nucleação de 0,38 moléculas d-1 célula-1 a uma concentração de proteína intracelular de 1 mM. Duas réplicas biológicas independentes levaram a valores intimamente correspondentes para a taxa de nucleação e a ordem de reação, que estão de acordo com um estudo anterior utilizando um protocolo semelhante17 (Tabela 1).
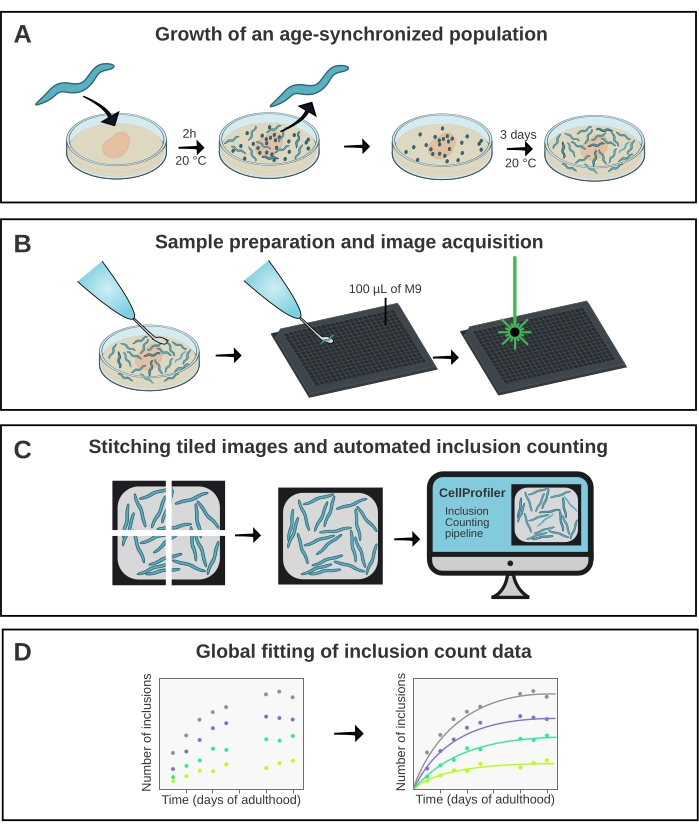
Figura 1: Visão geral esquemática do método. (A) As populações de C. elegans sincronizadas pela idade são geradas por um ovo-lay cronometrado. (B) Animais da mesma população são retratados em uma placa de 384 poços em diferentes pontos de tempo. (C) As telhas são costuradas juntas para formar imagens de todos os poços, que são analisadas no CellProfiler para quantificar os números de inclusão por animal. (D) Os dados são ajustados a um modelo matemático usando o AmyloFit. Clique aqui para ver uma versão maior desta figura.
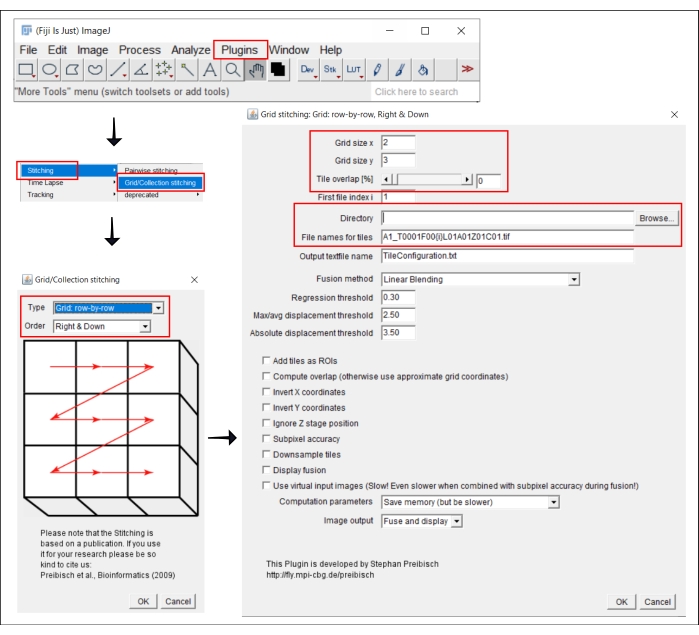
Figura 2: Capturas de tela do procedimento de costura no ImageJ usando a costura grade/coleção de plugin21. Clique aqui para ver uma versão maior desta figura.

Figura 3: Esquema do pipeline CellProfiler para quantificar números de inclusões. (A-C) A imagem de campo brilhante (A) é usada para identificar os worms (B, close-up em C). (D-G) A imagem de fluorescência (D, close-up em E) é usada para identificar as inclusões (F). Os vermes e inclusões estão relacionados para fornecer o número de inclusões para cada verme no poço (G). As imagens mostradas são de animais da linha Q40 A no terceiro dia da idade adulta. Clique aqui para ver uma versão maior desta figura.
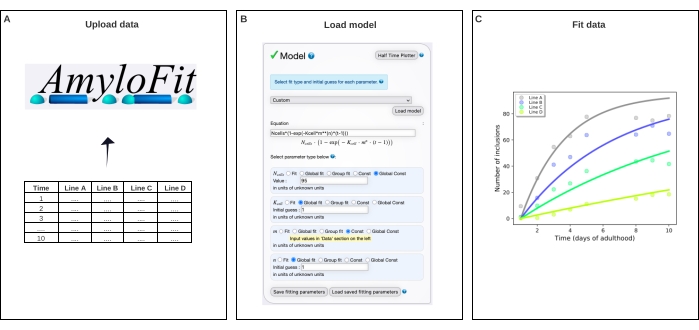
Figura 4: A montagem dos dados a um modelo matemático em AmyloFit. (A) Os dados são enviados para o AmyloFit. (B) Uma equação personalizada é inserida na formação de inclusão do modelo, assumindo eventos de nucleação independentes em cada célula. (C) Montagem da cinética de agregação para as linhas C. elegans A-D expressando diferentes níveis de Q40-YFP. Os dados são representativos de duas réplicas biológicas independentes. Clique aqui para ver uma versão maior desta figura.
| Conjunto de dados 1 | Conjunto de dados 2 | Sinnige et al.17 | |
| n | 2.1 | 1.9 | 1.6 |
| Célula K (moléculas M-n d-1 célula-1) | 9,9 x 105 | 1,4 x 105 | 3.1 x 104 |
| Taxa de nucleação em 1 mM (moléculas d-1 célula-1) | 0.38 | 0.21 | 0.35 |
Tabela 1: Valores da taxa de nucleação e ordem de reação da agregação Q40-YFP. Dados para duas réplicas biológicas independentes do protocolo e comparação com dados publicados anteriormente17.
Discussão
O método aqui apresentado facilita uma análise imparcial e quantitativa da cinética de agregação de proteínas no organismo modelo C. elegans. Depende de quatro elementos-chave (Figura 1): 1) manter uma população sincronizada por idade de nematoides; 2) microscopia de fluorescência em placas multiwell; 3) contagem automatizada de inclusão no CellProfiler; 4) encaixe de dados no AmyloFit. Em comparação com a contagem manual de inclusões em animais em movimento livre ou de imagens salvas26, a quantificação no CellProfiler é mais rápida e imparcial. O outro avanço fundamental do protocolo é a aquisição de dados cinéticos, em vez de pontos de tempo únicos, que fornece insights quantitativos sobre o mecanismo de agregação ao encaixar os dados em um modelo matemático.
Os quatro elementos do protocolo podem ser usados como módulos independentes que podem ser modificados dependendo da aplicação. Populações sincronizadas por idade também podem ser mantidas usando 5-fluoro-2'-desoxyuridina (FUDR) para esterilizar os animais. Este composto afeta a vida útil e a proteostase 24,25 e é altamente cancerígeno para o experimentador; no entanto, impede a transferência manual dos vermes, que podem ser intensivos em mão-de-obra quando grandes números são tratados. Outras alternativas são o uso de mutantes estéreis29 ou dispositivos de filtragem para separar a prole30.
A etapa de microscopia de fluorescência também pode ser ajustada, por exemplo, usando ampliações mais altas para monitorar a agregação de proteínas nos neurônios. A microscopia widefield pode ser suficiente para monitorar a agregação de polq em células musculares quando a diferença relativa entre as condições é mais importante do que o número absoluto de inclusões. O pipeline CellProfiler ainda pode ser usado nesses casos, embora as configurações para reconhecer worms e inclusões precisem ser ajustadas pelo usuário. Atualmente, o rendimento da técnica é limitado pela necessidade de colheita manual dos animais na placa de 384 poços. Isso pode ser potencialmente corrigido pelo uso de dispositivos microfluidos16. O azida de sódio é um anestésico relativamente severo, que pode ser substituído pela imobilização física por hidrogéis ou contas28,29.
A análise em Amilado apresentado aqui é baseada em um mecanismo de agregação composto por eventos de nucleação independentes em células individuais. Nos casos em que esse modelo não se encaixa, o usuário deve considerar uma alternativa como o modelo de agregação cooperativa desenvolvido anteriormente17. Uma limitação dessa abordagem é que as cepas que expressam a proteína do interesse em diferentes concentrações precisam estar disponíveis, embora estas possam ser geradas usando métodos de rotina C. elegans 24.
Ao todo, este protocolo fornece os meios para a obtenção de dados de alta qualidade para a cinética de agregação de proteínas em um sistema modelo in vivo , permitindo uma análise detalhada dos mecanismos de agregação17. Embora o método tenha sido demonstrado para agregação de poliq no tecido muscular C. elegans , futuras aplicações do protocolo podem incluir outras proteínas e tecidos e os efeitos de fatores de proteostase e pequenas moléculas.
Divulgações
Os autores não têm conflitos de interesse para divulgar.
Agradecimentos
Agradecemos ao laboratório Morimoto por cepas C. elegans e Esmeralda Bosman por assistência no microscópio confocal de alto rendimento. Este trabalho foi financiado por uma bolsa inicial da Universidade de Utrecht para t.S.
Materiais
| Name | Company | Catalog Number | Comments |
| 384-well plate | Greiner | 781091 | Black with flat clear bottom |
| AmyloFit | Knowles lab | v2.0 | Access at www.amylofit.ch.cam.ac.uk |
| C. elegans Q40 line A | Morimoto lab | AM1228 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line B | Morimoto lab | AM1229 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line C | Morimoto lab | AM1230 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line D | Morimoto lab | AM1231 | Genotype rmIs404[unc-54p::Q40::YFP] |
| CellProfiler | Broad Institute | 4.1.3 | Downloaded from https://cellprofiler.org |
| E. coli OP50 | Caenorhabditis Genetics Center (CGC) | OP50 | |
| FIJI | Open-source | (Fiji Is Just) ImageJ v2.1/1.5.3j | Downloaded from https://imagej.net/software/fiji/ |
| High-throughput confocal microscope | Yokogawa | CellVoyager CV7000S | |
| M9 buffer | Home-made | 3 g/L KH2PO4, 6 g/L Na2HPO4, 0.5 g/L NaCl, 1 mM MgSO4 | |
| NGM plates | Home-made | 17 g/L agar, 2.5 g/L bacto-peptone, 3 g/L NaCl, 25 mM KPO4 buffer pH 6.0, 1 mM MgSO4, 1 mM CaCl2, 5 mg/L cholesterol | |
| Pasteur pipette | WU Mainz | 250 | To make worm pick, 150 mm length |
| Platinum iridium wire | Alfa Aesar | 39383 | To make worm pick, 0.25 mm diameter |
| Sodium azide | Sigma-Aldrich | S2002 | |
| Stereomicroscope | Leica | S9 |
Referências
- Knowles, T. P. J., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nature Reviews. Molecular Cell Biology. 15 (6), 384-396 (2014).
- Chiti, F., Dobson, C. M. Protein misfolding, amyloid formation, and human disease: a summary of progress over the last decade. Annual Review of Biochemistry. 86 (1), 27-68 (2017).
- Knowles, T. P. J., et al. An analytical solution to the kinetics of breakable filament assembly. Science. 326 (5959), 1533-1537 (2009).
- Cohen, S. I. A., et al. Proliferation of amyloid-β42 aggregates occurs through a secondary nucleation mechanism. Proceedings of the National Academy of Sciences of the United States of America. 110 (24), 9758-9763 (2013).
- Meisl, G., et al. Molecular mechanisms of protein aggregation from global fitting of kinetic models. Nature Protocols. 11 (2), 252-272 (2016).
- Satyal, S. H., et al. Polyglutamine aggregates alter protein folding homeostasis in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 97 (11), 5750-5755 (2000).
- Morley, J. F., Brignull, H. R., Weyers, J. J., Morimoto, R. I. The threshold for polyglutamine-expansion protein aggregation and cellular toxicity is dynamic and influenced by aging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 99 (16), 10417-10422 (2002).
- Brignull, H. R., Moore, F. E., Tang, S. J., Morimoto, R. I. Polyglutamine proteins at the pathogenic threshold display neuron-specific aggregation in a pan-neuronal Caenorhabditis elegans model. Journal of Neuroscience. 26 (29), 7597-7606 (2006).
- Moronetti Mazzeo, L. E., Dersh, D., Boccitto, M., Kalb, R. G., Lamitina, T. Stress and aging induce distinct polyQ protein aggregation states. Proceedings of the National Academy of Sciences of the United States of America. 109 (26), 10587-10592 (2012).
- Prahlad, V., Morimoto, R. I. Neuronal circuitry regulates the response of Caenorhabditis elegans to misfolded proteins. Proceedings of the National Academy of Sciences of the United States of America. 108 (34), 14204-14209 (2011).
- Nollen, E. A. A. Genome-wide RNA interference screen identifies previously undescribed regulators of polyglutamine aggregation. Proceedings of the National Academy of Sciences of the United States of America. 101 (17), 6403-6408 (2004).
- Silva, M. C., et al. A genetic screening strategy identifies novel regulators of the proteostasis network. PLoS Genetics. 7 (12), 1002438 (2011).
- Brehme, M., et al. A chaperome subnetwork safeguards proteostasis in aging and neurodegenerative disease. Cell Reports. 9, 1-16 (2014).
- Calamini, B., et al. Small-molecule proteostasis regulators for protein conformational diseases. Nature Chemical Biology. 8 (2), 185-196 (2012).
- Sinnige, T., Stroobants, K., Dobson, C. M., Vendruscolo, M. Biophysical studies of protein misfolding and aggregation in in vivo models of Alzheimer's and Parkinson's diseases. Quarterly Reviews of Biophysics. 49, 22 (2020).
- Mondal, S., et al. Large-scale microfluidics providing high-resolution and high-throughput screening of Caenorhabditis elegans poly-glutamine aggregation model. Nature Communications. 7, 13023 (2016).
- Sinnige, T., et al. Kinetic analysis reveals that independent nucleation events determine the progression of polyglutamine aggregation in C. elegans. Proceedings of the National Academy of Sciences of the United States of America. 118 (11), 202188118 (2021).
- Brenner, S. Caenorhabditis elegans. Methods. 77 (1), 71-94 (1974).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- . FIJI/ImageJ Available from: https://imagej.net/downloads (2012)
- Preibisch, S., Saalfeld, S., Tomancak, P. Globally optimal stitching of tiled 3D microscopic image acquisitions. Bioinformatics. 25 (11), 1463-1465 (2009).
- Lamprecht, M. R., Sabatini, D. M., Carpenter, A. E. CellProfiler: free, versatile software for automated biological image analysis. BioTechniques. 42 (1), 71-75 (2007).
- CellProfiler. Broad Institute Available from: https://cellprofiler.org/releases (2021)
- Mello, C. C., Kramer, J. M., Stinchcomb, D., Ambros, V. Efficient gene transfer in C. elegans: extrachromosomal maintenance and integration of transforming sequences. EMBO Journal. 10 (12), 3959-3970 (1991).
- . Knowles group, University of Cambridge Available from: https://amylofit.com/amylofitmain/login/ (2021)
- Lazaro-Pena, M. I., Cornwell, A. B., Samuelson, A. V. Quantifying tissue-specific proteostatic decline in Caenorhabditis elegans. Journal of Visualized Experiments: JoVE. (175), (2021).
- Feldman, N., Kosolapov, L., Ben-Zvi, A. Fluorodeoxyuridine improves Caenorhabditis elegans proteostasis independent of reproduction onset. PLoS One. 9 (1), 85964 (2014).
- Brunquell, J., Bowers, P., Westerheide, S. D. Fluorodeoxyuridine enhances the heat shock response and decreases polyglutamine aggregation in an HSF-1-dependent manner in Caenorhabditis elegans. Mechanisms of Ageing and Development. 141-142, 1-4 (2014).
- David, D. C., et al. Widespread protein aggregation as an inherent part of aging in C. elegans. PLoS Biology. 8 (8), 47-48 (2010).
- Hunter, S., Maulik, M., Scerbak, C., Vayndorf, E., Taylor, B. E. Caenorhabditis sieve: A low-tech instrument and methodology for sorting small multicellular organisms. Journal of Visualized Experiments: JoVE. (137), (2018).
- Burnett, K., Edsinger, E., Albrecht, D. R. Rapid and gentle hydrogel encapsulation of living organisms enables long-term microscopy over multiple hours. Communications Biology. 1, 73 (2018).
- Dong, L., et al. Reversible and long-term immobilization in a hydrogel-microbead matrix for high-resolution imaging of Caenorhabditis elegans and other small organisms. PLoS One. 13 (3), 0193989 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados