É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Caracterização Viscoelástica de Fantasmas de Gelatina Imitadores de Tecidos Moles usando Recuo e Elastografia por Ressonância Magnética
Neste Artigo
Resumo
Este artigo apresenta uma demonstração e um resumo dos protocolos de confecção de simuladores de gelatina que imitam tecidos moles e a caracterização viscoelástica correspondente usando indentação e elastografia por ressonância magnética.
Resumo
A caracterização das propriedades biomecânicas dos tecidos biológicos moles é importante para entender a mecânica dos tecidos e explorar os mecanismos relacionados à biomecânica da doença, lesão e desenvolvimento. O método de ensaio mecânico é a forma mais simples de caracterização tecidual e é considerado como verificação para medição in vivo. Entre as muitas técnicas de teste mecânico ex vivo , o teste de recuo fornece uma maneira confiável, especialmente para amostras pequenas, difíceis de fixar e viscoelásticas, como o tecido cerebral. A elastografia por ressonância magnética (MRE) é um método clinicamente utilizado para medir as propriedades biomecânicas dos tecidos moles. Com base na propagação da onda de cisalhamento em tecidos moles registrada usando MRE, as propriedades viscoelásticas dos tecidos moles podem ser estimadas in vivo com base na equação de onda. Aqui, as propriedades viscoelásticas de simuladores de gelatina com duas concentrações diferentes foram medidas por ERM e recuo. Os protocolos de fabricação, teste e estimativa de módulo fantasma foram apresentados.
Introdução
A maioria dos tecidos biológicos moles parece ter propriedades viscoelásticas que são importantes para entender sua lesão e desenvolvimento 1,2. Além disso, as propriedades viscoelásticas são biomarcadores importantes no diagnóstico de uma variedade de doenças, como fibrose e câncer 3,4,5,6. Portanto, a caracterização das propriedades viscoelásticas dos tecidos moles é crucial. Dentre as muitas técnicas de caracterização utilizadas, o teste mecânico ex vivo de amostras de tecido e a elastografia in vivo utilizando imagens biomédicas são os dois métodos amplamente utilizados.
Embora várias técnicas de testes mecânicos tenham sido usadas para caracterização de tecidos moles, os requisitos para o tamanho da amostra e as condições de teste não são fáceis de serem satisfeitos. Por exemplo, o teste de cisalhamento precisa ter amostras fixadas firmemente entre as placas de cisalhamento7. O teste biaaxial é mais adequado para tecido de membrana e tem requisitos específicos de pinçamento 8,9. Um teste de compressão é comumente usado para testes de tecido, mas não pode caracterizar posições específicas dentro de uma amostra10. O teste de recuo não tem requisitos adicionais para fixar a amostra de tecido e pode ser usado para medir muitas amostras de tecido biológico, como o cérebro e o fígado. Além disso, com uma pequena cabeça de indenter, as propriedades regionais dentro de uma amostra poderiam ser testadas. Portanto, testes de recuo têm sido adotados para testar uma variedade de tecidos moles 1,3,11.
Caracterizar as propriedades biomecânicas dos tecidos moles in vivo é importante para estudos translacionais e aplicações clínicas da biomecânica. Modalidades de imagem biomédica, como ultrassom (US) e ressonância magnética (MR), são as técnicas mais utilizadas. Embora a imagem da US seja relativamente barata e fácil de realizar, ela sofre de baixo contraste e é difícil medir órgãos como o cérebro. Capaz de obter imagens de estruturas profundas, a elastografia por RM (EMR) pôde medir uma variedade de tecidos moles 6,12, especialmente o cérebro13,14. Com a vibração externa aplicada, a ERM poderia medir as propriedades viscoelásticas dos tecidos moles em uma frequência específica.
Estudos têm demonstrado que em 50-60 Hz, o módulo de cisalhamento do cérebro normal é ~1,5-2,5kPa 5,6,13,14,15 e ~2-2,5 kPa para fígado normal 16. Portanto, simuladores de gelatina que possuem propriedades biomecânicas semelhantes têm sido amplamente utilizados para mimetizar tecidos moles para teste e validação17,18,19. Neste protocolo, simuladores de gelatina com duas concentrações diferentes foram preparados e testados. As propriedades viscoelásticas dos simuladores de gelatina foram caracterizadas utilizando-se um dispositivo eletromagnético de ERM customizado14 e um dispositivo de recuo 1,3. Os protocolos de teste podem ser usados para testar muitos tecidos moles, como o cérebro ou o fígado.
Protocolo
1. Preparação fantasma de gelatina
- Pesar gelatina, glicerol e água de acordo com a Tabela 1. Misture o pó de gelatina com água para obter a solução de gelatina.
NOTA: As concentrações dos componentes individuais para a preparação dos dois simuladores são apresentadas no quadro 1. Quanto maior a concentração de gelatina, mais rígido o fantasma. - Aqueça a solução de gelatina a 60 °C em banho-maria. Adicione glicerol à solução de gelatina, mantendo a temperatura.
NOTA: O glicerol estabiliza as misturas de gelatina aumentando a sua temperatura de fusão e módulo de cisalhamento17. - Agitar a solução e aquecê-la novamente a 60 °C. Despeje a solução misturada em um recipiente que será usado para testes de ERM e recuo. Arrefecer a solução à temperatura ambiente e esperar até que a solução esteja solidificada.
2. Teste de ERM
- Coloque a placa de vibração em cima do simulador de gelatina. Certifique-se de que o contato entre o simulador e a placa de vibração é firme (Figura 1A).
NOTA: A placa de vibração é feita de poliamida com uma dimensão de 50 x 50 x 5 mm3. - Coloque o fantasma de gelatina na bobina da cabeça. Coloque esponjas e sacos de areia ao redor do fantasma de gelatina para se certificar de que o fantasma está firmemente colocado. Use um atuador eletromagnético personalizado com uma barra de transmissão14,18. Monte um atuador eletromagnético na bobina da cabeça. Conecte a barra de transmissão à placa de vibração (Figura 1B).
- Conecte as linhas de alimentação do atuador com o amplificador. Conecte as linhas de controle com o controlador (Figura 1C).
- Configurações de parâmetros de varredura do atuador e da ressonância magnética
- Defina a forma de onda, a frequência de vibração e a amplitude no gerador de funções. Defina a amplitude de vibração desejada ajustando o amplificador de potência.
NOTA: Aqui, a forma de onda é definida como sinusoidal no gerador de funções; a frequência de vibração é ajustada para 40 Hz ou 50 Hz, e a amplitude é ajustada para 1,5 Vpp. No amplificador de potência, a taxa de amplificação é ajustada para 40%. - Defina o gerador de funções para funcionar no modo de disparo. Conecte a linha de gatilho à porta de disparo externa da máquina de ressonância magnética.
- Defina a frequência de varredura (atuador) MRE da mesma forma que a do gerador de funções, de modo que o gradiente de codificação de movimento seja sincronizado com o movimento da placa de vibração.
- Defina a forma de onda, a frequência de vibração e a amplitude no gerador de funções. Defina a amplitude de vibração desejada ajustando o amplificador de potência.
- Medição e análise de dados
- Siga os procedimentos de posicionamento de imagem de rotina. Use uma sequência de ERM baseada em gradiente-eco 2D (GRE) para imagens do fantasma de gelatina20. Defina os parâmetros de imagem do GRE-MRE da seguinte forma: Flip-angle = 30°; TR/TE = 50/31 ms; Campo de visão = 300 mm; Espessura da fatia = 5 mm; Tamanho do Voxel = 2,34 x 2,34 mm2.
- Meça as imagens de fase em quatro pontos temporais em um ciclo sinusoidal. Aplique gradientes de codificação de movimento positivo e negativo em cada ponto de tempo.
- Com base na imagem de fase adquirida, remova a fase de fundo subtraindo as imagens de fase codificadas positivas e negativas. Desembrulhe a fase com um algoritmo baseado em classificação de confiabilidade21.
- Extraia o componente principal do movimento aplicando a Transformada de Fourier rápida às imagens de fase desencapsuladas. Filtre a imagem de fase com um filtro passa-banda digital. Estimar o módulo de cisalhamento com um algoritmo de inversão direta (DI) 2D para obter o módulo de armazenamento G' e o módulo de perda G''13,14.
NOTA: A frequência de corte do filtro passa-banda é [0,04 0,08]. O tamanho da janela de ajuste do algoritmo DI é 11 x 11.
3. Ensaio de recuo
- Use um punção circular ou lâmina cirúrgica para aparar o fantasma de gelatina em uma amostra cilíndrica ou cuboide, respectivamente. Certifique-se de que a espessura da amostra está entre 3 e 10 mm e o diâmetro da amostra cilíndrica ou do lado longo do cuboide é maior que 4 mm. Use uma lâmina afiada para aparar a superfície da amostra para torná-la o mais suave possível para o recuo.
- Ligue a alimentação do testador de recuo. Execute o seguinte usando o programa de controle indenter projetado para automatizar o procedimento de contato indenter (programa personalizado; consulte Tabela de materiais).
- Clique no botão Voltar para fora na GUI para inicializar o processo de calibração (Figura 2B). Leia o valor do sensor a laser e digite o valor na caixa Linha de Base .
NOTA: Durante o processo de calibração, a distância entre o sensor laser e a placa defletora é ajustada para um valor específico pré-definido. - Coloque uma lâmina de vidro na placa defletora e registre o valor mostrado pelo sensor a laser. Em seguida, coloque a amostra na lâmina de vidro e coloque-as juntas na placa defletora. Leia o valor do sensor a laser e digite esse valor na caixa Amostra+Slide .
NOTA: O sensor a laser é usado para registrar o deslocamento do recuo, mas também é usado para medir a espessura da amostra antes do teste. - Tomemos a diferença entre os dois valores obtidos na etapa 3.2.2 como a espessura da amostra na região de interesse (ROI).
- Coloque cuidadosamente a amostra junto com a lâmina de vidro subjacente logo abaixo do indenter e, em seguida, clique no botão Contato para iniciar o contato automático entre o indenter e a superfície da amostra.
NOTA: Se o contato automático não for satisfatório, ou seja, o indenter pressionar profundamente a amostra ou não tiver um contato, ajuste a posição do indenter digitando um valor no intervalo de 0,05-0,1 mm na caixa Offset e repita as etapas 1.2.1-1.2.4. - Com base na espessura da amostra medida (etapa 3.2.3), estime o deslocamento do recuo (ou seja, a profundidade total do recuo) multiplicando a espessura pela deformação de teste recuada (aqui, é definido como ≤8% para manter o recuo dentro da suposição de pequena deformação).
- Digite os valores de deslocamento (etapa 3.2.5) na caixa Deslocamento (mm ). Defina o tempo de relaxamento para 180 s na caixa Tempo de Morada . Clique no botão Recuo . O deslocamento e a força reativa durante o procedimento de retenção de rampa serão automaticamente registrados e salvos em um arquivo no Caminho do Arquivo especificado.
Observação : O caminho do arquivo pode ser predefinido como o caminho para salvar dados de teste.
- Clique no botão Voltar para fora na GUI para inicializar o processo de calibração (Figura 2B). Leia o valor do sensor a laser e digite o valor na caixa Linha de Base .
- Exporte os dados de recuo para uma planilha. Use uma série de Prony de dois termos
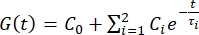 para ajustar a curva de relaxamento de força 1,3,11:
para ajustar a curva de relaxamento de força 1,3,11: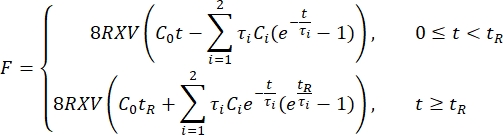
- Estimar o módulo de cisalhamento instantâneo (G 0) e o módulo de cisalhamento a longo prazo (G∞) com base nos parâmetros ajustados:
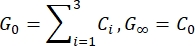
NOTA: Nas equações acima, C 0, C i e τi são parâmetros de modelo da série de Prony, F é a força de recuo, R é o raio do indenter, X é o fator de compensação para a suposição de meio espaço infinito, V é a velocidade de recuo, t é a variável de tempo e t R é o tempo de rampa.
Resultados
Seguindo o protocolo de ERM, observou-se uma clara propagação de onda de cisalhamento nos simuladores de gelatina a 40 e 50 Hz (Figura 3). As propriedades viscoelásticas medidas a partir do MRE e os testes de indentação são mostrados na Figura 4. Os valores estimados de G' e G" em cada teste para cada fantasma estão resumidos na Tabela 2. Seguindo o protocolo de recuo, as propriedades viscoelásticas de cada simulador em cada ponto de tes...
Discussão
Os simuladores de gelatina são comumente utilizados como materiais que imitam tecidos para testes e validação de algoritmos e dispositivos 17,19,22,23,24,25,26,27. Um dos estudos pioneiros utilizando o simulador de gelatina para comparar a ERM e o teste ...
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
O apoio financeiro da Fundação Nacional de Ciências Naturais da China (subsídio 31870941), da Fundação de Ciências Naturais de Xangai (subsídio 22ZR1429600) e da Comissão de Ciência e Tecnologia do Município de Xangai (subsídio 19441907700) é reconhecido.
Materiais
| Name | Company | Catalog Number | Comments |
| 24-channel head & Neck coil | United Imaging Healthcare | 100120 | Equipment |
| 3T MR Scanner | United Imaging Healthcare | uMR 790 | Equipment |
| Acquisition board | Advantech Co | PCI-1706U | Equipment |
| Computer-Windows | HP | 790-07 | Equipment |
| Electromagnetic actuator | Shanghai Jiao Tong University | Equipment | |
| Function generator | RIGOL | DG1022Z | Equipment |
| Gelatin | CARTE D’OR | Reagent | |
| Glycerol | Vance Bioenergy Sdn.Bhd | Reagent | |
| Indenter control program | custom-designed | Software; accessed via: https://github.com/aaronfeng369/FengLab_indentation_code. | |
| Laser sensor | Panasonic | HG-C1050 | Equipment |
| Load cell | Transducer Technique | GSO-10 | Equipment |
| MATLAB | Mathworks | Software | |
| Power amplifier | Yamaha | A-S201 | Equipment |
| Voice coil electric motor | SMAC Corporation | DB2583 | Equipment |
Referências
- Qiu, S., et al. Viscoelastic characterization of injured brain tissue after controlled cortical impact (CCI) using a mouse model. Journal of Neuroscience Methods. 330, 108463 (2020).
- Garcia, K. E., et al. Dynamic patterns of cortical expansion during folding of the preterm human brain. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 3156-3161 (2018).
- Qiu, S., et al. Characterizing viscoelastic properties of breast cancer tissue in a mouse model using indentation. Journal of Biomechanics. 69, 81-89 (2018).
- Yin, Z., et al. A new method for quantification and 3D visualization of brain tumor adhesion using slip interface imaging in patients with meningiomas. European Radiology. 31 (8), 5554-5564 (2021).
- Streitberger, K. -. J., et al. How tissue fluidity influences brain tumor progression. Proceedings of the National Academy of Sciences of the United States of America. 117 (1), 128 (2020).
- Bunevicius, A., Schregel, K., Sinkus, R., Golby, A., Patz, S. REVIEW: MR elastography of brain tumors. NeuroImage: Clinical. 25, 102109 (2020).
- Namani, R., et al. Elastic characterization of transversely isotropic soft materials by dynamic shear and asymmetric indentation. Journal of Biomechanical Engineering. 134 (6), 061004 (2012).
- Potter, S., et al. A novel small-specimen planar biaxial testing system with full in-plane deformation control. Journal of Biomechanical Engineering. 140 (5), 0510011 (2018).
- Zhang, W., Feng, Y., Lee, C. -. H., Billiar, K. L., Sacks, M. S. A generalized method for the analysis of planar biaxial mechanical data using tethered testing configurations. Journal of Biomechanical Engineering. 137 (6), 064501 (2015).
- Delaine-Smith, R. M., Burney, S., Balkwill, F. R., Knight, M. M. Experimental validation of a flat punch indentation methodology calibrated against unconfined compression tests for determination of soft tissue biomechanics. Journal of the Mechanical Behavior of Biomedical Materials. 60, 401-415 (2016).
- Chen, Y., et al. Comparative analysis of indentation and magnetic resonance elastography for measuring viscoelastic properties. Acta Mechanica Sinica. 37 (3), 527-536 (2021).
- Garteiser, P., Doblas, S., Van Beers, B. E. Magnetic resonance elastography of liver and spleen: Methods and applications. NMR in Biomedicine. 31 (10), 3891 (2018).
- Arani, A., Manduca, A., Ehman, R. L., Huston Iii, J. Harnessing brain waves: a review of brain magnetic resonance elastography for clinicians and scientists entering the field. British Journal of Radiolology. 94 (1119), 20200265 (2021).
- Qiu, S., et al. An electromagnetic actuator for brain magnetic resonance elastography with high frequency accuracy. NMR in Biomedicine. 34 (12), 4592 (2021).
- Hiscox, L. V., et al. Standard-space atlas of the viscoelastic properties of the human brain. Human Brain Mapping. 41 (18), 5282-5300 (2020).
- Seyedpour, S. M., et al. Application of magnetic resonance imaging in liver biomechanics: A systematic review. Frontiers in Physiology. 12, 733393 (2021).
- Okamoto, R. J., Clayton, E. H., Bayly, P. V. Viscoelastic properties of soft gels: comparison of magnetic resonance elastography and dynamic shear testing in the shear wave regime. Physics in Medicine and Biology. 56 (19), 6379-6400 (2011).
- Feng, Y., et al. A multi-purpose electromagnetic actuator for magnetic resonance elastography. Magnetic Resonance Imaging. 51, 29-34 (2018).
- Zeng, W., et al. Nonlinear inversion MR elastography with low-frequency actuation. IEEE Transactions on Medical Imaging. 39 (5), 1775-1784 (2020).
- Wang, R., et al. Fast magnetic resonance elastography with multiphase radial encoding and harmonic motion sparsity based reconstruction. Physics in Medicine and Biology. 67 (2), (2022).
- Herraez, M. A., Burton, D. R., Lalor, M. J., Gdeisat, M. A. Fast two-dimensional phase-unwrapping algorithm based on sorting by reliability following a noncontinuous path. Applied Optics. 41 (35), 7437-7444 (2002).
- Gordon-Wylie, S. W., et al. MR elastography at 1 of gelatin phantoms using 3D or 4D acquisition. Journal of Magnetic Resonance. 296, 112-120 (2018).
- McGarry, M., et al. Uniqueness of poroelastic and viscoelastic nonlinear inversion MR elastography at low frequencies. Physics in Medicine and Biology. 64 (7), 075006 (2019).
- Zampini, M. A., Guidetti, M., Royston, T. J., Klatt, D. Measuring viscoelastic parameters in Magnetic Resonance Elastography: a comparison at high and low magnetic field intensity. Journal of the Mechanical Behavior of Biomedical Materials. 120, 104587 (2021).
- Ozkaya, E., et al. Brain-mimicking phantom for biomechanical validation of motion sensitive MR imaging techniques. Journal of the Mechanical Behavior of Biomedical Materials. 122, 104680 (2021).
- Guidetti, M., et al. Axially- and torsionally-polarized radially converging shear wave MRE in an anisotropic phantom made via Embedded Direct Ink Writing. Journal of the Mechanical Behavior of Biomedical Materials. 119, 104483 (2021).
- Badachhape, A. A., et al. The relationship of three-dimensional human skull motion to brain tissue deformation in magnetic resonance elastography studies. Journal of Biomechanical Engineering. 139 (5), 0510021 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados