É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Triagem de alto rendimento de isolados microbianos com impacto na saúde de Caenorhabditis elegans
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Os micróbios intestinais podem impactar positiva ou negativamente a saúde de seu hospedeiro por meio de mecanismos específicos ou conservados. Caenorhabditis elegans é uma plataforma conveniente para rastrear esses micróbios. O presente protocolo descreve a triagem de alto rendimento de 48 isolados bacterianos quanto ao impacto na resistência ao estresse dos nematoides, usados como um proxy para a saúde dos vermes.
Resumo
Com seu tamanho pequeno, vida útil curta e genética fácil, Caenorhabditis elegans oferece uma plataforma conveniente para estudar o impacto de isolados microbianos na fisiologia do hospedeiro. Ele também fluoresce em azul ao morrer, fornecendo um meio conveniente de identificar a morte. Essa propriedade tem sido explorada para desenvolver ensaios de sobrevivência de C. elegans sem rótulo de alto rendimento (LFASS). Estes envolvem o registro de fluorescência de lapso de tempo de populações de vermes fixadas em placas de múltiplos poços, das quais o tempo médio de morte da população pode ser derivado. O presente estudo adota a abordagem LFASS para rastrear múltiplos isolados microbianos de uma só vez quanto aos efeitos sobre a suscetibilidade de C. elegans a calor severo e estresses oxidativos. Esse pipeline de triagem microbiana, que pode ser usado notavelmente para pré-rastrear probióticos, usando resistência ao estresse grave como um proxy para a saúde do hospedeiro é relatado aqui. O protocolo descreve como cultivar coleções de isolados de microbiota intestinal de C. elegans e populações de vermes síncronos em matrizes de vários poços antes de combiná-las para os ensaios. O exemplo fornecido abrange o teste de 47 isolados bacterianos e uma cepa de controle em duas cepas de vermes, em dois ensaios de estresse em paralelo. No entanto, o pipeline de abordagem é prontamente escalável e aplicável à triagem de muitas outras modalidades. Assim, ele fornece uma configuração versátil para pesquisar rapidamente uma paisagem multiparamétrica de condições biológicas e bioquímicas que afetam a saúde de C. elegans.
Introdução
O corpo humano abriga cerca de 10-100 trilhões de células microbianas vivas (bactérias, fungos archaea), que são encontradas principalmente nos ambientes intestinais, cutâneos e mucosas1. Em um estado saudável, estes fornecem benefícios ao seu hospedeiro, incluindo a produção de vitaminas, maturação do sistema imunológico, estimulação de respostas imunes inatas e adaptativas a patógenos, regulação do metabolismo da gordura, modulação das respostas ao estresse e muito mais, com impacto no crescimento e desenvolvimento, início da doença e envelhecimento 2,3,4,5 . A microbiota intestinal também evolui consideravelmente ao longo da vida. A evolução mais drástica ocorre durante a infância e a primeira infância6, mas mudanças significativas também ocorrem com a idade, incluindo uma diminuição na abundância de Bifidobacterium e um aumento nas espécies Clostridium, Lactobacillus, Enterobacteriaceae e Enterococcus 7. O estilo de vida pode alterar ainda mais a composição microbiana intestinal, levando à disbiose (perda de bactérias benéficas, crescimento excessivo de bactérias oportunistas), resultando em várias patologias, como doença inflamatória intestinal, diabetes e obesidade5, mas também contribuindo para as doenças de Alzheimer e Parkinson 8,9,10,11.
Essa constatação tem contribuído criticamente para refinar o conceito de eixo intestino-cérebro (GBA), onde as interações entre a fisiologia intestinal (agora incluindo os micróbios dentro dela) e o sistema nervoso são consideradas o principal regulador do metabolismo animal e das funções fisiológicas12. No entanto, o papel preciso da microbiota na sinalização intestino-cerebral e os mecanismos de ação associados estão longe de ser totalmente compreendidos13. Sendo a microbiota intestinal um determinante fundamental do envelhecimento saudável, a forma como as bactérias modulam o processo de envelhecimento tornou-se objeto de intensa pesquisa e controvérsia 6,14,15.
Com a demonstração de que a lombriga Caenorhabditis elegans hospeda uma microbiota intestinal genuína dominada - como em outras espécies - por Bacteroidetes, Firmicutes e Actinobacteria 16,17,18,19,20, sua rápida ascensão como plataforma experimental para estudar as interações comensais intestino-hospedeiro 21,22,23,24 ,25,26 expandiu significativamente nosso arsenal investigativo26,27,28,29. Em particular, as abordagens experimentais de alto rendimento disponíveis para C. elegans estudarem interações gene-dieta, gene-droga, gene-patógeno, etc., podem ser adaptadas para explorar rapidamente como os isolados bacterianos e coquetéis afetam a saúde e o envelhecimento de C. elegans.
O presente protocolo descreve um pipeline experimental para rastrear de uma só vez matrizes de isolados bacterianos ou misturas ajustadas em placas de múltiplos poços para efeitos sobre a resistência ao estresse de C. elegans como um proxy para a saúde, que pode ser usado para identificar probióticos. Ele detalha como cultivar grandes populações de vermes e lidar com matrizes bacterianas em formatos de placas de 96 e 384 poços antes de processar vermes para análise automatizada de resistência à tensão usando um leitor de placas de fluorescência (Figura 1). A abordagem é baseada em ensaios automatizados de sobrevivência sem rótulo (LFASS)30 que exploram o fenômeno da fluorescência da morte31, em que vermes moribundos produzem uma explosão de fluorescência azul que pode ser usada para identificar o momento da morte. A fluorescência azul é emitida por ésteres glucosilílicos de ácido antranílico armazenados em grânulos intestinais de C. elegans (um tipo de organela relacionada ao lisossomo), que estouram quando uma cascata necrótica é desencadeada no intestino do verme após a morte31.
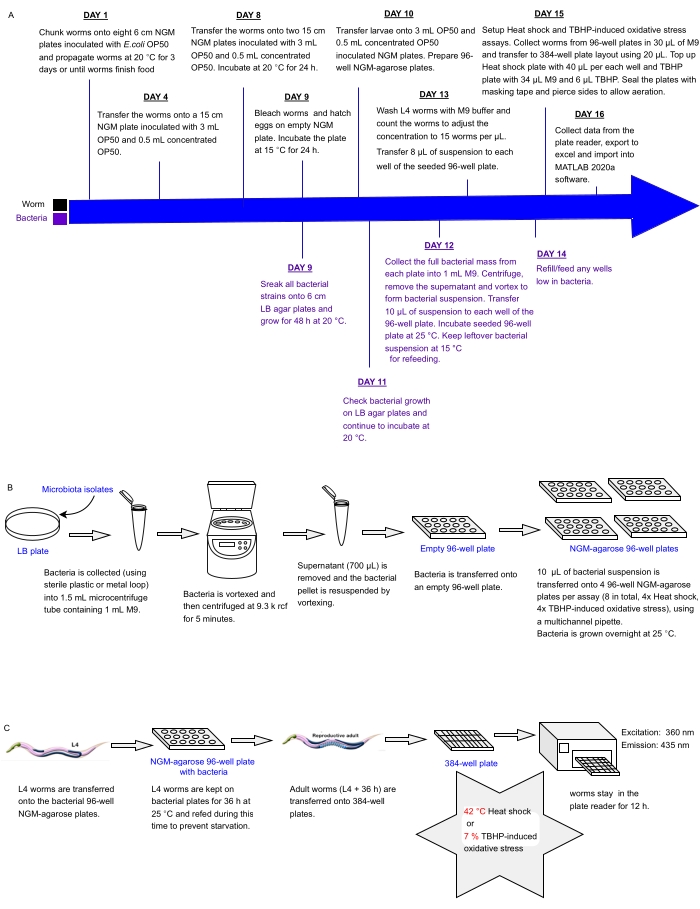
Figura 1: Fluxo de trabalho experimental para triagem de alto rendimento de isolados bacterianos com impacto na resistência de C. elegans ao estresse . (A) Linha do tempo para manutenção de vermes e bactérias e configuração de ensaios. (B) Configuração e manuseio de matriz de placas bacterianas de 96 poços. (C) Configuração da placa sem-fim de 384 poços. Por favor, clique aqui para ver uma versão maior desta figura.
Access restricted. Please log in or start a trial to view this content.
Protocolo
As duas cepas de C. elegans utilizadas em paralelo para o presente estudo foram Bristol N2 tipo silvestre e HT1890: daf-16(mgDf50), que crescem a taxas semelhantes. No entanto, o protocolo pode ser replicado com qualquer combinação de duas cepas que tenham taxas de crescimento semelhantes. Observe que, ao testar outras cepas em paralelo (por exemplo, mutantes daf-2 do tipo selvagem e de crescimento lento), diferentes taxas de crescimento devem ser consideradas e, consequentemente, o protocolo precisa ser ajustado. As escalas de tempo e quantidades de vermes e bactérias no protocolo a seguir são otimizadas para testes paralelos de 48 isolados bacterianos em duas cepas de vermes em dois ensaios LFASS em tetraplicatos. Serão necessários ajustamentos para que mais condições sejam testadas em paralelo. Escherichia coli A cepa da bactéria OP50 foi obtida do Caenorhabditis Genetics Center (CGC), Universidade de Minnesota. Os 48 isolados bacterianos foram obtidos do laboratório Schulenburg e mantidos em ágar LB.
1. C. elegans cultivando no OP50 (Dias 1 - 8)
NOTA: A abordagem atual visa cultivar hermafroditas de C. elegans em um meio sólido em todos os estágios e evita mudanças dietéticas desnecessárias (ou seja, usando cepas alternativas de E. coli de crescimento mais rápido, como NA22, ou meios de crescimento mais ricos, como placas de ovos) para permanecer o mais próximo possível das condições de crescimento padrão32,33 que ainda são amplamente utilizadas. A temperatura de crescimento do verme (aqui fixada em 15 °C) depende da(s) estirpe(ns) de C. elegans utilizada(s) e pode necessitar de ajustes (por exemplo, para evitar ou desencadear a expressão de um fenótipo ou biomarcador sensível à temperatura). Para obter informações sobre a criação de vermes, consulte a referência33.
- Preparar oito placas de NGM de 6 cm de diâmetro (10 mL de ágar meio de crescimento de nematoides, NGM, Arquivo Suplementar 1)32,33 por cepa de verme e deixá-las secar por 1 dia à temperatura ambiente.
- Preparar uma cultura líquida saturada de bactérias E. coli OP50 semeando um único clone bacteriano de uma placa de ágar caldo de lisogenia recém-cultivada (ágar LB, Arquivo Suplementar 1) em 25 mL de meio OP50 (Arquivo Suplementar 1) em um tubo cônico de 50 mL. Cultivar a cultura durante a noite a 37 °C em uma incubadora agitadora.
- Inocular as oito placas de NGM de 6 cm por cepa com 100 μL de cultura líquida saturada de E. coli OP50 por placa e manter as placas a 20 °C por 2 dias antes do uso.
- Usando um bisturi, corte e transfira um pedaço de ágar quadrado de 0,5 cm com vermes de uma placa NGM recentemente faminta em cada uma das oito placas de NGM de 6 cm inoculadas e incube essas placas a 20 °C por 3 dias (ou até que os vermes terminem o alimento).
- Preparar cinco placas de NGM de 15 cm por cepa de verme (30 mL de meio NGM por placa) e inocular com 3 mL de OP50. Deixar secar as placas antes de incubar a 37 °C durante a noite. Manter as placas a 20 °C até à sua utilização em etapas posteriores.
- Usando uma pipeta P-1000, adicione até 3 mL de tampão M9 estéril (Arquivo Suplementar 1) às placas NGM de 6 cm (Etapa 1.1.) para ressuspender os vermes e colete a solução de minhoca de todas as oito placas por cepa em um único tubo cônico de 15 mL.
- Centrífuga a 142 x g durante 2 min a 4 °C. Remova cuidadosamente o sobrenadante usando uma pipeta P-5000 ou uma bomba de água equipada com uma pipeta ou ponta Pasteur estéril. Adicione 10 mL de tampão M9 estéril para lavar o pellet de verme. Repita 2x.
- Remova o sobrenadante (tanto quanto possível) e transfira os vermes para uma placa NGM inoculada OP50 de 15 cm (Passo 1.5.) utilizando uma pipeta. Adicionar 0,5 mL de cultura concentrada de OP50.
- Para fazer OP50 concentrado, inocular cada um dos quatro frascos de 1 L de LB com 2 mL de cultura inicial de OP50 (preparados na Etapa 1.2.) e crescer em uma incubadora de agitação por 6 h a 37 °C e 160 x g. Pellet as bactérias a 3057 x g e 20 °C durante 15 min. Descarte o sobrenadante, ressuspenda os pellets bacterianos com 6 mL de meio OP50 e colete em um tubo cônico estéril de 50 mL.
NOTA: As bactérias podem ser armazenadas a 4 °C por até 1 semana.
- Cultivar cada cepa de verme em uma placa NGM de 15 cm de diâmetro por 3-4 dias a 15 °C, realimentando os vermes com 0,5 mL de OP50 concentrado diariamente.
- Uma vez que os vermes tenham quase terminado o alimento, colete e lave em tampão M9 (Etapa 1.6.1.), transfira cada cultura de cepa de verme para duas placas de NGM de 15 cm (Etapa 1.5.) e propague vermes a 20 °C até ~ 95% da população sejam adultos gravídicos (levará cerca de 24 horas para o tipo selvagem Bristol N2).
NOTA: Os adultos gravídicos são caracterizados pela presença de ovos dentro do verme, e a placa ideal também deve ter uma abundância de ovos não eclodidos colocados na placa sem muitas larvas33.
- Uma vez que os vermes tenham quase terminado o alimento, colete e lave em tampão M9 (Etapa 1.6.1.), transfira cada cultura de cepa de verme para duas placas de NGM de 15 cm (Etapa 1.5.) e propague vermes a 20 °C até ~ 95% da população sejam adultos gravídicos (levará cerca de 24 horas para o tipo selvagem Bristol N2).
2. Manutenção das coleções de isolados da microbiota intestinal (Dia 9)
- Estice os 48 isolados bacterianos em placas individuais de ágar LB de 6 cm e cresça por 48 h a 20 °C.
NOTA: As bactérias podem ser cultivadas a 25 °C durante 24-36 h, se necessário, mais cedo, mas o crescimento mais longo de 20 °C permite detectar potenciais contaminantes. - Sincronize um grande número de C. elegans.
- Alvejar vermes adultos gravídicos seguindo o método padrão de preparação de ovos33 e transferir os ovos para duas placas NGM de 15 cm não semeadas por 24 h a 15 °C para permitir que todas as larvas L1 eclodam e cresçam de forma síncrona nas etapas subsequentes.
CUIDADO: Tenha cuidado ao manusear soluções de água sanitária.
- Alvejar vermes adultos gravídicos seguindo o método padrão de preparação de ovos33 e transferir os ovos para duas placas NGM de 15 cm não semeadas por 24 h a 15 °C para permitir que todas as larvas L1 eclodam e cresçam de forma síncrona nas etapas subsequentes.
3. Cultivo de grandes culturas de C. elegans (Dia 10)
- Uma vez eclodidas, colete as larvas L1 (da Etapa 2.2.1.) em 3-4 mL de M9 em um tubo cônico limpo de 15 mL. Pipetar quatro gotas de 10 μL de solução de minhoca em uma lâmina ou uma placa e contar o número de vermes em cada gota sob um estereomicroscópio com ampliação de 16x. Determine a concentração de vermes da solução calculando a média do número de larvas de todas as gotas de solução de verme. Multiplique esse valor pelo volume deixado e estime a contagem total de vermes para cada cepa.
NOTA: 46.000-50.000 larvas L1 por cepa são necessárias nesta fase para preencher posteriormente uma placa de 384 poços ou duas meias-placas.- Para cada cepa, transfira todas as larvas de L1 para duas placas de NGM de 15 cm (23.000-25.000 L1 por placa) previamente inoculadas com 3 mL de OP50 (Etapa 1.5.) e re-semeadas com 0,5 mL de OP50 concentrado.
- Incubar a 15 °C, completando com 0,5 mL de OP50 concentrado diariamente, conforme necessário, até que os vermes atinjam o estágio L4.
NOTA: O estágio L4 é caracterizado por um intestino ligeiramente mais escuro e uma mancha branca em forma de meio disco ou crescente, onde a vulva acabará por se formar32,33. - Prepare placas de AGROSE NGM-96 poços seguindo os passos abaixo.
- Prepare oito placas de NGM-agarose de 96 poços enchendo cada poço com 125 μL de NGM-agarose (quatro placas por ensaio).
NOTA: Recomenda-se chapear algumas placas extras se algumas ficarem contaminadas nas etapas subsequentes. Dois leitores de placas serão necessários para executar ensaios em paralelo, mas eles também podem ser executados sucessivamente começando com o ensaio de estresse térmico, pois pode ser executado por apenas 6 h. Para essas placas, o ágar cinza <4% é substituído por agarose (consulte Tabela de Materiais), permitindo uma secagem mais lenta e uniforme nos plugues NGM e reduzindo a escavação de minhocas para melhor recuperação. - Certifique-se de que os poços estejam cheios uniformemente e livres de bolhas. Use um bloco de calor ajustado a 70 ° C (com transferência de calor lenta através do plástico da placa de vários poços, o NGM-agarose só pode aquecer até cerca de 55-60 ° C) para evitar que a mistura se solidifique durante o processo. Para remover bolhas dentro de poços, use uma agulha estéril aquecida por chama.
- Deixe as placas de 96 poços se ajustarem à temperatura ambiente em um ambiente estéril antes de inverter (tampa para baixo para evitar a condensação) e armazenar a 4 °C em uma caixa limpa até que seja necessário.
- Prepare oito placas de NGM-agarose de 96 poços enchendo cada poço com 125 μL de NGM-agarose (quatro placas por ensaio).
- No dia 11, verifique os vermes da Etapa 3.2., garantindo que nenhuma contaminação tenha aparecido e que os vermes ainda estejam repletos.
- No dia 12, verifique os vermes da Etapa 3.1., garantindo que nenhuma contaminação tenha aparecido e que os vermes ainda estejam repletos. Além disso, verifique o estágio de desenvolvimento dos vermes.
NOTA: O sexo/estirpe e a fase de desenvolvimento do verme, tais como os vermes L4 ou L4 + 24 h utilizados, dependem dos tratamentos a que os vermes são submetidos. Aqui, hermafroditas do tipo selvagem foram expostos a isolados bacterianos de L4 por 36 h.
4. Preparação de coleções de isolados de microbiota intestinal para realimentação de vermes
- Monitorize o crescimento bacteriano nas placas de ágar LB a partir da Etapa 2.1. e continuar a incubar a 20 °C.
NOTA: Embora não seja o ideal, no caso de alguns clones não crescerem ou revelarem contaminações, as bactérias podem ser re-riscadas de estoques limpos para placas LB de 6 cm e cultivadas a 25-28 °C por 24 h para estarem prontas para o experimento. - Definir um layout de matriz de 96 poços para a coleção bacteriana que está sendo testada, facilitando a semeadura sistemática de placas e a análise dos dados nas etapas subsequentes (Tabela Suplementar 1).
- Recolher a massa bacteriana de cada placa bacteriana de 6 cm (Passo 4.1.) e transferi-la para um tubo microcentrífugo de 1,5 ml marcado contendo 1 ml de tampão M9. Execute isso usando um laço de plástico estéril de 2 mm de diâmetro de uso único ou um laço de metal de 5 mm de diâmetro. Esterilize o laço de metal entre cepas bacterianas mergulhando em etanol 100%, inflamando e resfriando por 5 s.
- Vórtice os tubos de microcentrífuga até que os pellets bacterianos estejam totalmente ressuspensos (dependendo da cepa bacteriana, isso pode levar ~ 1-10 s).
- Gire para baixo a 9.300 x g por 5 min à temperatura ambiente, remova 700 μL de sobrenadante e ressuspenda o pellet bacteriano por vórtice.
- Transferir 200 μL de cada suspensão bacteriana para um único poço de uma placa estéril de 96 poços vazia, de acordo com o esquema estabelecido na etapa 4.2.
- A partir desta placa, inocular oito placas de NGM-agarose de 96 poços (preparadas na Etapa 3.3.) com 10 μL de solução bacteriana utilizando uma pipeta multicanal e incubar com a tampa ligada a 25 °C durante 24 h. Não sele as placas para permitir a secagem da placa e o crescimento aeróbico bacteriano e para evitar o excesso de condensação.
- Sele a placa de suspensão de 96 poços preparada na Etapa 4.6. com película de vedação adesiva limpa (ver Tabela de Materiais) e conservar a 15 °C até 5 dias. Isso será usado para a realimentação de vermes, conforme necessário.
5. Choque térmico LFASS e configuração do ensaio oxidativo (Dias 13 - 14)
- Olhando para as placas da Etapa 3.5., avalie o estágio de desenvolvimento dos vermes. Uma vez que >90% dos vermes tenham atingido L434, colete os vermes em até 10 mL de solução estéril de M9 em tubos cônicos de 15 mL.
- Lave os vermes extensivamente (pelo menos 4x) girando a 142 x g por 2 min a 4 °C, removendo o sobrenadante e adicionando 10 mL de M9 estéril fresco entre cada lavagem para se livrar da bactéria OP50. Ressussuscite o pellet de verme em 10 mL de M9.
- Transferir 50 μL de solução sem-fim para um tubo de ligação de superfície baixa (ver Tabela de Materiais) contendo 950 μL de M9. Depois de misturar suavemente o conteúdo do tubo para evitar a sedimentação de vermes, use rapidamente uma ponta de pipeta de baixa ligação molhada para transferir 3-4 gotas separadas de 10 μL para uma lâmina de vidro ou uma placa NGM e conte os números de minhocas sob um estereomicroscópio (ver Tabela de Materiais) com ampliação de 16x. Faça a média das contagens das 3-4 gotas e determine o número de vermes por microlitro na solução de minhoca (ver Passo 3.1.).
- Ajuste a concentração de vermes no tubo de 10 mL para atingir ~120 vermes em 8 μL. Se a solução for preparada na Etapa 5.2. não está concentrado o suficiente, gire os vermes para baixo e remova M9 de acordo para atingir 120 vermes por 8 μL.
- Transfira 8 μL de solução de minhoca (~120 vermes) para cada um dos poços das oito placas de AGM-agarose de 96 poços da Etapa 4.7., usando uma pipeta multicanal ou uma pipeta de repetição. Certifique-se de usar dicas de retenção baixas para limitar a perda de worms. Também pode ser necessário cortar as extremidades da ponta para permitir que vermes adultos grandes limitem o estresse mecânico em vermes adultos.
NOTA: O ensaio requer um mínimo de 30 vermes saudáveis vivos para funcionar de forma confiável, mas funciona melhor com cerca de 100 vermes por poço. - Incubar as placas de NGM-agarose de 96 poços semeadas por vermes e bactérias a 25 °C durante 36 h.
- Verifique as placas entre 12-24 h, garantindo que os vermes permaneçam repletos por toda parte. Se for necessária a realimentação, ressuspender as bactérias dentro da placa de matriz bacteriana de 96 poços armazenada a 15 °C na Etapa 4.8., e adicionar até 10 μL da solução bacteriana correspondente às placas de AGM-agarose de 96 poços onde os vermes estão em risco de fome antes do final do período de incubação de 36 h (vermes famintos produzirão resultados muito diferentes, então isso é muito importante).
NOTA: As etapas a seguir precisam ser realizadas no dia 15. Antes de iniciar o ensaio, pode ser necessário otimizar a altura de leitura. A leitura ideal será alcançada 20-50 μm acima do fundo do poço. Isso dependerá do modelo do leitor de placas. Alguns oferecem a possibilidade de um Z-scan, enquanto outros permitem a entrada manual de altura. Defina a altura ideal no nível em que o sinal de fluorescência azul mais alto (365 nm/430 nm) é detectado. Alguns leitores de placas podem operar a uma altura fixa otimizada para ensaios de células aderentes e podem não ser ideais para ensaios LFASS. - Após 36 h, dispensar 30 μL de M9 em cada poço da placa de 96 poços.
NOTA: Para ensaios de tensão térmica, o leitor de placas precisa ter atingido a temperatura necessária para executar o ensaio e pode precisar ser ligado com antecedência. O protocolo atual usa 42 °C para maximizar a velocidade de matança, mas a abordagem se aplica a outras temperaturas acima de 30 °C. - Transfira os vermes (cerca de 20 μL) para a placa de 384 poços de acordo com os layouts definidos, usando pontas de baixa retenção (considere cortar a extremidade das pontas para permitir que vermes grandes reduzam o estresse mecânico para vermes adultos).
NOTA: Para o presente estudo, duas configurações diferentes de leitor de placas são usadas para os dois ensaios descritos aqui (estresse térmico e estresse oxidativo) e, portanto, as amostras destinadas a esses dois ensaios não devem ser revestidas na mesma placa de 384 poços. - Certifique-se de que os leitores de placas estão configurados corretamente (Tabela 1).
- Complemente as placas de 384 poços com mais M9, visando um volume final de 60 μL por poço. Para o ensaio de tensão térmica, adicione 40 μL de M9 e, para o estresse oxidativo induzido por t-BHP, adicione 34 μl de M9 em 6 μl de t-BHP (ver Tabela de Materiais).
- Inicie o ensaio dentro de 2 minutos após a adição de t-BHP (idealmente, todos os vermes devem ser expostos a t-BHP simultaneamente, sendo a resolução do tempo de ensaio de 2 min). Se não for possível, use um temporizador para estimar o tempo gasto pipetando t-BHP antes do início do ensaio para permitir o ajuste posterior do tempo médio de morte.
- Feche as placas com a tampa transparente. Sele as bordas das placas de 384 poços com fita adesiva (colando sobre a placa e a tampa), garantindo que a fita não passe por cima da tampa ou sob a placa. Corte a fita entre a tampa e a placa em intervalos usando um bisturi para permitir a troca de ar, minimizando a evaporação durante o ensaio.
- Insira a placa no leitor de placas (consulte Tabela de materiais) e inicie a corrida. Apontar para excitar a 365 nm e detectar a emissão a 435 nm a cada 2 min por 6-12 h (Tabela 1).
NOTA: Normalmente, 6 h é suficiente para ensaios de estresse térmico de 42 °C e 8 h para ensaios de estresse oxidativo t-BHP a 7%.
6. Tratamento de dados do leitor de chapas
- Salve os dados brutos de fluorescência do leitor de placas como formatos de .txt separados por vírgulas ou tabulações, .csv ou .xls /.xlsx e converta para o formato xls /.xlsx. Dependendo do formato de dados, reorganize-os para corresponder ao layout da planilha do Excel necessário para a análise do LFASS. Siga as instruções detalhadas fornecidas na referência30.
NOTA: Embora os dados possam ser analisados manualmente, normalizando cada série temporal e procurando o momento em que a fluorescência da morte atinge a metade do máximo, a análise automatizada pode ser realizada no Matlab executando a rotina LFASS30. - Baixe e instale o Matlab (versão 2014a ou superior) e o pacote de software LFASS a partir de https://github.com/ABA80/LFASS. Siga as diretrizes e anotações fornecidas nele.
NOTA: A Figura 1C apresenta uma breve descrição da abordagem. O Matlab é necessário para executar a rotina do LFASS. Alternativamente, o código Matlab pode ser traduzido para Oracle, exceto para a função de ajuste, que é proprietária. Novas funções de suavização e sigmoid podem ser reescritas para permitir o uso em uma plataforma totalmente de código aberto. - Entre as análises do LFASS, mova os dados e os resultados para um novo local, pois a análise do LFASS processará todos os arquivos na pasta de dados e substituirá os arquivos na pasta Resultados.
7. Inspeção de dados
- Abra o arquivo excel e rotule as linhas de acordo com a posição do poço na placa de 384 poços. O Arquivo Suplementar 2 mostra um exemplo do arquivo excel dos dados brutos de fluorescência gerados para o ensaio de choque térmico. Use a posição do poço na placa de 384 poços para rotular o verme e as cepas bacterianas.
- Antes da análise do Matlab, inspecione visualmente os dados no excel, plotando a intensidade da fluorescência ao longo do tempo para um poço representativo. Dependendo do leitor de placas usado, os dados podem ser barulhentos, mas devem exibir um pico claro. Em particular:
- Determine um valor de fluorescência abaixo do qual um pico não seria significativamente diferente do ruído (a definição de tal limite no LFASS acelerará a análise excluindo poços vazios).
- Observe o primeiro ponto de tempo em que as flutuações de fluorescência amortecem antes de subir (os animais podem se debater vigorosamente por até 30 minutos, levando a leituras de fluorescência azul flutuantes rápidas).
NOTA: O ajuste de pico pode ser melhorado excluindo esses pontos de tempo iniciais da janela de ajuste da curva. - Observe os pontos de tempo entre os quais se espera que os valores mínimos e máximos de fluorescência caiam (observe vários poços para identificar esses intervalos), pois eles serão usados para ajuste de curvas.
- Verifique se as amplitudes dos picos de fluorescência variam significativamente entre os poços, normalize os dados antes de uma análise mais aprofundada usando a seguinte fórmula:
Poço de fluorescência normalizado n (t) = (Poço de fluorescêncian [t] - poço de fluorescência mínima [Dt]) / (poço de fluorescência máxima [Dt] - poço de fluorescência mínima [Dt])
onde "n" é o número atual do poço, "t" é o ponto de tempo e "Dt" é a série de pontos de tempo para o ensaio.
8. Processamento de dados LFASS
NOTA: Os detalhes são fornecidos em https://github.com/ABA80/LFASS e nos materiais complementares da Referência30.
- Crie duas subpastas dentro da pasta LFASSI, uma para os dados a serem analisados e outra para os resultados, por exemplo, "meus dados" e "resultados".
- Copie o arquivo de dados do Excel do ensaio para a subpasta LFASS "meus dados" após a inspeção de dados.
- Inicie o MATLAB, navegue até a pasta LFASS , digite e execute fitfolder na janela de comando (Arquivo Suplementar 3). Em seguida, siga as instruções na tela.
- Depois de digitar "fitfolder", o sistema pede o nome da pasta em que o arquivo do Excel está localizado, por exemplo, 'meus dados'. Digite o nome da pasta de dados (neste exemplo, "meus dados").
- Siga as instruções na tela, fornecendo os vários parâmetros solicitados.
- Digite "2" para o intervalo de tempo entre medições sucessivas no protocolo atual (especificar isso permite que os resultados sejam expressos em minutos em vez de unidades de ponto de tempo).
NOTA: O intervalo de tempo pode ser modificado para realizar medições de fluorescência com mais ou menos frequência (para diminuir ou aumentar a resolução do tempo) e também dependendo dos recursos do leitor de placas (ou seja, o intervalo de tempo pode precisar ser aumentado para leitores de placas que não podem realizar medições rápidas o suficiente). Certifique-se sempre de combinar o intervalo de tempo experimental com a rotina do LFASS. - Atribua o limite de tolerância superior digitando "0,95" (isso pode ser alterado conforme necessário para melhorar o ajuste) e o limite de tolerância inferior digitando "0,05" (isso pode ser alterado conforme necessário para melhorar o ajuste) para restringir o ajuste sigmoide.
NOTA: Outros parâmetros de tempo são baseados em notas do usuário da inspeção de dados (Etapa 7.2.).
- Digite "2" para o intervalo de tempo entre medições sucessivas no protocolo atual (especificar isso permite que os resultados sejam expressos em minutos em vez de unidades de ponto de tempo).
- Escolha se deseja exibir ou não curvas ajustadas e suavizadas digitando "y" para SIM ou "n" para NÃO quando solicitado. Para inspecionar visualmente os ajustes convergentes, selecione o primeiro.
Observação : o último é útil para visualizar todos os dados suavizados, mas geralmente não é selecionado porque gera muitos gráficos pop-up. Depois disso, o Matlab executará a rotina LFASS, que pode levar de 1 a 10 minutos se vários arquivos do Excel estiverem sendo processados de uma só vez. Janelas pop-up com curvas aparecerão de acordo com a seleção na Etapa 8.6. O Arquivo Suplementar 4A mostra um exemplo de uma curva ajustada. - Escolha se (1) analisar curvas identificadas como ruído ou (2) reformar curvas mal ajustadas com uma opção [s/n]. Tipo y para aprovar e n para rejeitar.
NOTA: Recomenda-se a aprovação do reajuste, especialmente se houver muitas curvas mal ajustadas ou não ajustadas. Isso permitirá que o usuário forneça parâmetros de ajuste de curva personalizados para cada curva à medida que aparecem na tela e solicite apenas limites anteriores e posteriores para o ajuste sigmoide. Pode ser tentado quantas vezes forem necessárias. - Depois que os dados forem analisados, feche o Matlab e abra a pasta LFASS .
- Clique na subpasta LFASS Meus resultados, pois os arquivos de resultados são salvos automaticamente na pasta Resultados conforme .txt.
NOTA: O Matlab gera três arquivos .txt: "Batch-fitted.txt", "Batch and noise-fitted.txt" e "Refitted.txt". Os dois primeiros são salvos como precaução em caso de falha do computador ou erro do usuário durante a remontagem. O arquivo que contém a análise completa mais precisa é "Refitted.txt". - Abra o arquivo Refitted.txt com o Microsoft Excel e salve como .xls para processamento adicional. O Arquivo Suplementar 4B mostra um exemplo de tal arquivo de resultado.
NOTA: Para cada poço (organizado em linhas), três valores são fornecidos nas colunas que fornecem estimativas do tempo mediano de morte da população de vermes: "Bruto": relata o tempo de interseção na metade máxima do pico de dados experimentais; "Lote-montado": comunica o tempo de intersecção a meio máximo da curva montada em lote; "Reajustado": informa o tempo de intersecção na metade máxima da curva reajustada. - Salve o arquivo no formato .xls como uma cópia em um local seguro. Não fazer isso correrá o risco de os arquivos serem substituídos durante a próxima execução da rotina LFASS.
NOTA: Os resultados podem então ser processados para análise gráfica ou estatística.
Access restricted. Please log in or start a trial to view this content.
Resultados
Os ensaios LFASS fornecem triagem robusta, de alto rendimento e rápida de várias condições de teste de uma só vez, como a triagem de inúmeros parâmetros genéticos e da microbiota que contribuem para a resistência ao estresse e o envelhecimento. Leva apenas 2-3 semanas para o experimento adquirir um extenso conjunto de dados de várias condições de teste. Populações adultas de vermes selvagens L4 + 36 h foram expostas a estresse térmico de 42 °C e estresse oxidativo induzido por t-BHP a 7% após um...
Access restricted. Please log in or start a trial to view this content.
Discussão
C. elegans oferece muitas vantagens para a triagem rápida de vários parâmetros experimentais de uma só vez, devido ao seu pequeno tamanho, transparência, rápido desenvolvimento, curta vida útil, baixo custo e facilidade de manuseio. Seu genoma, plano corporal, sistema nervoso, intestino e microbioma consideravelmente mais simples, mas complexo e semelhante o suficiente aos seres humanos, o tornam um poderoso modelo pré-clínico, onde a visão mecanicista pode ser obtida durante o teste de eficácia ou to...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos ao CGC Minnesota (Madison, EUA, NIH - P40 OD010440) por fornecer cepas de vermes e OP50 e ao Pr. Hinrich Schulenburg (CAU, Kiel, Alemanha) por fornecer todos os isolados microbianos ambientais aqui descritos. Este trabalho foi financiado por uma subvenção UKRI-BBSRC à AB (BB/S017127/1). O JM é financiado por uma bolsa de doutoramento FHM da Universidade de Lancaster.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
Referências
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood - Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977(2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer's disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98(2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson's disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921(2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095(2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323(2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38(2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364(2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545(2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849(2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998(2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613(2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019(2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer's disease: a systematic review. Nutrients. 14 (1), 20(2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson's disease. Ageing Research Reviews. 69, 101347(2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -E. H., Jung, Y., Lee, S. -J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190(2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652(2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583(2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398(2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448(2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35(2016).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados