É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Demonstração do alinhamento da sequência para prever a ferramenta de suscetibilidade entre espécies para avaliação rápida da conservação de proteínas
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para utilizar a versão mais recente da ferramenta Alinhamento de Sequência da Agência de Proteção Ambiental dos EUA para Prever a Suscetibilidade entre Espécies (SeqAPASS). Este protocolo demonstra a aplicação da ferramenta on-line para analisar rapidamente a conservação de proteínas e fornecer previsões personalizáveis e facilmente interpretáveis de suscetibilidade química entre espécies.
Resumo
A ferramenta Alinhamento de Sequência da Agência de Proteção Ambiental dos EUA para Prever a Suscetibilidade entre Espécies (SeqAPASS) é um aplicativo de triagem on-line rápido, disponível gratuitamente e que permite que pesquisadores e reguladores extrapolem informações de toxicidade entre espécies. Para alvos biológicos em sistemas modelo, como células humanas, camundongos, ratos e peixes-zebra, os dados de toxicidade estão disponíveis para uma variedade de produtos químicos. Através da avaliação da conservação do alvo de proteínas, esta ferramenta pode ser usada para extrapolar dados gerados a partir de tais sistemas modelo para milhares de outras espécies sem dados de toxicidade, produzindo previsões de suscetibilidade química intrínseca relativa. As versões mais recentes da ferramenta (versões 2.0-6.1) incorporaram novos recursos que permitem a rápida síntese, interpretação e uso dos dados para publicação, além de gráficos de qualidade de apresentação.
Entre esses recursos estão visualizações de dados personalizáveis e um relatório de resumo abrangente projetado para resumir os dados do SeqAPASS para facilitar a interpretação. Este artigo descreve o protocolo para orientar os usuários através do envio de trabalhos, navegando pelos vários níveis de comparações de sequências de proteínas e interpretando e exibindo os dados resultantes. Novos recursos do SeqAPASS v2.0-6.0 são destacados. Além disso, são descritos dois casos de uso focados na conservação da transtirretina e da proteína do receptor opioide usando essa ferramenta. Finalmente, os pontos fortes e limitações do SeqAPASS são discutidos para definir o domínio de aplicabilidade da ferramenta e destacar diferentes aplicações para extrapolação entre espécies.
Introdução
Tradicionalmente, o campo da toxicologia tem se baseado fortemente no uso de testes em animais inteiros para fornecer os dados necessários para as avaliações de segurança química. Tais métodos são tipicamente caros e consomem muitos recursos. No entanto, devido ao grande número de produtos químicos atualmente utilizados e ao ritmo acelerado em que novos produtos químicos estão sendo desenvolvidos, globalmente há uma necessidade reconhecida de métodos mais eficientes de triagem química 1,2. Essa necessidade e a mudança de paradigma resultante dos testes em animais levaram ao desenvolvimento de muitos novos métodos de abordagem, incluindo ensaios de triagem de alto rendimento, transcriptômica de alto rendimento, sequenciamento de próxima geração e modelagem computacional, que são estratégias alternativas de teste promissoras 3,4.
Avaliar a segurança química em toda a diversidade de espécies potencialmente impactadas por exposições químicas tem sido um desafio duradouro, não apenas com os testes tradicionais de toxicidade, mas também com novos métodos de abordagem. Os avanços na toxicologia comparativa e preditiva forneceram estruturas para a compreensão da sensibilidade relativa de diferentes espécies, e os avanços tecnológicos nos métodos computacionais continuam a aumentar a aplicabilidade desses métodos. Várias estratégias têm sido discutidas ao longo da última década que alavancam os bancos de dados de sequências de genes e proteínas existentes, juntamente com o conhecimento de alvos moleculares químicos específicos, para apoiar abordagens preditivas para extrapolação entre espécies e melhorar as avaliações de segurança química além dos organismos modelo típicos 5,6,7,8.
Para avançar a ciência em ação, com base nesses estudos fundamentais em toxicologia preditiva, priorizar os esforços de testes químicos e apoiar a tomada de decisões, foi criada a ferramenta Alinhamento de Sequência da Agência de Proteção Ambiental dos EUA para Prever a Suscetibilidade entre Espécies (SeqAPASS). Esta ferramenta é um aplicativo público e disponível gratuitamente baseado na web que usa repositórios públicos de informações de sequência de proteínas em constante expansão para prever a suscetibilidade química em toda a diversidade de espécies9. Com base no princípio de que a suscetibilidade intrínseca relativa de uma espécie a um determinado produto químico pode ser determinada avaliando a conservação dos alvos proteicos conhecidos desse produto químico, esta ferramenta compara rapidamente sequências de aminoácidos proteicos de uma espécie com sensibilidade conhecida a todas as espécies com dados de sequência de proteínas existentes. Esta avaliação é completada através de três níveis de análise, incluindo (1) sequência de aminoácidos primários, (2) domínio funcional e (3) comparações críticas de resíduos de aminoácidos, cada um exigindo um conhecimento mais aprofundado da interação químico-proteína e proporcionando maior resolução taxonômica na predição de susceptibilidade. Uma grande força do SeqAPASS é que os usuários podem personalizar e refinar sua avaliação, adicionando linhas adicionais de evidência para a conservação do alvo com base na quantidade de informações disponíveis sobre a interação química-proteína ou proteína-proteína de interesse.
A primeira versão foi lançada em 2016, que permitiu aos usuários avaliar sequências de aminoácidos primários e domínios funcionais de maneira simplificada para prever a suscetibilidade química e continha recursos mínimos de visualização de dados (Tabela 1). Diferenças individuais de aminoácidos têm se mostrado determinantes importantes de diferenças entre espécies nas interações químico-proteína, o que pode afetar a suscetibilidade química das espécies10,11,12. Portanto, versões subsequentes foram desenvolvidas para considerar os aminoácidos críticos que são importantes para a interação química direta13. Respondendo ao feedback das partes interessadas e dos usuários, essa ferramenta passou por lançamentos de versões anuais com novos recursos adicionais projetados para atender às necessidades de pesquisadores e comunidades regulatórias para enfrentar os desafios na extrapolação entre espécies (Tabela 1). O lançamento do SeqAPASS versão 5.0 em 2020 trouxe recursos centrados no usuário que incorporam opções de visualização e síntese de dados, links externos, opções de tabela de resumo e relatório e recursos gráficos. No geral, os novos atributos e capacidades desta versão melhoraram a síntese de dados, a interoperabilidade entre bancos de dados externos e a facilidade de interpretação de dados para previsões de suscetibilidade entre espécies.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Primeiros passos
NOTA: O protocolo apresentado aqui é focado no utilitário da ferramenta e nos principais recursos. Descrições detalhadas de métodos, recursos e componentes podem ser encontradas no site em um Guia do Usuário abrangente (Tabela 1).
Tabela 1: Evolução da ferramenta SeqAPASS. Uma lista de recursos e atualizações adicionados à ferramenta SeqAPASS desde sua implantação inicial. abreviaturas: seqAPASS = alinhamento de sequência para prever a suscetibilidade entre espécies; ECOTOX = Base de conhecimento ECOTOXicology. Por favor, clique aqui para baixar esta Tabela.
- Acesse https://seqapass.epa.gov/seqapass usando o Chrome. Selecione Login para usar uma conta existente ou siga as instruções para criar uma conta SeqAPASS, que permitirá que os usuários executem, armazenem, acessem e personalizem seus trabalhos concluídos.
- Antes de realizar uma análise, primeiro identifique uma proteína de interesse e uma espécie-alvo ou sensível, revisando a literatura existente ou dados pré-existentes (Figura 1). Como o SeqAPASS contém links para recursos externos para ajudar a identificar a proteína de consulta, clique nos botões suspensos em Identificar um alvo de proteína para acessar recursos relevantes.
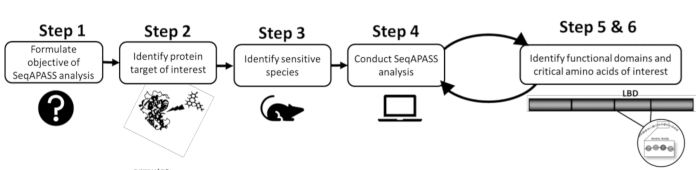
Figura 1: Formulação do problema SeqAPASS: diagrama esquemático das informações preliminares necessárias para uma análise bem-sucedida. abreviaturas: seqAPASS = alinhamento de sequência para prever a suscetibilidade entre espécies; LBD = domínio de ligação de ligantes. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 2: Interoperabilidade do SeqAPASS entre bancos de dados. Diagrama esquemático de ferramentas, bancos de dados e recursos externos integrados ao SeqAPASS. abreviaturas: seqAPASS = alinhamento de sequência para prever a suscetibilidade entre espécies; POA = via de desfecho adverso; NCBI = Centro Nacional de Informação em Biotecnologia; ECOTOX = Base de conhecimento ECOTOXicology. Por favor, clique aqui para ver uma versão maior desta figura.
Tabela 2: Links, recursos e ferramentas integrados à ferramenta SeqAPASS. Uma lista das várias fontes de dados, links e recursos aproveitados na ferramenta SeqAPASS. Abreviação: SeqAPASS = alinhamento de sequência para prever a suscetibilidade entre espécies. Por favor, clique aqui para baixar esta Tabela.
2. Desenvolvendo e executando uma consulta SeqAPASS: Nível 1
NOTA: Em uma análise de Nível 1, toda a sequência de aminoácidos primários de uma proteína de consulta é comparada com as sequências de aminoácidos primários de todas as espécies com informações de sequência disponíveis. Essa ferramenta usa algoritmos para minerar, coletar e compilar dados disponíveis publicamente para alinhar e comparar rapidamente sequências de aminoácidos entre espécies. O back-end armazena informações dos bancos de dados do National Center for Biotechnology Information (NCBI) e usa estrategicamente as versões autônomas da Protein Basic Local Alignment Search Tool (BLASTp)54 e da Constraint-based Multiple Alignment Tool (COBALT)55.
- Em Comparar Sequências de Aminoácidos Primários, clique em Por Espécie ou Por Acesso. Use a seleção Por espécie para digitar ou selecionar em uma lista de espécies para escolher o alvo de proteína de interesse.
- Envie acessos de proteína (ou seja, ID de proteína NCBI) diretamente inserindo a(s) adesão(ões) na caixa de texto Por Adesão .
- Selecione Executar solicitação para enviar a consulta. Uma vez enviado, aguarde até que uma notificação apareça no canto superior direito da janela do navegador, indicando um envio bem-sucedido.
- Selecione a guia Status da Execução do SeqAPASS na parte superior da página para exibir uma lista de todas as execuções do SeqAPASS realizadas nessa conta de usuário e verificar a porcentagem de conclusão.
- Clique em Atualizar Dados enquanto o botão de opção apropriado está selecionado para verificar o status das execuções de Nível 2 e Nível 3.
- Selecione a guia Exibir Relatórios do SeqAPASS na parte superior da página para acessar uma lista de todos os relatórios concluídos nessa conta.
- Na guia Exibir Relatórios do SeqAPASS , selecione a proteína de consulta de interesse. Clique em Solicitar Relatório Selecionado para abrir a página Informações de Proteína de Consulta de Nível 1 e exibir resultados, opções de personalização de dados, visualizações e relatórios de resumo.
- Por padrão, selecione Exibir Relatório para exibir dados no navegador da Web. Como alternativa, selecione Salvar relatório para baixar dados brutos como um arquivo .zip.
NOTA: O tempo necessário para uma análise de Nível 1 variará (média de 23 minutos para a versão 5.1) dependendo da demanda global do usuário naquele momento, do número de trabalhos enviados para a fila e da quantidade de informações de proteína que existem para um trabalho enviado. Se um alvo de proteína tiver sido previamente concluído, os dados estarão disponíveis em segundos após a submissão.
3. Desenvolvendo e executando uma consulta SeqAPASS: Nível 2
NOTA: Como toda a sequência de proteínas não está diretamente envolvida em uma interação química, uma análise de Nível 2 compara apenas a sequência de aminoácidos do domínio funcional para fazer previsões de suscetibilidade em classificações taxonômicas mais baixas (por exemplo, classe, ordem, família).
- Na página Informações de Proteína de Consulta de Nível 1, clique no sinal de adição + ao lado do cabeçalho Nível 2 para preencher o menu Consulta de Nível 2 .
- Identifique o(s) domínio(s) apropriado(s) na proteína de interesse (proteína de consulta).
- Se um domínio não tiver sido identificado, clique no link integrado para o NCBI Conserved Domains Database (CDD) (Tabela 1), que pode ajudar na identificação da seleção de domínio apropriada.
Observação : normalmente, apenas domínios de hit específicos são selecionados como consultas no nível 2.
- Se um domínio não tiver sido identificado, clique no link integrado para o NCBI Conserved Domains Database (CDD) (Tabela 1), que pode ajudar na identificação da seleção de domínio apropriada.
- Clique na caixa Selecionar Domínio para preencher automaticamente uma lista de domínios funcionais para a proteína de consulta.
- Selecione a( s) acessão(ões) de domínio na lista suspensa e inicie a consulta de Nível 2 clicando no botão Solicitar Execução de Domínio . Uma vez enviado, aguarde até que uma notificação apareça indicando um envio bem-sucedido.
- Clique em Atualizar execuções de Nível 2 e 3 para preencher os dados de Nível 2 , que estarão disponíveis em segundos após o envio.
- Em Exibir Dados de Nível 2, selecione a(s) adesão(ões) de domínio concluída(s) na lista suspensa e clique no botão Exibir Dados de Nível 2 para abrir os resultados em uma nova página.
4. Acesso e compreensão dos dados: SeqAPASS Nível 1 e Nível 2
- Role até a parte inferior da página Informações sobre a Proteína de Consulta para exibir um relatório dos resultados - um Relatório Primário é fornecido com análises de Nível 1 e 2 por padrão. Selecione o botão de opção Relatório completo para exibir um relatório mais detalhado que forneça todos os hits de sequência e métricas de alinhamento. Clique na acessão/ID/nome apropriado em ambos os relatórios para acessar as informações transparentes de alinhamento e taxonomia de proteínas no banco de dados NCBI.
- Role até o lado direito da tabela de resultados para exibir a coluna ECOTOX . Clique em links para a base de conhecimento ECOTOXicology (ECOTOX) para coletar rapidamente dados de toxicidade correspondentes para espécies com previsões de suscetibilidade.
NOTA: O ECOTOX é uma base de conhecimentos abrangente e publicamente disponível que fornece dados únicos de toxicidade química para plantas aquáticas e terrestres e animais selvagens. O SeqAPASS v6.0 inclui um widget ECOTOX para se conectar com dados ECOTOX relevantes mais rapidamente por produtos químicos e espécies de interesse. - Clique em Baixar Tabela para salvar a tabela como um arquivo de planilha. Clique no botão Exibir Relatório de Resumo para exibir e baixar uma tabela de relatório de resumo que apresenta dados classificados por grupo taxonômico.
NOTA: As tabelas de resumo de dados estão disponíveis para Relatórios Principais e Completos e fornecem uma visão geral das previsões para um determinado destino.
5. Manipulando configurações de dados: SeqAPASS Nível 1 e Nível 2
NOTA: Em ambas as análises de Nível 1 e Nível 2, assume-se que quanto maior a similaridade da proteína, maior a probabilidade de um produto químico interagir com a proteína de maneira semelhante à espécie/proteína de consulta, tornando-os suscetíveis a potenciais impactos de produtos químicos com esse alvo molecular. Devido à semelhança desses dados, as etapas para a compreensão dos dados de Nível 1 e 2 são descritas juntas em um único protocolo.
- Consulte os submenus na parte superior de Consultar informações de proteína para acessar e manipular as configurações de relatório e usar as configurações padrão para todas as opções de relatório para a maioria das análises. Se houver justificativa científica para alterar a configuração padrão, siga estas etapas opcionais:
- (OPCIONAL) Clique no sinal de adição + ao lado de Corte de suscetibilidade para exibir e ajustar as configurações de corte de suscetibilidade em uma nova guia. Selecione um novo valor de corte em uma lista suspensa ou insira um valor de corte definido pelo usuário.
- (OPCIONAL) Altere o número no campo E-Value (o número de alinhamentos diferentes esperados por acaso) se algo diferente do padrão for desejado.
NOTA: Qualquer proteína com um valor E maior que o número na caixa será eliminada do relatório principal. - (OPCIONAL) Use a opção Classificar por Grupo Taxonômico para escolher o Nível de hierarquia taxonômica a ser exibida na coluna Grupo Taxonômico Filtrado na tabela de resultados.
NOTA: Alterar a hierarquia taxonômica também alterará a previsão de suscetibilidade com base nas espécies de cada grupo filtrado que são encontradas acima do ponto de corte. - (OPCIONAL) Altere o campo Domínio Comum (quantos domínios comuns uma proteína deve compartilhar com a proteína de consulta a ser incluída nos resultados) se algo diferente do padrão for desejado.
Observação : como a configuração padrão é 1, qualquer sequência que não compartilha pelo menos um domínio comum com a proteína de consulta será excluída. - (OPCIONAL) Selecione Não em Espécies lidas para recuperar as previsões de suscetibilidade de Y somente se a Similaridade Percentual for maior ou igual ao ponto de corte ou se o acerto for identificado como um candidato a ortolog.
NOTA: Esta configuração padrão é Sim, o que significa que uma previsão de suscetibilidade de Y será relatada para todos os candidatos a ortolog, todas as espécies listadas acima do ponto de corte de suscetibilidade e todas as espécies abaixo do ponto de corte do mesmo grupo taxonômico com uma ou mais espécies acima do ponto de corte.
- Clique no botão Baixar configurações atuais do relatório para baixar um arquivo que capture as configurações atuais aplicadas.
NOTA: O nível de avaliação específico (1, 2 ou 3) selecionado ditará as configurações apresentadas no relatório.
6. Visualizando os dados: SeqAPASS Nível 1 e Nível 2
- Clique no sinal de adição + ao lado de Visualização e clique no botão Visualizar Dados para abrir uma guia separada exibindo as informações definidas pelo usuário e a opção para selecionar um gráfico interativo de resultados.
- Clique em Boxplot para abrir os controles de plotagem e boxplot interativos e permitir que a visualização de boxplot seja atualizada ativamente para refletir as alterações na tabela de dados e fornecer gráficos de qualidade de publicação e apresentação.
Observação : O boxplot padrão exibe grupos de espécies no eixo x e semelhança percentual no eixo y. Os boxplots exibem o ponto de corte de suscetibilidade (linha tracejada), a semelhança percentual entre as espécies em comparação com as espécies de consulta e os valores médios e medianos para cada grupo taxonômico, juntamente com os percentis 25 e 75 e a faixa interquartil. Dependendo do objetivo da análise e das necessidades do usuário, muitos recursos do boxplot podem ser modificados por meio das seguintes etapas opcionais.- (OPCIONAL) Para personalizar os grupos taxonômicos exibidos, consulte a caixa Grupos Taxonômicos na seção Controles . Remova grupos rolando sobre os nomes e selecionando x ou usando o menu suspenso Grupos taxonômicos .
- (OPCIONAL) Para adicionar uma legenda que identifique uma espécie de interesse ou grupos predefinidos específicos (por exemplo, espécies ameaçadas ou ameaçadas), passe o mouse sobre um nome de grupo taxonômico no eixo x para ativar uma caixa pop-up listando as três principais espécies ordenadas por maior similaridade percentual. Passe o mouse sobre as espécies na legenda para gerar uma caixa pop-up com as informações de espécies correspondentes. Clique na caixa de um grupo taxonômico específico para gerar uma tabela de resumo para download listando espécies e previsões.
- Clique em Download Boxplot para escolher um tipo de arquivo, personalizar a resolução de largura/altura e salvar a visualização.
7. Desenvolvendo e executando uma análise SeqAPASS: Nível 3
NOTA: Uma análise de Nível 3 avalia os resíduos de aminoácidos identificados pelo usuário dentro da proteína de consulta e compara rapidamente a conservação desses resíduos entre as espécies. Presume-se que as espécies em que esses resíduos são conservados são mais propensas a interagir com um produto químico de maneira semelhante à espécie/proteína modelo. Como o Nível 3 se concentra em aminoácidos individuais, uma análise só pode ser realizada quando o conhecimento detalhado dos resíduos de aminoácidos críticos para a interação química-proteína ou proteína-proteína estiver disponível.
- Clique no sinal de adição + ao lado do cabeçalho Nível 3 na página Informações de Proteína de Consulta de Nível 1 para preencher o menu Consulta de Nível 3.
- Clique no sinal de mais + ao lado do Reference Explorer para abrir a ferramenta de explorador de referência, que gera uma cadeia de caracteres booleana predefinida para consultar a literatura disponível e auxilia os usuários na identificação de literatura apropriada para apoiar a identificação de aminoácidos críticos a serem usados na avaliação de Nível 3 (Tabela 2 e Figura 2).
- (OPCIONAL) Depois que a proteína de consulta for preenchida automaticamente, use a função Adicionar nome da proteína para adicionar proteínas adicionais.
- Clique em Gerar link do Google Acadêmico para abrir um pop-up contendo uma cadeia de caracteres de pesquisa gerada automaticamente que inclui termos de pesquisa relevantes.
- Clique em Pesquisar no Google Acadêmico para consultar o banco de dados de literatura usando a cadeia de caracteres de pesquisa.
- Como alternativa, clique em Copiar para a Área de Transferência e personalize a cadeia de caracteres de pesquisa adicionando ou removendo termos usando as funções no Gerenciador de Referências.
8. Identificar resíduos críticos de aminoácidos usando a literatura identificada
- Selecione a sequência de modelos à qual as espécies selecionadas pelo usuário serão alinhadas no menu Consulta de Nível 3.
NOTA: Esta sequência de modelos é comumente escolhida com base na literatura para a qual os aminoácidos críticos foram identificados e podem ser da mesma espécie ou de uma espécie diferente das consultadas nos níveis 1 e 2.- (OPCIONAL) Use a caixa Comparações Adicionais para comparar quaisquer acessos/sequências que não apareçam nas tabelas Relatório Principal/Completo .
- Insira um nome definido pelo usuário para a execução de Nível 3 na caixa de texto Inserir Nome de Execução de Nível 3 para identificar a execução de Nível 3 concluída. Escolha um nome exclusivo para cada avaliação.
- Selecione o grupo taxonômico de interesse no campo Escolher grupo(s) taxonômico(s ). Selecione um grupo taxonômico para filtrar automaticamente a tabela por esse grupo taxonômico.
- Na tabela de resultados, clique manualmente na caixa de seleção ao lado de qualquer espécie a ser alinhada à sequência do modelo.
NOTA: Para garantir o alinhamento apropriado, um grupo taxonômico de cada vez deve ser comparado ao modelo. Selecione apenas proteínas anotadas de forma semelhante para as espécies de interesse. Ao selecionar sequências para comparação, é importante prestar atenção a certas sequências (por exemplo, hipotéticas, de BAIXA QUALIDADE ou parciais). A menos que haja uma justificativa transparente para a inclusão, é melhor excluir essas sequências, pois elas podem distorcer as previsões devido a informações de sequência incompletas ou inadequadas. - Repita as etapas para alinhar todos os grupos taxonômicos de interesse.
- Clique em Atualizar execuções de Nível 2 e 3 depois que todas as espécies tiverem sido alinhadas para preencher o menu Selecionar Nome de Execução de Nível 3 com os trabalhos de Nível 3 concluídos e obter os dados de um alinhamento de Nível 3 imediatamente.
- Clique em Combinar Dados de Nível 3 para combinar alinhamentos de vários grupos taxonômicos.
- Como alternativa, para exibir um único relatório, selecione o nome definido pelo usuário em Escolher Consulta a Exibir e clique em Exibir Dados de Nível 3.
- Selecione o modelo de Nível 3 a ser usado como base para a comparação de resíduos de aminoácidos no menu Combinar Relatórios de Nível 3 e clique em Avançar.
- Em Trabalhos de Nível 3, selecione trabalhos concluídos para comparação e clique em Avançar. Use a função Trabalhos de Nível 3 de Ordem para reordenar os grupos taxonômicos, se desejado. Clique em Exibir Dados de Nível 3 para produzir uma página de relatório de Nível 3 com os grupos taxonômicos combinados alinhados.
- Selecione as posições de aminoácidos previamente identificadas para as espécies-modelo digitando a(s) posição(ões) de aminoácidos, separada por vírgulas, na caixa Inserir Posições de Resíduos de Aminoácidos e, em seguida, selecionando Copiar para Lista de Resíduos . Selecione diretamente os resíduos na sequência do modelo na caixa de transporte.
- Clique em Atualizar Relatório para atualizar a página e exibir as previsões de suscetibilidade de Nível 3.
NOTA: O nível 3 usa um conjunto simples de regras derivadas de descritores básicos de propriedades funcionais de cadeia lateral (por exemplo, alifáticas, aromáticas) e dimensões moleculares (diferenças de peso molecular >30 g/mol) para determinar se as diferenças em posições-chave provavelmente afetarão as interações proteicas13.
9. Visualizando dados SeqAPASS de nível 3
NOTA: Como nos níveis anteriores, os relatórios principal e completo estão disponíveis. Além de dados idênticos aos dados dos Níveis 1 e 2, o Relatório Primário exibe posições de aminoácidos, abreviações e uma suscetibilidade sim/não (S/N) semelhante à previsão do modelo. Da mesma forma, o Relatório Completo contém informações sobre a classificação da cadeia lateral dos aminoácidos e o peso molecular.
- Na página Relatório de Nível 3, role até a parte inferior para exibir um relatório dos resultados. Clique em Baixar Tabela na parte inferior do relatório para salvá-la.
- Clique em Exibir Relatório de Resumo de Nível 3 para exibir e baixar uma tabela de relatório de resumo que apresenta dados classificados por grupo taxonômico. Clique no sinal de adição + ao lado de Visualização na página Relatório de Nível 3 para abrir uma guia separada do navegador exibindo as informações definidas pelo usuário e a opção de exibir os resultados na forma de um mapa de calor interativo.
- Clique em Mapa de calor na página Informações de visualização para abrir o gráfico interativo e os controles e permitir que a visualização do mapa de calor seja atualizada ativamente para refletir as alterações na tabela de dados. Execute as seguintes etapas opcionais para personalizar o mapa de calor.
- (OPCIONAL) Selecione Opções de Relatório para alternar entre um Relatório Simples, que exibe a posição do aminoácido, a abreviação de uma letra e a semelhança de aminoácidos, ou um Relatório Completo, que exibe informações detalhadas sobre cada aminoácido selecionado.
- (OPCIONAL) Selecione Opções de Relatório para alterar a forma como as espécies são exibidas, seja por Nome Comum ou Nome Científico.
NOTA: No Relatório Simples, os aminoácidos são categorizados como Correspondência Total (azul escuro), Correspondência Parcial (azul claro, substituições que atendem a apenas um critério) ou Não é uma Correspondência (amarelo, substituições que não atendem a nenhum critério) para o aminoácido modelo. O Relatório Completo exibe comparações como uma Correspondência Total (azul escuro) ou Não é uma Correspondência (amarelo). - (OPCIONAL) Selecione Seleções Opcionais para destacar informações úteis, como Candidatos a Ortolog, Espécies Ameaçadas, Espécies Ameaçadas ou Organismos Modelo Comuns.
- (OPCIONAL) Selecione Configurações do Mapa de Calor para selecionar opções de personalização adicionais, incluindo adicionar ou remover colunas, legendas e texto.
- Clique em Download Boxplot para escolher um tipo de arquivo e salvar a visualização.
10. Interpretação dos Resultados do SeqAPASS: Linhas de evidência para a conservação de proteínas
NOTA: Para facilitar a interpretação, esta ferramenta inclui um Relatório de Resumo de Decisão (Relatório DS) concebido para integrar dados entre Níveis. O Relatório DS contém os resultados (ou seja, tabelas de dados e/ou visualizações) que o usuário selecionou e permite a rápida avaliação de previsões de suscetibilidade em vários níveis para várias espécies simultaneamente.
- Clique em Enviar número de nível para o relatório DS nas páginas de resultados ou visualização de dados e aguarde até que os dados sejam "enviados" e a guia Relatório DS se torne ativa.
Observação : se os resultados ou quaisquer alterações não tiverem sido enviadas por push para o relatório DS, o nível de envio # para o relatório DS permanecerá ativo até que seja selecionado. Se uma configuração tiver sido alterada, o texto Clique para enviar novas alterações por push será exibido até que as alterações sejam enviadas por push para o relatório. As visualizações podem ser enviadas por push para o Relatório DS a qualquer momento durante a avaliação. - Selecione a guia Relatório DS a qualquer momento para acessar a página DS.
NOTA: Para todas as espécies alinhadas no Nível 1, a tabela Relatório de Resumo da Decisão Final contém os dados importantes e as previsões de suscetibilidade para cada análise. Se uma espécie na tabela DS não foi incluída no relatório de Nível 3, mas foi encontrada em trabalhos de Nível 1 e/ou Nível 2, a célula na tabela receberá uma designação não aplicável (NA) para a previsão de suscetibilidade de Nível 3.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para demonstrar a aplicação da ferramenta SeqAPASS e destacar novos recursos, dois estudos de caso são descritos representando casos em que a conservação de proteínas prevê que há diferenças na suscetibilidade química entre as espécies (transtirretina humana) e que não há diferenças (μ receptor opioide [MOR]). O primeiro desses exemplos aborda comparações estruturais / sequência de proteínas para prever o domínio da aplicabilidade para vias de desfechos adversos (POAs, ver Tabela 2 pa...
Access restricted. Please log in or start a trial to view this content.
Discussão
Há um reconhecimento generalizado de que não é viável testar empiricamente espécies suficientes para capturar a diversidade genômica, fenotípica, fisiológica e comportamental de organismos vivos que podem ser expostos a produtos químicos de interesse toxicológico. O objetivo do SeqAPASS é maximizar o uso de dados estruturais e de sequência de proteínas existentes e em contínua expansão para auxiliar e informar a extrapolação de dados/conhecimentos de toxicidade química de organismos testados para centen...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Os autores agradecem ao Dr. Daniel L. Villeneuve (EPA dos EUA, Centro de Toxicologia Computacional e Exposição) e ao Dr. Jon A. Doering (Departamento de Ciências Ambientais, Louisiana State University) por fornecerem comentários sobre um rascunho anterior do manuscrito. Este trabalho foi apoiado pela Agência de Proteção Ambiental dos EUA. As opiniões expressas neste artigo são as dos autores e não refletem necessariamente as opiniões ou políticas da Agência de Proteção Ambiental dos EUA, nem a menção de nomes comerciais ou produtos comerciais indica endosso pelo governo federal.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Spreadsheet program | N/A | N/A | Any program that can be used to view and work with csv files (e.g. Microsoft Excel, OpenOffice Calc, Google Docs) can be used to access data export files. |
| Basic computing setup and internet access | N/A | N/A | SeqAPASS is a free, online tool that can be easily used via an internet connection. No software downloads are required. |
Referências
- Krewski, D., et al. Toxicity testing in the 21st century: a vision and a strategy. Journal of Toxicology and Environmental Health, Part B. 13 (2-4), 51-138 (2010).
- Wang, Z., Walker, G. W., Muir, D. C. G., Nagatani-Yoshida, K. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories. Environmental Science & Technology. 54 (5), 2575-2584 (2020).
- Brooks, B. W., et al. Toxicology advances for 21st century chemical pollution. One Earth. 2 (4), 312-316 (2020).
- Kostal, J., Voutchkova-Kostal, A. Going all in: A strategic investment in in silico toxicology. Chemical Research in Toxicology. 33 (4), 880-888 (2020).
- Cheng, W., Doering, J. A., LaLone, C., Ng, C. Integrative computational approaches to inform relative bioaccumulation potential of per- and polyfluoroalkyl substances (PFAS) across species. Toxicology Sciences. 180 (2), 212-223 (2021).
- Kostich, M. S., Lazorchak, J. M. Risks to aquatic organisms posed by human pharmaceutical use. Science of the Total Environment. 389 (2-3), 329-339 (2008).
- Gunnarsson, L., Jauhiainen, A., Kristiansson, E., Nerman, O., Larsson, D. G. Evolutionary conservation of human drug targets in organisms used for environmental risk assessments. Environmental Science & Technology. 42 (15), 5807-5813 (2008).
- LaLone, C. A., et al. Evidence for cross species extrapolation of mammalian-based high-throughput screening assay results. Environmental Science & Technology. 52 (23), 13960-13971 (2018).
- LaLone, C. A., et al. Editor's highlight: Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS): A web-based tool for addressing the challenges of cross-species extrapolation of chemical toxicity. Toxicology Sciences. 153 (2), 228-245 (2016).
- Head, J. A., Hahn, M. E., Kennedy, S. W. Key amino acids in the aryl hydrocarbon receptor predict dioxin sensitivity in avian species. Environmental Science & Technology. 42 (19), 7535-7541 (2008).
- Bass, C., et al. Mutation of a nicotinic acetylcholine receptor β subunit is associated with resistance to neonicotinoid insecticides in the aphid Myzus persicae. BMC Neuroscience. 12, 51-51 (2011).
- Erdmanis, L., et al. Association of neonicotinoid insensitivity with a conserved residue in the loop d binding region of the tick nicotinic acetylcholine receptor. Biochemistry. 51 (23), 4627-4629 (2012).
- Doering, J. A., et al. et al. In silico site-directed mutagenesis informs species-specific predictions of chemical susceptibility derived from the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) tool. Toxicology Sciences. 166 (1), 131-145 (2018).
- Noyes, P. D., et al. Evaluating chemicals for thyroid disruption: Opportunities and challenges with in vitro testing and adverse outcome pathway approaches. Environmental Health Perspectives. 127 (9), 95001(2019).
- Park, G. Y., Jamerlan, A., Shim, K. H., An, S. S. A. Diagnostic and treatment approaches involving transthyretin in amyloidogenic diseases. Int J Mol Sci. 20 (12), 2982(2019).
- Rabah, S. A., Gowan, I. L., Pagnin, M., Osman, N., Richardson, S. J. Thyroid hormone distributor proteins during development in vertebrates. Front Endocrinol (Lausane). 10, 506(2019).
- Richardson, S. J. Cell and molecular biology of transthyretin and thyroid hormones. International Review of Cytology. 258, 137-193 (2007).
- Yamauchi, K., Ishihara, A. Transthyretin and Endocrine Disruptors. Recent Advances in Transthyretin Evolution, Structure and Biological Functions. Richardson, S. J., Cody, V. , Springer. Berlin Heidelberg, Germany. 159-171 (2009).
- Iakovleva, I., et al. Tetrabromobisphenol A is an efficient stabilizer of the transthyretin tetramer. PLoS One. 11 (4), 0153529(2016).
- Ishihara, A., Sawatsubashi, S., Yamauchi, K. Endocrine disrupting chemicals: Interference of thyroid hormone binding to transthyretins and to thyroid hormone receptors. Molecular and Cellular Endocrinology. 199 (1), 105-117 (2003).
- Kar, S., Sepúlveda, M. S., Roy, K., Leszczynski, J. Endocrine-disrupting activity of per- and polyfluoroalkyl substances: Exploring combined approaches of ligand and structure based modeling. Chemosphere. 184, 514-523 (2017).
- Morais-de-Sa, E., Pereira, P. J., Saraiva, M. J., Damas, A. M. The crystal structure of transthyretin in complex with diethylstilbestrol: A promising template for the design of amyloid inhibitors. Journal of Biological Chemistry. 279 (51), 53483-53490 (2004).
- Morgado, I., Campinho, M. A., Costa, R., Jacinto, R., Power, D. M. Disruption of the thyroid system by diethylstilbestrol and ioxynil in the sea bream (Sparus aurata). Aquatic Toxicology. 92 (4), 271-280 (2009).
- Yamauchi, K., Prapunpoj, P., Richardson, S. J. Effect of diethylstilbestrol on thyroid hormone binding to amphibian transthyretins. General and Comparative Endocrinology. 119 (3), 329-339 (2000).
- Zhang, J., et al. Structure-based virtual screening protocol for in silico identification of potential thyroid disrupting chemicals targeting transthyretin. Environmental Science & Technology. 50 (21), 11984-11993 (2016).
- Ren, X. M., et al. Binding interactions of perfluoroalkyl substances with thyroid hormone transport proteins and potential toxicological implications. Toxicology. 366-367, 32-42 (2016).
- Wilson, N., Mbabazi, K., Seth, P., Smith, H., Davis, N. L. Drug and opioid-involved overdose deaths - United States, 2017-2018. Morbidity and Mortality Weekly Report. 69 (11), 290-297 (2020).
- EPA. National Pollutant Discharge Elimination System (NPDES). United States Environmental Protection Agency. , Available from: https://www.epa.gov/npdes/npdes-resources (2018).
- Duvallet, C., Hayes, B. D., Erickson, T. B., Chai, P. R., Matus, M. Mapping community opioid exposure through wastewater-based epidemiology as a means to engage pharmacies in harm reduction efforts. Preventing Chronic Disease. 17, 200053(2020).
- Gushgari, A. J., Venkatesan, A. K., Chen, J., Steele, J. C., Halden, R. U. Long-term tracking of opioid consumption in two United States cities using wastewater-based epidemiology approach. Water Research. 161, 171-180 (2019).
- Lau, B., Bretaud, S., Huang, Y., Lin, E., Guo, S. Dissociation of food and opiate preference by a genetic mutation in zebrafish. Genes Brain Behave. 5 (7), 497-505 (2006).
- Bossé, G. D., Peterson, R. T. Development of an opioid self-administration assay to study drug seeking in zebrafish. Behavioural Brain Research. 335, 158-166 (2017).
- Mottaz, H., et al. Dose-dependent effects of morphine on lipopolysaccharide (LPS)-induced inflammation, and involvement of multixenobiotic resistance (MXR) transporters in LPS efflux in teleost fish. Environmental Pollution. 221, 105-115 (2017).
- Manglik, A., et al. Crystal structure of the µ-opioid receptor bound to a morphinan antagonist. Nature. 485 (7398), 321-326 (2012).
- Comer, S. D., Cahill, C. M. Fentanyl: Receptor pharmacology, abuse potential, and implications for treatment. Neuroscience & Biobehavioral Reviews. 106, 49-57 (2019).
- Podlewska, S., Bugno, R., Kudla, L., Bojarski, A. J., Przewlocki, R. Molecular modeling of µ opioid receptor ligands with various functional properties: PZM21, SR-17018, morphine, and fentanyl-simulated interaction patterns confronted with experimental data. Molecules. 25 (20), 4636(2020).
- Huang, W., et al. Structural insights into µ-opioid receptor activation. Nature. 524 (7565), 315-321 (2015).
- Lipiński, P. F. J., et al. Fentanyl family at the mu-opioid receptor: Uniform assessment of binding and computational analysis. Molecules. 24 (4), 740(2019).
- Boland, L. A., Angles, J. M. Feline permethrin toxicity: Retrospective study of 42 cases. Journal of Feline Medicine and Surgery. 12 (2), 61-71 (2010).
- Stevenson, B. J., Pignatelli, P., Nikou, D., Paine, M. J. Pinpointing P450s associated with pyrethroid metabolism in the dengue vector, Aedes aegypti: developing new tools to combat insecticide resistance. PLoS Neglected Tropical Diseases. 6 (3), 1595(2012).
- Ankley, G. T., Gray, L. E. Cross-species conservation of endocrine pathways: A critical analysis of tier 1 fish and rat screening assays with 12 model chemicals. Environmental Toxicology and Chemistry. 32 (5), 1084-1087 (2013).
- Meteyer, C. U., Rideout, B. A., Gilbert, M., Shivaprasad, H. L., Oaks, J. L. Pathology and proposed pathophysiology of diclofenac poisoning in free-living and experimentally exposed oriental white-backed vultures (Gyps bengalensis). Journal of Wildlife Diseases. 41 (4), 707-716 (2005).
- EPA. ECOTOX User Guide: ECOTOXicology Knowledgebase System. EPA, United States Environmental Protection Agency. , Available from: https://cfpub.epa.gov/ecotox/index.cfm (2021).
- ECOS Environmental Conservation Online System. U.S. Fish & Wildlife Service. , Available from: https://ecos.fws.gov/ecp/ (2021).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados