É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Desenvolvimento de linhas de células musculares knock-out usando edição de genes CRISPR/Cas9 mediados por lentivírus
Neste Artigo
Resumo
O protocolo descreve como gerar myoblasts knock-out usando CRISPR/Cas9, desde o design de guide-RNAs até a clonagem celular e caracterização dos clones knock-out.
Resumo
Uma importante aplicação de repetições palindômicas interespaçadas interespaçadas (CRISPR)/Cas 9 é o desenvolvimento de linhas celulares knock-out, especificamente para estudar a função de novos genes/proteínas associados a uma doença, identificada durante o diagnóstico genético. Para o desenvolvimento dessas linhas celulares, duas questões principais devem ser desemaranhadas: inserção das ferramentas CRISPR (o Cas9 e o guia RNA) com alta eficiência nas células escolhidas, e restrição da atividade Cas9 à exclusão específica do gene escolhido. O protocolo descrito aqui é dedicado à inserção das ferramentas CRISPR em células difíceis de transfeito, como células musculares. Este protocolo baseia-se no uso de lentivírus, produzidos com plasmídeos disponíveis publicamente, para os quais todas as etapas de clonagem são descritas para atingir um gene de interesse. O controle da atividade cas9 tem sido realizado usando uma adaptação de um sistema descrito anteriormente chamado KamiCas9, no qual a transdução das células com um lentivírus codificando um guia de RNA visando o Cas9 permite a abolição progressiva da expressão Cas9. Este protocolo tem sido aplicado ao desenvolvimento de uma linha de células musculares humanas RYR1-knock out, que tem sido ainda caracterizada no nível proteico e funcional, para confirmar o nocaute deste importante canal de cálcio envolvido na liberação de cálcio intracelular muscular e no acoplamento excitação-contração. O procedimento descrito aqui pode ser facilmente aplicado a outros genes em células musculares ou em outras células difíceis de transfeito e produzir ferramentas valiosas para estudar esses genes em células humanas.
Introdução
Com o progresso do sequenciamento genético e a identificação de mutações em genes de funções desconhecidas em um tecido específico, o desenvolvimento de modelos celulares relevantes para entender a função de um novo gene alvo e confirmar seu envolvimento nos mecanismos fisiodicos relacionados constitui uma ferramenta essencial. Além disso, esses modelos são de grande importância para futuros desenvolvimentos terapêuticos 1,2, constituindo uma alternativa interessante para o desenvolvimento de modelos animais nocautes em linha reta com as recomendações internacionais para redução do uso de animais na experimentação. A edição de genes usando CRISPR/Cas9 está entre as ferramentas mais poderosas disponíveis atualmente, o que permitiu o desenvolvimento de muitos modelos knock-out/knock-in, e a validação de genes direcionada usando CRISPR/Cas9 está entre as aplicações mais utilizadas do CRISPR/Cas93. O sucesso da edição de genes baseia-se na capacidade de introduzir as ferramentas CRISPR (o guia RNAs e o nuclease Cas9) no modelo de célula alvo, o que pode ser um desafio em muitas células difíceis de transfectar, como as células musculares4. Esse desafio pode ser superado com o uso do vírus, geralmente o lentivírus, que tem a grande vantagem de transduzir eficientemente muitos tipos de células e entregar seu transgene. Mas sua maior desvantagem é a integração do transgene no genoma da célula hospedeira, levando a uma possível alteração de genes localizados no local de integração e à expressão permanente do transgene, o que no caso do nuclease Cas9 resultaria em consequências prejudiciais5. Uma solução inteligente foi proposta por Merienne e seus colegas6, que consiste em introdução nas células de um guia-RNA visando o próprio gene Cas9, levando à inativação cas9. Uma adaptação dessa estratégia é apresentada aqui como um protocolo fácil de usar e versátil, permitindo eliminar praticamente qualquer gene em células difíceis de transfetar.
O objetivo do protocolo aqui apresentado é induzir a inativação de um gene de interesse em células musculares imortalizadas. Pode ser usado para eliminar qualquer gene de interesse.em diferentes tipos de células imortalizadas. O protocolo descrito aqui contém etapas para projetar os RNAs guia e sua clonagem em plasmídeos lentiviral, para produzir as ferramentas CRISPR em vetores lentivirais, para transdutar as células com os diferentes lentivírus, e para clonar as células para produzir uma linha celular homogênea editada.
Usando este protocolo, células musculares esqueléticas humanas imortalizadas foram desenvolvidas com a exclusão do receptor ryanodine tipo 1 (RyR1), um canal essencial de cálcio envolvido na liberação intracelular de cálcio e contração muscular7. O knock-out (KO) do gene foi confirmado no nível de proteína usando a mancha ocidental, e no nível funcional usando imagens de cálcio.
Protocolo
As biópsias musculares foram obtidas do Banco de Tecidos para Pesquisa (Myobank, um parceiro da rede europeia EuroBioBank, Paris, França) de acordo com as recomendações europeias e a legislação francesa. O consentimento informado por escrito foi obtido de todos os indivíduos. Myoblasts imortalizados foram gentilmente produzidos pelo Dr. V. Mouly (Myology Institute, Paris, França), e os protocolos foram aprovados pelo comitê de ética do Myology Institute (MESRI, n AC-2019-3502).
1. Design do guia CRISPR
- Identifique a região do gene a ser excluída. Pesquise sua sequência genômica usando ferramentas de navegador de genoma, como ensembl.org ou genome.ucsc.edu e determine as coordenadas cromossômicas das duas regiões para procurar o RNA guia (gRNA), em ambos os lados da região a ser excluído.
- Para o gene usado aqui, obtenha a sequência FASTA do gene RYR1 e a sequência do exon 101 da seguinte forma. Em ensembl, pesquise RYR1 na versão mais recente do genoma humano, selecione a primeira entrada e clique na transcrição da sequência de codificação de proteínas. Em seguida, clique em Exons para redirecionar para a lista de exons do gene.
- Clique em Sequência de Download e selecione Somente Sequência Genômica para baixar a sequência de consenso completa de todo o gene. Role para baixo a lista dos exons e introns do gene e selecione os alvos.
- Encontre a sequência de nucleotídeos correspondentes no gene. Selecione as sequências de nucleotídeos dos introns imediatamente rio acima e rio abaixo do exon a ser excluído, que será usado para procurar os gRNAs.
- Projete os dois gRNA, chamados guia 1 e guia 2 aqui, nas regiões identificadas na etapa 1.1 (introns rio acima e rio abaixo da região a serem excluídos) usando ferramentas online como Crispor.tefor.net8. Escolha os dois gRNAs separados por algumas centenas de pares de base (bp), sendo a sequência de cada gRNA exatamente 20 nucleotídeos longos sem o motivo adjacente protoespaço (PAM). Escolha os melhores guias disponíveis para limitar o limite do alvo. Consulte a Figura 1 como um exemplo de design guia.
- Espera-se que a sequência entre os dois guias seja suprimida e, para resultar na eliminação do gene de interesse, escolha assim a posição dos dois guias de tal forma que ele exclua uma sequência essencial ou um exon essencial no gene de interesse. Certifique-se de que a sequência/exon excluído não esteja presente exclusivamente em uma transcrição alternativa do gene e/ou que ela codifica uma parte importante da proteína, portanto sua exclusão resultará em um knock-out funcional.
NOTA: Embora o local do decote Cas9 esteja previsto para ocorrer 3 bp upstream do motivo adjacente protoespaço (PAM), o decote a uma distância maior também pode ocorrer, portanto, uma boa solução é não depender da localização precisa do local do decote, como o decote em intron.
- Espera-se que a sequência entre os dois guias seja suprimida e, para resultar na eliminação do gene de interesse, escolha assim a posição dos dois guias de tal forma que ele exclua uma sequência essencial ou um exon essencial no gene de interesse. Certifique-se de que a sequência/exon excluído não esteja presente exclusivamente em uma transcrição alternativa do gene e/ou que ela codifica uma parte importante da proteína, portanto sua exclusão resultará em um knock-out funcional.
- Determine a sequência de complemento reverso (RC) para cada gRNA, sem o PAM, a fim de ter as seguintes sequências: Guia 1 e Guia 1-RC, Guia 2 e Guia 2-RC.
- Ordene os primers apresentados na Tabela 1 para realizar a clonagem dos plasmídeos. Ao longo do protocolo, use primers a uma concentração de 10 nM em H2O estéril.
NOTA: As sequências adicionadas aos gRNAs nesses primers (em negrito e sublinhados) correspondem à sequência do plasmídeo antes e depois do guia, promotor e trans-ativação-Crispr RNA (tracrRNA), respectivamente e não devem ser modificados a fim de garantir uma boa sobreposição entre os primers e o plasmid.
2. Clonagem plasmida
NOTA: Nesta etapa, os gRNAs serão inseridos na espinha dorsal plasmida para a produção de lentivírus. Um codificando os dois gRNAs é produzido pela primeira vez por sucessivas reações em cadeia de polimerase (PCR), usando os primers sobrepostos. O novo é então inserido no plasmídeo lentiviral nº 87919.
- Obtenha os seguintes plasmídeos: plasmídeo #87919, codificação para RNAs guia CRISPR em um vetor lentiviral e codificação plasmida #87904 para a sequência SpCas9 em um vetor lentiviral.
- Construção de
NOTA: O protocolo de clonagem é resumido em Figura 2.- Execute uma reação PCR (A), com 2 μL de plasmídeo #87919, 2 μL de primer_XmaIF, 2 μL de primer_Guide1R, 25 μL de mistura de polimerase e 19 μL de H2O. Execute o seguinte programa PCR (programa 1): denaturação inicial 5 min a 98 °C, seguido por 30 ciclos de: 30 s a 98 °C, 30 s a 60 °C, 1 min 45 s a 72 °C, e um alongamento final de 7 min a 72 °C. O Tm dos primers descritos na etapa 1.4 é de 60 °C.
NOTA: Embora o tempo de alongamento no programa 1 pareça bastante longo, este tempo de alongamento foi escolhido para garantir a produção de material suficiente do tamanho certo. De fato, devido às repetidas sequências no plasmídeo (a sequência de tracrRNA após guias de RNA é repetida três vezes no plasmídeo #87919) a amplificação pcr do DNA esperado é difícil, e bandas menores adicionais são produzidas nos SUCESSIVOs PCRs. Assim, devido à concorrência entre os diferentes produtos pcr, tanto o tempo de alongamento foi aumentado (para favorecer o mais longo e ter material purificado suficiente no final), ou um pcr (programa 2) de toque para o fragmento longo (como descrito para o PCR (final) na etapa 2.2.6). - Separe os produtos PCR em um gel de 1% de agarose no buffer Tris-Borate-EDTA (TBE), extirpar e purificar o fragmento de 300 bp. Realize a purificação utilizando um kit dedicado seguindo as instruções do fabricante, com eluição em um volume final de 20 μL. Utilize o fragmento purificado diretamente ou armazene a -20 °C para a etapa 2.2.5.
- Execute uma reação PCR (B), com 2 μL de plasmídeo #87919, 2 μL de primer_Guide1F, 2 μL de primer_Guide2R, 25 μL de mistura de polimerase e 19 μL de H2O usando o programa PCR 1. Separe os produtos PCR em um gel de 1% de agarose em TBE, extirpar e purificar o fragmento de 400 bp em 20 μL de tampão de eluição. Use o fragmento purificado diretamente ou armazene a -20 °C para a etapa 2.2.5.
- Execute uma reação PCR (C), com 2 μL de plasmídeo #87919, 2 μL de primer_Guide2F, 2 μL de primer_BlpIR, 25 μL de mistura de polimerase e 19 μL de H2O usando pcr programa 1. Separe os produtos PCR em um gel de 1% de agarose no buffer TBE. Extir esse e purifique o fragmento de 600 bp em 20 μL de tampão de eluição. Use o fragmento purificado diretamente ou armazene a -20 °C para a etapa 2.2.6.
NOTA: Outro fragmento a 900 bp pode ser visível, devido à hibridização da cartilha nas regiões repetidas do plasmídeo, conforme descrito na NOTA da etapa 2.2.1. Se estiver presente, esta banda deve ser descartada. - Execute uma reação pcr (D), com 2 μL de elução PCR A (a partir da etapa 2.2.2), 2 μL de elução PCR B (a partir da etapa 2.2.3), 2 μL de primer_XmaIF, 2 μL de primer_Guide2R, 25 μL de mistura polimerase e 19 μL de H2O, com programa pcr 1. Separar os produtos PCR em um gel de 1% de agarose no buffer TBE; excisar e purificar o fragmento de 700 bp em 20 μL de tampão de eluição. Use o fragmento purificado diretamente ou armazene a -20 °C para a etapa 2.2.6.
NOTA: Outras bandas podem ser visíveis em > 1.000 bp, 400 bp e 300 bp, devido à hibridização dos primers nas regiões repetidas e devem ser descartadas. - Execute uma reação PCR (final), com 4 μL de elução PCR C (a partir da etapa 2.2.4), 4 μL de elução PCR D (a partir da etapa 2.2.5), 4 μL de primer_XmaIF, 4 μL de primer_BlpIR, 50 μL de mistura de polimerase e 34 μL de H2O. O programa utilizado é o seguinte (programa PCR 2): desnaturação inicial por 5 min a 98 °C; seis ciclos de: 30 s a 98 °C, 30 s a 66 °C (redução na temperatura de hibridização de 1 °C por ciclo), 1 min 45 a 72 °C; 35 ciclos de: 30 s a 98 °C, 30 s a 60 °C, 1 min 45 a 72 °C, e um alongamento final de 5 min a 72 °C.
NOTA: O programa PCR para esta amplificação final é um PCR touch down, diferente do anterior, devido ao grande tamanho da amplificação final que contém duas sequências repetidas de tracrRNA logo após cada guia. - Separar os produtos PCR em um gel de 1% de agarose no buffer TBE; excisar e purificar o final, que migra a cerca de 1.300 bp em 20 μL de tampão de eluição. Quantifique o produto elucido. Use o fragmento purificado diretamente ou armazene a -20 °C para a etapa 2.3.2.1. Este é o produto final que será inserido no plasmídeo lentiviral.
NOTA: Outros fragmentos podem ser visíveis em >1000 bp, 400 bp e 300 bp, correspondentes a fragmentos incompletos de PCR que devem ser descartados.
- Execute uma reação PCR (A), com 2 μL de plasmídeo #87919, 2 μL de primer_XmaIF, 2 μL de primer_Guide1R, 25 μL de mistura de polimerase e 19 μL de H2O. Execute o seguinte programa PCR (programa 1): denaturação inicial 5 min a 98 °C, seguido por 30 ciclos de: 30 s a 98 °C, 30 s a 60 °C, 1 min 45 s a 72 °C, e um alongamento final de 7 min a 72 °C. O Tm dos primers descritos na etapa 1.4 é de 60 °C.
- Inserção do gRNAs na espinha dorsal plasmida lentiviral.
- Linearize o plasmídeo por dupla digestão do plasmídeo #87919 com as enzimas de restrição XmaI e BlpI.
- Prepare a reação com 15 μL de tampão recomendado, 15 μL de plasmídeo (1μg/μL), 7,5 μL de enzima BlpI (a 10 U/μL), 7,5 μL de enzima XmaI (a 10 U/μL) e 112,5 μL de H O.Incubar por 1h a 37 °C e, em seguida, para 20 min a 65 °C. Carregue a quantidade total em um gel de 1% de agarose, corte e purifique o plasmídeo ~10 kb com um kit apropriado. A elução é realizada em 20 μL de tampão, e o produto elucido é quantificado por meio da medição da densidade óptica.
NOTA: Use um protocolo de purificação adequado para grande fragmento de DNA, como o protocolo descrito por Sun e o coll9.
- Prepare a reação com 15 μL de tampão recomendado, 15 μL de plasmídeo (1μg/μL), 7,5 μL de enzima BlpI (a 10 U/μL), 7,5 μL de enzima XmaI (a 10 U/μL) e 112,5 μL de H O.Incubar por 1h a 37 °C e, em seguida, para 20 min a 65 °C. Carregue a quantidade total em um gel de 1% de agarose, corte e purifique o plasmídeo ~10 kb com um kit apropriado. A elução é realizada em 20 μL de tampão, e o produto elucido é quantificado por meio da medição da densidade óptica.
- Ligate o gRNA e o plasmid. Prepare a mistura de reação com o gRNA da etapa 2.2.7 e o plasmídeo linearizado da etapa 2.3.1.1, adicione 2 μL de enzima e H2O tenha um volume final de 10 μL. Incubar por 15 min a 50 °C para produzir o plasmídeo final chamado p_guides.
NOTA: A quantidade de de DNA deve ser entre 50-100 ng e quantidade plasmida entre 100-200 ng, com uma razão molar de 2:1.
- Linearize o plasmídeo por dupla digestão do plasmídeo #87919 com as enzimas de restrição XmaI e BlpI.
- Use 2 μL do plasmídeo recém-preparado para transformar E.Coli quimicamente competente, como Stbl3 (50 μL) ou XL10-Gold e espalhe na placa de ágar LB com 100 μg/mL ampicillin após 1 h de crescimento a 37 °C sem antibiótico. Incubar a 37 °C durante a noite. Escolha algumas colônias e realize uma mini preparação usando um kit comercial seguindo as instruções do fabricante.
- Realize uma amplificação pcr no DNA miniprep com Primer_XmaI e Primer_BlpR usando o programa PCR 1 (ver Figura 2). Separe os produtos PCR em um gel de 1% de agarose no buffer TBE. Selecione algumas colônias (~5) com uma banda no tamanho esperado de cerca de 1300 bp.
- Realize o sequenciamento de DNA das colônias selecionadas, utilizando o Primer_XmaI ou Primer_BlpR, para confirmar a inserção correta do gRNA.
NOTA: Uma das colônias verificadas por sequência é mais utilizada no estudo, e o plasmídeo é chamado de p_guides. - Repita a partir da etapa 2.2 (construção de) com os Primers-Killer F e R, e os Primers-mCherry F e R. Use uma colônia verificada por sequência para análise posterior. O plasmid é chamado p_Killer.
3. Produção de lentivírus
- Produza e purifique uma grande quantidade de todos os plasmídeos necessários usando um kit maxi-prep livre de endotoxina seguindo as instruções do fabricante. Prepare as alíquotas a 2 μg/μL. Armazene a -20 °C
- Preparação de células (Dia 1)
- Prepare 18 placas de 145 cm semeadas com 1 x 106 células HEK293 por placa em 16 mL de meio composto pelo piruvato de alta glicose (DMEM) de Dulbecco, suplementado com 10% de soro bovino fetal (FBS) e 1% penicilina/estreptomicina. Amplie as células a 37 °C, em uma incubadora de 5% de CO2 por 3 dias.
NOTA: A produção de lentivírus deve ser realizada com cautela em um laboratório de Biossegurança Nível 2, utilizando engrenagens de proteção adaptadas, incluindo traje de proteção descartável, tampa de proteção e luvas. Todos os experimentos devem ser feitos sob um capô de fluxo laminar (gabinete de segurança BSLII) com pontas de filtro. Toda a solução contendo lentivírus e todos os resíduos de plástico/vidro usados devem ser inativados com etanol 70% ou outro inativador de vírus.
- Prepare 18 placas de 145 cm semeadas com 1 x 106 células HEK293 por placa em 16 mL de meio composto pelo piruvato de alta glicose (DMEM) de Dulbecco, suplementado com 10% de soro bovino fetal (FBS) e 1% penicilina/estreptomicina. Amplie as células a 37 °C, em uma incubadora de 5% de CO2 por 3 dias.
- Transfecção de células (Dia 4)
- Verifique a confluência das células, para ter certeza de que atingiram 60%-65% de confluência.
- Prepare a solução de transfecção contendo para cada placa: 20,8 μg do plasmídeo de interesse (p-guias, ou p-Killer, ou pCas9 #87904), 4,8 μg da codificação plasmida do envelope (VSV-G, #8454), 20,8 μg do plasmid psPAX2 (#12260) para embalagem lentiviral, 136 μL de fosfa de cálcio ajustar e ajustar com H2O a um volume final de 1.000 μL. Adicione esta solução dropwise e sob agitação a 1 mL de soro fisiológico 2x hepes-tampão (HBS).
NOTA: Não prepare uma mistura para todas as placas ao mesmo tempo para garantir uma preparação ideal dos reagentes; preparar uma mistura para seis placas ao mesmo tempo, por exemplo, para 18 placas, preparar três vezes uma mistura para seis placas. - Incubar à temperatura ambiente (RT) por pelo menos 10 min e adicionar 2 mL de solução dropwise às células. Homogeneize o reagente de transfecção com agitação suave da placa para trás, para frente, para cima e para baixo, incubar a 37 °C, 5% de CO2 por pelo menos 5 h.
NOTA: A partir deste ponto e até o final da produção de lentivírus, use equipamentos de proteção adicionais, incluindo um segundo par de luvas, mangas de proteção e um plastron descartável. - Cinco horas após a transfecção, retire o meio das placas e enxágue com PBS para se livrar dos reagentes de transfecção. Adicione 12 mL de meio fresco e incubar 48 h a 37 °C, 5% de CO2.
- Coleta das partículas virais (Dia 6)
- Colete e junte o meio de todas as placas. Centrifugar a 800 x g por 5 min a 4 °C, para pelotar detritos celulares. Filtre o supernatante usando um filtro de 0,45 μm (múltiplos filtros necessários).
- Centrifugar a 68.300 x g por 2 h a 4 °C em um rotor de balde balançando. Remova o supernatante e deixe os tubos de cabeça para baixo sob o gabinete de segurança em um papel por 5-10 min para remover o máximo de líquido possível e, em seguida, adicione 100 μL de meio de proliferação HEK por pelota. Depois de pelo menos 2h a 4 °C, resuspense as pelotas pipetando para cima e para baixo. Acumule todas as pelotas resuspended.
NOTA: As pelotas podem ser deixadas no meio durante a noite a 4 °C antes de aliquotar. - Lentivírus de alíquota em tamanho amostral de 10 μL ou 25 μL (dependendo do uso) e snap-freeze com nitrogênio líquido. Armazém a -80 °C. Não congele uma alíquota que tenha sido descongelada.
- Repita as etapas 3.2 a 3.4 com outros plasmídeos de interesse para produzir guias LV, LV-Killer e LV-Cas9.
NOTA: Como alternativa, os lentivírus podem ser adquiridos em uma empresa ou em uma instalação de vírus.
4. Titulação de lentivírus
NOTA: A titulação do vírus é realizada em células HEK293. A titulação é importante para incorporar nas etapas subsequentes um número preciso de lentivírus por célula (qualquer que seja o lote de lentivírus), para as células de interesse. O número de partículas virais que transduem eficientemente uma célula é chamado de multiplicidade de infecção (MOI): MOI 10 correspondem assim à introdução de 10 partículas virais por célula. Como o ciclo de congelamento/descongelamento afeta a viabilidade do lentivírus, a titulação é realizada com um aliquot lentivírus congelado, e cada experimento subsequente será realizado com uma nova alíquota da mesma piscina. Um método de titulação é descrito aqui, mas outros métodos podem ser usados.
- No dia 1, sementes 1 x 105 células por placa em cinco placas de 35 mm com tampas de vidro na parte inferior e duas placas de 35 mm sem tampas.
- No dia 2, das duas placas sem tampas, colete e conte o número de células após a experimentação, e determine a quantidade média de células por placa (N).
- Prepare 100 μL de lentivírus diluído a 1/10 em meio de proliferação. Transduza as cinco placas cultivadas com diferentes volumes de vírus diluídos, de 1 a 50 μL de vírus diluído. Para este protocolo, utilize os seguintes volumes para transduzir as cinco placas: 1 μL, 5 μL, 10 μL, 20 μL, 50 μL. Incubar por 48 h a 37 °C, em um CO2 de 5%.
- No dia 4, fixar as células por incubação das tampas na RT por 20 a 30 min em 4% de paraformaldeído. Para o LV-Cas9, permeabilize as células com 0,1% Triton X100 em Salina Tampão fosfato (PBS) por 10 min na RT, saturado em PBS-0,1% Triton X100-2% Soro de Cabra- 0,5% Bovine Serum Albumin (BSA) por 20 min na RT, e rótulo para 45 min na RT com anticorpos de anticorpo primário V5 (diluição 1/400) seguido de incubação de 30 min na RT com anticorpo fluorescente secundário. Rotule os núcleos com Hoescht (10 μg/mL em PBS) por 10 min na RT.
- Monte as tampas em um slide e observe usando um microscópio fluorescente equipado com um objetivo de 20x. Para guias LV e LV-Killer, após a fixação proceda diretamente para a rotulagem dos núcleos e montar as tampas. Para cada deslizamento de cobertura, conte o número total de núcleos (número total de células) no campo de visão e o número de células rotuladas (com V5 ou mCherry), e determine a razão de células rotuladas para cada deslizamento de cobertura.
- Selecione um deslizamento em que a razão de células transduzidas seja de pelo menos 10% e não exceda 50%. Determine a eficiência de transdução (X) para este deslizamento de cobertura, e observe o volume do vírus diluído (V, em μL) utilizado para obter esta eficiência de transdução.
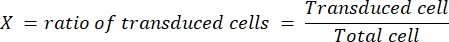
- Determine o título (em partículas infecciosas, representadas como ip/mL) do vírus de acordo com a seguinte fórmula:

O fator de diluição é a diluição do lentivírus realizado na etapa 4.3. Nós rotineiramente obtivemos um título final de 1 x 109 ip/mL para LV-Cas9 e 1 x 10ip /mL para LV-guide, determinado em células HEK.
5. Transdução de myoblast
NOTA: Os míbios imortalizados são sucessivamente transduzidos com os três lentivírus produzidos anteriormente. Eles são mantidos em uma densidade abaixo de 50% em um meio de proliferação composto de F10 de Ham suplementado com 20% de FBS, 2% penicilina/estreptomicina, 2% Ultroser G, e cultivado a 37 °C, 5% CO2.
- Determine o volume de lentivírus necessário para tratar o número escolhido de células com MOI 10 para LV-Cas9 e LV-guias e MOI 20 para LV-killer, de acordo com a seguinte fórmula:
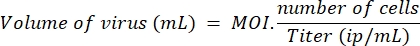
NOTA: Em um experimento paralelo, a eficiência de transdução foi comparada em myoblasts e HEK, usando um lentivírus de controle (lenti-GFP), e determinamos que cinco vezes mais lentivírus são necessários para transduzir um myoblast eficientemente em comparação com uma célula HEK. Assim, moi 10 medido em HEK corresponde a duas partículas virais por myoblast. O MOI usado aqui é calculado em células HEK. - No primeiro dia, sementes 96-poços placas com 10.000 células em 100 μL de meio de proliferação por poço. No dia 2, transduza as células sob o gabinete de segurança adicionando o volume apropriado de guias LV e LV-Cas9 calculado na etapa 5.1. Devolva as células à incubadora até o dia 7.
NOTA: O uso de MOI superior a 25, especialmente para LV-Cas9, pode não melhorar o número de células editadas devido à maior morte celular em uma alta concentração de lentivírus. - No dia 7, realize a experimentação e conte as células. Semear as células a uma confluência de 40% a 50% em uma nova placa e devolver as células à incubadora. Cinco horas depois, transduzi as células com LV-Killer em um MOI de 20 (volume calculado na etapa 5.1). Amplifique as células por 5 a 10 dias após a transfecção, por pelo menos duas passagens, e mantenha-as sempre em baixa confluência de <50%. As células estão prontas para o próximo passo, clonagem celular, quando seu crescimento é devolvido ao normal (estimado pelo tempo de divisão).
NOTA: A proliferação pode ser um pouco mais lenta após a transdução. O crescimento normal das células pode ser determinado antes deste procedimento experimental pela estimativa do tempo de divisão das células.
6. Clonagem celular
NOTA: Como a transdução myoblast é difícil e nunca atinge 100% de eficiência, mesmo quando se usa lentivírus, a clonagem celular é necessária para obter uma linha celular totalmente corrigida. Isso só é possível com células imortalizadas, ou células que podem ser cultivadas e amplificadas durante algumas semanas/meses.
- Tentepsinizar e contar as células. Diluir as células em meio de proliferação a 10 células/mL e semear as células em 1 célula/poço em placas de 96 poços contendo 100 μL/bem de médio.
NOTA: O número de placas a serem semeadas depende da probabilidade de edição de genes esperado, 2 a 10 placas são usadas rotineiramente. - Monitore as células para crescimento e amplie progressivamente cada poço em uma placa maior até atingir pelo menos uma placa de 35 mm, mantendo a confluência dos míobios abaixo de 50%. Esta etapa pode durar de 2 a 6 semanas, dependendo das células usadas e sua capacidade de crescer uma vez isolada em um poço.
7. Seleção de clones
NOTA: Esta etapa é realizada para identificar quais dos clones em crescimento foram adequadamente modificados.
- Projete um conjunto de primers compostos por uma cartilha localizada antes do primeiro guia (Primer_BeforeGuide1F) e outra cartilha localizada após o segundo guia (Primer_AfterGuide2R) a fim de amplificar a região que inclui a sequência putativamente modificada. Consulte a Tabela 1 para os primers usados aqui.
- Coletar células de cada clone, poupar pelo menos 300.000 células para amplificação futura e extrair DNA genômico usando qualquer protocolo padrão nas células restantes.
- Se um grande número de clones estão crescendo, para descartar os não editados, realize um teste rápido reunindo células de cinco clones no mesmo tubo para extrair o DNA desta piscina e testar com PCR. Repita com quantos clones for necessário. Em seguida, separe ainda mais as piscinas que continham células editadas para realizar análises individuais.
- Controle a edição pelo PCR da seguinte forma. Prepare a reação do PCR com 1 μL de Primer_BeforeGuide1F, 1 μL de Primer_AfterGuide2R, 12,5 μL de mistura de polimerase, 3 μL de DNA genômico e 7,5 μL de H2O. Amplificar em um termociclador de acordo com as instruções e parâmetros do fabricante. Execute um gel de 1% de agarose para identificar os clones editados.
- Controle a edição sequenciando da seguinte forma. Execute o sequenciamento Sanger dos clones selecionados para confirmar a exclusão e identificar como a edição foi realizada em cada clone. Mantenha mais de um clone editado para garantir que apenas o gene alvo tenha sido modificado e seja responsável pelo efeito fisiológico observado e mantenha um clone não editado que será usado como um clone de controle (CTRL) nos experimentos subsequentes.
- Expanda os clones selecionados. Uma vez que a confluência de cada clone tenha atingido cerca de 50%, tente as células e plaque as células em um prato maior, até que células suficientes tenham sido produzidas para realizar as caracterizações bioquímicas e funcionais (geralmente mais de 1 x 106 por clone), e armazenar alíquotas congeladas de cada clone para uso futuro.
8. Caracterização de clones editados
NOTA: Uma vez que alguns clones tenham sido colhidos e confirmados pelo sequenciamento de DNA, a exclusão do gene alvo pode ser confirmada no nível de proteína usando a mancha ocidental, e no nível funcional se um ensaio celular funcional estiver disponível para este gene. No caso do RYR1-KO, como o RyR1 é um canal de cálcio, a caracterização funcional tem sido realizada usando imagens de cálcio em células cultivadas.
- Expressão proteica em clones editados
NOTA: O RyR1 é expresso apenas em myotubesdiferenciados 10. Sua expressão foi avaliada em mobotos usando mancha ocidental, para confirmar a exclusão ao nível proteico de RyR1, bem como a exclusão da proteína Cas9.- Placa 200.000 células em meio de proliferação (descrito acima, passo 5) em uma superfície de cerca de 1,76 cm2 em uma placa de 35 mm revestida com laminina (superfície correspondente a uma gota de 200 μL de laminina a 10 mg/mL em PBS com cálcio). Uma vez que as células estejam presas à placa após serem incubadas por 2-3 h a 37 °C, 5% de CO2, mude o meio de cultura para um meio de diferenciação composto de DMEM baixa glicose + 10% soro de cavalo + 1% penicilina/estreptomicina, e devolva as células à incubadora por 6 dias.
- Após 6 dias de diferenciação, colete elise as células com 200 μL de RIPA suplementados com inibidores de protease. Determine a concentração de proteínas usando o método Folin Lowry11.
- Carregar 15 μg de proteína, após a desinaturação por 30 min no RT em tampão de desnaturação laemmli, em um gel de acrilamida gradiente de 5%-15%. Após a separação eletroforética, transfira as proteínas em Immobilon P a 0,8 V por 4 h11.
- Após a saturação da membrana por 30 min na RT em PBS contendo 0,1% Tween 20 e 5% leite seco sem gordura, incubar a membrana com os anticorpos primários diluídos no mesmo tampão por 2h no RT ou durante a noite a 4 °C, lavar a membrana 5x por 5 min com PBS-0,1% Tween 20 e incubar a membrana com os anticorpos secundários por 1 h na RT. Os anticorpos primários utilizados são: anticorpos contra V5-tag (diluição: 1/5000) para detectar Cas9, anti-GAPDH (diluição: 1/1000) como um controle de carregamento, anticorpo anti-RyR112,13 (diluição: 1/10.000), anticorpo contra a subunidade alfa 1 de DHPR (diluição: 1/1000) e anticorpo contra a cadeia pesada de miosina MF20 (diluição: 1/1000).
- Lave a membrana 5x por 5 min com PBS-0,1% Tween 20, seque o excesso de líquido e adicione o substrato quimiluminescente. Proceda conforme recomendado pelo provedor de substrato para detectar o sinal quemiluminescente.
- Caracterização funcional de clones editados
NOTA: A função do RyR1 foi avaliada utilizando-se imagens de cálcio em miotutos diferenciados, produzidos a partir de clones CTRL ou KO14.- Placa 50.000 células em uma superfície de 0,2 cm2 no centro de 35 mm de pratos revestidos com laminina (superfície coberta por uma gota de laminina de 50 μL, a 10 mg/mL em PBS com cálcio) e induzir diferenciação por 6 dias como descrito na etapa 8.1.1. Prepare três pratos para cada estimulação, para ter um triplicado biológico.
- Carregue os miotubes com 50 μL de fluo 4-direto, diluído 1:1 em meio de diferenciação e incubado por 30 min a 37 °C. Enxágüe as células duas vezes com tampão KREBS suplementado com glicose a 1 mg/mL.
- Meça as variações de fluorescência com um microscópio fluorescente invertido ou um microscópio confocal usando um objetivo de 10x. Instale a placa no estágio do microscópio e inicie a aquisição a 1 quadro por segundo para 90 s.
- Remova o KREBS restante e estimule as células no quadro 25 por adição de 2 mL de KCl para despolarização de membrana (concentração final de 140 mM) ou 2 mL de 4 CmC (concentração final de 500 μM) para estimulação direta de RyR1. Certifique-se de que pelo menos 10 miotubes estão presentes no campo gravado.
- Quantifique a variação da fluorescência em cada miotube, usando um software dedicado. Selecione para análise pelo menos 10 miótubos por prato (idealmente 20-30 miotubes por prato), desenhe uma linha (ou uma Região de Interesse (ROI)) no longo eixo de cada miotube e colete a fluorescência F ao longo desta linha para todos os quadros.
- Determine o valor fluorescente inicial, F0, correspondente aos quadros 1 a 24. Plote a variação fluorescente (F-F0)/F0 em função do tempo de 0 a 90 s. Repita o experimento três vezes para obter a variação de fluorescência de pelo menos 90 miotubes de três culturas diferentes. Acumule todos os resultados para os 90 miotubes e calcule a média ± SEM de (F-F0)/F0 em cada período de tempo. Quantifique o pico de amplitude da liberação de cálcio para cada estimulação e cada clone.
Resultados
Este protocolo foi aplicado a míbios imortalizados de um sujeito saudável15 (as chamadas células HM, para míbios humanos), em que o RyR1 foi anteriormente caracterizado16, a fim de derrubar o gene RYR1 codificando a proteína RyR1. O design dos guias RNA foi feito para excluir a sequência que abrange parte do exon 101 e intron 101 do gene. A exclusão de parte do exon 101 está prevista para resultar em interrupção do quadro de leitura. Além disso, o exon 1...
Discussão
Um grande passo no caminho para a caracterização de genes de função desconhecida envolvidos em patologias é o desenvolvimento de modelos celulares relevantes para estudar a função desses genes. O uso da edição de genes usando CRISPR/Cas9 é um campo de pesquisa em crescimento exponencial, e o desenvolvimento de modelos de knock-out como apresentado aqui está entre suas aplicações mais utilizadas. Nesse contexto, propomos aqui um protocolo versátil para desenvolver uma linha celular humana em qualquer gene de...
Divulgações
Os autores não têm conflitos de interesse para divulgar.
Agradecimentos
Este trabalho foi financiado por subsídios da Associação Francesa contre les myopathies (AFM-Téléthon) e da Auvergne-Rhône Alpes Région (AURA).
Materiais
| Name | Company | Catalog Number | Comments |
| Anti-CACNA1S antibody | Sigma-Aldrich | HPA048892 | Primary antibody |
| Blp I | NE BioLabs | R0585S | Restriction enzyme |
| CalPhos Mammalian Transfection Kit | Takara | 631312 | Transfection kit |
| Easy blot anti Mouse IgG | GeneTex | GTX221667-01 | HRP secondary antibody |
| Easy blot anti Rabbit IgG | GeneTex | GTX221666 | HRP secondary antibody |
| Fluo-4 direct | Molecular Probes | F10472 | Calcium imaging |
| GAPDH(14C10) Rabbit mAb | Cell Signaling Technology | #2118 | Primary antibody |
| HindIII | Fermentas | ER0501 | Restriction enzyme |
| InFusion HD Precision Plus | Takara | 638920 | Ligation kit |
| MasterMix Phusion High Fidelity with GC | ThermoFisher Scientific | F532L | Mix for PCR reaction with High fidelity Taq polymerase and dNTPs |
| Myosin Heavy Chain antibody | DHSB | MF20 | Primary antibody |
| NucleoBond Xtra Maxi EF | Macherey-Nagel | REF 740424 | Maxipreparation kit for purification of plasmids |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609 | DNA purification |
| NucleoSpin Tissue | Macherey-Nagel | 740952 | Kit for DNA extraction from cell |
| One Shot Stbl3 Chemically Competent E. coli | ThermoFisher Scientific | C737303 | Chemically competent cells |
| Plasmid #87904 | Addgene | 87904 | Lentiviral plasmid encoding the SpCas9 (for LV-Cas9) |
| Plasmid #87919 | Addgene | 87919 | Lentiviral backbone for insertion of cassette with guides (for LV-guide-target) |
| Plasmid #12260 | Addgene | 12260 | Lentiviral plasmid encoding lentiviral packaging GAG POL |
| Plasmid #8454 | Addgene | 8454 | Lentiviral plasmid encoding envelope protein for producing lentiviral and MuLV retroviral particles |
| V5 Tag Monoclonal Antibody | Invitrogene | R96025 | Primary antibody |
| XL10-Gold Ultracompetent Cells | Agilent | 200317 | Chemically competent cells |
| Xma I | NE BioLabs | R0180S | Restriction enzyme |
Referências
- Claussnitzer, M., Susztak, K. Gaining insight into metabolic diseases from human genetic discoveries. Trends in Genetics. 37 (12), 1081-1094 (2021).
- Fuster-García, C., García-Bohórquez, B., Rodríguez-Muñoz, A., Millán, J. M., García-García, G. Application of CRISPR tools for variant interpretation and disease modeling in inherited retinal dystrophies. Genes. 11 (5), 473 (2020).
- Modell, A. E., Lim, D., Nguyen, T. M., Sreekanth, V., Choudhary, A. CRISPR-based therapeutics: current challenges and future applications. Trends in Pharmacological Sciences. 43 (2), 151-161 (2022).
- Olson, E. N. Toward the correction of muscular dystrophy by gene editing. Proceedings of the National Academy of Sciences of the United States of America. 118 (22), (2021).
- Wu, X., Kriz, A. J., Sharp, P. A. Target specificity of the CRISPR-Cas9 system. Quantitative Biology. 2 (2), 59-70 (2014).
- Merienne, N., et al. The self-inactivating KamiCas9 system for the editing of CNS disease genes. Cell Reports. 20 (12), 2980-2991 (2017).
- Marty, I., Fauré, J. Excitation-contraction coupling alterations in myopathies. Journal of Neuromuscular Diseases. 3 (4), 443-453 (2016).
- Concordet, J. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Research. 46, 242-245 (2018).
- Sun, Y., Sriramajayam, K., Luo, D., Liao, D. J. A quick, cost-free method of purification of DNA fragments from agarose gel. Journal of Cancer. 3, 93-95 (2012).
- Flucher, B. E., Conti, A., Takeshima, H., Sorrentino, V. Type 3 and type 1 ryanodine receptors are localized in triads of the same mammalian skeletal muscle fibers. The Journal of Cell Biology. 146 (3), 621-630 (1999).
- Hess, H. H., Lees, M. B., Derr, J. E. A linear Lowry--Folin assay for both water-soluble and sodium dodecyl sulfate-solubilized proteins. Analytical Biochemistry. 85 (1), 295-300 (1978).
- Garibaldi, M., et al. Dusty core disease' (DuCD): expanding morphological spectrum of RYR1 recessive myopathies. Acta Neuropathologica Communications. 7 (1), 3 (2019).
- Marty, I., et al. Biochemical evidence for a complex involving Dihydropyridine receptor and Ryanodine receptor in triad junctions of skeletal muscle. Proceedings of the National Academy of Sciences of the United States of America. 91 (6), 2270-2274 (1994).
- Oddoux, S., et al. Triadin deletion induces impaired skeletal muscle function. Journal of Biological Chemistry. 284 (50), 34918-34929 (2009).
- Mamchaoui, K., et al. Immortalized pathological human myoblasts: towards a universal tool for the study of neuromuscular disorders. Skeletal Muscle. 1, 34 (2011).
- Cacheux, M., et al. Functional characterization of a central core disease RyR1 mutation (p.Y4864H) associated with quantitative defect in RyR1 protein. Journal of Neuromuscular Diseases. 2 (4), 421-432 (2015).
- Luis, A. The old and the new: Prospects for non-integrating lentiviral vector technology. Viruses. 12 (10), 1103 (2020).
- Leenay, R. T., Beisel, C. L. Deciphering, communicating, and engineering the CRISPR PAM. Journal of Molecular Biology. 429 (2), 177-191 (2017).
- Salmon, P., Trono, D. Production and titration of lentiviral vectors. Current Protocols in Neurosciences. , (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados