É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Análise da Forma da Linha de Espectros Dinâmicos de RMN para Caracterização de Rearranjos de Esferas de Coordenação em um Complexo de Polihidreto de Rênio Quiral
Neste Artigo
Resumo
A análise da forma da linha dos espectros de RMN coletados em uma faixa de temperaturas serve como um guia para o rearranjo de átomos da esfera de coordenação interna em um complexo de polihidreto de rênio(V) quiral, de oito coordenadas, ReH5(PPh3)2(sec-butil amina). A análise da forma da linha também é usada para determinar os parâmetros de ativação ΔH‡, ΔS‡ e ΔG‡ para esses rearranjos de átomos.
Resumo
A espectroscopia de ressonância magnética nuclear (RMN) de solução dinâmica é o método típico de caracterização dos rearranjos dinâmicos de átomos dentro da esfera de coordenação para complexos de polihidreto metálico de transição. O ajuste da forma da linha dos espectros dinâmicos de RMN pode levar a estimativas para os parâmetros de ativação dos processos de rearranjo dinâmico. Uma combinação de espectroscopia dinâmica de RMN 31 P-{1 H} de átomos de fósforo ligados a metais com espectroscopia dinâmica de RMN de 1H-{31P} de ligantes de hidreto pode identificar rearranjos de ligantes de hidreto que ocorrem em conjunto com um rearranjo de átomos de fósforo. Para moléculas que exibem tal par acoplado de rearranjos, a espectroscopia dinâmica de RMN pode ser usada para testar modelos teóricos para os rearranjos de ligantes. A espectroscopia dinâmica de RMN H-{31P} e o ajuste da forma da linha também podem identificar a presença de um processo detroca que move um ligante de hidreto específico para além da esfera de coordenação interna do metal através de uma troca de prótons com uma molécula de solvente, como água adventícia. A preparação de um novo composto, ReH5(PPh3)2(sec-butil amina), que exemplifica múltiplos processos de rearranjo dinâmico é apresentada juntamente com o ajuste da forma da linha dos espectros dinâmicos de RMN do complexo. Os resultados do ajuste da forma da linha podem ser analisados pela equação de Eyring para estimar os parâmetros de ativação para os processos dinâmicos identificados.
Introdução
A espectroscopia de RMN é comumente usada para caracterizar processos dinâmicos que ocorrem dentro ou entre moléculas. Para muitos rearranjos intramoleculares simples, a estimativa de ΔG‡ é tão direta quanto medir a diferença de frequência, Δν, entre duas ressonâncias no limite de troca lenta e determinar a temperatura de coalescência para essas mesmas ressonâncias (Figura 1)1. A relação,
ΔG‡ = 4,575 x 10-3 kcal/mol x T c [9,972 + log (Tc/Δν)]
onde Tc é a temperatura de coalescência para um par de ressonâncias que representam a forma de troca lenta de uma amostra dinâmica, pode ser usada para resolver a energia livre de ativação para tal rearranjo dinâmico. Sistemas dinâmicos mais complexos requerem ajuste de forma de linha de espectros dinâmicos de RMN ou outra técnica de RMN, como espectroscopia de troca bidimensional (2D-EXSY) ou espectroscopia bidimensional de efeito Overhauser de quadro rotativo (2D-ROESY) para estimar parâmetros de ativação.
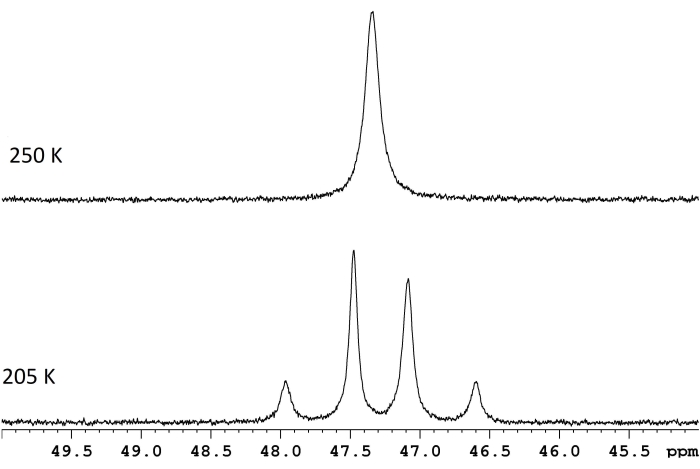
Figura 1: Espectros de RMN para uma solução de d 8-tolueno de ReH5(PPh3)2(sec-butil amina) a duas temperaturas. A diferença de frequência entre os dois duplos de troca lenta (traço inferior, 117,8 Hz) e uma temperatura de coalescência de 250 K (traço superior) correspondem a uma barreira de energia (ΔG‡) de 11,8 kcal/mol. Clique aqui para ver uma versão maior desta figura.
O ajuste da forma da linha dos espectros dinâmicos de RMN é uma técnica comum que tem sido usada há muito tempo para a estimativa de parâmetros de ativação que descrevem rearranjos dinâmicos para substâncias com uma energia de ativação de aproximadamente 5 a 25 kcal/mol 2,3,4,5. A determinação das barreiras energéticas à troca de prótons entre as moléculas de água e amina6, a barreira de energia à rotação sobre a ligação C-N na dimetilformamida7 ou o tamanho geral das metades orgânicas8 são apenas alguns exemplos das muitas propriedades que foram avaliadas através do ajuste da forma da linha de espectros dinâmicos de RMN. Este manuscrito demonstra o uso do ajuste da forma da linha para caracterizar os processos dinâmicos intermoleculares e intramoleculares que ocorrem para o complexo ReH5(PPh3)2(sec-butil amina). Os objetivos deste e de experimentos similares de RMN de forma de linha de ajuste são: 1) caracterizar todos os processos de troca de átomos dinâmicos intramoleculares observáveis de RMN, se presentes, 2) identificar e caracterizar processos de troca de átomos dinâmicos intramoleculares observáveis de RMN, se presentes, 3) identificar trocas de átomos intramoleculares correlacionadas que ocorrem para, neste exemplo, átomos de hidrogênio e fósforo, e 4) para o exemplo aqui apresentado, comparar dois modelos publicados para os processos dinâmicos que ocorrem no complexo ReH5(PPh3)2(sec-butil amina).
Sistemas de polihidreto de rênio(V) de oito coordenadas são sistemas dinâmicos complexos nos quais os ligantes participam de múltiplos processos dinâmicos e os átomos de fósforo podem participar de um único processo dinâmico que é um segundo aspecto de um processo de troca de ligantes de hidreto 9,10,11,12,13,14,15,16,17,18 ,19,20,21,22,23,24,25,26,
27,28,29. Complexos de polihidreto de rênio(V) pseudododecaédricos de oito coordenadas adotam uma geometria molecular (Figura 2), que pode ser descrita como um par de trapézios ortogonais de ligantes17,26. Os vértices nas bordas longas dos trapézios são comumente rotulados como locais B e, em complexos de polihidreto de rênio, são geralmente os locais ocupados por ligantes doadores neutros de dois elétrons, como fosfinas terciárias ou ligantes de amina. Os vértices nas bordas curtas dos trapézios são comumente rotulados como locais A e são tipicamente ocupados por ligantes aniônicos de dois elétrons doadores e hidretos. Os espectros de RMN à temperatura ambiente dos complexos de polihidreto de rênio(V) são, tipicamente, enganosamente simples devido aos vários processos dinâmicos que ocorrem em soluções de temperatura ambiente.
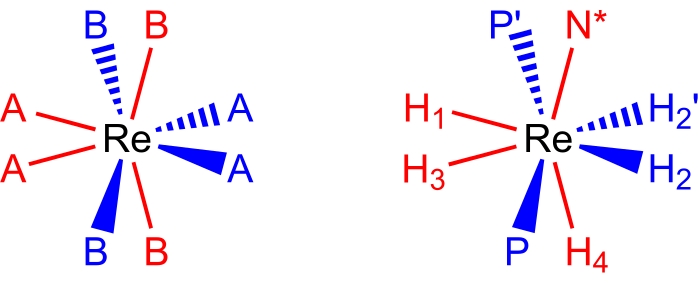
Figura 2: Um conjunto de coordenação dodecaédrica (esquerda) e o complexo ReH5(PPh3)2(sec-butil amina) da mesma perspectiva (direita). Os sítios de cor vermelha representam locais de coordenação que formam um trapézio vertical, e os locais de cor azul representam locais de coordenação que formam um trapézio horizontal. Por favor, clique aqui para ver uma versão maior desta figura.
Complexos da forma ReH5(PPh3)2(amina) são a classe mais estudada de complexos de polihidreto de rênio em relação a processos dinâmicos 9,10,12,13,16,30,31. Três processos dinâmicos (Figura 3) foram identificados para os complexos ReH5(PPh3)2(amina): 1) uma troca de prótons entre o ligante de hidreto do único sítio B e um próton de uma molécula de água (adventícia ou intencional)9,13, 2) uma troca de catraca de um par de ligantes de hidreto do sítio A com um ligante de hidreto do sítio B adjacente 9, 11,13,30,31 e 3) uma inversão estérica (ou pseudo-rotação) que se manifesta como uma troca par a par dos ligantes de hidreto do sítio A e um movimento par a par dos átomos do sítio B para o lado oposto do centro do rênio (conforme descrito na Figura 4)4,5,6,8,26,27 . O movimento de átomos do sítio B para o lado oposto do rênio é observável por espectroscopia dinâmica de RMN como: 1) um processo que torna os inequivalentes 3 e 5 prótons de N = equivalente piridina equivalente à temperatura ambiente10,30,31, 2) um processo que faz com que os isômeros E e Z de N = ligantes de amina aromática substituídos assimetricamente sofram troca rápida à temperatura ambiente9, 10,13,30,31, ou 3) processo que provoca uma rápida troca das perspectivas estéricas de um par diastereotópico de átomos de fósforo em relação a um centro quiral localizado no ligante amina9,30,31. O complexo quiral ReH5(PPh3)2(sec-butil amina) não relatado anteriormente oferece uma oportunidade para descrever geralmente os métodos que podem ser usados para identificar e caracterizar os rearranjos dinâmicos de complexos de polihidreto de rênio.
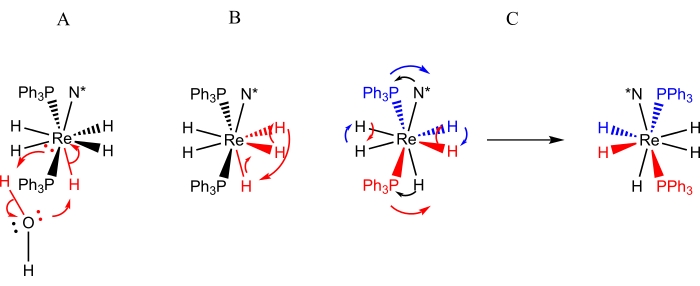
Figura 3: Representações dos processos dinâmicos que são observados por espectroscopia de RMN para soluções de ReH5(PPh3)2(sec-butil amina). A representação A retrata a troca de um único próton de água adventícia pelo ligante de hidreto exclusivo do sítio B. A representação B representa a troca de catracas de três ligantes de hidreto adjacentes, dois dos quais residem no local A, enquanto o terceiro é o ligante de hidreto do sítio B único. A representação C descreve tanto a troca par a par de ligantes de hidreto do sítio A quanto a inversão estérica dos átomos de fósforo em relação ao ligante de amina quiral (N*). Deve-se notar que a troca par de ligantes de hidreto do sítio A não requer um deslocamento dos ligantes de hidreto do sítio A para o lado oposto do centro do rênio. Por favor, clique aqui para ver uma versão maior desta figura.
Para sistemas químicos como os complexos de polihidreto de rênio, que exibem um conjunto complexo de processos dinâmicos, o ajuste da forma de linha dos espectros dinâmicos de RMN é a técnica de RMN mais utilizada para caracterizar os processos 9,11,13,16,21,29. EXSY 9,32 bidimensional ou 2D-ROESY11 são técnicas alternativas de RMN dinâmica que também podem ser usadas para caracterizar quantitativamente os processos dinâmicos. Espectros bidimensionais de EXSY são tipicamente medidos no domínio da temperatura de troca lenta; espectros ROESY bidimensionais são tipicamente medidos no domínio da temperatura de troca rápida. Ambas as técnicas bidimensionais podem exigir um tempo considerável no espectrômetro para a aquisição de dados, na medida em que cada uma das técnicas está adquirindo um conjunto de dados muito maior, a uma determinada temperatura, do que os conjuntos de dados unidimensionais necessários para a análise de ajuste da forma da linha. Processos dinâmicos simples que são bem compreendidos, como a troca dinâmica dos dois grupos metil da dimetilformamida, podem ser prontamente caracterizados por qualquer uma das três técnicas de RMN. Sistemas mais complexos, como o ReH5(PPh3)2(sec-butil amina), no qual ligantes de hidreto individuais participam de múltiplos processos dinâmicos, ou sistemas que não são necessariamente bem compreendidos, como um novo complexo de polihidreto de metal de transição que pode ou não trocar prótons entre um ligante de hidreto e água adventícia, são mais facilmente caracterizados quantitativamente pelo método de RMN de ajuste da forma da linha do que pelos métodos de RMN bidimensional. Ao contrário dos métodos de RMN bidimensional, o método de ajuste da forma da linha fornece uma visualização facilmente interpretável da correspondência entre um modelo testado e os dados experimentais, bem como evidências visuais de uma troca que move um ligante de hidreto além da esfera de coordenação interna do rênio. Com base em alturas de pico e formas de pico em espectros de troca lenta, mesmo um sistema dinâmico complexo como o ReH5 (PPh3) 2 (sec-butil amina) pode levar a um conjunto inicial facilmente testado de modelos de troca. Além disso, quando vários modelos teóricos foram relatados para uma transformação molecular, o ajuste da forma da linha dos espectros dinâmicos de RMN pode permitir uma comparação visual de cada modelo versus os espectros observados.
Além das três técnicas de RMN mencionadas acima, experimentos de RMN de substituição isotópica envolvendo D2O ou HD têm sido utilizados para demonstrar qualitativamente a troca intermolecular de átomos para sistemas complexos de polihidreto de rênio, mas não têm sido utilizados para caracterizações quantitativas 9,33,34,35. Os cálculos teóricos apresentam um método adicional para caracterizar os processos dinâmicos de sistemas dinâmicos complexos30,31,36. Os cálculos teóricos têm a vantagem sobre o ajuste da forma da linha, pois podem ser usados para diferenciar entre possibilidades que não podem ser distinguidas pela análise do ajuste da forma da linha. Por exemplo, cálculos teóricos têm sido usados para descrever uma troca que envolve três ligantes de hidreto adjacentes em certos complexos de rênio (V) como uma troca de catraca de todos os três ligantes de hidreto, em vez de um par alternado de trocas pareadas com cada troca pareada, incluindo um ligante de hidreto único e um dos dois ligantes de hidreto quimicamente equivalentes30, 31. Os resultados dos cálculos teóricos são tipicamente comparados com caracterizações quantitativas observadas experimentalmente a partir de uma das três técnicas de RMN mencionadas acima como uma verificação da validade dos resultados calculados.
O ajuste da forma da linha dos espectros dinâmicos de RMN aproveita a mudança na aparência dos espectros de RMN que ocorre quando os núcleos ativos de RMN se movem entre diferentes ambientes químicos durante uma medição de RMN. Espectros de RMN de troca lenta (espectros com ressonâncias lorentzianas independentes para cada conjunto de núcleos de troca) ocorrem em temperaturas em que a diferença de frequência entre as ressonâncias para núcleos que trocam é grande em comparação com a taxa de troca dos núcleos37. Espectros de RMN de troca rápida (espectros com uma única ressonância lorentziana para troca de núcleos) ocorrem em temperaturas em que a taxa de troca dos núcleos é muito maior do que a diferença de frequência entre as ressonâncias de troca lenta37. Taxas de câmbio intermediárias ocorrem para temperaturas entre o domínio temperatura de troca lenta e o domínio de temperatura de troca rápida37. Se os parâmetros fundamentais da frequência de Larmor, deslocamento químico dos núcleos de troca, constantes de acoplamento (se houver) para os núcleos de troca e populações relativas de cada tipo de núcleo são conhecidos, constantes de taxa para trocas putativas entre núcleos podem ser determinadas comparando espectros simulados com espectros observados a várias temperaturas intermediárias. Bons ajustes para simulações em várias temperaturas resultam em dados constantes de temperatura e taxa que podem ser usados com a equação de Eyring para estimar parâmetros de ativação para a(s) troca(s) putativa(s). Os resultados do método foram considerados precisos e reprodutíveis.
Protocolo
1. Preparação da amostra
- Preparação de ReH7(PPh3)235
- Combinar 0,15 g de borohidreto de sódio e 0,41 g de ReOCl 3(PPh3)2 num balão de fundo redondo de 100 ml de duas ou três garras equipado com um septo de borracha e uma porta de gás, ou num balão de Kjeldahl de 100 ml (com uma porta de gás de braço lateral) equipado com um septo de borracha (figura suplementar 1).
- Adicione uma barra de rotação ao vaso de reação.
- Em um exaustor, use um pedaço de tubo de pressão de borracha para conectar a porta de gás do recipiente de reação com uma das torneiras de um coletor de vidro duplo para vácuo e gás nitrogênio. Conecte o coletor de vácuo de vidro a uma bomba de vácuo com tubulação de pressão de borracha e conecte o coletor de nitrogênio de vidro a um cilindro de gás nitrogênio regulado.
- Conecte o gás de saída do coletor de gás nitrogênio a uma torneira que pode ser usada para direcionar o gás ventilado através de uma coluna de 2 cm de óleo mineral ou uma coluna de 2 cm de mercúrio.
- Abra a torneira no cilindro de nitrogênio e ajuste a pressão sobre o gás que flui para 34 libras por polegada quadrada. Ventile o fluxo de gás nitrogênio através do borbulhador de mercúrio.
- Evacue o gás dentro do recipiente de reação ajustando a torneira no coletor de vidro para conectar o recipiente ao coletor de vácuo. Encha o recipiente de reação com gás nitrogênio trocando a torneira do coletor de vidro para que ele conecte o coletor de gás com o recipiente de reação.
- Repetir as etapas 1.1.5 e 1.1.6 mais duas vezes para substituir completamente o ar no recipiente de reação por gás nitrogênio. Arrefecer o balão e o seu conteúdo num banho de gelo.
- Adicione 8 mL de água desoxigenada e 8 mL de tetraidrofurano desoxigenado aos sólidos no vaso de reação através de uma seringa. Alterne a torneira de ventilação de gás para que o gás seja ventilado através do borbulhador de óleo mineral. Mexa a suspensão levemente no banho de gelo por 15 min. Remova o recipiente de reação do banho de gelo após os 15 minutos iniciais de agitação.
- Deixe a mistura continuar mexendo por mais 45 min. Observe a cor da mistura de reação como um indicador de quando a reação foi concluída. Uma cor de mistura de reação bronzeada a laranja (Figura 1 Suplementar) indica que a reação atingiu seu ponto final.
- Ao obter uma cor laranja a bronzeada para a mistura de reação, filtre a mistura através de um funil de vidro sinterizado médio de 30 mL. Lave o sólido recuperado três vezes cada com porções de 15 mL de água, metanol e éter etílico. Seque o sólido sob vácuo para remover qualquer solvente adsorvido.
NOTA: A reacção produz geralmente entre 0,20 g e 0,25 g de produto.
- Preparação de ReH5(PPh3)2(sec-butil amina)
- Pesar 0,070 g de ReH7(PPh3)2 e transferi-lo para um balão de fundo redondo de pescoço único de 50 ml que contenha uma barra de spin. Colocar o balão num condensador equipado com uma porta de gás. Desoxigenar o recipiente de reação usando o método da bomba e do enchimento das etapas 1.1.3-1.1.7.
- Adicionar um volume de 8 ml de tetraidrofurano desoxigenado ao recipiente de reacção através de uma seringa, rachando a junção entre o balão de fundo redondo e o condensador. Adicione um volume de 0,2 mL de sec-butil amina de maneira semelhante. Mude a torneira de ventilação de gás para que o gás seja ventilado para o borbulhador de óleo mineral.
- Aqueça a mistura de reacção ao refluxo a 65 °C com um manto de aquecimento ligado a um transformador CA variável regulado para 40 numa escala de 0 a 140 durante 40 minutos. Arrefecer a mistura de reacção a uma temperatura que permita um manuseamento conveniente do balão.
- Despeje a mistura de reação em 25 mL de metanol em um balão de Erlenmeyer de 125 mL. Mexa a mistura vigorosamente durante 5 min. Adicione 5 mL de água para induzir a formação de um precipitado amarelo floculante.
- Recolher o precipitado amarelo por filtração a vácuo num funil de vidro sinterizado. Lave o sólido com 15 mL de metanol. Seque o sólido sob vácuo. Após este processo, o rendimento típico do produto é de 0,035 g.
2. Aquisição e análise de espectros de RMN
- Medição de espectros dinâmicos de RMN
- Preparar uma amostra de RMN com aproximadamente 8 mg do complexo ReH5(PPh3)2(sec-butil amina) em cerca de 0,8 ml de d8-tolueno. Insira a amostra no instrumento.
- Clique na guia Arquivo e selecione Novo nas opções que aparecem para abrir uma caixa de diálogo usada para criar um experimento de RMN.
- Crie um experimento de 1H concluindo as etapas a seguir.
- Atribua um nome de pasta para o novo experimento preenchendo a caixa de entrada Nome com um nome de arquivo exclusivo. Atribua um número de experimento como 1 para o experimento de 1H na caixa EXPNO .
- Atribua um número de processo de 1 para o experimento na caixa PROCN . Atribua a pasta a um diretório usando a lista suspensa para DIR. Identifique o solvente que o instrumento bloqueará a partir da lista suspensa Opções de solvente .
- Escolha o diretório que contém os parâmetros para o experimento 1H na lista suspensa de diretórios em Dirs do experimento. Selecione o experimento Proton nas opções na lista suspensa Experimento e (opcional) adicione um título para os dados na caixa Preenchimento do título.
- Insira um comando Eda na linha de comando e ajuste os parâmetros conforme necessário para atender às descrições do experimento fornecidas no segundo parágrafo da seção Discussão abaixo.
- Clique na guia Janela, selecione Nova Janela na lista e repita as Etapas 2.1.3.1-2.1.3.8 para preparar um experimento de 1 H-{31P} usando um valor EXPNO de 2 para diferenciar o experimento do experimento de 1H construído anteriormente.
- Clique na guia Janela, selecione Nova Janela na lista e repita as Etapas 2.1.3.1-2.1.3.8 para preparar um experimento 31 P-{1 H} usando um valor EXPNO de 3 para diferenciar o experimento dos experimentos 1 H e 1 H-{31P} construídos anteriormente (consulte a Tabela Suplementar 1para obter informações detalhadas sobre parâmetros).
- Digite um comando Bloquear na linha de comando e selecione a opção d8-tolueno na lista. Clique em OK para aceitar a opção de solvente. Digite um comando Atma na linha de comando, se necessário, por causa de uma sonda de banda X de núcleo variável, para minimizar a energia refletida nas frequências de Larmor para 1H e 31P no instrumento.
- Digite um comando Ro na linha de comando, digite um valor de 20 na caixa e clique no botão Iniciar rotação . Insira um comando Shim na linha de comando. Escolha uma rotina de autoshim apropriada, como Topshim , na lista de rotinas de shim e clique no botão Iniciar .
- Insira um comando Rga na linha de comando. Escolha a seleção Ajuste automático do receptor e clique em OK. Por sua vez, meça os três espectros da amostra à temperatura ambiente usando 64 varreduras para cada espectro com um comando Go na linha de comando.
- Transforme os dados de um experimento em um espectro com um comando Efp inserido na linha de comando.
- Ajuste o faseamento do espectro usando os seguintes comandos.
- Clique na guia Fase, seguido de um clique na guia Ajustar Fase. Passe o cursor sobre o botão 0 na barra de ferramentas de faseamento e mantenha pressionado o botão esquerdo do mouse para baixo para que o botão 0 fique verde.
- Com o botão esquerdo do mouse pressionado, role o mouse para frente ou para trás até que a linha de base fique plana em todo o espectro e todas as ressonâncias sejam exibidas como absorvâncias (os picos se elevam acima da linha de base).
- Se a linha de base não puder ser nivelada apenas com o botão 0, ajuste o botão 1 conforme descrito nas etapas 2.1.10.1 e 2.1.10.2, bem como o botão 0, até que a linha de base fique plana para toda a janela espectral.
- Salve o ajuste de fase com os dados clicando no botão Salvar e Retornar na barra de ferramentas de faseamento.
- Ajuste o número de varreduras para cada medição, conforme necessário, com base na relação sinal-ruído no espectro, tendo em mente que o sinal-ruído normalmente diminui em temperaturas mais baixas devido à descoalescência dos sinais em ressonâncias individuais (Figura 4).
- Prepare o espectrômetro para o controle de temperatura de acordo com as instruções do fornecedor. Insira uma vazão de 200 L/h para o gás de resfriamento e uma temperatura alvo de 290 K para a sonda. Permita que o espectrômetro se estabilize na temperatura alvo por 2 min. Aumente a taxa de fluxo de gás de resfriamento, se necessário, para 210 ou 220 L / h para estabilizar a temperatura.
- Coloque a amostra a 290 K como na Etapa 2.1.7. Altere o nome do arquivo para cada um dos espectros medidos anteriormente adicionando a temperatura ao final do nome do arquivo (Etapas 2.1.2 e 2.1.3.1) e adquira um conjunto de três espectros a 290 K.
- Aumente a taxa de fluxo de gás de resfriamento em ≥ 30 L / h, conforme necessário para estabilizar na próxima temperatura, e diminua a temperatura alvo em 10 K. Deixe o espectrômetro se estabilizar na próxima temperatura por 2 min e, em seguida, retire a amostra como na Etapa 2.1.7. Meça o conjunto de três espectros.
- Repita as etapas 2.1.13 e 2.1.14 conforme necessário para adquirir espectros até a temperatura mais baixa desejada.
NOTA: Uma temperatura de 200 K é geralmente suficiente para um conjunto completo de dados que é adequado para determinar os parâmetros de ativação para os processos dinâmicos da amostra. - Aqueça a amostra de volta à temperatura ambiente em incrementos de 10 K. Estabilize a temperatura por 2 minutos a cada temperatura antes de aquecer a amostra novamente para evitar danos ao revestimento de vidro da sonda.
- Análise da forma da linha dos espectros medidos
- Dentro do programa NMR, clique na barra de comandos no canto superior esquerdo da janela e selecione Abrir no menu suspenso. Selecione Abrir dados NMR armazenados no formato padrão. Clique em OK para abrir a janela do explorador de arquivos do programa.
- Navegue até a pasta para os dados a serem analisados pelo ajuste da forma de linha. Selecione o número do arquivo que corresponde ao espectro a ser analisado e clique no botão Exibir . O espectro (se processado anteriormente) ou a curva de decaimento de indução livre (FID) é exibido no software de RMN.
- Processe o FID, se necessário, inserindo um comando Efp (multiplicação exponencial, transformação de Fourier e correção de fase) na linha de comando. Ajustar a fase do espectro (Etapa 2.1.10).
- Ajustar a linha de base do espectro; se não for plano em todo o espectro, nivele-se com a linha de intensidade 0, da seguinte forma.
- Clique na guia Processo e, em seguida, clique na guia Linha de Base . Passe o cursor sobre o botão A. Pressione o botão esquerdo do mouse e role o mouse para frente ou para trás para nivelar a linha de ajuste vermelha com a extremidade esquerda (downfield) do espectro.
- Se a linha de base ainda não estiver nivelada com a linha de ajuste vermelha, repita o processo com os botões de letra restantes até que a linha de ajuste vermelha se ajuste à linha de base do espectro. Use o botão Salvar e retornar para salvar o ajuste quando a linha de base ajustada em vermelho corresponder à linha de base real.
- Selecione a guia Analisar dentro do software NMR. Nas opções de análise, selecione a opção Formas de Linha seguida pela opção Ajustar Modelos Dinâmicos de RMN .
- O espectro agora é exibido na janela do módulo de ajuste da forma de linha. Use as barras de ferramentas acima do espectro para ajustar como o espectro é exibido. A janela à esquerda do espectro lida com o ajuste da forma de linha do espectro.
- Ajuste a exibição do espectro com a Ferramenta de zoom suave para que a parte do espectro a ser ajustada seja exibida na janela do espectro. Use o botão da barra de ferramentas Shift Spectrum Left and Right para centralizar uma parte do espectro na janela de exibição.
- Acesse a janela de deslocamento químico para ajuste da forma da linha selecionando a guia Espectro na janela de ajuste da forma da linha.
- Clique no botão Editar intervalo . Insira os deslocamentos químicos superiores e inferiores para o ajuste da forma da linha e clique no botão OK para aceitar esses limites.
- Inicie um modelo para ajuste de forma de linha clicando na guia Sistema de rotação na janela de ajuste de forma de linha. Clique no botão Adicionar para permitir a construção de um sistema de rotação de modelo.
- Desmarque LB (para ampliação de linha) e insira o valor para ampliação de linha manualmente com o mouse e o botão LB na barra de ferramentas de ajuste da forma de linha.
- Adicione o primeiro núcleo ao modelo clicando na guia Núcleo, seguido clicando no botão Adicionar . Um conjunto de valores padrão aparecerá para o Núcleo 1. Ajuste o deslocamento químico para o Núcleo 1 inserindo um valor para deslocamento químico na caixa Nu(iso) ou com a ferramenta de deslocamento químico na barra de ferramentas de ajuste da forma da linha.
NOTA: Se a caixa de seleção for deixada no formulário marcado, o deslocamento químico deste núcleo será variado para alcançar o melhor ajuste. As variáveis não verificadas não serão variadas no processo de ajuste de linha. - Use a caixa Pseudospin para o Núcleo 1 para inserir o número de núcleos equivalentes para o Núcleo 1 com cada núcleo de spin 1/2 equivalente a 0,5 na contagem. Digite a soma dos spins na caixa Pseudospin para contabilizar todos os núcleos equivalentes.
- Use a caixa In Molecule para acomodar modelos que exigem mais de uma única molécula para participar de um processo dinâmico. Atribua ressonâncias que surgem de moléculas diferentes para moléculas separadas usando designações como 1, 2, etc. para moléculas diferentes. Para ressonâncias que surgem de uma única molécula, atribua 1 para todos os valores In Molecule .
- Adicione o segundo e todos os núcleos subsequentes ao modelo clicando na guia Núcleo, seguido de clicar no botão Adicionar . Inclua o acoplamento spin-spin entre núcleos inserindo o acoplamento na caixa JN apropriada (onde N é o núcleo com o qual o núcleo adicionado está acoplado, N = 1, 2, ...) ou ajustando o botão de acoplamento escalar na barra de ferramentas de ajuste da forma de linha.
- Comece o processo de descrição das trocas de átomos clicando na guia Reação . Clique na caixa de seleção se a constante de taxa para a troca deve ser variada no ajuste da forma da linha. Insira o número de núcleos a serem trocados (número em relação às suas guias de identificação, como Núcleo 1 e Núcleo 2) na caixa Trocas para a primeira troca no modelo.
- Descreva as trocas a serem testadas nas caixas abaixo da caixa Trocas . Defina as trocas entre as guias Nucleus nas caixas abaixo. Uma troca de dois núcleos seria inserida como núcleo 1 para o núcleo 2 e núcleo 2 para o núcleo 1. Certifique-se de que as trocas são cíclicas em que, se um núcleo é movido do Núcleo 1, outro núcleo tem que ser movido para o Núcleo 1.
- Use o botão de velocidade do Exchange na barra de ferramentas de ajuste da forma de linha para alterar o valor inicial de k para ajustar iterativamente o valor de k, mesmo que a caixa de seleção esteja marcada para a constante de taxa.
- Adicione mais trocas ao modelo clicando na guia Reação seguida do botão Adicionar . Adicione trocas ao modelo conforme necessário. Use as ferramentas na barra de ferramentas de ajuste da forma da linha para ajustar as variáveis iniciais, incluindo a intensidade do espectro para uma boa correspondência para que o espectro seja ajustado.
- Comece o ajuste iterativo da forma da linha clicando no botão Iniciar o ajuste do espectro na barra de ferramentas de ajuste da forma da linha. Continue o ajuste iterativo até que nenhuma alteração seja encontrada na melhor sobreposição entre espectro e modelo ou até que 1000 iterações sejam alcançadas. Se o encaixe parar em 1000 iterações, continue outras iterações com o botão Iniciar o ajuste do espectro . O espectro do modelo é exibido com o espectro real para comparação.
- Registre os valores de melhor ajuste das guias apropriadas. Salve o espectro de melhor ajuste clicando na guia Espectro na janela de ajuste da forma da linha, seguida clicando no botão Salvar .
NOTA: O espectro de melhor ajuste será salvo na mesma pasta que foi usada para coletar os dados. O espectro de melhor ajuste será distinguido dos dados originais por ser salvo com um número de processamento diferente que é inserido quando o salvamento ocorre. - Salve o modelo usado para o ajuste da forma de linha clicando na guia Principal seguido do botão Salvar. Insira um nome para o modelo.
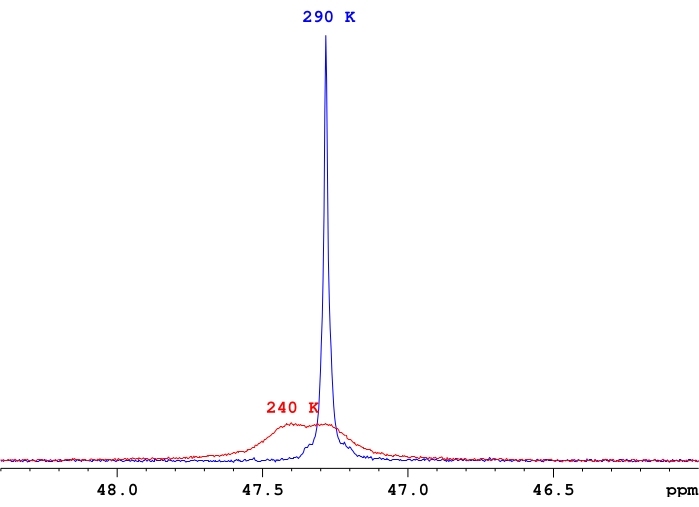
Figura 4: Comparação de 31intensidades de sinal P-{1H} para uma única amostra de ReH5(PPh3)2(sec-butil amina) em d8-tolueno. Uma demonstração representativa da diferença nas intensidades de sinal entre uma ressonância de fósforo único de troca rápida e um par de ressonâncias de fósforo perto da temperatura de coalescência para essas ressonâncias. Por favor, clique aqui para ver uma versão maior desta figura.
3. Determinação dos parâmetros de ativação a partir de uma parcela de Eyring 1
- Insira dados do ajuste da forma de linha para um processo dinâmico modelado em uma planilha com a variável independente inserida como 1/T e a variável dependente inserida como ln(k/T).
- Insira um gráfico de dispersão dos dados na planilha. Adicione uma linha de tendência através dos dados. Use a inclinação e o intercepto da linha de tendência para resolver ΔH‡ e ΔS‡. A inclinação da linha de tendência é -ΔH‡/R, enquanto o intercepto da linha de tendência é ΔS‡/R + 23,76.
- Resolva para ΔG‡ a uma determinada temperatura usando a relação
ΔG‡(T) = ΔH‡ - TΔS‡.
NOTA: Para uma troca simples de dois núcleos com ressonâncias que se aglutinam, uma verificação dos valores de ΔH‡ e ΔS‡ pode ser realizada comparando ΔG‡ calculado na temperatura de coalescência com o valor de ΔG‡ que surge da diferença de frequência de troca lenta entre ressonâncias e a temperatura de coalescência.
Resultados
As caracterizações de ambos os produtos de polihidreto de rênio descritas neste manuscrito são melhor realizadas por espectroscopia de RMN de 1H-{31P} e 31P-{1H}. Em uma solução de 6-benzeno d à temperatura ambiente, a ressonância do ligante hidreto de ReH7(PPh3)2 aparece como um tripleto binomial a δ = -4,2 ppm com 2 JPH = 18 Hz por espectroscopia de RMN de 1H (Figura Suplementar 2).
Discussão
Existem quatro itens na preparação do ReH7(PPh3)2 que podem impactar a quantidade e a pureza do material produzido. Primeiro, o uso de um banho de gelo durante os primeiros 15 minutos da reação é importante para remover o calor da reação que ocorre entre o borohidreto de sódio e a água. Temperaturas iniciais mais altas levam a uma diminuição do rendimento do produto ReH7(PPh 3)2 devido à formação do produto de decomposição térmica Re 2 H
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Os autores agradecem ao Departamento de Química e Física e ao Programa de Bolsas de Criatividade e Pesquisa (Naik, Moehring) da Universidade de Monmouth pelo apoio financeiro a este trabalho.
Materiais
| Name | Company | Catalog Number | Comments |
| Bruker Avance II 400 MHz NMR spectrometer | Bruker Biospin | The instrument includes a two channel probe (1H and X) with the X channel tunable from 162 MHz to 10 Mhz. The instrument is also VT capable with a dewar and heat exchanger for VT work. | |
| d8-toluene | MilliporeSigma | 434388 | |
| Powerstat variable transformer | Powerstat | ||
| sec-butyl amine | MilliporeSigma | B89000 | |
| Sodium borohydride | MilliporeSigma | 452882 | |
| Tetrahydrofuran | MilliporeSigma | 186562 | |
| Thermowell C3AM 100 mL | Thermowell | ||
| Topspin 3.0 or 4.1.4 with dNMR | Bruker Biospin | Data was acquired with Topspin version 3.0 and data handling was performed on a second computer that was running Topspin version 4.1.4.. | |
| Trichlorooxobis(triphenylphosphine) rhenium(V) | MilliporeSigma | 370193 | |
| Vacuubrand PC3000 vacuum pump with a CVC 3000 controller | Vacuubrand |
Referências
- Zimmer, K. D., Shoemaker, R., Ruminski, R. R. Synthesis and characterization of a fluxional Re(I) carbonyl complex fac-[Re(CO)3(dpop')Cl] with the nominally tri-dentate ligand dipyrido(2,3-α:3',2'-j)phenazine (dpop). Inorganica Chimica Acta. 359 (5), 1478-1484 (2006).
- McGlinchey, M. J. Symmetry breaking in NMR spectroscopy: the elucidation of hidden molecular rearrangement processes. Symmetry. 6 (3), 622-654 (2014).
- Casarini, D., Luazzi, L., Mazzanti, A. Recent advances in stereodynamics and conformational analysis by dynamic NMR and theoretical calculations. European Journal of Organic Chemistry. 2010 (11), 2035 (2010).
- Palmer, A. G., Williams, J., McDermott, A. Nuclear magnetic resonance studies of biopolymer dynamics. Journal of Physical Chemistry. 100 (31), 13293-13310 (1996).
- Kern, D., Kern, G., Scherer, G., Fischer, G., Drakenberg, T. Kinetic analysis of cyclophilin-catalyzed prolyl cis/trans isomerization by dynamic NMR spectroscopy. Biochemistry. 34 (41), 13594-13602 (1995).
- Menger, F. M., Lynn, J. L. Fast proton transfer at a micelle surface. Journal of the American Chemical Society. 97 (4), 948-949 (1975).
- Pines, A., Rabinovitz, M. A nuclear magnetic resonance total line-shape treatment of internal rotation in dimethylformamide. Tetrahedron Letters. 9 (31), 3529-3532 (1968).
- Mancinelli, M., Bencivenni, G., Pecorari, D., Mazzanti, A. Stereochemistry and recent applications of axially chiral organic molecules. European Journal of Organic Chemistry. 2020 (27), 4070-4086 (2020).
- Streisel, D. J., et al. Fluxionality, substitution, and hydrogen exchange at eight-coordinate rhenium(V) polyhydride centers. Inorganica Chimica Acta. 496 (1), 119028 (2019).
- Jimenez, Y., Strepka, A. M., Borgohain, M. D., Hinojosa, P. A., Moehring, G. A. Ortho-metalation, rotational isomerization, and hydride-hydride coupling at rhenium(V) polyhydride complexes stabilized by aromatic amine ligands. Inorganica Chimica Acta. 362 (9), 3259-3266 (2009).
- Lee, J. C., Yao, W., Crabtree, R. H., Ruegger, H. Fluxionality in [ReH5(PPh3)2(pyridine)]. Inorganic Chemistry. 35 (3), 695-699 (1996).
- Patel, B. P., Kavallieratos, K., Crabtree, R. H. Effects of dihydrogen bonding on fluxionality in ReH5(PPh3)2L. Journal of Organometallic Chemistry. 528 (1), 205-207 (1997).
- Geetha, B., et al. Chiral amine ligands at rhenium(V) pentahydride complexes allow for characterization of an energetically accessible and reversible steric inversion of diastereotopic phosphorus atoms. Inorganica Chimica Acta. 531 (1), 120741 (2022).
- Paulo, A., Ascenso, J., Domingos, A., Galvao, A., Santos, I. Rhenium-(III) and -(V) hydride complexes with modified poly(pyrazolyl)borates. Journal of the Chemical Society, Dalton Transactions. 1999 (8), 1293-1300 (1999).
- Bianchini, C., et al. Synthesis and characterization of rhenium polyhydrides stabilized by the tripodal ligand MeC(CH2PPh2)3. Journal of Organometallic Chemistry. 451 (1), 97-106 (1993).
- Scorzelli, A. G., Macalush, B. E., Naik, D. V., Moehring, G. A. Comparative study of fluxional processes at two different classes of eight-coordinate rhenium(V) polyhydride complexes. Inorganica Chimica Acta. 516 (1), 120120 (2021).
- Luo, X. -. L., Crabtree, R. H. Synthesis and spectroscopic characterization of rhenium complexes ReH5(triphos)] and [ReH6(triphos)]+ [triphos = PPh(CH2CH2PPh2)2]. Journal of the Chemical Society. 1991 (5), 587-590 (1991).
- Kim, Y., Deng, H., Gallucci, J. C., Wojcicki, A. Rhenium polyhydride complexes containing PhP(CH2CH2CH2PCy2)2 (Cyttp): protonation, insertion, and ligand substitution reactions of ReH5(Cyttp) and structural characterization of ReH5(Cyttp) and [ReH4(η2-H2)(Cyttp)]SbF6. Inorganic Chemistry. 35 (24), 7166-7173 (1996).
- Bolano, S., et al. Synthesis, characterization, protonation studies and X-ray crystal structure of ReH5(PPh3)2(PTA) (PTA = 1,3,5-triaza-7-phosphaadamantane). Journal of Organometallic Chemistry. 691 (4), 629-637 (2006).
- Ginsberg, A. P., Abrahams, S. C., Jamieson, P. B. Nonrigid stereochemistry in eight-coordinate pentahydridorhenium complexes. Journal of the American Chemical Society. 95 (14), 4751-4752 (1973).
- Bolano, S., Bravo, J., Garcia-Fontan, S. Mono- and dinuclear rhenium polyhydride complexes bearing the chelating ligand 1,2-bis(dicyclohexylphosphinanyloxy)ethane. European Journal of Inorganic Chemistry. 2004 (24), 4812-4819 (2004).
- Leeaphon, M., Rohl, K., Thomas, R. J., Fanwick, P. E., Walton, R. A. Reactions of the polyhydride complex ReH7(PPh3)2 with quinoline, 2-hydroxyquinoline, and 2-mercaptoquinoline. The preparation and characterization of hydrido complexes of rhenium(V) and chloro complexes of rhenium(III). Inorganic Chemistry. 32 (24), 5562-5568 (1993).
- Mejia, E., Togni, A. Rhenium complexes containing the chiral tridentate ferrocenyl ligand pigiphos. Organometallics. 30 (17), 4765-4770 (2011).
- Moehring, G. A., Walton, R. A. Reactions of heptahydrobis(triphenylphosphine)rhenium with bidentate aromatic heterocycles. Inorganic Chemistry. 26 (17), 2910-2912 (1987).
- Kosanovich, A. J., Reibenspies, J. H., Ozerov, A. V. Complexes of high-valent rhenium supported by the PCP pincer. Organometallics. 35 (4), 513-519 (2016).
- Emge, T. J., Koetzle, T. F., Bruno, J. W., Caulton, K. G. Pentahydridorhenium: crystal and molecular structure of ReH5(PMePh2)3. Inorganic Chemistry. 23 (24), 4012-4017 (1984).
- Costello, M. T., Fanwick, P. E., Green, M. A., Walton, R. A. Reactions of Heptahydridobis(triphenylphosphine)rhenium with 1-(diphenylphosphino)-2-(diphenylarsino)ethane (arphos) and 1,2-bis(diphenylarsino)ethane (dpae). Structural characterization of ReH5(PPh3)2(arphos-As) and ReH5(PPh3)2(dpae-As). Inorganic Chemistry. 30 (4), 861-864 (1991).
- Alvarez, D., Lundquist, E. G., Ziller, J. W., Evans, W. J., Caulton, K. G. Synthesis, structure and applications of transition-metal polyhydride anions. Journal of the American Chemical Society. 111 (22), 8392-8398 (1989).
- Albinati, A., et al. Synthesis, characterization, and interconversion of the rhenium polyhydrides ReH3(η4-NP3)] and [ReH4(η4-NP3)]+ {NP3 = tris[2-(diphenylphosphanyl)ethyl]amine}. European Journal of Inorganic Chemistry. 2002 (6), 1530-1539 (2002).
- Bosque, R., et al. Site preference energetics, fluxionality, and intramolecular M−H···H−N hydrogen bonding in a dodecahedral transition metal polyhydride. Inorganic Chemistry. 36 (24), 5505-5511 (1997).
- Tao, Y., Sou, W., Luo, G. -. G., Kraka, E. Describing polytopal rearrangement processes of octacoordinate structures. I. renewed insights into fluxionality of the rhenium polyhydride complex ReH5(PPh3)2(Pyridine). Inorganic Chemistry. 60 (4), 2492-2502 (2021).
- Beringhelli, T., D'Alfonso, G., Minoja, A. P. Rhenium-platinum mixed metal clusters. Characterization in solution and dynamic behavior of the isomers of [Re3Pt(µ-H3)(CO)14]. An example of a labile metal fragment that undergoes intermolecular exchange. Organometallics. 13 (2), 663-668 (1994).
- Grieco, G., Blacque, O. Solution and solid-state structure of the first NHC-substituted rhenium heptahydrides. European Journal of Inorganic Chemistry. 2019 (34), 3810-3819 (2019).
- Wazio, J. A., Jimenez, V., Soparawalla, S., John, S., Moehring, G. A. Hydrogen exchange of rhenium(VII) heptahydridobis(triphenylphosphine) with water, aniline, methanol, and itself. Inorganica Chimica Acta. 362 (1), 159-165 (2009).
- Chatt, J., Coffey, R. S. Hydrido-complexes of rhenium-containing tertiary phosphines. Journal of the Chemical Society, A. 1969, 1963-1972 (1969).
- Tao, Y., Wang, X., Zou, W., Luo, G. -. G., Kraka, E. Unusual intramolecular motion of ReH92- in K2ReH9 crystal: circle dance and three-arm turnstile mechanisms revealed by computational study. Inorganic Chemistry. 61 (2), 1041-1050 (2022).
- Berger, X., Braun, S. . 200 and More NMR Experiments a Practical Course. , (2004).
- He, G., Chen, J., Sung, H. H. -. Y., Williams, I. D., Jia, G. Substituent effect on reactions of ReH5(PMe2Ph)3 with propargyl alcohols. Inorganica Chimica Acta. 518 (1), 120239 (2021).
- Donnelly, L. J., Parsons, S., Morrison, C. A., Thomas, S. P., Love, J. B. Synthesis and structures of anionic rhenium polyhydride complexes of boron-hydride ligands and their application in catalysis. Chemical Science. 11 (9), 9994-9999 (2020).
- Donnelly, L. J., et al. C-H borylation catalysis of heteroaromatics by a rhenium boryl polyhydride. ACS Catalysis. 11 (12), 7394-7400 (2021).
- Jin, H., et al. CO-enabled rhenium hydride catalyst for directed C(sp2)-H bond alkylation with olefins. Organic Chemistry Frontiers. 2 (4), 378-382 (2015).
- Takaya, H., Ito, M., Murahashi, S. -. I. Rhenium-catalyzed addition of carbonyl compounds to the carbon−nitrogen triple bonds of nitriles: α-C−H activation of carbonyl compounds. Journal of the American Chemical Society. 131 (31), 10824-10825 (2009).
- Carr, S. W., Fowles, E. H., Fontaine, X. L. R., Shaw, B. L. Multihydride complexes of rhenium, osmium or iridium containing monodentate ditertiary phosphine ligands: selective hydrogen-deuterium exchanges of the rhenium multihydrides. Journal of the Chemical Society, Dalton Transactions. 1990 (2), 573-579 (1990).
- Jin, H., et al. Rhenium-catalyzed acceptorless dehydrogenative coupling via dual activation of alcohols and carbonyl compounds. ACS Catalysis. 3 (10), 2195-2198 (2013).
- Loza, M. L., de Gala, S., Crabtree, R. H. Steric crowding in a rhenium polyhydride induced by a chelating disilyl ligand: synthesis, characterization, and reactivity of ReH5(disil)(PPh3)2 (disil = 1,2-Bis(dimethylsilyl)benzene and 1,2-Bis(dimethylsilyl)ethane). Inorganic Chemistry. 33 (22), 5073-5078 (1994).
- Lin, Y., Zhu, X., Xiang, M. Transition metal polyhydrides-catalyzed addition of activated nitriles to aldehydes and ketones via Knoevenagel condensation. Journal of Organometallic Chemistry. 448 (1-2), 215-218 (1993).
- Abdukader, A., Jin, H., Cheng, Y., Zhu, C. Rhenium-catalyzed amination of alcohols by hydrogen transfer process. Tetrahedron Letters. 55 (30), 4172-4174 (2014).
- Lin, Y., Zhou, Y. Selective transfer hydrogenation catalyzed by transition metal polyhydrides. Fenzi Cuihua. 5 (2), 119-124 (1991).
- Green, M. A., Huffman, J. C., Caulton, K. G., Rybak, W. K., Ziolkowski, J. J. Ligand scavenging and catalytic utilization of the phototransient ReH5(PMe2Ph)2. Journal of Organometallic Chemistry. 218 (2), 39-43 (1981).
- Komiya, S., Chigira, T., Suzuki, T., Hirano, M. Polymerization of alkyl methacrylate catalyzed by hydridorhenium complexes. Chemistry Letters. 4 (4), 347-348 (1999).
- Michos, D., Luo, X. L., Faller, J. W., Crabtree, R. H. Tungsten(VI) hexahydride complexes supported by chelating triphosphine ligands: protonation to give η2-dihydrogen complexes and catalytic dehydrogenation of cyclooctane to cyclooctene. Inorganic Chemistry. 32 (8), 1370-1375 (1993).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados