É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Projetando um Biorreator para Melhorar a Aquisição de Dados e Modelar o Rendimento de Tecidos Cardíacos Projetados
Neste Artigo
Erratum Notice
Resumo
Tecidos cardíacos tridimensionais bioprojetados usando cardiomiócitos derivados de células-tronco têm emergido como modelos promissores para estudar o miocárdio humano saudável e doente in vitro , enquanto recapitulam aspectos-chave do nicho cardíaco nativo. Este manuscrito descreve um protocolo para fabricação e análise de tecidos cardíacos projetados de alto conteúdo gerados a partir de cardiomiócitos derivados de células-tronco pluripotentes induzidos por humanos.
Resumo
A insuficiência cardíaca continua sendo a principal causa de morte em todo o mundo, criando uma necessidade premente de melhores modelos pré-clínicos do coração humano. A engenharia de tecidos é crucial para a pesquisa em ciência básica cardíaca; a cultura de células humanas in vitro elimina as diferenças interespécies de modelos animais, enquanto um ambiente 3D mais semelhante a tecidos (por exemplo, com matriz extracelular e acoplamento heterocelular) simula condições in vivo em maior extensão do que a cultura bidimensional tradicional em placas de Petri plásticas. No entanto, cada sistema modelo requer equipamentos especializados, por exemplo, biorreatores personalizados e dispositivos de avaliação funcional. Além disso, esses protocolos são muitas vezes complicados, trabalhosos e atormentados pela falha dos tecidos pequenos e delicados.
Este artigo descreve um processo para gerar um sistema modelo robusto de tecido cardíaco humano projetado (hECT) usando cardiomiócitos derivados de células-tronco pluripotentes induzidos para a medição longitudinal da função tecidual. Seis hECTs com geometria de tira linear são cultivadas em paralelo, com cada hECT suspensa de um par de postes de polidimetilsiloxano (PDMS) com sensor de força acoplados a racks PDMS. Cada postagem é coberta com um rastreador de postagem estável PDMS preto (SPoT), um novo recurso que melhora a facilidade de uso, a taxa de transferência, a retenção de tecido e a qualidade dos dados. A forma permite o rastreamento óptico confiável de deflexões de poste, produzindo traçados de força de contração aprimorados com tensão ativa e passiva absoluta. A geometria da tampa elimina a falha tecidual devido ao deslizamento de hECTs dos postes e, como eles envolvem uma segunda etapa após a fabricação do rack PDMS, os SPoTs podem ser adicionados aos projetos pós-baseados em PDMS existentes sem grandes alterações no processo de fabricação do biorreator.
O sistema é usado para demonstrar a importância de medir a função hECT em temperaturas fisiológicas e mostra função tecidual estável durante a aquisição de dados. Em resumo, descrevemos um sistema modelo de última geração que reproduz condições fisiológicas fundamentais para promover a biofidelidade, eficiência e rigor de tecidos cardíacos projetados para aplicações in vitro .
Introdução
Os modelos de tecido cardíaco projetados vêm em uma gama diversificada de geometrias e configurações para recapitular vários aspectos do nicho cardíaco nativo que são difíceis de alcançar com a cultura de células bidimensional tradicional. Uma das configurações mais comuns é a tira de tecido linear, com âncoras flexíveis em cada extremidade para induzir a automontagem do tecido e fornecer ao tecido uma pré-carga definida e uma leitura das forças de contração resultantes 1,2,3,4,5,6,7,8,9,10,11, 12,13,14,15,16,17,18,19,20,21
,22,23,24,25,26,27. A força gerada pode ser determinada de forma robusta através do rastreamento óptico do encurtamento tecidual e utilizando a teoria do feixe elástico para calcular a força a partir das deflexões medidas e a constante de mola das âncoras 1,2,3,4,5,6,7,8,9,10,11, 12,13,14,15,16,17,18,19,20,
21,22,25,26,28.
No entanto, a engenharia de tecidos cardíacos ainda é um campo em evolução, e alguns desafios permanecem. Equipamentos especializados, como biorreatores sob medida e dispositivos de avaliação funcional, são necessários para cada sistema modelo 10,29,30,31. O tamanho e a complexidade do microambiente dessas construções são frequentemente limitados pelo baixo rendimento devido a protocolos trabalhosos, alto número de células e fragilidade tecidual. Para resolver isso, alguns grupos se voltaram para a fabricação de microtecidos contendo apenas centenas ou milhares de células para facilitar ensaios de alto rendimento que são úteis para a descoberta de drogas. No entanto, essa escala reduzida dificulta a avaliação precisa da função12, elimina aspectos importantes do nicho cardíaco nativo (como gradientes de difusão de nutrientes/oxigênio e arquitetura complexa36) e limita a quantidade de material disponível para análises moleculares e estruturais subsequentes (muitas vezes exigindo o agrupamento dos tecidos). A Tabela 1 resume algumas das configurações dos modelos lineares de tiras de tecido na literatura 1,2,3,4,5,6,7,8,9,10,11,12,13,14,15, 16,17,18,19,20,
21,22,23,24,25,26,37,38,39,40.
| Grupo | Células por tecido | Tecidos por placa | Formato da chapa | Recurso de ancoragem | Método de aquisição de dados funcionais | Banho de mídia compartilhado? | Medida funcional- E in loco? | ||||
| Yoshida (ECT)38 | 4 milhões | 6 | placa de 6 poços modificada* | transdutor de força | medição direta de força | Não | Não | ||||
| Chan (hESC-CM-ECTs)26 | 310 mil | 6 | prato personalizado de 6 poços | Postagens do PDMS | medição direta de força | Sim | Não | ||||
| Feinberg (dyn-EHT)16 | 1,5 milhão | 6 | prato personalizado de 6 poços | Fio PDMS | forma do tecido | Não | Sim | ||||
| RADÍSICO (BioWire)39, 40 | 110 mil | 8 | fio de polímero | forma do fio | Sim | Sim | |||||
| Costa (hECT simples)1, 2 | 1-2 milhões | 4** | Placa de Petri de 10 cm** | Postagens do PDMS | deflexão óptica (rastreamento de borda/objeto) | Sim | Sim | ||||
| Costa (multi-hECT)3–9 | 500 k-1 milhão | 6 | 6 cm Placa de Petri | Postagens do PDMS | deflexão óptica (rastreamento de borda/objeto) | Sim | Sim | ||||
| Costa (multi-hECT com SPoT) | 1 milhão | 6 | 6 cm Placa de Petri | Postes PDMS com tampas pretas | deflexão óptica (rastreamento de objetos) | Sim | Sim | ||||
| Passier (EHT)17 | 245 mil | 36 | Placa de 12 poços | Postes PDMS com tampas pretas | deflexão óptica (rastreamento de objetos) | Sim | Sim | ||||
| Vunjak-Novakovic13, 18 | 1 milhão | 12 | 6 cm Placa de Petri | Postes PDMS com tampas | deflexão óptica (detecção de borda) | Sim | Sim | ||||
| Vunjak-Novakovic (MilliPilar)14 | 550 mil | 6 | prato personalizado de 6 poços | Postes PDMS com tampas | deflexão óptica (rastreamento de objetos); imagem do cálcio | Não | Sim | ||||
| Eschenhagen (EHT)10, 19–21 | 1 milhão | 12 | Placa de 12 poços | Postes PDMS com tampas | deflexão óptica (detecção de borda de pós deflexão); imagem do cálcio | Não | Sim | ||||
| Zandstra (CaMiRi)22 | 25-150 mil | 96 | Placa de 96 poços | Postes PDMS com ganchos | deflexão óptica (detecção de borda) | Não | Sim | ||||
| Murry23, 24 | 900 mil | 24 | Placa de 24 poços | Postes PDMS com tampas, ímã integrado | sensor magnético | Não | Sim | ||||
| Reich (μTUG)11, 12, 25 | indefinido | 156 | Prato de 156 poços | Postes PDMS com tampas, ímã integrado | rastreamento óptico (cordão fluorescente) | Sim | Sim | ||||
Tabela 1: Características de alguns modelos lineares de tecido cardíaco na literatura. Os modelos de tecido cardíaco de engenharia linear variam em tamanho, rendimento, projetos de recursos de ancoragem e a facilitação de banhos médios compartilhados, bem como os requisitos para um sistema de banho muscular separado para caracterização funcional. * Os pesquisadores usaram um sistema de tecido projetado comercialmente disponível com base nas dimensões de uma placa padrão de 6 poços. ** Um sistema modular no qual biorreatores de tecido único são ancorados a qualquer placa de cultura plástica no número e local desejados.
Este trabalho descreve o protocolo mais recente para a fabricação de nosso modelo estabelecido de tecido cardíaco humano linear projetado (hECT)1,2,3,4,5,6,7,8,9,15,27 e métodos de avaliação da função contrátil da hECT. Cada biorreator multitecidual acomoda até seis hECTs em um banho médio compartilhado e é composto por duas peças de "rack" feitas do elastômero de silicone polidimetilsiloxano (PDMS) montado em uma estrutura rígida de polissulfona. Cada rack PDMS contém seis pinos flexíveis integrados de detecção de força que têm 0,5 mm de diâmetro e 3,25 mm de comprimento e, juntos, dois racks fornecem seis pares de postes, cada um dos quais contém um hECT. A inversão do biorreator ajuda a superar qualquer obstáculo à visualização dos hECTs por baixo devido à condensação de água do meio de cultura ou distorções do menisco da interface ar-líquido. Cada contração de uma hECT causa deflexão dos postes terminais integrados, e a medida óptica do sinal de deflexão é processada em um traçado força versus tempo representando a função contrátil da hECT 1,2,3,4,5,6,7,8,9,15,27 . Comparado aos biorreatores de tecido único tipicamente usados para tecidos desse tamanho, o desenho multitecidual melhora o rendimento experimental e permite o estudo da sinalização parácrina entre tecidos adjacentes de composição celular potencialmente diferente. Esse sistema foi validado em estudos publicados descrevendo aplicações em modelagem dedoenças4,8, sinalização parácrina6,7, cultura heterocelular5,9 e triagemterapêutica7,9.
Neste sistema, os hECTs são projetados para ter aproximadamente 6 mm de comprimento e 0,5 mm de diâmetro para facilitar o rastreamento óptico robusto de medições de força com baixo ruído. Além disso, aspectos da complexidade tecidual, como gradientes de difusão e organização celular, são equilibrados com uma necessidade gerenciável de 1 milhão de células por tecido. Com a tecnologia de câmera CCD padrão, forças tão fracas quanto 1 μN (representando menos de 5 μm pós-deflexão) geram um sinal claro, garantindo que mesmo a função contrátil extremamente fraca, como observado com alguns modelos de doença hECT, possa ser medida com precisão. Isso também facilita a análise detalhada da curva de força de contração, permitindo a análise de alto conteúdo de até 16 métricas de contratilidade41, incluindo força desenvolvida, taxas de contração (+dF/dt) e relaxamento (−dF/dt) e variabilidade da taxa de batimento.
Este protocolo começa com instruções para a fabricação dos componentes do biorreator. Atenção especial é dada às etapas para maximizar o rendimento da hECT, reduzir a variabilidade técnica na função tecidual e otimizar a qualidade e a profundidade da avaliação tecidual. A maioria dos estudos de engenharia de tecidos cardíacos não relata taxas de perda tecidual durante a fabricação e testes de longo prazo, embora seja um desafio bem conhecido na área e reduza o rendimento e a eficiência dos estudos27. Os métodos de engenharia de tecidos descritos aqui foram refinados ao longo dos anos para garantir a retenção de todos os hECTs na maioria dos biorreatores (independentemente de como os racks PDMS são fabricados). No entanto, mesmo uma perda de 5%-20% de tecidos pode afetar significativamente o poder estatístico, particularmente em experimentos menores limitados pelo número de cardiomiócitos disponíveis (por exemplo, devido a desafios de diferenciação com algumas linhagens celulares doentes4 ou devido ao alto custo dos cardiomiócitos comprados comercialmente), ou pela condição de tratamento (por exemplo, disponibilidade limitada ou alto custo de vários compostos de tratamento).
Este protocolo descreve a fabricação de rastreadores de poste estável (SPoTs), um novo recurso dos racks PDMS, que funcionam como tampas nas extremidades dos postes de detecção de força que seguram os hECTs27. Demonstra-se como a geometria da tampa reduz significativamente a perda de hECT por queda ou arrancamento dos postes, abrindo assim novas oportunidades para a cultura de hECTs com uma maior variedade de rigidezes e tensões, que são desafiadoras para a cultura em pinos sem tampa. Além disso, os SPoTs fornecem um objeto de alto contraste para melhorar o rastreamento óptico da contração da hECT por meio de uma forma consistente e bem definida27. Segue-se uma descrição da cultura de células-tronco pluripotentes induzidas humanas (iPSCs) e diferenciação de cardiomiócitos com base em protocolos previamente publicados 3,42,43 e uma explicação da fabricação, cultura e medidas funcionais da hECT.
Este artigo também aborda a necessidade de medir a função tecidual em temperatura fisiológica. O miocárdio humano (tanto tecido fetal como adulto saudável e doente), bem como tecido cardíaco de uma ampla gama de espécies animais (incluindo ratos, gatos, camundongos, furões e coelhos)44,45, exibe um aumento acentuado na força de contração compatível com a frequência em temperaturas de 28 °C-32 °C em comparação com a temperatura fisiológica - um fenômeno conhecido como inotropia hipotérmica45, 46º. No entanto, os efeitos da temperatura sobre a função do tecido miocárdico modificado permanecem pouco estudados. Muitos modelos recentes de tecido cardíaco projetados na literatura são projetados para serem avaliados funcionalmente a 37 °C para aproximar condições fisiológicas 13,14,37. No entanto, até onde sabemos, os efeitos dependentes da temperatura sobre a força gerada pelos tecidos cardíacos modificados não têm sido sistematicamente investigados. Esse protocolo descreve um projeto de eletrodo de estimulação que minimiza a perda de calor durante o teste, além de permitir a incorporação de um elemento de aquecimento isolado no setup para medidas funcionais, o que pode manter os hECTs em temperatura fisiológica sem comprometer a esterilidade27. Em seguida, relatamos alguns dos efeitos observados da temperatura na função hECT, incluindo sobre a força desenvolvida, frequência de batimento espontâneo, +dF/dt e −dF/dt. Em conjunto, este artigo fornece os detalhes necessários para fabricar este sistema de biorreator multitecidual com sensor de força para fabricar tecidos cardíacos humanos projetados e avaliar sua função contrátil, e um conjunto de dados é apresentado que fornece uma base de comparação para medições à temperatura ambiente e a 37 °C27.
Protocolo
Esse protocolo utilizou uma linha iPSC não identificada, SkiPS 31.3 (originalmente reprogramada com fibroblastos dérmicos de um homem saudável de 45 anos)47, sendo, portanto, isento de aprovação específica do Comitê de Ética em Pesquisa da instituição, em concordância com as diretrizes do comitê de ética em pesquisa com seres humanos da instituição. Realizar toda a manipulação celular e hECT em condições assépticas em um gabinete de segurança biológica classe II filtrado por HEPA ou bancada de trabalho de fluxo laminar. Esterilizar todas as soluções não estéreis por filtração através de um filtro de 0,22 μm e manter todas as células e hECTs em estufa a 37 °C, 95% de umidade relativa e 5% de CO2.
1. Fabricação de biorreatores
- Fabricação de componentes de biorreatores e master cast de alumínio
Observação : arquivos de projeto auxiliado por computador (CAD) são fornecidos no arquivo suplementar 1. O protocolo pode ser pausado em qualquer lugar entre essas etapas. Recomenda-se recrutar um maquinista profissional para fabricar os moldes mestres descritos nesta seção, pois altas tolerâncias (≤5 μm) e um acabamento liso são necessários para a geometria precisa do poste e para o ajuste adequado dos quadros de polissulfona às placas de base de politetrafluoretileno (PTFE) (visando um ajuste de atrito confortável, mas não muito apertado).- Usando um moinho de controle numérico computadorizado (CNC), usine a placa de base para fora de PTFE de acordo com os esquemas da Figura 1A. As hECTs serão formadas nos seis poços uniformemente espaçados (setas brancas).
- Utilizando um moinho CNC, usine o mestre mestre negativo de polidimetilsiloxano (PDMS) fundido em alumínio de acordo com os esquemas da Figura 1B, com três suportes de quadro (asteriscos verdes). Faça seis furos uniformemente espaçados (pontas de seta magenta) de 0,5 mm de diâmetro para criar os postes PDMS.
- Usando um moinho CNC, usine a estrutura do biorreator de polissulfona de acordo com os esquemas da Figura 1C. Os suportes de quadro (asteriscos verdes) correspondem aos suportes de quadro vistos no rack fundido (Figura 1B, asteriscos verdes).
- Usando um moinho CNC, usine o suporte fundido de alumínio de acordo com os esquemas da Figura 1D. Cada slot contém uma prateleira triangular (triângulo laranja) que tem 0,25 mm de altura para fornecer um espaço morto para o PDMS fluir através dos orifícios nos moldes de rack do PDMS (Figura 1B, pontas de seta magenta).
- Fundição do rack PDMS a partir dos mestres negativos de alumínio
- Usando uma impressora 3D termoplástica de modelagem de deposição fundida, imprima dois suportes de aparelhos de fundição em rack PDMS (Arquivo Suplementar 1). Use as seguintes configurações de impressão: uma altura de camada de 0,1 mm, uma espessura de parede/fundo/topo de 1 mm, uma densidade de preenchimento de 90% com triângulos, uma temperatura de impressão de 230 °C, uma temperatura de placa de construção de 70 °C e uma borda para aderência.
- Coloque quatro moldes mestres negativos de alumínio no suporte fundido (Figura 2AI) de modo que os orifícios do poste se alinhem com o espaço morto oposto às prateleiras do triângulo (ver Figura 1D). Embrulhe o aparelho em uma peça retangular de chapa de silicone de 0,5 mm de espessura (Figura 2B, setas brancas) como uma junta para evitar vazamento do PDMS líquido e fixe-o entre dois suportes paralelos impressos em 3D usando uma braçadeira de parafuso.
- Adicionar 0,5 mL de agente de cura PDMS a 5 mL de base de elastômero PDMS (proporção 1:10, de acordo com as instruções do fabricante) em um recipiente raso e misturar vigorosamente por 5 min. Desgaseifique a mistura de PDMS em uma câmara de vácuo e aplique um vácuo forte (0,1-1 kPa) por 20-60 min à temperatura ambiente ou até que as bolhas desapareçam.
- Despeje a mistura de PDMS sobre o aparelho de fundição, enchendo demais para garantir a cobertura completa de cada slot (Figura 2AII). Se desejar, adicione pequenas esferas de vidro coloridas ao corpo dos racks PDMS (Figura 2AII), opostos ao lado com os postes (Figura 2B), para a identificação exclusiva de cada rack PDMS. Retorne o aparelho de fundição para a câmara de vácuo (garantindo que ele esteja nivelado horizontalmente) e aplique um vácuo forte por pelo menos 12 h. Deixe o PDMS curar à temperatura ambiente por cerca de 48 h longe da poeira para permitir a cura completa e a máxima resistência dos pinos delicados. Evite usar um forno, pois isso deforma os componentes impressos em 3D.
NOTA: O protocolo pode ser pausado aqui.
- Remoção do rack PDMS dos moldes mestres negativos de alumínio
- Remova a braçadeira, os suportes e as chapas de silicone do aparelho de fundição. Usando uma lâmina de barbear de aço inoxidável, corte a película PDMS na parte superior do aparelho de fundição e dos suportes da armação e use suavemente os dedos para separar os racks PDMS dos lados do suporte do molde. Insira uma lâmina de barbear de aço inoxidável embotada no espaço morto entre o molde e o suporte fundido e separe-os (Figura 2CI, II), garantindo que o PDMS que preenche o espaço morto permaneça com o suporte fundido (pois este está preso aos postes). Usando uma lâmina afiada de aço inoxidável, corte os filmes PDMS restantes e corte o PDMS de espaço morto das pontas dos pinos (Figura 2C III-V).
- ETAPA CRÍTICA: Liberte o rack PDMS do molde (Figura 2D). Começando pelo lado oposto aos postes, use os dedos para separar lentamente o rack PDMS do elenco, trabalhando em lados alternados até que os postes estejam livres dos moldes mestres.
- Repita a etapa anterior até que todos os racks do PDMS e todas as postagens sejam liberadas. Use uma lâmina de barbear afiada para aparar qualquer excesso de PDMS restante dos racks. O resultado é um rack PDMS (Figura 2E) com seis pinos intactos (pontas de seta laranja) e contas coloridas (seta azul) para identificação.
NOTA: O protocolo pode ser pausado aqui.
- Fabricação de pós-rastreador estável (SPoT)
- Usando uma impressora 3D termoplástica com modelagem de deposição fundida, imprima os componentes do aparelho de fundição SPoT (Arquivo Suplementar 2 e Figura 3AI, II). Use as seguintes configurações de impressão: uma altura de camada de 0,1 mm, uma espessura de parede/fundo/topo de 1 mm, uma densidade de preenchimento de 80% com triângulos, uma temperatura de impressão de 230 °C, uma temperatura de placa de construção de 70 °C e uma borda para aderência.
- Garanta um encaixe seguro da prensa entre as peças impressas em 3D, bem como entre os racks PDMS e o gabarito de três pontas, e confirme se os racks PDMS se encaixam perfeitamente com os postes que acabam atingindo o fundo dos poços sem serem dobrados. Apague/arquive o plástico, se necessário.
- Adicionar 0,5 mL de PDMS preto parte A a 0,5 mL de parte B (proporção 1:1, de acordo com as instruções do fabricante) a um barco de pesagem pequeno (ou um recipiente pequeno e raso semelhante) e misturar cuidadosamente até obter uma cor uniforme. Desgaseifique o PDMS preto misto em uma câmara de vácuo sob um vácuo forte por 20 min. Despeje o PDMS preto desgaseificado na base impressa em 3D para preencher os orifícios e toque para garantir que não restem bolhas. Raspe o máximo possível de PDMS em excesso da base.
- Encaixe a peça de três pontas na base e coloque os racks PDMS nos sulcos do gabarito de três pontas (Figura 3AII, retângulo turquesa), garantindo que as extremidades dos postes mergulhem no PDMS preto nos poços circulares (Figura 3B, C). Curar o PDMS preto à temperatura ambiente e protegido da poeira por 48 h.
NOTA: O protocolo pode ser pausado aqui. - Deslize a peça de três pontas, minimizando a tensão nos postes. Use pinças pequenas para raspar a fina película de PDMS preto que envolve cada SPoT; em seguida, insira pinças dobradas de ponta fina no poço SPoT para liberá-lo da base impressa em 3D (Figura 3D).
- Inspecione os SPoTs (Figura 3E) e corte qualquer filme PDMS preto restante do processo de fundição não removido na etapa 1.4.5 usando uma tesoura fina de Vannas. Certifique-se de que os postes acabados tenham o comprimento correto, encaixando os racks PDMS na estrutura de polissulfona e, em seguida, deslizando-a na placa de base preta (Figura 4A).
NOTA: O protocolo pode ser pausado aqui. - Emparelhe os racks PDMS e adicione-os ao quadro usando as guias de quadro (Figura 4A). Autoclave em um saco com uma placa de base de PTFE por pelo menos um ciclo de 30 min (<122 °C para reduzir o empenamento).
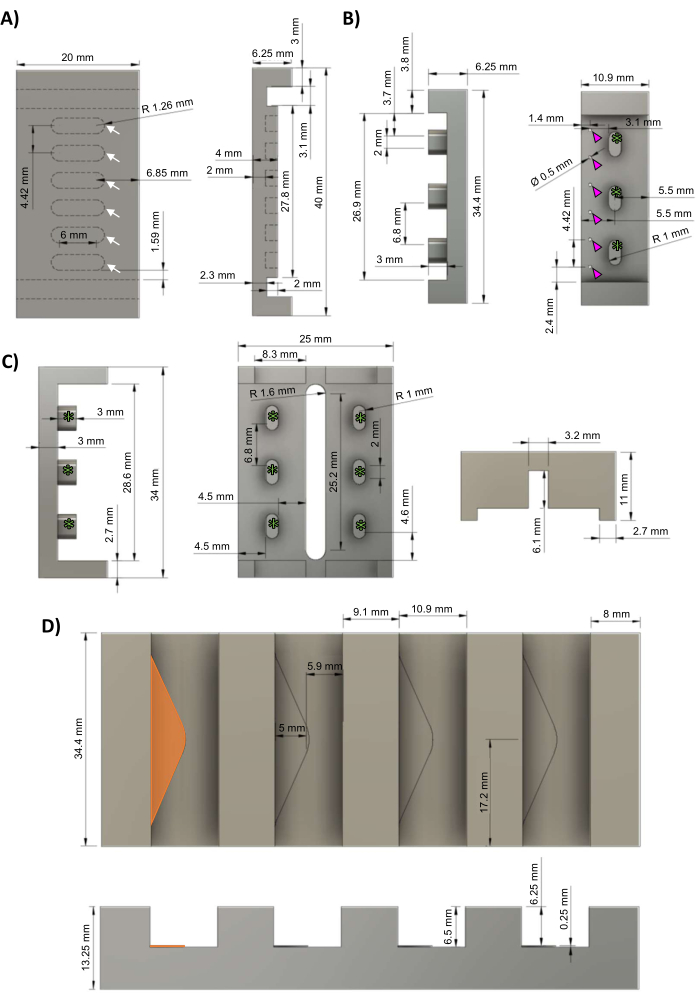
Figura 1: Componentes do biorreator hECT. (A) Vista superior (esquerda) e vista lateral (direita) da placa de base de PTFE com seis poços uniformemente espaçados para formação de hECTs (setas brancas). (B) Vista lateral (esquerda) e vista superior (direita) dos moldes mestres negativos de alumínio para os racks PDMS com seis postes uniformemente espaçados (pontas de seta magenta) e três vãos para fixação à estrutura do biorreator (asteriscos verdes). (C) Vista lateral (esquerda) e visão inferior (direita) dos quadros de polissulfona para os racks PDMS com três suportes de quadro uniformemente espaçados (asteriscos verdes) correspondentes aos suportes de quadro no rack cast PDMS (painel B). (D) Vista superior (superior) e vista lateral (inferior) do suporte fundido em alumínio com quatro ranhuras para os moldes de rack PDMS, cada um com uma prateleira triangular de 0,25 mm de altura (prateleira mais à esquerda destacada em laranja). Este valor foi modificado a partir de van Neste27. Abreviações: hECT = tecido cardíaco humano; Ø = diâmetro; PTFE = politetrafluoretileno; PDMS = polidimetilsiloxano; R = raio. Clique aqui para ver uma versão maior desta figura.
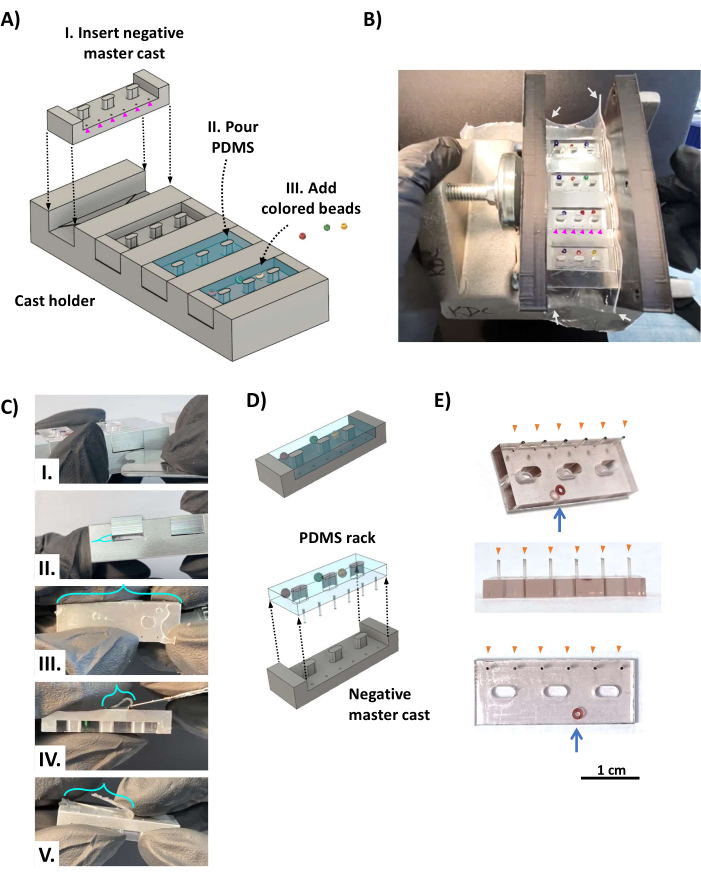
Figura 2: Fabricação dos racks PDMS. (A) As renderizações CAD mostram uma visão oblíqua do aparelho de fundição. (I) Um molde mestre de rack PDMS negativo é inserido em cada um dos quatro slots do suporte fundido com os orifícios que formam os postes PDMS (pontas de seta magenta) colocados sobre o espaço morto oposto à prateleira triangular (Figura 1D, triângulo laranja). (II) O PDMS é despejado em cada cavidade do molde mestre negativo. (III) Contas coloridas são adicionadas ao PDMS não curado como um sistema de identificação codificado por cores. (B) Foto mostrando o aparelho de fundição em rack PDMS montado, que é fixado em ambos os lados com dois suportes impressos em 3D mantidos no lugar por uma braçadeira de parafuso e envolvido com chapas de silicone de 0,5 mm de espessura (setas brancas) para selar as laterais presas. As contas coloridas são colocadas de forma que não cubram os orifícios de 0,5 mm de diâmetro que formam os postes (pontas de seta magenta). (C) Uma vez curado o PDMS, o molde é removido do suporte do molde. (I) Uma lâmina de barbear de aço inoxidável embotada ou uma ferramenta metálica fina semelhante é inserida entre o molde e o suporte do molde para arrancar o molde do suporte do molde (II). (III) A película (braquesas turquesa) formada pelo PDMS que flui através dos orifícios dos postes é fixada às pontas dos postes e deve ser cortada com lâmina afiada (IV,V). (D) O rack PDMS é separado do molde. (E) Fotos mostrando vistas oblíquas (superior), lateral (meio) e inferior (inferior) do rack PDMS com uma conta de vidro embutida no corpo para identificação (seta azul). As pontas dos postes (pontas de seta laranja) foram marcadas com tinta preta. Barra de escala = 1 cm. Este valor foi modificado a partir de van Neste27. Abreviações: CAD = computer-aided design; PDMS = polidimetilsiloxano. Clique aqui para ver uma versão maior desta figura.
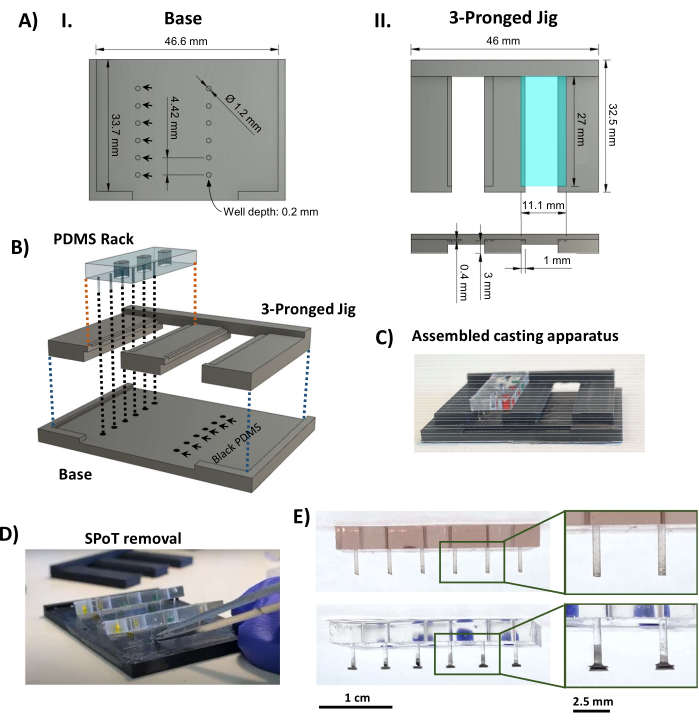
Figura 3: Fabricação do SPoT. (A) renderizações CAD indicando as dimensões-chave da base (I) e (II) peça de três pontas do gabarito de fundição SPoT. As dimensões das formas circulares SPoT (AI, setas pretas) são definidas como 0,2 mm de profundidade x 1,2 mm de diâmetro, e cada uma contém o PDMS preto para uma SPoT individual. A prateleira de 11,1 mm x 27 mm vista na vista superior (AII, superior, retângulo turquesa) é pressionada em 0,4 mm (como visto na vista lateral abaixo) para manter o rack PDMS no lugar durante a cura. (B) Renderização CAD mostrando a montagem do aparelho de fundição SPoT. (C) Uma foto do aparelho de fundição SPoT montado. (D) Após a cura do PDMS, o gabarito de três pontas é deslizado para fora de baixo dos racks do PDMS, e os SPoTs são liberados de seus poços usando pinças finas. (E) Fotos do rack PDMS sem (superior) e com (inferior) SPoTs. As inserções mostram visualizações ampliadas das postagens. Barras de escala = 1 cm (E), 2,5 cm (imagens ampliadas em E). Este valor foi modificado a partir de van Neste27. Abreviações: CAD = computer-aided design; Ø = diâmetro; PDMS = polidimetilsiloxano; R = raio; SPoT = rastreador de post estável. Clique aqui para ver uma versão maior desta figura.
2. Cultura celular
- Cultivando os iPSCs
NOTA: Diferentes linhagens celulares podem exigir ajustes na diluição e frequência de passagem e/ou na titulação dos aditivos do meio.- Revestir uma placa de 6 poços tratada com cultura celular com matriz de membrana basal qualificada (diluída em meio de águia modificado 1:1 Dulbecco: solução nutritiva F12 de Ham's [DMEM/F12] de acordo com as instruções do fabricante) e incubar a placa a 37 °C por pelo menos 30 min. Preparar 500 mL de meio de cultura iPSC de acordo com as instruções do fabricante e adicionar 5 mL de solução estoque de penicilina-estreptomicina (10.000 UI/mL a 10.000 μg/mL).
- Para passar as iPSCs, aspirar o meio dos poços e lavar cada poço uma vez com 1 mL de solução salina tamponada com fosfato (PBS). Adicionar 1 mL de solução de dissociação iPSC por poço e incubar em uma capela de fluxo laminar por 1 min.
- Aspirar a solução de dissociação iPSC e incubar as células a 37 °C (sem qualquer meio) durante 5 minutos. Adicionar 1 mL de tiazovivina 2 μM em meio iPSC para neutralizar a solução de dissociação iPSC.
- Use uma pipeta sorológica de 2 mL para dissociar as colônias em aglomerados de aproximadamente 10 células e lave cada poço com mais 1 mL de tiazovivina 2 μM em meio iPSC. Adicionar 2 ml de suspensão celular a cada poço da placa recém-revestida com matriz de membrana basal (passo 2.1.1).
- Após 24 h, retire o meio e adicione o meio iPSC fresco (sem tiazovivina). Alimentar as iPSCs a cada 48 h com 2 mL de meio iPSC ou a cada 72 h com 4 mL de meio. Replaquear as células em diluição de 1:6 para passagem a cada 3 dias ou quando atingirem 80% de confluência.
NOTA: Diferentes linhas celulares podem exigir o ajuste da diluição e da frequência de passagem.
- Diferenciação cardiomiocitária
- Comece a diferenciação quando as monocamadas iPSC forem 80%-90% confluentes.
- Preparar o meio de diferenciação adicionando 10 mL de suplemento B27 sem insulina e 5 mL de solução estoque de penicilina-estreptomicina a 500 mL de meio Roswell Park Memorial Institute 1640 (RPMI). Preparar o meio de manutenção de cardiomiócitos adicionando 10 mL de suplemento B27 e 5 mL de solução estoque de penicilina-estreptomicina a 500 mL de RPMI 1640.
NOTA: O meio de diferenciação e o meio de manutenção do cardiomiócito podem ser armazenados a 4 °C por até 2 semanas. - Dia 0: Lavar as células com 1 mL de DMEM/F12 e adicionar 2 mL de CHIR99021 10 μM e matriz de membrana basal diluída em meio de diferenciação.
- Dia 1: Após 24 h, ou quando a confluência celular tiver diminuído para menos de 70%, lavar as células com 1 mL de DMEM/F12, adicionar 2 mL de meio de diferenciação e incubar por 48 h.
- Dias 3-4: Lavar as células com 1 mL de DMEM/F12 e adicionar 2 mL de 5 μM de IWR-1 em meio de diferenciação. Repita no dia 4.
- Dias 5-6: Lavar as células com 1 mL de DMEM/F12 e adicionar 2 mL de meio de diferenciação. Repita no dia 6.
- Dias 7-10: Lavar as células com 1 mL de DMEM/F12 e adicionar 2 mL de meio de manutenção de cardiomiócitos. Repita a cada 24 h.
- Dias 11+: Substituir o meio por 4 mL de meio de manutenção de cardiomiócitos frescos a cada 48-72 h. Aspirar e pipetar lentamente para evitar danificar as monocamadas que batem vigorosamente.
3. Cultura hECT
- Colheita dos cardiomiócitos
- Colher as monocamadas de cardiomiócitos para uso na fabricação do hECT 8-60 dias após a indução da diferenciação. Espere 2-5 milhões de células por poço.
NOTA: Se as células não começaram a bater até o dia 10, é improvável que a diferenciação seja bem-sucedida. As monocamadas que batem vigorosamente geralmente se desprendem após 11-15 dias na diferenciação e compactam em tecidos densos. Recomenda-se usar ou repor essas células neste momento. - Enxaguar cada poço de cardiomiócitos 2x com 2 mL de PBS. Adicionar 1 mL de tripsina-EDTA 0,25% à temperatura ambiente. Incubar a 37 °C durante 5-10 minutos até que as células pareçam arredondadas e se desprendam com um leve toque da placa.
- Adicionar 1 mL de SFB a 10% em meio de manutenção de cardiomiócitos a cada poço para neutralizar a dissociação. Pipetar suavemente as monocamadas usando uma ponta de pipeta sorológica de 5 mL e transferir para um tubo cônico de 50 mL para quebrar o pellet em aglomerados de 10-20 células.
- Misturar a suspensão celular invertendo o tubo cónico antes de transferir 10 μL de células para 10 μL de azul de tripano. Conte as células usando um contador de células automatizado ou hemocitômetro de vidro. Separe a suspensão celular adequadamente se nem todas as células forem usadas ou se algumas células forem reservadas para citometria de fluxo.
NOTA: Células suplementares (como fibroblastos) podem ser adicionadas neste ponto. - Centrifugar as células a 250 × g durante 5 min. Aspirar imediatamente o máximo de sobrenadante possível, sem perturbar a pelota celular, e manter no gelo. Trabalhe rapidamente para minimizar o tempo que as células passam no pellet.
- Colher as monocamadas de cardiomiócitos para uso na fabricação do hECT 8-60 dias após a indução da diferenciação. Espere 2-5 milhões de células por poço.
- Fabricação de hECT
- Use os volumes da Tabela 2 e ajuste de acordo com o número de células no pellet de modo que cada hECT contenha 1 milhão de células. Após cada passo, misture pipetando lentamente para evitar bolhas.
Observação : execute as etapas 3.2.2-3.2.3 blindado contra luz direta, pois alguns componentes são sensíveis à luz. - Preparar uma solução de colágeno tipo 1 de 2,9 g/mL em um microtubo de 1,7 mL adicionando 13,442 μL de água destilada, 4,4 μL de PBS 10x e 0,638 μL de NaOH 1M. Adicionar 25,52 μL de 5 mg/mL de solução estoque de colágeno e misturar lentamente.
- Preparar mistura de matriz extracelular (mistura de MEC da Tabela 2): Adicionar 5,5 μL de solução de HEPES 0,2 N pH 9 seguido de 5,5 μL de 10x MEM. Misture bem até observar uma cor uniforme amarelo-claro a rosa-claro. Transferir 35,2 μL da solução de mistura de MEC para o pellet de células e adicionar 4,4 μL de matriz de membrana basal.
- Abra o saco autoclavado das peças do biorreator (passo 1.4.7, Figura 4A). Enquanto estiver usando luvas esterilizadas com etanol 70%, retire a placa de base preta do saco de autoclave e coloque em uma placa de 60 mm com os poços voltados para cima. Pipetar 44 μL da mistura celular em cada poço lentamente para evitar a introdução de bolhas. Se necessário, use a pipeta para remover quaisquer bolhas que foram introduzidas por pipetagem ou se formaram devido à hidrofobicidade do PTFE. Restaurar o volume da hECT de forma que a superfície do líquido fique nivelada com o lábio do poço (Figura 4BI).
- Use um novo par de luvas esterilizadas e remova a estrutura de polissulfona com racks PDMS do saco de autoclave. Abaixe o quadro sobre a placa de base de modo que as extremidades do quadro se encaixem nos sulcos nas extremidades da placa de base (Figura 4BII, III). Inspecione o biorreator para garantir que os postes estejam todos retos e que a estrutura não seja inclinada antes de colocar em uma placa de 60 mm.
- Adicione 1 mL de FBS a 10% em meio de manutenção de cardiomiócitos à placa de 60 mm (tome cuidado para não perturbar os hECTs) para aumentar a umidade na placa à medida que os hECTs se solidificam. Coloque a placa de 60 mm sem tampa em uma placa de perfil alto (20 mm de altura) de 100 mm, cubra com uma tampa de prato de 100 mm e devolva o biorreator à incubadora de 37 °C, 5% de CO2 para permitir que o colágeno forme um gel com as células em suspensão.
- Após 2 h, retire o prato da incubadora. Adicionar 13 mL de SFB a 10% no meio de manutenção do cardiomiócito, inclinando a placa para estimular o fluxo do meio entre a placa de base de PTFE e os racks do PDMS.
- Inspecione o biorreator do lado para garantir que nenhuma bolha de ar fique presa entre as superfícies hidrofóbicas da placa de base de PTFE e os racks PDMS, e devolva a placa à incubadora. Se houver ar aprisionado, incline o biorreator para fora do meio para deixar a bolha se romper e baixá-la lentamente novamente, ou use uma micropipeta com uma ponta de carregamento de gel para sifão do ar, tomando cuidado para não atrapalhar os postes.
- Use os volumes da Tabela 2 e ajuste de acordo com o número de células no pellet de modo que cada hECT contenha 1 milhão de células. Após cada passo, misture pipetando lentamente para evitar bolhas.
- Remoção da placa de base
- Inspecione a compactação hECT através da folga no quadro. Ao longo de 24-96 h, as hECTs compactam-se e tornam-se mais opacas (Figura 4IC-III). Uma vez que haja um espaço visível entre os hECTs e a parede da placa basal (Figura 4CII), realize duas trocas de meio volume para trocar o meio de manutenção do meio para cardiomiócitos sem SFB. Remova a placa basal quando as hECTs estiverem compactadas em pelo menos 30% em relação ao diâmetro original (Figura 4CIII). Encher a placa de 60 mm que contém o biorreator com meio de manutenção de cardiomiócitos até que o líquido esteja nivelado com o lábio da placa e adicionar 14 mL a uma nova placa de 60 mm.
- PASSO CRÍTICO: Ao usar luvas estéreis, vire o biorreator em sua placa para que a placa de base fique em cima (Figura 4BIV). Inspecionar a existência de bolhas de ar aprisionadas, como na etapa 3.2.8. Levante lentamente a placa basal, mantendo-a nivelada (Figura 4BV).
- Se uma hECT cair durante a remoção da placa de base, mas permanecer na placa de base, use pinças finas curvas estéreis para transferir a hECT da placa de base para a placa de 60 mm. Utilize a pinça para guiar a extremidade da hECT até o seu poste. Use um segundo par de pinças para manter o poste firme e enfie-o através do orifício no hECT. Repita para o segundo post, se necessário.
- Com todos os hECTs presos aos postes, transfira o quadro com os hECTs para a nova placa de 60 mm e coloque o quadro com os postes apontando para baixo (Figura 4BVI). Inspecione o biorreator para garantir que os hECTs permaneçam em seus postes apenas próximos aos SPoTs.
- Se uma hECT tiver sido empurrada por tensão superficial para a base de seus postes, estabilize o quadro com um par de pinças curvas estéreis. Insira o outro par de pinças através da ranhura no quadro, mantendo-o fechado. Uma vez que a ponta da pinça tenha sido abaixada para além dos racks PDMS, torça-a para que ela atinja o poste e use as pontas fechadas para empurrar suavemente a hECT em direção ao final do poste até que ela fique apoiada na SPoT (Figura 4BVI, inset).
- Manutenção hECT
- Realizar trocas de meio volume com meio de manutenção de cardiomiócitos a cada 24-48 h (após 2 semanas de cultura, a frequência pode ser reduzida para duas vezes por semana).
- Quando os hECTs exibem agrupamentos de batimentos espontâneos, tipicamente no dia 3, e batimento coordenado com pós-deflexão visível no dia 5, inicie as medidas funcionais e repita quantas vezes desejar.
NOTA: hECTs que não iniciaram o batimento coordenado até o dia 7 são improváveis de fazê-lo.
| Componente | Volume (μL) | |||||||
| destilado H2O | 13.442 | 2,9 mg/mL de solução de colágeno | "Mix de ECM" | mistura final de células hECT | ||||
| NaOH 1N | 0.638 | |||||||
| PBS 10x | 4.4 | |||||||
| 5 mg/mL de colágeno | 25.52 | |||||||
| 0,2 N pH 9 HEPES | 5.5 | |||||||
| 10x MEM | 5.5 | |||||||
| Volume da mistura de ECM para transferir para a pastilha celular | 35.2 | |||||||
| Volume de Matrigel | 4.4 | |||||||
Tabela 2: Reagentes de hECT. Os componentes devem ser adicionados na ordem listada e mantidos no gelo.
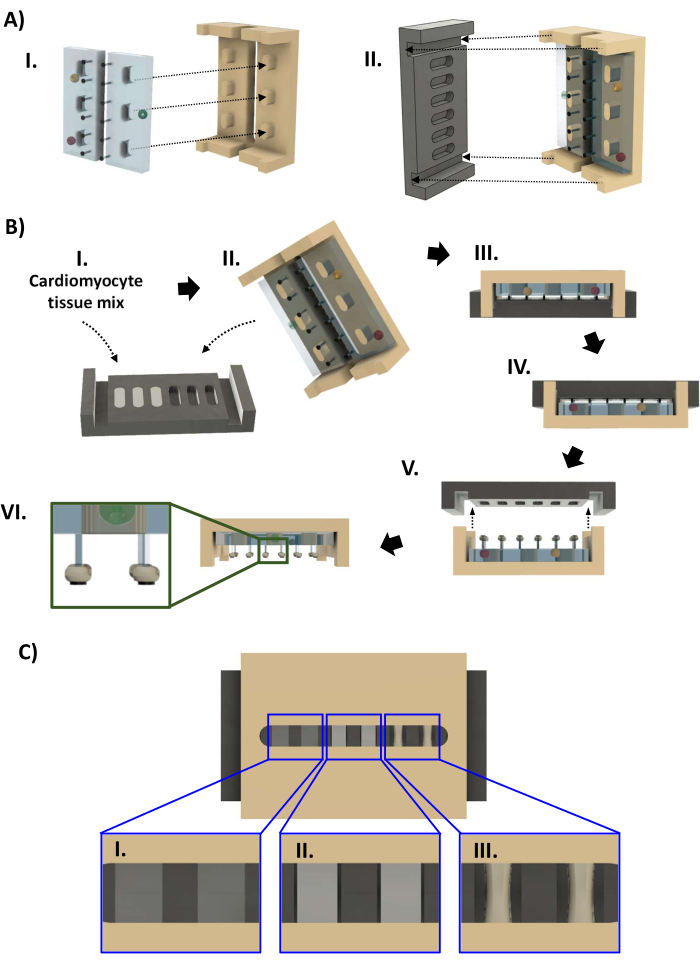
Figura 4: Montagem do biorreator e fabricação do hECT. (A) (I) Dois racks PDMS (esquerda, azul claro) instalados na estrutura de polissulfona (direita, bronzeado). (II) A placa de base de PTFE (preta, esquerda) então se encaixa no quadro (direita) de tal forma que cada par de postes se encaixe em um poço da placa de base. (B) (I) Quarenta e quatro microlitros de suspensão de cardiomiócitos em matriz extracelular à base de colágeno são adicionados a cada um dos seis poços da placa basal. (II,III) O quadro com racks PDMS é encaixado na placa de base. Após 1-4 dias, os hECTs podem ser removidos da placa basal. (IV) Primeiro, o biorreator é invertido antes que (V) a placa de base seja levantada do quadro. (VI) Vista lateral do biorreator com seis hECTs. Inset: visualização ampliada mostrando a posição hECT nos postes em relação aos SPoTs (inset). (C) Renderização CAD mostrando três níveis de compactação hECT ([I] baixo, [II] médio e [III] alto) como visto através da lacuna no quadro de polissulfona. Este valor foi modificado a partir de van Neste27. Abreviações: CAD = computer-aided design; PDMS = polidimetilsiloxano; PTFE = politetrafluoretileno; SPoT = rastreador de pós estável; hECT = tecido cardíaco humano projetado. Clique aqui para ver uma versão maior desta figura.
4. Equipamento de estimulação hECT
- Jaqueta para o palco aquecido
- Use uma máquina de corte a laser para cortar os componentes da camisa isolante acrílica de uma folha de acrílico transparente de 0,635 cm de espessura (Arquivo Suplementar 3), uma cada da Figura 5A-D e duas cada da Figura 5E, F.
- Montar as partes (B), (C), (D) e uma das (E) da Figura 5 e unir usando cola acrílica como mostra a Figura 5GI. Fixe o painel superior (Figura 5GII), aguarde várias horas para que a cola se fixe e, em seguida, deslize o palco aquecido para o lado da jaqueta (Figura 5GIII).
- Uma vez que a jaqueta esteja no lugar, use fita adesiva para prender as pastilhas entre as pernas do palco aquecido e adicione o painel frontal (Figura 5GIV) para finalizar a montagem (Figura 5GV).
- Fabricação de eletrodos de grafite
- Cortar barras de grafite de 6,25 mm de espessura e 25 mm de largura usando uma serra de fita em blocos de 35 mm de comprimento; Em seguida, corte cada bloco longitudinalmente em uma linha curva de modo que cada eletrodo tenha 13-16 mm de altura em uma extremidade e 8-10 mm de altura na outra extremidade. Faça dois furos de 0,7 mm de diâmetro no canto superior (Figura 6AI). Polir as peças com papel toalha e sonicar em água por 20 min para remover o pó de grafite. Certifique-se de que os eletrodos fiquem entre as paredes da placa e o biorreator de 25 mm de largura para garantir uma distância consistente entre os eletrodos (Figura 6AII).
- Rosqueie um fio de aço de 150 mm de comprimento x 0,25 mm de diâmetro através dos orifícios dos eletrodos e dobre-o para encaixar sobre o lábio da placa de 60 mm e ao redor das paredes da placa de 100 mm para que a tampa possa ser fechada (Figura 6AII).
- Limpe os eletrodos mergulhando em água destilada por 1-2 h após cada uso para remover qualquer meio absorvido, deixe secar durante a noite e, em seguida, autoclave a 132 °C por 30 min. Antes de iniciar as medições, colocar um eletrodo de cada lado do biorreator (Figura 6AII). Posicione os fios de forma que a tampa da placa de 100 mm possa ser fechada e retorne o biorreator à incubadora para equilibrar.
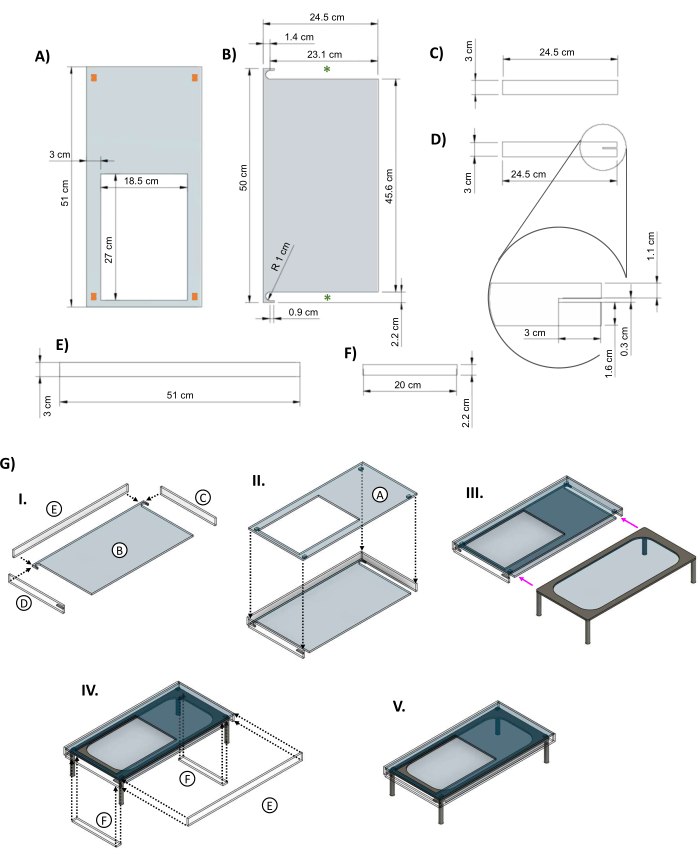
Figura 5: Jaqueta acrílica para isolamento do palco de vidro aquecido. Imagens CAD mostrando as principais dimensões das peças da jaqueta de acrílico projetada para a mesa de vidro. (A) O painel superior tem um recorte de furo de 27 cm x 18,5 cm para permitir que a placa do biorreator fique sobre o elemento de aquecimento. Os retângulos alaranjados nos cantos indicam a colocação sugerida de pequenos espaçadores para proporcionar espaço entre a parte superior da jaqueta e o elemento de aquecimento. (B) A parte inferior da jaqueta tem dois recortes para permitir que as pernas do palco aquecido deslizem (asteriscos verdes). (C&D) Dois painéis laterais cabem sob a peça superior. (D) O painel lateral esquerdo inclui um recorte de 3 cm x 0,3 cm (inset) para o cabo de alimentação do palco. (E) Painéis longos cabem na frente e atrás. (F) Inserções são adicionadas para preencher as lacunas quando a mesa estiver dentro. (G) (I) Os painéis laterais e traseiros são fixados à peça inferior e, em seguida, (II) o painel superior é adicionado. (III) A mesa de vidro é deslizada para dentro da jaqueta (setas magenta). (IV) As pastilhas são fixadas entre as pernas da mesa, e o encosto se encaixa na abertura para fechar a caixa. (V) O conjunto completo da jaqueta. Este valor foi modificado a partir de van Neste27. Abreviações: CAD = computer-aided design; R = raio; Ø = diâmetro. Clique aqui para ver uma versão maior desta figura.

Figura 6: Aquisição dos dados da contração da hECT. (A) (I) Fotos dos eletrodos cortados a partir de barras de grafite. As setas magenta indicam furos para fixação dos fios de aço inoxidável. Barra de escala = 1 cm. (II) Vista oblíqua (esquerda) e vista superior (direita) mostrando a colocação dos eletrodos de grafite no biorreator. Os eletrodos ocupam o espaço entre o biorreator de 25 mm de largura e a parede da placa para garantir uma distância consistente entre os eletrodos. Os fios são dobrados para permitir o fechamento da tampa do prato. (B) Foto da configuração de estimulação hECT dentro do equipamento de bancada limpa de fluxo laminar - todos os equipamentos são colocados na mesa de isolamento de vibração para reduzir o ruído de vibração da bancada limpa. O biorreator (ponta de seta magenta) fica no palco aquecido encamisado, iluminado por uma fonte de luz LED de cima. O microscópio dissecante é apontado horizontalmente para um espelho de ângulo direito (asterisco laranja) para visualizar o biorreator de baixo e é equipado com uma câmera CCD (esquerda). O suporte turquesa indica um banho de água para monitoramento contínuo da temperatura para fornecer feedback ao controlador de estágio aquecido de circuito fechado. Este valor foi modificado a partir de van Neste27. Abreviações: hECT = tecido cardíaco humano; LED = diodo emissor de luz. Clique aqui para ver uma versão maior desta figura.
5. Medidas funcionais da hECT
- Configurando o espaço de trabalho de ritmo
- Ligue o palco aquecido a 39,5 °C e configure o equipamento de estimulação em uma mesa de isolamento de vibração dentro de uma bancada limpa de fluxo laminar de acordo com a Figura 6B. Monte o microscópio dissecante em um suporte de lança e aponte-o para um espelho de ângulo reto (Figura 6B, asterisco laranja) localizado em um macaco de laboratório abaixo da mesa de vidro para visualizar o biorreator de baixo. Afixe uma câmera CCD de alta velocidade no microscópio e conecte-se ao computador. Irradie o setup com luz UV por 15 min para esterilizar o espaço de trabalho.
- Coloque o biorreator (Figura 6B, ponta de seta magenta) no palco aquecido encamisado, iluminado por uma fonte de luz LED de pescoço de ganso de cabeça dupla de cima (os gargalos das lâmpadas LED podem ser fixados com mais segurança à unidade principal em comparação com as lâmpadas de fibra óptica). Minimize o ruído adicional garantindo que o equipamento de estimulação na mesa de vibração (e na própria mesa) não esteja tocando em nenhuma parte da bancada limpa de fluxo laminar.
- Adicione um segundo prato de 60 mm preenchido com água pré-aquecida dentro de um prato de 100 mm sobre a mesa aquecida (Figura 6B, suporte turquesa) e vista com uma sonda de temperatura para monitoramento contínuo da temperatura. Ajustar a regulação da temperatura do estádio aquecido conforme necessário para manter a temperatura da placa de referência em 36-37 °C.
- Ajuste a ampliação do microscópio para 1,5x (ou outra ampliação desejada com a qual uma hECT possa ser visualizada em sua totalidade com resolução adequada).
- Ajustando as configurações da câmera
- Abra o software da câmera. Redimensione o feed de vídeo para cortar o campo de visão o máximo possível enquanto ainda visualiza um hECT inteiro. Isso maximiza a velocidade da câmera.
- Defina a taxa de captura para 90 quadros por segundo. Ajuste o tempo de exposição e a posição da fonte de luz para otimizar a uniformidade das condições de iluminação em todo o campo de visão e maximizar o contraste dos SPoTs.
- Configuração do software de aquisição
- Ligue o estimulador de pulso quadrado e conecte-o ao computador. Ajuste as configurações para fornecer pulsos bifásicos com uma amplitude de 12 V e uma duração de 5 ms.
- Abra o software de aquisição de dados e, em seguida, abra o arquivo "AutomatedPostTracking3.vi" (Arquivo Suplementar 4). Depois de carregar, clique na seta branca no lado esquerdo da barra de ferramentas para inicializar o programa (Figura 7A).
- Calibrar o software usando um hemocitômetro de vidro no palco aquecido. Na barra de ferramentas (Figura 7B), clique na ferramenta de linha para traçar uma linha em 1 mm das marcações do hemocitômetro (não mostradas). Na caixa Calibração de distância (pixels/mm) (Figura 7C), defina o Comprimento de referência (mm) como 1 e clique no botão Calcular .
- Meça a área de secção transversa do hECT usando a ferramenta de linha para desenhar uma linha em toda a largura do tecido. Clique no 1 na caixa Área de secção transversal (mm^2) (Figura 7D) para calcular a área (assumindo uma geometria cilíndrica das tiras de tecido lineares, conforme estabelecido na literatura 1,2,3,4,5,6,7,8,9,10,11, 12,15,16,18,19,20,
21,22,23,24,25,26,37,38). Repita ao longo de diferentes partes do hECT e registre os valores sob os outros dois botões na caixa. O arquivo de tabela de dados de saída informa a média desses três valores para calcular o diâmetro do tecido.
- Caracterização funcional da hECT
- Certifique-se de que as dicas de postagem estejam em foco. Ligue o interruptor de limiar (Figura 7E) e ajuste o controle deslizante (Figura 7F) até que os SPoTs (Figura 7G) estejam bem demarcados e não mudem de forma à medida que o hECT se contrai.
- Use a ferramenta retângulo para desenhar um retângulo ao redor de um dos SpoTs (Figura 7, retângulo verde) e clique no botão Set 1 dentro da caixa Post boundaries (Figura 7H) para definir a posição do retângulo ao redor do SPoT, garantindo que o SPoT permaneça sempre dentro do limite do retângulo. Repita para a outra postagem e registre-a em Conjunto 2.
- Ajuste as configurações de tamanho do objeto (Figura 7I) para impedir que o programa rastreie objetos menores. Certifique-se de que o número de objetos rastreados em cada retângulo permaneça constante. A interface (Figura 7J) mostra a distância medida entre os objetos rastreados em tempo real. Use este gráfico para monitorar o ruído.
- Selecione um diretório para salvar os arquivos (Figura 7K). Armazene dados de dias diferentes em pastas separadas. Selecione o número de tecido atual e escreva os comentários desejados na caixa Comentários .
- No cabeçalho Pacing Frequency (Hz) (Figura 7L), indique a faixa de frequências desejada (Min e Max) e o Intervalo desejado para passar de Min para Max. Se estiver acompanhando os hECTs em toda a sua faixa de captura, teste diferentes frequências de estimulação para encontrar a menor frequência na qual uma relação estímulo:pico de 1:1 é alcançada e continue a aumentar a frequência até que essa razão seja perdida. Meça a função espontânea escolhendo uma faixa de frequência arbitrária (por exemplo, 0,01 Hz a 0,01 Hz) e mantendo a saída do estimulador de pulso quadrado desligada.
- Nas caixas à direita, escolha o (s) tempo(s) de configuração desejado(s) (um intervalo de tempo após a frequência ter sido definida, mas os dados não são registrados) para permitir que o hECT se ajuste à nova frequência de ritmo. Especifique o (s) tempo(s) de gravação e a tensão de estimulação (V). Inicie o programa clicando no botão Start Program (Iniciar programa ) (Figura 7M).
Observação : os resultados são salvos automaticamente no diretório selecionado. Após cada gravação, observe que o script exibe a transformação de Fourier dos dados (Figura 7N), onde os picos correspondem à frequência de batimento detectada. - Se desejar, execute o programa "Excitation Threshold Finding" para encontrar a tensão mínima necessária para estimular o hECT em vista (Figura 7O). Se desejar, calcule as deflexões máxima e mínima dos postes (Figura 7P).
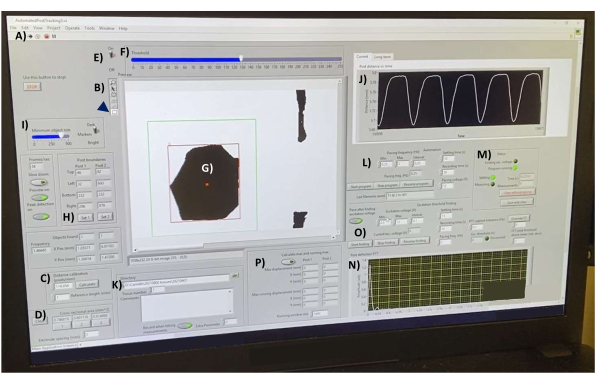
Figura 7: Interface de aquisição de dados pós-deflexão. (A) Botão para executar o software. (B) Barra de ferramentas contendo as ferramentas de linha e retângulo para as medidas de comprimento e seleção de objetos, respectivamente. (C) Controles de calibração à distância. (D) Ferramentas para medir a área de secção transversa da hECT em três pontos diferentes. (E) Interruptor de limiar e (F) controle deslizante para converter o feed de vídeo em imagens de alto contraste em tempo real. (G) Um SPoT visível na janela de visualização. (H) Ferramentas para selecionar os SPoTs. (I) Controle deslizante para filtrar os objetos por tamanho. (J) Gráfico mostrando a distância medida entre os objetos rastreados em tempo real. (K) Opções para selecionar o diretório para salvar os arquivos de saída. (L) Opções para definir a faixa de frequência, o intervalo de frequência, o tempo de gravação e o tempo de ajuste entre as gravações para o programa de pós-rastreamento (M). (N) Saída gráfica da transformação de Fourier da curva de deflexão do último registro salvo. (O) Programa para encontrar a tensão mínima necessária para estimular as hECTs. (P) Programa de cálculo das deflexões máxima e mínima dos postes. Abreviações: hECT = tecido cardíaco humano; SPoT = rastreador de post estável. Clique aqui para ver uma versão maior desta figura.
6. Medições em rack PDMS
- Distâncias descarregadas
- Antes da fabricação do hECT, monte o par desejado de racks PDMS em um quadro. Use a configuração de estimulação e o software descritos na etapa 5.1 para as medidas funcionais. Selecione os SPoTs estacionários nas extremidades dos posts.
- Ajustar a fonte de luz e/ou o limiar, se necessário, para reduzir o ruído para <2 μm. Registre o valor y médio ao vivo indicado no gráfico em uma planilha.
- Alturas dos postes e alturas do hECT
- A partir da configuração de ritmo descrita na etapa 5.2, remova o espelho angulado e o estágio aquecido. Coloque o biorreator diretamente no macaco de laboratório para uma visão lateral do biorreator.
- Abra o software da câmera. Ajuste o tempo de exposição e a posição da fonte de luz para otimizar a uniformidade das condições de iluminação em todo o campo de visão e maximizar a visibilidade dos postes.
- Abra o software de aquisição de dados e, em seguida, abra o arquivo "PostMeasurement_PB3.vi" (Arquivo Suplementar 5). Depois de carregar, clique na seta branca no lado esquerdo da barra de ferramentas para inicializar o programa.
- Calibrar o software usando um hemocitômetro de vidro. Clique na ferramenta de linha na barra de ferramentas vertical à esquerda da janela de visualização e desenhe uma linha em 1 mm das marcas do hemocitômetro. Na caixa Calibração de distância (pixels/mm) no canto inferior esquerdo da tela, defina o Comprimento de referência (mm) como 1 e clique no botão Calcular .
- Abaixo dos campos de calibração, defina o número de tecido desejado (para identificação) no campo Número de tecido . Focalize a câmera no poste esquerdo do hECT e selecione Esquerda na caixa Lado da publicação .
- Use a ferramenta de linha para desenhar uma linha da base do post (superior) até a ponta do SPoTs (inferior) e registre clicando em Medir Post Ht.
- Desenhe uma linha da base do poste até a borda mais distante do hECT e registre clicando em Medir Tissue Top Ht. Desenhe uma linha da base do poste até a borda próxima do hECT e registre clicando em Medir Base de Tecido Ht.
- Neste ponto, gire o biorreator para medir a altura do poste direito. Selecione a opção de postagem correta para registrar as mesmas medidas. Clique no botão Adicionar para preencher a planilha com os valores medidos e calcular automaticamente a altura média do hECT, que será usado na etapa 7.
- Quando terminar de gravar as alturas do tecido, clique no botão Salvar para salvar os valores em um arquivo de texto.
7. Processamento de dados funcionais usando scripts de análise personalizados
- Em um editor de planilhas, preencha o arquivo de resumo usando o modelo (Arquivo Suplementar 6). Utilizar os valores de comprimento pós e altura média do tecido adquiridos nos passos 6.2. Certifique-se de que todos os hECTs que têm dados na pasta estejam representados no arquivo de resumo. Nomeie o arquivo como "resumo #.csv", onde # refere-se ao número de dias no experimento.
NOTA: Os dados funcionais do hECT devem estar em pastas separadas de acordo com o dia do experimento. - Verifique se a pasta que contém os scripts AnalyzeLogsGUI (Arquivo Suplementar 7) e a pasta com as gravações hECT foram adicionadas ao caminho.
- Abra o software de análise de dados. À esquerda da barra de diretórios, clique no botão Procurar pasta para navegar até a pasta pai que contém a pasta AnalyzeLogsGUI e os dados funcionais hECT. Na barra lateral Janela atual , clique com o botão direito do mouse nessas pastas para Adicionar ao caminho | Adicione pastas e subpastas selecionadas.
- Abra o arquivo "AnalyzeLogsGui_SC.m". Na guia Editor, pressione o botão Executar e aguarde até que a interface gráfica do usuário (GUI) apareça em uma nova janela.
- Na caixa Log Selection (Figura 8AI), clique no botão Select Directory e navegue até a pasta que inclui os dados funcionais do hECT. Selecione o hECT desejado a ser processado no menu suspenso Tecido .
- Na caixa Entradas de Dados (Figura 8AII), insira a distância descarregada entre os postes registrados a partir dos passos 6.1 no campo Distância Diastólica. Entrada 0,25 no campo Raio de Post (mm).
- Na caixa Restrições de análise (Figura 8AIII), escolha as frequências a serem omitidas da análise ou selecione as frequências específicas a serem incluídas (separadas por vírgulas). A hora de início e a hora de término são definidas como 0 e −1, respectivamente, por padrão para processar toda a duração das gravações. Altere esses valores para cortar as gravações, se necessário.
- Altere os parâmetros de filtro (Figura 8AIV) de Ordem Polinomial e Tamanho do Quadro para alterar o nível de suavização durante o processo de filtragem e o controle deslizante Limite de Detecção de Pico para definir o tamanho mínimo de pico que será reconhecido pelos scripts.
Observação : o script contém a opção de remoção de pico , que corta picos altos causados por artefatos; No entanto, isso não é recomendado, pois altera a forma das contrações. Em vez disso, remova os artefatos cortando a gravação (Figura 8AIII). - Use opções adicionais (Figura 8AV) para saída de análise de dados adicionais: Análise pós-deflexão para executar um algoritmo de detecção de pico adicional, Autoscale y-axis em gráficos de zoom para ajustar automaticamente os eixos na curva de força de contração (Figura 8B), Salvar curvas de traço de força para salvar cada figura de força de contração em um arquivo .fig e Salvar dados de tempo de força para salvar as coordenadas x e y dos dados filtrados plotados na figura da curva de força do Twitch.
- Clique em Executar análise para gerar um arquivo de .txt que contém os atributos da curva de força de contração (Arquivo Suplementar 8) em média em toda a gravação.
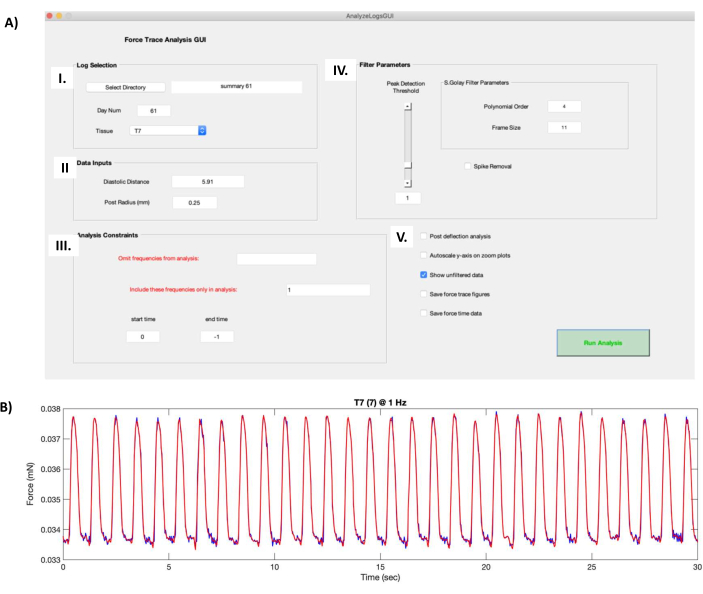
Figura 8: Cálculos da curva de força do Twitch. (A) Executar o arquivo "AnalyzeLogsGUI.m" no software de processamento de dados abre a janela GUI. (I) A caixa Seleção de log permite que o usuário selecione o diretório para a pasta que contém os dados funcionais do hECT. O campo Número do Dia é preenchido automaticamente a partir do título do arquivo de resumo criado na etapa de protocolo 7.1. O hECT a ser processado é selecionado usando o menu suspenso Tecido . (II) A caixa Entradas de Dados contém informações sobre o par de postes PDMS que suportam o hECT, como a distância descarregada (obtida na etapa de protocolo 6.1) e o raio do poste (0,25 mm). (III) A caixa Restrições de Análise permite ao usuário escolher as frequências a serem omitidas ou incluídas e aparar as gravações. (IV) A caixa de parâmetros do filtro contém as opções para escolher como a curva de força de contração bruta é filtrada. A ordem polinomial e o tamanho do quadro alteram o nível de suavização durante o processo de filtragem. O controle deslizante Limite de Detecção de Pico decide o tamanho mínimo de pico que será reconhecido pelos scripts. A opção Remoção de Spike clipa picos altos causados por artefatos. (V) As opções adicionais incluem a análise pós-deflexão, que executa um algoritmo adicional de detecção de picos, o eixo y Autoscale em gráficos de zoom, que atua na curva de força de contração, as curvas Save force-trace, que salva os números de força de contração, e Save force-time data, que salva os dados plotados de força de contração. (B) Exemplo da curva de força de contração de uma gravação de 30 s de um hECT cadenciado a 1 Hz produzido pela captura de tela GUI do painel A. A curva de força de contração vermelha mostra a força filtrada produzida pelos parâmetros em AIV, sobreposta à curva de força de contração bruta (curva azul escura, aparece quando a opção Mostrar dados não filtrados em AV é selecionada). Abreviações: hECT = tecido cardíaco humano; GUI = interface gráfica do usuário; PDMS = polidimetilsiloxano. Clique aqui para ver uma versão maior desta figura.
Resultados
Seguindo o protocolo acima, os cardiomiócitos foram gerados a partir de uma linhagem iPSC saudável usada previamente por nosso grupo 9,15 e fabricados em hECTs após 8-61 dias em cultura. A Figura 9A mostra imagens representativas de hECTs vistas de baixo, que foram criadas sem SPoTs (superior) e com SPoTs (inferior). Medidas funcionais foram realizadas à temperatura ambiente (23 °C) e fisiológica (36 °C) entre 37 dias e 52 dia...
Discussão
Existem inúmeros modelos lineares de tecido cardíaco modificados publicados na literatura, alguns dos quais estão descritos na Tabela 1. Alguns modelos envolvem a medida direta da força tecidual, mas estes tipicamente requerem a transferência do construto para um banho muscular separado38. A maioria dos modelos é desenhada com os tecidos ancorados permanentemente em ambas as extremidades, mais comumente aos pinos do PDMS1,2,3,4,5,6,7,8,9,10,11,12,13,14,15,16
Divulgações
K.D.C. é cofundador e diretor científico da Novoheart e detém participação acionária na holding Medera Biopharmaceutical. A Novoheart não contribuiu para o financiamento, planejamento ou execução deste estudo; no entanto, os resultados do estudo poderiam potencialmente ter um impacto financeiro sobre Novoheart e Medera. Os demais autores declaram não ter interesses concorrentes.
Agradecimentos
Os autores agradecem ao Dr. Timothy Cashman por trabalhos anteriores sobre este método. Este estudo foi financiado pelo National Institutes of Health (NIH) (R01-HL132226 e K01 HL133424) e pelo Leducq Foundation International Networks of Excellence Program (CURE-PLaN).
Materiais
| Name | Company | Catalog Number | Comments |
| 0.25 mm diamete 304 Stainless Steel Wire | McMaster Carr | 6517K61 | |
| 0.25% trypsin-EDTA | Gibco | 25200056 | |
| 1.7 mL Microtubes | Axygen | MCT-175-C | |
| 10 cm dishes (20 mm tall) | Corning | 353003 | |
| 10 mL Serological Pipette | Drummond | 6-000-010 | |
| 10 N NaOH | Fisher Scientific | SS225-1 | dilute 1:10 in sterile distilled water |
| 10X Modified Eagle Medium | Sigma Aldrich | M0275 | |
| 20 - 200 μL Micropipette | Eppendorf | 3123000055 | |
| 200 μL MicroPipette Tips | VWR | 76322-150 | |
| 5 mL Serological Pipette | Drummond | 6-000-005 | |
| 50 mL Conical Centrifuge Tubes | Falcon | 352070 | |
| 6 cm Petri Dish | Corning | 353002 | |
| 6 Watt LED Dual Gooseneck Illuminator | AmScope | LED-6W | |
| 6-Well Plates | Corning | 353046 | |
| 90 degree angle mirror | Edmund Optics | 45-594 | |
| Acrylic bonding glue | SCIGRIP | #4 | |
| Adjustable 10 cm x 10 cm jack | Fisher Scientific | 14-673-50 | |
| Aluminum 6061 | McMaster Carr | 9008K82 | |
| A-Plan 10X Objective Lens | ZEISS | 1020-863 | |
| Autoclave Bags | Propper | 21002 | |
| B-27 supplement | ThermoFisher | 17504044 | |
| B-27 supplement (without insulin) | ThermoFisher | A1895601 | |
| Benchtop Centrifuge | Eppendorf | 5810 R | |
| Black ABS | Ultimaker | 2.85 mm wide | |
| Bovine Collagen I | Gibco | A1064401 | |
| CHIR99021 | Tocris | 4423 | |
| Class II Biosafety Cabinet | Labconco | 3430009 | |
| Clear Acrylic Sheeting | estreetplastics | 1002502436 | 6.25 mm thick |
| CNC Vertical Mill | Haas | VF-1 | |
| Conductive Graphite Bars | McMaster Carr | 1763T33 | |
| Dissection microscope | Olympus | SZ61 | |
| Dulbecco's Modified Eagle Medium/Ham's F-12 Nutrient Mix | ThermoFisher | 11330032 | |
| Ethanol | Fisher Scientific | A4094 | Dilute to 70% in water |
| EVE Automated Cell counter | NanoEntek | E1000 | |
| EVE Cell Counting Slide | NanoEntek | EVS-050 | |
| Fetal Bovine Serum | Life Technologies | 10438026 | |
| Fine Curved Forceps | Fine Science Tools | 11253-25 | |
| Forma Series II Water Jacketed CO2 Incubator | Thermo Electron Corporation | 3110 | AKA "incubator". With HEPA class 100 filter |
| Fusion360 software | Autodesk | AKA "CAD software" | |
| Glass Hemocytometer | Reichert | 1475 | 0.1 mm deep |
| HEPES | Sigma Aldrich | H3784 | |
| hESC qualified matrigel | Corning | 354277 | AKA "basement membrane matrix". Store in frozen aliquots |
| High Speed CCD Camera | PixelLINK | P7410 | |
| Inverted Microscope | Carl Zeiss Werk | Axiovert 40 CFL | 10X phase contrast objective |
| IWR-1 | Selleck Chem | S7086 | |
| LabView Software | National Instruments | 2016 | |
| Laminar flow clean bench | NuAire | NU-201-330 | necessary for hECT functional analysis |
| Laptop | AsusTek | Strix | Intel Core i& processor ,CPU 2.8GHz, 16GB RAM |
| Laser Cutting Machine | Epilog | Helix 24 | |
| Magnification headset | ExcelBlades | 70020 | Recommended for steps requiring fine manipulations |
| Matlab | Mathworks | Version 2019b or later | AKA "data analysis software" |
| Micro Vannas Scissors, 3 mm blade | WPI Instruments | 501839 | |
| Microscope Boom Stand | Olympus | SZ2-STU1 | |
| Penicillin-Streptomycin stock solution | ThermoFisher | 15140122 | 10,000 IU/ml penicillin; 10,000 μg/ml streptomycin |
| Phosphate-buffered saline without divalent cations | Sigma Aldrich | P3813 | Diluted in distilled water to 1X and 10X concentrations |
| Pipette Controller | Drummond | 4-000-100 | |
| PixelLINK Capture OEM | PixelLINK | 10.2.1.6 | AKA "Camera Software" |
| Polysulfone | McMaster Carr | 86735K73 | translucent amber color |
| Polytetrafluoroethylene (PTFE) | McMaster Carr | 8545K176 | Black, molded |
| ReLeSR | Stem Cell Technologies | 5872 | AKA "iPSC dissociation media" |
| Rosewell Park Memorial Institute 1640 Media | ThermoFisher | 11875135 | |
| Silicone Sheeting | SMI manufacturing | glossy, 0.02 in thickness, durometer 40 | |
| Size 10/0 Blue, Green, Red, and Yellow Glass Seed Beads | Michael's | color should withstand autoclaving | |
| Spatula | Fisher Scientific | 14-373 | used for mixing PDMS |
| Square Pulse Stimulator | Astro-Med / Grass Technologies | S88X | |
| Stainless Steel Razoblades | GEM | 62-0179-CTN | preferred over non-stainless steel due to lower hardness |
| Stemflex | ThermoFisher | A3349401 | AKA "iPSC culture media" |
| Sterile distilled water | ThermoFisher | 5230 | |
| Sylgard 170 - Silicone Elastomer Encapsulant Black 0.9 kg Kit | Dow | DOWSIL 170 2LB KIT | AKA black Polydimethylsiloxane (black PDMS) |
| Sylgard 184 - Silicone Elastomer Clear 1 lb Kit | Dow | DC 184 SYLGARD 0.5KG 1.1LB KIT | AKA Polydimethylsiloxane (PDMS) |
| Temperature-controlled heated stage | Okolab | H401-HG-SMU | Set height to 10 cm |
| Thermoplastic 3D printer | Ultimaker | Ultimaker 3 | |
| Thiazovivin | Selleck Chem | S1459 | |
| Trypan Blue | NanoEntek | EBT-001 | |
| Vacuum Chamber | Bel-Art Parts | F42027-0000 | |
| Variable Speed Mini Band Saw | Micro-Mark | 82203 | |
| Variable Speed Miniature Drill Press | Micro-Mark | 82959 | |
| Vibration Isolation Table | Labconco | 3618000 | |
| Weighing Boats | VWR | 10803-140 | |
| Talon Cylinder Bench Clamp | VWR | 97035-528 | AKA screw clamp |
Referências
- Serrao, G. W., et al. Myocyte-depleted engineered cardiac tissues support therapeutic potential of mesenchymal stem cells. Tissue Engineering. Part A. 18 (13-14), 1322-1333 (2012).
- Turnbull, I. C., et al. Advancing functional engineered cardiac tissues toward a preclinical model of human myocardium. FASEB Journal. 28 (2), 644-654 (2014).
- Cashman, T. J., et al. Construction of defined human engineered cardiac tissues to study mechanisms of cardiac cell therapy. Journal of Visualized Experiments. (109), e53447 (2016).
- Stillitano, F., et al. Genomic correction of familial cardiomyopathy in human engineered cardiac tissues. European Heart Journal. 37 (43), 3282-3284 (2016).
- Mayourian, J., et al. Experimental and computational insight into human mesenchymal stem cell paracrine signaling and heterocellular coupling effects on cardiac contractility and arrhythmogenicity. Circulation Research. 121 (4), 411-423 (2017).
- Mayourian, J., et al. therapeutic paracrine modulation of cardiac excitation-contraction coupling. Circulation Research. 122 (1), 167-183 (2018).
- Mayourian, J., et al. Exosomal microRNA-21-5p mediates mesenchymal stem cell paracrine effects on human cardiac tissue contractility. Circulation Research. 7 (122), 933-944 (2018).
- Turnbull, I. C., et al. Cardiac tissue engineering models of inherited and acquired cardiomyopathies. Methods in Molecular Biology. 1816, 145-159 (2018).
- Murphy, J. F., et al. Adult human cardiac stem cell supplementation effectively increases contractile function and maturation in human engineered cardiac tissues. Stem Cell Research & Therapy. 10 (1), 373 (2019).
- Breckwoldt, K., et al. Differentiation of cardiomyocytes and generation of human engineered heart tissue. Nature Protocols. 12 (6), 1177-1197 (2017).
- Huang, C. Y., et al. Enhancement of human iPSC-derived cardiomyocyte maturation by chemical conditioning in a 3D environment. Journal of Molecular and Cellular Cardiology. 138, 1-11 (2020).
- Ramade, A., Legant, W. R., Picart, C., Chen, C. S., Boudou, T. Microfabrication of a platform to measure and manipulate the mechanics of engineered microtissues. Methods in Cell Biology. 121, 191-211 (2014).
- Ronaldson-Bouchard, K., et al. Engineering of human cardiac muscle electromechanically matured to an adult-like phenotype. Nature Protocols. 14 (10), 2781-2817 (2019).
- Tamargo, M. A., et al. milliPillar: A platform for the generation and real-time assessment of human engineered cardiac tissues. ACS Biomaterials Science & Engineering. 7 (11), 5215-5229 (2021).
- Ceholski, D. K., et al. CXCR4 and CXCR7 play distinct roles in cardiac lineage specification and pharmacologic β-adrenergic response. Stem Cell Research. 23, 77-86 (2017).
- Bliley, J. M., et al. Dynamic loading of human engineered heart tissue enhances contractile function and drives a desmosome-linked disease phenotype. Science Translational Medicine. 13 (603), (2021).
- Ribeiro, M. C., et al. A new versatile platform for assessment of improved cardiac performance in human-engineered heart tissues. Journal of Personalized Medicine. 12 (2), 214 (2022).
- Ronaldson-Bouchard, K., et al. Advanced maturation of human cardiac tissue grown from pluripotent stem cells. Nature. 556 (7700), 239-243 (2018).
- Mannhardt, I., et al. Human engineered heart tissue: Analysis of contractile force. Stem Cell Reports. 7 (1), 29-42 (2016).
- Mannhardt, I., et al. Blinded contractility analysis in hiPSC-cardiomyocytes in engineered heart tissue format: Comparison with human atrial trabeculae. Toxicological Sciences. 158 (1), 164-175 (2017).
- Saleem, U., et al. Force and calcium transients analysis in human engineered heart tissues reveals positive force-frequency relation at physiological frequency. Stem Cell Reports. 14 (2), 312-324 (2020).
- Thavandiran, N., et al. Functional arrays of human pluripotent stem cell-derived cardiac microtissues. Scientific Reports. 10 (1), 6919 (2020).
- Bielawski, K. S., Leonard, A., Bhandari, S., Murry, C. E., Sniadecki, N. J. Real-time force and frequency analysis of engineered human heart tissue derived from induced pluripotent stem cells using magnetic sensing. Tissue Engineering. Part C, Methods. 22 (10), 932-940 (2016).
- Leonard, A., et al. Afterload promotes maturation of human induced pluripotent stem cell derived cardiomyocytes in engineered heart tissues. Journal of Molecular and Cellular Cardiology. 118, 147-158 (2018).
- Bose, P., Huang, C. Y., Eyckmans, J., Chen, C. S., Reich, D. H. Fabrication and mechanical properties measurements of 3D microtissues for the study of cell-matrix interactions. Methods in Molecular Biology. 1722, 303-328 (2018).
- Zhang, W., et al. Maturation of human embryonic stem cell-derived cardiomyocytes (hESC-CMs) in 3D collagen matrix: Effects of niche cell supplementation and mechanical stimulation. Acta Biomaterialia. 49, 204-217 (2017).
- van Neste, C. Advances in bioreactor design and multi-dimensional analysis for assessing maturation phenotype of human engineered cardiac tissues. PhD thesis. Icahn School of Medicine at Mount Sinai. , (2022).
- Sala, L., et al. MUSCLEMOTION: A versatile open software tool to quantify cardiomyocyte and cardiac muscle contraction in vitro and in vivo. Circulation Research. 122 (3), e5-e16 (2018).
- Salazar, B. H., Cashion, A. T., Dennis, R. G., Birla, R. K. Development of a cyclic strain bioreactor for mechanical enhancement and assessment of bioengineered myocardial constructs. Cardiovascular Engineering and Technology. 6 (4), 533-545 (2015).
- Putame, G., et al. Application of 3D printing technology for design and manufacturing of customized components for a mechanical stretching bioreactor. Journal of Healthcare Engineering. 2019, 3957931 (2019).
- Akintewe, O. O., Roberts, E. G., Rim, N. -. G., Ferguson, M. A. H., Wong, J. Y. Design approaches to myocardial and vascular tissue engineering. Annual Review of Biomedical Engineering. 19, 389-414 (2017).
- Chen, G., et al. Phospholamban as a crucial determinant of the inotropic response of human pluripotent stem cell-derived ventricular cardiomyocytes and engineered 3-dimensional tissue constructs. Circulation. Arrhythmia and Electrophysiology. 8 (1), 193-202 (2015).
- Giacomelli, E., et al. Human-iPSC-derived cardiac stromal cells enhance maturation in 3D cardiac microtissues and reveal non-cardiomyocyte contributions to heart disease. Cell Stem Cell. 26 (6), 862-879 (2020).
- Beauchamp, P., et al. 3D co-culture of hiPSC-derived cardiomyocytes with cardiac fibroblasts improves tissue-like features of cardiac spheroids. Frontiers in Molecular Biosciences. 7, 14 (2020).
- Campostrini, G., et al. functional analysis and applications of isogenic three-dimensional self-aggregating cardiac microtissues from human pluripotent stem cells. Nature Protocols. 16 (4), 2213-2256 (2021).
- Swiatlowska, P., Iskratsch, T. Tools for studying and modulating (cardiac muscle) cell mechanics and mechanosensing across the scales. Biophysical Reviews. 13 (5), 611-623 (2021).
- Zhao, Y., et al. Engineering microenvironment for human cardiac tissue assembly in heart-on-a-chip platform. Matrix Biology. 85-86, 189-204 (2020).
- Fujiwara, Y., Deguchi, K., Miki, K., Nishimoto, T., Yoshida, Y. A method for contraction force measurement of hiPSC-derived engineered cardiac tissues. Methods in Molecular Biology. 2320, 171-180 (2021).
- Wang, E. Y., et al. Biowire model of interstitial and focal cardiac fibrosis. ACS Central Science. 5 (7), 1146-1158 (2019).
- Zhao, Y., et al. A platform for generation of chamber-specific cardiac tissues and disease modeling. Cell. 176 (4), 913-927 (2019).
- Lee, E. K., et al. Machine learning of human pluripotent stem cell-derived engineered cardiac tissue contractility for automated drug classification. Stem Cell Reports. 9 (5), 1560-1572 (2017).
- Batalov, I., Feinberg, A. W. Differentiation of cardiomyocytes from human pluripotent stem cells using monolayer culture. Biomarker Insights. 10, 71-76 (2015).
- Lian, X., et al. Robust cardiomyocyte differentiation from human pluripotent stem cells via temporal modulation of canonical Wnt signaling. Proceedings of the National Academy of Sciences of the United States of America. 109 (27), E1848-E1857 (2012).
- Penefsky, Z. J., Buckley, N. M., Litwak, R. S. Effect of temperature and calcium on force-frequency relationships in mammalian ventricular myocardium. Pflugers Archiv. 332 (4), 271-282 (1972).
- Bers, D. M. . Excitation-Contraction Coupling and Cardiac Contractile Force. , (2001).
- Kanaya, N., Gable, B., Wickley, P. J., Murray, P. A., Damron, D. S. Experimental conditions are important determinants of cardiac inotropic effects of propofol. Anesthesiology. 103 (5), 1026-1034 (2005).
- Galende, E., et al. Amniotic fluid cells are more efficiently reprogrammed to pluripotency than adult cells. Cellular Reprogramming. 12 (2), 117-125 (2010).
- Wacker-Gussmann, A., Strasburger, J. F., Cuneo, B. F., Wakai, R. T. Diagnosis and treatment of fetal arrhythmia. American Journal of Perinatology. 31 (7), 617-628 (2014).
- Federmann, M., Hess, O. M. Differentiation between systolic and diastolic dysfunction. European Heart Journal. 15, 2-6 (1994).
- Knight, W. E., et al. Maturation of pluripotent stem cell-derived cardiomyocytes enables modeling of human hypertrophic cardiomyopathy. Stem Cell Reports. 16 (3), 519-533 (2021).
- Ma, Z., et al. Contractile deficits in engineered cardiac microtissues as a result of MYBPC3 deficiency and mechanical overload. Nature Biomedical Engineering. 2 (12), 955-967 (2018).
- de Lange, W. J., et al. Human iPSC-engineered cardiac tissue platform faithfully models important cardiac physiology. American Journal of Physiology. Heart and Circulatory Physiology. 320 (4), H1670-H1686 (2021).
- Hiranandani, N., Varian, K. D., Monasky, M. M., Janssen, P. M. L. Frequency-dependent contractile response of isolated cardiac trabeculae under hypo-, normo-, and hyperthermic conditions. Journal of Applied Physiology. 100 (5), 1727-1732 (2006).
- Puglisi, J. L., Bassani, R. A., Bassani, J. W., Amin, J. N., Bers, D. M. Temperature and relative contributions of Ca transport systems in cardiac myocyte relaxation. The American Journal of Physiology. 270 (5), H1772-H1778 (1996).
- Puglisi, J. L., Yuan, W., Bassani, J. W., Bers, D. M. Ca(2+) influx through Ca(2+) channels in rabbit ventricular myocytes during action potential clamp: Influence of temperature. Circulation Research. 85 (6), e7-e16 (1999).
- Li, R. A., et al. Bioengineering an electro-mechanically functional miniature ventricular heart chamber from human pluripotent stem cells. Biomaterials. 163, 116-127 (2018).
- Sharma, A., et al. Biomanufacturing in low Earth orbit for regenerative medicine. Stem Cell Reports. 17 (1), 1-13 (2022).
- Strauss, D. G., Wu, W. W., Li, Z., Koerner, J., Garnett, C. Translational models and tools to reduce clinical trials and improve regulatory decision making for QTc and proarrhythmia risk (ICH E14/S7B updates). Clinical Pharmacology & Therapeutics. 109 (2), 319-333 (2021).
- Gintant, G., et al. Repolarization studies using human stem cell-derived cardiomyocytes: Validation studies and best practice recommendations. Regulatory Toxicology and Pharmacology. 117, 104756 (2020).
Erratum
Formal Correction: Erratum: Designing a Bioreactor to Improve Data Acquisition and Model Throughput of Engineered Cardiac Tissues
Posted by JoVE Editors on 1/10/2024. Citeable Link.
An erratum was issued for: Designing a Bioreactor to Improve Data Acquisition and Model the Throughput of Engineered Cardiac Tissues. The title was corrected from:
Designing a Bioreactor to Improve Data Acquisition and Model the Throughput of Engineered Cardiac Tissues
to:
Designing a Bioreactor to Improve Data Acquisition and Model Throughput of Engineered Cardiac Tissues
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados