Method Article
Uso de métodos minimamente invasivos para avaliar a utilização de combustível e ritmos circadianos em idosos
Neste Artigo
Resumo
Um protocolo novo e minimamente invasivo é apresentado para avaliar o impacto sinérgico da utilização de combustível e ritmos circadianos em indivíduos idosos, utilizando células mononucleares do sangue periférico.
Resumo
O envelhecimento está associado a múltiplas alterações fisiológicas que contribuem de forma sinérgica e independente para a incapacidade física e o risco de doenças crônicas. Embora a etiologia da incapacidade física relacionada à idade seja complexa e multifatorial, o declínio da função mitocondrial parece coincidir com a progressão do declínio funcional em muitos idosos. A razão pela qual há uma diminuição na função mitocondrial com o envelhecimento permanece indefinida, mas a ciência emergente indica que tanto o metabolismo do combustível quanto os ritmos circadianos podem influenciar a função mitocondrial.
Estudos recentes estabeleceram que os ritmos circadianos se tornam perturbados com o envelhecimento e que os ritmos circadianos interrompidos têm consequências patológicas que afetam a função mitocondrial e se sobrepõem a muitas doenças crônicas associadas à idade. Os métodos quantitativos atuais para avaliação direta da função mitocondrial são invasivos e normalmente requerem uma biópsia muscular, o que pode representar dificuldades no recrutamento dos participantes e na adesão ao estudo, dados os níveis percebidos de dor e risco potenciais. Assim, um protocolo inovador e relativamente não invasivo para avaliar mudanças na função mitocondrial no nível celular e nos padrões circadianos em adultos mais velhos foi adaptado. Especificamente, um analisador de fluxo metabólico em tempo real é usado para avaliar a função bioenergética mitocondrial dos glóbulos brancos sob disponibilidade diferencial de substrato.
A expressão dos genes do relógio circadiano nos glóbulos brancos para se correlacionar com a bioenergética mitocondrial e os resultados do ritmo circadiano também são analisados. Acredita-se que essas abordagens metodológicas inovadoras ajudarão futuros ensaios clínicos, fornecendo métodos minimamente invasivos para estudar a preferência de substrato mitocondrial e os ritmos circadianos em adultos mais velhos.
Introdução
Os avanços no século passado levaram a um aumento na expectativa de vida e na população de adultos idosos. Olhando para o futuro, a porcentagem de adultos com 65 anos ou mais deve aumentar 5% de 2020 a 2050 nos Estados Unidos1. Esse aumento na expectativa de vida não implica em aumento do tempo de saúde - o período da vida associado ao funcionamento independente. A realidade é que o envelhecimento é acompanhado por inúmeras alterações biológicas que afetam o metabolismo e a fisiologia celular, produzindo declínios graduais no funcionamento cognitivo e físico 2,3. À medida que a expectativa de vida humana continua a aumentar, há uma necessidade maior de preservar a capacidade funcional e a independência aos4 anos de idade.
Sabe-se há muito tempo que o declínio da função física e da independência com a idade é multifatorial, embora esteja frequentemente associado ao aparecimento de doenças crônicas e eventos desencadeantes agudos5. Por outro lado, foi demonstrado que esses declínios no desempenho físico e nas características musculares estão associados ao desenvolvimento de incapacidade com a idade, sem conexão clara com uma única doença6. Com as dificuldades em conhecer a etiologia exata da doença crônica e da incapacidade física, acredita-se que os prejuízos na função mitocondrial coincidam com o início e a progressão da doença crônica e a perda da função física em adultos idosos 7,8.
As mitocôndrias fornecem a maior parte do trifosfato de adenosina (ATP), necessário para muitos processos celulares9. Tecidos altamente oxidativos dependem das mitocôndrias para produção adequada de energia; com o envelhecimento, a capacidade oxidativa e o declínio da síntese mitocondrial de ATP. Esse declínio se deve em parte ao dano oxidativo ao DNA mitocondrial (mtDNA), que resulta em um acúmulo incremental de mutações e deleções do mtDNA10. O acúmulo de mutações e deleções do mtDNA causa uma diminuição na formação de proteínas funcionais da cadeia de transporte de elétrons, causando assim uma redução da capacidade das células de produzir ATP. O declínio da função mitocondrial associado à idade é mais notável em tecidos altamente oxidativos, como o coração e o músculo esquelético11. Estudos demonstraram que as mitocôndrias do músculo gastrocnêmio em amostras de ratos mais velhos exibem uma redução de aproximadamente 50% na produção e conteúdo de ATP em comparação com amostras mais jovens12. Além disso, foi demonstrado que a capacidade de produção de ATP mitocondrial no músculo esquelético humano diminui em aproximadamente 8% por década de vida13. Esses achados sugerem que declínios relacionados à idade na função mitocondrial podem contribuir para a diminuição da produção de energia nos organismos.
Acredita-se que um regulador chave da atividade mitocondrial seja o coativador do receptor γ ativado por proliferador de peroxissomo (PPARγ) 1 (PGC-1α) 14 . A deterioração da atividade do PGC-1α ou o declínio de sua abundância leva à redução da atividade oxidativa mitocondrial e, consequentemente, à diminuição da produção de energia. Além disso, um declínio na qualidade mitocondrial pode afetar a qualidade do músculo esquelético e, subsequentemente, levar ao desenvolvimento ou exacerbação de sarcopenia, dinapenia e declínio da capacidade funcional15,16. Evidências para o declínio concomitante relacionado à idade na função mitocondrial e na qualidade do músculo esquelético sugerem uma conexão entre o comprometimento mitocondrial e a patogênese do declínio funcional17. Recentemente, isso foi confirmado em idosos funcionais residentes na comunidade, mostrando que reduções no metabolismo das mitocôndrias do músculo esquelético predizem o declínio da mobilidade nessa população18. Embora o mecanismo exato que leva ao declínio mitocondrial com a idade não seja claro, evidências recentes destacaram uma interação recíproca entre o relógio circadiano e a função mitocondrial, com consequências para a utilização de combustível mitocondrial e biogênese19.
Utilização de combustível
A função mitocondrial parece ser influenciada pelo metabolismo do combustível e pelo tipo de combustível utilizado no nível celular no tecido muscular esquelético11. Durante os períodos de esgotamento do combustível, especificamente o esgotamento de carboidratos em humanos, a preferência do combustível pela produção de energia (mitocondrial) muda. Em baixos níveis de glicose, a preferência por combustível muda da glicose para ácidos graxos e corpos cetônicos derivados de ácidos. Essa mudança metabólica é marcada pela regulação positiva do metabolismo lipídico nos adipócitos, seguida por um aumento da liberação de cetonas no sangue4. A mudança na utilização de combustível de glicose para cetonas com uma dieta cetogênica parece ter um efeito benéfico na produção de espécies reativas de oxigênio mitocondrial, defesa antioxidante, síntese de ATP e biogênese20.
A mudança metabólica do metabolismo de carboidratos para lipídios ocorre em períodos de baixa disponibilidade de nutrientes ambientais e quando os estoques de glicogênio estão esgotados. Quando essa mudança é iniciada, os triglicerídeos armazenados são decompostos em glicerol, um substrato para a gliconeogênese, e ácidos graxos livres, que são transportados para o fígado para serem oxidados via β-oxidação em acetil coenzima A (acetil CoA). Os corpos cetônicos são sintetizados, principalmente no fígado, por uma condensação em duas etapas de três moléculas de acetil CoA em β-hidroxi-β-metilglutaril-CoA, que são então processadas em corpos cetônicos, incluindo acetoacetato e 3-βeta hidroxibutirato21. Esses corpos cetônicos são distribuídos pelos tecidos por todo o corpo, com o maior consumo ocorrendo no coração, cérebro e músculo esquelético21. Com o envelhecimento, a oxidação de ácidos graxos mitocondriais fica prejudicada, impactando assim o interruptor metabólico22. Foi proposto que deficiências na utilização de combustível mitocondrial levam a mais disfunção mitocondrial, que por sua vez contribui para doenças relacionadas à idade e declínio funcional23.
Alterações no consumo de oxigênio mitocondrial de células mononucleares do sangue periférico (PBMCs) têm sido estudadas para avaliar os padrões associados à disfunção e vascularização. Hartman et al. realizaram um estudo que teve como objetivo determinar a correlação entre o consumo de oxigênio e a dilatação diversamente mediada, o que sugeriu uma ligação entre disfunção mitocondrial e disfunção vascular de células musculares lisas24. Em relação a outros órgãos, as PBMCs têm sido correlacionadas com maior funcionamento cognitivo e cerebral, conforme determinado pela respirometria25. Assim, a bioenergética e a capacidade respiratória do PBMC podem servir como potenciais biomarcadores para avaliar a capacidade funcional de órgãos ou tecidos em todo o corpo.
Ritmo circadiano
Outro fator importante que afeta a saúde mitocondrial é o ritmo circadiano. Os ritmos circadianos são oscilações de ~ 24 h no comportamento e na fisiologia que ocorrem na ausência de pistas ambientais26. Esses ritmos funcionam de forma preditiva para apoiar a homeostase do sistema e do tecido. O mecanismo subjacente aos ritmos circadianos é um ciclo de feedback de transcrição-tradução chamado relógio circadiano27. Foi demonstrado nos últimos 15 anos que o mecanismo do relógio circadiano existe em praticamente todas as células do corpo28. Além de manter o tempo, o mecanismo do relógio molecular também contribui para um programa diário de expressão gênica, conhecido como saída do relógio circadiano29. Os genes de saída do relógio são exclusivos para cada tipo de tecido e estão funcionalmente associados a vias importantes para o metabolismo celular, autofagia, reparo e homeostase. Evidências recentes mostraram que a saúde mitocondrial depende da função do relógio circadiano e influencia a função mitocondrial, incluindo biogênese mitocondrial, utilização de combustível e mitofagia30.
Evidências emergentes em estudos pré-clínicos e clínicos demonstraram que, ao longo do envelhecimento, há distúrbios nos ritmos circadianos31. Isso inclui interrupções nos ciclos normais de sono e vigília, uma amplitude diminuída nos ritmos da temperatura corporal central e uma capacidade atrasada de se ajustar às mudanças na fase31. Um estudo, por exemplo, desafiou o sistema circadiano de camundongos adultos e velhos (20+ meses), mudando o horário de luz em 6 h. Verificou-se que os camundongos velhos demoraram mais para reintroduzir seus padrões de atividade para o novo cronograma de luz32. Consistente com as mudanças no comportamento circadiano, a análise dos relógios teciduais descobriu que os relógios teciduais centrais e periféricos estavam prejudicados na coorte de envelhecimento.
Mais recentemente, vários grupos realizaram análises transcriptômicas do relógio circadiano e da saída do relógio em diferentes tecidos com33 anos. Os resultados desses estudos destacam que há uma reprogramação em larga escala da saída do relógio circadiano com a idade. Isso significa que, embora o relógio central mantenha uma função de temporização, os genes direcionados para a expressão diária são muito diferentes. Por exemplo, dois estudos coletaram biópsias musculares de seres humanos a cada 4 h por 24 h, os resultados determinando que o pico e o vale da expressão gênica do relógio são revertidos entre roedores noturnos e humanos diurnos34 , 35 , 36 . Isso indica que, quando a expressão do gene do relógio é comparada com base apenas na fase ativa versus fase de repouso (e não clara versus escura), os padrões de expressão do gene do relógio nos músculos são praticamente os mesmos entre as espécies. Propõe-se que essa mudança associada à idade na saída do relógio resulte em deficiências na regulação das vias que incluem as características conhecidas do envelhecimento, como função mitocondrial, danos e reparos no DNA e autofagia37.
Justificativa do estudo
A conexão entre a função mitocondrial e o declínio da função física está bem estabelecida. No entanto, a causa subjacente da disfunção mitocondrial continua sendo um assunto de debate. Pesquisas recentes sugerem que a utilização de combustível celular e os ritmos circadianos podem desempenhar um papel nesse processo. Os métodos tradicionais de avaliação da função mitocondrial, como a medição do consumo de oxigênio mitocondrial em uma amostra de biópsia muscular, são frequentemente percebidos como dolorosos e invasivos, o que pode desencorajar a participação, principalmente em populações com baixa massa muscular, como adultos frágeis e sarcopênicos38.
Dadas essas limitações, há necessidade de um método menos invasivo para avaliar as mudanças na utilização de combustível celular e no ritmo circadiano em idosos. Este estudo tem como objetivo avaliar um novo protocolo minimamente invasivo que pode ser usado para avaliar o metabolismo do combustível e o ritmo circadiano nessa população. Os resultados deste estudo contribuirão para uma melhor compreensão das alterações relacionadas com a idade e da resposta a intervenções médicas ou comportamentais, servindo de modelo para futuros estudos neste campo.
Protocolo
Os procedimentos envolvendo participantes humanos foram aprovados pelo comitê de ética em pesquisa (Florida Ethics Policy 1.0104) e pelo Conselho de Revisão Institucional da Universidade da Flórida.
1. Função mitocondrial
- Isolamento de células mononucleares do sangue periférico (PBMCs)
- Colete PBMCs usando tubos especiais de coleta de sangue de 8 mL (16 mm x 125 mm; contendo anticoagulante citrato de sódio 0,1 M ou anticoagulante de heparina sódica) (consulte a Tabela de Materiais).
NOTA: Os tubos de coleta de sangue contêm meios de separação de sangue compostos por um gel de poliéster tixotrópico e um meio gradiente (consulte a Tabela de Materiais) para melhorar a separação dos glóbulos brancos. - Processe os tubos de preparação de células de 8 mL dentro de 2 h após a coleta para obter resultados viáveis.
- Misture os tubos de coleta de sangue depois de enchê-los com sangue (por inversão suave ou em um misturador) e mantenha em temperatura ambiente (RT) por no máximo 2 h.
- Centrifugue os tubos de coleta de sangue em um rotor basculante de encaixe a 2.000 × g por 15 min (ou equivalente a 30.000 G-min; não exceda 2.000 × g) em RT.
NOTA: Esses tubos são mais altos do que um tubo de centrífuga regular de 15 mL; portanto, atenção adicional deve ser dada ao uso do rotor correto. - Aspire e descarte em um recipiente de resíduos de risco biológico ~ 80% da camada de plasma e colete a camada de células do tubo de coleta de sangue (usando uma pipeta de transferência) em um tubo de centrífuga de 15 mL.
NOTA: Ao usar um tubo de 15 mL, adicione 7 mL de camada de células e preencha até 14 mL com solução salina estéril tamponada com fosfato (PBS). A proporção de células para PBS deve ser de 1:1. Use vários tubos, se necessário. Misture delicadamente invertendo. - Centrifugar num rotor basculante a 900 × g durante 5 min (ou equivalente a 4.500 G-min) a RT.
- Sem perturbar o pellet celular, aspire a solução de PBS e descarte-a em um recipiente de resíduos de risco biológico.
- Ressuspenda o pellet celular em 1 mL de meio basal (BM; consulte a Tabela de Materiais) triturando suavemente. Adicione o BM a um total de 10 mL e misture delicadamente invertendo. Execute uma contagem de células.
- Centrifugar a suspensão da célula num rotor basculante a 900 × g durante 5 min à RT.
NOTA: Quando as células são usadas no mesmo dia para a avaliação bioenergética, prossiga para a etapa 1.1.10; Se as células estiverem congeladas para armazenamento, prossiga para a etapa 1.1.13. - Calcule o volume do meio de ensaio (AM; consulte a Tabela de Materiais) para ressuspensão do próximo pellet celular para atingir a concentração celular desejada.
NOTA: Por exemplo, para uma densidade de semeadura de 150.000 células/poço em 50 μL, a concentração desejada da suspensão celular é de 3 milhões de células/mL. - Sem perturbar o sedimento celular, aspirar o sobrenadante e descartá-lo para um recipiente de resíduos com risco biológico.
- Ressuspenda o pellet celular suavemente em 1 mL ou menos de AM. Adicione AM ao volume total calculado para a concentração de células desejada. As células estão agora prontas para serem semeadas em uma placa de cultura de células revestida (ver Tabela de Materiais).
NOTA: As etapas a seguir são para congelar as células para armazenamento (1.1.13-1.1.16) e não se aplicam ao procedimento no mesmo dia. - Para congelar células, aspirar o sobrenadante sem perturbar o sedimento celular e descartá-lo em um recipiente para resíduos de risco biológico.
- Ressuspenda suavemente as células em meios de congelamento (consulte a Tabela de Materiais) na concentração desejada (5-10 milhões de células / mL). Alíquota volumes desejados em criogeniais marcados.
- Congelar lentamente a suspensão da célula utilizando um recipiente de congelação (ver Tabela de Materiais) durante um mínimo de 4 h ou durante a noite a -80 °C.
- Após 4 h, ou na manhã seguinte, transfira os criogenianos para armazenamento de nitrogênio líquido, onde devem residir na fase de vapor.
- No dia anterior à medição, cubra o fundo do poço de uma microplaca de cultura de células (consulte a Tabela de Materiais) com adesivo celular (consulte a Tabela de Materiais). Prepare o adesivo de acordo com as instruções do fabricante, com uma concentração recomendada de 22,4 μg/μL, diluindo o volume apropriado de solução estoque adesiva em bicarbonato de sódio 0,1 M (pH 8,0). Aplique 25 μL de adesivo em cada poço da microplaca de cultura de células.
- Após 20 min, sifão o líquido restante de cada poço e lave os poços duas vezes com 200 μL de água purificada estéril. Deixe a placa secar no gabinete de biossegurança (por ~2 h).
- Embrulhe o prato em papel alumínio ou coloque-o em um recipiente secundário fechado e guarde-o na geladeira a 4 °C.
- Colete PBMCs usando tubos especiais de coleta de sangue de 8 mL (16 mm x 125 mm; contendo anticoagulante citrato de sódio 0,1 M ou anticoagulante de heparina sódica) (consulte a Tabela de Materiais).
- Avaliação bioenergética de PBMCs com analisador de fluxo metabólico em tempo real
NOTA: Usando um analisador de fluxo e o leitor multimodo (consulte Tabela de Materiais), uma densidade de semeadura ideal de 150.000 PBMCs por poço, uma concentração final de carbonilcianeto-4 (trifluorometoxi) fenilhidrazona (FCCP) do desacoplador de 2 μM e uma concentração final de Hoechst 33342 de 4 μM (com um tempo de incubação de pelo menos 20 min pós-injeção) foram determinados.- Para hidratar os cartuchos do sensor com o hydrobooster, remova o cartucho do sensor com a placa utilitária da caixa e coloque o cartucho "sensor para cima" na bancada. Pipetar 200 μL de calibrante (consulte a Tabela de Materiais) em cada poço da placa de utilidade e coloque o hydrobooster firmemente na placa de utilidade, seguido pelo cartucho do sensor. Inspecione e remova quaisquer bolhas de ar presas e coloque em uma incubadora não-CO2 a 37 °C durante a noite.
- Ligue o analisador de fluxo e, em seguida, abra o software de análise de ensaio (consulte a Tabela de Materiais). Defina a temperatura para 37 °C. Deixe o instrumento ligado durante a noite para permitir a estabilização da temperatura definida.
NOTA: As seguintes etapas de programação (1.2.4-1.2.10) podem ser feitas no dia anterior ou no dia do experimento - Para programar o arquivo do software de análise de ensaio (consulte Tabela de materiais), abra o modelo para o Teste de Tensão de Oxidação do Substrato. Vá para Definições de Grupo, abra a guia Estratégias de Injeção , edite o Inibidor + Teste de Estresse Sub Ox para Etomoxir + Teste de Estresse Sub Ox e adicione mais duas estratégias de injeção: UK5099 + Teste de Estresse Sub Ox e BTPES + Teste de Estresse Sub Ox. Dentro de cada estratégia de injeção, dentro da janela Condição de Injeção , clique em A para a Porta A e insira a concentração final do respectivo inibidor (Etomoxir: 4 μM; UK5099: 2 μM; BTPES: 3 μM).
- Clique nas portas B, C e D e insira as respectivas concentrações finais (porta B: concentração de oligomicina [1.50 μM], porta C: FCCP [2.0 μM]; porta D: Rotenona + Antimicina A [0,5 μM]). Adicione Hoechst 33342, 4 μM à janela de compostos para a porta D.
- Abra a guia Pré-tratamentos e escolha pré-tratamentos, se aplicável. Por exemplo: Controle, Experimental ou habitual (por exemplo, Linha de Base e Acompanhamento). Adicione descrições na janela abaixo, se necessário.
- Abra a guia Mídia de ensaio e, no menu suspenso Mídia , escolha Meio RPMI, pH 7,4, insira o número do lote, o pessoal que prepara a mídia, o tempo de preparação e os suplementos adicionados à mídia (glicose 10 mM, glutamina 2 mM, piruvato 1 mM; consulte a Tabela de Materiais).
- Abra a guia Tipo de célula e insira PBMC-ID do assunto como o nome, PBMC como o tipo de célula, 150000 como a densidade de propagação, ID do sujeito como a origem, pessoal que preparou as células, data de preparação e dia do descongelamento da célula, se aplicável.
- Clique em Gerar Grupos, vá para Mapa de Placas e atribua os poços da placa de 96 poços aos respectivos grupos. Mantenha os poços A1, A12, H1 e H12 atribuídos como poços de fundo.
- Vá para Protocolo, marque a caixa Equilibrar (configuração padrão) e insira as seguintes informações: Linha de base: cinco ciclos de medição: 3 min mix, 0 min wait e 3 min measure; Meio ou Inibidor (Porta A): seis ciclos de medição: 3 min de mistura, 0 min de espera e 3 min de medição; Oligomicina (Porta B): três ciclos de medição: 3 min de mistura, 0 min de espera e 3 min de medição; FCCP (Porta C): três ciclos de medição: 3 min de mixagem, 0 min de espera e 3 min de medição; Rotenona + Antimicina A + Hoechst 33342 (Porta D): três ciclos de medição: 3 min de mistura, 0 min de espera e 3 min de medição.
- No dia da avaliação bioenergética, retire a microplaca de cultura de células revestida da geladeira e deixe-a aquecer até o RT na cabine de biossegurança.
- Prepare o meio de ensaio (AM) misturando 97 mL de MO pré-aquecido e suplementos: 1 mL de piruvato (concentração final: 1 mM), 1 mL de glicose (concentração final de 10 mM) e 1 mL de glutamina (concentração final: 2 mM). Colocar o AM a 37 °C numa incubadora sem CO2 até à utilização.
- Para semear as células, adicione 50 μL de suspensão celular (de 1.1.12) contendo 150.000 PBMCs a cada poço, exceto A1, H1, A12 e H12 (atribuídos como poços de fundo).
CRÍTICO: A densidade ideal de semeadura foi determinada anteriormente. - Cubra a placa celular com sua tampa e deixe 60 min de repouso celular em RT no gabinete de biossegurança para facilitar a distribuição uniforme das células.
CRÍTICO: Visualize as células ao microscópio para garantir a distribuição celular homogênea desejada pelo poço. - Para imagens celulares, ligue o leitor multimodo (consulte a Tabela de Materiais) algumas horas antes da primeira incubação celular; aguarde a conclusão do protocolo de iniciação do leitor antes de abrir o software de imagem (consulte a Tabela de Materiais). Abra o software de imagem e ajuste a temperatura de pré-aquecimento para 37 °C.
- Para preparar os reagentes de ensaio, siga as instruções do kit de ensaio para reconstituir os reagentes, com pequenas modificações conforme descrito na Tabela 1.
CRÍTICO: As concentrações finais ideais de FCCP e Hoechst33342 foram determinadas anteriormente. - Após o repouso celular de 60 minutos, inspecione os poços com um microscópio de cultura de células e anote quaisquer anormalidades visuais, como agregações celulares.
- Centrifugar a placa de cultura de células em RT numa centrífuga com rotor basculante para microplacas a 60 × g durante 1 min, com aceleração mínima e ruptura mínima; Gire a placa e centrifugue novamente a 40 × g por 1 min. Defina a aceleração e a pausa ao mínimo. Incubar as células durante 25-30 min a 37 °C numa incubadora sem CO2 .
NOTA: Quando a imagem celular é realizada, a placa celular é incubada no leitor multimodo pré-aquecido (consulte a Tabela de Materiais) e uma imagem de campo claro é tirada de cada poço durante este período de incubação (primeira incubação celular). - Digitalize o código de barras da placa de cultura de células com o leitor de código de barras (consulte Tabela de materiais), selecione Iniciar leitura de campo claro, coloque a placa de cultura de células na bandeja da placa e inicie a retração da bandeja. Selecione os poços contendo células para varredura e inicie a varredura.
- Durante a primeira incubação celular, carregue as portas do cartucho do sensor com os reagentes, conforme indicado na Tabela 1: porta (A): 20 μL de AM (controles), etomoxir (inibidor do transporte de ácidos graxos de cadeia longa para as mitocôndrias), UK 5099 (ácido 2-ciano-3-(1-fenil-1H-indol-3-il)-2-propenóico; inibidor do transportador de piruvato mitocondrial) ou BPTES (Bis-2-(5-fenilacetamido-1, 3,4-tiadiazol-2-il)sulfureto de etilo; inibidor da conversão de glutamina em glutamato); porta (B): 22 μL de oligomicina (inibidor da ATP sintase); porta (C): 25 μL de FCCP (desacoplador carbonilcianeto-4 (trifluorometoxi) fenilhidrazona); porta (D): 27 μL de rotenona/antimicina A/Hoechst 33342 (inibidores do complexo mitocondrial I e III, respectivamente, e corante nuclear). Retorne o cartucho do sensor para a incubadora de 37 °C não-CO2 por mais 5 min.
NOTA: Siga as instruções do fabricante para carregamento na porta. - Retire a placa de cultura de células da incubadora ou leitor de placas após a primeira incubação celular e após a conclusão da imagem celular (se aplicável) e adicione AM quente a cada poço até um volume final de 180 μL por poço. Colocar a placa celular a 37 °C numa incubadora sem CO2 durante mais 15-25 min (incubação da segunda célula).
- Inicie o ensaio metabólico iniciando a calibração do sensor durante a incubação da segunda célula. Clique em Executar ensaio quando o ensaio estiver pronto para ser iniciado com a calibração do cartucho do sensor. Quando solicitado, transfira o cartucho do sensor da incubadora de 37 °C não-CO2 para a bandeja do analisador de fluxo e inicie a calibração. Siga as instruções do software de análise de ensaio e substitua a placa de utilidade da placa celular após a conclusão da calibração.
NOTA: A calibração leva aproximadamente 20 min. - Após o ensaio metabólico, siga o prompt do software e remova o cartucho do sensor e a placa celular do instrumento. Remova o cartucho do sensor da placa celular e cubra a placa celular com a tampa.
- Depois que o tempo de incubação do corante Hoechst tiver sido determinado em experimentos preliminares, escaneie o código de barras da placa e siga as instruções do software para adquirir uma imagem fluorescente de cada poço contendo células com o leitor multimodo.
NOTA: O software de imagem e análise de ensaio tem interface, permitindo a importação dos dados de contagem de células para o arquivo de dados de análise de ensaio para normalização de dados para a contagem de células.
2. Expressão gênica do relógio circadiano
NOTA: A expressão dos genes do relógio dos participantes de PBMCs será revisada isolando o RNA usando o kit de sangue de RNA (consulte a Tabela de Materiais).
- Retire 3 mL do sangue do participante diretamente em um tubo de RNA (consulte a Tabela de Materiais) com 6 mL de reagentes estabilizadores. Vórtice por 10 s para misturar completamente.
NOTA: Se os reagentes estabilizadores não forem completamente misturados com a amostra de sangue do participante, há um risco aumentado de erro do estudo. - Transferir 1,0 mL da amostra de sangue estabilizada para um tubo de 15 mL, com 5 mL de tampão de lise eritrocitária. Incube no gelo por 10-15 min. Vortex o tubo 2x durante a incubação. Centrifugar o tubo a 4 °C a 400 × g durante 10 min.
- Identifique o pellet e despeje cuidadosamente o sobrenadante contendo glóbulos vermelhos lisados. Adicione 2 mL de tampão de lise eritrocitária (consulte a Tabela de Materiais), ressuspenda as células, faça um vórtice brevemente e centrifugue a 400 × g por 10 min a 4 ° C.
- Despeje o sobrenadante e deixe o tubo invertido em papel absorvente por 1-2 min. Seque qualquer líquido ao redor da borda do tubo com papel limpo antes da próxima etapa.
- Adicione cuidadosamente 600 μL de tampão de lise de RNA (consulte a Tabela de Materiais). Seguindo o protocolo do fabricante, adicionar β-mercaptoetanol e ressuspender o sedimento.
- Pipetar o lisado diretamente em um homogeneizador de lisado de célula descartável (ver Tabela de Materiais), coluna de centrifugação colocada em um tubo de coleta de 2 mL e centrifugar por 2 min na velocidade máxima para homogeneizar. Descarte a coluna de rotação do homogeneizador de lisado celular descartável e guarde o lisado homogeneizado.
- Adicione 1 volume (600 μL) de etanol a 70% ao lisado homogeneizado e misture por pipetagem. Pipetar cuidadosamente a amostra, incluindo qualquer precipitado que se possa ter formado, para uma nova coluna de centrifugação num tubo de recolha de 2 ml, mas não humedecer a borda. Centrífuga por 15 s a >8.000 × g.
- Transfira a coluna de centrifugação (consulte a Tabela de Materiais) para um novo tubo de coleta de 2 mL. Aplique 700 μL de tampão de lavagem rigoroso (consulte a Tabela de Materiais) na coluna de centrifugação e centrifugue por 15 s a >8.000 × g para lavar. Descarte o fluxo.
- Abra cuidadosamente a coluna de centrifugação e adicione 500 μL de tampão de lavagem suave (consulte a Tabela de Materiais). Feche a tampa e centrifugue em velocidade máxima (20.000 × g) por 3 min.
- Transfira a coluna de centrifugação para um tubo de microcentrífuga de 1,5 mL e pipete 30-50 μL de água livre de RNase diretamente para a membrana de sílica. Centrifugue por 1 min a >8.000 × g para eluir o RNA. Repita mais uma vez.
NOTA: O RNA pode ser armazenado a -80 °C. - Execute o tratamento DNase em uma coluna usando o conjunto DNase livre de RNase (consulte a Tabela de Materiais), de acordo com o protocolo do fabricante.
- Gere cDNA usando 500 ng de RNA total e um sistema de reação em cadeia da polimerase (PCR) em tempo real (ver Tabela de Materiais), de acordo com o protocolo do fabricante. Dilua todas as amostras de cDNA 1:25 em água livre de RNase e use 4 mL para realizar PCR de transcrição reversa quantitativa (qRT-PCR).
- Use um método de adição de primer (consulte a Tabela de Materiais) para completar qRT-PCR com 10 mM de cada um dos primers mostrados na Tabela 2. Preencha o qRT-PCR usando um sistema em tempo real (consulte a Tabela de Materiais).
- Normalize os níveis de mRNA dos genes escolhidos usando os níveis de mRNA Rpl26 . Use o método 2-DDCt para calcular a quantificação relativa.
- Para determinar se a expressão de um determinado mRNA exibiu uma oscilação circadiana, utilize um teste baseado em verossimilhança LR_rhythmicity39 (usando p≤ 0,01) para detectar ritmicidade circadiana em uma condição experimental40.
3. Plano de análise de dados
NOTA: Um inventário médico será usado para categorizar os participantes com base no uso de medicamentos43.
- Use um modelo linear de efeitos mistos, no qual idade, sexo, peso, altura, pressão arterial, frequência cardíaca e outros fatores de risco devem ser incluídos como covariáveis.
- Um termo de efeito aleatório deve ser incluído para explicar a correlação de dados dentro do sujeito.
- Para ajuste de modelo, uma seleção de variável reversa com o princípio hierárquico deve ser implementada.
- A partir do modelo ajustado, meça as alterações do ponto de tempo 1 para o ponto de tempo 2, considerando todas as covariáveis no modelo ajustado.
Resultados
O protocolo proposto inclui dados preliminares que servem como validação para a metodologia. O protocolo incorpora um analisador de fluxo metabólico em tempo real para examinar a função mitocondrial e a utilização de combustível celular, e extração de RNA e qRT-PCR para analisar genes de ritmo circadiano (por exemplo, BMAL1, CLOCK, Nfil2, Nr1d1, Dbp, Cry1, Per2).
A taxa de consumo de oxigênio (OCR) de PBMCs humanas isoladas de cinco participantes do controle, 10 dias após uma análise inicial, é apresentada na Figura 1. Os dados são usados para comparar pré e pós-valores e mostram os valores médios de respiração basal, resposta aguda, respiração máxima e capacidade sobressalente após a injeção de um controle, etomoxir, UK5099 e BPTES. Notavelmente, a Figura 1C mostra uma resposta aguda negativa significativa após a injeção de etomoxir, mas não foram observados efeitos significativos para a respiração basal, respiração máxima ou capacidade ociosa.
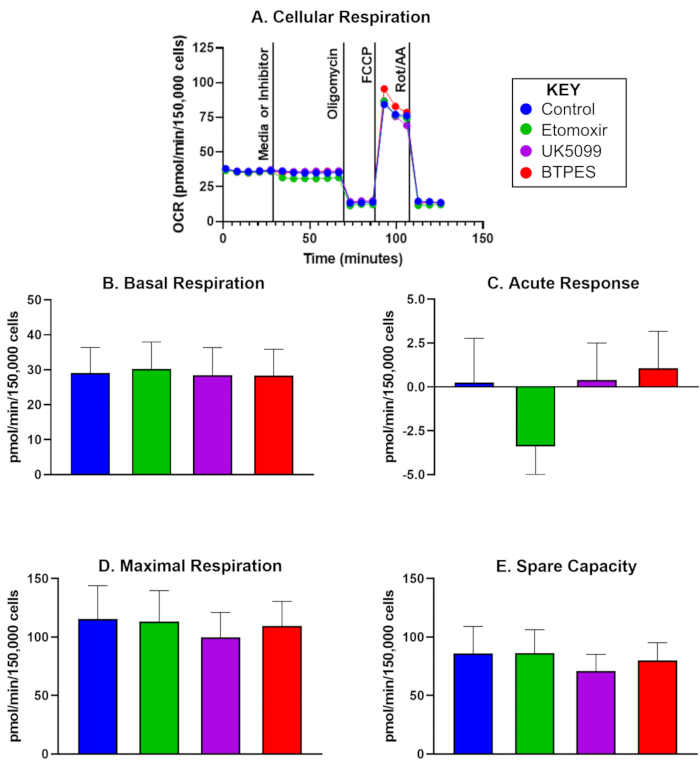
Figura 1: Consumo de oxigênio de células mononucleares isoladas do sangue periférico humano (PBMCs). (A) Taxa de consumo de oxigênio em tempo real (OCR; pmol/(min∙150.000 células) de PBMCs isoladas de um sujeito controle, medido com um analisador de fluxo e avaliado com o ensaio de oxidação do substrato. As células foram semeadas a uma densidade de 150.000 células/poço. A primeira injeção foi média (controle) ou inibidora (etomoxir, UK5099 ou BPTES; consulte o texto para obter detalhes) e ocorreu após a medição da frequência respiratória celular basal. A resposta aguda à limitação do substrato mitocondrial foi determinada como a diferença do OCR basal antes e após a injeção do inibidor. A oligomicina, o inibidor da ATP sintase, inibe a respiração acoplada à produção de ATP e produz respiração por vazamento de prótons. FCCP, o desacoplador, induz respiração máxima e desacoplada; rotenona e antimicina A (inibidores do complexo I e III, respectivamente) inibem tudo, exceto a respiração não mitocondrial (consulte o texto para obter detalhes). (BE) Quantificação da respiração celular (n = 5; os dados são representados como média ± DP). (B) OCR basal antes da injeção do inibidor, (C) resposta aguda ao inibidor (mudança no OCR em relação à taxa basal antes da injeção do inibidor), (D) OCR máximo e (E) capacidade sobressalente (diferença entre OCR máximo e OCR basal após a primeira injeção). A resposta aguda (C) à injeção de etomoxir pode sugerir uma maior dependência do OCR de ácidos graxos como substrato energético em condições basais em comparação com os outros grupos de substrato, sem um efeito perceptível no OCR durante alta demanda de energia (D). Clique aqui para ver uma versão maior desta figura.
| Composto | AM (μL) adicionado ao composto | Estoque (μM) | estoque (μL) para estoque de giro | AM (μL) para material de giro | Material de giro (μM) | Material de trabalho (μL) [porta] | Conc. final (μM) |
| Etomoxir | 700 | 160 | 500 | 1500 | 40 | 20 [UMA] | 4 |
| UK5099 | 700 | 80 | 500 | 1500 | 20 | 20 [UMA] | 2 |
| BPTES | 700 | 120 | 500 | 1500 | 30 | 20 [UMA] | 3 |
| oligo | 420 | 150 | 300 | 2700 | 15 | 22 [B] | 1.5 |
| FCCP | 720 | 100 | 600 | 2400 | 20 | 25 [C] | 2 |
| Podridão / AA / H | 540 | 50 | 300 | 2700 | 5 | 27 [D] | 0.5 |
Tabela 1: Preparação de reagentes para o teste de oxidação do substrato e concentrações de soluções de estoque, trabalho e finais. Todos os reagentes fazem parte do teste de estresse de mito celular ou dos kits de teste de estresse de oxidação do substrato. Abreviaturas: oligo = oligomicina; FCCP = desacoplador carbonilcianeto-4 (trifluorometoxi) fenil-hidrazona; Rot/AA/H = rotenona/antimicina A/Hoechst 33342. Etomoxir, UK5099, BTPES: inibidores da oxidação de ácidos graxos, glicose e glutamina, respectivamente.
| Bmal1 | Avançar – GCACGACGTTCTTTCTTCTGT |
| Reverso – GCAGAAGCTTTTTCGATCTGCTTTTTT | |
| Relógio | Avançar – CGTCTCAGACCCTTCCTCAAC |
| Reverso – GTAAATGCTGCCTGGGTGGA | |
| Choro 1 | Avançar – ACTGCTATTGCCCTGTTGGT |
| Reverso – GACAGGCAAATAACGCCTGA | |
| Por1 | Avançar – ATTCGGGTTACGAAGCTCCC |
| Reverso – GGCAGCCCTTTCATCCACAT | |
| Per2 | Avançar – CATGTGCAGTGGAGCAGATTC |
| Reverso – GGGGTGGTAGCGGATTTCAT | |
| Rev-erb α | Avançar – ACAGATGTCAGCAATGTCGC |
| Reverso – CGACCAAACCGAACAGCATC |
Tabela 2: Primers do gene do relógio circadiano.
Discussão
O declínio da função mitocondrial e a regulação do ritmo circadiano com a idade são cada vez mais vistos como fatores que contribuem para doenças relacionadas à idade. Alterar os ritmos circadianos por meio de modificações no estilo de vida, como dieta e atividade física, representa uma estratégia potencial para promover o envelhecimento saudável e reduzir o declínio da mobilidade associado ao envelhecimento. No entanto, os métodos atuais para avaliar diretamente a função mitocondrial são invasivos e muitas vezes requerem uma biópsia muscular, o que pode representar desafios no recrutamento e retenção dos participantes devido à dor e aos riscos percebidos.
A avaliação de marcadores de saúde circadiana e metabólica por meio de métodos menos invasivos, como coleta de sangue, forneceria resultados valiosos para explorar e testar alvos terapêuticos em estudos futuros. Esses métodos minimamente invasivos têm o potencial de avançar muito no campo, fornecendo novos insights sobre a complexa interação entre o ritmo circadiano e a saúde metabólica e seu impacto na função. O objetivo deste estudo é avaliar a relação entre o metabolismo energético celular e o ritmo circadiano. Em particular, a análise do fluxo bioenergético é usada para avaliar a função mitocondrial sob várias condições de disponibilidade de substrato, juntamente com o monitoramento da expressão gênica de um grupo de genes circadianos nos glóbulos brancos dos participantes. Ao empregar os dois braços da análise, bioenergética e expressão gênica, uma compreensão abrangente da relação entre esses dois processos fundamentais pode ser alcançada.
A análise estatística desses dados de séries temporais de uma perspectiva circadiana oferece informações sobre a força, o alcance e o tempo dos ritmos circadianos. Em conclusão, a integração da análise da expressão gênica, bioenergética celular e medidas metabólicas no nível do organismo constitui uma abordagem nova e inovadora que lançará luz sobre a interação entre o metabolismo energético e os ritmos circadianos em humanos.
Em um estudo piloto, detectamos uma resposta aguda no OCR de PBMCs à limitação da utilização de ácidos graxos (após a injeção de etomoxir, um inibidor da carnitina palmitoil transferase 1a). Esse achado sugere que em PBMCs desse grupo específico de participantes, pode haver uma dependência de ácidos graxos como substrato energético durante a respiração basal. No entanto, a respiração máxima não foi afetada, sugerindo que fontes alternativas de energia, como glicose e glutamina, podem compensar a redução da utilização de ácidos graxos durante a alta demanda de energia. Estudos futuros devem investigar se a) a bioenergética dos PBMCs reflete a energia de todo o corpo e b) se intervenções como alimentação com restrição de tempo podem afetar as preferências de substrato energético.
Existem várias etapas críticas relativas à análise de fluxo de PBMCs. Primeiro, antes que as amostras experimentais sejam avaliadas, a densidade de semeadura celular (células por poço) deve ser otimizada, certificando-se de que haja uma distribuição uniforme contínua de células dentro de cada poço e em cada placa, a concentração final de FCCP deve ser otimizada executando testes de concentrações usando as concentrações 0, 0,125, 0,25, 0,5, 1,0 e 2,0 μM, e, se aplicável, a coloração Hoechst 33342 deve ser otimizada seguindo as instruções do fabricante. Em segundo lugar, a normalização dos dados metabólicos para parâmetros celulares é crítica para a comparabilidade dos dados entre os experimentos. No presente protocolo, a contagem de células após a conclusão do ensaio do analisador de fluxo usando células coradas com Hoechst 33342 e um dispositivo de imagem celular é descrita. Se um dispositivo apropriado não estiver disponível, métodos alternativos de normalização podem ser aplicados, como proteína celular total ou conteúdo de DNA nuclear por poço. Há uma modificação notável que pode ser utilizada dentro do protocolo, em comparação com as que foram propostas. Especificamente, o protocolo pode ser concluído utilizando um kit de ensaio individual para cada um dos três inibidores, em comparação com apenas os dois kits propostos aqui (ver Tabela de Materiais).
O uso de PBMCs como substitutos para estudar a interação entre o metabolismo energético e os ritmos circadianos em adultos mais velhos é limitado pela suposição de que sua resposta ao tratamento pode refletir com precisão a resposta em outros tecidos e órgãos. Embora essa abordagem seja nova e minimamente invasiva, é importante reconhecer que diferentes tecidos e órgãos, como cérebro, fígado e músculo esquelético, podem reagir de maneira diferente sob várias condições. Um estudo pré-clínico demonstrou que a expressão do gene do relógio foi alterada em camundongos alimentados e em jejum, levando à regulação positiva parcial dos genes-alvo BMAL1 no fígado e no tecido muscular, mas à regulação negativa de outros41. Esses tecidos e órgãos periféricos são altamente representativos dos processos metabólicos e podem ser influenciados por pistas ambientais que afetam os mecanismos de expressão gênicado relógio 42. Mais pesquisas são necessárias para entender completamente a relação entre tecidos periféricos, órgãos e o relógio circadiano central.
Outra limitação é que os participantes não são desqualificados para tomar qualquer prescrição, o que pode representar limitações para a análise estatística. Para contrariar essa limitação, em pesquisas futuras, será utilizado um inventário médico, que foi validado em populações de idosos que fazem uso de medicamentos43. Os participantes serão categorizados com base nos medicamentos recomendados registrados na seção de revisão de dados do protocolo. Há um total de três categorias, em termos de medicamentos que demonstraram 1) acelerar o declínio funcional, 2) retardar o declínio funcional e 3) influenciar a função do músculo esquelético.
Por fim, a capacidade oxidativa mitocondrial do músculo esquelético humano exibe um ritmo dia-noite, atingindo o pico entre 18:00 e 23:00 e diminuindo entre 08:00 e 11:0044. Ainda não está claro se isso vale para a capacidade oxidativa mitocondrial dos PBMCs. No entanto, dados preliminares sugerem que as PBMCs e o metabolismo mitocondrial estão relacionados45. Como as informações sobre biópsias musculares e as alterações nas PBMCs não são tão claras, deve-se ter cautela na análise dos resultados. Dada essa limitação, é importante ter essas informações em mente ao avaliar e desenvolver um protocolo, pois elas podem fornecer contexto e informações valiosas que podem ajudar a garantir a validade e a eficácia do protocolo.
Até onde sabemos, nenhum estudo anterior avaliou os padrões de utilização de combustível ou ritmos circadianos por meio dos métodos propostos neste projeto. Nosso objetivo é examinar a capacidade de resposta dos marcadores de utilização de combustível mitocondrial e saúde circadiana às mudanças. Este estudo apresenta um método minimamente invasivo para mensuração de um biomarcador altamente sensível, que pode servir como alternativa em futuros estudos intervencionistas onde a biópsia muscular não é viável.
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Este estudo foi financiado pelo Older American's Independence Center (NIH / NIA P30AG028740), com a assistência do Clinical and Translational Science Institute (NIH / NCRR UL1TR000064).
Materiais
| Name | Company | Catalog Number | Comments |
| Agilent Technologies Cell Imaging | Agilent Technologies | Cell image software | |
| Agilent/Seahorse Wave desktop program | Agilent Technologies | 5994-0039EN | Software used to analyse data from the celluar analyser and stress test assay |
| Agilent/Seahorse XFe96 Flux Analyzer | Agilent Technologies | S7800B | Real-time cellular flux analyzer; flux analyser |
| Bar Code Reader | Agilent Technologies | G2615-90007 | |
| Seahorse Wave Desktop Software | Agilent technologies | Data acquisition software; assay analysis; wave program | |
| Seahorse XF 1.0 M Glucose solution | Agilent Technologies | 103577-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 100 mM Pyruvate solution | Agilent Technologies | 103578-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 200 mM Glutamine solution | Agilent Technologies | 103579-100 | Supplement to basal medium to make assay medium |
| Seahorse XF Cell Mito Stress Test | Agilent Technologies | 103015-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF Mito Fuel Flex Test | Agilent Technologies | 103260-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF RPMI Medium | Agilent Technologies | 103576-100 | Basal medium for PBMCs |
| Seahorse XFe96 FluxPak mini | Agilent Technologies | 102601-100 | Sensor cartridges and cell culture microplates |
| Cytation 1 Cell Imaging Multi-Mode Reader | Agilent/BioTek | Multimode reader to image cells | |
| CPT Sodium Heparin Tube, 16 x 125 mm x 8.0 mL | Becton Dickinson | 362753 | Blood collection tubes for isolation of peripheral blood mononuclear cells |
| CellTak Cell and Tissue Adhesive | Corning | 354240 | Cell adherent to coat cell culture microplate |
| Phosphate Buffered Saline | Corning | 21-040-CV | Buffer to wash blood cells |
| Ficoll Paque Plus | Cytiva | GE17-1440-02 | Gradient medium |
| Lunar Prodigy DXA scanner | General Electric | EN 60601-2-7 5.1 | Whole body lean mass and fat/lean tissue mass ratio |
| Freezing container, Nalgene Mr. Frosty | MilliporeSigma | C1562 | Freezing container used to slow-freeze cell suspension |
| Buffer EL. | Qiagen | 79217 | Erythrocyte lysis buffer |
| Buffer RLT | Qiagen | 79216 | RNA lysis buffer |
| Buffer RPE | Qiagen | 1018013 | Mild washing buffer |
| Buffer RW1 | Qiagen | 1053394 | Stringent washing buffer |
| QIAamp DNA Micro Kit | Qiagen | 56304 | DNA preps: QIAamp MinElute Columns, Proteinase K, Carrier RNA, Buffers, Collection Tubes (2 ml) |
| QIAamp RNA Blood Mini Kit | Qiagen | 52304 | RNA blood kit; Used to isolate RNA |
| QIAshredder | Qiagen | 79656 | disposable cell-lysate homogenizers |
| RNase-Free DNase Set | Qiagen | 79254 | Used to perform DNA digest |
| 2-Mercaptoethanol (Reagent) | Thermo Fisher Scientific | MFCD00004890 | |
| 2-mL collection tubes, 100 count | Thermo Fisher Scientific | AM12480 | |
| Fast SYBR Green Master Mix | Thermo Fisher Scientific | 4385612 | Primers are added to this and used to carry out qRT-PCR |
| Microcentrifuge Tubes, 1.5 mL | Thermo Fisher Scientific | 69715 | Used to hold RNA purification filter during RNA purification |
| Narrow p1000 pipette tips | Thermo Fisher Scientific | 02-707-402 | |
| QuantStudio 3 Real-Time PCR System, MiniAmp Plus Thermal Cycler, and 96-Well Plates Package | Thermo Fisher Scientific | A40393 | |
| Tempus Blood RNA Tube | Thermo Fisher Scientific | 4342792 | RNA Tube |
| Tempus Spin RNA Isolation kit | Thermo Fisher Scientific | 4380204 | RNA extraction and isolation |
Referências
- UN Department of Economic and Social Affairs. Population Division 2019, World Population Prospects. UN Department of Economic and Social Affairs. , (2019).
- Anton, S., Leeuwenburgh, C. Fasting or caloric restriction for healthy aging. Experimental Gerontology. 48 (10), 1003-1005 (2013).
- Dziechciaż, M., Filip, R. Biological psychological and social determinants of old age: Bio-psycho-social aspects of human aging. Annals of Agricultural and Environmental Medicine. 21 (4), 835-838 (2014).
- Anton, S. D., et al. Flipping the metabolic switch: understanding and applying the health benefits of fasting. Obesity. 26 (2), 254-268 (2018).
- Fried, L. P., Guralnik, J. M. Disability in older adults: evidence regarding significance, etiology, and risk. Journal of the American Geriatrics Society. 45 (1), 92-100 (1997).
- Manini, T. Development of physical disability in older adults. Current Aging Science. 4 (3), 184-191 (2011).
- Chung, H. Y., et al. Molecular inflammation: underpinnings of aging and age-related diseases. Ageing Research Reviews. 8 (1), 18-30 (2009).
- Sun, N., Youle, R. J., Finkel, T. The mitochondrial basis of aging. Molecular Cell. 61 (5), 654-666 (2016).
- Tarasov, A. I., Griffiths, E. J., Rutter, G. A. Regulation of ATP production by mitochondrial Ca2. Cell Calcium. 52 (1), 28-35 (2012).
- Chistiakov, D. A., Sobenin, I. A., Revin, V. V., Orekhov, A. N., Bobryshev, Y. V. Mitochondrial aging and age-related dysfunction of mitochondria. Biomed Research International. 2014, 238463 (2014).
- Boengler, K., Kosiol, M., Mayr, M., Schulz, R., Rohrbach, S. Mitochondria and ageing: role in heart, skeletal muscle and adipose tissue. Journal of Cachexia, Sarcopenia, and Muscle. 8 (3), 349-369 (2017).
- Drew, B., et al. Effects of aging and caloric restriction on mitochondrial energy production in gastrocnemius muscle and heart. American Journal of Physiology Regulatory, Integrative and Comparative Physiology. 284 (2), R474-R480 (2003).
- Short, K. R., et al. Decline in skeletal muscle mitochondrial function with aging in humans. Proceedings of the National Academy of Sciences. 102 (15), 5618-5623 (2005).
- Musci, R. V., Hamilton, K. L., Miller, B. F. Targeting mitochondrial function and proteostasis to mitigate dynapenia. European Journal of Applied Physiology. 118 (1), 1-9 (2018).
- Picca, A., et al. Targeting mitochondrial quality control for treating sarcopenia: lessons from physical exercise. Expert Opinion on Therapeutic Targets. 23 (2), 153-160 (2019).
- Fernandez-Marcos, P. J., Auwerx, J. Regulation of PGC-1α, a nodal regulator of mitochondrial biogenesis. The American Journal of Clinical Nutrition. 93 (4), 884-890 (2011).
- Kim, Y., Triolo, M., Hood, D. A. Impact of aging and exercise on mitochondrial quality control in skeletal muscle. Oxidative Medicine and Cellular Longevity. 2017, 3165396 (2017).
- Wang, H., Hiatt, W. R., Barstow, T. J., Brass, E. P. Relationships between muscle mitochondrial DNA content, mitochondrial enzyme activity and oxidative capacity in man: alterations with disease. European Journal of Applied Physiology and Occupational Physiology. 80 (1), 22-27 (1999).
- Tian, Q., et al. Muscle mitochondrial energetics predicts mobility decline in well-functioning older adults: The baltimore longitudinal study of aging. Aging Cell. 21 (2), e13552 (2022).
- Sardon Puig, L., Valera-Alberni, M., Cantó, C., Pillon, N. J. Circadian rhythms and mitochondria: connecting the dots. Frontiers in Genetics. 9, 452 (2018).
- Gano, L. B., Patel, M., Rho, J. M. Ketogenic diets, mitochondria, and neurological diseases. Journal of Lipid Research. 55 (11), 2211-2228 (2014).
- Liesa, M., Shirihai, O. S. Mitochondrial dynamics in the regulation of nutrient utilization and energy expenditure. Cell Metabolism. 17 (4), 491-506 (2013).
- Lesnefsky, E. J., Chen, Q., Hoppel, C. L. Mitochondrial metabolism in aging heart. Circulation Research. 118 (10), 1593-1611 (2016).
- Hartman, M. L., et al. Relation of mitochondrial oxygen consumption in peripheral blood mononuclear cells to vascular function in type 2 diabetes mellitus. Vascular Medicine. 19 (1), 67-74 (2014).
- Mahapatra, G., et al. Blood-based bioenergetic profiling is related to differences in brain morphology in African Americans with Type 2 diabetes. Clinical Science. 132 (23), 2509-2518 (2018).
- Moore-Ede, M. C. Physiology of the circadian timing system: predictive versus reactive homeostasis. The American Journal of Physiology. 250 (5), R737-R752 (1986).
- Young, M. W. Life's 24-hour clock: molecular control of circadian rhythms in animal cells. Trends in Biochemical Sciences. 25 (12), 601-606 (2000).
- Yoo, S. H., et al. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proceedings of the National Academy of Sciences. 101 (15), 5339-5346 (2004).
- Zhang, R., Lahens, N. F., Ballance, H. I., Hughes, M. E., Hogenesch, J. B. A circadian gene expression atlas in mammals: implications for biology and medicine. Proceedings of the National Academy of Sciences. 111 (45), 16219-16224 (2014).
- de Goede, P., Wefers, J., Brombacher, E. C., Schrauwen, P., Kalsbeek, A. Circadian rhythms in mitochondrial respiration. Journal of Molecular Endocrinology. 60 (3), R115-R130 (2018).
- Hood, S., Amir, S. The aging clock: circadian rhythms and later life. The Journal of Clinical Investigation. 127 (2), 437-446 (2017).
- Sellix, M. T., et al. Aging differentially affects the re-entrainment response of central and peripheral circadian oscillators. The Journal of Neuroscience. 32 (46), 16193-16202 (2012).
- Sato, S., et al. Circadian reprogramming in the liver identifies metabolic pathways of aging. Cell. 170 (4), 664-677 (2017).
- Lundell, L. S., et al. et al. feeding alters lipid and amino acid metabolite rhythmicity without perturbing clock gene expression. Nature Communications. 11 (1), 4643 (2020).
- Perrin, L., et al. Transcriptomic analyses reveal rhythmic and CLOCK-driven pathways in human skeletal muscle. eLife. 7, e34114 (2018).
- Gutierrez-Monreal, M. A., Harmsen, J. -. F., Schrauwen, P., Esser, K. A. Ticking for metabolic health: the skeletal-muscle clocks. Obesity. 28, S46-S54 (2020).
- Wolff, C. A., et al. Defining the age-dependent and tissue-specific circadian transcriptome in male mice. bioRxiv. 42 (1), 111982 (2023).
- Wilson, D., Breen, L., Lord, J. M., Sapey, E. The challenges of muscle biopsy in a community based geriatric population. BMC Research Notes. 11 (1), 830 (2018).
- Ding, H., et al. Likelihood-based tests for detecting circadian rhythmicity and differential circadian patterns in transcriptomic applications. Briefings in Bioinformatics. 22 (6), 224 (2021).
- Ding, Z., Lamb, T. M., Boukhris, A., Porter, R., Bell-Pedersen, D. Circadian clock control of translation initiation factor eIF2α activity requires eIF2γ-dependent recruitment of rhythmic PPP-1 phosphatase in Neurospora crassa. mBio. 12 (3), e00871 (2021).
- Di Francesco, A., Di Germanio, C., Bernier, M., de Cabo, R. A time to fast. Science. 362 (6416), 770-775 (2018).
- Kalfalah, F., et al. Crosstalk of clock gene expression and autophagy in aging. Aging. 8 (9), 1876-1895 (2016).
- Psaty, B. M., et al. Assessing the use of medications in the elderly: methods and initial experience in the cardiovascular health study. Journal of Clinical Epidemiology. 45 (6), 683-692 (1992).
- van Moorsel, D., et al. Demonstration of a day-night rhythm in human skeletal muscle oxidative capacity. Molecular Metabolism. 5 (8), 635-645 (2016).
- Janssen, J. J. E., et al. Extracellular flux analyses reveal differences in mitochondrial PBMC metabolism between high-fit and low-fit females. American Journal of Physiology. Endocrinology and Metabolism. 322 (2), E141-E153 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados