É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Ensaio de Sedimentação à Base de Concanavalina A para Medir a Ligação ao Substrato de Fosfatases Glucanas
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este método descreve um ensaio de sedimentação in vitro baseado em lectina para quantificar a afinidade de ligação da fosfatase glucana e amilopectina. Este ensaio de co-sedimentação é confiável para medir a ligação do substrato glucana fosfatase e pode ser aplicado a vários substratos glucanos solubilizados.
Resumo
As fosfatases glucanas pertencem à maior família de fosfatases de dupla especificidade (DSP) que desfosforilam substratos glucanos, como glicogênio em animais e amido em plantas. As estruturas cristalinas da fosfatase glucana com substratos modelo de glucana revelam distintas interfaces de ligação glucanas feitas de DSP e domínios de ligação a carboidratos. No entanto, medidas quantitativas das interações glucano-glucana fosfatase com substratos fisiologicamente relevantes são fundamentais para o entendimento biológico da família de enzimas glucanas fosfatases e para a regulação do metabolismo energético. Este manuscrito relata um ensaio de sedimentação in vitro baseado em Concanavalina A () projetado para detectar a afinidade de ligação ao substrato das fosfatases glucanas contra diferentes substratos glucanos. Como prova de conceito, determinou-se a constante de dissociação (KD) da fosfatase de glucana Arabidopsis thaliana Starch Excess4 (SEX4) e amilopectina. A caracterização dos mutantes SEX4 e de outros membros da família de enzimas glucanas fosfatases demonstra ainda mais a utilidade deste ensaio para avaliar a ligação diferencial das interações proteína-carboidrato. Estes dados demonstram a adequação deste ensaio para caracterizar uma ampla gama de proteínas interagindo amido e glicogênio.
Introdução
As fosfatases glucanas são membros de uma subfamília funcionalmente diversa de fosfatases de dupla especificidade (DSPs) dentro dasuperfamília 1 da proteína tirosina fosfatase (PTP). Eles têm sido encontrados na maioria das formas de vida, incluindo organismos fotossintéticos amplamente divergentes, humanos, vertebrados e alguns invertebrados e protistas 2,3,4. As plantas contêm três fosfatases glucanas conhecidas: Starch Excess4 (SEX4), Like Sex Four1 (LSF1) e Like Sex Four2 (LSF2)5,6,7. Plantas que não possuem fosfatases glucanas apresentam taxas reduzidas de degradação transitória do amido e acúmulo de amido nas folhas 8,9. A laforina é o membro fundador da família das fosfatases glucanas, que desfosforila glicogênio em vertebrados e humanos 3,10. As mutações da laforina resultam em doença de Lafora neurodegenerativa, uma forma autossômica recessiva fatal de epilepsia11. As fosfatases glucanas são necessárias para o metabolismo do glicogênio e amido e têm emergido como enzimas importantes para a modulação do conteúdo de amido em plantas e tratamento da doença neurodegenerativa deLafora12,13. Estudos recentes de cristalografia de raios X em fosfatases de glucanas com substratos modelo de glucanas têm lançado luz sobre a ligação de substratos e o mecanismo catalítico da desfosforilação de glucanos14,15,16,17. No entanto, a compreensão atual de como as fosfatases glucanas se ligam a seus substratos fisiológicos é incompleta.
O amido é um polímero insolúvel de glicose à base de amilopectina a 80%-90% e amilose a 10%-20%18. Os substratos para as fosfatases glucanas vegetais são moléculas de carboidratos fosforilados, como glicogênio e grânulos de amido. Os resíduos de glucosil fosforilados estão presentes a uma razão fosfato:resíduo de glucosil de 1:600. Curiosamente, os fosfatos estão presentes apenas nas moléculas de amilopectina19. A principal planta glucana fosfatase SEX4 atua no grânulo de amido para desfosforilar moléculas de amilopectina. A estrutura cristalina de raios X do SEX4 combinada com estudos de mutagênese guiada por estrutura demonstrou as especificidades únicas do substrato do SEX4 para diferentes posições dentro de uma estrutura glucana15. Recentemente, demonstramos que a atividade biologicamente relevante do SEX4 só pode ser observada quando atua sobre seus substratos de amilopectina solubilizados20. No entanto, o entendimento das interações glucano-SEX4 tem se mostrado difícil devido à complexidade estrutural do substrato, especificidades de ligação mais amplas e baixas afinidades de ligação entre a proteína e seus substratos. Essas questões têm dificultado a capacidade de utilizar métodos comumente usados em interações proteína-ligante, tais como calorimetria de titulação isotérmica (ITC), espectroscopia de ressonância magnética nuclear (RMN) e ensaios baseados em ensaio imunoenzimático (ELISA).
Curiosamente, muito da nossa compreensão das interações carboidrato-proteína veio do estudo de lectinas. Concanavalina A () é uma leguminosa da família de proteínas extraídas originalmente do feijão-de-porco. O liga carboidratos com alta especificidade, o que é vantajoso para seu uso em aplicações de direcionamento e liberação de medicamentos. A ligação do a uma variedade de substratos contendo α-D-manosil e α-D-glicosil não redutores tem sido extensivamente estudada19,20. Esferas de Sepharose ligadas a comercialmente disponíveis são comumente usadas para purificar glicoproteínas e glicolipídios21. O liga-se a essas glucanas via grupos hidroxila C3, C4 e C6 dos resíduos de glicose. As esferas de-Sepharose também têm sido usadas com sucesso para medir a ligação das interações glicogênio-proteína e amido-proteína22,23. Neste estudo, usamos esferas-Sepharose para desenvolver um ensaio de ligação para medir as especificidades de ligação das interações fosfatase-amilopectina glucana.
Anteriormente, um ensaio de sedimentação baseado em foi empregado para avaliar a capacidade de ligação ao substrato da fosfatase glucana14,20,24. Neste estudo, a mesma estratégia foi usada para desenvolver um novo método para determinar a afinidade de ligação das interações glucano-glucana fosfatase e carboidrato. Este método também tem uma vantagem para investigar várias interações carboidrato-proteína solubilizadas.
Protocolo
1. Preparação de contas de-Sepharose
- Completar 250 mL de um tampão de ligação contendo 67 mM HEPES (pH 7,5), 10 mM MgCl 2 e 0,2 mM CaCl2. Ajustar o pH utilizando solução de NaOH 1 M.
- Pipetar 250 μL de suspensão de esferas de-Sepharose para um tubo de microcentrífuga de 1,5 mL. Centrifugar o conteúdo a 10.000 x g durante 30 s a 4 °C. Descarte o sobrenadante.
NOTA: 250 μL de esferas de-Sepharose em um tubo de microcentrífuga de 1,5 mL são necessários para cada concentração de amilopectina usada para o ensaio. - Adicionar 750 μL do tampão de ligação a cada tubo contendo 250 μL de esferas de-Sepharose. Centrifugar os tubos a 10.000 x g por 1 min a 4 °C. Remova o sobrenadante. Repita esta etapa 2x para garantir que as contas sejam adequadamente lavadas e equilibradas com o buffer de ligação.
2. Preparação de soluções de amilopectina
- Fazer uma solução-mãe de 10 mg/mL de amilopectina de batata. A amilopectina é insolúvel em água e pode ser solubilizada pelo calor. Para solubilizar, adicionar 0,1 g de amilopectina de batata a 10 mL de água destilada. Aquecer a suspensão em banho-maria a 80 °C durante 1 h ou até que a solução deixe de estar turva.
- Permitir que a solução retorne à temperatura ambiente (TR), com vórtices repetidos para evitar aglomeração.
- O tratamento álcool-alcalino é um método alternativo para solubilizar substratos de amilopectina. Para solubilizar usando este método, siga as etapas abaixo.
- Suspender 0,5 g de substrato amilopectina em 5 mL de etanol a 20% e 5 mL de NaOH 2 M. Mexa o conteúdo vigorosamente por 15-20 min em RT.
- Em seguida, adicione 10 mL de água e ajuste o pH da solução para 6,5 adicionando 2 M de HCl. Aumente o volume da solução resultante para 50 mL com água destilada para fazer uma solução de amilopectina de 10 mg/mL.
- Diluir a solução de amilopectina solubilizada a 10 mg/ml para fazer uma série de 2 ml de soluções diluídas de amilopectina. Por exemplo, realizar meia diluição de 10 mg/mL para preparar uma série de concentrações de amilopectina (5 mg/mL, 2,5 mg/mL, 1,25 mg/mL, 0,625 mg/mL, 0,3125 mg/mL, 0,156 mg/mL, 0,078 mg/mL, 0,039 mg/mL, 0,019 mg/mL e 0 mg/mL).
3. Preparação de-Sepharose: contas de amilopectina
- Adicionar 250 μL de cada solução diluída de amilopectina a tubos de microcentrífuga de 1,5 ml contendo 250 μL de esferas de-Sepharose pré-equilibradas em tampão de ligação. Misture bem o conteúdo. Rotular os tubos com a concentração de amilopectina correspondente.
- Incubar o conteúdo numa roda giratória a 4 °C durante 30 min.
NOTA: Não há nenhuma alteração no complexo ligado a-Sepharose:amilopectina ao longo do tempo após 20 min. O tempo de incubação de 30 min foi escolhido variando os tempos de incubação de 10 min a 1 h para garantir que o equilíbrio fosse alcançado. - Centrifugar os tubos a 10.000 x g por 1 min. Recolher o sobrenadante num tubo de microcentrífuga de 1,5 ml recentemente marcado. Salve essas frações sobrenadantes para realizar o ensaio de D-glicose12 (hidrólise ácida da amilopectina seguida de determinação UV de glicose via ensaio enzimático). Este passo é necessário para garantir que toda a amilopectina está ligada às contas.
- Adicionar 750 μL de tampão de ligação às esferas de-Sepharose:amilopectina. Centrifugar os tubos a 10.000 x g por 1 min. Descarte o sobrenadante para remover quaisquer moléculas de amilopectina não ligadas.
- Repita o passo 3.4 para garantir uma lavagem suficiente. Cada tubo agora contém esferas de-Sepharose ligadas a quantidades variáveis de substratos de amilopectina.
4. Incubação SEX4 com-Sepharose: contas de amilopectina
- Misturar 250 μL de esferas de-Sepharose:amilopectina com 100 μL do tampão de ligação, que inclui 10 μg de proteína SEX4, 10 mM de ditiotreitol (DTT) e 10 μM de coquetel inibidor de protease (PIC). Note que o volume total em cada tubo é de 350 μL.
NOTA: Um coquetel inibidor de protease é adicionado como uma medida de precaução para evitar qualquer degradação desnecessária do SEX4. Esta é uma etapa opcional. Neste ensaio, a proteína recombinante Arabidopsis thaliana SEX4 (AtSEX4) é utilizada. A proteína purificada contém uma etiqueta de histidina N-terminal necessária para detectar a proteína através da quimioluminescência. Informações detalhadas sobre purificações de fosfatase glucana são descritas em publicações anteriores14,20,24. - Incubar a suspensão de proteína e-Sepharose:amilopectina a 4 °C durante 45 minutos com rotação suave.
NOTA: O tempo de incubação de 45 min é escolhido para garantir que o equilíbrio seja alcançado para o complexo. - Centrifugar os tubos a 10.000 x g por 1 min. Pipetar 50 μL do sobrenadante cuidadosamente usando uma ponta de carregamento de gel em um novo tubo de microcentrífuga de 1,5 mL. Adicionar 20 μL de corante 4x SDS-PAGE e 10 μL de água a cada tubo contendo 50 μL das frações sobrenadantes coletadas. Aquecer as amostras a 95 °C durante 10 min. Salve esses exemplos para executar os géis SDS-PAGE. Certifique-se de que 10 novos tubos rotulados como "sobrenadante (S)" tenham as concentrações de substrato correspondentes.
- Adicionar 750 μL do tampão de ligação às esferas-Sepharose:amilopectina: SEX4 para remover qualquer proteína não ligada das contas. Centrifugar os tubos a 10.000 x g por 1 min. Repita este passo mais uma vez para garantir a lavagem adequada. Descarte o sobrenadante.
- Adicionar 20 μL de corante 4x SDS-PAGE e 80 μL de água destilada nos tubos contendo esferas lavadas de-Sepharose:amilopectina:SEX4. Aquecer as amostras a 95 °C durante 10 min e centrifugar a 10.000 x g durante 1 min.
- Descarte o pellet e salve o sobrenadante para executar os géis SDS-PAGE. Pipetar 80 μL do sobrenadante em novos tubos e rotulá-los como "pellet (P)".
5. Executando géis SDS-PAGE
- Coloque 40 μL das amostras de proteína não ligadas (feitas na etapa 2.3, marcadas com S) em poços de gel de poliacrilamida pré-moldada de 4%-12% da menor concentração de substrato para a mais alta, mas mantenha a primeira faixa livre para carregar o marcador de peso molecular da proteína. Use um segundo gel para carregar 10 amostras de proteína ligadas feitas na etapa 2.5 (rotuladas como P).
- Adicione o buffer de funcionamento 1x SDS-PAGE recém-preparado a ambas as câmaras do aparelho. Passe o gel a 150 V durante 35 minutos ou até que a frente do corante atinja o fundo do gel.
- Retire o gel de corrida do aparelho e remova os espaçadores e placas de vidro. Use o gel separado para executar uma análise de western blot.
6. Western blotting para detecção por quimioluminescência14,15
NOTA: Este método pode ser facilmente modificado/adaptado dependendo do equipamento de western blotting que os usuários têm em seus laboratórios.
- Fazer 1 L de tampão de transferência contendo 5,8 g de Tris base, 2,9 g de glicina, 0,37 g de SDS e 200 mL de metanol.
- Transfira as proteínas separadas pelo tamanho do gel de poliacrilamida para uma membrana de nitrocelulose. Montar brevemente as esponjas, papéis de filtro, gel e membrana de nitrocelulose de acordo com o protocolo de transferência ocidental14,15. Funcione a 70 V por 1 h.
- Para evitar a ligação inespecífica às proteínas, incubar a membrana de nitrocelulose contendo a solução proteica de albumina de soro bovino (BSA) a 1%-5% ou proteína do leite em 50 mL de tampão TBST (20 mM Tris [pH 7,5], 150 mM NaCl, 0,1% Tween 20) por 1 h. Lave a membrana 3x usando o tampão TBST para remover qualquer solução de bloqueio não ligada.
- Incubar a membrana com um anticorpo ligado à peroxidase de raiz forte (HRP) específico para proteína marcada com His por 1 h. Lave a membrana 3x em tampão TBST para remover quaisquer anticorpos não ligados. Use uma diluição de 1:2.000 de anticorpo para TBST para obter uma ótima reprodutibilidade e sensibilidade.
- O anticorpo ligado à enzima HRP liga-se especificamente à etiqueta histidina da proteína SEX4, que produz uma banda na presença de reagentes de quimioluminescência. Para imagens digitais, fazer uma solução de partes iguais de soluções de substrato quimioluminescente (750 μL cada) em um tubo de 1,5 mL. Incubar a membrana durante pelo menos 5 minutos na solução.
- Coloque a proteína de membrana de lado para baixo no scanner de manchas e execute o software de aquisição para quantificar a proteína nas frações de pellet e sobrenadante.
7. Análise dos dados
- Realizar as medições quantitativas do sinal utilizando o software de aquisição com o blot scanner. Normalizar todas as medidas quantitativas nas frações sobrenadante e pellet para a proteína total carregada.
NOTA: O software permite quantificar a intensidade de cada banda proteica nas frações sobrenadante e pellet. - No experimento de ligação de saturação, plotar a porcentagem de proteína ligada versus concentração de amilopectina. Ajustar os dados a Y = Bmax x X/(K D + X), utilizando software de análise de dados para calcular KD.
NOTA: Bmax é a ligação específica máxima, o eixo Y é a porcentagem de proteína ligada, o eixo X é a concentração de amilopectina.
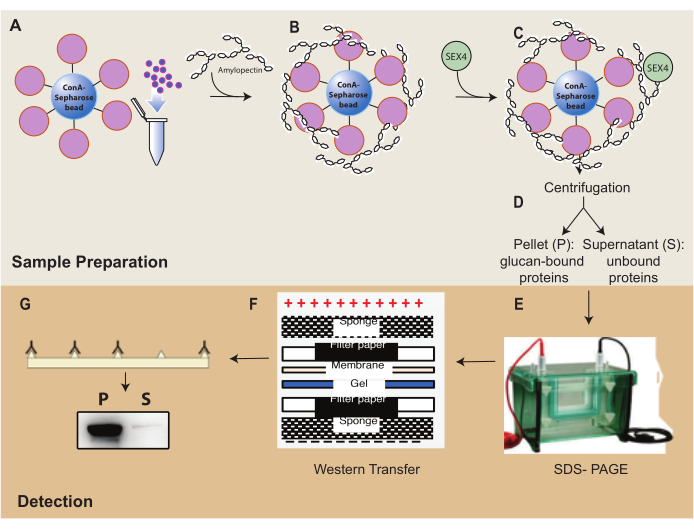
Figura 1: Visão geral do fluxo de trabalho do ensaio de sedimentação -Sepharose. (A) Preparação de contas de-Sepharose. (B) Incubação com substrato amilopectina. (C) Incubação com proteína SEX4. (D) Separação das frações proteicas ligadas e não ligadas por centrifugação. (E) Separação de proteínas através de SDS-PAGE. (F) Análise de Western-blot. (G) Detecção por quimioluminescência da proteína SEX4 marcada com His. Clique aqui para ver uma versão maior desta figura.
Resultados
Uma das principais características da família de proteínas glucanas fosfatases é a sua capacidade de se ligar a substratos glucanos. Primeiramente, a capacidade de ligação do SEX4 às esferas de-Sepharose:amilopectina foi analisada usando SDS-PAGE (Figura 2A). A albumina de soro bovino (BSA) serviu como controle negativo para detectar qualquer ligação inespecífica de proteínas às esferas de-Sepharose:amilopectina. A análise das proteínas em SDS-PAGE mostrou a presença da prote?...
Discussão
Este estudo demonstra o desenvolvimento bem-sucedido de um novo ensaio de sedimentação in vitro que permite determinar a afinidade de ligação das interações glucano-glucana fosfatase. O projeto do ensaio aproveita a ligação específica da lectina aos glucanos através dos resíduos hidroxila de glicose para capturar indiretamente substratos de carboidratos solubilizados em esferas de Sepharose. Isso permite a separação de frações proteicas ligadas e não ligadas via centrifugação ...
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Este estudo foi apoiado pelo prêmio da National Science Foundation MCB-2012074. Os autores agradecem ao Dr. Craig W. Vander Kooi, do Departamento de Bioquímica e Biologia Molecular da Universidade da Flórida, pelas valiosas discussões e apoio. Os autores também agradecem ao Dr. Matthew S. Gentry, do Departamento de Bioquímica e Biologia Molecular da Universidade da Flórida, por seu apoio. Gostaríamos de agradecer à Dra. Sara Lagalwar, presidente do programa de neurociência do Skidmore College, por nos permitir usar o scanner de blot de dígitos C LICOR para imagens de western blot.
Materiais
| Name | Company | Catalog Number | Comments |
| 6x-His Tag monoclonal antibody (HIS.H8), HRP | Therm Fisher Scientific | MA1-21315-HRP | |
| Biorad gel electrophoresis and Western blot kit | Biorad | 1703930 | |
| Calcium chloride | Sigma-Aldrich | 208291 | |
| C-Digit blot scanner | LICOR | 3600-00 | Blot scanner |
| Complete protease inhibitor cocktail | Sigma-Aldrich | 11836170001 | |
| Concanavalin A-sepharose beads | Sigma-Aldrich | C9017 | This product contains in 0.1 M acetate buffer, pH 6, containing 1 M NaCl, 1 mM CaCl2, 1 mM MnCl2, and 1 mM MgCl2 in 20% ethanol |
| Centrifuge | Eppendorf | 5425R | |
| Glycine | Fisher Scientific | BP381-5 | |
| GraphPad Prism 8.0 software | GraphPad | Version 8.0 | Data analysis software |
| HEPES | Sigma-Aldrich | H8651 | |
| Image Studio | LICOR | 3600-501 | Acquisition Software |
| Magnesium chloride | Sigma-Aldrich | M2670 | |
| Methanol | Fisher Scientific | A452SK-4 | |
| Sodium dodecyl sulfate | Fisher Scientific | PI28312 | |
| Potato amylopectin | Sigma-Aldrich | A8515 | |
| Precast SDSPAGE Gels | Genscript | M00653S | |
| Tris base | Fisher Scientific | BP154-1 | |
| Tween 20 | Fisher Scientific | MP1TWEEN201 | |
| Westernsure premium chemiluminescence substrate | LI-COR | 926-95000 |
Referências
- Meekins, D. A., Vander Kooi, C. W., Gentry, M. S. Structural mechanisms of plant glucan phosphatases in starch metabolism. The FEBS Journal. 283 (13), 2427-2447 (2016).
- Gentry, M. S., et al. The phosphatase laforin crosses evolutionary boundaries and links carbohydrate metabolism to neuronal disease. The Journal of Cell Biology. 178 (3), 477-488 (2007).
- Worby, C. A., Gentry, M. S., Dixon, J. E. Laforin, a dual specificity phosphatase that dephosphorylates complex carbohydrates. The Journal of Biological Chemistry. 281 (41), 30412-30418 (2006).
- Gentry, M. S., Pace, R. M. Conservation of the glucan phosphatase laforin is linked to rates of molecular evolution and the glucan metabolism of the organism. BMC Evolutionary Biology. 9, 138 (2009).
- Niittyla, T., et al. Similar protein phosphatases control starch metabolism in plants and glycogen metabolism in mammals. The Journal of Biological Chemistry. 281 (17), 11815-11818 (2006).
- Kotting, O., et al. STARCH-EXCESS4 is a laforin-like Phosphoglucan phosphatase required for starch degradation in Arabidopsis thaliana. The Plant Cell. 21 (1), 334-346 (2009).
- Comparot-Moss, S., et al. A putative phosphatase, LSF1, is required for normal starch turnover in Arabidopsis leaves. Plant Physiology. 152 (2), 685-697 (2010).
- Zeeman, S. C., Northrop, F., Smith, A. M., Rees, T. A starch-accumulating mutant of Arabidopsis thaliana deficient in a chloroplastic starch-hydrolysing enzyme. The Plant Journal: For Cell and Molecular Biology. 15 (3), 357-365 (1998).
- Kotting, O., et al. Identification of a novel enzyme required for starch metabolism in Arabidopsis leaves. The phosphoglucan, water dikinase. Plant Physiology. 137 (1), 242-252 (2005).
- Tagliabracci, V. S., et al. Laforin is a glycogen phosphatase, deficiency of which leads to elevated phosphorylation of glycogen in vivo. Proceedings of the National Academy of Sciences. 104 (49), 19262-19266 (2007).
- Gentry, M. S., Guinovart, J. J., Minassian, B. A., Roach, P. J., Serratosa, J. M. Lafora disease offers a unique window into neuronal glycogen metabolism. The Journal of Biological Chemistry. 293 (19), 7117-7125 (2018).
- Brewer, M. K., et al. Targeting pathogenic lafora bodies in lafora disease using an antibody-enzyme fusion. Cell Metabolism. 30 (4), 689-705 (2019).
- Santelia, D., Zeeman, S. C. Progress in Arabidopsis starch research and potential biotechnological applications. Current Opinion in Biotechnology. 22 (2), 271-280 (2011).
- Raththagala, M., et al. Structural mechanism of laforin function in glycogen dephosphorylation and lafora disease. Molecular Cell. 57 (2), 261-272 (2015).
- Meekins, D. A., et al. Phosphoglucan-bound structure of starch phosphatase Starch Excess4 reveals the mechanism for C6 specificity. Proceedings of the National Academy of Sciences. 111 (20), 7272-7277 (2014).
- Vander Kooi, C. W., et al. Structural basis for the glucan phosphatase activity of Starch Excess4. Proceedings of the National Academy of Sciences. 107 (35), 15379-15384 (2010).
- Meekins, D. A., et al. Structure of the Arabidopsis glucan phosphatase like sex four2 reveals a unique mechanism for starch dephosphorylation. The Plant Cell. 25 (6), 2302-2314 (2013).
- Smith, A. M., Zeeman, S. C. Starch: A flexible, adaptable carbon store coupled to plant growth. Annual Review of Plant Biology. 71, 217-245 (2020).
- Jane, J., Kasemuwan, T., Chen, J. F., Juliano, B. O. Phosphorus in rice and other starches. Cereal Foods World. 41 (11), 827-832 (1996).
- Mak, C. A., et al. Cooperative kinetics of the glucan phosphatase starch excess4. Biochemistry. 60 (31), 2425-2435 (2021).
- Campbell, K. P., MacLennan, D. H. Purification and characterization of the 53,000-dalton glycoprotein from the sarcoplasmic reticulum. The Journal of Biological Chemistry. 256 (9), 4626-4632 (1981).
- Campbell, K. P., MacLennan, D. H., Jorgensen, A. O., Mintzer, M. C. Purification and characterization of calsequestrin from canine cardiac sarcoplasmic reticulum and identification of the 53,000 dalton glycoprotein. The Journal of Biological Chemistry. 258 (2), 1197-1204 (1983).
- Davey, M. W., Sulkowski, E., Carter, W. A. Binding of human fibroblast interferon to concanavalin A-agarose. Involvement of carbohydrate recognition and hydrophobic interaction. Biochemistry. 15 (3), 704-713 (1976).
- Meekins, D. A., et al. Mechanistic insights into glucan phosphatase activity against polyglucan substrates. The Journal of Biological Chemistry. 290 (38), 23361-23370 (2015).
- Wilkens, C., et al. Plant α-glucan phosphatases SEX4 and LSF2 display different affinity for amylopectin and amylose. FEBS Letters. 590 (1), 118-128 (2016).
- Atanasova, M., Bagdonas, H., Agirre, J. Structural glycobiology in the age of electron cryo-microscopy. Current Opinion in Structural Biology. 62, 70-78 (2020).
- Doyle, M. L. Characterization of binding interactions by isothermal titration calorimetry. Current Opinion in Biotechnology. 8 (1), 31-35 (1997).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados