É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Métodos otimizados para a imobilização superficial de colágenos e ensaios de ligação ao colágeno
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este trabalho apresenta um protocolo otimizado para imobilizar e quantificar de forma reprodutível o colágeno tipo I e III em microplacas, seguido por um protocolo aprimorado de ensaio de ligação in vitro para estudar as interações colágeno-composto usando um método de fluorescência resolvido no tempo. A análise e interpretação de dados passo a passo subsequentes são fornecidas.
Resumo
A fibrose ocorre em vários tecidos como uma resposta reparadora a lesões ou danos. Se excessiva, no entanto, a fibrose pode levar a cicatrizes teciduais e falência de órgãos, o que está associado a alta morbidade e mortalidade. O colágeno é um dos principais impulsionadores da fibrose, sendo o colágeno tipo I e tipo III os principais tipos envolvidos em muitas doenças fibróticas. Ao contrário dos protocolos convencionais usados para imobilizar outras proteínas (por exemplo, elastina, albumina, fibronectina, etc.), protocolos abrangentes para imobilizar de forma reprodutível diferentes tipos de colágenos a fim de produzir revestimentos estáveis não estão prontamente disponíveis. A imobilização do colágeno é surpreendentemente desafiadora porque várias condições experimentais podem afetar a eficiência da imobilização, incluindo o tipo de colágeno, o pH, a temperatura e o tipo de microplaca usada. Aqui, é fornecido um protocolo detalhado para imobilizar e quantificar de forma reprodutível os colágenos tipo I e III, resultando em géis/filmes estáveis e reprodutíveis. Além disso, este trabalho demonstra como realizar, analisar e interpretar estudos in vitro de ligação de fluorescência resolvidos no tempo para investigar as interações entre colágenos e compostos candidatos a ligação ao colágeno (por exemplo, um peptídeo conjugado a um quelato metálico carregando, por exemplo, európio [Eu(III)]). Essa abordagem pode ser universalmente aplicada a várias aplicações biomédicas, incluindo o campo da imagem molecular para desenvolver sondas de imagem direcionadas, desenvolvimento de medicamentos, estudos de toxicidade celular, estudos de proliferação celular e imunoensaios.
Introdução
O acúmulo de tecido conjuntivo fibroso como parte do processo natural de cicatrização de feridas após a lesão tecidual é conhecido como fibrose. No entanto, se a deposição de tecido fibroso não terminar e continuar além do necessário para o reparo tecidual, a fibrose se torna excessiva 1,2. A fibrose excessiva prejudica a fisiologia e a função dos órgãos e pode levar a danos aos órgãos e potencialmente falência dos órgãos 3,4,5. Dois principais impulsionadores da fibrose são as proteínas da matriz extracelular (MEC) colágeno tipo I e tipoIII 6. O colágeno é uma proteína estrutural encontrada em vários órgãos que compõe aproximadamente um terço do conteúdo total de proteínas do corpo humano1. Existem 28 tipos diferentes de colágenos identificados pelo sequenciamento do genoma humano, e os mais abundantes são os colágenos fibrilares7. O colágeno fibrilar primário é o colágeno tipo I, que fornece à MEC resistência à tração e resistência à deformação8. O colágeno tipo III é um componente estrutural que fornece elasticidade e colocaliza com o colágeno tipo I. É expresso durante a embriogênese e é encontrado naturalmente em pequenas quantidades na pele, músculos e vasos sanguíneos adultos9.
A síntese de colágeno in vivo começa com um processo intracelular no qual o mRNA é transcrito no núcleo e depois se move para o citoplasma, onde é traduzido. Após a tradução, a cadeia formada sofre modificação pós-traducional no retículo endoplasmático, onde o pró-colágeno (o precursor do colágeno) é formado. O pró-colágeno então viaja para o aparelho de Golgi para modificação final antes de ser excretado para o espaço extracelular10. Através da clivagem proteolítica, o pró-colágeno é transformado em tropocolágeno. Este é então reticulado por meio de uma via de reticulação mediada por enzima catalisada pela enzima lisil oxidase (LOX) ou por meio de uma via de reticulação não mediada por enzima envolvendo a reação de Maillard11. Os protocolos in vitro para imobilizar o colágeno dependem principalmente da capacidade do colágeno de se auto-montar. O colágeno é extraído dos tecidos com base em sua solubilidade, que depende em grande parte da extensão da reticulação das fibrilas de colágeno individuais7. O colágeno fibrilar é dissolvido em ácido acético e as fibrilas podem se reformar quando o pH e a temperatura são ajustados12. In vitro, a fibrilogênese do colágeno pode ser vista como um processo de dois estágios7. O primeiro estágio é a fase de nucleação, onde as fibras de colágeno formam dímeros e trímeros A segunda fase é a fase de crescimento, onde as fibrilas começam a crescer lateralmente e resultam na formação característica da banda D, que geralmente é observada por mudanças na turbidez7. Estudos de microscopia de força atômica (AFM) também revelaram que o colágeno tipo I e tipo III têm características diferentes (Tabela 1)13.
Para estudar as interações de ligação entre o colágeno e outros compostos, o colágeno precisa ser imobilizado de forma reprodutível nos poços das microplacas. Existem vários protocolos para imobilização do colágeno solúvel 14,15,16. Microplacas disponíveis comercialmente que são pré-revestidas com colágeno são normalmente usadas para cultura de células. No entanto, as microplacas pré-revestidas têm uma camada muito fina de uma quantidade desconhecida de colágeno revestida nos poços, o que as torna inadequadas para ensaios de ligação in vitro. Existem vários desafios ao imobilizar colágeno nos poços da placa. Um dos principais desafios é escolher um tipo adequado de microplaca, porque diferentes tipos de colágenos (por exemplo, tipo I e III) têm propriedades químicas diferentes e, portanto, imobilizam de forma mais estável e eficaz, dependendo do material da microplaca. Outro desafio são as condições experimentais do protocolo de imobilização, pois o processo de fibrilogênese depende de múltiplos fatores, incluindo temperatura, pH, concentração estoque de colágeno e concentração iônica do tampão7.
Para estudar as interações entre o colágeno (o alvo) e outros compostos (ou seja, um peptídeo alvo), também é necessário desenvolver um ensaio de triagem robusto para investigar a especificidade e seletividade do composto em direção ao alvo, medindo a constante de dissociação, Kd. A posição do equilíbrio de formação de um complexo bimolecular entre uma proteína (colágeno) e um ligante é expressa em termos da constante de associação Ka, cuja magnitude é proporcional à afinidade de ligação. No entanto, mais comumente, os bioquímicos expressam relações de afinidade em termos da constante de dissociação de equilíbrio, Kd, do complexo bimolecular, que é definido como Kd = 1 / Ka (Kd e é o inverso de Ka). Quanto menor o valorde K d, mais forte é a força de ligação entre a proteína e o ligante. A vantagem de usar Kd para comparar a afinidade de ligação de diferentes ligantes para a mesma proteína (e vice-versa) está ligada ao fato de que as unidades de Kd para um complexo bimolecular são mol / L (ou seja, unidade de concentração). Na maioria das condições experimentais, o valor de Kd corresponde à concentração do ligante que leva a 50% de saturação dos sítios de ligação disponíveis no alvo no equilíbrio17,18. A constante de dissociação é normalmente extraída analisando a ocupação fracionada do receptor (FO), que é definida como a razão entre os locais de ligação ocupados e o total de locais de ligação disponíveis, em função da concentração do ligante. Isso pode ser feito desde que um ensaio analítico capaz de distinguir e medir a quantidade de ligante ligado esteja disponível.
Os ensaios de ligação ao ligante in vitro podem ser realizados usando vários métodos bioanalíticos, incluindo fotometria óptica, métodos de radioligante, espectrometria de massa com plasma indutivamente acoplado (ICP-MS) e ressonância plasmônica de superfície (SPR). Dentre os métodos fotométricos, aqueles baseados na emissão de fluorescência normalmente requerem a marcação de ligantes ou proteínas com fluoróforos para aumentar a sensibilidade e melhorar o limite de detecção do ensaio. Os quelatos de certos íons lantanídeos (III), como Eu (III), são muito atraentes como fluoróforos, pois têm grandes deslocamentos de Stokes, bandas de emissão estreitas (fornecendo uma boa relação sinal-ruído), fotobranqueamento limitado e longa vida útil de emissão. É importante ressaltar que a última propriedade permite o uso de fluorescência resolvida no tempo (TRF) de fluoróforos Eu(III) para abolir a autofluorescência de fundo19. Na versão de imunoensaio fluorescente de lantanídeo com dissociação aprimorada (DELFIA) do ensaio TRF baseado em Eu(III), os ligantes marcados com um quelato de Eu(III) não luminescente são incubados com o receptor imobilizado em microplacas. O complexo ligante/receptor marcado é separado do ligante não ligado, e a fluorescência Eu(III) é ativada pela dissociação do complexo Eu(III) em um pH ácido, seguida de recomplexação com um quelante que aumenta a fluorescência para formar um complexo Eu(III) altamente fluorescente embebido em micela20.
A etapa de descomplexação pode ser razoavelmente alcançada com quelantes, como o pentaacetato de dietilenotriamina (DTPA), que apresentam cinética de descomplexação rápida. No entanto, complexos Eu(III) com certos quelantes macrocíclicos, como DOTA (ácido 1,4,7,10-tetraazaciclododecano1,4,7,10-tetracético) e seus derivados monoamida (DO3AAm), apresentam alta estabilidade termodinâmica e inércia cinética muito alta. Nesse caso, as etapas de descomplexação devem ser otimizadas com precisão para obter ativação suficiente e reprodutível do TRF21 baseado em Eu(III). Vale ressaltar que os complexos lantanídeos (Ln(III))-DOTA e Ln(III)-DO3AAm são os mais comumente empregados como agentes de contraste para imagens moleculares in vivo por técnicas de ressonância magnética (RM)22. Assim, o ensaio TRF baseado em Ln(III) é a ferramenta de escolha para estudar in vitro a afinidade de ligação de sondas moleculares de ressonância magnética com seus alvos biológicos pretendidos. Atualmente, faltam protocolos abrangentes e reprodutíveis para imobilizar colágeno tipo I e tipo III e um pipeline reprodutível para realizar experimentos de TRF Eu(III) de ligação in vitro . Para superar essas limitações, foram desenvolvidos métodos reprodutíveis para automontar e imobilizar o colágeno tipo I e tipo III e gerar géis e filmes estáveis, respectivamente, com a concentração suficiente de colágeno necessária para ensaios de ligação in vitro . Um protocolo otimizado para Eu(III) TRF de complexos à base de Eu(III)-DO3Aam altamente inertes é apresentado. Finalmente, um ensaio TRF de microplaca Eu(III) otimizado in vitro para medir o Kd de ligantes marcados com Eu(III) em direção ao colágeno tipo I e tipo III imobilizado é demonstrado (Figura 1).
Protocolo
NOTA: Todas as informações do produto usadas para este trabalho são apresentadas na Tabela de Materiais.
1. Imobilização de colágeno
NOTA: Certifique-se de que cada poço na microplaca usada durante o ensaio de ligação tenha poços adjacentes livres para evitar fluorescência cruzada. Realize esta parte do protocolo no gelo porque o colágeno se auto-monta em temperaturas e níveis de pH crescentes. Realize este procedimento em uma coifa de cultura de tecidos e em condições estéreis, pois as microplacas são posteriormente incubadas em uma incubadora de cultura de tecidos (TC).
- Imobilização de colágeno tipo I nas microplacas de 96 poços (Figura 2)
Dia 1- Prepare uma bandeja de silicone com gelo. Coloque o frasco contendo colágeno tipo I, a solução salina fria 10x tamponada com fosfato (PBS) e as microplacas no gelo e borrife tudo com etanol a 70%. Coloque o material sob o capô TC.
- Neutralize o colágeno usando volumes iguais de colágeno tipo I e 10x PBS (pH 7,4).
- Inverta a solução algumas vezes, certificando-se de que não se formem bolhas.
- Adicione 100 μL de colágeno neutralizado a todos os outros poços e a todas as outras fileiras da microplaca e incube a 37 ° C por 18-20 h para evaporar o colágeno até a secura.
Dia 2 - Lave as microplacas com 100 μL de 1x PBS, pH 7,4, duas vezes para remover qualquer colágeno não ligado.
- Transferir as microplacas para a incubadora a 37 °C durante mais 2 h para secar antes de as utilizar para outras experiências de ligação.
- Imobilização de colágeno tipo III nas microplacas de 96 poços (Figura 3)
Dia 1- Prepare uma bandeja de silicone com gelo. Coloque o frasco contendo colágeno tipo III, o PBS frio 10x e as microplacas no gelo e borrife tudo com etanol 70%. Coloque o material sob o capô TC.
- Neutralize o colágeno usando volumes iguais de colágeno tipo III e 10x PBS (pH 7,4).
- Adicione 70 μL de colágeno neutralizado a todos os outros poços e a todas as outras fileiras da microplaca e incube a 37 ° C por 2 h, colocando a microplaca sob a capa de cultura de tecidos para evaporar o colágeno até a secura.
Dia 2 - Lave as microplacas com 70 μL de 1x PBS, pH 7,4, duas vezes para remover qualquer colágeno não ligado.
- Transfira as microplacas para a incubadora por 1 h a 37 ° C e, em seguida, transfira as microplacas para a bancada e deixe secar por 1 h antes de usá-las em outros experimentos de ligação.
2. Avaliação da estabilidade dos géis/filmes de colágeno imobilizados
- Incubação com PBS por 1 h
NOTA: Durante o experimento de ligação, incube o colágeno imobilizado com o composto de interesse. É importante investigar a estabilidade do gel ou filme de colágeno resultante. Para fazer isso, meça a estabilidade de três condições: sem lavagem = mede o colágeno imobilizado diretamente após a incubação; lavagem = mede o colágeno imobilizado após lavagem da placa duas vezes com 100 μL de PBS; e 1 h PBS imita e lava = mede o colágeno imobilizado após incubação por 1 h com PBS seguido de duas lavagens com PBS. Abaixo, o método de incubação PBS é explicado.- Adicione 70 μL de PBS (1x) a cada um dos poços revestidos com colágeno e incube a microplaca em temperatura ambiente por 1 h.
- Aspirar o excesso de líquido de cada alvéolo com uma pipeta e lavar com PBS (1x) duas vezes antes de efectuar o ensaio de quantificação das proteínas a seguir descrito.
- Quantificação da quantidade de colágeno imobilizado usando um ensaio de ácido bicinconínico (BCA)
NOTA: Use o kit de ensaio de proteína Pierce BCA (tabela de materiais) seguindo as instruções do fabricante. Faça os respectivos padrões de colágeno para este ensaio. A faixa de concentração para colágeno I é de 0-3.000 μg/mL e para colágeno III de 0-750 μg/mL. No total, faça 11 padrões por colágeno.- Prepare o volume total de reagente de trabalho (WR) necessário seguindo as instruções do fabricante.
- Adicione 25 μL de cada um dos padrões de colágeno nos poços das microplacas (em duplicata). Essas soluções são usadas para desenhar a curva padrão.
- Adicionar 200 μL de solução reagente de trabalho a cada um dos alvéolos que contêm os padrões e os alvéolos revestidos com concentrações desconhecidas de colagénio.
- Coloque a microplaca em um agitador de placas por 30 s. Cobrir as microplacas e incubar a 37 °C durante 30 min.
- Remova as microplacas e deixe esfriar em temperatura ambiente. Medir a absorvância a 560 nm utilizando um leitor de placas.
- Traçar uma curva de calibração traçando A560 (UA) em relação à concentração (μg/ml) das 11 soluções-padrão e utilizar a curva de calibração para calcular a quantidade de colagénio.
3. Ensaio de ligação do ligante TRF de európio (III) (figura 1)
NOTA: O composto usado é um peptídeo candidato a ligação ao colágeno (CBP) marcado com um único complexo Eu(III)-DO3AAm, conhecido como Eu(III)-DO3AAm-CBP (Figura 4).
- Incubação das placas revestidas de colágeno com o composto Eu(III)-DO3AAm-CBP
- Prepare soluções do composto Eu(III)-DO3AAm-CBP com concentrações variando entre 0,1-15 μM (0,1 μM, 0,5 μM, 1 μM, 3 μM, 5 μM, 7 μM, 10 μM e 15 μM) em 1x PBS.
- Adicione 75 μL de cada concentração de composto nos poços revestidos de colágeno (Placa A). Realize o experimento em triplicado para calcular a quantidade de composto que se liga ao colágeno.
- Use uma segunda placa não revestida (Placa B) e adicione 75 μL de cada composto aos poços vazios para calcular a ligação não específica do composto à placa. Use triplicados para cada concentração.
- Incube as microplacas por 1 h em temperatura ambiente.
- Usando uma pipeta, aspire e descarte o excesso de solução de cada poço e lave os poços com 1x PBS duas vezes para remover o excesso de composto não ligado. Execute esta etapa usando as microplacas revestidas com e sem revestimento de colágeno.
- A uma terceira placa não revestida (Placa C), adicione 10 μL da mesma faixa de concentrações de Eu(III)-DO3AAm-CBP (em duplicata). Use a leitura de fluorescência do Eu(III)-DO3AAm-CBP em solução para fazer uma curva de calibração.
NOTA: Não lave ou aspire a solução desta placa.
- Extração ácida de leituras de európio (III) e fluorescência resolvida no tempo (TRF)
NOTA: Consulte as informações suplementares sobre a preparação e calibração dos volumes da solução ácida (AS) e da solução tampão (BS). Os volumes de AS e BS necessários para atingir reprodutivelmente um pH ótimo foram de 54 μL e 46 μL, respectivamente, neste trabalho. Execute a seguinte operação na placa A, placa B e placa C.- Adicione 54 μL de solução ácida (AS) a cada poço e coloque a placa na incubadora a 37 °C por 90 min, cobrindo as microplacas com papel alumínio para evitar a evaporação. A temperatura e o tempo de incubação devem ser cuidadosamente controlados para obter uma descomplexação reprodutível.
- Adicione 46 μL de solução tampão (BS) a cada poço e agite suavemente a placa por 30 s.
- Adicione 100 μL de solução de realce (ES) e agite a placa por 30 s.
- Aguarde 30 minutos antes de ler a placa usando um leitor de placas TRF. Use os parâmetros listados na Tabela 2.
4. Análise dos dados
- Quantificação da concentração de colágeno imobilizado nos poços
- Obter a equação da curva de calibração da A560 (UA) em função da concentração (μg/ml) das 11 soluções-padrão.
- Use as leituras de absorbância adquiridas dos poços contendo os padrões de colágeno.
- Tabular os valores médios dos poços duplicados e traçar a absorbância média em relação às concentrações conhecidas de proteína (colágeno) (μg / mL) para obter a equação para a curva padrão.
- Utilizar os valores de absorvância para calcular a massa (μg) e a concentração (M) do colagénio imobilizado.
- Calcule os valores médios de absorbância nos três poços que continham colágeno imobilizado e registre o desvio padrão.
- Utilizar a equação da curva padrão obtida a partir da curva padrão do colagénio (passo 2.2.6) para converter a absorbância medida a partir dos alvéolos revestidos de colagénio em concertação. A partir disso, calcule a concentração de colágeno que foi imobilizada dentro dos poços experimentais em μg/mL.
- Converter a concentração calculada no passo 4.1.2.2 (μg/ml) primeiro em gramas/litro e, em seguida, em molar (M), com base na massa molecular do colagénio.
- Por fim, calcule a massa de colágeno imobilizado em cada poço dividindo a concentração pelo volume de colágeno adicionado ao poço (100 μL para colágeno tipo I e 70 μL para colágeno tipo III).
- Obter a equação da curva de calibração da A560 (UA) em função da concentração (μg/ml) das 11 soluções-padrão.
- Cálculo da constante de dissociação (Kd) (Figura 4)
- Extraia as leituras de fluorescência.
- Exporte as leituras de fluorescência do leitor de placas para uma planilha.
NOTA: Em ensaios de ligação, é importante levar em conta a potencial ligação não específica de um composto à superfície plástica das placas. - Calcular os valores médios das medições triplicadas de cada concentração de composto para as três placas diferentes: as leituras de ligação específica dos alvéolos revestidos (Placa A), a ligação não específica dos alvéolos não revestidos (Placa B) e o Eu(III)-DO3AAm-CBP total em solução nos alvéolos não revestidos (Placa C).
- Determine os valores de fluorescência para o composto ligado subtraindo as leituras de fluorescência dos poços não revestidos (Placa B) das leituras dos poços revestidos (Placa A).
Equação 1: Determinando a fluorescência ligada17:
Fluorescência ligada = Específica (poços revestidos) - Inespecífica (poços não revestidos) - Gere uma curva de calibração usando as leituras do composto marcado com Eu(III) em solução (Placa C). Representar graficamente as leituras de fluorescência obtidas em relação à concentração do composto marcado com Eu(III). Execute um ajuste de regressão linear.
- Exporte as leituras de fluorescência do leitor de placas para uma planilha.
- Converta as leituras de fluorescência em concentrações.
- Converter as leituras da fluorescência ligada (passo 4.2.3) em concentração utilizando a curva de fluorescência padrão a partir dos dados gerados utilizando as concentrações do composto em solução (passo 4.2.1.4).
NOTA: Ao comparar as propriedades de ligação de um composto em relação a diferentes proteínas-alvo que imobilizam em diferentes concentrações, estas últimas precisarão ser consideradas ao calcular a quantidade de composto ligado ao alvo (ou seja, composto/proteína ligado). - Divida a concentração do composto ligado pela concentração da proteína imobilizada no poço.
NOTA: Para este cálculo, use a concentração de colágeno imobilizado que foi calculada após os poços terem sido incubados com PBS por 1 h (o chamado experimento de imitação de PBS; seção 2.1 acima). Isso é para contabilizar as perdas potenciais de colágeno durante a etapa de incubação e a etapa de lavagem que não contribuirão para o sinal de fluorescência final. - Plote os dados usando um gráfico de dispersão que tenha as concentrações do composto no eixo x (μM) e o composto/proteína ligado no eixo y.
- Converter as leituras da fluorescência ligada (passo 4.2.3) em concentração utilizando a curva de fluorescência padrão a partir dos dados gerados utilizando as concentrações do composto em solução (passo 4.2.1.4).
- Obtenha os valoresde K d .
- Ajustar os dados adquiridos no passo 4.2.2.3 utilizando dois modelos cinéticos de ligação possíveis: ligação de um sítio e ligação de um sítio com declive de colina. As equações para cada modelo são mostradas na Figura 6.
- Escolha o modelo que fornece um ajuste não ambíguo com o maior valor de R-quadrado ao ajustar os dados.
- Exclua o(s) outlier(s) para cada conjunto de leituras de fluorescência por concentração por placa.
- Calcular o valor final de Kd e apresentar os dados como a média ± o desvio-padrão de experiências independentes.
NOTA: Para obter resultados robustos, realize medições triplicadas dentro de cada placa e pelo menos três experimentos independentes com diferentes microplacas.
- Calcule a ocupação fracionada (FO).
NOTA: A partir da Equação 2, a concentração do alvo é desconhecida e, portanto, usando álgebra e o Kd, da Equação 3, surge uma equação viável para calcular a ocupação fracionária na forma da Equação 4.
Equação 2: Definição de ocupação fracionada17:
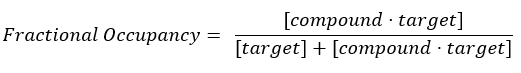
Equação 3: A constante de dissociação, Kd, que é a concentração na qual o composto ocupa 50% do alvo no equilíbrio17:
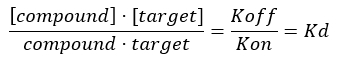
Equação 4: Equação rearranjada para calcular a equação FO17:
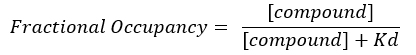
- Calcular o FO utilizando os valores Kd independentes obtidos para cada placa individual. Plote os resultados, média e desvios padrão do FO em relação à concentração do composto.
- Relate o FO com valores que variam de 0 a 1 ou como uma porcentagem com valores que variam de 0% a 100%.
- Extraia as leituras de fluorescência.
Resultados
Avaliação da estabilidade e concentração de colágeno tipo I e tipo III imobilizado em géis/filmes
A quantificação da concentração de colágeno imobilizado por poço foi realizada em três condições diferentes: a) em poços sem lavagem com PBS após imobilização das proteínas (sem lavagem); b) em poços com etapa de lavagem (duas vezes com PBS) após a imobilização para remover qualquer proteína não revestida; c) em poços após incubação com PBS p...
Discussão
Este trabalho apresenta um método reprodutível para imobilização de colágeno tipo I e tipo III. Ele também demonstra um protocolo para adquirir, analisar e interpretar dados de ligação de TRF Eu(III) in vitro para caracterizar as propriedades de ligação de um ligante candidato ao colágeno tipo I e III. Os protocolos de imobilização do colágeno tipo I e tipo III aqui apresentados foram desenvolvidos e otimizados considerando trabalhos publicados anteriormente sobre ...
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Somos gratos aos seguintes financiadores por apoiar este trabalho: (1) o Conselho de Pesquisa Médica do Reino Unido (MR / N013700 / 1) e o King's College London, membro da Parceria de Treinamento de Doutorado em Ciências Biomédicas do MRC; (2) Concessão do programa BHF RG / 20/1/34802; (3) Concessão do Projeto BHF PG/2019/34897; (4) Concessão do Centro de Excelência em Pesquisa King's BHF RE/18/2/34213; (5) o Programa de Iniciativa Científica do Milênio ANID - ICN2021_004; e (6) FB210024 de concessão ANID Basal.
Materiais
| Name | Company | Catalog Number | Comments |
| 10x PBS | Gibco | 14200075 | Use this to make 1x PBS by diluting in water (1:10) |
| 10x PBS | Gibco | 14200075 | Use this to make 1x PBS by diluting in water (1:10) |
| 2M HCL | Made in house and details are in the supporting document | ||
| 2M HCL | Made in house and details are in the supporting document | ||
| 2M Sodium hydroxide +2M Glycine | Made in house and details are in the supporting document | ||
| 2M Sodium hydroxide +2M Glycine | Made in house and details are in the supporting document | ||
| Cell-star 96 well microplate | Greiner Bio-One | 655 160 | |
| Cell-star 96 well microplate | Greiner Bio-One | 655 160 | |
| DELFIA enhacement solution | Perkin Elmer | 1244-104 | |
| DELFIA enhacement solution | Perkin Elmer | 1244-104 | |
| Ice | |||
| Ice | |||
| Infinite 200 PRO NanoQuant microplate reader | TECAN | ||
| Infinite 200 PRO NanoQuant microplate reader | TECAN | ||
| Non-binding (NBS) 96 well microplates | Corning | 3641 | |
| Non-binding (NBS) 96 well microplates | Corning | 3641 | |
| pH electrode Inlab Routine | Mettler Toledo | 51343050 | |
| pH electrode Inlab Routine | Mettler Toledo | 51343050 | |
| pH meter (sevenCompact) | Mettler Toledo | ||
| pH meter (sevenCompact) | Mettler Toledo | ||
| Pierce BCA protein assay kit | Thermofisher | 23227 | |
| Pierce BCA protein assay kit | Thermofisher | 23227 | |
| Tissue culture incubator (37 °C, 5% CO2) | |||
| Type I bovine collagen, 3 mg/mL | Corning | 354231 | |
| Type III human placenta collagen, 0.99 mg/mL | Advanced Biomatrix | 5021 |
Referências
- Distler, J. H. W., et al. Review: Frontiers of antifibrotic therapy in systemic sclerosis. Arthritis and Rheumatology. 69 (2), 257-267 (2017).
- Wynn, T. A. Fibrotic disease and the TH1/TH2 paradigm. Nature Reviews Immunology. 4 (8), 583-594 (2004).
- Saha, P., et al. Magnetic resonance T1 relaxation time of venous thrombus is determined by iron processing and predicts susceptibility to lysis. Circulation. 128 (7), 729-736 (2013).
- Mirshahi, M., et al. Defective thrombolysis due to collagen incorporation in fibrin clots. Thrombosis Research. 8, 73-80 (1988).
- Comerota, A. J. The ATTRACT trial: Rationale for early intervention for iliofemoral DVT. Perspectives in Vascular Surgery and Endovascular Therapy. 21 (4), 221-225 (2009).
- Bateman, E. D., Turner-Warwick, M., Adelmann-Grill, B. C. Immunohistochemical study of collagen types in human foetal lung and fibrotic lung disease. Thorax. 36 (9), 645-653 (1981).
- Pawelec, K. M., Best, S. M., Cameron, R. E. Collagen: A network for regenerative medicine. Journal of Materials Chemistry B. 4 (40), 6484-6496 (2016).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Copes, F., Pien, N., Van Vlierberghe, S., Boccafoschi, F., Mantovani, D. Collagen-based tissue engineering strategies for vascular medicine. Frontiers in Bioengineering and Biotechnology. 7, 166 (2019).
- Veis, A. The biochemistry of collagen. Annals of Clinical and Laboratory Science. 5 (2), 123-131 (1975).
- Bielajew, B. K., Hu, J. C., Athanasiou, K. A. Collagen: Quantification, biomechanics, and role of minor subtypes in cartilage. Nature Reviews Materials. 5, 730-747 (2020).
- Zhao, Z., et al. Structural and functional plasticity of collagen fibrils. DNA and Cell Biology. 38 (4), 367-373 (2019).
- Eryilmaz, E., Teizer, W., Hwang, W. In vitro analysis of the co-assembly of type-I and type-III collagen. Cellular and Molecular Bioengineering. 10 (1), 41-53 (2017).
- Jagnow, J., Clegg, S. Klebsiella pneumoniae MrkD-mediated biofilm formation on extracellular matrix- and collagen-coated surfaces. Microbiology. 149 (9), 2397-2405 (2003).
- O'Sullivan, D., O'Neill, L., Bourke, P. Direct plasma deposition of collagen on 96-well polystyrene plates for cell culture. ACS Omega. 5 (39), 25069-25076 (2020).
- Caravan, P., et al. Collagen-targeted MRI contrast agent for molecular imaging of fibrosis. Angewandte Chemie - International Edition. 46 (43), 8171-8173 (2007).
- Copeland, R. A. . Enzymes: A Practical Introduction to Structure, Mechanism, and Data Analysis. , (2000).
- Salahudeen, M. S., Nishtala, P. S. An overview of pharmacodynamic modelling, ligand-binding approach and its application in clinical practice. Saudi Pharmaceutical Journal. 25 (2), 165-175 (2017).
- Bünzli, J. C. G., Piguet, C. Taking advantage of luminescent lanthanide ions. Chemical Society Reviews. 34 (12), 1048-1077 (2005).
- Hemmilii, I. Luminescent lanthanide chelates - A way to more sensitive diagnostic methods. Journal of Alloys and Compounds. 225 (1-2), 480-485 (1995).
- De Silva, C. R., Vagner, J., Lynch, R., Gillies, R. J., Hruby, V. J. Optimization of time-resolved fluorescence assay for detection of europium-tetraazacyclododecyltetraacetic acid-labeled ligand-receptor interactions. Analytical Biochemistry. 398 (1), 15-23 (2010).
- Digilio, G., Lacerda, S., Lavin Plaza, B., Phinikaridou, A. Extracellular matrix targeted MRI probes. Analysis & Sensing. 3 (1), (2022).
- Phinikaridou, A., et al. Tropoelastin: A novel marker for plaque progression and instability. Circulation. Cardiovascular imaging. 11 (8), 007303 (2018).
- Guzaeva, T. V., et al. Protein A used in DELFIA for the determination of specific antibodies. Immunology Letters. 35 (3), 285-289 (1993).
- Nasiri, A. H., Nasiri, H. R. Polymerase assays for lead discovery: An overall review of methodologies and approaches. Analytical Biochemistry. 563, 40-50 (2018).
- Capuana, F., et al. Imaging of dysfunctional elastogenesis in atherosclerosis using an improved gadolinium-based tetrameric MRI probe targeted to tropoelastin. Journal of Medicinal Chemistry. 64 (20), 15250-15261 (2021).
- Drescher, D. G., Drescher, M. J., Ramakrishnan, N. A. Surface plasmon resonance (SPR) analysis of binding interactions of proteins in inner-ear sensory epithelia. Methods in Molecular Biology. 493, 323-343 (2009).
- Murali, S., Rustandi, R. R., Zheng, X., Payne, A., Shang, L. Applications of surface plasmon resonance and biolayer interferometry for virus-ligand binding. Viruses. 14 (4), 717 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados