Triagem de alto rendimento para obter hits de cristal para cristalografia de proteínas
Neste Artigo
Resumo
Este protocolo detalha a triagem de cristalização de alto rendimento, desde a preparação da placa de microensaio de 1.536 até o final de uma janela de tempo experimental de 6 semanas. Detalhes são incluídos sobre a configuração da amostra, as imagens obtidas e como os usuários podem realizar análises usando uma interface gráfica de usuário habilitada por inteligência artificial para identificar de forma rápida e eficiente as condições de cristalização macromolecular.
Resumo
A cristalografia de raios X é a técnica mais comumente empregada para discernir estruturas macromoleculares, mas a etapa crucial de cristalizar uma proteína em uma rede ordenada passível de difração permanece desafiadora. A cristalização de biomoléculas é amplamente definida experimentalmente, e esse processo pode ser trabalhoso e proibitivo para pesquisadores de instituições com recursos limitados. No Centro Nacional de Cristalização de Alto Rendimento (HTX), métodos altamente reprodutíveis foram implementados para facilitar o crescimento de cristais, incluindo uma configuração automatizada de placa de microlote sob óleo de 1.536 poços de alto rendimento projetada para amostrar uma ampla gama de parâmetros de cristalização. As placas são monitoradas usando modalidades de imagem de última geração ao longo de 6 semanas para fornecer informações sobre o crescimento de cristais, bem como para distinguir com precisão golpes de cristal valiosos. Além disso, a implementação de um algoritmo de pontuação de inteligência artificial treinado para identificar ocorrências de cristais, juntamente com uma interface de código aberto e amigável para visualização de imagens experimentais, agiliza o processo de análise de imagens de crescimento de cristais. Aqui, os principais procedimentos e instrumentação são descritos para a preparação dos coquetéis e placas de cristalização, imageando as placas e identificando acertos de forma a garantir a reprodutibilidade e aumentar a probabilidade de cristalização bem-sucedida.
Introdução
Mesmo em uma era de enorme progresso nos métodos de biologia estrutural, a cristalografia de raios X continua a ser um método confiável e popular para gerar modelos estruturais de macromoléculas de alta qualidade. Mais de 85% de todos os modelos estruturais tridimensionais depositados no Protein Data Bank (PDB) são de métodos estruturais baseados em cristais (em janeiro de 2023). 1 Além disso, a cristalografia de raios X permanece indispensável para a resolução de estruturas proteína-ligante, um componente crucial do processo de descoberta e desenvolvimento defármacos 2. Apesar da cristalização de proteínas ter permanecido a técnica dominante de biologia estrutural por mais de meio século, métodos para prever a probabilidade de cristalização com base em propriedades físicas3 ou sequência 4,5 ainda estão em sua infância.
A previsão das condições de cristalização é ainda mais obscura; Progressos limitados têm sido feitos para prever prováveis condições de cristalização mesmo para proteínas modelo 6,7. Outros estudos têm tentado identificar condições de cristalização baseadas na homologia de proteínas e condições extraídas da PDB 8,9,10. O poder preditivo a ser encontrado no PDB é limitado, no entanto, como apenas as condições finais e bem-sucedidas de cristalização são depositadas, o que, por necessidade, perde os experimentos de otimização, muitas vezes extensos, necessários para ajustar o crescimento de cristais. Além disso, muitas entradas PDB carecem de metadados contendo esses detalhes, incluindo as fórmulas do coquetel, o formato de cristalização, a temperatura e o tempo para cristalizar11,12. Portanto, para muitas proteínas de interesse, a maneira mais acessível de determinar as condições de cristalização é experimentalmente, usando o maior número possível de condições em uma ampla gama de possibilidades químicas.
Várias abordagens para tornar a triagem de cristalização o mais frutífera e completa possível têm sido exploradas com grande efeito, incluindo matrizes esparsas 13, triagem fatorial incompleta 14, aditivos 15,16, semeadura 17 e agentes nucleantes 18. O National HTX Center do Hauptman-Woodward Medical Research Institute (HWI) desenvolveu um pipeline eficiente para triagem de cristalização usando a abordagem de microlote sob óleo19, que utiliza manuseio automatizado de líquidos e modalidades de imagem para agilizar a identificação das condições iniciais de cristalização usando volumes comparativamente mínimos de amostra e coquetel (Figura 1). O conjunto de 1.536 coquetéis exclusivos são baseados em condições previamente determinadas como propícias ao crescimento de cristais de proteína e são projetados para serem quimicamente diversos, a fim de amostrar uma ampla gama de condições de cristalização possíveis20,21,22. A ampla amostragem das condições de cristalização aumenta a probabilidade de observar um ou mais eletrodos de cristalização.
Poucas análises formais de quantas condições são necessárias para o rastreamento têm aparecido na literatura. Um estudo focalizou o layout amostral de diferentes telas e verificou que a amostragem aleatória de componentes (semelhante a um fatorial incompleto) representou o método de amostragem mais completo e eficiente23. Outro estudo de triagem observou que houve inúmeros casos em que a tela muito completa de 1.536 produziu apenas um único acerto de cristal24, e um estudo muito recente destacou que a maioria das telas comerciais subestima o espaço de cristalização conhecido por estar associado a acertos de triagem25. Nem todos os condutores de cristalização produzirão um cristal de qualidade de difração adequado para coleta de dados devido à desordem inerente dentro do cristal, limitações de difração ou falhas cristalinas; Portanto, fundir uma rede mais ampla para condições tem o benefício adicional de fornecer formas alternativas de cristal para otimização.
O formato dos experimentos de cristalização de proteínas também tem impacto no sucesso da tela. A difusão de vapor é a configuração mais comumente usada para aplicações de cristalização de alto rendimento e é utilizada em centros de cristalização de última geração, incluindo os centros de triagem de alto rendimento EMBL Hamburg e Institut Pasteur26,27,28. O HTX Center utiliza o método de microbatch-under-oil; Embora menos utilizado, é um método robusto que minimiza o consumo de amostras e coquetéis de cristalização20,21,22. Uma vantagem do método de microlote sob óleo, particularmente quando se usa um óleo de parafina de alta viscosidade, é que apenas uma pequena evaporação ocorre dentro da gota durante o experimento, o que significa que a concentração de equilíbrio é alcançada após a mistura de gotas. Se resultados positivos de cristalização são observados no método de microbatch-under-oil, a reprodução dessas condições é tipicamente mais direta do que em configurações de difusão de vapor, nas quais a cristalização ocorre em algum ponto indefinido durante o equilíbrio entre a gota de cristalização e o reservatório. A reprodutibilidade de acertos é desejável para abordagens de cristalização de alto rendimento, que produzem cristais de proteína proibitivamente minúsculos que normalmente precisam ser otimizados para experimentos de raios X de cristal único.
A tela de cristalização de alto rendimento para proteínas solúveis é composta por coquetéis preparados internamente, telas comerciais prontas e telas comerciais modificadas internamente22. Os coquetéis foram inicialmente desenvolvidos utilizando a estratégia fatorial incompleta utilizando coquetéis de cristalização previamente bem sucedidos20. Os reagentes na tela que estão comercialmente disponíveis incluem arranjos de polímeros, sais de cristalização, PEG, e combinações de íons e telas que utilizam matriz esparsa e abordagens fatoriais incompletas. Há também reagentes que são modificados antes da inclusão na tela: uma tela aditiva, uma tela de pH e tampão, uma tela de aditivo líquido iônico e uma tela de polímero.
O poder das condições e estratégias de cristalização conhecidas foi aproveitado nos 1.536 coquetéis de cristalização, juntamente com os benefícios do sistema de microlote sob óleo para gerar um oleoduto que emprega manuseio automatizado de líquidos, imagens automatizadas de campo brilhante e imagens não lineares de segunda ordem de cristais quirais (SONICC). A automação do manuseio de líquidos e da geração de imagens oferece os benefícios de menos horas de laboratório molhado e maior reprodutibilidade. A natureza de alto rendimento da triagem automatizada de cristalização requer a automação do processo de monitoramento do crescimento de cristais. Esses avanços são alcançados com tecnologias de imagem de última geração para auxiliar na identificação de impactos positivos de cristais. Tanto a imagem padrão de campo claro de placas, bem como métodos de multifótons para detecção aprimorada, são usados através de um sistema de imagem de cristal com SONICC (Figura 2). SONICC combina microscopia de segunda geração harmônica (SHG)29 e microscopia de fluorescência excitada de dois fótons no ultravioleta (UV-TPEF)30 para detectar cristais muito pequenos, bem como aqueles obscurecidos por precipitado. A imagem SONICC informa se os poços contêm proteínas (via UV-TPEF) e cristais (via SHG). Além da identificação positiva dos cristais de proteínas, informações adicionais também podem ser obtidas usando métodos de imagem de última geração. A imagem somente com coquetel antes da adição da amostra serve como um controle negativo; Essas imagens podem identificar a aparência do poço antes da adição da amostra, inclusive em termos de cristais de sal e detritos. Além disso, as imagens SHG e UV-TPEF ajudam a diferenciar cristais de proteína de cristais de sal e podem ser usadas para visualizar material complexado proteína-ácido nucleico31.
Experimentos de cristalização de alto rendimento submetidos a monitoramento repetido via imagem resultam em um volume muito grande de imagens que necessitam de exame. Métodos automatizados de pontuação de cristais foram desenvolvidos para reduzir a carga sobre o usuário e aumentar a probabilidade de identificar acertos positivos de cristal. O HTX Center participou do desenvolvimento do algoritmo de pontuação MAchine Recognition of Crystallization Outcomes (MARCO), uma arquitetura de rede neural convolucional profunda treinada desenvolvida por um consórcio de parceiros acadêmicos, sem fins lucrativos, governamentais e da indústria para classificar imagens de poços de campo brilhante32. O algoritmo foi treinado em quase meio milhão de imagens de campo brilhante de experimentos de cristalização de várias instituições usando diferentes métodos de cristalização e diferentes imageadores. O algoritmo produz um escore probabilístico indicando se uma determinada imagem se enquadra em quatro classes de imagens possíveis: "cristalina", "clara", "precipitada" e "outra". O MARCO tem uma precisão de classificação relatada de 94,5%. A detecção de cristais é aprimorada com um software que implementa o algoritmo e fornece uma interface gráfica do usuário (GUI) para visualização de imagens simples e acessível, habilitada com os recursos de pontuação habilitados para IA32,33. A GUI MARCO Polo foi projetada para funcionar perfeitamente com a configuração do sistema de gerenciamento de imagens e dados no HTX Center para identificar acertos na tela de 1.536 poços, com envolvimento humano para examinar a saída de listas classificadas. Além disso, como software de código aberto disponível no GitHub, a GUI está prontamente disponível para modificação para refletir as necessidades específicas de outros grupos de laboratório.
Aqui, o processo de criação de um experimento de microlote sob óleo de alto rendimento usando manuseio robótico de líquidos para fornecer o coquetel e a proteína é descrito. O HTX Center possui uma gama única de instrumentação e recursos que não são encontrados em outras instituições, com o objetivo de fornecer serviços de triagem e recursos educacionais aos usuários interessados. Demonstrar os métodos e as capacidades das técnicas de alto rendimento habilitadas para robótica permitirá que a comunidade tenha conhecimento das tecnologias disponíveis e tome decisões para seus próprios esforços de determinação de estrutura.
Protocolo
1. Preparação ou compra de coquetéis para dezesseis blocos de poço profundo de 96 poços
- Prepare coquetéis químicos gerados internamente distribuindo em blocos de poço profundo (DW) de 96 poços. Use um manipulador robótico de líquidos para dispensar e misturar soluções estoque de sais, tampões, polímeros e água.
- Prepare coquetéis químicos modificados internamente usando um manipulador robótico de líquidos ou pipeta multicanal para adicionar componentes adicionais às telas de bloco DW de 96 poços que foram compradas comercialmente.
- Compre blocos DW disponíveis comercialmente.
- Armazene blocos DW de 96 poços rotulados a -20 °C por 12-18 meses.
NOTA: Os cocktails preparados no passo 1.1. e 1.2. preencher blocos DW de 96 poços 10/16 e blocos DW de 96 poços 5/16 são usados como comprados. Um bloco DW de 96 poços na tela é configurado no momento da dispensação da placa de 1.536 poços para evitar a precipitação da tela de aditivo (ver seção 3).
2. Distribuindo os coquetéis para pratos de 384 poços
- Descongele os blocos DW de 96 poços a 4 °C durante a noite. Levar à temperatura ambiente (20-23 °C) antes de iniciar a preparação das placas de 384 poços.
NOTA: A temperatura ambiente é adequada para o preparo dos pratos de coquetel. A principal preocupação no preparo desses pratos é evitar precipitados, que podem entupir dispositivos de manuseio de líquidos e levar a mudanças imprevisíveis nas concentrações dos ingredientes do coquetel. - Misture bem os blocos por inversão, conforme necessário, para dissolver qualquer precipitado opaco persistente. Se algum poço contiver precipitado, aqueça os blocos a 30 °C para dissolver.
- Forneça 50 μL de solução de coquetel de quatro blocos DW de 96 poços para uma placa de 384 poços usando um robô de manuseio de líquidos equipado com uma seringa de 96 ou cabeça de pipetador. Os quatro blocos DW de 96 poços são estampados na placa de 384 poços de tal forma que os quadrantes são preenchidos (por exemplo, A1 de 96-DW1 a A1 de 384-placa1, A1 de 96-DW2 a B1 de 384-placa1, etc.) (Figura 3).
- Entregue 15 dos 16 blocos DW de 96 poços a placas de 384 poços para armazenamento.
- Armazene as placas de 384 poços a -20 °C por até 6 meses para uso na preparação das placas de 1.536 poços.
3. Preparar as placas de 1.536 poços com óleo e coquetéis de cristalização
- Forneça 5 μL de óleo de parafina para cada poço de uma placa de 1.536 poços usando um sistema robótico de manuseio de líquidos com capacidade de aspiração e entrega lentas. Conservar as placas de óleo a 4 °C até 6 meses.
- Descongele as placas de 384 poços da seção 2 a 4 °C durante a noite. Inverter as placas para misturar as soluções e dissolver o precipitado. Incubar as placas a 30 °C para dissolver os precipitados persistentes.
- Para preparar os componentes da tela aditiva, use o bloco DW final de 96 poços contendo 0,1 M HEPES pH 6,8, 30% PEG3350 para misturar com a tela de aditivo comercial usando um robô de manuseio de líquidos ou uma pipeta multicanal.
- Preparar as soluções da peneira de aditivos dispensando uma mistura 1:1 da solução tamponada de PEG3350 preparada no passo 3.3 e a peneira de aditivos para um volume final de 50 μL na placa apropriada de 384 poços.
- Use um robô de manuseio de líquidos equipado com uma seringa 384 ou cabeça de pipettor para fornecer 200 nL de solução de coquetel em cada poço da placa de 1.536 poços. Eliminar quatro placas de 384 poços na placa de 1.536 poços de modo que os quadrantes sejam preenchidos (por exemplo, A1 da placa de 384 poços 1 a A1 da placa de 1.536 poços, A2 da placa de 384 poços 1 a A3 da placa de 1.536 poços, etc.) (Figura 3).
- Centrifugar as placas a 150 × g durante 5 minutos antes de armazenar a 4 °C durante um período máximo de 4 semanas.
4. Submissão da amostra
- Para enviar uma amostra, envie um e-mail de reserva antes do prazo de reserva para a próxima corrida de triagem. Inclua o número de experimentos de triagem, o nome, o IP e a instituição, bem como quaisquer requisitos especiais de manuseio para a amostra. As sessões de triagem são realizadas aproximadamente uma vez por mês, resultando em 12 corridas anuais.
- Preencha um formulário de envio de amostra antes de enviá-la.
- Para novos usuários, escolha uma senha que será usada para baixar as imagens de cristalização na seção 7.
- Para usuários estabelecidos, use uma senha existente ou altere a senha nesta etapa.
- Submeter a amostra em um tubo de 1,5 mL. Certifique-se de que a macromolécula esteja homogênea e adequadamente concentrada para promover a cristalização. Use um teste de pré-cristalização, tipicamente composto de sulfato de amônio ou PEG 4.000, para investigar a concentração apropriada da amostra, observando se as concentrações da amostra testada resultam em gotas claras ou precipitam34.
NOTA: Os testes de qualidade adequados que podem ser realizados antes do envio da amostra para verificar a pureza e homogeneidade incluem SDS-PAGE, filtração em gel e espalhamento dinâmico de luz (DLS), entre outros. A cristalização pode ser afetada pela presença de impurezas ainda menores. Um volume de amostra de 500 μL é atualmente necessário para configurar uma placa de 1.536 poços. Os testes estão em andamento para diminuir a necessidade de volume de amostra.- Evite usar concentrações de tampão superiores a 50 mM, bem como fosfatos, que podem cristalizar dentro da tela.
- Evitar agentes solubilizadores excessivos, incluindo concentrações de glicerol superiores a 10% p/v.
- Embale a amostra para manter com segurança uma temperatura adequada usando gelo seco, gelo úmido ou embalagens de resfriamento em um recipiente selado.
- Prioridade de amostra de navio durante a noite de segunda a quarta-feira durante a corrida.
- Envie o número de rastreamento por e-mail assim que a amostra for enviada.
5. Configuração da amostra nas placas preparadas de 1.536 poços
- Desembale as amostras e incube imediatamente à temperatura solicitada pelo utilizador.
- Uma vez descongelado, centrifugar a amostra a 10.000 × g por 2 min à temperatura ambiente. Observe visualmente a amostra para identificar a precipitação, a cor e a condição da amostra antes da instalação.
- Aqueça a placa de 1.536 poços a 23 °C e centrifugue a 150 × g por 5 min. Imagem da placa somente de coquetel usando imagens de campo brilhante como um controle negativo.
NOTA: Todas as placas são fotografadas com imagens de campo brilhante antes da configuração da amostra, o que permite a identificação de poços que já possuem cristais ou detritos antes da adição da amostra como um controle negativo. Além disso, permite a identificação de poços nos quais o coquetel de cristalização não foi entregue. O aquecimento da placa à temperatura ambiente elimina a condensação na superfície da placa, levando a imagens nítidas. - Distribua 200 nL de amostra para cada poço na placa de 1.536 poços usando um robô de manuseio de líquidos. Centrifugar a placa a 150 × g e incubar as placas a 4 °C, 14 °C ou 23 °C.
NOTA: Experimentos de microlote sob óleo podem ser configurados manualmente, dispensando a proteína e o coquetel sob o óleo desejado. No entanto, recomenda-se usar pelo menos 1 μL de cada proteína e coquetel para alcançar resultados reprodutíveis.
6. Monitorar placas de 1.536 poços para formação de cristais
- Após a amostra ter sido adicionada às placas de 1.536 poços, imagem com imagens de campo claro no dia 1 e semana 1, semana 2, semana 3, semana 4 e semana 6.
- Realizar imagens SONICC com SHG e UV-TPEF no ponto de tempo de 4 semanas para placas sendo incubadas a 23 °C e no ponto de tempo de 6 semanas para placas sendo incubadas a 14 °C ou 4 °C.
NOTA: O tempo para a imagem SONICC é agendado nos pontos de tempo de 4 semanas e 6 semanas para a placa de microensaio de alto rendimento 1.536 porque, normalmente, os cristais aparecerão nesses pontos de tempo. Para modificação em um microlote de 96 poços sob experimentos de difusão de óleo ou vapor, é aconselhável realizar a imagem SONICC mais cedo na janela de tempo. - Acesse as imagens experimentais que foram transferidas automaticamente para a conta do usuário usando um sistema LIMS interno. Notifique os usuários por meio de um daemon de e-mail htslab automatizado de que a imagem ocorreu.
7. Análise das imagens
- Recupere as imagens de triagem do site ftp HWI para cada arquivo .rar.
NOTA: A saída de imagem da tela 1.536 resulta em vários arquivos contendo imagens de campo brilhante, imagens SHG e imagens UV-TPEF. Cada modalidade de imagem ou ponto de tempo é um arquivo .rar separado. Cada arquivo .rar, quando descompactado, contém uma imagem de cada poço da placa de 1.536 poços em um ponto de tempo específico usando uma modalidade de imagem específica.- Use o FileZilla Client ou outras opções para acessar os dados ftp.
NOTA: O FileZilla Client é a maneira recomendada de gerenciar o grande volume de transferência de arquivos para minimizar falhas computacionais.- Se o FileZilla Client precisar ser instalado no computador do usuário, baixe o software FileZilla.
- Se o FileZilla Client já estiver instalado ou após a instalação, clique no ícone FileZilla para abrir o software.
- Faça login no servidor ftp remoto do FileZilla inserindo o site ftp do host, nome de usuário e senha.
- Baixe os arquivos .rar para o diretório desejado.
- Use o FileZilla Client ou outras opções para acessar os dados ftp.
- Use a GUI de código aberto habilitada para IA para exibir, pontuar e analisar as imagens de cristalização.
NOTA: A GUI pode ser usada na maioria dos sistemas operacionais (SO) Windows, Mac e Linux, e as instruções específicas do sistema operacional para download estão localizadas no site do GitHub. MARCO Polo é uma GUI de código aberto que incorpora metadados da tela de cristalização de alto rendimento 1.536 implementada no HTX Center. Ele está disponível para qualquer pessoa baixar do GitHub para modificação para refletir as necessidades específicas de outros grupos de laboratório.- Abra o arquivo .rar na GUI depois que o arquivo tiver sido baixado (consulte a Figura Suplementar S1).
- Clique em Importar, selecione Imagens no menu suspenso e, em seguida, selecione Do Arquivo/Diretório Rar.
- Clique em Procurar pasta na janela pop-up e, em seguida, navegue até a pasta que contém as imagens.
- Selecione o(s) arquivo(s) desejado(s) e importe para a GUI clicando em Abrir. Aguarde até que o(s) arquivo(s) apareça(m) na janela Caminhos Selecionados . Selecione um ou mais arquivos para baixar na GUI e clique em Importar execuções.
- Visualize a imagem do primeiro poço na janela do Visualizador de Apresentação de Slides na GUI clicando no símbolo de > à esquerda do nome da amostra e, em seguida, selecionando a leitura apropriada clicando duas vezes nele (as leituras são listadas pela data e tipo de campo brilhante da imagem, UV-TPEF ou SHG).
- Amplie a imagem redimensionando toda a janela. A caixa Detalhes da imagem inclui informações sobre a imagem , incluindo as informações de pontuação (vazias até que a leitura tenha sido pontuada). A caixa Detalhes do Coquetel contém metadados sobre os componentes do coquetel .
- Vá para o próximo poço clicando no botão Avançar no painel de navegação ou pressionando a tecla de seta para a direita no teclado. Navegue até um poço específico inserindo o número do poço na janela Por Número do Poço .
- Exiba todas as leituras (daquelas importadas para a GUI) marcando a caixa Mostrar todas as datas .
- Exiba todos os espectros (daqueles importados para a GUI) marcando a caixa Mostrar todos os espectros . Clique no botão Swap Spectrum para visualizar cada imagem de espectro individualmente.
- Abra o arquivo .rar na GUI depois que o arquivo tiver sido baixado (consulte a Figura Suplementar S1).
- Classifique as imagens de cristal usando o algoritmo MARCO destacando primeiro uma execução específica da lista no lado esquerdo da janela. Em seguida, clique no botão Classificar Execução Selecionada . Exiba as informações de pontuação do MARCO na janela Detalhes da imagem depois que uma leitura de imagem for marcada para todos os 1.536 poços.
NOTA: A classificação normalmente levará entre 2 e 5 minutos, dependendo da velocidade do computador e da memória disponível. O algoritmo gera pontuações que classificam o conteúdo em classes "cristalinas", "claras", "precipitadas" ou "outras". Os valores numéricos associados à classificação de cada poço refletem a probabilidade de o poço conter objetos dessa classe.- Visualize um subconjunto das imagens pontuadas marcando a(s) caixa(s) desejada(s) no painel Filtragem de imagem e clicando no botão Enviar filtros . Por exemplo, visualize apenas as imagens classificadas pelo MARCO como cristais marcando as caixas Cristais e MARCO e clicando em Enviar Filtros.
- Pontuar manualmente as imagens de cristal para gerar o conjunto "pontuação humana". Atribua uma pontuação a um poço clicando no botão apropriado (os botões "cristal", "claro", "precipitado" ou "outros" estão localizados no painel Classificação na parte inferior da janela). Alternativamente, use o teclado numérico no teclado para atribuir a pontuação (1 = "cristal", 2 = "claro", 3 = "precipitado", 4 = "outro"). Designar uma imagem marcada por humanos como "favorita" marcando a Favorita? caixa.
NOTA: Visualize apenas as imagens classificadas por um ser humano como cristais, marcando as caixas Cristais e Humanos e clicando em Enviar Filtros. Clicar na caixa Favoritos no painel Filtragem reduz ainda mais as imagens retornadas, retornando apenas as imagens de cristal com pontuação humana que também são favoritas. - Use a guia Visualizador de placas para exibir vários poços ao mesmo tempo. Na segunda guia Visualizador de chapas no painel Controles , selecione 16, 64 ou 96 imagens no menu suspenso na seção Imagens por placa . Use a guia Filtragem de imagem para ocultar imagens que não são de interesse. Selecione a caixa Aplicar filtro para filtrar as imagens.
NOTA: Por exemplo, selecione as caixas "humano" e "cristal", e apenas os poços que foram marcados como um cristal por um humano serão facilmente visíveis.- Navegue na guia Plate Viewer , clicando no botão Next para visualizar o próximo conjunto de imagens 16/64/96. Por padrão, as imagens marcadas como cristais são vermelhas, as pontuadas como claras são azuis, as pontuadas como precipitadas são verdes e as pontuadas como outras são laranjas. Altere as cores usando os menus suspensos.
- Selecione as informações a serem exibidas nos poços marcando várias caixas na guia Rótulos .
- Clique em Save View para salvar um arquivo de imagem da visualização atual.
- Clique em Swap Spectrum para alternar entre imagens de campo brilhante, SHG e UV-TPEF para a imagem de vários poços.
- Clique em Exportar e selecione o tipo de arquivo apropriado no menu suspenso para exportar os arquivos pontuados para uso em outros programas.
NOTA: Os arquivos CSV (valores separados por vírgula) são compatíveis com programas de planilha, como o Microsoft Excel ou o Planilhas Google. Os arquivos JSON (JavaScript Object Notation) podem ser abertos com a maioria dos editores de texto. PPTX (PowerPoint Presentation) pode ser usado para exibir imagens do Polo, incluindo uma comparação de imagens de campo brilhante, UV-TPEF e SHG. Os arquivos são salvos no formato .xtal para serem reabertos na GUI do MARCO Polo.- Salve um arquivo de formato .xtal clicando em Arquivo na parte superior da página e selecionando Salvar execução ou Salvar executar como. Forneça um nome de arquivo e um local de diretório.
- Abra arquivos de formato .xtal clicando em Importar e selecionando Imagens e, em seguida, De Execução Salva. Procure o local apropriado do arquivo, clique no nome do arquivo e, em seguida, clique em Abrir.
Resultados Representativos
Os resultados do experimento de triagem de cristais de 1.536 poços consistem em sete conjuntos completos de imagens de campo claro coletadas no dia 0 (controle negativo), dia 1, semana 1, semana 2, semana 3, semana 4 e semana 6 (Figura 4). As imagens SONICC são coletadas no ponto de tempo de 4 semanas para placas incubadas a 23 °C e no ponto de tempo de 6 semanas para placas incubadas a 4 °C ou 14 °C. No total, uma vez que uma amostra tenha sido enviada, os usuários podem antecipar ter suas placas configuradas dentro de 1 dia após a chegada. As imagens serão carregadas à medida que forem coletadas. O experimento de triagem de cristalização é concluído após 6 semanas.
A configuração da placa de 1.536 poços permite que todos os experimentos de triagem sejam realizados dentro da mesma placa, limitando o consumo de amostra e facilitando a aquisição de imagens e a comparação direta entre as modalidades de imagem. Resultados representativos para o curso temporal do crescimento do cristal para uma única condição de coquetel são mostrados na Figura 4. A imagem automatizada de placas ao longo do experimento permite a identificação de cristais de crescimento rápido e lento por imagens de campo claro. As imagens de UV-TPEF e SHG permitem validação cruzada dos acertos observados pela imagem de campo claro e indicam que os cristais observados são proteináceos e cristalinos, respectivamente (Figura 5A,B). Além disso, a imagem SONICC permite a identificação de cristais que são visualmente obscurecidos por precipitados ou filmes (Figura 5C) ou microcristais que podem ser confundidos com precipitados (Figura 5D). Para alguns cristais, a falta de sinal de SHG não é desqualificante, pois alguns grupos de pontos não produzem um sinal de SHG35,36, como exemplificado pelo cristal tetragonal de taumatina na Figura 5C. Por outro lado, a falta de sinal UV-TPEF para proteínas sem resíduos de triptofano deve ser antecipada. A observação dos sinais UV-TPEF e SHG também facilita a identificação de cristais de sal não proteicos, que aparecerão em campo claro e exibirão um forte sinal positivo de SHG, mas não terão um sinal UV-TPEF (Figura 5E).
A análise de imagem para a configuração da placa é simplificada com a GUI MARCO Polo, que também agrupa a transferência de dados ftp dos servidores HWI (como uma alternativa à transferência de arquivos com o FileZilla). A GUI MARCO Polo permite a visualização fácil de placas e imagens navegáveis e realiza pontuação computacional de imagens usando o algoritmo MARCO para que os resultados das imagens possam ser rapidamente baixados, visualizados e analisados a partir do HTX Center. O algoritmo de pontuação MARCO, conforme implementado na GUI do MARCO Polo, é capaz de pontuar imagens de toda a placa de 1.536 poços em menos de 5 minutos. As imagens sinalizadas como cristalinas pelo algoritmo MARCO podem ser posteriormente classificadas pela GUI do Polo para exibição. Como o algoritmo MARCO foi otimizado para identificação de cristais e minimização de falsos negativos para não perder nenhum acerto positivo, a pontuação pode resultar em sinalizadores falsos positivos. No entanto, a capacidade do MARCO de limitar o conjunto de imagens que precisam ser examinadas, concentrando a atenção nos poços com alta probabilidade de conter cristais, resulta em uma redução substancial na carga de processamento de dados para os usuários. A implementação conveniente do algoritmo na plataforma de visualização amigável MARCO Polo, com sua capacidade de classificar imagens com base nas pontuações MARCO, melhora muito a capacidade do usuário de analisar o conjunto de dados rapidamente e determinar com precisão os acertos de cristal.
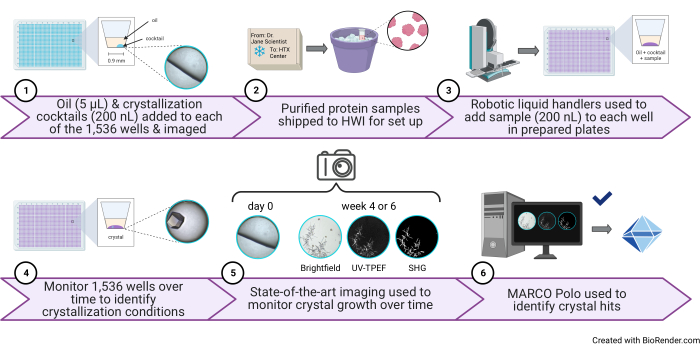
Figura 1: Esquema de um experimento de triagem de cristalização de 1.536 poços de alto rendimento realizado no HTX Center. (1) Nesta etapa, 5 μL de óleo de parafina e 200 nL de coquetel são adicionados a cada poço (protocolo passo 3.1 e passo 3.5). Uma ilustração de desenho animado de um poço contendo apenas óleo e coquetel e uma imagem representativa são mostrados à direita. (2) As amostras chegam ao HTX Center (protocolo passo 5.1). 3) Nesta etapa, 200 nL de amostra são adicionados a cada poço (passo do protocolo 5.4). (4) Todos os 1.536 poços são monitorados ao longo do tempo usando imagens de campo claro, 5) bem como as modalidades UV-TPEF e SHG (etapa 6 do protocolo). 6) A GUI de código aberto habilitada para IA é usada para visualizar, pontuar e analisar as imagens de cristalização (etapa 7 do protocolo). Abreviações: HTX = cristalização de alto rendimento; UV-TPEF = fluorescência excitada por UV-dois fótons; SHG = segunda geração harmônica; IA = inteligência artificial; GUI = interface gráfica do usuário. Clique aqui para ver uma versão maior desta figura.

Figura 2: Placas únicas de 1.536 poços contendo experimentos de triagem, obtidas com imagens de campo claro, UV-TPEF e SHG. As placas de 1.536 poços são mostradas com um centavo americano para escala (topo). Cada experimento de triagem é fotografado uma vez antes da configuração e seis vezes após a adição da amostra com imagens de campo claro (sete conjuntos de imagens de campo claro total, à esquerda). As placas são submetidas a exames de UV-TPEF (centro) e SHG (direita) em 4 semanas ou 6 semanas. Abreviações: UV-TPEF = UV-fluorescência excitada de dois fótons; SHG = segunda geração harmônica. Clique aqui para ver uma versão maior desta figura.
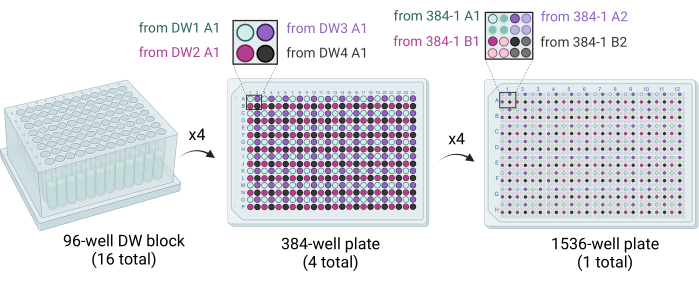
Figura 3: Esquema mostrando como as placas de 1.536 poços são geradas. Dezesseis blocos DW de 96 poços são usados para carimbar quatro placas de 384 poços, com cada quadrante de cada placa de 384 poços preenchido por coquetéis de cristalização. Quatro blocos DW de 96 poços preenchem uma placa de 384 poços (meio). Quatro placas de 384 poços são usadas para carimbar a placa única de 1.536 poços (à direita). Abreviação: DW = poço profundo. Clique aqui para ver uma versão maior desta figura.
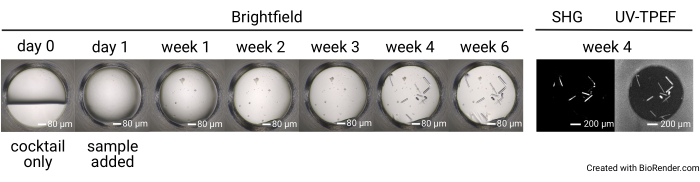
Figura 4: Curso temporal representativo de um único poço em um experimento de triagem de 1.536 poços. As placas são fotografadas antes da configuração da amostra (dia 0), bem como com imagens de campo claro no dia 1, semana 1, semana 2, semana 3, semana 4 e semana 6. As placas incubadas a 23 °C são imageadas com SONICC na semana 4. Barras de escala = 80 μm (campo claro), 200 μm (SHG, UV-TPEF). Abreviações: SONICC = imagem não linear de segunda ordem de cristais quirais; UV-TPEF = fluorescência excitada por UV-dois fótons; SHG = segunda geração harmônica. Clique aqui para ver uma versão maior desta figura.
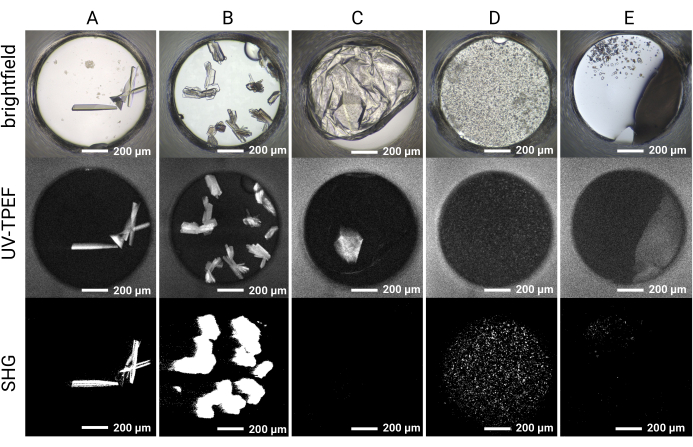
Figura 5: Resultados de imagem representativos para os experimentos de triagem de cristais HT 1.536. Os resultados de imagem de campo brilhante, UV-TPEF e SHG são mostrados para cinco poços de exemplo. (A,B) Os cristais de proteína observados por imagens de campo claro, UV-TPEF e SHG são claramente aparentes em todas as três modalidades de imagem. (C) Um cristal de proteína obscurecido por filme em imagem de campo claro é visível por imagem UV-TPEF; o cristal não é observado pela imagem de SHG devido à incompatibilidade do grupo de pontos. (D) Exemplo de microcristais verificados por imagens UV-TPEF e SHG que podem ser considerados precipitados. (E) Exemplo de cristais de sal que aparecem cristalinos por imagem de campo brilhante e SHG, mas não exibem um sinal UV-TPEF. Barras de escala = 200 μm. Diâmetro do poço = 0,9 mm. Abreviações: UV-TPEF = UV-fluorescência excitada de dois fótons; SHG = segunda geração harmônica. Clique aqui para ver uma versão maior desta figura.
Figura suplementar S1: Abrindo arquivos de imagem no MARCO Polo. Os arquivos de imagem podem ser abertos dentro da GUI do MARCO Polo navegando até a opção Importar | Guia Imagens na parte superior (a). Observe que os arquivos também podem ser transferidos através da ferramenta From FTP diretamente no MARCO Polo (a) ou podem ser transferidos via FileZilla conforme descrito na etapa de protocolo 7.2. Para importar arquivos que já foram baixados, selecione Imagens | Do arquivo/diretório Rar. Na janela pop-up exibida, selecione Procurar pasta (b) e navegue até o diretório de arquivos onde os arquivos de imagem de placa são salvos. Quando os arquivos estiverem na janela Caminhos Selecionados (c), realce um arquivo e clique em Importar Execuções (d). A GUI do MARCO Polo identificará os metadados corretos do Cocktail File para importar com as imagens. Clique aqui para baixar este arquivo.
Discussão
O método descreve um pipeline de alto rendimento para triagem de cristalização de proteínas que requer apenas 500 μL de amostra para 1.536 experimentos individuais de cristalização no formato de microlote sob óleo. O pipeline conta com robótica de manuseio de líquidos para ajudar de forma rápida e reprodutível a configuração experimental, bem como o recurso de análise de imagem computacional MARCO Polo, que é personalizado para analisar imagens de placas de 1.536 poços usando o algoritmo MARCO para identificar e isolar ocorrências de cristais.
O pequeno volume de gotas de triagem individuais (total de 400 nL com uma proporção de amostra :coquetel de 1:1) significa que volumes de amostra extremamente pequenos são necessários para identificar condições positivas de cristalização. Esses pequenos tamanhos de gotas necessariamente produzem pequenos cristais que não podem ser pescados pelo looping tradicional. Foram desenvolvidos métodos de colheita das 1.536 placas37; Adicionalmente, as placas com cristais têm sido utilizadas diretamente em fontes síncrotron para coleta de dados in situ 38. Se um método robusto para a colheita desses cristais fosse desenvolvido, os avanços na tecnologia síncrotron e feixes microfocados permitiriam ainda mais a obtenção de conjuntos de dados úteis. Adicionalmente, os cristais obtidos podem potencialmente ser utilizados como sementes para esforços de otimização.
A imagem SONICC é claramente vantajosa na identificação de cristais de proteína pequenos e cristais de proteína escondidos sob o precipitado. Apesar dessas vantagens, nem todos os tipos de amostra são passíveis de imagens por SHG e UV-TPEF. Por exemplo, proteínas com poucos ou nenhum resíduo aromático de triptofano mostrarão um sinal UV-TPEF ambíguo. Além disso, cristais em grupos espaciais específicos, incluindo grupos centrossimétricos ou grupo de pontos 432, não serão detectados por imagens de SHG. Amostras com fluoróforos às vezes interferem com o sinal de SHG, resultando no cancelamento do sinal ou aumento da intensidade, o que significa que uma interpretação cuidadosa dos sinais de SHG é necessária para proteínas contendo metais e proteínas contendo metades contendo metais. No entanto, em muitos casos, é possível racionalizar a ausência de um sinal SHG ou UV-TPEF, e a falta desses sinais não deve necessariamente descartar a presença de um cristal de proteína.
O formato microbatch-under-oil fornece uma alternativa ao método de difusão de vapor mais comum usado para cristalografia de alto rendimento. É importante ressaltar que o formato de cristalização impacta a identificação de acertos39, o que fornece uma justificativa para o uso de diferentes formatos de cristalização para esforços de triagem de alto rendimento. Imagens automatizadas e modalidades habilitadas para SONICC auxiliam na rápida identificação de cristais de proteína ao longo do curso experimental de 6 semanas. Finalmente, a GUI MARCO Polo permite que os usuários analisem rapidamente imagens de 1.536 condições para identificar poços de impacto promissores para otimização. Os recursos do HTX Center, incluindo a configuração experimental de alto rendimento habilitada para robótica, juntamente com as ferramentas computacionais e de imagem de última geração para análises, fornecem uma grande contribuição para a comunidade de biologia estrutural, capacitando os pesquisadores a abordar efetivamente um gargalo primário no trabalho estrutural baseado em cristais: encontrar condições de cristalização.
Divulgações
Os autores não têm interesses financeiros concorrentes ou outros conflitos de interesse.
Agradecimentos
Gostaríamos de estender nossa gratidão aos nossos usuários por confiarem suas preciosas amostras a nós para triagem de cristais, bem como por fornecer feedback crítico e solicitações que nos ajudaram a refinar e desenvolver nossos recursos para melhor servir a comunidade de biologia estrutural. Também gostaríamos de agradecer a Ethan Holleman, Dra. Lisa J Keefe e Dra. Erica Duguid, que conduziram o desenvolvimento da GUI MARCO Polo. Gostaríamos de agradecer aos colegas do HWI pelo apoio e sugestões, especialmente à Dra. Diana CF Monteiro. Agradecemos o apoio financeiro dos Institutos Nacionais de Saúde, R24GM141256.
Materiais
| Name | Company | Catalog Number | Comments |
| 1536 Well Imp@ct LBR LoBase | Greiner Bio-One | 790 801 | |
| Acetic acid | Hampton Research | HR2-853 | |
| AlumaSeal II Sealing Film | Hampton Research | HR8-069 | |
| Ammonium bromide | Molecular Dimensions | MD2-100-247 | |
| Ammonium chloride | Hampton Research | HR2-691 | |
| Ammonium hydroxide | Hampton Research | HR2-855 | |
| Ammonium nitrate | Hampton Research | HR2-665 | |
| Ammonium phosphate dibasic | Hampton Research | HR2-629 | |
| Ammonium phosphate monobasic | Hampton Research | HR2-555 | |
| Ammonium sulfate | Hampton Research | HR2-541 | |
| Ammonium thiocyanate | Molecular Dimensions | MD2-100-301 | |
| Bicine pH 9.0 | Hampton Research | HR2-723 | |
| Bis-tris propane pH 7.0 | Hampton Research | HR2-993-08 | |
| Calcium acetate | Hampton Research | HR2-567 | |
| Calcium chloride dihydrate | Hampton Research | HR2-557 | |
| CAPS pH 10.0 | Rigaku Reagents | none given | |
| ClearSeal Film | Hampton Research | HR4-521 | |
| Cobalt sulfate heptahydrate | Molecular Dimensions | MD2-100-42 | |
| Crystal Screen HT screen | Hampton Research | HR2-130 | |
| Formulator | Formulatrix | ||
| Glycerol | Hampton Research | HR2-623 | |
| Gryphon liquid handling robot | Art Robbins Instruments | ||
| HEPES pH 7.0 | Hampton Research | HR2-902-03 | |
| HEPES pH 7.5 | Hampton Research | HR2-902-08 | |
| HWI HTX Center sample submission form | https://hwi.buffalo.edu/high-throughput-crystallization-screening-center-sample-submission-form/ | ||
| Hydrochloric acid | Hampton Research | HR2-581 | |
| Index HT screen | Hampton Research | HR2-134 | |
| Ionic Liquid screen | Hampton Research | HR2-214 | |
| Lithium bromide | Molecular Dimensions | MD2-100-312 | |
| Lithium chloride | Hampton Research | HR2-631 | |
| Lithium sulfate monohydrate | Hampton Research | HR2-545 | |
| Magnesium acetate tetrahydrate | Hampton Research | HR2-561 | |
| Magnesium chloride hexahydrate | Hampton Research | HR2-559 | |
| Magnesium nitrate hexahydrate | Hampton Research | HR2-657 | |
| Magnesium sulfate heptahydrate | Hampton Research | HR2-821 | |
| Manganese chloride tetrahydrate | Millipore Sigma | 63535-50G | |
| Manganese sulfate monohydrate | Molecular Dimensions | MD2-100-310 | |
| MARCO Polo GUI download | https://hauptman-woodward.github.io/Marco_Polo/ | ||
| Matrix Platemate 2 x 3 liquid handling robot | Thermo Scientific | ||
| MES pH 6.0 | Hampton Research | HR2-943-09 | |
| Mosquito liquid handling robot | SPTLabtech | ||
| Paraffin Oil/White Mineral Oil Saybolt Viscosity 340-365 at 100 °F | Sigma Aldrich | PX0045-3 | |
| PEG 1000 | Hampton Research | HR2-523 | |
| PEG 2000 | Hampton Research | HR2-592 | |
| PEG 20000 | Hampton Research | HR2-609 | |
| PEG 3350 | Hampton Research | HR2-527 | |
| PEG 400 | Hampton Research | HR2-603 | |
| PEG 4000 | Hampton Research | HR2-529 | |
| PEG 6000 | Hampton Research | HR2-533 | |
| PEG 8000 | Hampton Research | HR2-535 | |
| PEG/Ion HT screen | Hampton Research | HR2-139 | |
| PEGRx HT screen | Hampton Research | HR2-086 | |
| Plate reservations | htslab@hwi.buffalo.edu | ||
| Potassium acetate | Hampton Research | HR2-671 | |
| Potassium bromide | Hampton Research | HR2-779 | |
| Potassium carbonate | Molecular Dimensions | MD2-100-311 | |
| Potassium chloride | Hampton Research | HR2-649 | |
| Potassium nitrate | Hampton Research | HR2-663 | |
| Potassium phosphate dibasic | Hampton Research | HR2-635 | |
| Potassium phosphate-monobasic | Hampton Research | HR2-553 | |
| Potassium phosphate-tribasic | Molecular Dimensions | MD2-100-309 | |
| Potassium thiocyanate | Hampton Research | HR2-695 | |
| Rock Imager 1000 with SONICC | Formulatrix | ||
| Rock Imager 54 | Formulatrix | ||
| Rubidium chloride | Millipore Sigma | R2252-10G | |
| SaltRx HT screen | Hampton Research | HR2-136 | |
| Silver Bullets screen | Hampton Research | HR2-096 | |
| Slice pH screen | Hampton Research | HR2-070 | |
| Sodium acetate pH 5.0 | Hampton Research | HR2-933-15 | |
| Sodium bromide | Hampton Research | HR2-699 | |
| Sodium chloride | Hampton Research | HR2-637 | |
| Sodium citrate pH 4.2 | Hampton Research | HR2-935-01 | |
| Sodium citrate pH 5.6 | Hampton Research | HR2-735 | |
| Sodium hydroxide | Hampton Research | HR2-583 | |
| Sodium molybdate dihydrate | Molecular Dimensions | MD2-100-207 | |
| Sodium nitrate | Hampton Research | HR2-661 | |
| Sodium phosphate monobasic | Hampton Research | HR2-551 | |
| Sodium thiosulfate pentahydrate | Molecular Dimensions | MD-100-307 | |
| StockOptions Polymer screen | Hampton Research | HR2-227 | |
| Tacsimate pH 7 | Hampton Research | HR2-755 | |
| TAPS pH 9.0 | bioWORLD | 40121071 | |
| Tris pH 8 | Hampton Research | HR2-900-11 | |
| Tris pH 8.5 | Hampton Research | HR2-725 | |
| ViaFLO 384 | Integra | ||
| ViaFLO 384 384 channel pipettor head (0.5-12.5µL) | Integra | ||
| ViaFLO 384 96 channel pipettor head (300µL) | Integra | ||
| Zinc acetate dihydrate | Hampton Research | HR2-563 |
Referências
- PDB data distribution by experimental method and molecular type. RCSB Protein Data Bank Available from: https://www.rcsb.org/stats/summary (2022)
- Maveyraud, L., Mourey, L. Protein X-ray crystallography and drug discovery. Molecules. 25 (5), 1030 (2020).
- Dupeux, F., Röwer, M., Seroul, G., Blot, D., Márquez, J. A. A thermal stability assay can help to estimate the crystallization likelihood of biological samples. Acta Crystallographica Section D: Biological Crystallography. 67 (11), 915-919 (2011).
- Elbasir, A., et al. DeepCrystal: A deep learning framework for sequence-based protein crystallization prediction. Bioinformatics. 35 (13), 2216-2225 (2019).
- Zucker, F. H., et al. Prediction of protein crystallization outcome using a hybrid method. Journal of Structural Biology. 171 (1), 64-73 (2010).
- George, A., Wilson, W. W. Predicting protein crystallization from a dilute solution property. Acta Crystallographica Section D: Biological Crystallography. 50 (4), 361-365 (1994).
- Jia, Y., Liu, X. -. Y. From surface self-assembly to crystallization: prediction of protein crystallization conditions. The Journal of Physical Chemistry B. 110 (13), 6949-6955 (2006).
- Slabinski, L., et al. XtalPred: A web server for prediction of protein crystallizability. Bioinformatics. 23 (24), 3403-3405 (2007).
- Abrahams, G. J., Newman, J. BLASTing away preconceptions in crystallization trials. Acta Crystallographica Section F: Structural Biology Communications. 75 (3), 184-192 (2019).
- Rosa, N., et al. Tools to ease the choice and design of protein crystallisation experiments. Crystals. 10 (2), 95 (2020).
- Newman, J., et al. On the need for an international effort to capture, share and use crystallization screening data. Acta Crystallographica Section F: Structural Biology and Crystallization Communications. 68 (3), 253-258 (2012).
- Lynch, M. L., Dudek, M. F., Bowman, S. E. A searchable database of crystallization cocktails in the PDB: analyzing the chemical condition space. Patterns. 1 (4), 100024 (2020).
- Jancarik, J., Kim, S. -. H. Sparse matrix sampling: a screening method for crystallization of proteins. Journal of Applied Crystallography. 24 (4), 409-411 (1991).
- Carter, C. W. Efficient factorial designs and the analysis of macromolecular crystal growth conditions. Methods. 1 (1), 12-24 (1990).
- McPherson, A., Cudney, B. Searching for silver bullets: An alternative strategy for crystallizing macromolecules. Journal of Structural Biology. 156 (3), 387-406 (2006).
- McPherson, A., Nguyen, C., Cudney, R., Larson, S. The role of small molecule additives and chemical modification in protein crystallization. Crystal Growth & Design. 11 (5), 1469-1474 (2011).
- Luft, J. R., DeTitta, G. T. A method to produce microseed stock for use in the crystallization of biological macromolecules. Acta Crystallographica Section D: Biological Crystallography. 55 (5), 988-993 (1999).
- Thakur, A. S., et al. Improved success of sparse matrix protein crystallization screening with heterogeneous nucleating agents. PLoS One. 2 (10), 1091 (2007).
- Chayen, N. E., Stewart, P. D. S., Blow, D. M. Microbatch crystallization under oil-a new technique allowing many small-volume crystallization trials. Journal of Crystal Growth. 122 (1-4), 176-180 (1992).
- Luft, J. R., et al. A deliberate approach to screening for initial crystallization conditions of biological macromolecules. Journal of Structural Biology. 142 (1), 170-179 (2003).
- Luft, J. R., Snell, E. H., DeTitta, G. T. Lessons from high-throughput protein crystallization screening: 10 years of practical experience. Expert Opinion on Drug Discovery. 6 (5), 465-480 (2011).
- Lynch, M. L., Snell, M. E., Potter, S. A., Snell, E. H., Bowman, S. E. 20 years of crystal hits: Progress and promise in ultrahigh-throughput crystallization screening. Acta Crystallographica Section D: Biological Crystallography. , (2023).
- Segelke, B. W. Efficiency analysis of sampling protocols used in protein crystallization screening. Journal of Crystal Growth. 232 (1-4), 553-562 (2001).
- Luft, J. R., Newman, J., Snell, E. H. Crystallization screening: the influence of history on current practice. Acta Crystallographica Section F. 70 (7), 835-853 (2014).
- Mlynek, G., Kostan, J., Leeb, S., Djinovic-Carugo, K. Tailored suits fit better: Customized protein crystallization screens. Crystal Growth & Design. 20 (2), 984-994 (2019).
- Mueller-Dieckmann, J. The open-access high-throughput crystallization facility at EMBL Hamburg. Acta Crystallographica Section D: Biological Crystallography. 62 (12), 1446-1452 (2006).
- Weber, P., et al. High-throughput crystallization pipeline at the crystallography core facility of the Institut Pasteur. Molecules. 24 (24), 4451 (2019).
- Lin, Y. What's happened over the last five years with high-throughput protein crystallization screening. Expert Opinion on Drug Discovery. 13 (8), 691-695 (2018).
- Haupert, L. M., Simpson, G. J. Screening of protein crystallization trials by second order nonlinear optical imaging of chiral crystals (SONICC). Methods. 55 (4), 379-386 (2011).
- Madden, J. T., DeWalt, E. L., Simpson, G. J. Two-photon excited UV fluorescence for protein crystal detection. Acta Crystallographica Section D: Biological Crystallography. 67 (10), 839-846 (2011).
- Fleming, A. M., et al. Second harmonic generation interrogation of the endonuclease APE1 binding interaction with G-quadruplex DNA. Analytical Chemistry. 94 (43), 15027-15032 (2022).
- Bruno, A. E., et al. Classification of crystallization outcomes using deep convolutional neural networks. PLoS One. 13 (6), 0198883 (2018).
- Holleman, E. T., Duguid, E., Keefe, L. J., Bowman, S. E. Polo: An open-source graphical user interface for crystallization screening. Journal of Applied Crystallography. 54 (2), 673-679 (2021).
- Niesen, F. H., et al. An approach to quality management in structural biology: Biophysical selection of proteins for successful crystallization. Journal of Structural Biology. 162 (3), 451-459 (2008).
- Padayatti, P., Palczewska, G., Sun, W., Palczewski, K., Salom, D. Imaging of protein crystals with two-photon microscopy. Biochemistry. 51 (8), 1625-1637 (2012).
- Haupert, L. M., DeWalt, E. L., Simpson, G. J. Modeling the SHG activities of diverse protein crystals. Acta Crystallographica Section D: Biological Crystallography. 68 (11), 1513-1521 (2012).
- Luft, J. R., Grant, T. D., Wolfley, J. R., Snell, E. H. A new view on crystal harvesting. Journal of Applied Crystallography. 47 (3), 1158-1161 (2014).
- Bruno, A. E., Soares, A. S., Owen, R. L., Snell, E. H. The use of haptic interfaces and web services in crystallography: An application for a 'screen to beam' interface. Journal of Applied Crystallography. 49 (6), 2082-2090 (2016).
- Baldock, P., Mills, V., Stewart, P. S. A comparison of microbatch and vapour diffusion for initial screening of crystallization conditions. Journal of Crystal Growth. 168 (1-4), 170-174 (1996).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados