É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Bioimpressão 3D de hidrogéis fototáveis para estudo da ativação de fibroblastos
Neste Artigo
Resumo
Este artigo descreve como bioimprimir hidrogéis fototáveis em 3D para estudar o enrijecimento da matriz extracelular e a ativação de fibroblastos.
Resumo
Os hidrogéis fotosajustáveis podem transformar-se espacial e temporalmente em resposta à exposição à luz. A incorporação desses tipos de biomateriais em plataformas de cultura celular e o desencadeamento dinâmico de mudanças, como o aumento da rigidez microambiental, permite aos pesquisadores modelar as mudanças na matriz extracelular (MEC) que ocorrem durante a progressão da doença fibrótica. Neste trabalho, é apresentado um método para bioimpressão 3D de um biomaterial hidrogel fototável capaz de duas reações sequenciais de polimerização dentro de um banho suporte de gelatina. A técnica de bioimpressão Freeform Reversible Embedding of Suspended Hydrogels (FRESH) foi adaptada ajustando-se o pH do banho de suporte para facilitar uma reação de adição de Michael. Primeiro, a biotinta contendo poli(etilenoglicol)-alfa metacrilato (PEGαMA) foi reagida fora da estequiometria com um reticulante degradável por células para formar hidrogéis moles. Esses hidrogéis moles foram posteriormente expostos ao fotoinibidor e à luz para induzir a homopolimerização dos grupos não reagidos e enrijecer o hidrogel. Este protocolo abrange síntese de hidrogel, bioimpressão 3D, fotoenrijecimento e caracterizações de desfechos para avaliar a ativação de fibroblastos em estruturas 3D. O método aqui apresentado permite aos pesquisadores bioimprimir em 3D uma variedade de materiais que sofrem reações de polimerização catalisadas por pH e pode ser implementado para projetar vários modelos de homeostase, doença e reparo tecidual.
Introdução
A bioimpressão 3D é uma tecnologia transformadora que permite aos pesquisadores depositar com precisão células e biomateriais em volumes 3D e recriar a complexa estrutura hierárquica dos tecidos biológicos. Na última década, avanços na bioimpressão 3D criaram tecidos cardíacos humanos batidos1, modelos funcionais de tecidos renais2, modelos de trocas gasosas dentro do pulmão3 e modelos tumorais para pesquisa de câncer4. A invenção de técnicas de bioimpressão 3D incorporadas, como a bioimpressão Freeform Reversible Embedding of Suspended Hydrogel (FRESH), tornou possível reproduzir estruturas complexas de tecidos moles, como vasos sanguíneos pulmonares5 e até coração humano6 em 3D. A bioimpressão 3D FRESH facilita a impressão camada por camada de biotintas macias e de baixa viscosidade através da extrusão em um banho de suporte de desbaste de cisalhamento. O banho de suporte consiste em um material como micropartículas de gelatina bem embaladas que atua como um plástico Bingham e mantém a forma e a estrutura pretendidas da biotinta após a impressão. Uma vez que a construção impressa tenha se solidificado, o banho de suporte pode então ser dissolvido aumentando a temperatura para 37 °C7.
Um artigo de revisão recente resumiu os materiais que foram bioimpressos em 3D em várias publicações usando a técnica FRESH. Esses materiais de origem natural variam desde colágeno tipo I até ácido hialurônico metacrilado e representam vários mecanismos diferentes de gelificação7. A maioria das pesquisas realizadas com essa técnica de bioimpressão 3D emprega biomateriais estáticos que não se alteram em resposta a estímulos externos. Biomateriais dinâmicos de hidrogel fotosajustáveis têm sido utilizados por nosso laboratório e outros 8,9,10,11,12 para modelar uma variedade de doenças fibróticas. Ao contrário dos biomateriais estáticos, as biotintas fotosajustáveis permitem que um modelo amolecido com menor valor de módulo de elasticidade seja criado e posteriormente enrijecido para explorar respostas celulares a aumentos no enrijecimento microambiental.
As doenças fibróticas caracterizam-se pelo aumento da produção de matriz extracelular, que pode causar cicatrizes e enrijecimento13. O enrijecimento tecidual pode iniciar novas lesões e destruição do tecido impactado, causando danos permanentes aos órgãos e até a morte; As desordens fibróticas são responsáveis por um terço da mortalidade mundial. Os fibroblastos produzem excesso e matriz extracelular aberrante nesse estadopatológico 14,15. O aumento da proliferação fibroblástica e a deposição de matriz extracelular enrijecem ainda mais o tecido e ativam uma alça de feedback positivo profibrótico16,17,18,19. O estudo da ativação de fibroblastos é vital para a compreensão das doenças fibróticas. Aqui apresentamos a hipertensão arterial pulmonar (HAP) humana como um exemplo de um distúrbio fibrótico no qual é importante imitar a geometria 3D do vaso sanguíneo usando bioimpressão 3D e introduzir as capacidades de enrijecimento dinâmico dos hidrogéis fotosajustáveis. A HAP é uma condição na qual a pressão nas artérias pulmonares principais ultrapassa os níveis normais e aplica tensão no coração, aumentando a ativação do fibroblasto adventício da artéria pulmonar humana (FAAPH) e enrijece os tecidos dos vasos sanguíneos16,17,18,19. Uma formulação de biotinta de poli(etilenoglicol)-alfa metacrilato (PEGαMA) fototunable permite o enrijecimento temporal em construtos e ajuda a modelar tanto o tecido saudável quanto a progressão da doença 5,8,9,10. A exploração desta característica única permite a quantificação da ativação e proliferação de HPAAF em resposta ao enrijecimento microambiental em 3D e pode fornecer informações valiosas sobre os mecanismos celulares envolvidos nesta doença. O protocolo aqui descrito permitirá aos pesquisadores criar modelos 3D que recapitulam mudanças no microambiente extracelular durante a progressão da doença ou reparo tecidual e estudar a ativação de fibroblastos.
Protocolo
1. Síntese e caracterização de PEGαMA
NOTA: A síntese de poli(etilenoglicol)-alfametacrilato (PEGαMA) foi adaptada de Hewawasam e col. e realizada em condições livres de umidade9.
- Pese os reagentes.
NOTA: Por exemplo, pesar 5 g de PEG-hidroxila de 8 braços de 10 kg/mol (PEG-OH) e 0,38 g de hidreto de sódio (NaH) (ver Tabela de Materiais). - Adicionar uma barra de agitação a 250 ml de balão de Schlenk e purgar com argónio.
- Dissolver o PEG-OH no menor volume de tetraidrofurano anidro (THF) necessário para a dissolução no frasco de Schlenk.
NOTA: Aproximadamente 80 mL de THF dissolverão 5 g de PEG-OH. Adicione a quantidade mínima de THF necessária para dissolver o PEG-OH. - Adicionar 3 vezes o excesso molar de NaH à mistura de reação e agitar à temperatura ambiente (TR) por 30 min.
- Adicionar 6 vezes o excesso molar de Ethyl 2-(bromomethyl)acrilate (EBrMA, ver Tabela de Materiais) gota a gota ao frasco de Schlenk e cobrir o recipiente de reação com folha de alumínio para protegê-lo da luz. Agite a reação à temperatura ambiente durante aproximadamente 48 horas.
NOTA: Para 5 g de PEG-OH e 0,38 g de NaH, utilizar 3,68 mL de EBrMA para esta reação. - Adicione algumas gotas de 1 N de ácido acético para apagar a reação. Filtre a vácuo a solução através de um auxiliar de filtração.
NOTA: A adição de ácido acético produzirá bolhas de gás. Pare de adicionar gotas de ácido acético quando as bolhas pararem de se formar, pois isso indica que a mistura foi extinta com sucesso. - Concentrar o filtrado num evaporador rotativo e precipitar em éter dietílico a 4 °C. Deixe o precipitado protegido da luz a 4 °C durante 12-18 h.
- Adicione um papel de filtro Whatman a um funil Buchner. Despeje lentamente a mistura de reação sobre o papel de filtro e use sucção a vácuo para separar o precipitado do éter dietílico. Recolher o precipitado num balão de filtração seco e limpo.
- Secar o produto a vácuo por pelo menos 5 h ou durante a noite à temperatura ambiente e dissolver no volume mínimo de água deionizada necessário. Transfira o produto dissolvido para a tubulação de diálise (ver Tabela de Materiais) e dialise contra 3,5 L de água deionizada por pelo menos quatro dias. Troque a água da diálise a cada 12 h.
NOTA: O produto aparecerá como pó sólido branco puro completamente seco após a secagem a vácuo. - Congelar o produto e liofilizar por aproximadamente 72 h ou até secar completamente.
- Dissolver o produto em clorofórmio D (CDCl3). Execute a amostra usando RMN de 1H com um protocolo que executa 248 varreduras com tempo de relaxamento de 2,5 s.
- Verifique a funcionalização e pureza do produto calibrando o pico de solvente CDCl3 para 7,26 PPM. Integre o pico para prótons de backbone PEG (d3.71) e calibre a integração para 114.
- Integrar os picos restantes: PEGαMA 1 H RMN (300 MHz, CDCl 3): d (ppm) 1,36(t, 3H, CH 3-),3,71 (s, 114H, PEG CH 2-CH 2), 4,29 (t, s, 4H, -CH 2-C(O)-O-O, -O-CH 2-C(=CH 2)-), 5,93 (q, 1H, -C=CH2), 6,34 (q, 1H, -C=CH 2) e comparar a integração para os picos do grupo final de alcenos αMA com o valor esperado (1H) com base na calibração do backbone do PEG (Gráfico 1).
OBS: Faça a média dos dois picos rotulados como "a" (Figura 1) e multiplique por 100 para obter a porcentagem média de funcionalização do PEGαMA.
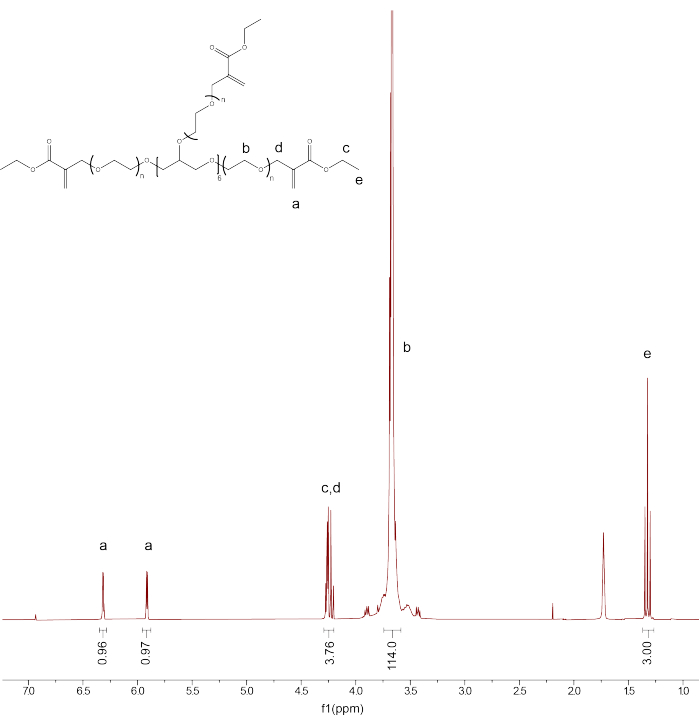
Figura 1: A RMN de prótons confirmou o sucesso da funcionalização do PEGαMA. A análise de RMN foi realizada em clorofórmio-D (CDCl3) e mostrou a funcionalização de 96,5%. PEGαMA 1 H RMN (300 MHz, CDCl3): d (ppm) 1,36(t, 3H, CH 3-),3,71 (s, 114H, PEG CH 2-CH 2), 4,29 (t, s, 4H, -CH 2-C(O)-O-O, -O-CH2-C(=CH 2)-), 5,93 (q, 1H, -C=CH 2), 6,34 (q, 1H, -C=CH 2). Clique aqui para ver uma versão maior desta figura.
2. Projeto do modelo e configuração da bioimpressora 3D
NOTA: Uma impressora 3D comercialmente disponível (ver Tabela de Materiais) foi modificada substituindo a extrusora termoplástica por uma extrusora de bomba de seringa personalizada e adaptada de Hinton et al.20. Projetos de código aberto estão disponíveis on-line: https://3d.nih.gov/users/awfeinberg.
- Abra o software Fusion 360 (consulte a Tabela de Materiais) e faça um projeto de cilindro oco assistido por computador 3D.
Observação : um arquivo para download que pode ser usado para esta etapa e imita a geometria do vaso sanguíneo pode ser encontrado no arquivo suplementar 1. - Salve o arquivo e abra-o no software Slic3r (consulte Tabela de Materiais). Verifique se todos os parâmetros estão conforme desejado e, em seguida, pressione o botão de código G de exportação. Salve o código G no computador.
- Abra o software Pronterface (consulte Tabela de Materiais) e carregue o arquivo de código G.
NOTA: O software Pronterface faz interface com a bioimpressora e fornece controle de entrada de hardware suficiente. Um arquivo de código G utilizável pode ser encontrado no Arquivo Suplementar 2. - Transfira a bioimpressora e todas as peças associadas para um gabinete de biossegurança (BSC) usando técnicas assépticas.
- Monte uma ponta de agulha romba de 30 G e 0,5" de comprimento (consulte Tabela de Materiais) na seringa de vidro de impressão e reserve.
- Conecte o cabo de alimentação da bioimpressora a uma tomada. Pressione o botão liga/desliga vermelho na parte frontal da bioimpressora para ligá-la. Conecte o cabo USB (barramento serial universal) entre o computador e a bioimpressora e certifique-se de que todas as conexões de fio estejam estabelecidas e conectadas.
3. Preparo do banho de apoio e reagentes
OBS: Executar todas as etapas em um gabinete de biossegurança utilizando técnicas assépticas.
- Preparar meio de cultura celular composto por meio basal SmBM (CC-3181) e suplementos SmGM-2 SingleQuots (CC-4149), excluindo soro fetal bovino (FBS), de acordo com as instruções do fabricante. Conservar a 4 °C até à utilização.
- Alíquota 50 mL do meio de cultura celular e adicionar 1% v/v de FBS (CC-4149) (ver Tabela de Materiais) para fazer um meio de soro baixo. Conservar a 4 °C até à utilização.
- Ressuspenda o pó de pasta de gelatina de acordo com as instruções do fabricante usando meios de cultura de células estéreis sem FBS como solvente (consulte a Tabela de Materiais). Imediatamente antes da utilização, ajustar o pH final da pasta de gelatina para pH 9 utilizando hidróxido de potássio (KOH) 2 M e/ou ácido clorídrico 2 M (HCl) para ajustar o pH da solução, conforme necessário, utilizando um medidor de pH.
- Encha o número desejado de poços de uma placa de 24 poços cada um aproximadamente meio cheio usando 1 mL de pasta de gelatina por poço usando uma seringa sem a agulha.
NOTA: Preencha uniformemente o centro dos poços e confirme que não existem bolsões de ar. Bata na placa para ajudar a distribuir uniformemente a lama de micropartículas. Ajuste a altura e o volume da lama conforme necessário para acomodar cada tamanho e forma de bioimpressão. Os usuários podem criar uma seringa caseira para transferir a pasta de gelatina para cada poço. Isso pode ser feito adicionando um êmbolo de seringa de tamanho correto em um tubo de ensaio de 50 mL já contendo a pasta de gelatina compactada na parte inferior. Ao inserir o êmbolo, insira um pequeno fio-guia ao lado do tubo de ensaio para ajudar a escapar do ar e, em seguida, remova-o quando o êmbolo estiver em contato com a pasta de gelatina. Imediatamente antes de usar, corte a ponta do tubo de ensaio com uma lâmina de barbear para criar um orifício para que a pasta de gelatina saia e pressione o êmbolo. - Coloque a placa cheia de 24 poços no centro do palco da bioimpressora e fixe-a no palco.
NOTA: A Figura 2 mostra uma configuração genérica da bioimpressora. Coloque um elástico ao redor do palco de impressão para prender uma placa de 24 poços à plataforma e evitar o movimento.

Figura 2: Configuração básica da bioimpressão 3D. A bioimpressora foi montada dentro de um ambiente estéril, como um gabinete de biossegurança, e a cabeça de impressão foi montada de modo que a seringa de vidro e a agulha fossem abaixadas verticalmente na área de impressão do banho de apoio abaixo. Clique aqui para ver uma versão maior desta figura.
4. Cultura celular
OBS: Executar todas as etapas em um gabinete de biossegurança utilizando técnicas assépticas.
- Descongelar células HPAAF (obtidas comercialmente, ver Tabela de Materiais) e expandi-las em frascos plásticos tratados com cultura de tecidos T-75 contendo Meio Basal SmBM (CC-3181) e todos os suplementos SmGM-2 SingleQuots (CC-4149) de acordo com as instruções do fabricante (ver Tabela de Materiais).
NOTA: Protocolos padrão de cultura de células para células aderentes devem ser usados, mantendo as células a 37 °C e 5% CO2 e repondo o meio a cada poucos dias. - Uma vez que os HPAAFs tenham atingido ~80-90% de confluência, aspirar o meio e enxaguar as células uma vez com solução salina tamponada com fosfato (PBS).
- Adicionar aproximadamente 4 mL de tripsina-EDTA a 0,05% pré-aquecido a cada frasco T-75. Inclinar o balão para garantir que toda a superfície de cultura celular seja coberta com solução de tripsina-EDTA a 0,05%. Incubar os frascos durante 3-5 minutos a 37 °C e verificar o desprendimento celular.
- Uma vez que as células estejam flutuando, adicionar pelo menos 6 mL de meio de águia modificada de Dulbecco (DMEM) a cada frasco e transferir as células para um tubo cônico de 50 mL.
- Centrifugar a suspensão celular a 300 x g por 5 min à temperatura ambiente para peletizar as células. Aspirar o sobrenadante da pastilha celular e ressuspender as células em meio de 1-3 mL com FBS usando uma pipeta de 1000 μL, garantindo uma suspensão de célula única.
- Transferir 10 μL da suspensão celular para um tubo de microcentrífuga. Adicione 10 μL de solução de Azul de Tripano e misture bem. Use 10 μL dessa mistura para contar células dentro de um hemocitômetro usando um microscópio de luz invertida.
NOTA: Para atingir uma concentração final de biotinta de 4 x 106 células/mL, 800.000 fibroblastos foram reservados para cada 200 μL de biotinta.
5. Preparação da biotinta hidrogel
NOTA: A preparação de biotinta foi adaptada de Davis-Hall et al.5. As etapas 5.1-5.2 podem ser concluídas em paralelo com as etapas 4.1-4.3 para minimizar o tempo entre a coleta celular e a ressuspensão na biotinta. Executar etapas em um gabinete de biossegurança usando uma técnica asséptica.
- Preparar uma solução de fosfina tris(2-carboxietil) de 20 mM (TCEP, ver Tabela de Materiais) pH 7 e filtro estéril usando um filtro de seringa de 0,2 μm. Imediatamente antes da utilização, adicionar 2 M KOH e/ou 2 M HCl para ajustar o pH da solução, conforme necessário. Meça com medidor de pH e ajuste de acordo.
NOTA: O CEP reduz as ligações dissulfeto. - Preparar uma solução-mãe de 0,25 mg/mL de PEGαMA ressuspensa em meio de cultura celular estéril sem FBS, soluções-estoque de 250 mM de 1,4-ditiotreitol (DTT), reticulante degradável de MMP2 e peptídeo CGRGDS (RGD) (ver Tabela de Materiais), ressuspendendo tudo em TCEP estéril de 20 mM e uma solução estoque de 15% em peso de poli(óxido de etileno) (PEO) em água destilada (DI) usando pipetas conforme necessário.
- Seguindo a Tabela 1 como guia, combine as respectivas quantidades necessárias de PEGαMA, TDT, reticulante degradável para MMP2, PEO, CGRGDS e meios de cultura de células de baixo soro juntamente com os fibroblastos em um tubo cônico de 50 mL.
NOTA: Recomenda-se verificar o pH com tiras de pH depois de adicionar todos os meios de cultura celular, exceto o meio de cultura celular, pois esta combinação deve resultar no pH muito próximo de 6,2. Se forem necessários mais ajustes de pH, acompanhe quanto volume adicional é necessário para ajustar o pH da solução precursora. Aumentar o volume total para 200 μL adicionando o volume restante do meio de cultura celular menos qualquer volume adicionado durante o ajuste final do pH. - Misture a biotinta usando uma pipeta de deslocamento positivo para garantir que as células sejam células únicas e confirme se a solução precursora final é pH 6,2 para evitar a polimerização catalisada por base durante a bioimpressão 3D.
- Coloque a biotinta na seringa de vidro removendo o êmbolo e usando uma seringa separada com uma ponta de agulha romba de 1,5" de comprimento de calibre 15 (ver Tabela de Materiais) conectada para transferir a biotinta do tubo da centrífuga para a seringa, tomando cuidado para evitar a formação de bolhas de ar dentro da solução.
- Coloque a seringa de vidro dentro da cabeça de impressão e conecte os componentes da cabeça de impressão para que tudo esteja firmemente montado e pronto para impressão.
NOTA: Neste ponto, a seringa de vidro dentro da cabeça de impressão deve ter uma ponta de agulha romba de 30 gauge de 0,5" de comprimento acoplada a ela para impressão.
| Componente | Concentração da Solução Estoque | Valor a ser adicionado |
| PEGαMA | 0,25 mg/ml | 140 μL |
| TDT | 250 mM | 12,24 μL |
| Reticulante Degradável MMP2 | 250 mM | 5,25 μL |
| RGD | 250 mM | 1,6 μL |
| PEO | 15% em peso | 33,33 μL |
| Meio de Ativação e/ou Reagentes de Ajuste de pH | - | 7,58 μL |
| Fibroblastos | - | 800000 células |
Tabela 1: Exemplos de volumes necessários para preparar 200 μL de biotinta (solução precursora de hidrogel e células de fibroblastos).
6.3D bioimpressão
OBS: Executar todas as etapas em um gabinete de biossegurança utilizando técnicas assépticas.
- Usando as setas direcionais dentro do software Pronterface, ajuste manualmente a posição da agulha de extrusão no centro de um poço e dentro da lama do banho de suporte. Deixe pelo menos 1 mm de lama de banho de apoio abaixo da ponta da agulha.
NOTA: O software não tem capacidade de dizer onde a agulha está dentro do espaço. Cabe completamente ao usuário mover a agulha clicando manualmente nas setas dentro do software (por exemplo, clicar na seta para cima moverá a agulha para cima ou para longe da plataforma de impressão, etc.). Manobrar a agulha cuidadosamente para garantir que ela não atingirá nenhum limite do poço. - Uma vez que a ponta da agulha esteja situada no centro da lama dentro do poço, aperte o botão de partida dentro do Pronterface e aguarde a impressão ser concluída para obter construções, como demonstrado na Figura 3A.
Observação : para bioimprimir uma construção usando o arquivo fornecido (arquivo suplementar 1), levará aproximadamente 3 min. Leva aproximadamente 5 minutos para orientar e mover a agulha e, em seguida, imprimir uma construção completamente do início ao fim. - Repita as etapas 6.1-6.2 até que o número de construções bioimpressas desejadas seja atingido.
Observação : é recomendável fazer mais construções do que o necessário para contabilizar quaisquer impressões malsucedidas. Se ocorrer uma falha, vá para o próximo poço, redefina tudo e repita as etapas 6.1-6.2 novamente. - Deixe a placa do poço à temperatura ambiente e cubra-a no BSC por 1 h após o término da impressão para permitir a polimerização catalisada por base do hidrogel fotosintonizável.
- Coloque a placa de poço contendo construções bioimpressas em 3D em uma incubadora estéril a 37 °C e deixe-as por 12-18 h para derreter a lama do banho de suporte.
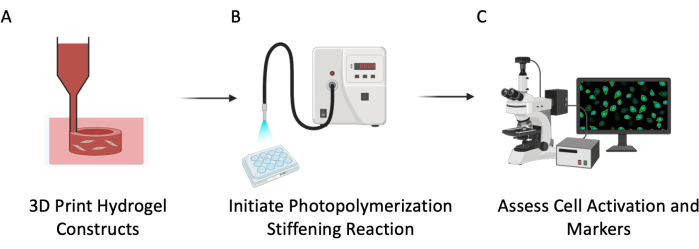
Figura 3: Esquema experimental. Esse protocolo foi descrito em três grandes etapas: (A) bioimpressão 3D de tubos ocos de PEGαMA com células emblocadas para mimetizar a vasculatura pulmonar. (B) Fotoiniciação da reação de homopolimerização para enrijecimento do microambiente celular. (C) Avaliação de marcadores celulares de proliferação e ativação. Clique aqui para ver uma versão maior desta figura.
7.3D cultura de construção bioimpressa e fotoenrijecimento
OBS: Todas as etapas devem ser realizadas em gabinete de biossegurança utilizando técnicas assépticas.
- Preparar 2,2 mM de fenil-2,4,6-trimetilbenzoilfosfinato de lítio (LAP) (ver Tabela de Materiais) solução-mãe em PBS e filtro estéril usando um filtro de seringa de 0,2 μm. Mantenha a solução de LAP protegida da luz.
- Após 12-18 h, mude a mídia em torno das construções bioimpressas. Remova manualmente o meio e o banho de suporte de gelatina derretida dentro dos poços e tenha cuidado para não perturbar as construções bioimpressas.
NOTA: É útil remover lentamente a mídia enquanto segura a placa em um ângulo de 45° para que as construções surjam dentro do poço e possam ser vistas. Um cilindro de hidrogel transparente deve ser identificável em cada poço com impressão bem-sucedida (Figura 4A). - Adicione um volume apropriado de meio de soro baixo a cada poço.
NOTA: Para uma placa de 24 poços, o meio de 700 μL por poço deve cobrir completamente as construções bioimpressas. Ajuste conforme necessário. - Retornar a placa à incubadora e trocar o meio das amostras a cada 3 dias ou de acordo com o planejamento experimental.
- Vinte e quatro horas antes do tempo de enrijecimento desejado, retirar os meios das amostras e substituí-los por meios de soro baixo suplementados com 2,2 mM de LA estéril.
NOTA: Para rotular fluorescentemente a estrutura, inche construções bioimpressas em 3D em PBS suplementadas com 10 μM de metacriloxietil tiocarbamoilrodamila B (verT capaz de Materiais) durante a noite, em seguida, enrijeça conforme descrito na etapa 7.6 para rotular fluorescentemente a estrutura. Transferir para PBS por 2 dias a 4 °C para remover o excesso de rodamina e imagem usando um filtro TRITC (Figura 4B,C). - No momento de enrijecimento desejado, retire metade do meio dos poços a serem enrijecidos e coloque a placa com a tampa desligada sob luz UV. Ligue a luz UV e enrijeça essas construções aplicando 10 mW/cm2 365 nm de luz por 5 min usando Omnicure (consulte Tabela de Materiais) e um filtro passa-banda de 365 nm (Figura 3B).
NOTA: Use o radiômetro/fotômetro para confirmar se a intensidade da luz está correta antes de expor as células à luz UV. - Remova o meio restante desses poços e adicione meios frescos de baixo soro a cada poço. Devolva a placa à incubadora.
- Retire a placa da incubadora e realize o estudo de ativação de fibroblastos no momento desejado seguindo o passo 9.
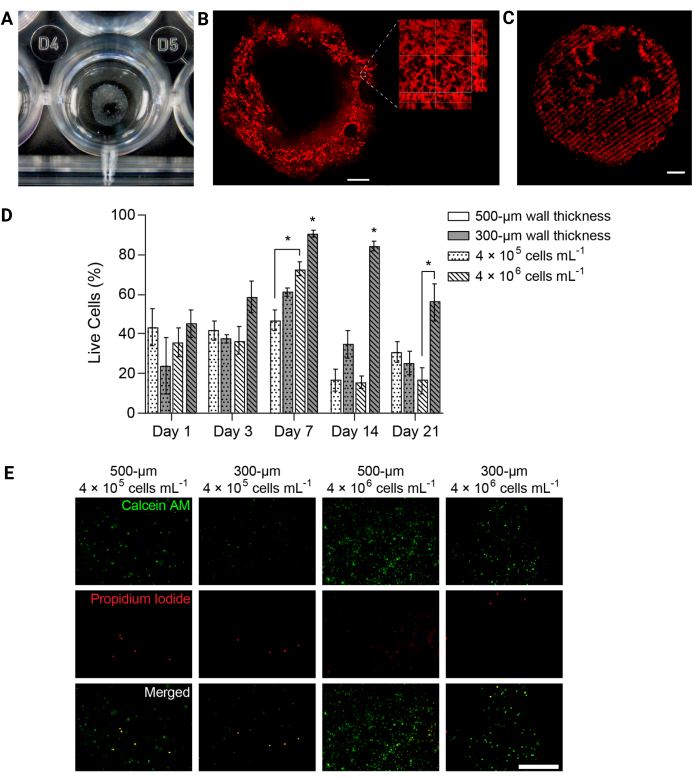
Figura 4: Estruturas de hidrogel bioimpressas em 3D suportaram a viabilidade celular ao longo do tempo. (A) Fotografia da estrutura de hidrogel impressa em 3D em uma placa de 24 poços. (B) Projeção de intensidade máxima do hidrogel impresso em 3D PEGαMA fluorescentemente marcado. Barra de escala = 1 mm. A microscopia de maior magnificação mostrou poros dentro da estrutura do hidrogel induzidos por micropartículas de gelatina no banho suporte de bioimpressão FRESH. (C) O tubo PEGαMA impresso em 3D com regiões endurecidas marcadas fluorescentemente em microscópio confocal (100 μm z-stack exibido como projeção de intensidade máxima) mostrou controle espacial sobre o enrijecimento em 3D. Barra de escala = 500 μm. (D) Viabilidade da HPAAF em construções bioimpressas em 3D medidas por ensaios vivos/mortos. Construções com 300 μm de espessura e 4 × 106 células/mL superaram todas as outras condições em todos os momentos. A viabilidade atingiu o pico no dia 7. Essa condição e o momento foram selecionados para experimentos futuros. As colunas mostram a média ± EPM, n = 3. *, p < 0,05, ANOVA, Tukey HSD. (E) Imagens confocais representativas de células em construções 3D coradas com reagente vivo/morto no dia 7, o momento com maior viabilidade global. Calceína AM marcou células vivas em verde e iodeto de propídio marcou células mortas em vermelho. A coluna mais à direita mostra que a condição de melhor desempenho tinha uma distribuição celular uniforme e uma alta porcentagem de células vivas. Barra de escala = 500 μm. Reproduzido com permissão de Davis-Hall et al.5. Clique aqui para ver uma versão maior desta figura.
8. Avaliação da viabilidade de fibroblastos
- Nos momentos de viabilidade desejados, corar usando calceína AM e iodeto de propídio (ver Tabela de Materiais).
NOTA: Para resumir, os meios devem ser removidos de cada poço e as construções devem ser lavadas com PBS estéril. Incubar as construções em uma solução de coloração viva/morta por 40 min a 37°C em um balancim. A solução corante deve conter calceína AM (diluição 1:1000) e iodeto de propídio (diluição 1:1000) para identificar células vivas ou mortas nos exames de imagem. - Transfira as construções para PBS estéril e imediatamente a imagem em um microscópio confocal de fluorescência. Adquira três diferentes imagens z-stack de 100 μm por amostra por ponto de tempo e expresse viabilidade como a porcentagem média de células vivas (Figura 4D,E).
9. Avaliação da ativação fibroblástica
- Preparar 3% p/v de albumina de soro bovino (BSA) e 0,1% v/v Tween 20 em PBS. Esta solução será referida como a solução de imunofluorescência (IF).
- Nos momentos desejados, remova o meio dos poços de amostra e lave as construções com PBS. Substitua o PBS por paraformaldeído (PFA) a 4% e mantenha essas amostras a 37 °C por 30 min em um balancim. Em seguida, substitua o PFA 4% por glicina 100 mM em PBS e deixe essas amostras em balancim à temperatura ambiente (TR) por mais 15 min.
- Em seguida, transfira essas amostras para criomoldes Tissue-Tek, cubra a amostra completamente com solução de temperatura de corte ideal (OCT) (consulte Tabela de Materiais) e permita que a OCT se difunda nas amostras por 12-18 h a 4 °C.
- Congelar rapidamente as amostras embebidas em OCT em 2-metilbutano utilizando azoto líquido. Encha uma caixa de isopor ou outro recipiente apropriado com nitrogênio líquido e, em seguida, coloque um segundo recipiente cheio de 2-metilbutano dentro do nitrogênio líquido para que ele fique pelo menos submerso pela metade. Use pinças para segurar cada criomold contendo uma amostra coberta de OCT em nitrogênio líquido resfriado com 2-metilbutano até congelar visivelmente. Estas amostras podem ser armazenadas a -80 °C até estarem prontas para a criossecção.
CUIDADO: Equipamentos de proteção individual (EPIs), como luvas protetoras a frio, avental protetor contra frio e protetor facial fornecido no Kit de Segurança Criogênica (consulte Tabela de Materiais) devem ser usados ao manusear nitrogênio líquido. - Criosecção das amostras congeladas de OCT a -22 °C e anexar cortes de 10 μm de espessura a lâminas de microscópio de vidro carregadas positivamente. Prepare três lâminas de microscópio com pelo menos 3-5 criosecções por lâmina para cada amostra de hidrogel 3D.
NOTA: As lâminas do microscópio podem ser armazenadas neste ponto a -80 °C, se necessário, como ponto de parada. - Fixe as criosecções em acetona gelada por 15 min para ajudar as criosecções a aderirem às lâminas. Enxágue suavemente as criossecções com água RT para remover qualquer OCT restante. Deixe essas amostras secarem e contorne as criossecções com uma caneta hidrofóbica (consulte a Tabela de Materiais).
- Permeabilizar as amostras à temperatura ambiente com Triton X-100 0,2% v/v em PBS por 10 min e, em seguida, bloquear os cortes com 5% BSA p/v em PBS por 1 h em TR.
- Adicione o anticorpo primário anti-actina de músculo liso alfa humano (αSMA) de rato (diluição de 1:250) (ver Tabela de Materiais) à solução FI. Armazenar estas amostras seccionadas com o anticorpo primário sobre elas durante a noite a 4 °C. Enxaguar as amostras 3x com solução FI.
- Incubar as secções em solução de IF contendo o anticorpo secundário de cabra anti-rato Alexa Fluor 555 (diluição de 1:250) e duas gotas de ActinGreen 488 ReadyProbe (ver Tabela de Materiais) por mililitro de solução de FI. Cubra as amostras para todas as etapas subsequentes com papel alumínio para protegê-las da luz e deixe a solução de anticorpos secundários permanecer nas amostras por 1 h no RT.
- Enxaguar as secções 3x com solução IF. Incubá-los em 300 nM 4',6-diamidino-2-filindolo (DAPI) em água DI por 15 min em RT. Realizar um enxágue final das seções 3x com água DI.
- Usando 10 μL de um reagente antifade disponível comercialmente (ver Tabela de Materiais), cubra as seções usando métodos padrão.
NOTA: As lâminas montadas podem ser armazenadas protegidas da luz em um freezer de -80 °C até que seja necessário para a obtenção de imagens. - Imagem das criossecções em microscópio de fluorescência (Figura 3C e Figura 5B). Imagine três seções aleatórias por slide usando uma objetiva de 10x.
NOTA: As imagens devem ser obtidas nos canais DAPI, FITC e TRITC. - Carregue as imagens no ImageJ (NIH). Quantificar a porcentagem de células positivas para αSMA como medida da ativação fibroblástica (Figura 5A) dividindo o número total de células positivas para αSMA pelo número total de núcleos celulares para cada campo de visão.
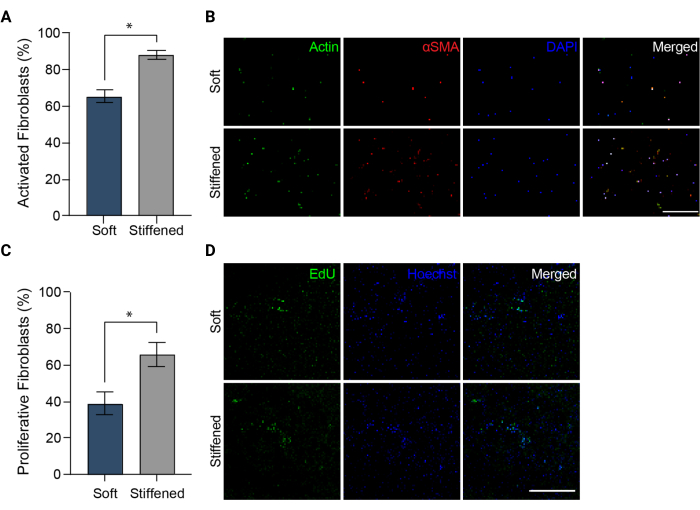
Figura 5: Ativação fibroblástica em modelos bioimpressos 3D de adventícia arterial pulmonar . (A) Ativação fibrótica em hidrogéis 3D moles e enrijecidos medidos pela expressão de αSMA. HPAAFs em construções enrijecidas foram significativamente mais positivas para αSMA do que células em construções moles. As colunas representam a média ± EPM, n = 3. *, p < 0,05, teste U de Mann-Whitney. (B) Imagens confocais representativas de imunomarcação para αSMA, actina e DAPI em hidrogéis 3D moles e enrijecidos. HPAAFs em construções enrijecidas mostraram imunofluorescência αSMA mais prevalente do que células em construções moles. Barra de escala = 250 μm. (C) Proliferação fibroblástica em construções bioimpressas 3D macias e enrijecidas medidas pela positividade de EdU. HPAAFs em construções enrijecidas foram significativamente mais positivas para EdU do que células em construções moles. As colunas representam a média ± EPM, n = 3. *, p < 0,05, teste U de Mann-Whitney. (D) Imagens confocais representativas de imunomarcação para EdU e corante Hoechst em hidrogéis 3D moles e enrijecidos. HPAAFs em construções enrijecidas mostraram imunofluorescência EdU mais prevalente do que células em construções moles. Barra de escala = 300 μm. Reproduzido com permissão de Davis-Hall et al.5. Clique aqui para ver uma versão maior desta figura.
10. Avaliação da proliferação fibroblástica
- Vinte e quatro horas antes de um ponto de tempo de proliferação desejado, remova o meio de cultura celular de cada poço e substitua por meios de soro baixo suplementados com solução de EdU de 10 μM do kit de proliferação celular disponível comercialmente (ver Tabela de Materiais). Retorne as amostras à incubadora para incubação durante a noite.
- No ponto de tempo de proliferação desejado, fixar as amostras incubadas com EdU usando PFA 4% a 37 °C por 30 min em um balancim. Substitua a solução de PFA a 4% por glicina 100 mM em PBS e incube as amostras a 37 °C durante, pelo menos, 15 minutos. Adicione o Hoechst numa concentração adequada durante 30 min e, em seguida, lave as construções 2x com PBS.
NOTA: As amostras podem ser armazenadas protegidas da luz a 4 °C até à obtenção de imagens. - Imagens de todas as amostras de EdU fixadas e armazenadas usando um microscópio de fluorescência e as configurações e filtros sugeridos pelo fabricante do kit de proliferação celular (Figura 3C e Figura 5D). Adquira três imagens z-stack diferentes de 100 μm por amostra e crie projeções máximas de cada uma dessas z-stacks. Meça a proliferação de HPAAF contando o número de células EdU-positivas e dividindo-se pelo número total de células identificadas pela contracoloração Hoechst dentro das imagens de projeção máxima (Figura 5C).
Resultados
Este protocolo descreve como bioimprimir hidrogéis fototáveis em 3D dentro de um banho de suporte para criar construções capazes de enrijecimento dinâmico e temporal para estudar a ativação de fibroblastos em geometrias que mimetizam tecidos humanos. Primeiro, o protocolo explicou como sintetizar PEGαMA, a espinha dorsal desse sistema polimérico fotosintonizável. Medidas de espectroscopia de ressonância magnética nuclear (RMN) mostraram sucesso na funcionalização do PEGαMA em 96,5% (Fi...
Discussão
Reações de polimerização em duplo estágio em resposta à exposição controlada à luz podem enrijecer biomateriais com controle espacial e temporal. Vários estudos têm utilizado essa técnica para avaliar interações célula-matriz em diversas plataformas5,8,9,10,11,21,22,23.
Divulgações
Os autores não têm conflitos de interesse a declarar. Partes deste manuscrito são reproduzidas com permissão da © IOP Publishing https://doi.org/10.1088/1758-5090/aca8cf. 5 Todos os direitos reservados.
Agradecimentos
Os autores gostariam de agradecer ao Dr. Adam Feinberg (Carnegie Mellon University) e àqueles que organizaram o 3D Bioprinting Open-Source Workshop. Esses indivíduos possibilitaram o aprendizado das técnicas de bioimpressão FRESH e a construção da bioimpressora 3D utilizada para esses estudos. Além disso, os autores gostariam de agradecer Biorender.com, que foi usado para produzir figuras neste manuscrito. Este trabalho foi apoiado por vários grupos ou fontes de financiamento, incluindo a Rose Community Foundation (DDH e CMM), um Colorado Pulmonary Vascular Disease Research Award (DDH e CMM), a National Science Foundation sob Award 1941401 (CMM), o Departamento do Exército sob o Prêmio W81XWH-20-1-0037 (CMM), o Instituto Nacional do Câncer do NIH sob o Prêmio R21 CA252172 (CMM), o Ludeman Family Center for Women's Health Research da University of Colorado Anschutz Medical Campus (DDH e CMM), o National Heart, Lung, and Blood Institute dos Institutos Nacionais de Saúde sob os prêmios R01 HL080396 (CMM), R01 HL153096 (CMM), F31 HL151122 (DDH) e T32 HL072738 (DDH e AT).
Materiais
| Name | Company | Catalog Number | Comments |
| AccuMax Radiometer/Photometer Kit | Spectronics Corporation | XPR-3000 | To measure light intensity, used for photostiffening |
| Acetic Acid | Fisher Scientific | BP2401-500 | Used during PEGaMA synthesis |
| Acetone | Fisher Scientific | A184 | Used with the cryosections |
| ActinGreen 488 ReadyProbes | Fisher Scientific | R37110 | Used for staining |
| Aluminum Foil | Reynolds | F28028 | |
| Anhydrous Tetrahydrofuran (THF) | Sigma-Aldrich | 401757-1L | Used during PEGaMA synthesis |
| Argon Compressed Gas | Airgas | AR R300 | Used during PEGaMA synthesis |
| 8 Arm Poly(ethylene glycol)-hydroxyl (PEG-OH) | JenKem Technology | 8ARM-PEG-10K | Used during PEGaMA synthesis |
| 365 nm Bandpass Filter | Edmund Optics | 65-191 | Used for photostiffening |
| Bovine Serum Albumin (BSA) | Fisher Scientific | BP9700-100 | Used during staining process |
| Buchner Funnel | Quark Glass | QFN-8-14 | Used during PEGaMA synthesis |
| Calcein AM | Invitrogen | 65-0853-39 | Used during staining process |
| Celite 545 (Filtration Aid) | EMD Millipore | CX0574-1 | Used during PEGaMA synthesis |
| Charged Microscope Slides | Globe Scientific | 1358W | |
| Chloroform-d | Sigma-Aldrich | 151823-10X0.75ML | Used to characterize PEGaMA |
| Click-iT Plus EdU Cell Proliferation Kit | Invitrogen | C10637 | Used for staining |
| 50 mL Conical Tubes | CELLTREAT | 667050B | |
| Cryogenic Safety Kit | Cole-Parmer | EW-25000-85 | |
| Cryostat | Leica | CM 1850-3-1 | |
| Dialysis Tubing | Repligen | 132105 | |
| 4’,6-Diamidino-2-Phylindole (DAPI) | Sigma-Aldrich | D9542-1MG | Used for staining |
| Diethyl Ether | Fisher Scientific | E1384 | Used during PEGaMA synthesis |
| 1,4-Dithiothreitol (DTT) | Sigma-Aldrich | 10197777001 | Bioink component |
| Dulbecco's Modified Eagle's Medium (DMEM) | Cytiva | SH30271.FS | |
| Filter Paper | Whatman | 1001-090 | Used during PEGaMA synthesis |
| Freezone 2.5L Freeze Dry System | Labconco | LA-2.5LR | Lyophilizer |
| Fusion 360 | Autodesk | N/A | Software download |
| 2.5 mL Gastight Syringe | Hamilton | 81420 | Used for bioprinting |
| 15 Gauge 1.5" IT Series Tip | Jensen Global | JG15-1.5X | Used for bioprinting |
| 30 Gauge 0.5" HP Series Tip | Jensen Global | JG30-0.5HPX | Used for bioprinting |
| Goat Anti-Mouse Alexa Fluor 555 Antibody | Fisher Scientific | A21422 | Used for staining |
| Glycine | Fisher Scientific | C2H5NO2 | Used during staining process |
| Hemocytometer | Fisher Scientific | 1461 | |
| Hoechst | Thermo Scientific | 62249 | Used during staining process |
| Human Pulmonary Artery Adventitial Fibroblasts (HPAAFs) | AcceGen | ABC-TC3773 | From a 2-year-old male patient |
| Hydrochloric Acid (HCl) | Fisher Scientific | A144-500 | Used to pH adjust solutions |
| ImageJ | National Institutes of Health (NIH) | N/A | Free software download |
| ImmEdge® Pen | Vector Laboratories | H-4000 | Used during staining process |
| Incubator | VWR | VWR51014991 | |
| LifeSupport Gelatin Microparticle Slurry (Gelatin Slurry) | Advanced Biomatrix | 5244-10GM | Used for bioprinting |
| Light Microscope | Olympus | CKX53 | Inverted light microscope |
| Lithium Phenyl-2,4,6-Trimethylbenzoylphosphinate (LAP) | Sigma-Aldrich | 900889-5G | Photoinitiator used for photostiffening |
| Liquid Nitrogen | N/A | N/A | |
| LulzBot Mini 2 | LulzBot | N/A | Bioprinter adapted |
| Methacryloxyethyl Thiocarbamoyl Rhodamine B | Polysciences Inc. | 669775-30-8 | |
| 2-Methylbutane | Sigma-Aldrich | M32631-4L | |
| Microman Capillary Pistons CP1000 | VWR | 76178-166 | Positive displacement pipette tips |
| MMP2 Degradable Crosslinker (KCGGPQGIWGQGCK) | GL Biochem | N/A | Bioink component |
| Mouse Anti-Human αSMA Monoclonal Antibody | Fisher Scientific | MA5-11547 | Used for staining |
| OmniCure Series 2000 | Lumen Dynamics | S2000-XLA | UV light source used for photostiffening |
| Paraformaldehyde (PFA) | Electron Microscopy Sciences | 15710 | Used to fix samples |
| pH Meter | Mettler Toledo | FP20 | |
| pH Strips | Cytiva | 10362010 | |
| Phosphate Buffered Saline (PBS) | Hyclone Laboratories, Inc. | Cytiva SH30256.FS | |
| Pipette Set | Fisher Scientific | 14-388-100 | |
| 10 µL Pipette Tips | USA Scientific | 1120-3710 | |
| 20 µL Pipette Tips | USA Scientific | 1183-1510 | |
| 200 µL Pipette Tips | USA Scientific | 1111-0700 | |
| 1000 µL Pipette Tips | USA Scientific | 1111-2721 | |
| Poly(Ethylene Glycol)-Alpha Methacrylate (PEGαMA) | N/A | N/A | Refer to manuscript for synthesis steps |
| Poly(Ethylene Oxide) (PEO) | Sigma-Aldrich | 372773-250G | Bioink component |
| Positive Displacement Pipette | Fisher Scientific | FD10004G | 100-1000 µL |
| Potassium Hydroxide (KOH) | Sigma-Aldrich | 221473-500G | Used to pH adjust solutions |
| ProLong Gold Antifade Reagent | Invitrogen | P36930 | Used during staining process |
| Pronterface | All3DP | N/A | Software download |
| Propidium Iodide | Sigma-Aldrich | P4864-10ML | Used for staining |
| RGD Peptide (CGRGDS) | GL Biochem | N/A | Bioink component |
| Rocker | VWR | 10127-876 | |
| Rotary Evaporator | Thomas Scientific | 11100V2022 | Used during PEGaMA synthesis |
| Rubber Band | Staples | 808659 | |
| Schlenk Flask | Kemtech America | F902450 | Used during PEGaMA synthesis |
| Slic3r | Slic3r | N/A | Software download |
| Smooth Muscle Cell Growth Medium-2 (SmGM-2) BulletKit | Lonza | CC-3182 | Kit contains CC-3181 and CC-4149 components |
| Sodium Hydride | Sigma-Aldrich | 223441-50G | Used during PEGaMA synthesis |
| Sorvall ST 40R Centrifuge | Fisher Scientific | 75-004-525 | |
| Stir Bar | VWR | 58948-091 | |
| Syringe Filter | VWR | 28145-483 | Used to sterile filter solutions |
| T-75 Tissue-Cultured Treated Flask | VWR | 82050-856 | Used for cell culture work |
| Tissue-Tek Cyromold | Sakura | 4557 | |
| Tissue-Tek O.C.T Compound (OCT) | Sakura | 4583 | |
| Tris(2-Carboxyethyl) Phosphine (TCEP) | Sigma-Aldrich | C4706-2G | |
| Triton X-100 | Fisher Bioreagents | C34H622O11 | Used during staining process |
| Trypan Blue | Sigma-Aldrich | T8154-20ML | Used for cell culture work |
| 0.05% Trypsin-EDTA | Gibco | 25-300-062 | Used for cell culture work |
| Tween 20 | Fisher Bioreagents | C58H114O26 | Used during staining process |
| Upright Microscope | Olympus | BX63F | Fluorescent microscope capabilities |
| Water Bath | PolyScience | WBE20A11B | |
| 24-Well Tissue Culture Plates | Corning | 3527 |
Referências
- Ahrens, J. H., et al. Programming cellular alignment in engineered cardiac tissue via bioprinting anisotropic organ building blocks. Advanced Materials. 34 (26), e2200217 (2022).
- Lin, N. Y. C., et al. Renal reabsorption in 3D vascularized proximal tubule models. Proceedings of the National Academy of Sciences of the United States of America. 116 (12), 5399-5404 (2019).
- Grigoryan, B., et al. Multivascular networks and functional intravascular topologies within biocompatible hydrogels. Science. 364 (6439), 458-464 (2019).
- Kang, Y., Datta, P., Shanmughapriya, S., Ozbolat, I. T. 3D bioprinting of tumor models for cancer research. ACS Applied Biomaterials. 3 (9), 5552-5573 (2020).
- Davis-Hall, D., Thomas, E., Pena, B., Magin, C. M. 3D-bioprinted, phototunable hydrogel models for studying adventitial fibroblast activation in pulmonary arterial hypertension. Biofabrication. 15 (1), (2022).
- Mirdamadi, E., Tashman, J. W., Shiwarski, D. J., Palchesko, R. N., Feinberg, A. W. FRESH 3D bioprinting of a full-size model of the human heart. ACS Biomaterials Science & Engineering. 6 (11), 6453-6459 (2020).
- Shiwarski, D. J., Hudson, A. R., Tashman, J. W., Feinberg, A. W. Emergence of FRESH 3D printing as a platform for advanced tissue biofabrication. APL Bioengineering. 5 (1), 010904 (2021).
- Petrou, C. L., et al. Clickable decellularized extracellular matrix as a new tool for building hybrid hydrogels to model chronic fibrotic diseases in vitro. Journal of Materials Chemistry B. 8 (31), 6814-6826 (2020).
- Hewawasam, R. S., Blomberg, R., Serbedzija, P., Magin, C. M. Chemical modification of human decellularized extracellular matrix for incorporation into phototunable hybrid hydrogel models of tissue fibrosis. ACS Applied Materials & Interfaces. 15 (12), 15071-15083 (2023).
- Saleh, K. S., et al. Engineering hybrid hydrogels comprised healthy or diseased decellularized extracellular matrix to study pulmonary fibrosis. Cellular and Molecular Bioengineering. 15 (5), 505-519 (2022).
- Guvendiren, M., Burdick, J. A. Stiffening hydrogels to probe short- and long-term cellular responses to dynamic mechanics. Nature Communications. 3, 792 (2012).
- Rosales, A. M., Vega, S. L., DelRio, F. W., Burdick, J. A., Anseth, K. S. Hydrogels with reversible mechanics to probe dynamic cell microenvironments. Angewandte Chemie International Edition English. 56 (40), 12132-12136 (2017).
- Wynn, T. A., Ramalingam, T. R. Mechanisms of fibrosis: therapeutic translation for fibrotic disease. Nature Medicine. 18 (7), 1028-1040 (2012).
- Huertas, A., Tu, L., Humbert, M., Guignabert, C. Chronic inflammation within the vascular wall in pulmonary arterial hypertension: more than a spectator. Cardiovascular Research. 116 (5), 885-893 (2020).
- Kendall, R. T., Feghali-Bostwick, C. A. Fibroblasts in fibrosis: novel roles and mediators. Frontiers in Pharmacology. 5, 123 (2014).
- Parker, M. W., et al. Fibrotic extracellular matrix activates a profibrotic positive feedback loop. The Journal of Clinical Investigation. 124 (4), 1622-1635 (2014).
- Habiel, D. M., Hogaboam, C. Heterogeneity in fibroblast proliferation and survival in idiopathic pulmonary fibrosis. Frontiers in Pharmacology. 5, 2 (2014).
- Hu, C. J., Zhang, H., Laux, A., Pullamsetti, S. S., Stenmark, K. R. Mechanisms contributing to persistently activated cell phenotypes in pulmonary hypertension. The Journal of Physiology. 597 (4), 1103-1119 (2019).
- Li, M., et al. Emergence of fibroblasts with a proinflammatory epigenetically altered phenotype in severe hypoxic pulmonary hypertension. The Journal of Immunology. 187 (5), 2711-2722 (2011).
- Hinton, T. J., et al. Three-dimensional printing of complex biological structures by freeform-reversible embedding of suspended hydrogels. Science Advances. 1 (9), e1500758 (2015).
- Brown, T. E., et al. Secondary photocrosslinking of click hydrogels to probe myoblast mechanotransduction in three dimensions. Journal of the American Chemical Society. 140 (37), 11585-11588 (2018).
- Ondeck, M. G., et al. Dynamically stiffened matrix promotes malignant transformation of mammary epithelial cells via collective mechanical signaling. Proceedings of the National Academy of Sciences of the United States of America. 116 (9), 3502-3507 (2019).
- Caliari, S. R., et al. Stiffening hydrogels for investigating the dynamics of hepatic stellate cell mechanotransduction during myofibroblast activation. Scientific Reports. 6, 21387 (2016).
- Liu, F., et al. Feedback amplification of fibrosis through matrix stiffening and COX-2 suppression. Journal of Cell Biology. 190 (4), 693-706 (2010).
- Tschumperlin, D. J., Ligresti, G., Hilscher, M. B., Shah, V. H. Mechanosensing and fibrosis. The Journal of Clinical Investigation. 128 (1), 74-84 (2018).
- Chelladurai, P., Seeger, W., Pullamsetti, S. S. Matrix metalloproteinases and their inhibitors in pulmonary hypertension. European Respiratory Journal. 40 (3), 766-782 (2012).
- Caracena, T., et al. Alveolar epithelial cells and microenvironmental stiffness synergistically drive fibroblast activation in three-dimensional hydrogel lung models. Biomaterials Science. 10 (24), 7133-7148 (2022).
- Ruskowitz, E. R., DeForest, C. A. Proteome-wide analysis of cellular response to ultraviolet light for biomaterial synthesis and modification. ACS Biomaterials Science & Engineering. 5 (5), 2111-2116 (2019).
- Kruse, C. R., et al. The effect of pH on cell viability, cell migration, cell proliferation, wound closure, and wound reepithelialization: In vitro and in vivo study. Wound Repair and Regeneration. 25 (2), 260-269 (2017).
- Filippi, M., et al. Perfusable biohybrid designs for bioprinted skeletal muscle tissue. Advanced Healthcare Materials. , e1500758 (2023).
- Matthiesen, I., et al. Astrocyte 3D culture and bioprinting using peptide-functionalized hyaluronan hydrogels. Science and Technology of Advanced Materials. 24 (1), 2165871 (2023).
- Xu, L., et al. Bioprinting a skin patch with dual-crosslinked gelatin (GelMA) and silk fibroin (SilMA): An approach to accelerating cutaneous wound healing. Materials Today Bio. 18, 100550 (2023).
- Bliley, J. M., Shiwarski, D. J., Feinberg, A. W. 3D-bioprinted human tissue and the path toward clinical translation. Science Translational Medicine. 14 (666), eabo7047 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados