É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Análise espacial de alta resolução dos níveis de expressão gênica em tendões
Neste Artigo
Resumo
Este artigo descreve como executar um protocolo in situ otimizado para tendões. Este método discute a preparação do tecido, a permeabilização da seção, o design da sonda e os métodos de amplificação de sinal.
Resumo
Nos últimos anos, muitos protocolos foram desenvolvidos para transcriptômica de alta resolução em muitos campos médicos e biológicos diferentes. No entanto, os tecidos ricos em matriz e, especificamente, os tendões foram deixados para trás devido ao seu baixo número de células, baixa quantidade de RNA por célula e alto conteúdo de matriz, o que os tornava complicados de analisar. Uma das ferramentas recentes e mais importantes de uma única célula é a análise espacial dos níveis de expressão gênica em tendões. Essas ferramentas espaciais de RNA têm especificamente alta importância em tendões para localizar células específicas de populações novas e desconhecidas, validar resultados de RNA-seq de célula única e adicionar contexto histológico aos dados de RNA-seq de célula única. Esses novos métodos permitirão a análise de RNA em células com sensibilidade excepcional e a detecção de alvos de RNA de molécula única no nível de célula única, o que ajudará a caracterizar molecularmente os tendões e promover a pesquisa de tendões.
Neste artigo de método, vamos nos concentrar nos métodos disponíveis para analisar os níveis espaciais de expressão gênica em seções histológicas usando novos ensaios de hibridização in situ para detectar RNA alvo dentro de células intactas em níveis de célula única. Primeiro, vamos nos concentrar em como preparar o tecido tendinoso para os diferentes ensaios disponíveis e como amplificar sinais específicos do alvo sem ruído de fundo, mas com alta sensibilidade e alta especificidade. Em seguida, o artigo descreverá métodos específicos de permeabilização, os diferentes designs de sondas e as estratégias de amplificação de sinal atualmente disponíveis. Esses métodos exclusivos de análise dos níveis de transcrição de diferentes genes em resolução de célula única permitirão a identificação e caracterização das células do tecido tendinoso em populações jovens e envelhecidas de vários modelos animais e tecidos tendinosos humanos. Este método também ajudará a analisar os níveis de expressão gênica em outros tecidos ricos em matriz, como ossos, cartilagens e ligamentos.
Introdução
Os tendões são tecidos conjuntivos que permitem a transmissão de força entre o músculo e o osso1. Em termos de desenvolvimento, os tenócitos axiais são derivados de células mesenquimais dentro do esclerótomo dos somitos2; os tendões dos membros derivam do mesoderma da placa lateral; e tendões cranianos surgem da linhagem da crista neural craniana 3,4. O tendão pode ser caracterizado pela expressão do fator de transcrição do escleraxia5, embora vários marcadores também desempenhem um papel fundamental no desenvolvimento do tendão, incluindo tenomodulina, moicano e resposta de crescimento precoce 1/2 6,7,8,9.
Apesar dos poucos marcadores conhecidos do tendão, em geral, uma caracterização mais aprofundada continua sendo um desafio porque o tendão contém células que se estendem por um gradiente de propriedades biomecânicas. Da junção miotendínea, do meio do corpo do tendão e da êntese mais calcificada, as células do tendão residem em matrizes extracelulares que variam em propriedades de tração. Como o tendão deve suportar o estresse de tração imposto pela diferença de resistência mecânica entre o tecido mole e o duro, a organização espacial das células no tendão é particularmente importante para sua função. No entanto, pouco se sabe sobre essas subpopulações tendíneas.
Muitas ferramentas transcriptômicas espaciais de alta resolução podem ser usadas para começar a elucidar subpopulações celulares, incluindo, mas não se limitando a, RNA Seq de célula única ou hibridização in situ . No entanto, embora esses ensaios de perfil espacial ajudem a descobrir a expressão de RNA no tecido após a microdissecção ou secção, esses métodos podem ser desafiadores quando realizados no tecido do tendão. Os tendões são tecidos ricos em matriz compostos por quase 86% de colágeno em massa seca10, dificultando a extração das células para sequenciamento. Devido às complicações no isolamento das células da matriz, à natureza hipocelular do tendão11 e à contagem relativamente baixa de RNA, o tendão é um tecido difícil de analisar.
Neste artigo, apresentamos um método para otimizar novos ensaios de hibridização in situ para aproveitá-los para tendões, fornecendo métodos de preparação de tecido, permeabilização e design de sonda. Juntamente com as tecnologias de sequenciamento existentes, isso pode ajudar os pesquisadores a caracterizar espacialmente as subpopulações de tendões em tendões em desenvolvimento, adultos ou lesionados com maior sensibilidade e especificidade do ensaio.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos os experimentos com animais foram realizados de acordo com as diretrizes do Comitê Institucional de Cuidados e Uso de Animais (IACUC) e AAALAC. Os experimentos foram realizados sob protocolo aprovado #2013N000062 no Massachusetts General Hospital. Neste estudo, foram utilizados camundongos C57BL/J6 (5 semanas de idade e P0). Consulte a Tabela de Materiais para obter detalhes relacionados a todos os materiais, reagentes e instrumentos usados neste protocolo.
1. Preparação e fixação da amostra
- Eutanásia de camundongos em uma câmara de CO2 seguida de luxação cervical como método secundário de eutanásia. Em seguida, use uma tesoura para cortar os membros posteriores do camundongo ao longo da articulação do quadril12. Em um frasco de cintilação, mergulhe o membro posterior do camundongo em solução suficiente de paraformaldeído (PFA) a 4% para cobrir completamente a amostra e fixe o tecido por 24 h.
NOTA: Em vez de 4% de PFA, 10% de formalina também pode ser usada para fixação. Após a fixação, a amostra pode ser armazenada em solução salina tamponada com fosfato (PBS) até processamento posterior. Ao fixar todo o membro posterior, somos capazes de preservar a tensão no tendão e temos a oportunidade de examinar a êntese, o meio do corpo do tendão ou a junção miotendínea. - Depois de remover o PFA do frasco de cintilação, lave a amostra por 3 x 10 min com 1x PBS adicionando PBS suficiente para cobrir a amostra. Em seguida, mergulhe os membros posteriores em EDTA 0,5 M a 4 ° C com agitação por 1-2 semanas, substituindo por EDTA fresco a cada 2-3 dias.
- Após remover o EDTA, lave a amostra por 3 x 10 min com 1x PBS, desidrate as amostras em EtOH 70% e embeba-as em parafina na orientação desejada. Use técnicas convencionais de seccionamento FFPE para seccionar o tecido com uma espessura de 3 μm e transferir os cortes para uma lâmina de microscópio tratada e carregada (ver Tabela de Materiais)13.
NOTA: As seções podem ser armazenadas em 4 °C antes de prosseguir para as próximas etapas. - Desparafinização
- Asse as lâminas em uma placa quente a 60 °C por 1 h e depois deixe as lâminas atingirem a temperatura ambiente (RT). Para remover a cera de parafina das lâminas, coloque-as em um suporte de lâminas e abaixe-as em uma mancha cheia de xileno. Repita em xileno fresco por 3 x 5 min.
CUIDADO: O xileno é perigoso; manuseie com cuidado. - Para reidratar as seções, mergulhe as lâminas no seguinte por 5 min por lavagem, tudo em RT: lavar 2x em 100% de EtOH, 1x em 75% de EtOH diluído em dH2O, 1x em 50% de EtOH diluído em PBS, 1x em 25% de EtOH diluído em PBS e 2x em PBS.
NOTA: Todas as etapas de reidratação são feitas com as lâminas em um rack de lâminas e com as várias soluções de EtOH em placas de coloração.
- Asse as lâminas em uma placa quente a 60 °C por 1 h e depois deixe as lâminas atingirem a temperatura ambiente (RT). Para remover a cera de parafina das lâminas, coloque-as em um suporte de lâminas e abaixe-as em uma mancha cheia de xileno. Repita em xileno fresco por 3 x 5 min.
2. Adaptação do protocolo 14 do RNAscope (ISH comercializado)
- Preparando soluções e sondas
- Preparar a solução TEG para o pré-tratamento das amostras seccionadas, combinando 25 mM de Tris-HCl a pH 8, 10 mM de EDTA e 50 mM de glicose. Fazer um volume suficiente para que todas as amostras sejam tratadas ou preparar o excesso; filtrar-esterilizar e armazenar em RT; Misture a solução corretamente antes de usá-la.
- Prepare 1x tampão de lavagem suficiente (consulte a Tabela de Materiais) para todas as lavagens e guarde-o em RT.
- Em banho-maria ou incubadora a 40 °C, aqueça as sondas de interesse por 10 min. Em seguida, prepare as sondas de acordo com o protocolo ISH14.
- Pré-tratamento de amostras
- Em vez de usar a solução de pré-tratamento recomendada, mergulhe as lâminas em uma placa de coloração contendo tampão TEG por 4 h a 60 ° C.
NOTA: A incubação do tampão TEG pode ser estendida por até 6 h se o tecido for particularmente denso ou se a recuperação do antígeno do motivo de ligação ao RNA de interesse for um desafio. Nesse caso, um ponto de parada pode ser adicionado aqui, as lâminas secas e mantidas em uma geladeira a 4 °C durante a noite. - Retire as amostras do tampão e deixe-as secar. Em seguida, aspire as amostras com uma caneta de barreira hidrofóbica, mergulhe-as em outra placa de coloração contendo protease IV e incube-as em um forno por 45 min a 40 ° C.
NOTA: Recomendamos protease IV para camundongos adultos. Se estiver usando camundongos mais jovens, como P0, recomendamos o uso de uma enzima mais suave, como a protease III, por 30 min. Ao solucionar problemas para diferentes idades, observe a integridade do tendão e verifique se há degradação. Se o tecido estiver se dissociando ou se degradando, diminua o tempo de incubação ou altere a protease usada. - Lave as amostras novamente em tampão TEG por 30 min e, em seguida, hibridize as sondas de acordo com o protocolo ISH14.
- Adicione 50-100 μL de reagente de montagem em cima da amostra. Coloque uma lamínula em cima das amostras para microscopia. Ao criar imagens, use uma ampliação alta (objetiva de 40x ou superior) para visualizar o sinal.
- Em vez de usar a solução de pré-tratamento recomendada, mergulhe as lâminas em uma placa de coloração contendo tampão TEG por 4 h a 60 ° C.
3. Adaptação do protocolo HCR ISH15
- Postfixação
- Prepare 4% de PFA (em PBS), solução de PBT (0,1% de Tween-20 em PBS) e proteinase K em PBS (concentração estoque de 10 mg / mL, concentração final de 5 μg / mL).
- Para iniciar a primeira etapa do sufixo, mergulhe as lâminas em uma placa de coloração contendo 4% de PFA por 5 min; em seguida, remova a solução de PFA a 4% e guarde-a para uso em etapas posteriores. Enxágue as lâminas por 2 x 5 min em solução PBT e remova a solução PBT. Coloque as lâminas em uma placa de coloração contendo solução de proteinase K (5 μg / mL) por 5 min e enxágue-as na solução PBT 2 x 5 min.
- Comece a segunda etapa de sufixo e mergulhe as lâminas no PFA a 4% por 5 min.
NOTA: Este é o mesmo PFA de 4% economizado na etapa 3.1.2. - Enxágue as lâminas por 3 x 5 min na solução PBT.
NOTA: Todas as etapas de pós-fixação são feitas com as lâminas em um suporte de lâminas e em placas de coloração contendo as respectivas soluções.
- Acetilação
- Prepare a solução de acetilação contendo 625 μL de anidrido acético, 3,3 mL de tampão trietanolamina (TEA) 1 M e 246 mL de ddH2O. Certifique-se de misturar bem e usar imediatamente após a preparação.
- Coloque as lâminas na solução de acetilação por 10 minutos e depois enxágue 3 x 5 min em solução PBT. Enxágue as lâminas em ddH2O e deixe-as secar ao ar por 30 min. Em seguida, desenhe ao redor das amostras com uma caneta de barreira hidrofóbica.
NOTA: Todas as etapas de acetilação são feitas com as lâminas em um suporte de lâminas e em placas de coloração contendo as respectivas soluções
- Hibridização
- Pré-aqueça o tampão de hibridização a 37 °C e pré-aqueça uma câmara umidificada a 37 °C.
CUIDADO: O tampão contém formamida, um material perigoso. Se as lâminas não estiverem suficientemente secas, seque as bordas com um lenço de laboratório para remover o excesso de dH2O. - Prepare soluções de sonda adicionando 0,4 pmol de cada mistura de sonda a 100 μL de tampão de hibridização.
NOTA: Se estiver usando imagens dHCR, use uma concentração mais alta de sonda para melhorar a eficiência da hibridização da sonda. - Prepare 4 L de Tampão Citrato de Sódio de Cloreto de Sódio (20x SSC) combinando 3 M NaCl (701,1 g de NaCl em um volume final de 4 L) e 0,3 M de Na3CH6H5O7•2H2O (352,8 g) e, em seguida, ajustando o pH final para 7 adicionando HCl ou 10 N NaOH. Adicione H2O suficiente para atingir 4 L. Prepare 5x SSCT diluindo 20x SSC para 5x e adicionando 10% Tween 20 suficiente para que seja 0,1% do volume total.
- Adicione 200 μL de tampão de hibridização à amostra, coloque as lâminas em uma câmara umidificada e deixe as lâminas incubarem por 10 min. Remova a solução de hibridização e drene o excesso de tampão na lâmina, enxugando as bordas com um lenço de laboratório.
- Adicione 100 μL da solução de sonda preparada em cima da amostra e coloque uma lamínula na amostra. Incubar em estufa durante a noite ou por cerca de 12-16 h em câmara umidificada com uma solução contendo 5x SSC e 50% de formamida. Defina a temperatura para 37 °C.
NOTA: Como a incubação é durante a noite, a adição da lamínula é para minimizar a evaporação. - Deixar uma alíquota de 5x solução de SSCT num banho-maria a 37 °C e uma segunda alíquota em RT. Antecipar os volumes destas alíquotas necessários com base no número de amostras e no número de lavagens no passo seguinte.
- Pré-aqueça o tampão de hibridização a 37 °C e pré-aqueça uma câmara umidificada a 37 °C.
- Lavagem
- Usando a solução SSCT 5x previamente pré-aquecida, prepare 75% de tampão de lavagem / 25% 5x SSCT, 50% de tampão de lavagem / 50% 5x SSCT e 25% de tampão de lavagem / 75% 5x SSCT soluções.
- Para remover o excesso de sondas, mergulhe as lâminas em série por 15 min por lavagem a 37 °C em 75% de tampão de lavagem/25% 5x SSCT, 50% de tampão de lavagem/50% 5x SSCT e 25% de tampão de lavagem/75% 5x SSCT. Deixar que as lamínulas flutuem das amostras.
- Incube as lâminas 2 x 15 min com 100% 5x SSCT e, em seguida, mergulhe-as em 5x SSCT por 5 min em RT.
NOTA: As etapas de lavagem podem ser feitas com as lâminas em um suporte deslizante e em pratos de coloração contendo as respectivas soluções ou com as soluções adicionadas como gotículas.
- Amplificação
- Preparar 6 pmol de grampo h1 e 6 pmol de grampo h2 arrefecendo 2 μL de um stock de 3 μM (aquecer a 95 °C durante 90 s e arrefecer a RT). Proteja os grampos de cabelo da luz durante este processo.
NOTA: Os grampos de cabelo HCR h1 e h2 são fornecidos em um buffer de armazenamento de grampos de cabelo e estão prontos para resfriamento instantâneo. Encaixe os grampos de cabelo h1 e h2 frios em tubos separados. - Seque as lâminas enxugando suas bordas com um lenço de laboratório. Em seguida, adicione 200 μL de tampão de amplificação em cima da amostra e coloque-a em uma câmara umidificada por 30 min em RT. Prepare a mistura de grampos de cabelo adicionando grampos de cabelo h1 resfriados e grampos de cabelo h2 resfriados a 100 μL de tampão de amplificação em RT.
- Remova o tampão de amplificação e drene o excesso de líquido na lâmina, enxugando as bordas com um lenço de laboratório. Adicionar 100 μL da mistura de grampos de cabelo previamente preparada e colocar o parafilme sobre a amostra. Incubar as lâminas durante um mínimo de 4 h ou durante a noite numa câmara escura e humidificada à temperatura de saída.
- Para remover o excesso de grampos de cabelo, mergulhe as lâminas em 5x SSCT em uma placa de coloração e incube em RT por 30 min. Repita a lavagem em SSCT 5x fresco por 30 min e uma terceira vez por 5 min.
- Seque as lâminas enxugando as bordas com um lenço de laboratório e adicione 50-100 μL de reagente de montagem em cima da amostra. Coloque uma lamínula em cima das amostras para microscopia. Ao criar imagens, use uma ampliação alta (objetiva de 40x ou superior) para visualizar o sinal.
- Preparar 6 pmol de grampo h1 e 6 pmol de grampo h2 arrefecendo 2 μL de um stock de 3 μM (aquecer a 95 °C durante 90 s e arrefecer a RT). Proteja os grampos de cabelo da luz durante este processo.
Access restricted. Please log in or start a trial to view this content.
Resultados
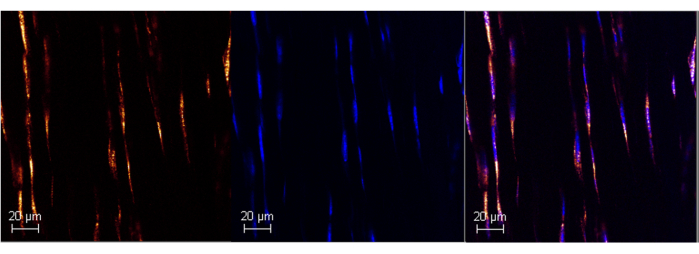
Figura 1: Expressão de RNA poli A no tendão de Aquiles de camundongos adultos usando RNAScope. Imagem representativa da marcação bem-sucedida de Poly A no tendão de Aquiles de camundongo (painel esquerdo) usando o ensaio ISH comercializado. A colocalização com DAPI confirma a especificidade da sonda (painéi...
Access restricted. Please log in or start a trial to view this content.
Discussão
Neste artigo, descrevemos as modificações feitas para alavancar as ferramentas ISH existentes, de modo que possam ser usadas em tecidos tendinosos com alto grau de especificidade e sensibilidade. Como o tendão é um tecido altamente denso em matriz, ajustes de protocolo geralmente devem ser feitos para atingir graus semelhantes de penetração e especificidade da sonda. Esses métodos específicos de permeabilização e estratégias de amplificação de sinal do tecido tendinoso são ...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Os autores agradecem a Jenna Galloway e aos membros do Galloway Lab por seu apoio e incentivo no desenvolvimento e solução de problemas desses protocolos.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 1 M triethanolamine buffer | |||
| 10% Formalin solution | |||
| 10% Tween-20 | |||
| 20x Saline Sodium Citrate buffer | |||
| 4% PFA | |||
| ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 | ACD | 323100 | |
| Acetic Anhydride | |||
| Axio Imager Microscope | ZEISS | ||
| C57BL/J6 mice | JAX ID: 000664 | ||
| Coverslips | Fisher | 12-541-042 | |
| ddH2O | |||
| ETDA | Thermofisher | AM9262 | |
| EtOH | |||
| Glucose | VWR Chemicals BDH | BDH9230-500G | |
| HCR RNA-FISH Bundle | Molecular Instruments Inc. | ||
| HybEZ II Hybridization System | ACD | ||
| Immedge Barrier Pen | Vector Laboratories | H4000 | |
| Leica SPE Confocal Microscope | Leica | ||
| Parafilm | Fisher | ||
| Phosphate-buffered saline (PBS, 1x) | Invitrogen | AM9625 | Dilute 10x PBS in milli-Q water to get 1x solution |
| Protease IV | |||
| Proteinase K | Roche | 3115836001 | |
| RNAscope H2O2 and Protease Reagents | ACD | PN 322381 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V3 |
| RNAscope Multiplex Fluorescent Detection Kit | ACD | PN 323110 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 |
| RNAscope Target Retrieval reagents | ACD | 322000 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V4 |
| RNAscope Wash Buffer | ACD | PN 310091 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V5 |
| RNAscope Probe Diluent | ACD | 300041 | |
| Slide holder | StatLab | 4465A | |
| Staining Dish with Lid | StatLab | LWS20WH | |
| Superfrost Plus Microscope slides | Fisher | 1255015 | treated, charged slides |
| Tris-HCl | |||
| Xylene | Sigma-Aldrich | 534056-4L |
Referências
- Sharma, P., Maffulli, N. Tendon injury and tendinopathy: healing and repair. Journal of Bone and Joint Surgery. 87 (1), 187-202 (2005).
- Brent, A. E., Schweitzer, R., Tabin, C. J. A somitic compartment of tendon progenitors. Cell. 113 (2), 235-248 (2003).
- Noden, D. M. The role of the neural crest in patterning of avian cranial skeletal, connective, and muscle tissues. Developmental Biology. 96 (1), 144-165 (1983).
- Chen, J. W., Galloway, J. L. The development of zebrafish tendon and ligament progenitors. Development. 141 (10), 2035-2045 (2014).
- Schweitzer, R., et al. Analysis of the tendon cell fate using Scleraxis, a specific marker for tendons and ligaments. Development. 128 (19), 3855-3866 (2001).
- Tsai, S., Nödl, M., Galloway, J. Bringing tendon biology to heel: Leveraging mechanisms of tendon development, healing, and regeneration to advance therapeutic strategies. Developmental Dynamics. 250 (3), 393-413 (2021).
- Kannus, P., et al. Location and distribution of non-collagenous matrix proteins in musculoskeletal tissues of rat. The Histochemical Journal. 30 (11), 799-810 (1998).
- Kannus, P. Structure of the tendon connective tissue. Scandinavian Journal of Medicine & Science in Sports. 10 (6), 312-320 (2000).
- Thorpe, C. T., Birch, H. L., Clegg, P. D., Screen, H. R. C. The role of the non-collagenous matrix in tendon function. International Journal of Experimental Pathology. 94 (4), 248-259 (2013).
- Lin, T. W., Cardenas, L., Soslowsky, L. J. Biomechanics of tendon injury and repair. Journal of Biomechanics. 37 (6), 865-877 (2004).
- Grinstein, M., et al. A distinct transition from cell growth to physiological homeostasis in the tendon. eLife. 8, e48689(2019).
- Villaseñor, S., Grinstein, M. Two-photon microscopy for the study of tendons. Journal of Visualized Experiments. , e65853(2023).
- Qin, C., et al. The cutting and floating method for paraffin-embedded tissue for sectioning. Journal of Visualized Experiments. (139), e58288(2018).
- Wang, F., et al. RNAscope®: A Novel In Situ RNA Analysis Platform for Formalin-Fixed Paraffin-Embedded Tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Choi, H. M. T., et al. Third-generation situ hybridization chain reaction: multiplexed, quantitative, sensitive, versatile, robust. Development. 145 (12), 165753(2018).
- Jackson, D. J., Herlitze, I., Hohagen, J. A whole mount in situ hybridization method for the gastropod mollusc Lymnaea stagnalis. Journal of Visualized Experiments. (109), e53968(2016).
- Young, A. P., Jackson, D. J., Wyeth, R. C. A technical review and guide to RNA fluorescence in situ hybridization. PeerJ. 8, e8806(2020).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados