Method Article
PyDesigner v1.0: Uma implementação Pythonic do pipeline DESIGNER para ressonância magnética de difusão
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui apresentamos o PyDesigner, um pipeline de processamento de ressonância magnética de difusão (dMRI) baseado em Python capaz de corrigir artefatos típicos de dMRI e produzir imagens de tensor de difusão (DTI), imagens de curtose de difusão (DKI), imagens de bola de fibra (FBI), modelagem de microestrutura (integridade da substância branca [WMTI] e substância branca da bola de fibra [FBWM]) e saídas de tractografia.
Resumo
O PyDesigner é um pacote de software baseado em Python baseado no parâmetro de difusão original EStImation com pipeline Gibbs and NoisE Removal (DESIGNER) (Dv1) para pré-processamento de dMRI e estimativa de tensor. Este software é fornecido abertamente para pesquisas não comerciais e não pode ser usado para atendimento clínico. O PyDesigner combina ferramentas de FSL e MRtrix3 para realizar redução de ruído, correção de toque de Gibbs, correção de movimento de correntes parasitas, mascaramento cerebral, suavização de imagem e correção de viés de Rician para otimizar a estimativa de múltiplas medidas de difusão. Ele pode ser usado em várias plataformas no Windows, Mac e Linux para derivar com precisão métricas comumente usadas de conjuntos de dados DKI, DTI, WMTI, FBI e FBWM, bem como ODFs de tractografia e arquivos .fib. Também é independente de formato de arquivo, aceitando entradas na forma de formato .nii, .nii.gz, .mif e dicom. Fácil de usar e fácil de instalar, este software também produz métricas de controle de qualidade que ilustram gráficos de relação sinal-ruído, voxels discrepantes e movimento da cabeça para avaliar a integridade dos dados. Além disso, esse pipeline de processamento de dMRI oferece suporte ao processamento de vários conjuntos de dados de tempo de eco e apresenta personalização de pipeline, permitindo que o usuário especifique quais processos são empregados e quais saídas são produzidas para atender a uma variedade de necessidades do usuário.
Introdução
A ressonância magnética de difusão (dMRI) é amplamente aplicada para o estudo não invasivo das propriedades microestruturais do cérebro. Embora muitos métodos de dMRI tenham sido propostos, dois comumente usados são a imagem por tensor de difusão (DTI) e a imagem de curtose difusional (DKI). Essas técnicas estão intimamente relacionadas, sendo o DKI uma extensão do DTI que inclui a quantificação da não-Gaussianidade difusional1. Ambos fornecem uma variedade de medidas de difusão escalar e permitem a construção da tractografia de fibras da substância branca. Uma vantagem importante do DTI e do DKI é que eles têm uma base sólida em física de difusão, de modo que sua validade não depende de suposições detalhadas sobre a microestrutura do tecido 2,3. Isso permite que o DTI e o DKI sejam aplicados em todo o cérebro e corpo para participantes de qualquer idade e estado de doença.
Como as imagens brutas ponderadas por difusão (DWIs) são degradadas por vários fatores, incluindo ruído de sinal, movimento, toque de Gibbs e distorção de correntes parasitas, o pré-processamento deve ser empregado antes do cálculo de quaisquer quantidades de difusão4. O pré-processamento de DWIs é um campo ativo de pesquisa que agora está altamente desenvolvido. No entanto, combinar as etapas de processamento necessárias em um único pipeline que forneça resultados consistentes é um desafio porque várias configurações definidas pelo usuário devem ser ajustadas dependendo dos detalhes da aquisição de dMRI e porque a ordem em que as etapas de pré-processamento são executadas afeta o resultado. Por esse motivo, o pipeline do parâmetro de difusão EStImation with Gibbs and NoisE Removal (DESIGNER, GitHub: NYU-DiffusionMRI/DESIGNER) foi inicialmente proposto em 2016 para otimizar, padronizar e agilizar o pré-processamento para DWIs5. O DESIGNER Dv1 conta com ferramentas de software incorporadas em FSL, MRtrix3, MATLAB e Python para criar um processo DWI contínuo e completo - que engloba a correção de imagem por meio de pré-processamento e estimativa de tensor de difusão/curtose5. Com sinalizadores de controle para ativar ou desativar as etapas de pré-processamento, as correções de DWI podem ser executadas seletivamente. O DESIGNER pré-processa em uma ordem específica - (i) análise de componentes principais de Marchenko-Pastur (MP-PCA) denoising6, (ii) correção de toque de Gibbs7, (iii) correção de distorção de imagem ecoplanar (EPI)8, correção de corrente parasita9, correção de movimento10 e substituição de outlier11, (iv) correção de campo de polarização B1, (v) geração de máscara cerebral, (vi) suavização, (vii) correção de viés de ruído Rician e (viii) normalização b0. O pré-processamento nesta ordem específica melhora a precisão e a relação sinal-ruído (SNR) efetiva5. Vale a pena notar que cada etapa do PyDesigner é opcional e pode ser empregada ou ignorada com base na preferência do usuário. A suavização, por exemplo, pode não ser uma etapa de pré-processamento necessária para alguns conjuntos de dados. Embora ajude a mitigar erros de filtragem, pode não ser necessário para conjuntos de dados de qualidade ultra-alta. Dessa forma, os usuários podem optar por empregar apenas as etapas necessárias para seus dados.
A implementação do DESIGNER em todas as plataformas é um desafio devido às diferenças nos sistemas operacionais e nas configurações do ambiente. Em particular, o fato de o DESIGNER Dv1 ser escrito principalmente em MATLAB cria problemas significativos de portabilidade decorrentes de requisitos de configuração complicados necessários para habilitar a interface Python-MATLAB. Além disso, diferentes combinações de MATLAB, Python e versões de dependência comprometem a reprodutibilidade desse pipeline. Por esse motivo, desenvolvemos o PyDesigner, que é totalmente baseado em Python. Esse pipeline de processamento de dMRI não apenas permite um pré-processamento contínuo, mas também permite que o pipeline seja incorporado a um contêiner do Docker, o que aumenta muito a portabilidade e a reprodutibilidade. Além disso, ao substituir o código MATLAB, o PyDesigner evita todas as taxas de licenciamento e melhora a acessibilidade.
Este pipeline de processamento de dMRI aumenta a abordagem mãos-livres introduzida pelo DESIGNER, adiciona vários novos recursos e incorpora ferramentas de FSL e MRtrix3 para realizar o pré-processamento. Bibliotecas matemáticas padrão do Python, como Numpy12, SciPy13 e CVXPY14,15, foram usadas para substituir as partes do MATLAB do DESIGNER pelo código Python. Este software é fornecido abertamente e está disponível no Github16. O objetivo deste artigo não é validar ou comparar nosso software com software semelhante, mas sim fornecer aos usuários um guia passo a passo para processar seus dados com o PyDesigner, caso optem por fazê-lo.
Protocolo
Todos os dados usados para desenvolver e testar o software foram coletados sob um protocolo de estudo aprovado pelo conselho de revisão institucional.
NOTA: O PyDesigner foi criado no MacOS 10.14 e requer um Windows Subsystem for Linux (WSL) para ser executado no Windows. Os mesmos comandos podem ser usados para sistemas Linux/Mac.
1. Compatibilidade do sistema operacional
- Para sistemas Windows, siga o link para instalar o WSL17 e prossiga para a etapa 2.6.
- Para sistemas baseados em Unix (como Linux e Mac), vá para a seção 2.
NOTA: Os sistemas baseados em Unix podem instalar nativamente o PyDesigner e suas dependências.
2. Instale o FSL
NOTA: Todos os testes do PyDesigner foram feitos com o FSL versão 6.0.2 e recomenda-se o uso da v6.0.2 ou superior.
- Vá para o wiki FSL18 e selecione Baixar/Instalar. Para usuários do Windows, vá para a etapa 2.6.
- Escolha Baixar FSL sob o título Instalando o FSL. Em seguida, selecione a Licença de Software de Instalação FSL, leia o contrato e selecione concordo.
- Preencha os campos obrigatórios e selecione Registrar.
- Escolha o link Baixar FSL (primeiro link na página). Em seguida, selecione o link de instruções de instalação (segundo link na página). Siga estas instruções para baixar o FSL para cada sistema operacional.
NOTA: O FSL leva cerca de 10 minutos para ser instalado. - Para verificar a instalação bem-sucedida, insira o seguinte em uma nova janela de terminal:
$ flirt -version. Isso deve retornar o número da versão. Prossiga para a etapa 3. - Para usuários do Windows, siga o guia de instalação do WSL para FSL19.
3. Crie um ambiente Conda
- Instale um pacote conda para gerenciar ambientes Python. Os usuários podem escolher Miniconda ou Anaconda.
- Baixar Miniconda/Anaconda20. Os usuários de Mac e Linux prosseguem para a etapa 3.2.
- Para Windows, configure o conda no WSL21.
NOTA: As instruções a seguir são para o Miniconda, mas os mesmos comandos funcionam para o Anaconda. Embora o conda não suporte mais o Python 3.7, ainda é possível criar um ambiente conda com o Python 3.7, permitindo que esse protocolo seja implementado exatamente como descrito.
- Insira o seguinte no terminal para criar o ambiente conda:
$ conda create -n mri python=3.7. Use esse ambiente conda para todas as atividades relacionadas ao PyDesigner.- Quando solicitado com
Proceed ([y]/n)?Entery.
NOTA: -n flag: Use isso para indicar o nome do ambiente conda. No vídeo, o ambiente conda é chamado de ressonância magnética.
NOTA:python=A versão 3.7 é recomendada, pois o PyDesigner foi construído e testado nesta versão.
- Quando solicitado com
4. Instale o MRtrix3
- Ative o ambiente conda digitando o seguinte comando:
$ conda activate mri - Abra a página de download do MRtrix322 e siga as instruções de download para o sistema operacional apropriado. O código de instalação solicitará a confirmação de certas dependências. Enter
ypara continuar o processo de download.- Imprima a documentação do MRtrix3 para confirmar a instalação digitando o seguinte no terminal:
$ mrinfo -h
- Imprima a documentação do MRtrix3 para confirmar a instalação digitando o seguinte no terminal:
5. Instale o PyDesigner
- Abra uma nova janela de terminal e ative o ambiente conda inserindo o seguinte no terminal:
$ conda activate mri - Execute o comando:
$ pip install PyDesigner-DWI - Para verificar a instalação bem-sucedida, imprima o número da versão digitando o seguinte no terminal:
$ pydesigner -v- Se o erro "módulo não encontrado" aparecer, consulte a seção Solução de problemas no final do protocolo (seção 10)
NOTA: $ pydesigner -h imprimirá a documentação de ajuda que explica os comandos e a sintaxe do software. O PyDesigner também pode ser baixado via Github. Consulte as instruções de instalação no website23.
- Se o erro "módulo não encontrado" aparecer, consulte a seção Solução de problemas no final do protocolo (seção 10)
6. Preparação de dados
NOTA: O PyDesigner funciona com vários tipos de arquivo, como .nii, .nii.gz, .mif e DICOM. No vídeo, os DICOMs são classificados e convertidos em NIfTI antes de usar esse pipeline de processamento de dMRI. Os arquivos NIfTI são mais fáceis de trabalhar porque são desidentificados e há um arquivo por sequência.
- Para classificar os DICOMs, use a ferramenta DicomSort24.
NOTA: Qualquer outra ferramenta de classificação DICOM disponível que realize a mesma tarefa é permitida. - Depois de classificar os DICOMs, converta-os em arquivos NIfTI usando dcm2niix25.
- Instale o dcm2niix digitando o seguinte comando no terminal:
$ sudo apt installdcm2niix - Uma vez baixado, digite o seguinte no terminal para verificar a instalação imprimindo a documentação do dcm2nii:
$ dcm2niix -h
- Instale o dcm2niix digitando o seguinte comando no terminal:
- Para executar o dcm2niix, digite o seguinte comando no terminal:
$ dcm2niix -f %s_%p_%d -o pathtosavefolder/ pathtorawdicoms/
NOTA: -f: Para especificar a estrutura de nome de arquivo para o arquivo de saída, siga esta tag com qualquer estrutura de nome de arquivo desejada. O vídeo usa: -f %s_%p_%d . %s, %p e %d são notações para série, número de protocolo e descrição, respectivamente. Qualquer estrutura de arquivo funcionará. As saídas esperadas do dcm2niix são arquivos .bval, .bvec, .json e .nii. Se os dados TOPUP foram adquiridos, haverá arquivos TOPUP .json e .nii.
7. Uso básico do PyDesigner
NOTA: O PyDesigner pode levar até 1,5 h para um único conjunto de dados, dependendo dos parâmetros de processamento e dos recursos do computador.
- Para executar o processamento padrão, use o sinalizador
-sou-standard. Esse comando executará o pipeline de pré-processamento recomendado na seguinte ordem: redução de ruído6, correção de toque de Gibbs7, correção de distorção EPI8, correção de movimento de corrente parasita9, criação de máscaras cerebrais e de líquido cefalorraquidiano (LCR), suavização, correção de viés de Rician, detecção de outlier11, estimativa de tensor e criação de mapas paramétricos. - Para executar o comando básico do PyDesigner, primeiro ative o ambiente conda digitando o seguinte comando no terminal:
$ conda activate mri - Imprima a documentação de ajuda do software digitando o seguinte comando:
$ pydesigner -h. Esta documentação explica todos os comandos e sintaxe possíveis para o PyDesigner. - Use o exemplo a seguir para estruturar o comando.
$ pydesigner -s --verbose --rpe_pairs 1 -o ./output_folder/ ./input_folder/input1.nii ./input_folder/input2.nii - Observe os seguintes sinalizadores importantes:
--verbose: Use isso para exibir o console do PyDesigner à medida que ele processa dados (não necessário).--rpe pairs #: Use isso para permitir que os usuários com uma sequência TOPUP acelerem o processo de correção de distorção EPI8 usando TOPUP. O # refere-se a quantos volumes B0 da sequência TOPUP serão usados. O uso de #=1 evita a superestimação do campo de distorção e reduz o tempo necessário para criar esse campo. Os usuários têm usado rotineiramente #=1 com resultados confiáveis. Ao usar #=1, o PyDesigner usará como padrão o primeiro volume.
8. Uso avançado do PyDesigner: Adaptando o software às necessidades individuais dos usuários
NOTA: Ao adaptar o PyDesigner, omita o -s sinalizador; esse sinalizador é o comando para pré-processamento automático.
- Ative o ambiente conda digitando o seguinte comando no terminal:
$ conda activate mri - Consulte o site do software23 para obter uma lista de sinalizadores; cada sinalizador permite que o usuário execute cada parte do PyDesigner individualmente.
- Estruture o comando. Comece com os sinalizadores para cada sequência que o usuário gostaria de executar. Em seguida, direcione o PyDesigner para os arquivos de entrada e saída. Por exemplo:
$ pydesigner flag1 flag2 flag3 -o ./output_folder/ ./input_folder/input1.nii - Exemplo 1: Execute o ajuste do tensor sozinho (sem pré-processamento adicional).
- Ative o ambiente conda digitando o seguinte comando no terminal:
$ conda activate mri - Consulte o site do PyDesigner23 para obter uma lista de sinalizadores. Este exemplo usa
-m. -m: Adicione este sinalizador para limitar o ajuste do tensor apenas aos voxels cerebrais. Isso acelera o ajuste do tensor + detecção de outliers.- Use o exemplo a seguir para estruturar o comando:
$ pydesigner -m -o ./output_folder/ ./input_folder/input1.nii
NOTA: A entrada para ajuste de tensor é um arquivo .nii pré-processado.
- Ative o ambiente conda digitando o seguinte comando no terminal:
- Exemplo 2: execute a redução de ruído sozinha (sem pré-processamento adicional).
- Ative o ambiente conda inserindo o seguinte no terminal:
$ conda activate mri - Consulte o site do PyDesigner23 para obter uma lista de sinalizadores. Este exemplo usa
-n. - -n: Adicione esse sinalizador para concluir a etapa de redução de ruído. Insira o arquivo DWI pré-processado anteriormente rotulado DWI_preproccessed.nii, que é encontrado na pasta de saída de cada sujeito.
- Use o exemplo a seguir para estruturar o comando:
$ pydesigner -n -o ./output_folder/ ./input_folder/input1.nii
- Ative o ambiente conda inserindo o seguinte no terminal:
9. Futuras atualizações do PyDesigner
NOTA: As atualizações podem ser encontradas no site23.
- Para atualizar o PyDesigner, insira o seguinte em uma nova janela de terminal:
$ pip install --upgrade PyDesigner-DWI
NOTA: Se o PyDesigner foi instalado por meio do Github16, ele também deve ser atualizado usando o git.
10. Solução de problemas de instalação
- Ao baixar o PyDesigner, o código de erro "módulo não encontrado" aparecerá se houver um módulo Python ausente.
- Corrija um módulo ausente inserindo o seguinte no terminal:
$ pip install nameofmodule - Em seguida, repita o primeiro comando de instalação digitando o seguinte no terminal :
$ pip install PyDesigner-DWI - O PyDesigner agora deve estar totalmente instalado. Por favor, volte para a etapa 5.3. Se o mesmo erro ocorrer, repita esse processo até que todos os módulos Python ausentes sejam instalados.
- Corrija um módulo ausente inserindo o seguinte no terminal:
11. Solução de problemas de erros de saída
- Se ocorrer algum problema, revise o arquivo log_command.json para identificar erros de pré-processamento. Revise os arquivos intermediários para determinar o erro específico.
- Para obter ajuda na solução de problemas, envie uma solicitação na página16 do GitHub.
12. Executando o PyDesigner com um conjunto de dados de exemplo
- Baixe o conjunto de dados de exemplo do OpenNeuro aqui26.
NOTA: Este conjunto de dados de exemplo contém dados brutos e pré-processados. Os dados brutos destinam-se a ser a entrada para o processamento de exemplo do PyDesigner. Os dados pré-processados consistem nas saídas geradas depois que os dados brutos foram processados usando o pipeline de processamento dMRI em um iMac OS v12.4. Os usuários podem baixar os dados brutos, processar esses dados usando o PyDesigner e comparar suas saídas com as saídas de exemplo fornecidas para garantir que o PyDesigner esteja sendo executado corretamente. Os dados brutos estão contidos na pasta principal (/PyDesigner Example Dataset/sub-01) e os dados pré-processados estão contidos na pasta de derivativos (/PyDesigner Example Dataset/derivatives/sub-01). A menos que especificado de outra forma, o conjunto de dados OpenNeuro será baixado na pasta de usuário padrão. - Abra um novo terminal no Mac/Linux ou o WSL no Windows.
NOTA: Para executar o pipeline de processamento padrão do PyDesigner, siga a estrutura de comando de exemplo abaixo. Insira os seguintes comandos abaixo no terminal/WSL. Neste exemplo, onde " ./user_download_folder/" indica o caminho para a pasta de dados de exemplo. Esse caminho será específico para onde os dados de exemplo são armazenados no computador de usuários individuais. - Para criar uma nova pasta para saídas, insira o seguinte no terminal:
$ mkdir ./user_download_folder/ ds004945-download /PyDesigner_Outputs - Para ativar o ambiente conda, insira o seguinte no terminal:
$ conda activate mri - Para executar o PyDesigner no conjunto de dados de exemplo, insira o seguinte no terminal:
$ pydesigner - o ./user_download_folder/ds004945-download/PyDesigner_Outputs -s ./user_download_folder/ds004945-download/sub-01/dwi/sub-01_dwi.nii
OBSERVAÇÃO: neste exemplo, ./user_download_folder/ indica o caminho para a pasta de dados de exemplo. Esse caminho será específico para o computador de cada usuário. Os comandos nas etapas 12.3 a 12.5 seguem a estrutura da etapa 8.3. Esse comando emprega o sinalizador -s, o sinalizador de processamento padrão do PyDesigner, que executa as seguintes etapas de processamento nesta ordem: redução de ruído6, correção de toque de Gibbs7, não distorção 8,9, mascaramento cerebral, suavização e correção de viés rician. - Se o software estiver instalado corretamente, todas as saídas serão preenchidas na pasta PyDesigner_Outputs recém-criada. Compare os arquivos de pré-processamento produzidos pelo PyDesigner com a pasta pré-processada do conjunto de dados de exemplo encontrada na pasta "derivados" baixada do OpenNeuro.
- Consulte a seção de resultados representativos para comparar seus mapas de métricas com os dos resultados do conjunto de dados de exemplo.
Resultados
O software PyDesigner aplica várias etapas de correção de imagem aos dados brutos de difusão e gera saídas usadas para melhorar a precisão dos arquivos brutos ao realizar análises. Cada etapa disponível no pipeline foi previamente validada por meio de publicações revisadas por pares 5,6,7,8,9,10,11, conforme discutido na introdução. As saídas do software podem ser usadas em análises como perfis de tractografia, matrizes de conectividade, análises de voxel, análises de ROI, análises de fODF, TBSS e análises baseadas em fixel.
O site do software23 lista todos os arquivos de saída gerados durante o pipeline de pré-processamento. Depois que cada prompt for executado, o console produzirá uma descrição de todos os processos concluídos. Existem 3 tipos de arquivos de saída: arquivos de processamento, métricas e controle de qualidade. A estrutura do diretório de saída é mostrada na Figura 1. Esses arquivos estão disponíveis ao usar o pré-processamento padrão (consulte a seção 7 do protocolo). Se o usuário precisar de uso mais avançado (consulte a seção 8 do protocolo), os arquivos de saída disponíveis dependerão de quais processos foram concluídos.
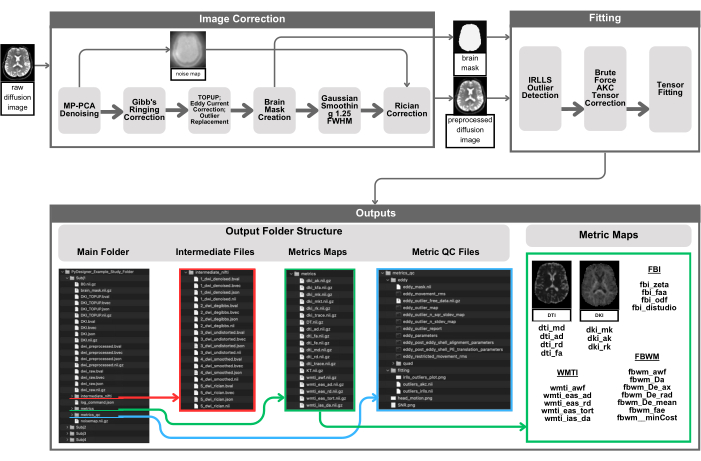
Figura 1: Representação visual do pipeline do PyDesigner. O pré-processamento começa fornecendo uma entrada 4D DWI para PyDesigner (canto superior esquerdo), que então passa por MP-PCA denoising para produzir um 4D DWI sem ruído e um mapa de ruído 3D. O DWI 4D com ruído passa por correção de toque de Gibbs, correção de polarização de Rician, TOPUP, correção de corrente parasita e correção de outlier. Uma máscara cerebral é então calculada para etapas subsequentes, detecção de outliers e ajuste de tensor para acelerar os cálculos, executando-os apenas dentro da máscara cerebral. As saídas podem ser encontradas em subdiretórios na pasta de processamento do assunto principal - intermediate_nifti, métricas e metrics_qc. Observe que as capturas de tela das saídas do PyDesigner não pretendem ser uma lista exaustiva de todas as saídas possíveis, mas sim fornecer um exemplo visual esquemático do que os usuários podem esperar. As saídas, tanto as saídas finais quanto os arquivos intermediários, variam de acordo com os dados de entrada do usuário e os sinalizadores de processamento empregados. Clique aqui para ver uma versão maior desta figura.
Processamento
Os arquivos de processamento são usados durante o pipeline do PyDesigner e são armazenados no diretório de saída raiz. Para cada etapa de pré-processamento, os arquivos DWI intermediários são salvos na pasta de saída "intermediate_nifti", conforme mostrado na Figura 1. Esses arquivos devem ser referenciados ao abordar quaisquer problemas com processamento ou saídas para avaliar cada etapa do pipeline separadamente.
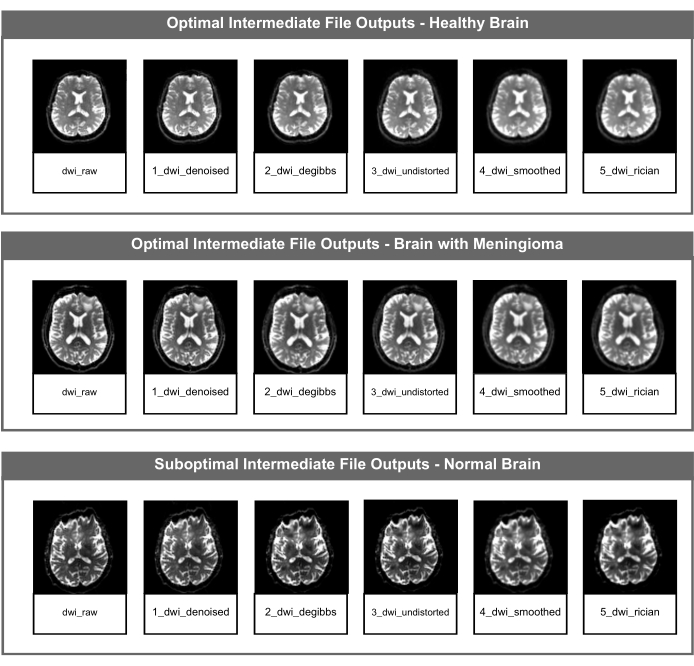
Figura 2: Arquivos DWI NifTI intermediários ótimos e abaixo do ideal. A figura mostra o arquivo NIfTI intermediário para cada etapa de correção de imagem do pipeline do PyDesigner. A linha superior é um exemplo de saídas de arquivo intermediário ideais usando dados de um cérebro adulto saudável, a linha do meio é um exemplo de saídas de arquivo intermediário ideais usando dados de um cérebro patológico (meningioma) e a linha inferior exibe saídas de arquivo intermediário abaixo do ideal usando dados de um cérebro adulto saudável com um artefato de suscetibilidade não relacionado à estrutura ou saúde do cérebro. Clique aqui para ver uma versão maior desta figura.
Métricas
Essa pasta contém todos os mapas paramétricos que o PyDesigner calcula (consulte a Figura 1). Isso inclui mapas paramétricos para DTI/DKI, imagem de bola de fibra (FBI)/substância branca de bola de fibra (FBWM) e métricas de integridade do trato da substância branca (WMTI) (Tabela 1)16.

Tabela 1: Intervalo esperado de valores para métricas DTI/DKI e FBI/FBWM. A tabela inclui uma lista de métricas robustas de DTI, DKI, FBI e FBWM geradas pelo PyDesigner e seus intervalos de valores esperados. As sequências e valores b necessários (s/mm2) para derivar cada métrica também são listados. As métricas de DTI listadas são FA, MD, AD e RD. As métricas DKI listadas são MK, AK, RK e KFA. A métrica do FBI listada é a FAA. As métricas FBWM listadas são AWF, DA, DE_AX, DE_RAD e FAE.
Os usuários podem realizar o controle de qualidade visual e de valor (QC) das métricas de difusividade média (MD), anisotropia fracionada (FA) e curtose média (MK) para identificar resultados abaixo do ideal. Se essas métricas não forem ideais pelos padrões descritos abaixo, os usuários deverão examinar cada arquivo intermediário descrito na Figura 2 para determinar qual etapa de pré-processamento não foi bem-sucedida.
O controle de qualidade visual é usado para identificar resultados abaixo do ideal (por exemplo, problemas de ajuste de tensor e artefatos). Recomendamos o uso do ImageJ para controle de qualidade visual para garantir que nenhuma manipulação seja feita na imagem por meio dos padrões do software. A linha superior da Figura 3 mostra mapas métricos típicos de MD, FA e MK usando um limite biologicamente plausível de 0-3 μm2/ms, 0-1 μm2/ms e 0-2 μm2/ms, respectivamente (Figura 3 [linha superior]). O mapa MD deve ter os valores mais altos nos ventrículos e valores altos na substância cinzenta cortical (Figura 3A [linha superior]). O mapa de FA deve ter tratos aparentes de substância branca que são claros em todo o cérebro (Figura 3B [linha superior]). O mapa MK deve ter valores altos em WM e valores mais baixos na substância cinzenta e no LCR (Figura 3C [linha superior]). A Figura 3D [linha superior] é um exemplo de um mapa de métricas com problemas de ajuste de tensor, o que resulta em clusters de voxels de valor zero. Se ocorrer algum problema, revise o arquivo log_command.json para encontrar erros de pré-processamento. Revise os arquivos intermediários para determinar o erro específico. Para obter ajuda na solução de problemas, envie uma solicitação na página do GitHub do PyDesigner.
O controle de qualidade de valor é usado para identificar se os voxels de um determinado mapa métrico são relativamente uniformes entre os sujeitos para cada conjunto de dados. O intervalo de valores esperado para cada mapa e conjunto de dados depende dos dados e dos parâmetros PyDesigner. Em nosso conjunto de dados de teste, tivemos picos consistentes nas faixas de 8000 a 10.000, 2500 a 4000 e 5000 a 13.000 para MD, FA e MK, respectivamente, usando o binning de histograma padrão FSLeyes. A linha inferior da Figura 3 fornece exemplos de variabilidade do histograma. A Tabela 1 contém os valores esperados do eixo x para essas métricas. Voxels mais altos ou mais baixos são característicos do conjunto de dados ou indicam um problema de artefato ou pré-processamento (Figura 3D [linha inferior]).
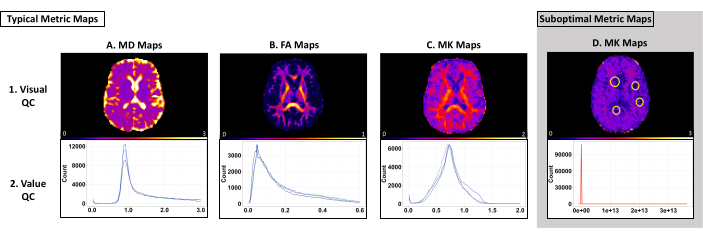
Figura 3: Exemplos de mapas de métricas e histogramas do PyDesigner com resultados ótimos e abaixo do ideal. A linha superior mostra exemplos de mapas MD, FA e MK de assunto único usados para controle de qualidade visual. A linha inferior mostra histogramas de vários assuntos usados para o valor QC. (AC) Exemplos típicos de mapas métricos e histogramas que passam no controle de qualidade visual e de valor. Cada linha azul nos histogramas para cada tipo de métrica representa um conjunto de dados individual. Observe que cada conjunto de dados segue uma curva semelhante e se enquadra em um intervalo semelhante. (D) Um exemplo de um mapa métrico que não passa no controle de qualidade visual ou de valor. Observe como a linha vermelha no histograma mostra uma curva diferente da de A-C. Os voxels zero circulados neste mapa métrico são devidos a problemas de ajuste do tensor durante o pré-processamento (Painel D, linha superior). Este histograma é um exemplo de voxels geralmente mais altos ou mais baixos em um conjunto de dados do que o esperado (Painel D, linha inferior). Clique aqui para ver uma versão maior desta figura.
Controle de qualidade
Depois que o PyDesigner processar os dados, a pasta metrics_qc (consulte a Figura 1) deverá ser usada para identificar conjuntos de dados abaixo do ideal. Para cada conjunto de dados, o PyDesigner gera três gráficos usados para controle de qualidade.
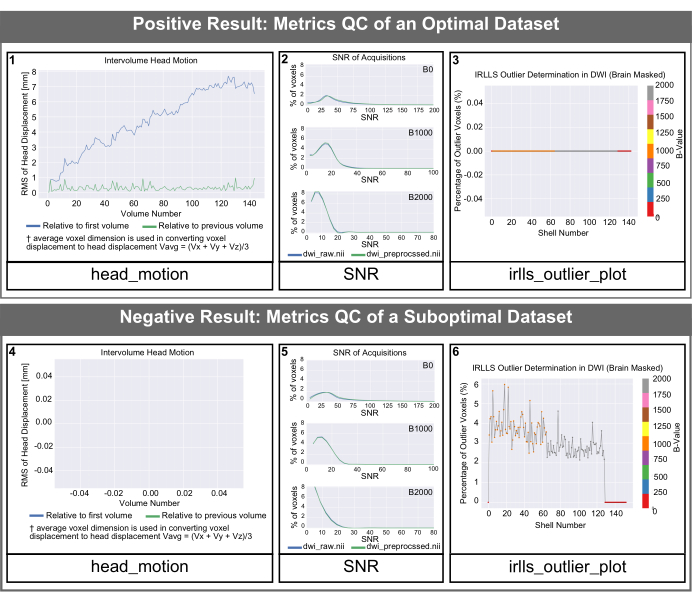
Figura 4: histogramas de controle de qualidade gerados pelo PyDesigner para conjuntos de dados ótimos e abaixo do ideal. SNR, movimento de cabeça entre volumes e histogramas atípicos gerados pelo PyDesigner. Ambas as linhas representam dados de um cérebro adulto saudável. A linha superior é um exemplo de histogramas de controle de qualidade para um conjunto de dados ideal. A linha inferior mostra as saídas de controle de qualidade de um conjunto de dados abaixo do ideal com um artefato de suscetibilidade não relacionado à estrutura ou saúde do cérebro. Observe que o tamanho do texto dos rótulos nas saídas padrão do PyDesigner é menor do que eles aparecerão nesta figura. Aumentamos o tamanho do texto nesta figura para facilitar a leitura. Clique aqui para ver uma versão maior desta figura.
Os gráficos head_motion na Figura 4 mostram o deslocamento da cabeça em relação ao primeiro volume e ao volume anterior. Como visto na Figura 4 (painel 1), o deslocamento da cabeça é normalmente pequeno e o PyDesigner se ajusta para esses artefatos de movimento no pipeline de processamento padrão usando o programa FSL Eddy junto com TOPUP para correção de movimento e corrente parasita9. Para conjuntos de dados abaixo do ideal, o gráfico head_motion pode parecer vazio, conforme mostrado na Figura 4 (painel 4). Isso indica que a correção de movimento por correntes parasitas não foi bem-sucedida, portanto, o PyDesigner não conseguiu gerar um gráfico. Os arquivos de log de correção de correntes parasitas podem ser encontrados na subpasta de redemoinhos dentro da pasta metrics_qc (consulte a Figura 1). O gráfico de relação sinal-ruído (SNR) exibe 3 gráficos. Cada gráfico é para um valor b diferente e mostra os dados pré-processados e brutos. Para um conjunto de dados ideal, o pico de SNR de dados brutos deve ser ≥5 (Figura 4 [painel 2]). Conjuntos de dados abaixo do ideal terão um pico de SNR de dados brutos de ≤3 (Figura 4 [painel 5]). Idealmente, os usuários devem ver que o pico de SNR para todos os valores b aumenta ligeiramente, mas não drasticamente. O gráfico de valores discrepantes é encontrado na pasta de ajuste dentro metrics_qc e mostra a porcentagem de valores discrepantes no conjunto de dados (Figura 4 [painel 3 e 6]). Um bom conjunto de dados deve ter uma baixa porcentagem de valores discrepantes, normalmente inferior a 5% (Figura 4 [painel 3]). Um conjunto de dados abaixo do ideal terá uma grande porcentagem de valores discrepantes, conforme mostrado na Figura 4 (painel 6).
Exemplos de resultados do conjunto de dados
Depois que o PyDesigner terminar de processar o conjunto de dados de exemplo, todas as saídas deverão estar contidas na pasta "PyDesigner_Outputs". Essas saídas podem ser comparadas com as encontradas na pasta "derivados" empacotada com o conjunto de dados de exemplo baixado do OpenNeuro (processado no MacOS 12.4). Se o software for executado corretamente, a estrutura de arquivos de "PyDesigner_Outputs" e "derivados" será exatamente a mesma. Da mesma forma, os gráficos SNR, movimento da cabeça e outliers encontrados na subpasta "metrics_qc" devem corresponder aos da Figura 5A. Os mapas métricos (encontrados na subpasta "métricas") podem ser comparados por meio de software de imagem como FSLeyes, MRIcron, ImageJ, etc. Histogramas de valores de FA, MD e MK de pré-processados/métricas podem ser vistos na Figura 5B. Observe que todos os histogramas de métrica mostrados na Figura 5B são dimensionados com base nas escalas de valores de métrica sugeridas na Tabela 1.
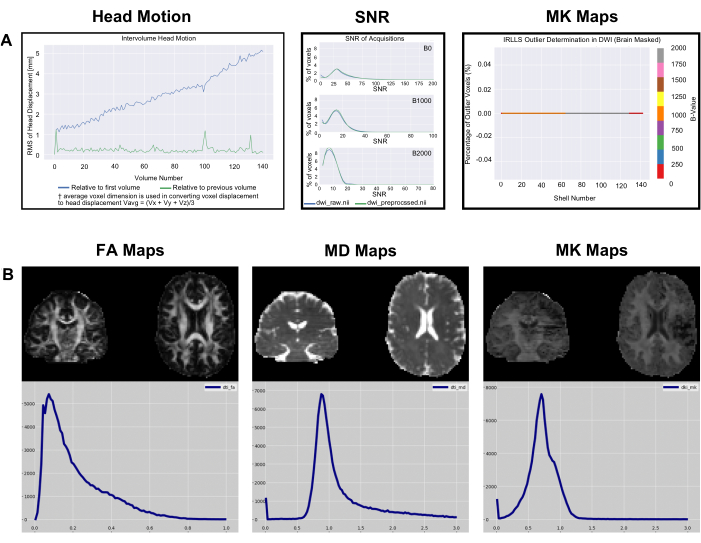
Figura 5: Exemplo de histogramas métricos e de controle de qualidade métricos. (A) SNR, movimento da cabeça entre volumes e histogramas discrepantes gerados pelo PyDesigner para os dados de exemplo baixados do OpenNeuro. Observe que o tamanho do texto foi aumentado nos gráficos nesta figura para facilitar a leitura. (B) Histogramas de mapas de métricas de assunto único de contagens de valores de voxel para FA, MD e MK para o mesmo conjunto de dados, visualizados por meio do FSLeyes v6.0. Clique aqui para ver uma versão maior desta figura.
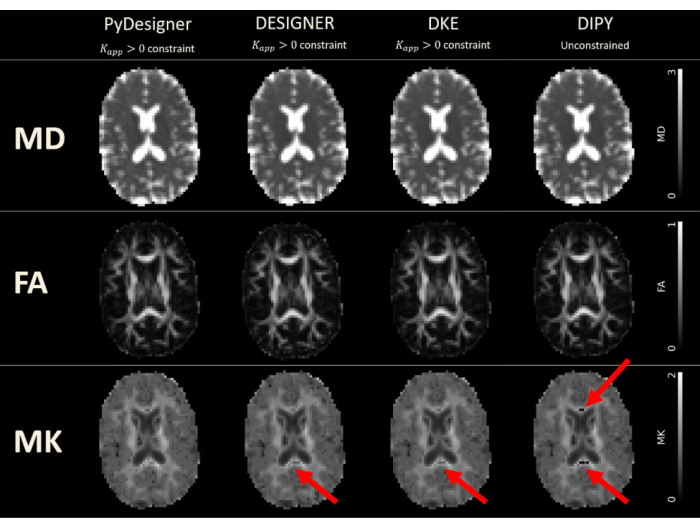
Figura 6: Mapas DTI e DKI derivados de PyDesigner, DESIGNER, DKE e DIPY. O ajuste do tensor foi realizado com a restrição Kapp > 0 no PyDesigner, DESIGNER5 e Diffusional Kurtosis Estimator (DKE)27, enquanto o ajuste irrestrito foi usado no Diffusion Imaging in Python (DIPY)28 devido a limitações de software. As unidades para MD são micrômetros quadrados por milissegundo (μm2/ms), enquanto as outras métricas são adimensionais. Desistências devido a um problema genérico da correção de toque de Gibbs podem ser vistas em mapas MK produzidos por DESIGNER, DKE e DIPY. Essa figura foi reproduzida com permissão de Dhiman et al.29. Clique aqui para ver uma versão maior desta figura.
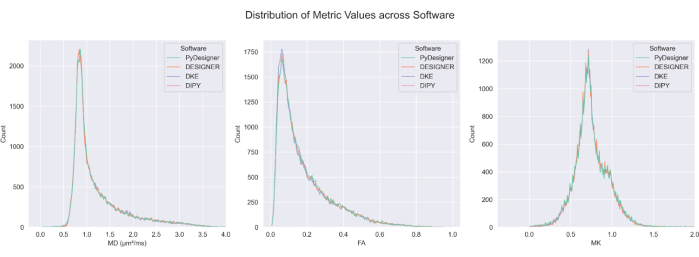
Figura 7: Comparação de FA, MD e MK entre pipelines. A distribuição dos valores calculados para FA, MD e MK de PyDesigner, DESIGNER5, DKE27 e DIPY28 em cérebros excluídos do LCR é semelhante na maioria dos voxels. O cálculo do mapa métrico entre os métodos é comparável. Essa figura foi reproduzida com permissão de Dhiman et al.29. Clique aqui para ver uma versão maior desta figura.
Discussão
A principal motivação para o desenvolvimento do PyDesigner foi implementar os elementos-chave do DESIGNER enquanto substituía todo o código MATLAB por Python, permitindo assim maior portabilidade e acessibilidade. PyDesigner e DESIGNER Dv1 produzem saídas quase idênticas29. No entanto, existem algumas opções adicionais, configurações padrão e pequenas correções de bugs incluídas no PyDesigner. A documentação online do PyDesigner16 descreve isso em detalhes.
O PyDesigner também produz resultados semelhantes às ferramentas de análise DKI Diffusional Kurtosis Estimator (DKE)27 e Diffusion Imaging in Python (DIPY)28 comumente usadas29 (ver Figura 6 e Figura 7), mas os mapas gerados com base no dMRI pré-processado pelo DESIGNER são considerados mais precisos devido ao seu algoritmo de ajuste, conforme demonstrado por Ades-Aron et al.5. Consulte a Figura 6 para obter uma comparação dos mapas de métricas MD, FA e MK do PyDesigner, DESIGNER5, DKE27 e DIPY28. A Figura 7 mostra a comparação dos histogramas MD, FA e MK de cada pipeline. A combinação de ajuste de tensor restrito, detecção de outliers e correção do coeficiente de curtose aparente produz um ajuste de tensor mais robusto e preciso, como visto no PyDesigner e no DESIGNER5.
Uma vantagem do PyDesigner sobre o DESIGNER Dv1 é que ele está disponível por meio do contêiner NeuroDock Docker30, o que aumenta muito a portabilidade e simplifica a instalação. Esse contêiner é executado em todas as principais plataformas de sistema operacional compatíveis com o Docker, incluindo Windows, Mac OS e várias distribuições Linux. Com base nos comentários do usuário, o PyDesigner v2.0 incluirá atualizações para o PyDesigner Docker Container. As melhorias introduzidas pela nova versão do PyDesigner e a introdução de um Dockerfile de vários estágios resolverão todos os problemas existentes que os usuários estão enfrentando. Se os usuários tiverem problemas com a instalação, recomendamos enviar perguntas para a páginade discussão 16 do PyDesigner. A tecnologia de contêiner do Docker também permite a implantação direta em clusters de alto desempenho (HPCs) para processamento em lote de DWIs rapidamente em clusters locais compatíveis com o Docker.
O PyDesigner também inclui cálculos de modelagem microestrutural que vão além do DKI, incluindo WMTI3, FBI e FBWM. Para WMTI, um conjunto de dados DKI padrão é adequado e os parâmetros microestruturais associados são calculados por padrão. No entanto, deve-se enfatizar que a validade do WMTI é restrita a regiões da substância branca com alta AF (ou seja, FA ≥ 0,4). Algumas métricas WMTI têm precisão limitada devido à suposição de alinhamento paralelo de axônios em qualquer voxel31. FBI 32,33,34 é um método distinto de dMRI aplicável em toda a substância branca cerebral, que requer um alto valor b (ou seja, b ≥ 4000 s/mm 2) e dados de dMRI amostrados com um mínimo de 64 direções de codificação de difusão (junto com dados para b = 0). As principais saídas do FBI são a função de densidade de orientação de fibra (fODF) para cada voxel de substância branca, que pode ser usada para tractografia de substância branca e serve como entrada para FBWM, bem como a anisotropia fracionada intra-axonal (FAA). O FBWM utiliza os dados de dMRI do DKI e do FBI para estimar os mesmos parâmetros do WMTI, mas com maior precisão, e pode ser aplicado em toda a substância branca, independentemente do valor de FA. Assim, se esses dados adicionais estiverem disponíveis, as estimativas do FBWM são preferidas às do WMTI35. Tal como acontece com o FBI, o FBWM só foi validado na substância branca cerebral adulta.
Além das invariantes rotacionais fornecidas pelo PyDesigner (orientação RAS), os arquivos .fib específicos do DSIstudio (orientação LPS) são produzidos separadamente para DTI, DKI e FBI. Os arquivos .fib contêm informações direcionais ODF para gerar perfis de tractografia para cada método. Os perfis de tractografia resultantes podem ser usados para gerar matrizes de conectividade nos níveis de assunto e grupo. Os arquivos .fib DKI e FBI contêm informações multidirecionais que retransmitem informações de fibra cruzada que não estão disponíveis com a tractografia DTI. Além disso, dentro de cada arquivo .fib, os vários invariantes rotacionais de cada método foram incluídos, que podem ser usados em várias combinações como critérios para semear, executar e parar a tractografia. Mais detalhes sobre o DSIstudio podem ser encontrados em seu site36.
Outro recurso notável do PyDesigner é a entrada de vários arquivos, que permite lidar com várias entradas de arquivo - NifTi (.nii), NifTi compactado (.nii.gz), DICOM (.dcm) e formato de arquivo MRtrix (.mif). O PyDesigner pode identificar automaticamente as informações de aquisição dos metadados do cabeçalho, independentemente do formato de entrada, e executar correções de acordo, dando suporte a uma abordagem sem intervenção. Independentemente das diferenças nos protocolos, o mesmo comando (veja acima) pode ser usado para processar uma ampla variedade de DWIs. Assim, este software economiza tempo e esforço, minimizando as etapas e comandos de pré-processamento manual. Em uma atualização lançada recentemente (v1.0-RC10), isso foi aprimorado com a introdução da compatibilidade para vários conjuntos de dados de tempo de eco (multi-TE). Isso permite que o PyDesigner execute etapas de pré-processamento de imagem, que são amplamente independentes do TE, em um DWI multi-TE para produzir uma imagem com o mínimo de ruído e artefatos. Os cálculos de tensores dependentes de TE são então realizados em cada TE separadamente para produzir métricas de difusão ou curtose.
Observamos que o PyDesigner v1.0 não representa um ponto de extremidade no design de pipelines de pré-processamento de imagem. O desenvolvimento e validação de ferramentas de processamento de imagens é uma área ativa de pesquisa. Em particular, durante a preparação deste manuscrito, novos desenvolvimentos em redução de ruído de imagem e remoção de anel de Gibbs foram propostos, resultando no lançamento de um pipeline Designer revisado, Designer Dv237, que inclui redução de ruído aprimorada e correção de artefatos de Gibbs para dMRI adquiridos com aquisições parciais de Fourier35, e está disponível no DESIGNER Github38. Da mesma forma, a conformidade organizacional do BIDS39 é uma faceta importante de qualquer pipeline de análise de ressonância magnética. O formato BIDS permite um compartilhamento de dados mais eficiente e a implementação de pipeline, estabelecendo uma estrutura de conjunto específica para todos os tipos de dados de MRI. Embora o PyDesigner não seja atualmente compatível com o BIDS, uma próxima versão do PyDesigner incluirá saídas compatíveis com o BIDS. Além disso, o PyDesigner foi originalmente escrito usando o Python versão 3.7. No momento desta publicação, o Python 3.7 agora é considerado desatualizado. No entanto, as informações contidas neste manuscrito são independentes da versão Python. Ou seja, os procedimentos descritos aqui que são funções importantes de pré-processamento (por exemplo, redução de ruído, correção de toque de Gibbs, etc.) seguirão o mesmo fluxo de trabalho operacional e conceitual, independentemente das alterações nos procedimentos de instalação. É importante ressaltar que, como acontece com todos os softwares baseados em Python, as versões futuras do PyDesigner serão atualizadas para uma nova versão do Python. Nós nos esforçamos para validar e integrar esses desenvolvimentos ainda mais ao longo do tempo.
Para obter a versão mais atualizada do PyDesigner, incluindo qualquer nova documentação relevante para as atualizações de versão do Python, os leitores são incentivados a consultar o site antes de iniciar uma nova análise e participar do fórum de discussão onde perguntas sobre o PyDesigner podem ser enviadas16. A implementação do Docker para portabilidade é chamada de NeuroDock30, que contém o PyDesigner e suas dependências para permitir o processamento em uma ampla variedade de plataformas.
Divulgações
Nenhum.
Agradecimentos
Somos gratos pelas discussões úteis com Olivia Horn, Daniel Lench e Graham Warner.
A pesquisa relatada nesta publicação foi apoiada, em parte, pelos subsídios do National Institutes of Health R01AG054159, R01AG057602, R01AG055132, R01DC014021, R01NS110347, R21DA050085, F31NS108623, P20GM109040, P50DC000422, T32GM008716 e T32DC014435. O conteúdo é de responsabilidade exclusiva dos autores e não representa necessariamente as opiniões oficiais dos Institutos Nacionais de Saúde. Financiamento adicional foi fornecido pela Fundação Litwin.
Materiais
| Name | Company | Catalog Number | Comments |
| Python version 3.7 or above | Python Software Foundation | https://www.python.org/ | |
| FMRIB Software Library (FSL) verison 6.0.2 or above | University of Oxford Centre for Integrative Neuroimaging | https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/ | |
| MRtrix3 version 3.0_RC3 or above | numerous contributors | https://www.mrtrix.org/ | |
| Anaconda | Anaconda | https://anaconda.org/ | |
| Computer | Apple | Mac OS 10.14 | Built on Mac OS 10.14; tested on Mac OS 12.4, Mac OS 13, Windows 11 via WSL |
Referências
- Jensen, J. H., Helpern, J. A. MRI quantification of non-Gaussian water diffusion by kurtosis analysis. NMR Biomed. 23 (7), 698-710 (2010).
- Basser, P. J. Relationships between diffusion tensor and q-space MRI. Magn Reson Med. 47 (2), 392-397 (2002).
- Fieremans, E., Jensen, J. H., Helpern, J. A. White matter characterization with diffusional kurtosis imaging. Neuroimage. 58 (1), 177-188 (2011).
- Le Bihan, D., Poupon, C., Amadon, A., Lethimonnier, F. Artifacts and pitfalls in diffusion MRI. J Magn Reson Imaging. 24 (3), 478-488 (2006).
- Ades-Aron, B., et al. Evaluation of the accuracy and precision of the diffusion parameter EStImation with Gibbs and NoisE removal pipeline. Neuroimage. 183, 532-543 (2018).
- Veraart, J., Novikov, D. S., Christiaens, D., Ades-aron, B., Sijbers, J., Fieremans, E. Denoising of diffusion MRI using random matrix theory. Neuroimage. 142, 394-406 (2016).
- Kellner, E., Dhital, B., Kiselev, V. G., Reisert, M. Gibbs-ringing artifact removal based on local subvoxel-shifts. Magn Reson Med. 76 (5), 1574-1581 (2016).
- Holland, D., Kuperman, J. M., Dale, A. M. Efficient correction of inhomogeneous static magnetic field-induced distortion in Echo Planar Imaging. Neuroimage. 50 (1), 175-183 (2010).
- Andersson, J. L. R., Sotiropoulos, S. N. An integrated approach to correction for off-resonance effects and subject movement in diffusion MR imaging. Neuroimage. 125, 1063-1078 (2016).
- Godenschweger, F., et al. Motion correction in MRI of the brain. Phys Med Biol. 61 (5), 32-56 (2016).
- Andersson, J. L. R., Graham, M. S., Zsoldos, E., Sotiropoulos, S. N. Incorporating outlier detection and replacement into a non-parametric framework for movement and distortion correction of diffusion MR images. Neuroimage. 141, 556-572 (2016).
- Harris, C. R., et al. Array programming with NumPy. Nature. 585 (7825), 357-362 (2020).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nat Methods. 17 (3), 261-272 (2020).
- Agrawal, A., Verschueren, R., Diamond, S., Boyd, S. A rewriting system for convex optimization problems. J Control Decis. 5 (1), 42-60 (2018).
- Diamond, S., Boyd, S. CVXPY: A Python-embedded modeling language for convex optimization. , Available from: http://arxiv.org/abs/1603.00943 (2016).
- PyDesigner. , Available from: https://github.com/muscbridge/PyDesigner (2023).
- How to install Linux on Windows with WSL. , Available from: https://learn.microsoft.com/en-us/windows/wsl/install (2023).
- FSL Wiki. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki (2024).
- Fsl Instillation. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/FslInstallation/Windows (2024).
- Installing conda. , Available from: https://conda.io/projects/conda/en/stable/user-guide/install/download.html (2024).
- Steps to install Anaconda on Windows Ubuntu terminal. , Available from: https://gist.github.com/kauffmanes/5e74916617f9993bc3479f401dfec7da (2024).
- Install MRtrix3. , Available from: https://www.mrtrix.org (2024).
- PyDesigner Read the Docs. , Available from: https://pydesigner.readthedocs.io/en/latest/ (2024).
- dicomSort. , Available from: https://github.com/muscbridgelab/dicomSort (2024).
- dcm2niix. , Available from: https://github.com/rordenlab/dcm2niix (2024).
- PyDesigner Example Dataset. , Available from: https://openneuro.org/datasets/ds004945 (2024).
- Tabesh, A., Jensen, J. H., Ardekani, B. A., Helpern, J. A. Estimation of tensors and tensor-derived measures in diffusional kurtosis imaging. Magn Reson Med. 65 (3), 823-836 (2011).
- Garyfallidis, E., et al. Dipy, a library for the analysis of diffusion MRI data. Front Neuroinform. 8, 8(2014).
- Dhiman, S., et al. PyDesigner: A Pythonic implementation of the DESIGNER pipeline for diffusion tensor and diffusional Kurtosis imaging. bioRxiv. , (2021).
- Neurodock. , Available from: https://hub.docker.com/r/dmri/neurodock (2024).
- Jelescu, I. O., et al. One diffusion acquisition and different white matter models: How does microstructure change in human early development based on WMTI and NODDI. Neuroimage. 107, 242-256 (2015).
- Jensen, J. H., Russell Glenn, G., Helpern, J. A. Fiber ball imaging. Neuroimage. 124, 824-833 (2016).
- Moss, H. G., Jensen, J. H. High fidelity fiber orientation density functions from fiber ball imaging. NMR Biomed. 35 (1), e4613(2022).
- Moss, H. G., McKinnon, E. T., Glenn, G. R., Helpern, J. A., Jensen, J. H. Optimization of data acquisition and analysis for fiber ball imaging. Neuroimage. 200, 690-703 (2019).
- McKinnon, E. T., Helpern, J. A., Jensen, J. H. Modeling white matter microstructure with fiber ball imaging. Neuroimage. 176, 11-21 (2018).
- DSIstudio. , Available from: https://dsi-studio.labsolver.org (2024).
- Chen, J., et al. Optimization and validation of the DESIGNER dMRI preprocessing pipeline in white matter aging. ArXiv. , arXiv:2305.14445v2 (2024).
- DESIGNER. , Available from: https://github.com/NYU-DiffusionMRI/DESIGNER (2024).
- Gorgolewski, K. J., et al. The brain imaging data structure, a format for organizing and describing outputs of neuroimaging experiments. Sci Data. 3, 160044(2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados