É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Condensados sintéticos e arquiteturas semelhantes a células de nanoestruturas de DNA anfifílico
Neste Artigo
Resumo
Apresentamos um protocolo para preparação de condensados biomoleculares sintéticos constituídos por nanoestrelas de DNA anfifílico a partir de seus oligonucleotídeos de DNA constituintes. Os condensados são produzidos a partir de um único componente de nanoestrela ou de dois componentes e são modificados para sustentar a transcrição in vitro de RNA a partir de um molde de DNA incorporado.
Resumo
Gotículas e condensados sintéticos estão se tornando constituintes cada vez mais comuns de sistemas biomiméticos avançados e células sintéticas, onde podem ser usados para estabelecer compartimentalização e sustentar respostas semelhantes à vida. As nanoestruturas de DNA sintético demonstraram potencial significativo como blocos de construção formadores de condensado devido à sua forma programável, funcionalização química e comportamento de automontagem. Recentemente, demonstramos que as "nanoestrelas" de DNA anfifílico, obtidas pela marcação de junções de DNA com porções hidrofóbicas, constituem uma solução particularmente robusta e versátil. Os condensados de DNA anfifílicos resultantes podem ser programados para exibir arquiteturas internas complexas e multicompartimentais, responder estruturalmente a vários estímulos externos, sintetizar macromoléculas, capturar e liberar cargas úteis, sofrer transformações morfológicas e interagir com células vivas. Aqui, demonstramos protocolos para a preparação de condensados de DNA anfifílicos a partir de oligonucleotídeos de DNA constituintes. Abordaremos (i) sistemas monocomponentes formando condensados uniformes, (ii) sistemas de dois componentes formando condensados núcleo-casca e (iii) sistemas nos quais os condensados são modificados para suportar a transcrição in vitro de nanoestruturas de RNA.
Introdução
As células sintéticas são dispositivos em escala micrométrica (10-50 μm) construídos de baixo para cima para replicar funções e estruturas de células biológicas existentes 1,2. As células sintéticas são frequentemente ligadas por membranas construídas a partir de vesículas de bicamada lipídica 3,4,5,6,7, polimerossomos 8,9 ou proteinossomos10,11, que também podem ser usados para estabelecer compartimentalização interna12,13. Inspiradas nas organelas sem membrana conhecidas por sustentar várias funcionalidades em células vivas14, estruturas como coacervados poliméricos, condensados biomoleculares e hidrogéis estão ganhando força como alternativas versáteis e robustas para estabelecer compartimentalização externa e interna em células sintéticas 15,16,17,18.
Aproveitando o versátil kit de ferramentas da nanotecnologia de DNA19, várias soluções foram desenvolvidas para projetar gotículas e condensados sintéticos a partir da automontagem de nanoestruturas artificiais de DNA, cujo tamanho, forma, funcionalidade, valência e interações mútuas podem ser programados com precisão20. Gotículas ou condensados de DNA são biocompatíveis e podem atuar como andaimes para células sintéticas e organelas, hospedando reações químicas e biomoleculares21, computando informações22,23, capturando e liberando cargas24,25 e sustentando respostas estruturais26.
Entre os diversos projetos de nanoestruturas de DNA formadoras de condensado, as nanoestrelas de DNA anfifílicas - apelidadas de estrelas C - provaram ser robustas e versáteis27. As estrelas C são motivos ramificados simples que consistem em uma junção fixa de DNA (normalmente de quatro vias), da qual emergem braços de DNA de fita dupla (ds)28. Os braços são então derrubados com porções hidrofóbicas, normalmente colesterol, tornando as nanoestruturas anfifílicas e conduzindo sua condensação após um simples recozimento de um pote. Os condensados C-star oferecem programabilidade estrutural e funcional precisa, incluindo a possibilidade de estabelecer arquiteturas multicompartimentais29,30, respondendo estruturalmente a gatilhos de DNA e cátions31, sintetizando macromoléculas29, capturando e liberando cargas úteis32 e interagindo com células vivas33. Abaixo, descreveremos e discutiremos protocolos para produzir condensados de estrela C a partir de seus oligonucleotídeos constituintes.
O protocolo resume a preparação de condensados unários (um componente) e binários (dois componentes), utilizando três designs diferentes de C-star (Figura 1) - "Não responsivo", "TMSD-responsivo" e "Modelagem de RNA". A estrela C "não responsiva" (painel A) consiste em quatro "fitas centrais" com sequências distintas formando a junção de quatro vias. Quatro oligonucleotídeos idênticos modificados por colesterol estão conectados à junção, garantindo que uma molécula de colesterol esteja presente no final de cada braço. As estrelas C não responsivas constituem andaimes simples e inertes para condensados unários e binários. Na estrela C "responsiva a TMSD" (painel B), a conexão entre as fitas colesterolizadas e a junção é garantida por uma fita "Toeholding bridge", que apresenta um domínio "toehold" de fita simples (ss) DNA. Na presença de uma fita de DNA invasora com um domínio de toehold complementar, uma reação de deslocamento da fita mediada pelo toehold pode ser desencadeada34, em que o invasor desloca a ponte Toeholding, quebrando a conexão entre a junção e as porções hidrofóbicas e desencadeando a desmontagem da rede de DNA32. Finalmente, a estrela C de "modelagem de RNA" (painel C) inclui uma modificação de "Base" complementar a uma fita de "Ponte", a última das quais liga o modelo de ssDNA transcritível para o aptâmero de brócolis29. Detalhes da sequência dos oligonucleotídeos constituintes para os três tipos de projetos de estrelas C mencionados aqui podem ser encontrados na Tabela Suplementar 1 e em trabalhos anteriores 29,30,32.
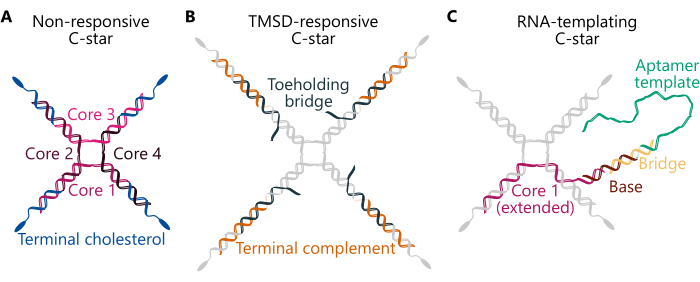
Figura 1. Esquemas de três designs diferentes de nanoestrelas de DNA anfifílico (estrelas C). Sequências de oligonucleotídeos para vários exemplos de estrelas C descritas aqui podem ser encontradas na Tabela Suplementar 1. (A) Esquema de uma estrela C projetada para formar condensados não responsivos, com as fitas de oligonucleotídeos componentes "Núcleo 1", "Núcleo 2", "Núcleo 3", "Núcleo 4" (colorido em tons de rosa) e "Colesterol terminal" (colorido em azul). Cada cor única representa uma fita de oligonucleotídeo de sequência única. "Núcleo 1" e "Núcleo 3" são parcialmente complementares a "Núcleo 2" e "Núcleo 4", mas não complementares entre si. (B) Esquema de uma estrela C projetada para se desmontar após a adição de uma fita invasora por meio do deslocamento da fita mediada pelo apoio do pé, conforme descrito no trabalho anterior32. Esta estrela C é composta por fitas "Core" e "Colesterol terminal" (coloridas em cinza), bem como uma fita "Complemento terminal" (mostrada em laranja) e uma fita "Toeholding bridge" (mostrada em azul-petróleo escuro). Este último contém uma saliência de seis nucleotídeos à qual uma fita invasora adequadamente projetada pode se ligar e, subsequentemente, deslocar totalmente a fita "Toeholding bridge", que causa a dissociação da junção central da nanoestrela (composta por "Core 1, 2, 3 e 4") dos duplexes compostos pelas fitas "Complemento terminal" e "Colesterol terminal". (C) Esquema de uma estrela C funcionalizada com um molde de DNA para um aptâmero de RNA. Este também é composto pela fita "Colesterol terminal" e "Núcleo 2, 3 e 4" (todos mostrados em cinza), bem como uma versão estendida da fita "Núcleo 1" (mostrada em rosa), uma fita "Base" (marrom), uma fita "Ponte" (amarelo) e o "Modelo de Aptâmero" (verde). O duplex de DNA composto pelas duas últimas fitas forma a região promotora da polimerase T7, que marca o local de início da transcrição. Clique aqui para ver uma versão maior desta figura.
Os condensados C-star se formam após o recozimento térmico dos oligonucleotídeos constituintes, que no protocolo aqui apresentado é conduzido dentro de capilares de vidro selados com uma seção transversal retangular de alta proporção. Esses recipientes oferecem várias vantagens principais: i) A vedação garante que a evaporação seja completamente evitada nas etapas de recozimento (às vezes lentas); ii) O fundo plano de qualidade óptica dos capilares permite a obtenção de imagens do transiente de automontagem (ou desmontagem); iii) a alta proporção dos capilares garante que os condensados pesados se depositem em uma área ampla e plana, reduzindo as chances de coalescência e agregação em estágios posteriores do transiente de automontagem que ocorreria em recipientes em forma de cunha (por exemplo, tubos de microcentrífuga) e produzindo populações de condensado relativamente monodispersas; iv) realizar o recozimento em um capilar de vidro alongado minimiza a exposição da amostra a interfaces hidrofóbicas (ar, plástico ou óleo), que foram observadas perturbando a automontagem ao recrutar os oligonucleotídeos anfifílicos colesterolizados. Uma vez concluído o protocolo de montagem, os condensados podem ser extraídos dos capilares de vidro para experimentos adicionais que envolvem reagentes adicionais.
Protocolo
NOTA: O protocolo é dividido em três seções. A Seção 1 descreve as etapas de pré-requisito, incluindo a preparação de oligonucleotídeos de DNA e capilares de vidro. A Seção 2 descreve a preparação de condensados de estrela C de vários projetos, incluindo projetos de um e dois componentes, e sua extração dos capilares de vidro. A Seção 3 descreve o uso de condensados de estrela C de modelagem de RNA de um componente para a síntese de um aptâmero de RNA. O usuário deve seguir as boas práticas de laboratório o tempo todo, garantir que todas as avaliações e mitigações de risco necessárias estejam em vigor e usar equipamento de proteção individual (EPI) adequado, incluindo luvas, óculos de segurança e jaleco. A limpeza dos tubos capilares de vidro requer sua sonicação, primeiro em uma solução surfactante e depois em isopropanol ou etanol. A extração de condensados de estrela C de tubos capilares requer o uso de uma caneta de riscagem de diamante para marcar e quebrar o vidro, com um risco associado de ferimentos causados por fragmentos de vidro. Os principais materiais, equipamentos e reagentes usados estão listados na Tabela de Materiais. A maioria dos oligonucleotídeos não funcionalizados é purificada pelo fornecedor usando dessalinização padrão, com exceção das fitas "núcleo estendido 1" e "modelo de Aptâmer", que são solicitadas com purificação por eletroforese em gel de poliacrilamida (PAGE). Os oligonucleotídeos modificados com colesterol são purificados pelo fornecedor usando cromatografia líquida de alta resolução (HPLC) de fase reversa.
1. Pré-requisitos
NOTA: As seguintes soluções devem ser preparadas em água ultrapura (Tipo I) e filtradas com filtros de seringa de 0,22 μm: Tampão Tris-EDTA (TE), compreendendo 10 mM Tris, 1 mM EDTA, a pH ~ 8,0; Tampão TE suplementado com NaCl 2 M; e tampão TE suplementado com NaCl 0,3 M. As soluções-tampão devem ser utilizadas no prazo de 2 semanas após a preparação e conservadas a 4 °C quando não estiverem a ser utilizadas. Além disso, uma solução de 1 vol% de detergente óptico alcalino em água ultrapura será usada para limpar os capilares de vidro.
- Preparação de oligonucleotídeos de DNA do estado liofilizado
- Centrifugue brevemente (2000 x g por 10-30 s) os tubos de oligonucleotídeos de DNA liofilizado para garantir que os pellets de DNA liofilizado se acumulem no fundo.
NOTA: A centrifugação é feita à temperatura ambiente (RT) por 10-30 s. A mini centrífuga usada aqui tem como padrão 2000 x g (6000 rpm). - Adicione a quantidade apropriada de tampão TE para reconstituir cada oligonucleotídeo a 100 μM. Vortex completamente para garantir a dissolução completa e, em seguida, gire os tubos para coletar todo o líquido conforme a etapa 1.1.1.
- Para cada solução-mãe, medir a absorvância a 260 nm e calcular a concentração final utilizando o coeficiente de extinção da sequência de oligonucleótidos em ensaio.
NOTA: O coeficiente de extinção para uma determinada sequência de oligonucleotídeos geralmente é encontrado na folha de dados do fornecedor, mas também pode ser calculado com ferramentas on-line usando o modelo de vizinho mais próximo35 ou valores tabulados do coeficiente de extinção de nucleotídeos individuais36. - Conservar os oligonucleótidos reidratados a 4 °C durante curtos períodos de tempo (até 1 semana) ou a -20 °C para armazenamento a longo prazo (até 6 meses).
- Centrifugue brevemente (2000 x g por 10-30 s) os tubos de oligonucleotídeos de DNA liofilizado para garantir que os pellets de DNA liofilizado se acumulem no fundo.
- Limpeza de tubos capilares de vidro
- Sonicate em detergente ótico alcalino a 1% para componentes óticos em água desionizada.
- Pegue o número necessário de tubos capilares de vidro a serem limpos e coloque-os em um recipiente alto e estreito (por exemplo, um béquer ou um tubo de centrífuga de 15/50 mL) de forma que os capilares não fiquem planos na base do recipiente.
- Adicione o detergente ao recipiente, enchendo-o logo acima do nível dos tubos capilares. Certifique-se de que não haja bolhas de ar presas dentro dos tubos capilares - toque para remover, se necessário. Cubra frouxamente o recipiente (por exemplo, em papel alumínio ou usando a tampa do tubo da centrífuga, certificando-se de que não esteja bem fechado).
- Defina um banho-maria de sonicação para 40 °C, coloque o recipiente coberto na vertical no banho e sonice por 30 min. Certifique-se de que a folha, se usada, não entre em contato com a água do banho de sonicação.
NOTA: O banho de sonicação usado neste estudo tem como padrão uma frequência de sonicação de 60 Hz e uma potência de sonicação de 150 W.
CUIDADO: Nunca adicione ou remova itens de um banho de sonicação durante a corrida; Sempre pause o protocolo primeiro. - Quando a sonicação estiver concluída, desligue o elemento de aquecimento do banho-maria sonicante e, em seguida, enxágue bem os capilares do recipiente com água deionizada ou ultrapura - no mínimo cinco vezes, descartando a água de enxágue a cada vez.
- Sonicate em detergente ótico alcalino a 1% para componentes óticos em água desionizada.
- Sonicar em isopropanol ou etanol.
- Nos tubos capilares de vidro no recipiente de limpeza, adicione isopropanol ou etanol (mínimo 70%), enchendo até um pouco acima do nível dos tubos capilares. Como na etapa 1.2.1.2, certifique-se de que não haja bolhas de ar presas nos tubos capilares e cubra frouxamente o recipiente.
- Coloque o recipiente na vertical no banho de sonicação e sonice por 15-30 min.
CUIDADO: Isopropanol e etanol são inflamáveis, com pontos de fulgor abaixo de RT. Minimize os volumes de solventes usados durante a limpeza escolhendo um recipiente de tamanho apropriado, certifique-se de que os recipientes estejam frouxamente cobertos e não deixe o banho de sonicação sem vigilância durante este stage. - Quando a sonicação estiver completa, descarte adequadamente o isopropanol ou etanol e seque os capilares sob nitrogênio, manuseando-os com um tecido sem fiapos.
2. Preparação e extração de condensados de estrela C (Figura 2)

Figura 2: Carregamento de misturas de estrelas C e extração de condensados de tubos capilares de vidro. Em todos os painéis, a mistura C-star foi substituída por uma solução aquosa de calceína 25 mM para ajudar na visibilidade. (A-E) Principais etapas, em ordem, a serem tomadas antes do recozimento, correspondendo às seções 2.1 e 2.2 do protocolo. (F-J) Principais etapas, em ordem, a serem seguidas após o recozimento, correspondendo à seção 2.3 do protocolo. Durante a extração (painéis (I-J)), os condensados de DNA sedimentarão do capilar para o reservatório tampão, desde que o tubo da microcentrífuga seja armazenado verticalmente. Os condensados não serão visíveis a olho nu. Clique aqui para ver uma versão maior desta figura.
- Preparação de misturas C-star para condensados monocomponentes
NOTA: Os detalhes da sequência para vários projetos de componentes individuais podem ser encontrados na Tabela Suplementar 1 e em trabalhos anteriores 29,30,32. Os protocolos de recozimento lento definidos nas etapas 2.1.9 e 2.2.2 foram desenvolvidos para garantir o relaxamento das redes de nanoestrelas em morfologias compactas, dadas as propriedades reológicas fortemente dependentes da temperatura das redes de nanoestrelas37.- Os componentes de oligonucleotídeos e tampão variam dependendo do design de estrela C desejado. Prepare uma mistura de 60 μL de condensados de estrela C de componente único na concentração de 5 μM para a estrela C de quatro braços não responsiva, a estrela C responsiva a TMSD ou a estrela C de modelagem de RNA.
- Para cada projeto, pipetar os componentes apropriados listados na Tabela Suplementar 2 em um tubo de microcentrífuga separado e misturar bem por pipetagem.
NOTA: Presume-se que todas as soluções de estoque de oligonucleotídeos estejam na concentração de 100 μM em TE. - Retire todos os 60 μL de solução C-star e pipete cuidadosamente para injetar a mistura no tubo capilar de vidro limpo e seco, conforme mostrado na Figura 2A, tomando cuidado para evitar a introdução de bolhas de ar.
- Usando uma pipeta, injete aproximadamente 9-12 μL de óleo mineral em cada extremidade do tubo capilar, tampando a amostra de forma que não haja interface livre entre a solução C-star e o ar (ver Figura 2B). Em seguida, seque cuidadosamente o tubo capilar com papel de seda para garantir que nenhum óleo esteja presente na parte externa dele (Figura 2C), tomando cuidado para não absorver óleo mineral ou solução C-star do tubo capilar. O resultado final é demonstrado na Figura 2D.
- Prepare um pequeno lote de cola epóxi de duas partes para aderir cada extremidade do tubo capilar, com o lado plano para baixo, a uma lamínula de vidro. Certifique-se de que as aberturas do tubo estejam completamente cobertas com cola, formando uma vedação contínua, conforme mostrado na Figura 2E.
- Reserve para curar por no mínimo 3 h, mas de preferência durante a noite.
- Após ~30 min de cura, inspecione a camada de cola quanto à presença de lacunas na vedação causadas por bolhas de ar. Se estiverem presentes, sele-os com uma pequena quantidade de cola.
- Enrole o tubo capilar colado à lamínula em papel alumínio, garantindo que o papel alumínio seja mantido plano na parte inferior da lamínula de vidro. A folha garante um bom contato térmico entre o mesmo e o elemento de aquecimento do termociclador (veja o próximo passo).
- Coloque a amostra embrulhada em um termociclador e recoze usando o seguinte protocolo: Mantenha a 95 ° C por 30 min, depois resfrie de 85 ° C a 50 ° C a -0,04 ° C · min-1 e, em seguida, resfrie de 50 ° C a RT a -0,5 ° C · min-1.
- Armazene os condensados C-star recozidos a 4 ° C ou RT por longos períodos (meses), desde que o capilar permaneça selado, pois o recozimento térmico esteriliza efetivamente a amostra e desnatura as nucleases. Mantenha o capilar plano durante o armazenamento e manuseio para evitar que os condensados sedimentem em uma extremidade e se agreguem.
- Preparação de misturas de estrelas C para condensados binários
NOTA: Para informar a seleção de populações de estrelas C para sistemas binários, consulte o trabalho de Malouf et al., que descreve as regras de projeto e o comportamento de fase esperado de condensados binários de estrelas C30.- Para preparar condensados binários, combine volumes de 30 μL das misturas C-star descritas na etapa 2.1 acima com um volume total de 60 μL, misture bem, carregue nos capilares e siga as etapas 2.1.4-2.1.8.
- Coloque a amostra embrulhada em um termociclador e recoze usando o seguinte protocolo: Mantenha a 95 ° C por 30 min, depois resfrie de 85 ° C a 40 ° C a -0,01 ° C · min-1 e, em seguida, resfrie de 40 ° C a RT a -0,1 ° C · min-1.
- Extração de condensados de estrela C de tubos capilares.
- Prepare um tubo de microcentrífuga grande (1,5 mL ou 2,0 mL) contendo 60 μL de NaCl 0,3 M em tampão TE.
- Desembrulhe a amostra capilar e use uma caneta de diamante para marcar a parte inferior da lamínula em cada extremidade interna da área colada (Figura 2F). Quebre a lamínula nesta região e descarte adequadamente.
- Limpe bem os tubos capilares com etanol e seque.
- Marque cada extremidade dos tubos capilares com a caneta de riscagem de diamante, primeiro com a parte inferior (Figura 2G), depois virando e marcando os tubos "com o lado direito para cima".
- Certifique-se de que as linhas de pontuação não se sobrepõem à interface óleo-água. Devem estar dentro da região aquosa para evitar que o óleo permaneça na amostra extraída.
- Retire as extremidades dos tubos capilares (Figura 2H), retendo a parte central do tubo e descartando o restante. Coloque o tubo capilar cortado no tubo de microcentrífuga preparado na etapa 2.3.1, certificando-se de que o fundo do tubo capilar entre em contato com a solução tampão (Figura 2I).
- Os condensados sedimentarão do tubo para o reservatório tampão por gravidade; permita que isso ocorra por um mínimo de 10 min (Figura 2J). Remova o tubo capilar cortado e descarte-o adequadamente. Essa abordagem de extração suave limita o risco de agregação de condensado.
3. Transcrição de um aptâmero de RNA a partir de condensados de estrela C de modelagem de RNA
NOTA: Para a produção do aptâmero de RNA de brócolis, é necessária uma solução de difluoro-4-hidroxibenzilideno imidazolidinona (DFHBI) - o pó de DFHBI é primeiro preparado como uma solução estoque a 10 mM em dimetilsulfóxido (DMSO), que é então diluído a 600 μM em água livre de RNase e DNase.
- Lavagem de condensados de estrela C de modelagem de RNA
NOTA: Os condensados de estrela C com modelagem de RNA são lavados três vezes para garantir que os oligonucleotídeos molde não ligados sejam removidos.- Deixe a solução de condensados extraídos repousar durante pelo menos 5 minutos e, em seguida, retire cerca de metade do volume do sobrenadante.
- Para um volume de 60 μL de condensados extraídos, remova entre 25-30 μL do sobrenadante.
- Pipete a partir do topo do nível do líquido para minimizar o número de condensados removidos durante esta etapa.
- Adicione o mesmo volume de NaCl 0,3 M em TE para substituir o sobrenadante removido e misture por pipetagem.
- Repita as duas etapas anteriores para um total de três ciclos.
- Deixe a solução de condensados extraídos repousar durante pelo menos 5 minutos e, em seguida, retire cerca de metade do volume do sobrenadante.
- Preparação da mistura de transcrição T7
NOTA: A mistura de transcrição T7 é preparada usando o kit de transcrição CellScript T7-FlashScribe. A etapa 3.2.2 é uma modificação do protocolo do fabricante, que pode ser encontrada aqui38. Aqui descrevemos a transcrição do aptâmero de RNA de brócolis, que induz fluorescência em DFHBI após a ligação. Para outros aptâmeros iluminados, substitua o volume de DFHBI descrito na etapa 3.2.2 pelo fluorogênio apropriado. Para outras transcrições, remova esse componente completamente.- Preparar uma solução-mãe de 10 mM de DFHBI em DMSO e, em seguida, diluir uma alíquota até obter uma concentração final de 600 μM utilizando água isenta de RNase e DNase.
- Descongele os componentes do kit de transcrição em gelo e, em seguida, pipete o seguinte em um tubo de microcentrífuga autoclavado à temperatura ambiente usando pontas de pipeta estéreis: 2 μL de tampão de transcrição 10x T7 (fornecido no kit de transcrição), 1,8 μL de 100 mM ATP, 1,8 μL de 100 mM CTP, 1,8 μL de 100 mM UTP, 2 μL de DTT 100 mM, 2 μL de DFHBI 600 μM, 0,5 μL de inibidor de RNase, 2 μL de solução enzimática T7 (fornecida no kit de transcrição).
- Misture suavemente a solução pipetando.
- Use a mistura de transcrição para a etapa 3.3 (síntese do transcrito de RNA) imediatamente após a preparação.
- Síntese de transcrição de RNA
- Em uma câmara adequada para imagens de microscopia, pipetar 3,3 μL dos condensados lavados preparados a partir de estrelas C de modelagem de RNA e adicionar o volume total da mistura de transcrição preparada anteriormente.
- Adquirir imagens microscópicas durante 18 h, começando imediatamente após a adição da mistura de transcrição aos condensados.
NOTA: As configurações de microscopia dependerão do sistema preparado. Certifique-se de que as configurações apropriadas de excitação e emissão sejam usadas para quaisquer fluoróforos na amostra, juntamente com tempos de exposição que não levarão à saturação à medida que a transcrição avança. Como em qualquer lapso de tempo, deve-se encontrar um compromisso entre um laser ou LED alto o suficiente para um bom sinal, ao mesmo tempo em que minimiza o risco de fotobranqueamento. O fotobranqueamento por longos períodos de tempo será minimizado em sistemas baseados em LED em comparação com sistemas baseados em laser. Intervalos de imagem sugeridos: 20 min nas primeiras 2 h e 30 min subsequentes.
Resultados
Após o recozimento, os condensados de estrela C podem ser visualizados diretamente no tubo capilar ou após a extração, para confirmar sua formação. Para todas as variações de projeto de estrela C, deve-se observar condensados esféricos ou poliédricos distintos com aproximadamente 10-50 μm de diâmetro, este último se formando quando ocorre a cristalização28,32. Para condensados de componente único, os condensados devem ser discretos e uniformes na ...
Discussão
O protocolo descrito aqui fornece uma abordagem para a preparação de condensados de um ou dois componentes a partir de nanoestrelas de DNA anfifílico, com variações de design para introduzir diferentes respostas nos condensados. O protocolo fornecido produz condensados em uma solução tampão de NaCl 0,3 M em TE, mas as condições tampão podem ser alteradas modificando adequadamente os volumes listados acima. Trabalhos anteriores estudaram a formação de condensados C-star em 0,2 M NaCl em TE e 0,1 M NaCl em TE ...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
LM, LDM e DT reconhecem o apoio do Conselho Europeu de Pesquisa (ERC) no âmbito do Programa de Pesquisa e Inovação Horizonte 2020 (ERC-STG No 851667 - NANOCELL). O LDM reconhece o apoio de uma bolsa de pesquisa da Royal Society para bolsistas de pesquisa (RGF / R1 / 180043) e o apoio de uma bolsa de pesquisa da Royal Society University (UF160152, URF / R / 221009).
Materiais
| Name | Company | Catalog Number | Comments |
| 0.22 μm syringe filters | Sigma-Aldrich | SLGVR33RB | |
| 24 x 60 mm #1.5 Rectangular cover glasses, Menzel Gläser | VWR | 631-0853 | |
| 2-Propanol | Sigma-Aldrich | 34683 | |
| 6 L Ultrasonic Cleaner with Digital Timer and Heat, 230 VAC | Cole-Parmer | WZ-08895-11 | |
| Araldite Rapid Adhesive 2 Part Epoxy Glue | RS | ARA-400005 | |
| Bio-Rad C1000 thermal cycler | Bio-Rad | 1851197 | |
| Brand Microcentrifuge Tube 2 mL with Locking Lid | Fisher Scientific | 15338665 | 2 mL microcentrifuge tubes for the extraction of C-star condensates |
| Diamond Scribing Pen | RS | 394-217 | |
| Difluoro-4-hydroxybenzylidene imidazolidinone (DFHBI) | Sigma-Aldrich | SML1627 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | 472301 | |
| Eppendorf PCR Clean Colorless Safe-Lock Centrifuge Tubes | Fisher Scientific | 0030123301 | 0.5 mL microcentrifuge tubes for the preparation of C-star mixtures |
| Ethanol Absolute 99.8+% | Fisher Scientific | 10437341 | 70% ethanol is sufficient for cleaning purposes |
| Fisherbrand ZX4 IR Vortex Mixer | Fisherbrand | 13284769 | |
| Hellmanex III | Hellma | 9-307-011-4-507 | |
| Hollow Rectangle Capillaries ID 0.40 x 4.00 mm, 50 mm in length | CM Scientific | 2540-50 | |
| Mineral oil | Sigma-Aldrich | 69794 | |
| Mini Centrifuge, 230 V | PRISM(TM) | Z763128 | |
| NaCl | Sigma-Aldrich | S3014 | |
| NanoDrop One Spectrophotometer | Thermo Fisher Scientific | ND-ONE-W | Used to measure absorbance of oligonucleotides for concentration calculations |
| Oligonucleotides | Integrated DNA Technologies | Custom | Oligonucleotide sequences are unique to the C-star design required. |
| ScriptGuard RNase inhibitor | CELLSCRIPT | C-SRI6310K | RNase inhibitor |
| T7-FlashScribe Transcription Kit | Cambio | C-ASF3507 | |
| Tris-EDTA buffer, 100x stock solution | Sigma-Aldrich | 574793 | |
| UltraPure DNase/RNase-Free Distilled Water | Invitrogen | 10977035 | |
| VWR Spec-Wipe 3 Wipers | VWR | 21914-758 |
Referências
- Buddingh', B. C., Hest, J. C. M. v. Artificial cells: Synthetic compartments with life-like functionality and adaptivity. Acc Chem Res. 50 (4), 769-777 (2017).
- Fanalista, F., et al. Shape and size control of artificial cells for bottom-up biology. ACS Nano. 13 (5), 5439-5450 (2019).
- Dora Tang, T. -. Y., et al. Fatty acid membrane assembly on coacervate microdroplets as a step towards a hybrid protocell model. Nat Chem. 6 (6), 527-533 (2014).
- Deshpande, S., et al. Spatiotemporal control of coacervate formation within liposomes. Nat Commun. 10 (1), 1800 (2019).
- Rubio-Sánchez, R., et al. Thermally driven membrane phase transitions enable content reshuffling in primitive cells. J Am Chem Soc. 143 (40), 16589-16598 (2021).
- Jahnke, K., Huth, V., Mersdorf, U., Liu, N., Göpfrich, K. Bottom-up assembly of synthetic cells with a DNA cytoskeleton. ACS Nano. 16 (5), 7233-7241 (2022).
- Tran, M. P., et al. A DNA segregation module for synthetic cells. Small. 19 (13), 2202711 (2023).
- Mason, A. F., Buddingh', B. C., Williams, D. S., Hest, J. C. M. v. Hierarchical self-assembly of a copolymer-stabilized coacervate protocell. J Am Chem Soc. 139 (48), 17309-17312 (2017).
- Gumz, H., et al. Toward functional synthetic cells: In-depth study of nanoparticle and enzyme diffusion through a cross-linked polymersome membrane. Adv Sci. 6 (7), 1801299 (2019).
- Huang, X., Patil, A. J., Li, M., Mann, S. Design and construction of higher-order structure and function in proteinosome-based protocells. J Am Chem Soc. 136 (25), 9225-9234 (2014).
- Booth, R., Qiao, Y., Li, M., Mann, S. Spatial positioning and chemical coupling in coacervate-in-proteinosome protocells. Angew Chem Int Ed Engl. 58 (27), 9120-9124 (2019).
- Hindley, J. W., et al. Light-triggered enzymatic reactions in nested vesicle reactors. Nat Commun. 9 (1), 1093 (2018).
- Zubaite, G., Hindley, J. W., Ces, O., Elani, Y. Dynamic reconfiguration of subcompartment architectures in artificial cells. ACS Nano. 16 (6), 9389-9400 (2022).
- Hirose, T., et al. A guide to membraneless organelles and their various roles in gene regulation. Nat Rev Mol Cell Biol. 24 (4), 288-304 (2023).
- Guindani, C., Silva, L. C. d., Cao, S., Ivanov, T., Landfester, K. Synthetic cells: From Simple Bio-Inspired Modules to Sophisticated Integrated Systems. Angew Chem Int Ed Engl. 61 (16), e202110855 (2022).
- Adamala, K. P., et al. Present and future of synthetic cell development. Nat Rev Mol Cell Biol. 25 (3), 162-167 (2023).
- Allen, M. E., et al. Biomimetic behaviors in hydrogel artificial cells through embedded organelles. Proc Natl Acad Sci U S A. 120 (35), e2307772120 (2023).
- Cook, A. B., Novosedlik, S., Hest, J. C. M. v. Complex coacervate materials as artificial cells. Acc Mater Res. 4 (3), 287-298 (2023).
- Seeman, N. C., Sleiman, H. F. DNA nanotechnology. Nat Rev Mater. 3, 17068 (2018).
- Takinoue, M. DNA droplets for intelligent and dynamical artificial cells: from the viewpoint of computation and non-equilibrium systems. Interface Focus. 13 (5), 20230021 (2023).
- Liu, W., Lupfer, C., Samanta, A., Sarkar, A., Walther, A. Switchable hydrophobic pockets in DNA protocells enhance chemical conversion. J Am Chem Soc. 145 (13), 7090-7094 (2023).
- Wilner, O. I., Willner, I. Functionalized DNA nanostructures. Chem Rev. 112 (4), 2528-2556 (2012).
- Gong, J., Tsumura, N., Sato, Y., Takinoue, M. Computational DNA droplets recognizing miRNA sequence inputs based on liquid-liquid phase separation. Adv Funct Mater. 32, 2202322 (2022).
- Jeon, B. -. J., Nguyen, D. T., Saleh, O. A. Sequence-controlled adhesion and microemulsification in a two-phase system of DNA liquid droplets. J Phys Chem. 124 (40), 8888-8895 (2020).
- Sato, Y., Sakamoto, T., Takinoue, M. Sequence-based engineering of dynamic functions of micrometer-sized DNA droplets. Sci Adv. 6 (23), 3471 (2020).
- Saleh, O. A., et al. Vacuole dynamics and popping-based motility in liquid droplets of DNA. Nat Commun. 14 (1), 3574 (2023).
- Rubio-Sánchez, R., Fabrini, G., Cicuta, P., Michele, L. D. Amphiphilic DNA nanostructures for bottom-up synthetic biology. Chem Commun. 57 (95), 12725-12740 (2021).
- Brady, R. A., Brooks, N. J., Cicuta, P., Di Michele, L. Crystallization of amphiphilic DNA C-Stars. Nano Lett. 17 (5), 3276-3281 (2017).
- Leathers, A., et al. Reaction-diffusion patterning of DNA-based artificial cells. J Am Chem Soc. 144 (38), 17468-17476 (2022).
- Malouf, L., et al. Sculpting DNA-based synthetic cells through phase separation and phase-targeted activity. Chem. 9 (11), 3347-3364 (2023).
- Fabrini, G., Minard, A., Brady, R. A., Antonio, M. D., Michele, L. D. Cation-responsive and photocleavable hydrogels from noncanonical amphiphilic DNA nanostructures. Nano Lett. 22 (2), 602-611 (2022).
- Brady, R. A., Brooks, N. J., Foderà, V., Cicuta, P., Di Michele, L. Amphiphilic-DNA platform for the design of crystalline frameworks with programmable structure and functionality. J Am Chem Soc. 140 (45), 15384-15392 (2018).
- Walczak, M., et al. Responsive core-shell DNA particles trigger lipid-membrane disruption and bacteria entrapment. Nat Commun. 12 (1), 4743 (2021).
- Zhang, D. Y., Winfree, E. Control of DNA strand displacement kinetics using Toehold exchange. J Am Chem Soc. 131 (47), 17303-17314 (2009).
- . Integrated DNA Technologies OligoAnalyzer Tool Available from: https://www.idtdna.com/pages/products/custom-dna-rna/dna-oligos/custom-dna-oligos (2024)
- Cavaluzzi, M. J., Borer, P. N. Revised UV extinction coefficients for nucleoside-5'-monophosphates and unpaired DNA and RNA. Nucleic Acids Res. 32 (1), e13 (2004).
- Lattuada, E., Caprara, D., Piazza, R., Sciortino, F. Spatially uniform dynamics in equilibrium colloidal gels. Sci Adv. 7 (49), (2021).
- . T7-FlashScribeTM Transcription Kit Available from: https://www.cellscript.com/products/018pl0617CS.pdf (2017)
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. J Comput Chem. 32 (1), 170-173 (2011).
- Walczak, M., Brady, R. A., Leathers, A., Kotar, J., Di Michele, L. Influence of hydrophobic moieties on the crystallization of amphiphilic DNA nanostructures. The Journal of Chemical Physics. 158 (8), 084501 (2023).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados