É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Extração de RNA de animal único e tubo único para comparação dos níveis relativos de transcrição via qRT-PCR no Tardígrado Hypsibius exemplaris
Neste Artigo
Resumo
Este trabalho apresenta um método de extração rápida de RNA e comparação de nível de transcrição para análise da expressão gênica no tardígrado Hypsibius exemplaris. Usando lise física, este método de alto rendimento requer um único tardígrado como material de partida e resulta em produção robusta de cDNA para reação em cadeia da polimerase de transcrição reversa quantitativa (qRT-PCR).
Resumo
O tardígrado Hypsibius exemplaris é um organismo modelo emergente conhecido por sua capacidade de sobreviver a extremos ambientais. Para explorar os mecanismos moleculares e a base genética de tal extremotolerância, muitos estudos contam com o sequenciamento de RNA (RNA-seq), que pode ser realizado em populações que variam de grandes coortes a animais individuais. A reação em cadeia da polimerase com transcrição reversa (RT-PCR) e a interferência de RNA (RNAi) são posteriormente usadas para confirmar os achados de RNA-seq e avaliar os requisitos genéticos para genes candidatos, respectivamente. Tais estudos requerem um método eficiente, preciso e acessível para extração de RNA e medição dos níveis relativos de transcrição por RT-PCR quantitativo (qRT-PCR). Este trabalho apresenta um método eficiente de extração de RNA de tubo único e tardígrado único (STST) que não apenas isola de forma confiável o RNA de tardígrados individuais, mas também reduz o tempo e o custo necessários para cada extração. Este método de extração de RNA produz quantidades de cDNA que podem ser usadas para amplificar e detectar vários transcritos por PCR quantitativo (qRT-PCR). O método é validado pela análise de mudanças dinâmicas na expressão de genes que codificam duas proteínas reguladas por choque térmico, Heat-Shock Protein 70 β2 (HSP70 β2) e Heat-Shock Protein 90α (HSP90α), possibilitando avaliar seus níveis relativos de expressão em indivíduos expostos ao calor usando qRT-PCR. O STST complementa efetivamente os métodos existentes de extração de RNA tardígrado único e em massa, permitindo o exame rápido e acessível dos níveis transcricionais de tardígrados individuais por qRT-PCR.
Introdução
Os tardígrados são pequenos animais multicelulares conhecidos por sua capacidade de sobreviver a condições extremas que são letais para a maioria das outras formas de vida1. Por exemplo, esses animais podem sobreviver quase 1000 vezes a dose de radiação ionizante que é letal para os humanos 2,3,4,5,6,7,8,9,10, dessecação quase completa 11,12,13,14,15, congelamento na ausência de adição de crioprotetores 16,17,18 e, em seu estado dessecado, até mesmo o vácuo do espaço 19,20. Devido à sua capacidade única de sobrevivência em ambientes extremos, esses animais tornaram-se modelos fundamentais para a compreensão da extrema tolerância em organismos multicelulares complexos 1,21,22,23.
A manipulação genética estável desses animais notáveis, incluindo transgênese e modificação genética da linha germinativa, permaneceu indescritível até recentemente 24,25. Como tal, a maioria dos experimentos para revelar mecanismos moleculares de extremotolerância são realizados por meio de perfis transcricionais via sequenciamento de RNA. Existem muitos conjuntos de dados de sequenciamento de RNA valiosos e informativos para tardígrados sob várias condições extremas, variando de radiação 8,9,26,27,28, estresse térmico29, estresse de congelamento12 e dessecação 27,30,31,32,33. Alguns desses estudos utilizaram métodos de extração e purificação de RNA em massa para iluminar nossa compreensão molecular da extremotolerância. No entanto, a extração em massa de transcritos de RNA de muitos animais impede a análise da variação na expressão gênica entre indivíduos, perdendo assim a riqueza potencial de conjuntos de dados mais refinados. É importante ressaltar que esses estudos geralmente analisam populações heterogêneas de animais que incluem animais que sobrevivem a estressores ambientais e aqueles que não sobrevivem. Como tal, esses estudos são confundidos pela média de dados de expressão de estados de resposta múltiplos e potencialmente dramaticamente diferentes. Para resolver esse problema, Arakawa et al., 201634 desenvolveram um elegante pipeline de RNA-seq de baixa entrada que aplica um kit de extração de RNA seguido por uma etapa de amplificação de PCR linear usando 34,35,36 ou vários 30,37,38 animais como entrada. Esses estudos foram fundamentais para nossa compreensão da extremotolerância ao tardígrado22. Curiosamente, este protocolo também foi aplicado ao qRT-PCR usando sete animais como material de partida24.
Na maioria dos organismos modelo, tendo identificado alvos potenciais via RNA-seq, o qRT-PCR é então realizado para confirmar as alterações transcricionais identificadas pelo RNA-seq e avaliar o curso do tempo de expressão dos genes candidatos de maneira de alta resolução. Para testar a função dos genes identificados, esses estudos são frequentemente seguidos por knockdown mediado por RNAi de alvos moleculares39,40 e análise da capacidade extremotolerante 12,41. A eficácia de cada knockdown de RNAi é normalmente confirmada por qRT-PCR, monitorando diretamente a diminuição na abundância de transcritos. No entanto, o RNAi é um processo trabalhoso em tardígrados, pois cada dsRNA deve ser administrado por meio de microinjeção manual de indivíduos39,40. Devido à natureza de baixo rendimento dessa estratégia, um método de extração de RNA rápido e de baixo custo adaptado para qRT-PCR de animais únicos seria altamente valioso para a pesquisa de tardígrados. Embora métodos anteriores tenham sido desenvolvidos para extrair RNA de tardígrados únicos, esses protocolos não combinaram sua extração com qRT-PCR, em vez disso, confiaram em métodos baseados em densidade óptica 12,40,41. Motivados por esses desafios, procuramos desenvolver um protocolo que produza RNA de forma confiável em quantidade e qualidade que possa ser usado para qRT-PCR a partir de um único H. exemplaris.
Adaptado de um protocolo de extração de RNA de um único animal desenvolvido para Caenorhabditis elegans42, o STST é otimizado para H. exemplaris. O método de extração consiste em seis etapas rápidas de congelamento e descongelamento, interrompendo fisicamente a cutícula, permitindo a extração de RNA e subsequente síntese de cDNA. O método STST diminui o tempo de extração em mais de 24 vezes em comparação com os métodos de extração de RNA em massa, conforme descrito por Boothby, 201843, e em 30% em comparação com os kits de extração de RNA tardígrado único, conforme descrito por Arakawa et al., 201634. Além disso, o número de interações amostra-experimentador é reduzido de 5 para apenas 1 em comparação com as preparações do kit de extração de RNA, reduzindo assim o risco de contaminação por ribonucleases exógenas. Ao consultar genes altamente expressos, o método STST produz cDNA suficiente para 25 reações quantitativas de RT-PCR por tardígrado único, exigindo apenas 1 μL do volume total de 25 μL de cDNA por reação. No entanto, as concentrações do modelo precisam ser determinadas empiricamente para transcritos de menor abundância.
A eficácia do método STST para analisar mudanças dinâmicas na expressão gênica foi avaliada investigando a expressão diferencial dos genes que codificam a proteína de choque térmico-90α (HSP90α) e a proteína de choque térmico 70β2 (HSP70β2) em resposta ao choque térmico de curto prazo a 35°C por 20 minutos. Tanto o HSP70β2 quanto o HSP90α na maioria dos organismos eucarióticos são rapidamente regulados positivamente após a exposição a choques térmicos de curto prazo (20 min) 42 . A análise em H. exemplaris revelou que os RNAs codificadores de HSP70β2 e HSP90α extraídos de tardígrados tratados termicamente mostraram aumentos estatisticamente significativos na expressão após exposição ao calor de curto prazo. Esses achados demonstram que o protocolo STST pode ser usado para analisar mudanças dinâmicas na expressão gênica em animais individuais ao longo do tempo.
O método de extração STST deve complementar os métodos experimentais existentes, como RNA-seq, facilitando a extração rápida e barata de RNA e a subsequente comparação dos níveis de transcrição por qRT-PCR. Este método também será valioso para avaliar a eficiência e penetrância do RNAi em indivíduos injetados manualmente de forma mais quantitativa do que a densidade óptica sozinha. Finalmente, devido às suas estruturas cuticulares e características físicas semelhantes, é provável que esse método também seja eficaz para analisar a expressão gênica em outras espécies de tardígrados44.
Protocolo
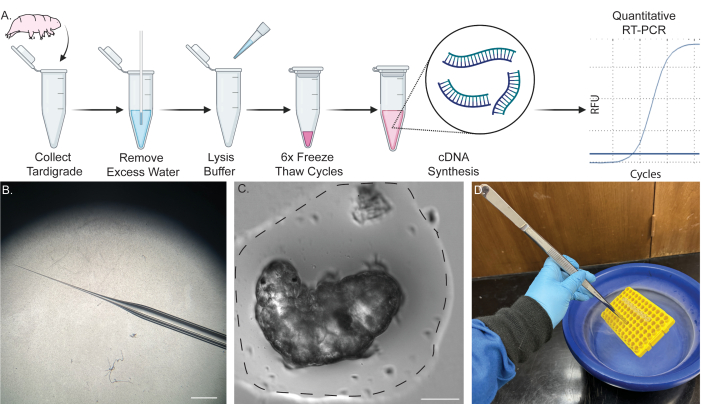
Figura 1: Tubulação de tubo único para extração de RNA de um único tardígrado. (A) Esquema mostrando o protocolo para extração de RNA de um único tardígrado, incluindo seis ciclos de congelamento e descongelamento e subsequente síntese de cDNA. As amostras podem ser posteriormente utilizadas para RT-PCR e qRT-PCR. (B) Imagem do cone da micropipeta usado para remoção de água. Barra de escala: 2 mm. (C) Imagem de campo claro de um tardígrado em um pequeno volume de água (linha pontilhada). A remoção da maior parte da água na medida mostrada é necessária para uma extração bem-sucedida e evita a diluição do tampão de lise. Barra de escala: 50 μm. (D) Imagem mostrando a imersão de amostras em nitrogênio líquido usando pinças longas para congelar e descongelar rapidamente as amostras com segurança. Parte do conteúdo foi criado em BioRender. Kirk, M. (2022) BioRender.com/d93s511 Clique aqui para ver uma versão maior desta figura.
NOTA: A Figura 1A mostra um esquema do procedimento. Para procedimentos detalhados de cultura de tardígrados e algas, consulte os relatórios publicados anteriormente 45,46,47.
1. Esterilização da água de nascente
- Despeje 2 L de água mineral de uma jarra de água de 5 galões (consulte a Tabela de Materiais para Detalhes) em uma garrafa de vidro própria para autoclave de 2 L.
- Coloque a tampa no frasco seguro para autoclave e feche com uma pequena quantidade de fita adesiva. Não aperte a garrafa; Coloque a tampa por cima.
- Autoclave a água da nascente por 50 min em um ciclo úmido sem etapa de secagem.
- Deixe a água atingir a temperatura ambiente (RT) e feche a tampa firmemente antes de armazená-la em RT.
2. Tração de micropipeta de vidro (com extrator de pipeta)
- Prenda uma micropipeta de vidro (D.E.: 1 mm, I.D.: 0.58 mm, Comprimento: 10 cm) em um extrator de micropipeta. Evite o contato com o filamento de aquecimento, pois isso alterará o formato da pipeta e danificará o filamento.
- Determinar empiricamente a tracção da pipeta para cada filamento e extractor de pipetas. No entanto, para servir como ponto de partida para a otimização, use 78 °C e uma única etapa de tração de peso de tração de 182,2 g.
- Deixe o filamento aquecer e a gravidade separar a micropipeta de vidro em duas micropipetas de vidro com pontas afiadas (Figura 1B).
- Armazene essas micropipetas de vidro puxado em uma placa de Petri fechada de 100 mm com cera ou argila para mantê-las no lugar e evitar que as pontas afiadas se quebrem.
3. Tração de micropipeta de vidro (sem extrator de pipeta)
- Acenda um bico de Bunsen ou outra fonte de chama controlada em uma configuração baixa.
- Pegue uma micropipeta de vidro com uma extremidade em cada mão.
- Segure o centro da micropipeta de vidro sobre a chama até que o vidro comece a derreter. Em seguida, separe rapidamente as duas extremidades. Isso criará duas pontas afiadas muito delicadas.
- Quebre levemente a ponta com uma pinça fina estéril.
- Armazene essas micropipetas de vidro puxado em uma placa de Petri fechada de 100 mm com cera ou argila para mantê-las no lugar e evitar que as pontas afiadas se quebrem.
4. Extração de RNA
- Obtenha 0,5 L de nitrogênio líquido em um recipiente crio-seguro.
CUIDADO: O nitrogênio líquido é criogênico e pode causar queimaduras se exposto à pele ou aos olhos. Ao manusear, use roupas de proteção, óculos de proteção, luvas de nitrilo, luvas criogênicas, jaleco e sapatos fechados. Verifique se o recipiente é seguro para nitrogênio líquido antes de transportar o líquido. O uso de um banho de gelo seco com etanol para esta etapa também pode ser possível. - Faça a mistura mestre de síntese de cDNA: uma solução de 10 μL contendo 1 μL de primer hexâmero aleatório, 2 μL de DNase, 4 μL de 5x RT Buffer, 1 μL de Enzyme Mix, 1 μL de H2O e 1 μL de 10 mM dNTPs. Armazene esta solução no gelo.
- Prepare o tampão de lise de tardígrados (5 mM Tris (pH = 8), 0,5% (v/v) Detergente 1, 0,5% (v/v) Detergente 2, 0,25 mM EDTA em água estéril sem nuclease).
NOTA: Esta solução pode ser armazenada na bancada por 6 meses. No entanto, mantenha a esterilidade e evite possíveis fontes de contaminação por RNAse. - Alíquota tampão de lise suficiente para extracções (2 μL/tardígrado).
- Adicionar inibidor de RNAse à solução tampão de lise de tardígrados até uma concentração final de 4 U/μL.
- Vortex e centrifugar a solução em RT em uma centrífuga de bancada a uma velocidade de 2000 x g por 5 s antes de armazenar a solução no gelo.
- Remova quantos tardígrados forem necessários para o experimento da cultura usando uma pipeta P1000 estéril com ponta de filtro e coloque-os em uma placa de Petri estéril de 35 mm.
NOTA: Qualquer número de tardígrados pode ser processado desta forma. Normalmente, três tardígrados por condição são processados para extração. - Lave os tardígrados três vezes, usando 1 mL de água de nascente estéril autoclavada e uma pipeta P1000 com ponta de filtro estéril. Pipetá-los lentamente para cima e para baixo ajuda a remover contaminantes de algas.
- Usando um microscópio de dissecação com aumento de 25x a 50x, transfira um único tardígrado desta cultura lavada para uma nova placa de Petri estéril de 35 mm usando uma pipeta P10 estéril com ponta de filtro.
- Use uma pipeta P200 com ponta de filtro estéril para lavar o tardígrado único em 100 μL de água estéril sem nuclease.
NOTA: Esta etapa de lavagem é usada para remover ainda mais contaminantes, incluindo ribonucleases. - Transfira o tardígrado lavado para o fundo de um tubo de PCR limpo e estéril em 1-2 μL de água estéril sem nuclease usando uma pipeta P10 com ponta de filtro estéril, garantindo cuidadosamente que o tardígrado não grude na lateral da ponta.
- Visualize o tardígrado sob um microscópio de dissecação com ampliação de 25x.
- Para facilitar a remoção de água, quebre levemente a ponta da micropipeta de vidro puxado fora do tubo. Certifique-se de que o furo seja grande o suficiente para puxar a água, mas não o tardígrado.
- Usando a ação capilar de uma micropipeta de vidro puxado, remova a água até que o animal esteja cercado por uma pequena bolha de água com aproximadamente dois comprimentos de tardígrados de diâmetro.
- Monitore o processo de remoção de água por meio do escopo de dissecação para garantir que o nível da água seja adequado e o tardígrado permaneça hidratado.
NOTA: A Figura 1C oferece um exemplo de quanta água remover. Este é um passo crítico. Uma pequena bolha de água envolverá o tardígrado para evitar que seque, mas o máximo de água em excesso possível deve ser removido para evitar a diluição do tampão de lise. Para obter um example dos níveis de água restantes, consulte a Figura 1C. - Imediatamente após a remoção da água, adicione 2 μL de tampão de lise de tardígrados ao fundo do tubo, faça um vórtice breve e centrifugue o tubo em RT por 5 s a 2000 x g em uma centrífuga de mesa.
- Coloque imediatamente as amostras contendo os tardígrados em um suporte de tubo de PCR e certifique-se de que estejam firmes pelo rack.
- Segure o rack usando um par de pinças longas e grossas e mergulhe suavemente o rack contendo as amostras no nitrogênio líquido até congelar completamente (Figura 1D).
- Remova o rack do nitrogênio líquido e coloque-o imediatamente no gelo. Deixe a amostra descongelar (leva ~ 45 s a 1 min). Monitore a amostra a cada 15 s, removendo-a do gelo e inspecionando-a visivelmente. Quando a amostra estiver visivelmente transparente, passe para a próxima etapa.
- Repita as etapas 4.18-4.19 mais cinco vezes. Um total de seis ciclos de congelamento e descongelamento são necessários para lise e extração máximas (Figura 2A, B).
- Quando o congelamento e descongelamento estiver concluído, coloque as amostras no gelo e avance imediatamente para a próxima etapa. Não congele as amostras neste ponto para armazenamento, pois isso diminuirá o RNA disponível para a preparação do cDNA.
5. Síntese de cDNA
- Adicionar 2 μL de mistura principal de síntese de cDNA ao tubo de PCR contendo lisado de tardígrado. Agite brevemente o tubo e gire-o em RT a 2000 x g por 5 s com uma centrífuga de mesa antes de recolocar as amostras no gelo.
- Coloque as amostras em um termociclador e incube a 25 ° C por 10 min para recozer primers, a 55 ° C por 30 min para realizar a transcrição reversa e, finalmente, enzimas inativadas pelo calor a 85 ° C por 5 min.
- Após a incubação, coloque imediatamente o tubo no gelo e dilua a amostra até um volume total de 25 μL adicionando 21 μL de água estéril sem nuclease. Para transcrições com baixo número de cópias, altere esta etapa de diluição conforme determinado empiricamente.
6. qPCR
- Determine a temperatura de recozimento do conjunto de primers usando RNA total preparado a partir de grandes quantidades de tardígrados, por exemplo, o método de extração em massa apresentado em Boothby, 201843.
- Execute um gradiente de temperatura de PCR para determinar a temperatura de recozimento ideal antes de executar qRT-PCR (para todas as configurações de PCR usadas neste protocolo, consulte a Tabela 1 e a Tabela 2).
- Descongele um tubo de corante indicador, super misture no gelo e isole da luz. Coloque uma placa de qPCR de 96 poços no gelo e coloque 5 μL de super mistura, 2 μL de água, 1 μL de cada primer (10 μM) e 1 μL de produto de cDNA no número de poços desejados.
- Sele a placa de PCR com a vedação da placa e execute o qRT-PCR usando uma temperatura de recozimento apropriada para o conjunto de primers (para todas as configurações de qRT-PCR usadas neste artigo, consulte a Tabela 3).
7. Quantificação e interpretação dos resultados
- Compare os resultados quantitativamente com um ou mais genes de manutenção de controle, cuja expressão deve ser constante nas condições impostas. Para este estudo, foi utilizado o gene da actina.
- Obter e comparar os valores Ct ou o limiar de ciclo de cada alvéolo com os valores Ct das reações genéticas de manutenção de controlo. Calcule a mudança de dobra na expressão gênica usando a seguinte equação:
NOTA: A expressão gênica da dobra é plotada para cada transcrito e tardígrado como um 2- (ΔΔCt) 48. - Para obter uma estimativa aproximada do número de transcritos a partir do valorC t, use a seguinte equação:
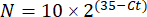
Onde N é o número de transcritos e 2 é a eficiência de PCR assumida ou o aumento de dobras na fluorescência por ciclo de PCR48.
Resultados
Desenvolvimento e otimização da extração de RNA de tardígrado único
Adaptando o protocolo de Ly et al., 201542 para extração de RNA em tardígrados, o sistema STST é otimizado para maximizar a quantidade e a qualidade da preparação (Figura 1A). RT-PCR foi realizado para transcritos de actina, quantificando o rendimento do transcrito amplificando uma região de 527 pb abrangendo os éxons 1 e 2 (as sequê...
Discussão
Este estudo apresenta um método eficiente para a extração de RNA para qRT-PCR de tardígrado único. A comparação direta da metodologia STST com um kit de extração de RNA tardígrado único existente revelou que a extração de RNA STST produz quantidades >200 vezes maiores de transcritos de RNA de actina, reduz o custo para menos de um dólar por amostra e reduz o tempo necessário para extração em 30%. Para aplicar o STST a uma questão biológica relevante, avaliamos o perfil...
Divulgações
Os autores declaram não haver conflitos de interesse a divulgar.
Agradecimentos
Queremos agradecer a bolsa NIH Ruth Kirschstein # 5F32AG081056-02 e a bolsa de pós-doutorado Errett Fisher, que apoiou a Dra. Molly J. Kirk, a Crowe Family Fellowship, que apoiou Chaoming Xu, e uma bolsa do Senado Acadêmico da Universidade da Califórnia, Santa Bárbara, e bolsas do NIH #R01GM143771 e #2R01HD081266, que apoiaram esses esforços de pesquisa. Os autores também reconhecem o uso do Laboratório de Nanoestruturas Biológicas dentro do Instituto de NanoSistemas da Califórnia, apoiado pela Universidade da Califórnia, Santa Bárbara, e pela Universidade da Califórnia, Gabinete do Presidente.
Materiais
| Name | Company | Catalog Number | Comments |
| 10 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-401 | Refered to as Sterile Filter-Tipped P 10 Pipette Tips |
| 1000 µL Premium Pipet Tips, Low Binding, Racked, Sterile | Genesee Scientific | 23-165RS | Refered to as Sterile Filter-Tipped P 1000 Pipette Tips |
| 200 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-412 | Refered to as Sterile Filter-Tipped P 200 Pipette Tips |
| 4 Star Straight Strong Medium Point Tweezer | Excelta | 00-SA-DC | Refered to as Long forceps |
| 96-Well PCR Rack with Lid Assorted, 5 Racks/Unit | Genesee Scientific | 27-202A | Refered to as PCR Rack |
| Andwin Scientific 3M LEAD FREE AUTOCLAVE TAPE 1" | Thermo Fisher Scientific | NC0802040 | Refered to as Autoclave Tape |
| Autoclave Tape | Thermo Fisher Scientific | AB1170 | Refered to as PCR Plate Seals |
| Benchling v8 | Benchling | N/A | Refered to as Benchling |
| BioRadHard-Shell 96-Well PCR Plate | BioRad | HSS9641 | Refered to as PCR Plate |
| BULWARK FR Lab Coat: | Grainger | 26CF64 | Refered to as Lab Coat |
| C1000 Touch Bio-rad Thermocycler | BioRad | 1851148 | Refered to as Thermocycler |
| C1000 Touch Bio-rad Thermocycler with CFX Optics Module | BioRad | 1845097 | Refered to as qPCR thermocycler |
| Chloroccoccum hypnosporum. | Carolina | 152091 | Refered to as Algae |
| Corning PYREX Reusable Media Storage Bottles | Thermo Fisher Scientific | 06-414-1E | Refered to as 2 L Autoclave-safe Glass Bottle |
| Daigger & Company Vortex-Genie 2 Laboratory Mixer | Thermo Fisher Scientific | 3030A | Refered to as Vortexer |
| Direct-zol Micro Prep | Zymo Research | R2060 | Refered to as RNA extraction kit |
| Dumont 5 Biology Tweezers | Fine Science Tools | 11254-20 | Refered to as Fine Forceps |
| EDTA | Fisher Scientific | S311-500 | Refered to as EDTA |
| FIJI v 2.14.0/1.54f | ImageJ, | N/A | Refered to as FIJI/ImageJ |
| Filament for pippette Puller | Tritech Research | PC-10H | Refered to as Filament |
| Fisherbrand Economy Impact Goggles | Fisher Scientific | 19-181-501 | Refered to as Splash Goggles |
| Glass Micropipette O.D. 1mm ID 0.58, Length 10 cm | TriTech Research | GD-1 | Reffered to as glass micropipette |
| Hypsibius exemplaris Z151 Strain | Carolina | 133960 | Refered to as Tardigrades or H. exemplaris |
| Liquid Nitrogen Dewar 1 L | Agar Scientific | AGB7475 | Refered to as Cryo-safe container |
| Maxima H Minus First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1651 | Refered to as cDNA Synthesis Master Mix |
| Narishige Dual-Stage Glass Micropipette Puller | Tritech Research | PC-10 | Refered to as micropipette puller |
| Nitrile Gloves | Fisher Scientific | 17-000-314 | Refered to as Nitrile Gloves |
| PETRI DISH, PS, 35/10 mm, WITH VENTS | Grenier | 627102 | Refered to as 35 mm Petri dish |
| PIPETMAN P10, 1–10 µL, Metal Ejector | Gilson | F144055M | Refered to as P 10 Pipette |
| PIPETMAN P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | Refered to as P 1000 Pipette |
| PIPETMAN P200, 20–200 µL, Metal Ejector | Gilson | F144058M | Refered to as P 200 Pipette |
| Pound This 4-Color Modeling Clay | American Science Surplus | 96517P001 | Refered to as Clay |
| Prism v10.0 | GraphPad | N/A | Refered to a Prism |
| RNAse-Free, 8 Strip 0.2 mL PCR Tubes with caps | Invitrogen | AM12230 | Refered to as Sterile PCR Tube |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Refered to as RNAse inhibitor |
| Spring water | Nestle Pure Life | 44221229 | Refered to as Spring Water |
| SsoAdvanced Universal SYBR Green Supermix | BIO RAD | 1725271 | Refered to as Indicator Dye Super mix |
| Stereo-Microscope System w/optics and illumination | TriTech Research | SMT1 | Refered to as Dissecting Microscope |
| Supertek Scientific Tirrill Burners | Thermo Fisher Scientific | S09572B | Refered to as Bunsen Burner |
| Table Top Centrifuge | Qualitron | DW-41-115-NEW | Refered to as Table Top Centrifuge |
| Tempshield Cryo-Gloves | Fisher Scientific | 11-394-305 | Refered to as Cryo Gloves |
| Thermo Scientific Nunc Petri Dishes | Thermo Fisher Scientific | 08-757-099 | Refered to as 100 mm Petri dish |
| Tris base | Fisher Scientific | T395-500 | Refered to as Tris or Tris Base |
| Triton X-100 | Fluka | 93443 | Refered to as Detergent 1 |
| TWEEN 20 | Sigma aldrich | P1379-500 | Refered to as Detergent 2 |
| Water - PCR/RT-PCR certified, nuclease-free | Growcells | PCPW-0500 | Refered to as Sterile Nuclease Free Water |
Referências
- Møbjerg, N., Neves, R. C. New insights into survival strategies of tardigrades. Comp Biochem Physiol Part A Mol Integr Physiol. 254, 110890 (2021).
- Jönsson, K. I., Harms-Ringdahl, M., Torudd, J. Radiation tolerance in the eutardigrade Richtersius coronifer. Int J Radiat Biol. 81 (9), 649-656 (2005).
- Horikawa, D. D., et al. Radiation tolerance in the tardigrade Milnesium tardigradum. Int J Radiat Biol. 82 (12), 843-848 (2006).
- Bruckbauer, S. T., Cox, M. M. Experimental evolution of extremophile resistance to ionizing radiation. Trends Genet. 37 (9), 830-845 (2021).
- Jönsson, K. I., Hygum, T. L., Andersen, K. N., Clausen, L. K. B., Møbjerg, N. Tolerance to gamma radiation in the marine heterotardigrade, Echiniscoides sigismundi. PLoS One. 11 (12), e0168884 (2016).
- Jönsson, K. I. Radiation tolerance in tardigrades: Current knowledge and potential applications in medicine. Cancers (Basel). 11 (9), 1333 (2019).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Clark-Hachtel, C. M., et al. The tardigrade Hypsibius exemplaris dramatically upregulates DNA repair pathway genes in response to ionizing radiation. Curr Biol. 34 (9), 1819-1830.e6 (2024).
- Anoud, M., et al. Comparative transcriptomics reveal a novel tardigrade specific DNA binding protein induced in response to ionizing radiation. Elife. 13, RP92621 (2024).
- Jönsson, K. I., Schill, R. O. Induction of Hsp70 by desiccation, ionising radiation and heat-shock in the eutardigrade Richtersius coronifer. Comp Biochem Physiol B Biochem Mol Biol. 146 (4), 456-460 (2007).
- Boothby, T. C. Desiccation of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 871-873 (2018).
- Boothby, T. C., et al. Tardigrades use intrinsically disordered proteins to survive desiccation. Mol Cell. 65 (6), 975-984.e5 (2017).
- Horikawa, D. D., Higashi, S. Desiccation tolerance of the tardigrade Milnesium tardigradum collected in Sapporo, Japan, and Bogor, Indonesia. Zoolog Sci. 21 (8), 813-816 (2004).
- Halberg, K. A., Jørgensen, A., Møbjerg, N. Desiccation tolerance in the tardigrade Richtersius coronifer relies on muscle mediated structural reorganization. PLoS One. 8 (12), e3330 (2013).
- Sørensen-Hygum, T. L., Stuart, R. M., Jørgensen, A., Møbjerg, N. Modelling extreme desiccation tolerance in a marine tardigrade. Sci Rep. 8 (1), 11495 (2018).
- Lyons, A. M., Roberts, K. T., Williams, C. M. Survival of tardigrades (Hypsibius exemplaris) to subzero temperatures depends on exposure intensity, duration, and ice-nucleation - as shown by large-scale mortality dye-based assays. bioRxiv. , (2024).
- Møbjerg, A., et al. Extreme freeze-tolerance in cryophilic tardigrades relies on controlled ice formation but does not involve significant change in transcription. Comp Biochem Physiol Part A Mol Integr Physiol. 271, 111245 (2022).
- Tsujimoto, M., Imura, S., Kanda, H. Recovery and reproduction of an Antarctic tardigrade retrieved from a moss sample frozen for over 30 years. Cryobiology. 72 (1), 78-81 (2016).
- Jönsson, K. I. Tardigrades as a potential model organism in space research. Astrobiology. 7 (5), 757-766 (2007).
- Jönsson, K. I., Rabbow, E., Schill, R. O., Harms-Ringdahl, M., Rettberg, P. Tardigrades survive exposure to space in low Earth orbit. Curr Biol. 18 (17), R729-R731 (2008).
- Kasianchuk, N., Rzymski, P., Kaczmarek, &. #. 3. 2. 1. ;. The biomedical potential of tardigrade proteins: A review. Biomed Pharmacother. 158, 113983 (2023).
- Arakawa, K. Examples of extreme survival: Tardigrade genomics and molecular anhydrobiology. Annu Rev Anim Biosci. 10 (1), 519-542 (2022).
- Hvidepil, L. K. B., Møbjerg, N. New insights into osmobiosis and chemobiosis in tardigrades. Front Physiol. 14, 1274522 (2023).
- Tanaka, S., Aoki, K., Arakawa, K. In vivo expression vector derived from anhydrobiotic tardigrade genome enables live imaging in Eutardigrada. Proc Natl Acad Sci U S A. 120 (5), e2216739120 (2023).
- Kondo, K., Tanaka, A., Kunieda, T. Single-step generation of homozygous knockout/knock-in individuals in an extremotolerant parthenogenetic tardigrade using DIPA-CRISPR. PloS Genet. 20 (6), e1011298 (2024).
- Yoshida, Y., Hirayama, A., Arakawa, K. Transcriptome analysis of the tardigrade Hypsibius exemplaris exposed to the DNA-damaging agent bleomycin. bioRxiv. , (2024).
- Yoshida, Y., et al. Time-series transcriptomic screening of factors contributing to the cross-tolerance to UV radiation and anhydrobiosis in tardigrades. BMC Genomics. 23 (1), 405 (2022).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Neves, R. C., et al. Differential expression profiling of heat stressed tardigrades reveals major shift in the transcriptome. Comp Biochem Physiol Part A Mol Integr Physiol. 267, 111143 (2022).
- Yoshida, Y., et al. Comparative genomics of the tardigrades Hypsibius dujardini and Ramazzottius varieornatus. PLoS Biol. 15 (7), e2002266 (2017).
- Wang, C., Grohme, M. A., Mali, B., Schill, R. O., Frohme, M. Towards decrypting cryptobiosis - analyzing anhydrobiosis in the tardigrade Milnesium tardigradum using transcriptome sequencing. PLoS One. 9 (3), e92663 (2014).
- Mali, B., et al. Transcriptome survey of the anhydrobiotic tardigrade Milnesium tardigradum in comparison with Hypsibius dujardini and Richtersius coronifer. BMC Genomics. 11 (1), 168 (2010).
- Förster, F., et al. Transcriptome analysis in tardigrade species reveals specific molecular pathways for stress adaptations. Bioinform Biol Insights. 6, 69-96 (2012).
- Arakawa, K., Yoshida, Y., Tomita, M. Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 3 (1), 160063 (2016).
- Arakawa, K. Transcriptome assembly of Richtersius coronifer with annotated BLAST result against Ramazzottius varieornatus. Figshare. Dataset. , (2019).
- Yoshida, Y., Konno, S., Nishino, R., Murai, Y., Tomita, M., Arakawa, K. Ultralow input genome sequencing library preparation from a single tardigrade specimen. J Vis Exp. (137), (2018).
- Murai, Y., et al. Multiomics study of a heterotardigrade, Echiniscus testudo, suggests convergent evolution of anhydrobiosis-related proteins in Tardigrada. bioRxiv. , (2020).
- Yoshida, Y., Sugiura, K., Tomita, M., Matsumoto, M., Arakawa, K. Comparison of the transcriptomes of two tardigrades with different hatching coordination. BMC Dev Biol. 19 (1), 24 (2019).
- Tenlen, J. R. Microinjection of dsRNA in tardigrades. Cold Spring Harb Protoc. 2018 (11), (2018).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223 (3), 171-181 (2013).
- Giovannini, I., et al. Production of reactive oxygen species and involvement of bioprotectants during anhydrobiosis in the tardigrade Paramacrobiotus spatialis. Sci Rep. 12 (1), 15888 (2022).
- Ly, K., Reid, S. J., Snell, R. G. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 2, 59-63 (2015).
- Boothby, T. C. Total RNA extraction from tardigrades. Cold Spring Harb Protoc. 2018 (11), 905-907 (2018).
- Czerneková, M., Vinopal, S. The tardigrade cuticle. Limnol Rev. 21 (3), 127-146 (2021).
- McNuff, R. Laboratory culture of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 867-870 (2018).
- Gabriel, W. N., et al. The tardigrade Hypsibius dujardini, a new model for studying the evolution of development. Dev Biol. 312 (2), 545-559 (2007).
- Ruiz-Villalba, A., Ruijter, J. M., van den Hoff, M. J. B. Use and misuse of cq in qPCR data analysis and reporting. Life (Basel). 11 (6), 508 (2021).
- Antonov, J., et al. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization. Lab Invest. 85 (8), 1040-1050 (2005).
- Toussaint, J., et al. Improvement of the clinical applicability of the genomic grade index through a qRT-PCR test performed on frozen and formalin-fixed paraffin-embedded tissues. BMC Genomics. 10, 424 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados