Dissecção de ovários, intestino médio e glândulas salivares de mosquitos para análises de microbioma no nível do órgão
Neste Artigo
Resumo
As comunidades microbianas em mosquitos são uma grande promessa para as estratégias de biocontrole de vetores. A maioria dos simbiontes não é cultivável, exigindo análises metagenômicas. Descrevemos um método para dissecar mosquitos fêmeas e separar ovários, intestino médio e glândulas salivares, evitando a contaminação cruzada, facilitando estudos de microbioma no nível do órgão e melhorando a compreensão dos papéis dos microrganismos na biologia do mosquito.
Resumo
A carga global de doenças transmitidas por mosquitos, incluindo malária, dengue, Nilo Ocidental, Zika, Usutu e febre amarela, continua a aumentar, representando uma ameaça significativa à saúde pública. Com o aumento da resistência a inseticidas e a ausência de vacinas eficazes, novas estratégias estão surgindo com foco na microbiota do mosquito. No entanto, a maioria dos simbiontes permanece resistente ao cultivo. Caracterizar a diversidade e a função dos genomas bacterianos em espécimes de mosquitos, portanto, depende da metagenômica e das estratégias subsequentes de montagem e binning. A obtenção e análise de genomas montados no metagenoma (MAGs) de órgãos separados podem fornecer informações importantes sobre o papel específico dos micróbios associados ao mosquito nos ovários (órgãos reprodutivos), no intestino médio (chave para a digestão e imunidade dos alimentos) ou nas glândulas salivares (essenciais para a transmissão de doenças transmitidas por vetores, pois os patógenos devem colonizá-los para entrar na saliva e atingir a corrente sanguínea durante uma refeição de sangue). Esses genomas recém-reconstruídos podem abrir caminho para o desenvolvimento de novas estratégias de biocontrole de vetores. Para isso, é necessário isolar os órgãos do mosquito, evitando a contaminação cruzada entre eles ou com microrganismos presentes em outros órgãos do mosquito. Aqui, descrevemos um protocolo de dissecação otimizado e livre de contaminação para estudar o microbioma do mosquito no nível do órgão.
Introdução
Os mosquitos espalham uma ampla gama de patógenos que causam doenças e são uma séria ameaça à saúde pública. Devido ao aumento da prevalência de resistência a inseticidas entre as populações de mosquitos e na ausência de vacinas eficazes contra esses patógenos, novos métodos de biocontrole que se concentram no microbioma do mosquito estão surgindo. Em particular, destaca-se a bactéria intracelular Wolbachia, que pode interferir na transmissão do patógeno e manipular a reprodução do hospedeiro 1,2,3. Além disso, outros simbiontes de mosquitos são centrais para a sobrevivência, desenvolvimento ou sistema imunológico de seu hospedeiro, bem como na infecção e transmissão de patógenos, e mostram grande promessa para sua exploração no combate a doenças transmitidas por vetores 4,5,6,7,8.
Os microrganismos associados aos mosquitos abrangem todos os domínios da vida microbiana (incluindo bactérias, eucariotos e fungos) que interagem intimamente com seu hospedeiro, mas também entre si nos diferentes compartimentos do corpo 9,10. Portanto, uma melhor compreensão da microbiota, sua potencial variação sazonal11 e os mecanismos pelos quais seus membros interagem diretamente em tecidos distintos de mosquitos podem ajudar a desenvolver novos métodos de biocontrole direcionados ou melhorar os existentes. No entanto, a maioria dos simbiontes permanece resistente ao cultivo, impossibilitando sua caracterização.
O advento dos métodos de sequenciamento de segunda e terceira geração, juntamente com abordagens de montagem e binning de última geração, permitiram a reconstrução de genomas microbianos e o acesso à diversidade e potencial funcional de micróbios não cultiváveis. Aqui, apresentamos um método para a dissecção dos ovários, intestino médio e glândulas salivares de mosquitos fêmeas, evitando a contaminação cruzada. Este protocolo pode ser seguido por extração de DNA genômico e subsequente metabarcoding ou sequenciamento metagenômico shotgun para explorar a diversidade e função da microbiota do mosquito no nível do órgão. Fornecemos um exemplo de dados de dissecação de mosquitos e microbioma para espécimes de Culex spp., embora este protocolo possa ser estendido a vetores de outros gêneros, como Anopheles ou Aedes.
Protocolo
NOTA: A Figura 1 mostra um esquema do método resumindo as diferentes etapas do protocolo.
1. Preparação do material
- Antes de iniciar o experimento, prepare uma solução de alvejante com 0,5% de cloro ativo (recomendado pela Organização Mundial da Saúde) para desinfetar o equipamento e a bancada.
- Em 900 mL de água, dissolva três comprimidos solúveis, cada um contendo 1,5 g de cloro ativo. Aguarde 15 minutos para que os comprimidos se dissolvam completamente, resultando em uma solução de alvejante a 0,5%.
- Coloque a solução de alvejante a 0,5% em um borrifador. Em seguida, pulverize a solução em todos os equipamentos de dissecação, incluindo lâminas microscópicas e bancada de trabalho.
NOTA: O equipamento e a bancada devem ser deixados secar por ~10 min antes do uso.
- Para enxaguar os mosquitos antes de cada dissecação para evitar a contaminação das amostras com bactérias externas, prepare quatro tubos recém-esterilizados de 1,5 mL: dois contendo etanol a 96% e outros dois contendo 1x tampão PBS (solução salina tamponada com fosfato).
- Preparar os tubos para armazenar as amostras após a dissecação de cada mosquito: quatro tubos estéreis de 1,5 mL, cada um contendo 100 μL de tampão de preservação.
NOTA: Prepare um tubo para as glândulas salivares, um para os ovários, um para o intestino médio e um para a carcaça do mosquito.
2. Coleta de mosquitos
- Colete e mantenha indivíduos adultos do sexo feminino vivos (de campo ou laboratório, 4-7 dias) até a esterilização da superfície.
3. Esterilização superficial de mosquitos
NOTA: Limpe as pinças e agulhas e use tubos novos e esterilizados entre cada órgão e indivíduo. Se o alvejante não estiver disponível, certifique-se de esterilizar completamente as pinças e agulhas de dissecação entre a dissecção de cada indivíduo e órgão para evitar a contaminação cruzada do material biológico. Os instrumentos de dissecção devem estar secos, sem resíduos de alvejante/etanol, antes do próximo uso.
- Para evitar que as fêmeas esvaziem o conteúdo intestinal quando expostas ao álcool, anestesieste-as com gelo ou em um freezer a -20 °C (por 4 min para que os tecidos e órgãos não congelem).
- Transfira os mosquitos individuais para um novo tubo esterilizado de 1,5 mL com 1 mL de etanol a 96%. Vortex suavemente (manualmente) por 30 s (agite bem o mosquito no tubo para destacar as escamas e potenciais contaminantes bacterianos na superfície dos insetos). Enxágüe duas vezes com tubos contendo etanol.
- Transfira o mosquito para um novo tubo esterilizado de 1,5 mL com 1 mL de solução estéril 1x PBS e vortex suavemente (manualmente) por 30 s (para evitar precipitação de DNA com etanol). Enxágue duas vezes usando tubos contendo PBS 1x estéril.
4. Dissecção de mosquitos
- Coloque várias pequenas gotas de PBS 1x estéril na lâmina para facilitar a colocação do mosquito e o enxágue dos órgãos dissecados.
NOTA: As gotas de PBS estéril devem ser grandes o suficiente para acomodar os órgãos do mosquito. - Pegue o mosquito limpo e coloque-o em uma gota de PBS 1x estéril na lâmina do microscópio.
- Disseque as glândulas salivares com pinça e agulha esterilizadas, conforme descrito abaixo.
- Segure o tórax do mosquito com uma pinça para evitar que ele se mova. Em seguida, coloque uma agulha no pescoço, abaixo da cabeça, e puxe suavemente a cabeça do mosquito para separá-lo do tórax.
NOTA: As glândulas salivares estão localizadas na parte superior do tórax e estão conectadas à cabeça pelo ducto salivar. Durante a dissecção, é crucial não romper esse ducto, pois as glândulas salivares não virão com a cabeça quando você a puxar do tórax. É por isso que é importante ter um mosquito fresco, não congelado. - Quando a cabeça se separa do tórax, observe as duas glândulas salivares emergirem, cada uma composta por três lobos. Corte as glândulas salivares ao nível do ducto salivar, recupere-as com uma agulha e coloque-as em uma nova gota de PBS 1x estéril para enxaguá-la e evitar contaminações entre órgãos.
- Recupere as glândulas salivares usando uma agulha e coloque-as em um novo tubo esterilizado contendo 100 μL de tampão de preservação.
- Segure o tórax do mosquito com uma pinça para evitar que ele se mova. Em seguida, coloque uma agulha no pescoço, abaixo da cabeça, e puxe suavemente a cabeça do mosquito para separá-lo do tórax.
- Disseque os ovários com pinça esterilizada e agulha da seguinte maneira.
- Coloque o mosquito de costas. Usando uma pinça para segurar o mosquito pelo tórax para evitar o movimento durante a dissecção, perfure o terceiro segmento abdominal (começando de baixo) com uma agulha.
NOTA: Opcionalmente, remova as pernas e asas do mosquito para facilitar a dissecação. - Ao perfurar o terceiro segmento abdominal, puxe o abdômen para baixo para abri-lo e revelar os órgãos internos. Remova todos os pedaços restantes de exoesqueleto de mosquito para facilitar a liberação dos órgãos.
NOTA: Órgãos no abdômen, como intestino, túbulos de Malpighi e ovários, são visíveis. - Recupere ambos os ovários com pinças esterilizadas e coloque-os em uma nova gota de PBS 1x estéril na lâmina do microscópio para enxaguá-los. Pegue os ovários e coloque-os no tubo de 1,5 mL com os 100 μL de tampão de preservação.
- Coloque o mosquito de costas. Usando uma pinça para segurar o mosquito pelo tórax para evitar o movimento durante a dissecção, perfure o terceiro segmento abdominal (começando de baixo) com uma agulha.
- Agora que o abdômen está aberto e os órgãos expostos, recupere o intestino médio com uma nova pinça esterilizada e/ou agulha.
- Separe o intestino médio do intestino anterior e posterior; em seguida, remova os túbulos de Malpighi do intestino médio. Pegue o intestino médio usando uma agulha estéril e coloque-o em uma nova gota estéril de 1x PBS para enxaguá-lo e evitar contaminações entre órgãos.
- Transfira para uma nova gota estéril de 1x PBS. Abra o intestino médio com uma pinça para liberar e enriquecer as bactérias, conforme descrito no Arquivo Suplementar 1. Pegue o intestino médio com uma pinça e pipete todas as bactérias contendo PBS para armazenar todo o material (líquido e tecido) em um novo tubo esterilizado de 1,5 mL com 100 μL de tampão de preservação.
- Adicione novamente 5 μL de 1x PBS no local usado anteriormente na lâmina do microscópio e pipete diretamente de volta o PBS para o mesmo tubo de 1,5 mL para recuperar bactérias que possam ter grudado na lâmina do microscópio.
- Use uma pinça estéril para agarrar a carcaça (corpo restante) e armazene-a em um novo tubo esterilizado de 1,5 mL com 100 μL de tampão de preservação.
- Armazenamento de material até processamento posterior
- Rotule os tubos com 'Espécie, Órgão, Data, Nome do Pesquisador'.
- Armazenar ovários, intestino médio, glândulas salivares e carcaça dissecados a -20 °C até ao processamento.
- Acompanhe em uma planilha todos os metadados associados às amostras, incluindo espécie (ou apenas nível de gênero), data de coleta, país, província, latitude, longitude, nome da pessoa que fez a dissecação, fêmea grávida ou não, refeição de sangue ou não.
Resultados Representativos
x'Dissecações de mosquito
Seguindo o protocolo, coletamos e isolamos os ovários, intestino médio, glândulas salivares e a carcaça de dois espécimes de Culex pipiens molestus (incluindo uma fêmea grávida) de uma colônia de laboratório. Confirmamos dissecções limpas após a observação de órgãos inteiros (ou seja, intactos) e bem isolados, sem detritos remanescentes sob o binóculo. Todo o corpo, cabeça e tórax, glândulas salivares dissecadas, ovários e intestino médio de um espécime de Culex pipiens molestus são mostrados na Figura 2. Como esperado, o intestino médio e as glândulas salivares eram uma ordem de magnitude menor do que os ovários do mosquito. Os ovos de um espécime de Culex grávida, juntamente com seus túbulos do intestino médio e de Malpighi, são apresentados na Figura 3. É importante notar que as dissecções são mais propensas a falhar quando o material não é fresco e o tecido provavelmente se romperá (Figura 4B, C). Portanto, sugerimos dissecar o material logo após a coleta (em menos de 12 h), quando possível, pois os tecidos ainda são elásticos (Figura 4A). Da mesma forma, a dissecção de material congelado pode ser realizada, mas há um risco muito maior de falha e contaminação cruzada entre órgãos devido a tecidos frágeis (Figura 4).
Dados do microbioma
Além disso, coletamos e separamos o intestino médio e os ovários de três espécimes individuais de Culex quinquefasciatus de Noumea, Nova Caledônia, seguindo o mesmo procedimento. Extraímos DNA de cada órgão, preparamos amostras para sequenciamento do genoma completo e realizamos análises de dados de microbioma conforme detalhado no Arquivo Suplementar 1. Uma análise de diversidade taxonômica bacteriana em leituras curtas filtradas por qualidade não montadas usando PhyloFlash12 mostrou táxons dominantes distintos no intestino médio em comparação com os ovários (Figura 5A). Notavelmente, as comunidades bacterianas ovarianas foram dominadas por Wolbachia, com a presença adicional de um filotipo de Spirochaetaceae nos ovários do indivíduo 3, enquanto as comunidades do intestino médio exibiram uma diversidade mais ampla, incluindo Gammaproteobacteria, Spirochaetaceae e Firmicutes. A partir dos mesmos dados de sequenciamento, reconstruímos quatro genomas montados no metagenoma (MAGs) com > de completação de 80% e redundância < 5%, pertencentes às classes Spirochaetia, Alpha- e Gammaproteobacteria (Tabela 1).
Como esperado, os MAGs reconstruídos aqui não cobriram toda a diversidade taxonômica prevista pelos resultados do PhyloFlash devido às deficiências específicas da reconstrução do genoma usando leituras curtas metagenômicas. O MAG atribuído a Wolbachia (Ref. MAG 3 na Tabela 1) foi detectado em todos os ovários e dois dos intestinos médios e teve maior cobertura nos ovários (Figura 5B). Também reconstruímos dois MAGs pertencentes à família Enterobacteriaceae , incluindo o gênero Pantoea (Ref. MAG 1 e 2 na Tabela 1), no intestino médio obtido dos indivíduos 2 e 3 de Culex , que não foram detectados nos ovários correspondentes (Figura 5B). Finalmente, também reconstruímos um genoma bacteriano de Spirochaetaceae , Ref. MAG 4 (Tabela 1), atribuído ao gênero BR149 que foi isolado com sucesso do intestino médio de Culex pipiens por Graña-Miraglia e colegas13. Curiosamente, esse MAG foi detectado no intestino médio dos indivíduos 1 e 3, bem como nos ovários do indivíduo 3 (Figura 5B).
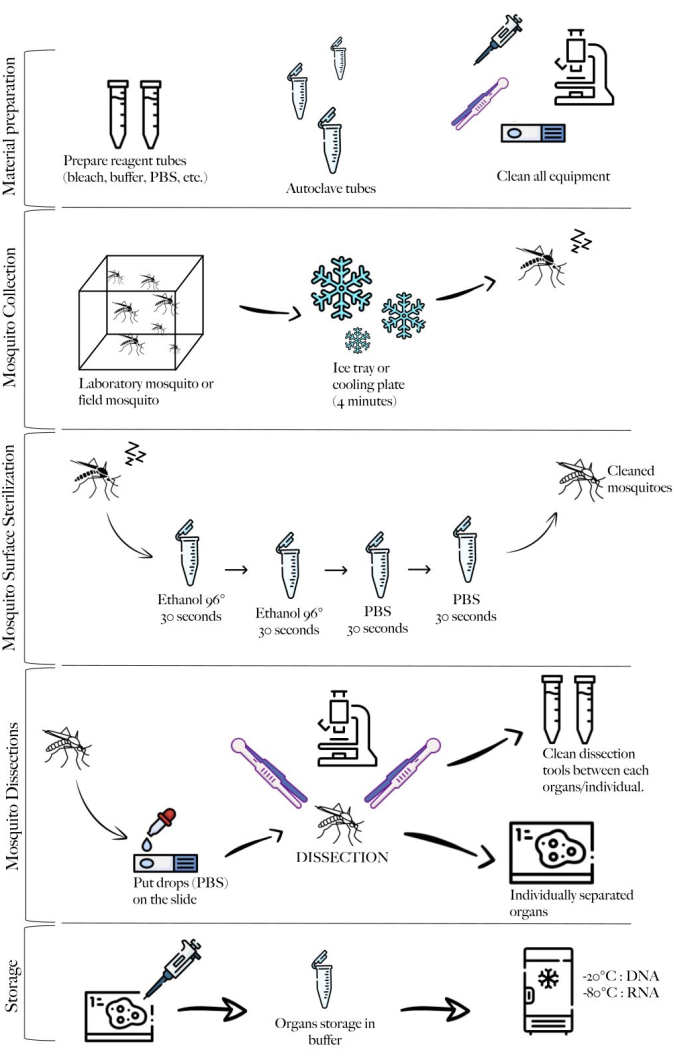
Figura 1: Esquema do método resumindo as diferentes etapas. Preparação de material, coleta de mosquitos, limpeza de mosquitos, dissecações de mosquitos e armazenamento. Clique aqui para ver uma versão maior desta figura.
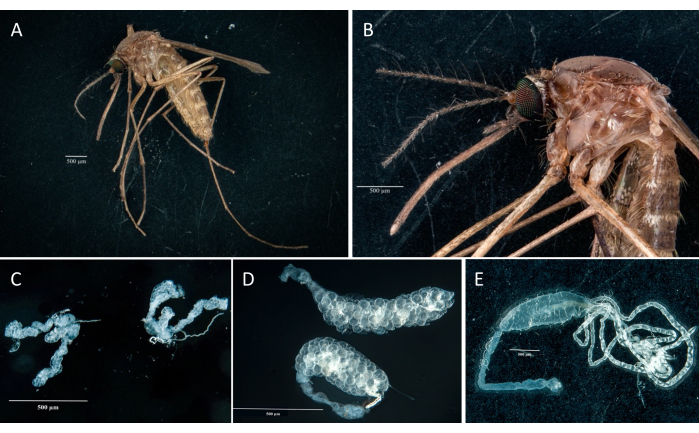
Figura 2: Culex pipiens molestus fêmea. (A) Corpo inteiro. (B) Cabeça e tórax. (C) Glândulas salivares dissecadas. (D) Ovários dissecados. (E) Intestino dissecado com túbulos de Malpighi. Barras de escala = 500 μm. Clique aqui para ver uma versão maior desta figura.
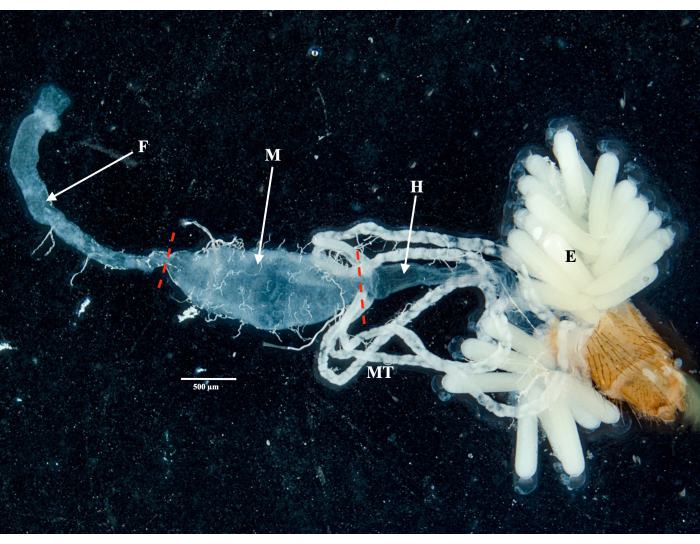
Figura 3: Abdômen dissecado de uma fêmea grávida de Culex pipiens molestus . O intestino anterior, o intestino médio, o intestino posterior, os túbulos de Malpighi e os ovos da amostra são mostrados. Traços vermelhos indicam onde cortar para separar o intestino médio do intestino anterior e do intestino posterior. Barra de escala = 500 μm. Abreviaturas: F = intestino anterior; M = intestino médio; H = intestino grosso; E = ovos; MT = túbulos de Malpighi. Clique aqui para ver uma versão maior desta figura.
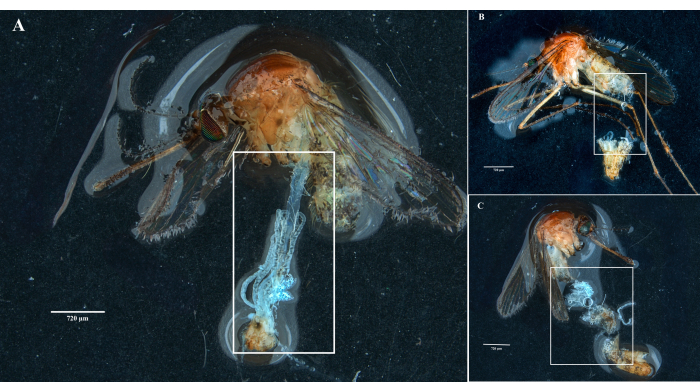
Figura 4: Dissecção de espécimes de mosquitos Culex frescos e congelados. Retângulos brancos ilustram (A) tecidos de mosquitos com órgãos intactos de uma amostra recém-dissecada e (B, C) material biológico quebrado de amostras que foram congeladas antes da dissecação. Barras de escala = 720 μm. Clique aqui para ver uma versão maior desta figura.
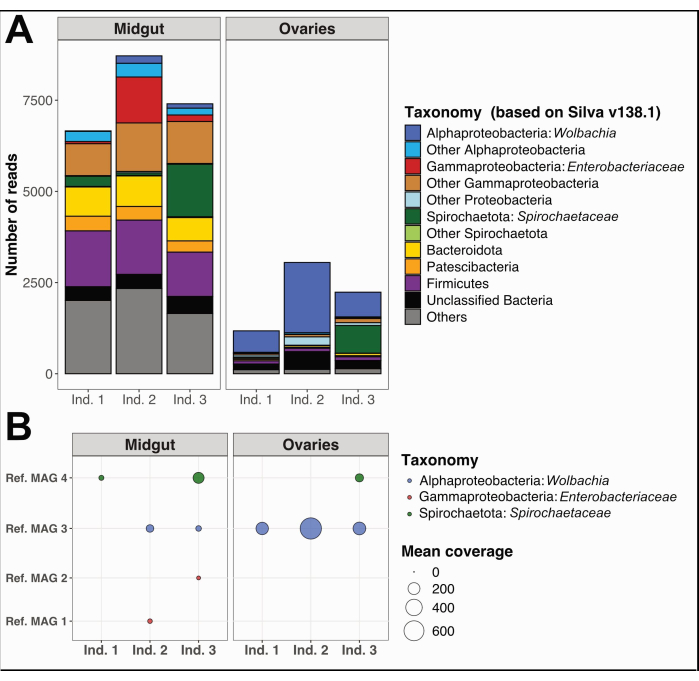
Figura 5: Exemplo de análise de microbioma em três indivíduos de Culex quinquefasciatus . (A) Visualização da diversidade bacteriana estimada através da extração de SSU rRNA lê com PhyloFlash12 no intestino médio e ovários dos três espécimes. (B) Cobertura média dos MAGs reconstruídos sobre as amostras. Abreviaturas: MAG = Genoma Montado no Metagenoma; Ind = Individual. Clique aqui para ver uma versão maior desta figura.
| MAG | Ref. MAG 4 | Ref. MAG 2 | Ref. MAG 1 | Ref. MAG 3 |
| Comprimento (bp) | 1,287,790 | 4,910,866 | 4,751,276 | 1,298,266 |
| Número de contigs | 9 | 162 | 149 | 123 |
| GC % | 34.05 | 55.45 | 54.05 | 34.15 |
| Conclusão (%) | 84.5 | 97.18 | 98.59 | 91.55 |
| Redundância (%) | 0 | 2.82 | 4.22 | 0 |
| Domínio | Bactéria | Bactéria | Bactéria | Bactéria |
| Filo | Spirochaetota | Proteobactérias | Proteobactérias | Proteobactérias |
| Classe | Spirochaetia | Gamaproteobactérias | Gamaproteobactérias | Alfaproteobactérias |
| Ordem | WRBN01 | Enterobacterales | Enterobacterales | Rickettsiales |
| Família | WRBN01 | Enterobacteriaceae | Enterobacteriaceae | Anaplasmataceae |
| Gênero | BR149 | Pantoea | - | Wolbachia |
Tabela 1: MAGs reconstruídos a partir dos três indivíduos de Culex quinquefasciatus . Tamanho do genoma, número de contigs, proporção de GC, estimativas de conclusão e redundância com base na coleção de genes de núcleo de cópia única disponível em Anvi'o17 e taxonomia obtida usando GTDB18.
Arquivo Suplementar 1: Exemplo detalhado de procedimento para análise de dados de microbioma a partir da coleta de amostras, extração de DNA e sequenciamento do genoma completo, seguido pelo fluxo de trabalho de bioinformática para reconstrução do genoma e estimativa da proporção de leitura procariótica e eucariótica Clique aqui para baixar este arquivo.
Discussão
Recomendamos prestar atenção especial à sequência de dissecção de órgãos, começando pelas glândulas salivares. De fato, observamos que eles eram mais facilmente extraídos do tórax de espécimes de Culex se a integridade do mosquito fosse preservada. Danos ao abdômen ou tórax podem reduzir a pressão no corpo do mosquito, impedindo o procedimento. No entanto, também é possível cortar entre o tórax e o abdômen e, em seguida, retirar as glândulas salivares da cabeça e do tórax (A. B. Failloux, comunicação pessoal). Além disso, adquirir habilidades de dissecação proficientes pode ser um desafio, por isso sugerimos praticar em um número adequado de espécimes antes do experimento.
Isolar os órgãos do mosquito e evitar a contaminação cruzada de maneira sistemática é crucial para uma ampla gama de análises da microbiota do mosquito a jusante. Um estudo metagenômico resolvido pelo genoma após a dissecção de ovários únicos de espécimes de Culex pipiens no sul da França permitiu a descoberta do primeiro plasmídeo de Wolbachia em Culex pipiens (pWCP14). Usando uma abordagem semelhante, investigamos a distribuição e a frequência de pWCP em espécimes de Culex pipiens e Culex quinquefasciatus de áreas continentais e insulares em todo o mundo, em várias condições ambientais e laboratoriais. No geral, os dados revelaram um elemento plasmídeo Wolbachia notavelmente conservado em mosquitos Culex , sugerindo um papel crucial para este elemento móvel na biologia endossimbionte15, justificando uma análise mais aprofundada.
Aqui, fornecemos exemplos adicionais de análises do microbioma do mosquito em amostras do intestino médio e do ovário obtidas usando este procedimento sistemático. Observamos uma clara diferença na microbiota entre os tecidos (Figura 5), com táxons bacterianos compartilhados e órgãos-específicos. Como esperado, a presença de Wolbachia foi detectada em ambos os órgãos, com maior abundância relativa (com base em leituras curtas não montadas) e cobertura média de MAGs nos ovários do mosquito em comparação com o intestino médio, consistente com a observação de que esse endossimbionte é transmitido através dos ovários e subsequentemente se espalha para os tecidos somáticos. Embora este estudo tenha sido restrito a amostras da Nova Caledônia, este protocolo pode facilitar a investigação da variabilidade genômica da Wolbachia em escala global, bem como seu papel em vários fenótipos, incluindo a regulação da densidade de si mesma e a proteção viral. Além disso, este trabalho exemplifica como o procedimento de dissecação apresentado aqui permite o exame da diversidade taxonômica e das potenciais capacidades funcionais dos simbiontes do mosquito em amostras do intestino médio.
Obtivemos dois genomas de rascunho de enterobactérias que estavam presentes apenas em duas amostras de intestino médio, confirmando a falta de contaminação entre os órgãos para esses dois espécimes. Em relação às espiroquetas, detectadas tanto no intestino médio quanto nos ovários do indivíduo 3, Juma e colegas, 2020 observaram a presença dessas bactérias na superfície das jangadas de ovos. Os autores sugeriram que as comunidades bacterianas encontradas nas jangadas de ovos podem ser herdadas principalmente maternalmente dos ovários, uma vez que as jangadas de ovos foram mantidas em água deionizada e livre de bactérias. No entanto, eles não puderam descartar a possibilidade de colonização bacteriana ocorrer logo após a oviposição e recomendaram mais estudos sobre o microbioma ovário16.
Embora inicialmente projetado para espécimes do complexo de espécies Culex pipiens , prevemos a aplicabilidade deste protocolo a uma comunidade mais ampla de entomologistas médicos que estudam outros vetores, como Anopheles ou Aedes. Ao operar no nível de órgãos individuais, esse método pode permitir comparações genômicas intra e interindividuais, oferecendo insights sobre a variabilidade genômica do simbionte em uma escala fina, com o potencial de avançar estratégias de controle de vetores. Este método de dissecção e isolamento das glândulas salivares, ovários e intestino médio, evitando a contaminação cruzada microbiana, também pode ser um protocolo útil para estudos de dinâmica de infecção viral nesses três órgãos.
Divulgações
Os autores declaram não ter conflitos de interesse.
Agradecimentos
Agradecemos a Gilbert Legoff por ensinar Jordan Tutagata a dissecar glândulas salivares de mosquitos e Giuliano Mucci por ajudar com fotos dos órgãos do mosquito. Agradecemos a Anna-Bella Failloux e Nonito Pages pela discussão útil sobre o protocolo. Este trabalho foi apoiado pelo ERC RosaLind Starting Grant "948135" para JR. Agradecemos à plataforma Vectopole (IRD, Montpellier) por fornecer suporte técnico e pela criação e manutenção das populações de mosquitos.
Materiais
| Name | Company | Catalog Number | Comments |
| Alcohol 96° | Fisher scientific | 10332562 | |
| Binocular magnifier | |||
| Bleach | RAJA | 145517 | 150 tablets of 1.5 g |
| DNA prep kit | Illumina | Provided by MGX sequencing platform | Previously known as Nextera DNA Flex |
| DNA-RNA Shield (50 mL) | Zymo research | ZR1100-50 | Preservation buffer |
| DNeasy Blood and Tissue Kit | Qiagen | 69504 | DNA extraction kit |
| Filter tips 20 µL | Starlab | S1120-3810 | |
| Filter tips 200 µL | Starlab | S1120-8710 | |
| Filter tips 1000 µL | Starlab | S1122-1730-C | |
| Forceps | FST (Fine Science Tools) | 11252-20 | Dumont Forceps #5 |
| Library quantification kit | Roche | Provided by MGX sequencing platform | KAPA Library Quanitification Kits |
| Micropipettes 2-20 µL | Eppendorf | 6.291704 | |
| Micropipettes 20-200 µL | Eppendorf | 6.291703 | |
| Micropipettes 100-1000 µL | Eppendorf | 7.648488 | |
| Microscope slides | Epredia | J1800BMNZ | dimension : 75 mm x 50 mm |
| Needles | Terumo | AN*2719R1 | |
| NGS kit | Agilent | Provided by MGX sequencing platform | Fragment Analyzer Systems HS Genomic DNA 50kb Kit |
| NovaSeq 6000 | Illumina | Provided by MGX sequencing platform | Sequencer |
| PBS Phosphate Buffered Saline (Sterile) | Fisher scientific | 10212990 | |
| Permanent black marker | |||
| Sterile Eppendorf | Dutscher | 33871 | 1.5 mL |
| Support for needles | FST (Fine Science Tools) | 26016-12 | Moria MC1 Pin Holder 12 cm |
Referências
- Achee, N. L., et al. Alternative strategies for mosquito-borne arbovirus control. PLOS Negl Trop Dis. 13 (1), e0006822 (2019).
- Hoffmann, A. A., et al. Successful establishment of Wolbachia in Aedes populations to suppress dengue transmission. Nature. 476 (7361), 454-457 (2011).
- Porter, J., Sullivan, W. The cellular lives of Wolbachia. Nat Rev Microbiol. 21 (11), 750-766 (2023).
- Coon, K. L., Brown, M. R., Strand, M. R. Mosquitoes host communities of bacteria that are essential for development but vary greatly between local habitats. Mol Ecol. 25 (22), 5806-5826 (2016).
- Didion, E. M., et al. Microbiome reduction prevents lipid accumulation during early diapause in the northern house mosquito, Culex pipiens pipiens. J Insect Physiol. 134, 104295 (2021).
- Gabrieli, P., et al. Mosquito trilogy: Microbiota, immunity and pathogens, and their implications for the control of disease transmission. Front Microbiol. 12, 630438 (2021).
- Garrigós, M., Garrido, M., Panisse, G., Veiga, J., Martínez-de La Puente, J. Interactions between West Nile Virus and the microbiota of Culex pipiens vectors: A literature review. Pathogens. 12 (11), 1287 (2023).
- Zheng, R., et al. Holobiont perspectives on tripartite interactions among microbiota, mosquitoes, and pathogens. ISME J. 17 (8), 1143-1152 (2023).
- Flores, G. A. M., et al. Wolbachia dominance influences the Culex quinquefasciatus microbiota. Sci Rep. 13 (1), 18980 (2023).
- Hegde, S., et al. Interkingdom interactions shape the fungal microbiome of mosquitoes. Anim Microbiome. 6 (1), 11 (2024).
- Suo, P., et al. Seasonal variation of midgut bacterial diversity in Culex quinquefasciatus populations in Haikou city, Hainan province, China. Biology. 11 (8), 1166 (2022).
- Gruber-Vodicka, H. R., Seah, B. K. B., Pruesse, E. phyloFlash: Rapid small-subunit rRNA profiling and targeted assembly from metagenomes. mSystems. 5 (5), e00920-e00920 (2020).
- Graña-Miraglia, L., et al. Spirochetes isolated from arthropods constitute a novel genus Entomospira genus novum within the order Spirochaetales. Sci Rep. 10 (1), 17053 (2020).
- Reveillaud, J., et al. The Wolbachia mobilome in Culex pipiens includes a putative plasmid. Nat Commun. 10 (1), 1051 (2019).
- Ghousein, A., et al. pWCP is a widely distributed and highly conserved Wolbachia plasmid in Culex pipiens and Culex quinquefasciatus mosquitoes worldwide. ISME Commun. 3 (1), 40 (2023).
- Juma, E. O., Kim, C. -. H., Dunlap, C., Allan, B. F., Stone, C. M. Culex pipiens and Culex restuans egg rafts harbor diverse bacterial communities compared to their midgut tissues. Parasit Vectors. 13 (1), 532 (2020).
- Lee, M. D. GToTree: a user-friendly workflow for phylogenomics. Bioinformatics. 35 (20), 4162-4164 (2019).
- Parks, D. H., et al. A complete domain-to-species taxonomy for bacteria and Archaea. Nat Biotechnology. 38 (9), 1079-1086 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
SOBRE A JoVE
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados